選択的スプライシング
選択的スプライシング(せんたくてき-、Alternative Splicing)とは、DNAからの転写過程において特定のエクソンをとばしてスプライシングを行うことである。択一的スプライシングとも呼ばれる。

遺伝子にはアミノ酸配列に関する情報を含む核酸塩基配列(エクソン)が遺伝情報を含まない配列(イントロン)によっていくつかに分断されている。通常、DNAからmRNAへの転写が行われる際にはこれらのすべてが順に転写されていく。その後、転写生成物(mRNA前駆体)からイントロン部分の切り捨てが行われてエクソン部分が連結し成熟mRNAが出来上がるが、この不要な部分の切り捨ての過程をスプライシングと呼んでいる。
しかし、時にスプライシングを行う部位・組み合わせが変化し、複数種の成熟mRNAが生成することがある。これを選択的スプライシングと呼び、ひとつの遺伝子から多数の生成物が生じてくることになる。選択的スプライシングによってスプライスバリアントまたはスプライシングバリアント(splice/splicing variant)と呼ばれる変異タンパク質が生成される。
選択的スプライシングは真核生物における正常な現象であり、ゲノムにコードされるタンパク質の多様性を大きく増大させる[1]。ヒトでは、複数のエクソンからなる遺伝子のうち約95%が選択的スプライシングを受ける[2]。選択的スプライシングには多数の形式が観察されているが、最も一般的な形式はエクソンスキッピングである。この形式では、特定のエクソンが特定の条件下や組織ではmRNAに組み込まれ、他の場合にはmRNAから省かれることとなる[1]。
選択的スプライシングを受けたmRNAの産生は、一次転写産物自身に存在するシスエレメント(シスに作用する)、そしてそれらに結合するタンパク質(トランスに作用する)のシステムによって調節される。関与するタンパク質には、特定のスプライス部位の利用を促進するスプライシング活性化因子や、特定の部位の利用を低下させるスプライシング抑制因子が含まれる。選択的スプライシングの機構はきわめて多様であり、特にハイスループットな技術の利用によって新たな例が発見され続けている。研究者らは、スプライシングに関与する調節システムを完全に解明し、ある遺伝子から特定の状況下で産生されるスプライシングバリアントが「スプライシング・コード」によって予測できるようになることを望んでいる[3][4]。
異常なスプライシングバリアントは疾患にも関与しており、ヒトの遺伝子疾患のかなりの部分がスプライシングバリアントによるものである[3]。異常なスプライシングバリアントはがんの発生にも寄与していると考えられており[5][6][7][8]、スプライシング因子の遺伝子はさまざまなタイプのがんで頻繁に変異が生じている[8]。
発見[編集]
選択的スプライシング最初に観察されたのは1977年である[9][10]。アデノウイルスはその感染サイクルの初期に、ウイルスDNAの複製に先立って5種類の一次転写産物を産生し、DNA複製の開始後に新たに1種類の産生を行う。感染の終盤に産生される新たな一次転写産物は巨大で、32kbのアデノウイルスゲノムの5/6に由来する。これは感染細胞に存在するどのアデノウイルスmRNAよりもかなり大きいものである。研究者らは、2型アデノウイルスが終盤に産生する一次転写産物は多くの異なる形へとスプライシングされ、異なるウイルスタンパク質をコードするmRNAが形成されていることを発見した。さらに、一次転写産物は複数のポリアデニル化部位を含んでおり、プロセシングされたmRNAはさまざまな3'末端を有する[11][12][13]。
1981年、正常な内在性遺伝子の転写産物での選択的スプライシングの最初の例が同定された[11]。甲状腺ホルモンであるカルシトニンをコードする遺伝子は、哺乳類細胞では選択的スプライシングを受けることが判明した。この遺伝子からの一次転写産物は6つのエクソンを含んでおり、カルシトニンのmRNAはエクソン1–4を含み、エクソン4のポリアデニル化部位の後で終わる。この一次転写産物からは他のmRNAも産生され、それはエクソン4をスキップしエクソン1–3、5、6を含んでいる。このmRNAはカルシトニン遺伝子関連ペプチド(CGRP)として知られるタンパク質をコードしている[14][15]。哺乳類の免疫グロブリン遺伝子での選択的スプライシングの例も1980年代の初期に観察された[11][16]。
その後、選択的スプライシングは真核生物で普遍的な現象であることが判明した[1]。選択的スプライシングの「記録保持者」はキイロショウジョウバエ Drosophila melanogaster のDscamと呼ばれる遺伝子であり、この遺伝子からは38,016種類のスプライスバリアントが生じる可能性がある[17]。
形式[編集]

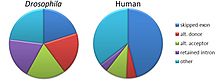
選択的スプライシングは5種類の基本的な形式が一般的に知られている[1][2][3][18]。
- エクソンスキッピング(exon skipping)またはカセットエクソン(cassette exon): エクソンは一時転写産物から除去されたり、保持されたままだったりする。哺乳類のmRNA前駆体では最も一般的な様式である[18]。
- 相互排他的エクソン(mutually exclusive exons): 2つのエクソンのうちどちらかがmRNAに保持されるが、両方保持されることはない。
- 選択的5'供与部位(alternative 5' donor sites): 選択的な5'スプライスジャンクション(供与部位)が利用され、上流のエクソンの3'側の境界が変化する。
- 選択的3'受容部位(alternative 3' acceptor sites): 選択的な3'スプライスジャンクション(受容部位)が利用され、下流のエクソンの5'側の境界が変化する。
- イントロン保持(intron retention): ある配列がイントロンとして除去されるか、そのまま保持されるかする。保持される配列がイントロンと隣接していないという点で、エクソンスキッピングとは区別される。保持されたイントロンがコーディング領域内であれば、隣接するエクソンと同じ読み枠でアミノ酸をコードしなければならない。終止コドンが存在したり読み枠がシフトしたりすれば、タンパク質は機能を失う。哺乳類では最も稀な形式である[18]。
これらの選択的スプライシングの主要な形式に加えて、同じ遺伝子から異なるmRNAが作り出される主要な機構が他に2つ存在する。多重プロモーター(multiple promoters)と多重ポリアデニル化部位(multiple polyadenylation sites)である。多重プロモーターの利用は選択的スプライシングではなく転写調節機構として説明されるが、異なる地点から転写が開始されることで最も5'側のエクソンが異なる転写産物が作り出されることがある。他方、多重ポリアデニル化部位は転写産物の3'末端の地点が異なる。どちらの機構も選択的スプライシングと組み合わせて用いられ、1つの遺伝子に由来するmRNAにさらなる多様性をもたらしている[1][3]

これらの形式は基本的なスプライシング機構を記述するものであるが、複雑なスプライシングの記述には不適切である可能性がある。例えば、右の図ではマウスのヒアルロニダーゼ3遺伝子の3種類のスプライシング形式を示している。1番目(緑)と2番目(黄)のエクソン構造を比較した場合の形式はイントロン保持となるが、2番目(黄)と3番目(青)を比較した場合はエクソンスキッピングとなる。すべての可能なスプライシングパターンを一意に示すための命名法が近年提唱されている[18]。
機構[編集]
一般的なスプライシング機構[編集]

DNAから転写されたmRNA前駆体(pre-mRNA)には、いくつかのイントロンとエクソンが含まれている(転写された1つのpre-mRNAには、線虫では平均して4–5個、ショウジョウバエでは100個以上のイントロンとエクソンが含まれていることもある)。mRNA中にどのエクソンが保持されるかは、スプライシングの過程で決定される。スプライス部位の調節と選択は、エクソン内スプライシングエンハンサー、エクソン内スプライシングサイレンサーといったpre-mRNA自身に存在するシス作用エレメント、そしてトランスに作用するスプライシング活性化因子とスプライシング抑制因子によって行われる。
真核生物の典型的なイントロンには、重要な領域を定義するコンセンサス配列が存在する。各イントロンは5'末端にGU配列を持っている。3'末端の近傍には分枝部位(branch site)が存在する。分枝地点のヌクレオチドは常にアデニン(A)であるが、その周辺配列にはいくぶん多様性が存在する。ヒトの分枝部位のコンセンサス配列はyUnAyである[19]。分枝部位に続いて一連のピリミジン配列(ポリピリミジントラクト)が存在し、それに3'末端のAG配列が続く[3]。
mRNAのスプライシングは、スプライソソームとして知られるRNA-タンパク質複合体によって行われる。スプライソソームは、U1、U2、U4、U5、U6と名付けられたsnRNPを含んでいる(U3はmRNAのスプライシングには関与しない)[20]。U1は5'末端のGU配列に結合し、U2はU2AFタンパク質因子の助けのもと、分枝部位内のAに結合する。この段階の複合体はスプライソソームA複合体として知られている。A複合体の形成は通常、スプライシングで除去されるイントロンの末端と保持されるエクソンの末端を決定する重要な段階である[3]。
U4、U5、U6が複合体に結合し、U6はU1に取って代わる。U1とU4が解離する。その後、残った複合体は2つのエステル交換反応を行う。最初の反応では、イントロンの5'末端が上流のエクソンから切り離され、分枝部位のAに対して2',5'-ホスホジエステル結合を形成する。2番目の反応では、イントロンの3'末端が下流のエクソンから切り離され、2つのエクソンがホスホジエステル結合で連結される。その後、投げ縄型のイントロンが解離して分解される[1]。
調節エレメントとタンパク質[編集]

スプライシングはトランスに作用するタンパク質(抑制因子と活性化因子)とpre-mRNA上に存在してシスに作用する調節部位(サイレンサーとエンハンサー)によって調節される。しかし、スプライシング因子の影響はしばしば位置依存的であることには留意すべきである。つまり、イントロンのエンハンサーエレメントへ結合した際にスプライシング活性化因子として働くスプライシング因子が、エクソンのスプライシングエレメントに結合した際には抑制因子として機能する場合もあり、逆もまた然りである[21]。pre-mRNA転写産物の二次構造も、スプライシングエレメントどうしを結び付けたり、スプライシング因子の結合エレメントを覆い隠したりといった形でスプライシングを調節する役割を持つ[22][23]。これらのエレメントは、さまざまな条件下でスプライシングがどのように起こるかを指示する「スプライシング・コード」を形成している[24][25]。
pre-mRNA上に存在するシス作用RNAエレメントには2つの主要なタイプが存在し、それらには対応するRNA結合タンパク質が存在する。スプライシングサイレンサーはスプライシング抑制タンパク質が結合する部位であり、近接する部位がスプライスジャンクションとして利用される可能性を低下させる。これらはイントロン自身に位置していることもあり(イントロン性スプライシングサイレンサー、ISS)、隣接するエクソンに位置していることもある(エクソン性スプライシングサイレンサー、ESS)。これらの配列は多様であり、結合するタンパク質の種類もまた多様である。スプライシング抑制因子の多くは、hnRNPA1やポリピリミジントラクト結合タンパク質(PTB)などのhnRNPである[3][24]。スプライシングエンハンサーはスプライシング活性化因子が結合する部位であり、近接する部位がスプライスジャンクションとして利用される可能性を高める。これらもまた、イントロンに位置するもの(イントロン性スプライシングエンハンサー、ISE)とエクソンに位置するもの(エクソン性スプライシングエンハンサー、ESE)がある。ISEとESEに結合する活性化タンパク質の大部分は、SRタンパク質ファミリーのメンバーである。これらのタンパク質はRNA認識モチーフとアルギニンセリンリッチドメイン(RSドメイン)を含んでいる[3][24]。
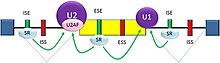
一般的に、スプライシングはコンテクストに依存した様式で決定される[25]。特定のシス作用RNAエレメントの存在は、ある場合には近接部位でのスプライシングの可能性を増加させるが他の場合では可能性を低下させることもあり、その効果はコンテクストに依存する。スプライシングを調節するコンテクストには、pre-mRNA上の他のRNA配列特徴の存在によって決定されるシス作用コンテクストと、細胞の条件によって決定されるトランス作用コンテクストが含まれる。例えば、一部のシス作用RNAエレメントは、複数のエレメントが同じ領域に存在するときにのみスプライシングに影響を与える。他の例としては、シス作用エレメントはその細胞でどのタンパク質が発現しているか(例えば神経型PTBか非神経型PTBか)によってスプライシングに反対の影響を与える。スプライシングサイレンサーとエンハンサーの適応的意義について研究がなされており、ヒトの遺伝子では新たなサイレンサーを生み出したり既存のエンハンサーを破壊することを防ぐ強い選択がかかっていることが示されている[26][27]。
社会性昆虫におけるDNAメチル化と選択的スプライシング[編集]
社会性昆虫では、CpGDNAメチル化が選択的スプライシングを調節する役割があることが示されている[28][29]。セイヨウミツバチ Apis mellifera では、ゲノム公開後に行われた最初のいくつかの研究によると、CpGメチル化がエクソンスキッピングを調節するようである[30][31][32]。CpGメチル化によって調節される選択的スプライシングは、エクソンスキッピングだけではなく、イントロン保持や他のスプライシングイベントでも広範な影響を与えている[33]。
例[編集]
エクソンスキッピング: ショウジョウバエdsx遺伝子[編集]
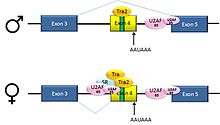
キイロショウジョウバエD. melanogasterのdsx遺伝子は、6つのエクソンを含んでいる。オスでは、エクソン1、2、3、5、6が連結されてmRNAを形成し、オスの発生に必要な転写調節タンパク質をコードする。メスでは、エクソン1、2、3、4が連結され、エクソン4のポリアデニル化シグナルがその地点でのmRNAの切断を引き起こす。その結果生じたmRNAはメスの発生に必要な転写調節タンパク質をコードする[34]。
これはエクソンスキッピングの例である。エクソン4の上流のイントロンは、コンセンサス配列とあまり一致しないポリピリミジントラクトを持つため、U2AFタンパク質はスプライシング活性化因子の補助がなければほとんど結合しない。そのため、この3'スプライス受容部位はオスでは利用されない。メスではスプライシング活性化因子Transformer(Tra)が産生されている(下を参照)。SRタンパク質Tra2は両性で産生されており、エクソン4のESEに結合する。Tra存在下では、TraはTra2へ結合し、他のSRタンパク質とともに弱いポリピリミジントラクトへのU2AFタンパク質の結合を補助する複合体を形成する。その結果U2が分枝点にリクルートされ、エクソン4がmRNAへ組み込まれる[34][35]。
選択的受容部位: ショウジョウバエTransformer遺伝子[編集]

キイロショウジョウバエD. melanogasterのTransformer遺伝子のpre-mRNAは、選択的受容部位形式の選択的スプライシングを受ける。この遺伝子がコードするTransformer(Tra)タンパク質は、メスでのみ発現する。この遺伝子の一次転写産物は、2つの受容部位を持つイントロンを含んでいる。オスでは、上流の受容部位が利用される。そのためプロセシングされた転写産物には長いバージョンのエクソン2が組み込まれ、そこには本来の終止コドンよりも上流に代替的終止コドンが含まれている。その結果、mRNAは切り詰められた不活性なタンパク質をコードすることとなる。メスは性決定タンパク質であるSxlを産生する。Sxlタンパク質はTra転写産物の上流の受容部位近傍のISSに結合するスプライシング抑制因子であり、U2AFタンパク質がポリピリミジントラクトに結合するのを防ぐ。これによってこのジャンクションの利用が防がれ、スプライソソームは下流の受容部位へ結合する。この地点でスプライシングが起こることで、上流の代替的終止コドンはイントロンの一部として除去される。その結果、mRNAは活性のあるTraタンパク質をコードし、他の性関連遺伝子の選択的スプライシングの調節因子となる(上を参照)[1]。
エクソンの定義: Fas受容体[編集]
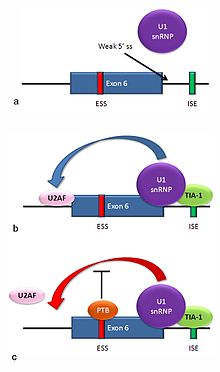
Fas受容体の複数のアイソフォームは選択的スプライシングによって生み出される。ヒトで通常生じる2つのアイソフォームはエクソンスキッピング機構によって生み出される。エクソン6を含むmRNAは膜結合型のFas受容体をコードし、アポトーシスまたはプログラム細胞死を促進する。慢性的に日光に曝された皮膚細胞ではFas受容体の発現が上昇しているのに対し皮膚がん細胞では発現がみられないことは、ヒトではこの機構が前がん状態の細胞の除去に重要であることを示唆している[36]。エクソン6がスキップされた場合、生じるmRNAは可溶性のFasタンパク質をコードし、これはアポトーシスを促進しない。エクソンが組み込まれるかスキップされるかは、TIA-1とPTBという拮抗する2つのタンパク質に依存している。
- pre-mRNAのエクソン6の下流のイントロンの5'供与部位はコンセンサス配列との間に弱い一致しかみられず、通常U1 snRNPは結合しない。U1が結合していないとき、エクソンはスキップされる(図のaを参照)。
- ISEにTIA-1タンパク質が結合すると、U1 snRNPの結合が安定化される[3]。その結果形成された5'供与部位の複合体はエクソンの上流の3'スプライス部位へのU2AFの結合を補助するが、その機構は不明である(bを参照)[37]。
- エクソン6はピリミジンに富むESS(ure6)を含んでおり、そこへPTBが結合する。PTBが結合した場合、PTBは5'供与部位複合体がU2AFの受容部位への結合に与える影響を阻害し、その結果エクソンはスキップされる(cを参照)。
この機構はスプライシングによってエクソンが定義される例である。通常スプライソソームはイントロンで組み立てられ、snRNPのサブユニットはイントロンの5'端と3'端が結合するようにRNAを折り畳む。しかし、このような近年研究されている例はエクソンの両端の相互作用も存在することを示している。このようなエクソンを定義する相互作用では、2つの近接するイントロンでスプライソソームが組み立てられる前にコアとなるスプライシング因子が結合することが必要である[37]。
抑制因子と活性化因子の競合: HIV-1 tat エクソン2[編集]

ヒトでAIDSを引き起こすレトロウイルスであるHIVは1本のRNA一次転写産物を産生し、複数通りの選択的スプライシングを受けて40以上の異なるmRNAが産生される[38]。異なるスプライシングを受けた転写産物間の平衡によって異なる産物をコードする複数のmRNAがもたらされており、この機構はウイルスの複製に必要とされる[39]。異なるスプライシングを受ける転写産物にはtat遺伝子が含まれ、その中のエクソン2がスキップされたり組み込まれたりするカセットエクソンである。エクソン2の組み込みはスプライシング抑制因子hnRNP A1とSRタンパク質SC35の間の競合によって調節されている。エクソン2内部に存在するESSとESEの配列は重複している。A1抑制タンパク質がESSに結合した場合、複数のA1分子が協調的に結合してエクソン2の上流の5'供与部位まで伸び、コアスプライシング因子U2AF35がポリピリミジントラクトに結合するのを防ぐ。SC35がESEに結合した場合、A1の結合は防がれて5'供与部位はスプライソソームの組み立てのためにアクセス可能な状態が維持される。活性化因子と抑制因子の競合は、両方のmRNAのタイプ(エクソン2を含むものと含まないもの)が産生されることを保証している[38]。
適応的意義[編集]
選択的スプライシングは、1つのDNA配列が1つのポリペプチドをコードするという概念(一遺伝子一酵素説)の例外の1つである。現在では「一遺伝子多ポリペプチド」とでもいう方が正確であるかもしれない[40]。あるDNA配列やあるpre-mRNAからどのポリペプチドが産生されるかを決定するためには、外部の情報が必要である。調節方法は遺伝するため、変異によって遺伝子発現に影響を与える新たな方法がもたらされている[7]。
真核生物においては、選択的スプライシングは情報をずっと効率的に保存するための非常に重要なステップであることが提唱されている。いくつかのタンパク質をそれぞれ別々の遺伝子ではなく1つの遺伝子にコードすることによって、限られたサイズのゲノムからより多様なプロテオームを作り出すことができるようになる[1]。また、選択的スプライシングによって進化的な柔軟性がもたらされる。1か所の点変異によって、あるエクソンが時折除去されたり組み込まれたりするようになる可能性があり、これによって元のタンパク質を失うことなく新たなアイソフォームを生み出すことができる[1]。非構成的(選択的)エクソンには天然変性領域(天然変性タンパク質を参照)が多く見られることが研究で示されており[41]、アイソフォーム間の機能的差異はこうした領域の機能的モジュールの変化によってもたらされていることが示唆されている。アイソフォーム間の機能的差異はそれらの発現パターンにも反映されており、機械学習によるアプローチによる予測も行われている[42][43]。また、進化の過程で選択的スプライシングは多細胞性よりも先に出現しており、この機構が多細胞生物の発達を補助するために採用されたものである可能性が示唆されている[44]。
ヒトゲノムプロジェクトや他のゲノムシーケンシングに基づいた研究によって、ヒトの遺伝子の数は線虫 Caenorhabditis elegans よりも30%多いだけであり、キイロショウジョウバエ Drosophila melanogaster のわずか2倍である。この発見はヒト、より一般的に脊椎動物でみられる複雑性は、無脊椎動物よりもヒトで選択的スプライシングが高率で起こるためではないか、という思索をもたらした[45][46]。しかし、ヒト、マウス、ラット、ウシ、ハエ(D. melanogaster)、線虫(C. elegans)、そしてシロイヌナズナ由来のそれぞれ100,000のESTサンプルを用いた研究では、ヒトと他の動物との間で選択的スプライシングを受ける遺伝子の頻度に大きな差は見られなかった[47]。一方別の研究では、これらの結果は生物種によって利用可能なESTの数が異なることによるアーティファクトであるとされた。各生物種からランダムに選ばれた遺伝子で選択的スプライシングを比較した際には、脊椎動物では無脊椎動物よりも高率で選択的スプライシングが起こっていると著者らは結論付けている[48]。
疾患[編集]
RNAプロセシング装置の変化は複数の転写産物に誤ったスプライシングを引き起こす一方、スプライシング部位やシス作用調節部位の一塩基置換は1つの遺伝子のスプライシングに変化をもたらす。確率的解析による2005年の研究では、ヒトの疾患の原因となる変異の60%以上が、コーディング配列に直接影響を与えるものではなく、スプライシングに影響を与えるものであることが示された[49]。より最近の研究では、すべての遺伝疾患のうち1/3がスプライシングの要素を有する可能性が高いことが示された[21]。正確な割合がどの程度であるかは置いておくとして、スプライシングと関連した疾患は多数存在する[50]。後述する通り、スプライシングと関連した疾患の有名な例はがんである。
異常なスプライシングを受けたmRNAは、がん細胞に高い割合で見つかる[5][6][8]。RNA-Seqとプロテオミクス解析を組み合わせた研究によって、がん経路に重要なタンパク質のスプライシングアイソフォームに大きな差が生じていることが明らかにされた[51]。このような異常なスプライシングパターンががんの成長に寄与しているのか、それとも単にがんと関連した細胞の異常の結果であるのかは必ずしも明らかではない。大腸がんや前立腺がんなど特定種のがんではスプライシングエラーの数が個々のがんで大きく異なることが示されており、この現象はtranscriptome instability(トランスクリプトームの不安定性)と呼ばれる[52][53]。さらに、transcriptome instabilityはスプライシング因子の遺伝子の発現レベルの低下と大きく相関することが示されている。DNMT3Aの変異は血液のがんに寄与することが示されており、変異細胞株では同系統の野生株と比較してtransriptome instabilityを示す[54]。
がん細胞では正常細胞と比較して選択的スプライシングの減少とスプライシングのタイプの変化が生じている。例えば、がん細胞ではイントロン保持は通常よりも高率であるが、エクソンスキッピングは低率である[55]。がん細胞でのスプライシングの差異は、スプライシング因子の遺伝子の体細胞変異が高率でみられるためである可能性があり[8]、またスプライシング因子のリン酸化の変化によるものである可能性もある[7]。他には、産生されるスプライシング因子の相対的な量の変化の可能性もあり、乳がんではスプライシング因子SF2/ASFのレベルが上昇している[56]。ある研究では、正常細胞より腫瘍細胞で高頻度に見られるスプライスバリアントの割合は比較的小さく(26000以上のうちの383種類)、誤ったスプライシングを受けたときに腫瘍の成長に寄与する遺伝子は限られていることが示唆されている[57]。一方で、誤ったスプライシングを受けた転写産物は通常ナンセンス変異依存mRNA分解機構(NMD)と呼ばれる転写後品質管理機構によって除去されると考えられている[58]。
特定のスプライシングバリアントががんと関係している例としては、ヒトのDNAメチルトランスフェラーゼ(DNMT)遺伝子の変異がある。3つのDNMT遺伝子はDNAにメチル基を付加する酵素をコードし、この修飾は多くの場合調節効果を有する。異常なスプライシングを受けたDNMT3BのmRNAが腫瘍やがん細胞株では見いだされる。2つの異なる研究において、これらの異常なスプライシングを受けたmRNAのうちの2種類で、哺乳類細胞での発現によってDNAメチル化のパターンの変化が引き起こされることが示された。異常なmRNAの1つを発現する細胞は対照細胞の2倍の速度で成長し、腫瘍成長への直接的な寄与が示された[7]。
他の例としては、Ron(MST1R)がん原遺伝子が挙げられる。がん細胞の重要な性質は、正常組織へ移動し侵入する能力である。Ronの異常スプライシング産物は、乳がん細胞でのSF2/ASFのレベルの上昇と関連していることが判明している。このmRNAにコードされるRonタンパク質の異常なアイソフォームは、細胞に運動性をもたらす[56]。
側坐核の特定のニューロン集団におけるFOSB遺伝子の切り詰められたスプライスバリアント(ΔFosB)の過剰発現は、薬物依存や行動嗜癖の誘導と維持の原因機構として同定されている[59][60][61][62]。
近年の研究では、選択的スプライシングの調節におけるクロマチン構造やヒストン修飾の重要な機能が指摘されている。これらの洞察はエピジェネティックな調節機構は、ゲノムのどの部分が発現するかだけでなく、それらがどのようにスプライシングされるかについても決定していることを示唆している[63]。
ゲノムワイド解析[編集]
選択的スプライシングのゲノムワイド解析は挑戦的な課題である。一般的には選択的スプライシングを受けた転写産物はESTの配列を比較することで発見されるが、これには非常に多数のESTのシーケンシングを必要とする。ESTライブラリの大部分は限られた数の組織に由来し、そのため組織特異的なスプライスバリアントは見逃されがちである。一方、DNAマイクロアレイベースの解析、RNA結合アッセイ、ディープシーケンシングといった、ハイスループットなアプローチでスプライシングを調査する手法も開発されている。これらの手法は、タンパク質結合に影響を与えるスプライシングエレメント周辺の多型や変異のスクリーニングにも利用される。In vivoでのレポーター遺伝子アッセイなどのスプライシングアッセイと組み合わせることで、多型や変異がpre-mRNAのスプライシングに与える機能的な影響を分析することができる[21][24][64]。
マイクロアレイ解析では、アレイには個々のエクソン(Affymetrix exon microarrayなど)またはエクソン-エクソン境界(ExonHitやJivanなど)のDNA断片が利用される。アレイは研究対象の組織由来のラベルされたcDNAとの結合が行われる。プローブとなるcDNAは、その組織でmRNAに組み込まれているエクソン由来のDNAまたはエクソン境界のDNAに結合する。これによって特定の選択的スプライシングを受けたmRNAの存在が明らかにされる[65]。
CLIP(cross-linking and immunoprecipitation、クロスリンクと免疫沈降)では、スプライシングが行われている組織でタンパク質とRNA分子の紫外線照射による架橋が行われる。そして、研究対象のトランス作用スプライシング調節タンパク質は特異的抗体を用いて沈降される。タンパク質に結合しているRNAの単離とクローニングが行われ、そのタンパク質の標的配列が明らかとなる[4]。RNA結合タンパク質を同定しそのpre-mRNA転写産物へのマッピングを行う他の手法としては、MEGAshift(Microarray Evaluation of Genomic Aptamers by shift)がある[66]。この手法は、SELEX法(Systematic Evolution of Ligands by Exponential Enrichment)[67] の応用であり、マイクロアレイベースの読み出しを組み合わせたものである。MEGAshift法によって選択的スプライシングが行われるエクソン周辺においてASF/SF2やPTBといったスプライシング因子が結合する配列が特定され、選択的スプライシングの調節に新たな洞察がもたらされた[68]。このアプローチはRNAの二次構造とスプライシング因子の結合の関係を明らかにするためにも利用されている[23]。
ディープシーケンシング技術は、プロセシングを受けていないmRNAや受けたmRNAのゲノムワイド解析を行うために利用されており、選択的スプライシングへ洞察をもたらしている。例えば、ディープシーケンシングを用いて得られた結果からは、ヒトでは複数のエクソンからなる遺伝子の転写産物の95%が選択的スプライシングを受けると推計され、多数のpre-mRNA転写産物は組織特異的なスプライシングを受けていることが示された[2]。機能ゲノミクスとmultiple instance learningベースの計算機アプローチが、RNA-seqのデータを統合し、選択的スプライシングを受けたアイソフォームの機能を予測するために開発されている[43]。ディープシーケンシングは、スプライシングの過程で放出される一時的な投げ縄構造のin vivoでの検出、分枝部位の配列の決定、そしてヒトのpre-mRNA転写産物における分枝点の大規模マッピングにも利用されている[69]。
レポーターアッセイは、特定の選択的スプライシングイベントに関与するスプライシングタンパク質の発見を可能にする。この場合、レポーター遺伝子はスプライシング反応が起こったかどうかによって2つの異なる蛍光タンパク質のいずれか1つを発現するように構築される。この手法はスプライシングに影響が生じている変異体を単離し、それらの変異体で不活性化されている新規スプライシング調節タンパク質を同定するために利用される[4]。
データベース[編集]
選択的スプライシングのデータベースは複数存在する。これらのデータベースは、選択的スプライシングを受けるpre-mRNAを産生する遺伝子を発見する際に有用である。
出典[編集]
- ^ a b c d e f g h i “Mechanisms of alternative pre-messenger RNA splicing”. Annual Review of Biochemistry 72 (1): 291–336. (2003). doi:10.1146/annurev.biochem.72.121801.161720. PMID 12626338.
- ^ a b c “Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing”. Nature Genetics 40 (12): 1413–5. (December 2008). doi:10.1038/ng.259. PMID 18978789.
- ^ a b c d e f g h i j “Understanding alternative splicing: towards a cellular code”. Nature Reviews. Molecular Cell Biology 6 (5): 386–98. (May 2005). doi:10.1038/nrm1645. PMID 15956978.
- ^ a b c “The search for alternative splicing regulators: new approaches offer a path to a splicing code”. Genes & Development 22 (3): 279–85. (February 2008). doi:10.1101/gad.1643108. PMC 2731647. PMID 18245441.
- ^ a b “Alternative splicing in cancer: noise, functional, or systematic?”. The International Journal of Biochemistry & Cell Biology 39 (7-8): 1432–49. (2007). doi:10.1016/j.biocel.2007.02.016. PMID 17416541.
- ^ a b Bauer, Joseph Alan, ed (2009). “A global view of cancer-specific transcript variants by subtractive transcriptome-wide analysis”. PLOS ONE 4 (3): e4732. Bibcode: 2009PLoSO...4.4732H. doi:10.1371/journal.pone.0004732. PMC 2648985. PMID 19266097.
- ^ a b c d “Aberrant RNA splicing and its functional consequences in cancer cells” (Free full text). Disease Models & Mechanisms 1 (1): 37–42. (2008). doi:10.1242/dmm.000331. PMC 2561970. PMID 19048051.
- ^ a b c d “Aberrant RNA splicing in cancer; expression changes and driver mutations of splicing factor genes”. Oncogene 35 (19): 2413–27. (May 2016). doi:10.1038/onc.2015.318. PMID 26300000.
- ^ “An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA”. Cell 12 (1): 1–8. (September 1977). doi:10.1016/0092-8674(77)90180-5. PMID 902310.
- ^ “Spliced segments at the 5' terminus of adenovirus 2 late mRNA”. Proceedings of the National Academy of Sciences of the United States of America 74 (8): 3171–5. (August 1977). Bibcode: 1977PNAS...74.3171B. doi:10.1073/pnas.74.8.3171. PMC 431482. PMID 269380.
- ^ a b c “Complex transcriptional units: diversity in gene expression by alternative RNA processing”. Annual Review of Biochemistry 55 (1): 1091–117. (1986). doi:10.1146/annurev.bi.55.070186.005303. PMID 3017190.
- ^ “The spliced structures of adenovirus 2 fiber message and the other late mRNAs”. Cell 15 (2): 497–510. (October 1978). doi:10.1016/0092-8674(78)90019-3. PMID 719751.
- ^ “Steps in the processing of Ad2 mRNA: poly(A)+ nuclear sequences are conserved and poly(A) addition precedes splicing”. Cell 15 (4): 1477–93. (December 1978). doi:10.1016/0092-8674(78)90071-5. PMID 729004.
- ^ “Altered expression of the calcitonin gene associated with RNA polymorphism”. Nature 290 (5801): 63–5. (March 1981). Bibcode: 1981Natur.290...63R. doi:10.1038/290063a0. PMID 7207587.
- ^ “Calcitonin mRNA polymorphism: peptide switching associated with alternative RNA splicing events”. Proceedings of the National Academy of Sciences of the United States of America 79 (6): 1717–21. (March 1982). Bibcode: 1982PNAS...79.1717R. doi:10.1073/pnas.79.6.1717. PMC 346051. PMID 6952224.
- ^ “The role of DNA rearrangement and alternative RNA processing in the expression of immunoglobulin delta genes”. Cell 24 (2): 353–65. (May 1981). doi:10.1016/0092-8674(81)90325-1. PMID 6786756.
- ^ “Drosophila Dscam is an axon guidance receptor exhibiting extraordinary molecular diversity”. Cell 101 (6): 671–84. (June 2000). doi:10.1016/S0092-8674(00)80878-8. PMID 10892653.
- ^ a b c d e Brent, Michael R., ed (August 2008). “A general definition and nomenclature for alternative splicing events”. PLoS Computational Biology 4 (8): e1000147. Bibcode: 2008PLSCB...4E0147S. doi:10.1371/journal.pcbi.1000147. PMC 2467475. PMID 18688268.
- ^ “Human branch point consensus sequence is yUnAy”. Nucleic Acids Research 36 (7): 2257–67. (April 2008). doi:10.1093/nar/gkn073. PMC 2367711. PMID 18285363.
- ^ Clark, David (2005). Molecular biology. Amsterdam: Elsevier Academic Press. ISBN 978-0-12-175551-5
- ^ a b c “Using positional distribution to identify splicing elements and predict pre-mRNA processing defects in human genes”. Proceedings of the National Academy of Sciences of the United States of America 108 (27): 11093–8. (July 2011). Bibcode: 2011PNAS..10811093H. doi:10.1073/pnas.1101135108. PMC 3131313. PMID 21685335.
- ^ “Role of RNA structure in regulating pre-mRNA splicing”. Trends in Biochemical Sciences 35 (3): 169–78. (March 2010). doi:10.1016/j.tibs.2009.10.004. PMC 2834840. PMID 19959365.
- ^ a b “Next-generation SELEX identifies sequence and structural determinants of splicing factor binding in human pre-mRNA sequence”. RNA 15 (12): 2385–97. (December 2009). doi:10.1261/rna.1821809. PMC 2779669. PMID 19861426.
- ^ a b c d “Splicing regulation: from a parts list of regulatory elements to an integrated splicing code” (Free full text). RNA 14 (5): 802–13. (May 2008). doi:10.1261/rna.876308. PMC 2327353. PMID 18369186.
- ^ a b “Deciphering the splicing code”. Nature 465 (7294): 53–9. (May 2010). Bibcode: 2010Natur.465...53B. doi:10.1038/nature09000. PMID 20445623.
- ^ “Positive selection acting on splicing motifs reflects compensatory evolution”. Genome Research 18 (4): 533–43. (April 2008). doi:10.1101/gr.070268.107. PMC 2279241. PMID 18204002.
- ^ “Single nucleotide polymorphism-based validation of exonic splicing enhancers”. PLoS Biology 2 (9): E268. (September 2004). doi:10.1371/journal.pbio.0020268. PMC 514884. PMID 15340491.
- ^ “The Function of DNA Methylation Marks in Social Insects”. Frontiers in Ecology and Evolution 4: 57. (2016). doi:10.3389/fevo.2016.00057.
- ^ “Physiological and molecular mechanisms of nutrition in honey bees.”. Advances in insect physiology. 49. Academic Press. (January 2015). pp. 25–58. doi:10.1016/bs.aiip.2015.06.002
- ^ “The honey bee epigenomes: differential methylation of brain DNA in queens and workers”. PLoS Biology 8 (11): e1000506. (November 2010). doi:10.1371/journal.pbio.1000506. PMC 2970541. PMID 21072239.
- ^ “Genome-wide association between DNA methylation and alternative splicing in an invertebrate”. BMC Genomics 13 (1): 480. (September 2012). doi:10.1186/1471-2164-13-480. PMC 3526459. PMID 22978521.
- ^ “Insights into social insects from the genome of the honeybee Apis mellifera”. Nature 443 (7114): 931–49. (October 2006). Bibcode: 2006Natur.443..931T. doi:10.1038/nature05260. PMC 2048586. PMID 17073008.
- ^ “RNA interference knockdown of DNA methyl-transferase 3 affects gene alternative splicing in the honey bee”. Proceedings of the National Academy of Sciences of the United States of America 110 (31): 12750–5. (July 2013). Bibcode: 2013PNAS..11012750L. doi:10.1073/pnas.1310735110. PMC 3732956. PMID 23852726.
- ^ a b “Assembly of specific SR protein complexes on distinct regulatory elements of the Drosophila doublesex splicing enhancer”. Genes & Development 10 (16): 2089–101. (August 1996). doi:10.1101/gad.10.16.2089. PMID 8769651.
- ^ “The role of U2AF35 and U2AF65 in enhancer-dependent splicing”. RNA 7 (6): 806–18. (June 2001). doi:10.1017/S1355838201010317. PMC 1370132. PMID 11421359.
- ^ “Expression of CD95 (Fas) in sun-exposed human skin and cutaneous carcinomas”. Cancer 94 (3): 814–9. (February 2002). doi:10.1002/cncr.10277. PMID 11857317.
- ^ a b “Regulation of Fas alternative splicing by antagonistic effects of TIA-1 and PTB on exon definition”. Molecular Cell 19 (4): 475–84. (August 2005). doi:10.1016/j.molcel.2005.06.015. PMID 16109372.
- ^ a b “SC35 and heterogeneous nuclear ribonucleoprotein A/B proteins bind to a juxtaposed exonic splicing enhancer/exonic splicing silencer element to regulate HIV-1 tat exon 2 splicing”. The Journal of Biological Chemistry 279 (11): 10077–84. (March 2004). doi:10.1074/jbc.M312743200. PMID 14703516.
- ^ “A second exon splicing silencer within human immunodeficiency virus type 1 tat exon 2 represses splicing of Tat mRNA and binds protein hnRNP H”. The Journal of Biological Chemistry 276 (44): 40464–75. (November 2001). doi:10.1074/jbc.M104070200. PMID 11526107.
- ^ “HHMI Bulletin September 2005: Alternative Splicing”. www.hhmi.org. 2009年6月22日時点のオリジナルよりアーカイブ。2009年5月26日閲覧。
- ^ “Alternative splicing in concert with protein intrinsic disorder enables increased functional diversity in multicellular organisms”. Proceedings of the National Academy of Sciences of the United States of America 103 (22): 8390–5. (May 2006). Bibcode: 2006PNAS..103.8390R. doi:10.1073/pnas.0507916103. PMC 1482503. PMID 16717195.
- ^ “The emerging era of genomic data integration for analyzing splice isoform function”. Trends in Genetics 30 (8): 340–7. (August 2014). doi:10.1016/j.tig.2014.05.005. PMC 4112133. PMID 24951248.
- ^ a b “Systematically differentiating functions for alternatively spliced isoforms through integrating RNA-seq data”. PLoS Computational Biology 9 (11): e1003314. (Nov 2013). Bibcode: 2013PLSCB...9E3314E. doi:10.1371/journal.pcbi.1003314. PMC 3820534. PMID 24244129.
- ^ “Functional and evolutionary analysis of alternatively spliced genes is consistent with an early eukaryotic origin of alternative splicing”. BMC Evolutionary Biology 7: 188. (October 2007). doi:10.1186/1471-2148-7-188. PMC 2082043. PMID 17916237.
- ^ “Analysis of expressed sequence tags indicates 35,000 human genes”. Nature Genetics 25 (2): 232–4. (June 2000). doi:10.1038/76115. PMID 10835644.
- ^ “Estimate of human gene number provided by genome-wide analysis using Tetraodon nigroviridis DNA sequence”. Nature Genetics 25 (2): 235–8. (June 2000). doi:10.1038/76118. PMID 10835645.
- ^ “Alternative splicing and genome complexity”. Nature Genetics 30 (1): 29–30. (January 2002). doi:10.1038/ng803. PMID 11743582.
- ^ “Different levels of alternative splicing among eukaryotes”. Nucleic Acids Research 35 (1): 125–31. (2006). doi:10.1093/nar/gkl924. PMC 1802581. PMID 17158149.
- ^ “Are splicing mutations the most frequent cause of hereditary disease?”. FEBS Letters 579 (9): 1900–3. (March 2005). doi:10.1016/j.febslet.2005.02.047. PMID 15792793.
- ^ “The pathobiology of splicing”. The Journal of Pathology 220 (2): 152–63. (January 2010). doi:10.1002/path.2649. PMC 2855871. PMID 19918805.
- ^ “A new class of protein cancer biomarker candidates: differentially expressed splice variants of ERBB2 (HER2/neu) and ERBB1 (EGFR) in breast cancer cell lines”. Journal of Proteomics 107: 103–12. (July 2014). doi:10.1016/j.jprot.2014.04.012. PMC 4123867. PMID 24802673.
- ^ “Transcriptome instability as a molecular pan-cancer characteristic of carcinomas”. BMC Genomics 15: 672. (August 2014). doi:10.1186/1471-2164-15-672. PMC 3219073. PMID 25109687.
- ^ “Transcriptome instability in colorectal cancer identified by exon microarray analyses: Associations with splicing factor expression levels and patient survival”. Genome Medicine 3 (5): 32. (May 2011). doi:10.1186/gm248. PMC 4137096. PMID 21619627.
- ^ “Abnormal RNA splicing and genomic instability after induction of DNMT3A mutations by CRISPR/Cas9 gene editing”. Blood Cells, Molecules & Diseases 69: 10–22. (March 2018). doi:10.1016/j.bcmd.2017.12.002. PMC 6728079. PMID 29324392.
- ^ “Insights into the connection between cancer and alternative splicing”. Trends in Genetics 24 (1): 7–10. (January 2008). doi:10.1016/j.tig.2007.10.001. PMID 18054115.
- ^ a b “Cell motility is controlled by SF2/ASF through alternative splicing of the Ron protooncogene”. Molecular Cell 20 (6): 881–90. (December 2005). doi:10.1016/j.molcel.2005.10.026. PMID 16364913.
- ^ “Identification of alternatively spliced mRNA variants related to cancers by genome-wide ESTs alignment”. Oncogene 23 (17): 3013–23. (April 2004). doi:10.1038/sj.onc.1207362. PMID 15048092.
- ^ “Abnormally spliced beta-globin mRNAs: a single point mutation generates transcripts sensitive and insensitive to nonsense-mediated mRNA decay”. Blood 99 (5): 1811–6. (March 2002). doi:10.1182/blood.V99.5.1811. PMID 11861299.
- ^ “Cellular basis of memory for addiction”. Dialogues in Clinical Neuroscience 15 (4): 431–43. (December 2013). PMC 3898681. PMID 24459410. "DESPITE THE IMPORTANCE OF NUMEROUS PSYCHOSOCIAL FACTORS, AT ITS CORE, DRUG ADDICTION INVOLVES A BIOLOGICAL PROCESS: the ability of repeated exposure to a drug of abuse to induce changes in a vulnerable brain that drive the compulsive seeking and taking of drugs, and loss of control over drug use, that define a state of addiction. ... A large body of literature has demonstrated that such ΔFosB induction in D1-type NAc neurons increases an animal's sensitivity to drug as well as natural rewards and promotes drug self-administration, presumably through a process of positive reinforcement"
- ^ “Molecular neurobiology of addiction: what's all the (Δ)FosB about?”. The American Journal of Drug and Alcohol Abuse 40 (6): 428–37. (November 2014). doi:10.3109/00952990.2014.933840. PMID 25083822. "ΔFosB is an essential transcription factor implicated in the molecular and behavioral pathways of addiction following repeated drug exposure. The formation of ΔFosB in multiple brain regions, and the molecular pathway leading to the formation of AP-1 complexes is well understood. The establishment of a functional purpose for ΔFosB has allowed further determination as to some of the key aspects of its molecular cascades, involving effectors such as GluR2 (87,88), Cdk5 (93) and NFkB (100). Moreover, many of these molecular changes identified are now directly linked to the structural, physiological and behavioral changes observed following chronic drug exposure (60,95,97,102). New frontiers of research investigating the molecular roles of ΔFosB have been opened by epigenetic studies, and recent advances have illustrated the role of ΔFosB acting on DNA and histones, truly as a ‘‘molecular switch’’ (34). As a consequence of our improved understanding of ΔFosB in addiction, it is possible to evaluate the addictive potential of current medications (119), as well as use it as a biomarker for assessing the efficacy of therapeutic interventions (121,122,124). Some of these proposed interventions have limitations (125) or are in their infancy (75). However, it is hoped that some of these preliminary findings may lead to innovative treatments, which are much needed in addiction."
- ^ “Epigenetic regulation in drug addiction”. Annals of Agricultural and Environmental Medicine 19 (3): 491–6. (2012). PMID 23020045. "For these reasons, ΔFosB is considered a primary and causative transcription factor in creating new neural connections in the reward centre, prefrontal cortex, and other regions of the limbic system. This is reflected in the increased, stable and long-lasting level of sensitivity to cocaine and other drugs, and tendency to relapse even after long periods of abstinence. These newly constructed networks function very efficiently via new pathways as soon as drugs of abuse are further taken"
- ^ “Natural rewards, neuroplasticity, and non-drug addictions”. Neuropharmacology 61 (7): 1109–22. (December 2011). doi:10.1016/j.neuropharm.2011.03.010. PMC 3139704. PMID 21459101.
- ^ “Epigenetics in alternative pre-mRNA splicing”. Cell 144 (1): 16–26. (January 2011). doi:10.1016/j.cell.2010.11.056. PMC 3038581. PMID 21215366.
- ^ “Predictive identification of exonic splicing enhancers in human genes”. Science 297 (5583): 1007–13. (August 2002). Bibcode: 2002Sci...297.1007F. doi:10.1126/science.1073774. PMID 12114529.
- ^ “Revealing global regulatory features of mammalian alternative splicing using a quantitative microarray platform”. Molecular Cell 16 (6): 929–41. (December 2004). doi:10.1016/j.molcel.2004.12.004. PMID 15610736.
- ^ “A rapid high-throughput method for mapping ribonucleoproteins (RNPs) on human pre-mRNA”. Journal of Visualized Experiments 34 (34): 1622. (December 2009). doi:10.3791/1622. PMC 3152247. PMID 19956082.
- ^ “Systematic evolution of ligands by exponential enrichment: RNA ligands to bacteriophage T4 DNA polymerase”. Science 249 (4968): 505–10. (August 1990). Bibcode: 1990Sci...249..505T. doi:10.1126/science.2200121. PMID 2200121.
- ^ “High-throughput binding analysis determines the binding specificity of ASF/SF2 on alternatively spliced human pre-mRNAs”. Combinatorial Chemistry & High Throughput Screening 13 (3): 242–52. (March 2010). doi:10.2174/138620710790980522. PMC 3427726. PMID 20015017.
- ^ “Large-scale mapping of branchpoints in human pre-mRNA transcripts in vivo”. Nature Structural & Molecular Biology 19 (7): 719–21. (June 2012). doi:10.1038/nsmb.2327. PMC 3465671. PMID 22705790.
参考文献[編集]
- 今堀 和友、山川 民夫 編集 『生化学辞典 第4版』 東京化学同人 2007年 ISBN 978-4-8079-0670-3
- Zheng ZM. "Regulation of alternative RNA splicing by exon definition and exon sequences in viral and mammalian gene expression." J Biomed Sci. 2004 May-Jun;11(3):278-94. [1]
- Norton PA. "Alternative pre-mRNA splicing: factors involved in splice site selection." J Cell Sci. 1994 Jan;107 ( Pt 1):1-7. [2]
関連項目[編集]
- AspicDB - ヒトタンパク質変異体のデータベース
- Exitron - イントロンとエキソンのデータベース
- Intronerator - 線虫の選択的スプライシング遺伝子とイントロンのデータベース
- ProSAS - スプライシングがタンパク質の構造に与える影響を記述したデータベース
- SpliceInfo - ゲノムに存在する4つの主要な選択的スプライシングモードのデータベース
- トランススプライシング - 異なる一時転写産物のエクソンが連結されるRNAスプライシングの特殊な形態
外部リンク[編集]
- A General Definition and Nomenclature for Alternative Splicing Events at SciVee
- AStalavista (Alternative Splicing landscape visualization tool), a method for the computationally exhaustive classification of Alternative Splicing Structures
- IsoPred: computationally predicted isoform functions
- Stamms-lab.net: Research Group dealing with alternative Splicing issues and mis-splicing in human diseases
- Alternative Splicing of ion channels in the brain, connected to mental and neurological diseases
- BIPASS: Web Services in Alternative Splicing
