「コピー数変化」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| (同じ利用者による、間の1版が非表示) | |||
| 1行目: | 1行目: | ||
英語名:copy number variations: CNVs | 英語名:copy number variations: CNVs | ||
| 12行目: | 5行目: | ||
類義語:コピー数多型、コピー数変異 | 類義語:コピー数多型、コピー数変異 | ||
コピー数変化とは、染色体上の1kb以上にわたるゲノムDNAが、通常2コピーのところ、1コピー以下(欠失)、あるいは3コピー以上(重複)となっている現象をいう。ヒトゲノム中の12%にわたる領域に見いだされる。正常ゲノムに多型的に存在する場合や疾患ゲノムの原因として存在する場合などがある。 | コピー数変化とは、染色体上の1kb以上にわたるゲノムDNAが、通常2コピーのところ、1コピー以下(欠失)、あるいは3コピー以上(重複)となっている現象をいう。ヒトゲノム中の12%にわたる領域に見いだされる。正常ゲノムに多型的に存在する場合や疾患ゲノムの原因として存在する場合などがある。 | ||
==コピー数変化とは== | ==コピー数変化とは== | ||
| 115行目: | 106行目: | ||
== 参考文献 == | == 参考文献 == | ||
<references /> | <references /> | ||
<br> (執筆者:深井綾子、三宅紀子、松本直通 担当編集委員:加藤忠史) | |||
2012年6月11日 (月) 11:00時点における版
英語名:copy number variations: CNVs
同義語:コピー数変動
類義語:コピー数多型、コピー数変異
コピー数変化とは、染色体上の1kb以上にわたるゲノムDNAが、通常2コピーのところ、1コピー以下(欠失)、あるいは3コピー以上(重複)となっている現象をいう。ヒトゲノム中の12%にわたる領域に見いだされる。正常ゲノムに多型的に存在する場合や疾患ゲノムの原因として存在する場合などがある。
コピー数変化とは
従来、核型検査により検出されるヒトゲノムの異常として、染色体の欠失、重複、逆位、転座等が知られていた。ゲノムのコピー数変化・多型(copy number variations: CNVs)という概念は、2004年Iafrate AJとSebatらにより提唱された[1] [2]。常染色体上のゲノムDNAは通常1体細胞当たり2コピーであるが、個々人により1コピー以下しか存在しない領域(欠失)、もしくは3コピー以上存在する領域(重複)があり、病的であるもの、病的でないものを含め、それらをCNVと提唱した。さらに、SchererらはCNVを対照ゲノムと比較してコピー数が異なる1 kb以上のDNA断片と定義し、その中でも1%以上の人口で認めるコピー数変化をcopy number polymorphism (CNP)としている[3]。
同時期から、コピー数変化を検出できる様々な解析法が開発され、より高密度の解析が可能となり、当初予想された以上にヒトゲノムにはCNVが存在することが判明した。国際HapMapプロジェクトで用いられたヨーロッパ、アフリカ、アジアの異なる祖先をもつ3系統270名のリンパ芽球細胞(lymphoblastoid cell lines: LCLs)由来のDNAを使用してCNVの検証が行われた。Affymetrix GeneChip Human Mapping 500K early access array (500K EA)と、whole genome TilePath (WGTP) arrayの2種類を用いた検証の結果、合計1447か所のCNVsが検出された。そのゲノムサイズの合計は約360 Mb でヒトゲノムの約12%に相当した[4] [5]。
また、2010年のConradらは、41人の女性のLCLs由来のDNAを用いてNimbleGen arrayを用いた解析を行い、11,700か所のCNV (サイズの中央値:2.7 kb)を検出した[6]。 2006年RedonらによりヒトゲノムCNVカタログが作成され[7]、現在ではヒト、マウス、ラット、チンパンジー、アカゲザル、キイロショウジョウバエ等でも同様のCNVカタログが作成されている[8]。
CNVの形成メカニズム
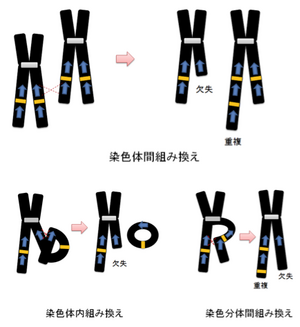
太青矢印はLCR/SDの位置と方向を、橙四角は遺伝子を示す。2本鎖DNA上に存在するLCR等の相同性の高い配列(青)間で異常な組み換え(赤点線)が起こり、相同配列間のゲノムが重複あるいは欠失する。
Wenli Gu et al 2008 より改変引用[9]

Wenli Gu et al 2008 より改変引用[9]
通常1Kb以上の長さで、90%以上の相同性を持つ配列はlow copy repeats (LCRs) またはsegmental duplications (SDs)と定義される[10]。このような配列はヒトハプロイドゲノムに3.6%存在するとされる[11]。特に10 kb以上の長さで97%以上の相同性を持つ場合LCRs領域では、ゲノム不安定性が高まり、組み換えが起こりやすくなるため欠失、重複、挿入、転座、逆位によるゲノム再構成 (genomic rearrangement) が生じやすい。これらのゲノム再編成を生じるメカニズムとして、生体内では主に以下の3つが考えられている[9]。
- NAHR (non-allelic homologous recombination) (図1)
- NHEJ (non-homologous end joining)[12] (図2)
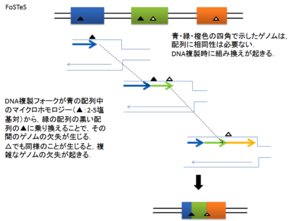
- FoSTeS (fork stalling and template switching) (図3)
NAHRは、高い相同性を持つ領域の間(同一染色体上、あるいは異なる染色体上)において、異常な相同組み換えが起きた結果、重複あるいは欠失が起きる場合である(図1)。
NEHJは、二本鎖DNA内の修復過程において、ゲノムの一部が欠失した状態で、両断端が連結された結果、ゲノム領域の一部が欠失する場合である(図2)。この場合、断端に相同領域の存在を要しない。
FoSTeSは、DNA複製途中で、DNA複製フォークが、微小な相同性(2-5塩基)を持つ離れた配列に乗り換えることによって、その間のゲノムが欠失する現象である(図3)。
NAHRは、異なった個体において独立に新たな欠失、重複が類似の構造を持つゲノム領域に生じうるのに対して、NHEJやFoSTeSでは、必ずしも特徴的なゲノム構造に依存するわけではないため、異なる個体で同様の欠失、重複が生じることはない(非常にまれ)という違いがある。
CNVの検出方法
一般的に用いるCNVの検出方法には、定量PCR法などの特定の遺伝子座(locus specific)を対象とする方法やアレイなどの全genome を対象とする方法がある[7]。
Array CGH

比較ゲノムハイブリダイゼーション法(Comparative Genomic Hybridization: CGH)は全ゲノムを対象にDNAコピー数変化を調べるための効率的な方法。
Agilent社ウェッブページより一部改変引用。
Array CGHとは、オリゴヌクレオチドあるいはプラズミドDNAにクローン化したヒトゲノムの一部(プローブ)をチップ上に配置したアレイを基盤とする。コピー数変化を調べたい検体DNAと対照とするDNAをそれぞれ異なる波長の蛍光色素で標識をする。標識した2つのDNAを同量混合させ、アレイ上のプローブと競合的にハイブリダイズさせる。プローブに結合した検体DNAと対照DNAの蛍光シグナルの強度の比から検体DNAと対照DNAコピー数の比を算出することができる。具体的には、両試料でコピー数が等しい場合は同等のシグナル強度を示し、片方の試料に染色体の欠失あるいは重複があれば異なったシグナル強度を示す。(図4)
SNP array

検体DNAを断片化した後に蛍光色素で標識し、熱変性条件下でチップと反応させる。
2004年頃Affymetrix社の一塩基多型(SNP)解析用arrayでコピー数解析ができるようになった。このアレイはreference genomeを必要とせず、疾患のゲノムだけで解析が可能である。アレイ上には対立遺伝子の25-merのプローブがあり、既知のSNPサイトに対して異なる塩基(例えばCもしくはT)を搭載している。相補的な配列を持つラベル化された検体DNAがプローブに結合する際、SNPサイトにミスマッチが存在すると結合しにくくなり、シグナルは弱くなる。最近のSNPアレイを用いるとgenotypeも同時に検出が可能で、更にアリルピーク(Genotype: 2 copyの場合 AA/AB/BB, 3 copyの場合 AAA/AAB/ABB/BBB, 1 copyの場合A/B)を見ることで情報性が付加されたコピー数変化としてとらえることが可能となり信頼性が増した。(図5)
定量PCR法
Quantitative real time polymerase chain reaction: qPCR
これは標的とするゲノム領域のCNVをスクリーニングするのに最も一般的に使用される方法の一つである。標的とする遺伝子(領域)に対してプライマーを設計し、蛍光色素をとりこませながらPCRで増幅させ、PCR産物の増幅効率を測定する。 テンプレートの量に応じて増幅効率が異なるのを利用して、DNAの定量(コピー数解析)が可能である。
MLPA法
Multiplex ligation-dependent probe amplification
この方法は標的とする遺伝子(領域)に対して特異的に結合するプローブを用い、標的ゲノムDNA上でligationを行い、ligationされたDNAを鋳型とし蛍光標識プライマーでPCR増幅させ、異なる長さの増幅断片を電気泳動解析により検出する。ピーク面積は標的遺伝子領域のコピー数を反映し重複や欠失を定量的に捉えることができる。
CNVと疾患関連性
欠失、重複などのゲノム再構成が起きる際、遺伝子そのものあるいは遺伝子発現に関与する領域を含む事がある。ヒトゲノムで一世代を経ることで点変異(SNP)は1.8~2.5x10-8、CNVは1.7x10-6~1.2x10-4の確率で起こるとされており、CNV発生はSNPのそれに比べて102~104倍高率であるため、遺伝子の点変異よりもCNVが原因となる遺伝性疾患が多いと推定される[13] 。CNVはいくつかの分子メカニズム(表1)を介して生じ、病気を惹起する場合もある。病的・非病的CNVがあり、例えばCNVが①罹患の血縁者に存在するか既報の疾患関連CNVに一致する、②疾患責任遺伝子や多数の遺伝子を含むか遺伝子発現領域を含む、③サイズが3Mb以上である、④増幅が3コピー以上である、などの場合、病的CNVである可能性が高いと考えられている[14]。
CNVの検索方法
正常人に認めるCNVsはToronto Database of Genomic VariantsやHuman Structural Variation Database に登録・一般公開されている。また、臨床学的な情報を含む染色体異常はDECIPHER (Database of Chromosomal Imbalance and Phenotype in Humans Using Ensembl Resources)、ECARUCA (European Cytogenetics Association Reigister of Unbalanced Chromosome Aberrations)、ISCA(The International Standards for Cytogenomic Arrays (ISCA) Consortium)などで検索可能である。
表1. CNVと疾患関連性の代表例と分子メカニズム[15] [16]
| 例[15] | 疾患の例[15] [16] | ||
| ① 量依存的遺伝子(dose-sensitive gene) | 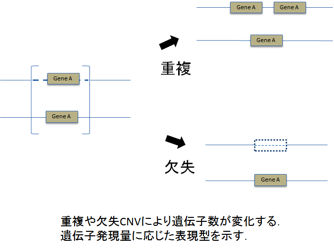
|
Charcot-Marie-Tooth病(CMT病1A型) | Chr 17 p11上のPMP22を含む約1.4 Kbの領域の重複 |
| 遺伝性圧脆弱性ニューロパチー(NHPP) | Chr 17 p11上のPMP22を含む約1.4 Kbの領域の欠失 | ||
| ② 位置効果(position effect) | 
|
Blepharophimosis症候群 | 責任遺伝子forkhead box protein L2(FOXL2)の230kb上流にある転写因子の結合部位がCNVにより欠失すると発症する。 |
| ③ 劣性アリルの顕在化(unmasking of recessive allele) | 
|
片方のアレルに劣性変異が存在する個体で、野生型アレルが欠失した場合、劣性変異が顕在化する。 | |
| ④ 遺伝子破壊/遺伝子融合(Gene interruption/Gene fusion) | 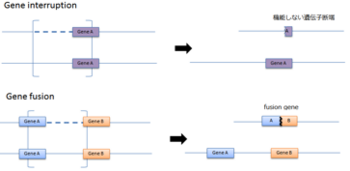
|
融合遺伝子:急性骨髄性白血病(AML)や骨髄異形成症候群(MDS) | CNV配列上に遺伝子が存在した場合遺伝子の破壊や融合遺伝子形成が起こる。 |
参考文献
- ↑
Sebat, J., Lakshmi, B., Troge, J., Alexander, J., Young, J., Lundin, P., ..., & Wigler, M. (2004).
Large-scale copy number polymorphism in the human genome. Science (New York, N.Y.), 305(5683), 525-8. [PubMed:15273396] [WorldCat] [DOI] - ↑
Bruno, D.L., Ganesamoorthy, D., Schoumans, J., Bankier, A., Coman, D., Delatycki, M., ..., & Slater, H.R. (2009).
Detection of cryptic pathogenic copy number variations and constitutional loss of heterozygosity using high resolution SNP microarray analysis in 117 patients referred for cytogenetic analysis and impact on clinical practice. Journal of medical genetics, 46(2), 123-31. [PubMed:19015223] [WorldCat] [DOI] - ↑
McCarroll, S.A., & Altshuler, D.M. (2007).
Copy-number variation and association studies of human disease. Nature genetics, 39(7 Suppl), S37-42. [PubMed:17597780] [WorldCat] [DOI] - ↑
Redon, R., Ishikawa, S., Fitch, K.R., Feuk, L., Perry, G.H., Andrews, T.D., ..., & Hurles, M.E. (2006).
Global variation in copy number in the human genome. Nature, 444(7118), 444-54. [PubMed:17122850] [PMC] [WorldCat] [DOI] - ↑
Feuk, L., Carson, A.R., & Scherer, S.W. (2006).
Structural variation in the human genome. Nature reviews. Genetics, 7(2), 85-97. [PubMed:16418744] [WorldCat] [DOI] - ↑
Conrad, D.F., Pinto, D., Redon, R., Feuk, L., Gokcumen, O., Zhang, Y., ..., & Hurles, M.E. (2010).
Origins and functional impact of copy number variation in the human genome. Nature, 464(7289), 704-12. [PubMed:19812545] [PMC] [WorldCat] [DOI] - ↑ 7.0 7.1
Fanciulli, M., Petretto, E., & Aitman, T.J. (2010).
Gene copy number variation and common human disease. Clinical genetics, 77(3), 201-13. [PubMed:20002459] [WorldCat] [DOI] - ↑
Henrichsen, C.N., Chaignat, E., & Reymond, A. (2009).
Copy number variants, diseases and gene expression. Human molecular genetics, 18(R1), R1-8. [PubMed:19297395] [WorldCat] [DOI] - ↑ 9.0 9.1 9.2
Gu, W., Zhang, F., & Lupski, J.R. (2008).
Mechanisms for human genomic rearrangements. PathoGenetics, 1(1), 4. [PubMed:19014668] [PMC] [WorldCat] [DOI] - ↑
Stankiewicz, P., & Lupski, J.R. (2010).
Structural variation in the human genome and its role in disease. Annual review of medicine, 61, 437-55. [PubMed:20059347] [WorldCat] [DOI] - ↑
Bailey, J.A., Yavor, A.M., Massa, H.F., Trask, B.J., & Eichler, E.E. (2001).
Segmental duplications: organization and impact within the current human genome project assembly. Genome research, 11(6), 1005-17. [PubMed:11381028] [PMC] [WorldCat] [DOI] - ↑
Symington, L.S., & Gautier, J. (2011).
Double-strand break end resection and repair pathway choice. Annual review of genetics, 45, 247-71. [PubMed:21910633] [WorldCat] [DOI] - ↑
Lupski, J.R. (2007).
Genomic rearrangements and sporadic disease. Nature genetics, 39(7 Suppl), S43-7. [PubMed:17597781] [WorldCat] [DOI] - ↑
Lee, C., Iafrate, A.J., & Brothman, A.R. (2007).
Copy number variations and clinical cytogenetic diagnosis of constitutional disorders. Nature genetics, 39(7 Suppl), S48-54. [PubMed:17597782] [WorldCat] [DOI] - ↑ 15.0 15.1 15.2
Lee, J.A., Carvalho, C.M., & Lupski, J.R. (2007).
A DNA replication mechanism for generating nonrecurrent rearrangements associated with genomic disorders. Cell, 131(7), 1235-47. [PubMed:18160035] [WorldCat] [DOI] - ↑ 16.0 16.1
Lupski, J.R., & Stankiewicz, P. (2005).
Genomic disorders: molecular mechanisms for rearrangements and conveyed phenotypes. PLoS genetics, 1(6), e49. [PubMed:16444292] [PMC] [WorldCat] [DOI]
(執筆者:深井綾子、三宅紀子、松本直通 担当編集委員:加藤忠史)