「ディファレンシャルディスプレイ」の版間の差分
細編集の要約なし |
Ykishimoto (トーク | 投稿記録) 細編集の要約なし |
||
| (3人の利用者による、間の40版が非表示) | |||
| 1行目: | 1行目: | ||
英:differential display 英略語:DDRT-PCR, DD-PCR 仏:l'affichage différentiel | 英:differential display 英略語:DDRT-PCR, DD-PCR 仏:l'affichage différentiel | ||
ディファレンシャルディスプレイ(別名、"ディファレンシャルディスプレイRT-PCR (DDRT-PCR, DD-PCR)")とは、[[wikipedia:ja:遺伝子|遺伝子]][[wikipedia:ja:発現|発現]]の量の差異を[[wikipedia:ja:ポリメラーゼ連鎖反応|PCR]]法により解析する方法の一種である。ここで、ディスプレイ(display)とは、目的遺伝子を[[wikipedia:ja:ゲル|ゲル]]上で視覚的に表示するとの意であり、多数のRNAにおいて、様々な発現パターンを示す[[wikipedia:ja:mRNA|mRNA]]を包括的に同定しその発現量を比較できることから、[[wikipedia:ja:ゲノム|ゲノム]]科学における遺伝子発現解析 に有力な手法である<ref name=Liang_Pardee_Science><pubmed>1354393</pubmed></ref><ref name=Liang_Biotech><pubmed>12188186</pubmed></ref><ref>'''野島博'''<br>最新生命科学キーワードブック<br>羊土社: 2007</ref>。 | |||
== 原理と適用例 == | == 原理と適用例 == | ||
[[ | 遺伝子から転写された[[wikipedia:ja:mRNA|mRNA]]の量は、遺伝子機能の量的な面を反映していると考えられるため、遺伝子の発現量から機能を推測しようというのが、この遺伝子発現解析の基本的な概念である。本技法は、1992年に、LiangとPardeeにより、[[wikipedia:ja:mRNA|mRNA]]レベルで[[wikipedia:ja:真核生物|真核細胞]]の試料間で、遺伝子発現の同定比較を行うことできる技術として発表された<ref name=Liang_Pardee_Science></ref><ref name=Liang_Biotech></ref>。形態的、遺伝学的、または実験的処置等に異なる試料間で、どの遺伝子発現に変化・差異があるかを見出すことができる。 | ||
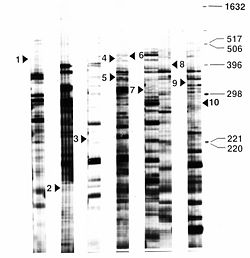
例えば、物質Aの添加により遺伝子Bの発現の増加/減少が見出された場合、この遺伝子Bの発現が物質Aにより誘導/抑制されていると考えらる。防御蛋白質や阻害作用の解析などに多くの適用が考えられる。脳神経科学領域では、ある[[学習]]課題が成立した動物個体としていない動物個体(脊椎動物および無脊椎動物)の細胞間で、発現に差のある遺伝子を同定することで、その学習に関与する遺伝子群が同定された適応例がある<ref><pubmed>10531455</pubmed></ref> <ref><pubmed>11168596</pubmed></ref>。図1に、Nakayaら9)が、ナメクジの臭い忌避学習で特異的に発現が増大する遺伝子(LAPS18)を同定した実験のDDの結果を示す <ref><pubmed>11168596</pubmed></ref>。比較的感度が良く、実験操作も簡便に行うことができ、また必要に応じて、3対以上の試料間での比較に拡張することも可能である。[[Image:DD-fig1.jpg|thumb|250px|図1 ディファレンシャルディスプレイ法の適応例]] | |||
原法では、検出方法としてRIを用いていた(RI-DD法)が<ref name=Liang_Pardee_Science></ref>、その後、蛍光標識された[[wikipedia:ja:プライマー (生物)|プライマー]]を用いて検出する[[wikipedia:ja:蛍光「|蛍光]]ディファレンシャルディスプレイ (FDD) 法」が開発され、検出感度、操作性や再現性が高められた<ref name=Liang_Biotech></ref><ref>'''伊藤隆司'''<br>Differential Display<br>実験医学別冊 新遺伝子工学ハンドブック改訂第4版(村松正實、 山本雅編)羊土社: 2003, pp. 60-64 (なお、第5版から本項目は削除されている)</ref>。 | |||
== 利点と不利点 == | == 利点と不利点 == | ||
本法は、単一ゲル上で多数の試料のバンド分布を同時に比較できることから効率良く遺伝子発現量の差が検出できるため、組織や細胞が発現する遺伝子とその発現量の解析、つまりtranscription imageの研究分野に急速に普及した。ただし、任意プライマーを用いてPCR反応を行うことから再現性が高いとは言えず、また、高感度であるが故にPCRの条件設定を厳密に行わないと擬陽性のバンドも出やすい。そのため、2000年代半ばには、高速かつ高感度で大量解析可能な[[DNAアレイ]](DNAチップ)、RNA-seq、定量的[[ | 本法は、単一ゲル上で多数の試料のバンド分布を同時に比較できることから効率良く遺伝子発現量の差が検出できるため、組織や細胞が発現する遺伝子とその発現量の解析、つまりtranscription imageの研究分野に急速に普及した。ただし、任意プライマーを用いてPCR反応を行うことから再現性が高いとは言えず、また、高感度であるが故にPCRの条件設定を厳密に行わないと擬陽性のバンドも出やすい。そのため、2000年代半ばには、高速かつ高感度で大量解析可能な[[DNAアレイ]](DNAチップ)、RNA-seq、定量的[[wikipedia:ja:リアルタイムPCR|リアルタイムPCR]]リアルタイムPCR(RT-PCR)などの技術の登場によってその相対的重要性は低下している<ref>英語版WikipediaのDifferential displayの項目。http://en.wikipedia.org/wiki/Differential_display</ref>。しかしながら、DNAアレイに比較して、ディファレンシャルディスプレイ法はコスト面、所要時間で未だ有利であり、出発材料が少量でも解析を行えるという利点も有する。また通常のDNAアレイと異なり、ディファレンシャルディスプレイ法では新規遺伝子の検出が可能であることなどの利点もあることから、今後も多くの分野で適用が期待される。 | ||
なお、蛋白質レベルにおける発現量を比較する「蛋白質ディファレンシャルディスプレイ法」という手法もある<ref>'''平野久''' <br>タンパク質のアミノ酸配列と翻訳後修飾の分析<br>蛋白質をみるー構造と挙動(長谷俊治、高尾敏文、高木淳一編)化学同人: 2009 pp. 1-32</ref>。これは、複数種の特定の細胞試料間で蛋白質の発現パターンの違いを分析する方法であり、一般的には二次元[[wikipedia:Gel_electrophoresis|ゲル電気泳動]]で各細胞の蛋白質を分離し、そのパターンの差を比較する方法である。 | |||
== 方法と手順 == | == 方法と手順 == | ||
以下、通常のmRNA試料を対象としたディファレンシャルディスプレイ法について説明する (図2) | 以下、通常のmRNA試料を対象としたディファレンシャルディスプレイ法について説明する (図2)。[[Image:Differentialdisplay2.jpg|thumb|400px|図2 ディファレンシャルディスプレイ法の手順]] | ||
細胞中で発現したmRNAすべてを[[ | 細胞中で発現したmRNAすべてを[[wikipedia:ja:逆転写|逆転写]]しcDNAを得た後、これを鋳型にして複数の混合プライマーを用いたPCRを行う。ここで、特異性を持たせずできるだけ多くの種類のcDNA断片を増幅するよう設定した複数のプライマーを用いることにより、非常に多数のバンドが得られる。1つの細胞で発現し得る約15,000種類の遺伝子すべての発現を網羅して探索するために、4種類のアンカープライマーと20種類の混成プライマー(10 mer)を用いてPCRを行う。得られたバンドの強弱を遺伝子発現の強度の指標とし、特定の物質添加の有無などさまざまな操作・環境の違いによりその細胞のどの遺伝子が変化しているかを見出す。 | ||
#アンカープライマーによるmRNAの選択的逆転写(第1鎖合成) | #アンカープライマーによるmRNAの選択的逆転写(第1鎖合成) | ||
#: | #:比較したい2種類(またはそれ以上)の細胞からそれぞれ抽出したmRNAを、ポリ(A)鎖と結合するアンカープライマーと逆転写酵素を用いた逆転写反応によってcDNAに変換する。アンカープライマーとしはT(チミジン)12個からなる[[wikipedia:ja:オリゴヌクレオチド|オリゴヌクレオチド]]の3’端に1個の任意の塩基を付加したものを用いる。 | ||
# | #任意プライマーによるmRNAの選択的第2鎖合成とPCRによる増幅 | ||
#:次に、合成されたcDNAを鋳型に、アンカープライマーと任意の混成塩基配列を持つ(通常10merの)オリゴヌクレオチド群をプライマーとしてPCRを行う。これによって任意の共通塩基配列をもつ複数のcDNA断片を、同時にPCR増幅する。この結果、両端をアンカープライマーと任意プライマーの配列で置換された分子が生じる。 | #:次に、合成されたcDNAを鋳型に、アンカープライマーと任意の混成塩基配列を持つ(通常10merの)オリゴヌクレオチド群をプライマーとしてPCRを行う。これによって任意の共通塩基配列をもつ複数のcDNA断片を、同時にPCR増幅する。この結果、両端をアンカープライマーと任意プライマーの配列で置換された分子が生じる。 | ||
#電気泳動(もしくは蛍光イメージアナライザー等)による発現の分類 | #電気泳動(もしくは蛍光イメージアナライザー等)による発現の分類 | ||
#:反応生成物をポリアクリルアミド電気泳動で展開すると、複数のcDNA断片からなるフィンガープリントが得られる。この中から、強度に変化のある、または特異的な泳動パターンを示すバンドの切り出しを行う。 | #:反応生成物をポリアクリルアミド電気泳動で展開すると、複数のcDNA断片からなるフィンガープリントが得られる。この中から、強度に変化のある、または特異的な泳動パターンを示すバンドの切り出しを行う。 | ||
#バンドの再増幅およびクローン化 | #バンドの再増幅およびクローン化 | ||
#:ゲルからサンプルを回収し、それを鋳型としてPCRを繰り返し行い増幅し、cDNA[[ | #:ゲルからサンプルを回収し、それを鋳型としてPCRを繰り返し行い増幅し、cDNA[[wikipedia:ja:クローン|クローン]]を回収する。 | ||
== 参考文献 == | == 参考文献 == | ||
| 48行目: | 36行目: | ||
<references /> | <references /> | ||
(執筆者:岸本泰司、中矢 正、桐野 豊、担当編集委員:林 康紀) | |||
<keywords content="ディファレンシャルディスプレイ,ディファレンシャルディスプレー,differential display,遺伝子発現,gene expression" /> | <keywords content="ディファレンシャルディスプレイ,ディファレンシャルディスプレー,differential display,遺伝子発現,gene expression" /> | ||
2011年12月30日 (金) 11:19時点における版
英:differential display 英略語:DDRT-PCR, DD-PCR 仏:l'affichage différentiel
ディファレンシャルディスプレイ(別名、"ディファレンシャルディスプレイRT-PCR (DDRT-PCR, DD-PCR)")とは、遺伝子発現の量の差異をPCR法により解析する方法の一種である。ここで、ディスプレイ(display)とは、目的遺伝子をゲル上で視覚的に表示するとの意であり、多数のRNAにおいて、様々な発現パターンを示すmRNAを包括的に同定しその発現量を比較できることから、ゲノム科学における遺伝子発現解析 に有力な手法である[1][2][3]。
原理と適用例
遺伝子から転写されたmRNAの量は、遺伝子機能の量的な面を反映していると考えられるため、遺伝子の発現量から機能を推測しようというのが、この遺伝子発現解析の基本的な概念である。本技法は、1992年に、LiangとPardeeにより、mRNAレベルで真核細胞の試料間で、遺伝子発現の同定比較を行うことできる技術として発表された[1][2]。形態的、遺伝学的、または実験的処置等に異なる試料間で、どの遺伝子発現に変化・差異があるかを見出すことができる。
例えば、物質Aの添加により遺伝子Bの発現の増加/減少が見出された場合、この遺伝子Bの発現が物質Aにより誘導/抑制されていると考えらる。防御蛋白質や阻害作用の解析などに多くの適用が考えられる。脳神経科学領域では、ある学習課題が成立した動物個体としていない動物個体(脊椎動物および無脊椎動物)の細胞間で、発現に差のある遺伝子を同定することで、その学習に関与する遺伝子群が同定された適応例がある[4] [5]。図1に、Nakayaら9)が、ナメクジの臭い忌避学習で特異的に発現が増大する遺伝子(LAPS18)を同定した実験のDDの結果を示す [6]。比較的感度が良く、実験操作も簡便に行うことができ、また必要に応じて、3対以上の試料間での比較に拡張することも可能である。
原法では、検出方法としてRIを用いていた(RI-DD法)が[1]、その後、蛍光標識されたプライマーを用いて検出する蛍光ディファレンシャルディスプレイ (FDD) 法」が開発され、検出感度、操作性や再現性が高められた[2][7]。
利点と不利点
本法は、単一ゲル上で多数の試料のバンド分布を同時に比較できることから効率良く遺伝子発現量の差が検出できるため、組織や細胞が発現する遺伝子とその発現量の解析、つまりtranscription imageの研究分野に急速に普及した。ただし、任意プライマーを用いてPCR反応を行うことから再現性が高いとは言えず、また、高感度であるが故にPCRの条件設定を厳密に行わないと擬陽性のバンドも出やすい。そのため、2000年代半ばには、高速かつ高感度で大量解析可能なDNAアレイ(DNAチップ)、RNA-seq、定量的リアルタイムPCRリアルタイムPCR(RT-PCR)などの技術の登場によってその相対的重要性は低下している[8]。しかしながら、DNAアレイに比較して、ディファレンシャルディスプレイ法はコスト面、所要時間で未だ有利であり、出発材料が少量でも解析を行えるという利点も有する。また通常のDNAアレイと異なり、ディファレンシャルディスプレイ法では新規遺伝子の検出が可能であることなどの利点もあることから、今後も多くの分野で適用が期待される。
なお、蛋白質レベルにおける発現量を比較する「蛋白質ディファレンシャルディスプレイ法」という手法もある[9]。これは、複数種の特定の細胞試料間で蛋白質の発現パターンの違いを分析する方法であり、一般的には二次元ゲル電気泳動で各細胞の蛋白質を分離し、そのパターンの差を比較する方法である。
方法と手順
以下、通常のmRNA試料を対象としたディファレンシャルディスプレイ法について説明する (図2)。

細胞中で発現したmRNAすべてを逆転写しcDNAを得た後、これを鋳型にして複数の混合プライマーを用いたPCRを行う。ここで、特異性を持たせずできるだけ多くの種類のcDNA断片を増幅するよう設定した複数のプライマーを用いることにより、非常に多数のバンドが得られる。1つの細胞で発現し得る約15,000種類の遺伝子すべての発現を網羅して探索するために、4種類のアンカープライマーと20種類の混成プライマー(10 mer)を用いてPCRを行う。得られたバンドの強弱を遺伝子発現の強度の指標とし、特定の物質添加の有無などさまざまな操作・環境の違いによりその細胞のどの遺伝子が変化しているかを見出す。
- アンカープライマーによるmRNAの選択的逆転写(第1鎖合成)
- 比較したい2種類(またはそれ以上)の細胞からそれぞれ抽出したmRNAを、ポリ(A)鎖と結合するアンカープライマーと逆転写酵素を用いた逆転写反応によってcDNAに変換する。アンカープライマーとしはT(チミジン)12個からなるオリゴヌクレオチドの3’端に1個の任意の塩基を付加したものを用いる。
- 任意プライマーによるmRNAの選択的第2鎖合成とPCRによる増幅
- 次に、合成されたcDNAを鋳型に、アンカープライマーと任意の混成塩基配列を持つ(通常10merの)オリゴヌクレオチド群をプライマーとしてPCRを行う。これによって任意の共通塩基配列をもつ複数のcDNA断片を、同時にPCR増幅する。この結果、両端をアンカープライマーと任意プライマーの配列で置換された分子が生じる。
- 電気泳動(もしくは蛍光イメージアナライザー等)による発現の分類
- 反応生成物をポリアクリルアミド電気泳動で展開すると、複数のcDNA断片からなるフィンガープリントが得られる。この中から、強度に変化のある、または特異的な泳動パターンを示すバンドの切り出しを行う。
- バンドの再増幅およびクローン化
- ゲルからサンプルを回収し、それを鋳型としてPCRを繰り返し行い増幅し、cDNAクローンを回収する。
参考文献
- ↑ 1.0 1.1 1.2
Liang, P., & Pardee, A.B. (1992).
Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction. Science (New York, N.Y.), 257(5072), 967-71. [PubMed:1354393] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2
Liang, P. (2002).
A decade of differential display. BioTechniques, 33(2), 338-44, 346. [PubMed:12188186] [WorldCat] [DOI] - ↑ 野島博
最新生命科学キーワードブック
羊土社: 2007 - ↑
Gomi, H., Sun, W., Finch, C.E., Itohara, S., Yoshimi, K., & Thompson, R.F. (1999).
Learning induces a CDC2-related protein kinase, KKIAMRE. The Journal of neuroscience : the official journal of the Society for Neuroscience, 19(21), 9530-7. [PubMed:10531455] [PMC] [WorldCat] - ↑
Nakaya, T., Kawahara, S., Watanabe, S., Lee, D., Suzuki, T., & Kirino, Y. (2001).
Identification and expression of a novel gene in odour-taste associative learning in the terrestrial slug. Genes to cells : devoted to molecular & cellular mechanisms, 6(1), 43-56. [PubMed:11168596] [WorldCat] - ↑
Nakaya, T., Kawahara, S., Watanabe, S., Lee, D., Suzuki, T., & Kirino, Y. (2001).
Identification and expression of a novel gene in odour-taste associative learning in the terrestrial slug. Genes to cells : devoted to molecular & cellular mechanisms, 6(1), 43-56. [PubMed:11168596] [WorldCat] - ↑ 伊藤隆司
Differential Display
実験医学別冊 新遺伝子工学ハンドブック改訂第4版(村松正實、 山本雅編)羊土社: 2003, pp. 60-64 (なお、第5版から本項目は削除されている) - ↑ 英語版WikipediaのDifferential displayの項目。http://en.wikipedia.org/wiki/Differential_display
- ↑ 平野久
タンパク質のアミノ酸配列と翻訳後修飾の分析
蛋白質をみるー構造と挙動(長谷俊治、高尾敏文、高木淳一編)化学同人: 2009 pp. 1-32
(執筆者:岸本泰司、中矢 正、桐野 豊、担当編集委員:林 康紀)