「機能欠失実験」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| (3人の利用者による、間の55版が非表示) | |||
| 1行目: | 1行目: | ||
<br> 英:loss of function | |||
ある遺伝子の機能を調べる際にその遺伝子の発現量や分子機能を減弱させることで機能を類推する実験手法。逆に遺伝子の機能や発現量を増強させる実験は[[機能獲得実験]]と呼ばれる。 | |||
== 機能欠失実験とは == | == 機能欠失実験とは == | ||
ある遺伝子の生理機能を解析する際、動物個体や細胞でその遺伝子の機能を減弱させて得られた変化を基に機能を類推する実験手法を機能欠失実験という。 機能欠失実験には発現量の減少または分子機能を失わせる方法がある。 | |||
<br> | |||
== | == 機能欠失実験の手法 == | ||
== | == 発現量の減少 == | ||
=== 遺伝子破壊(遺伝子ノックアウト) === | |||
目的とする遺伝子そのものを破壊することで目的遺伝子の発現あるいは目的遺伝子の機能を完全に抑制させる。 | |||
==== ジーンターゲティングによる遺伝子ノックアウト ==== | |||
目的の遺伝子に任意の変異を導入する手法をジーンターゲティングという。本法による遺伝子破壊を動物個体レベルで行うためにはES細胞が必要であったためマウスでのみ可能であったが近年ラットでもES細胞が樹立されジーンターゲティングによる遺伝子ノックアウトが可能になった。また、ES細胞の代わりにiPS細胞を用いたジーンターゲティングも行われるようになった。 | |||
==== | ==== 人工ヌクレアーゼによる遺伝子ノックアウト ==== | ||
[[Image: | 人工ヌクレアーゼは、任意の塩基配列に結合するようにデザインされたDNA結合ドメインとDNA切断酵素の切断ドメインを連結させたタンパク質であり、任意の塩基配列を切断すること可能な酵素である(図1)。 [[Image:Takahirohirabayashi fig 1.jpg|thumb|right|300px|図1 人工ヌクレアーゼの構造]] この人工ヌクレアーゼにはDNA配列を認識し、切断するという原理は共通だが、ジンクフィンガーのDNA結合ドメインを利用してDNA配列を認識するZinc Finger nuclease (ZFN), TALEsのDNA結合ドメインを利用しDNA配列を認識するTALENの2種類が主に使用されている。これら人工ヌクレアーゼを導入した細胞内では特定のDNAがdouble-strand breakするが、これを修復するためにNHEJ (Non-Homologous End Joining)機構が働く。この際、高頻度で塩基対の欠失、挿入などの修復エラーが生じ、結果的にフレームシフトを起こすことで遺伝子がノックアウトされる(図2)。[[Image:Takahirohirabayashi_fig_2.jpg|thumb|right|300px|図2 人工ヌクレアーゼによる遺伝子破壊]]この手法はES細胞を必要としないため、これまでES細胞が樹立されておらずジーンターゲティングによる遺伝子ノックアウトが不可能であった動物種でも使用例が報告されている。 | ||
<br> | |||
=== | === 遺伝子ノックダウン === | ||
特定の遺伝子の発現量を減少させるが遺伝子ノックアウトとは異なり、完全に発現が失われるわけではないので、遺伝子ノックアウトでは動物個体が目的の時期より以前に死に至るため解析が困難な場合などに有効な手法である。 | 特定の遺伝子の発現量を減少させるが遺伝子ノックアウトとは異なり、完全に発現が失われるわけではないので、遺伝子ノックアウトでは動物個体が目的の時期より以前に死に至るため解析が困難な場合などに有効な手法である。 | ||
| 47行目: | 34行目: | ||
==== ジーンターゲティングによる遺伝子発現量減少 ==== | ==== ジーンターゲティングによる遺伝子発現量減少 ==== | ||
ジーンターゲティングによる遺伝子ノックアウトが遺伝子中の翻訳領域を破壊するのに対し、遺伝子ノックダウンでは遺伝子中のpolyA付加配列など発現制御領域を欠損、あるいは変異を導入することでおこなわれる。本法も動物個体レベルで行うためにはES細胞が必要であるため、適用できる動物種に限りがある。 | |||
==== | ==== アンチセンスオリゴヌクレオチドによる遺伝子発現量減少 ==== | ||
標的とする遺伝子のmRNA配列に対し相補的な塩基配列を持つ短い核酸(アンチセンスオリゴヌクレオチド)を細胞外から導入すると、目的のmRNAと結合しその翻訳を阻害しその発現を抑制する。 このアンチセンスオリゴヌクレオチドは細胞内で不安定であるためリン酸基の酸素の一つがチオール(-SH)化(-SH)したホスホロチオエート結合オリゴ (s-oligo)や、RNAオリゴヌクレオチドのリボースをモルフォリノ環、リン酸基をホスホロジアミダイトへ、ウラシルをチミンにそれぞれ置換し、安定性の向上、細胞毒性の軽減させたモルフォリノアンチセンスオリゴなどが開発されてきた。しかし、現在では遺伝子発現抑制効果野高い2本鎖RNAによるRNA干渉を利用した方法に移行している。 | |||
==== RNA干渉==== | ==== RNA干渉(RNA interference, RNAi)を利用した遺伝子発現量減少 ==== | ||
RNA干渉とは21- | RNA干渉とは21-23塩基からなる対からなるsiRNA(short interfering RNA)と呼ばれる2本鎖RNAが相補的な配列を持つ生体内のmRNAと結合し、そのmRNAを分解する現象である。 この現象はウイルスなどに対する生体防御機構と考えられているが、近年ではこの現象を利用して特定の遺伝子発現の抑制を誘導する実験系が種々の動植物で確立され、さらにこれらを応用した疾患に対する治療法が開発されている。siRNAそのものを細胞内へ導入し、遺伝子発現抑制を誘導するためには大量のsiRNAが必要であることやその効果が一過性であることから、近年ではこのsiRNAを発現するベクターとして導入する方法が多く用いられている。 | ||
<br> | |||
== 機能の減弱 == | == 機能の減弱 == | ||
遺伝子変異 機能ドメインに変異 | |||
目的のタンパク質が持つアミノ酸がリン酸化されることでその機能が増強する場合、そのアミノ酸をリン酸化を受けない他のアミノ酸に置換した変異体を発現することでリン酸化による機能の増強を抑制することができる。 一般的にはセリン, スレオニンはアラニン、チロシンはフェニルアラニン、のように類似構造のアミノ酸に置換する。 | |||
2012年12月14日 (金) 19:38時点における版
英:loss of function
ある遺伝子の機能を調べる際にその遺伝子の発現量や分子機能を減弱させることで機能を類推する実験手法。逆に遺伝子の機能や発現量を増強させる実験は機能獲得実験と呼ばれる。
機能欠失実験とは
ある遺伝子の生理機能を解析する際、動物個体や細胞でその遺伝子の機能を減弱させて得られた変化を基に機能を類推する実験手法を機能欠失実験という。 機能欠失実験には発現量の減少または分子機能を失わせる方法がある。
機能欠失実験の手法
発現量の減少
遺伝子破壊(遺伝子ノックアウト)
目的とする遺伝子そのものを破壊することで目的遺伝子の発現あるいは目的遺伝子の機能を完全に抑制させる。
ジーンターゲティングによる遺伝子ノックアウト
目的の遺伝子に任意の変異を導入する手法をジーンターゲティングという。本法による遺伝子破壊を動物個体レベルで行うためにはES細胞が必要であったためマウスでのみ可能であったが近年ラットでもES細胞が樹立されジーンターゲティングによる遺伝子ノックアウトが可能になった。また、ES細胞の代わりにiPS細胞を用いたジーンターゲティングも行われるようになった。
人工ヌクレアーゼによる遺伝子ノックアウト
人工ヌクレアーゼは、任意の塩基配列に結合するようにデザインされたDNA結合ドメインとDNA切断酵素の切断ドメインを連結させたタンパク質であり、任意の塩基配列を切断すること可能な酵素である(図1)。
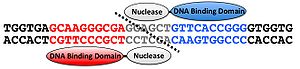
この人工ヌクレアーゼにはDNA配列を認識し、切断するという原理は共通だが、ジンクフィンガーのDNA結合ドメインを利用してDNA配列を認識するZinc Finger nuclease (ZFN), TALEsのDNA結合ドメインを利用しDNA配列を認識するTALENの2種類が主に使用されている。これら人工ヌクレアーゼを導入した細胞内では特定のDNAがdouble-strand breakするが、これを修復するためにNHEJ (Non-Homologous End Joining)機構が働く。この際、高頻度で塩基対の欠失、挿入などの修復エラーが生じ、結果的にフレームシフトを起こすことで遺伝子がノックアウトされる(図2)。
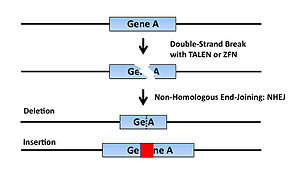
この手法はES細胞を必要としないため、これまでES細胞が樹立されておらずジーンターゲティングによる遺伝子ノックアウトが不可能であった動物種でも使用例が報告されている。
遺伝子ノックダウン
特定の遺伝子の発現量を減少させるが遺伝子ノックアウトとは異なり、完全に発現が失われるわけではないので、遺伝子ノックアウトでは動物個体が目的の時期より以前に死に至るため解析が困難な場合などに有効な手法である。
ジーンターゲティングによる遺伝子発現量減少
ジーンターゲティングによる遺伝子ノックアウトが遺伝子中の翻訳領域を破壊するのに対し、遺伝子ノックダウンでは遺伝子中のpolyA付加配列など発現制御領域を欠損、あるいは変異を導入することでおこなわれる。本法も動物個体レベルで行うためにはES細胞が必要であるため、適用できる動物種に限りがある。
アンチセンスオリゴヌクレオチドによる遺伝子発現量減少
標的とする遺伝子のmRNA配列に対し相補的な塩基配列を持つ短い核酸(アンチセンスオリゴヌクレオチド)を細胞外から導入すると、目的のmRNAと結合しその翻訳を阻害しその発現を抑制する。 このアンチセンスオリゴヌクレオチドは細胞内で不安定であるためリン酸基の酸素の一つがチオール(-SH)化(-SH)したホスホロチオエート結合オリゴ (s-oligo)や、RNAオリゴヌクレオチドのリボースをモルフォリノ環、リン酸基をホスホロジアミダイトへ、ウラシルをチミンにそれぞれ置換し、安定性の向上、細胞毒性の軽減させたモルフォリノアンチセンスオリゴなどが開発されてきた。しかし、現在では遺伝子発現抑制効果野高い2本鎖RNAによるRNA干渉を利用した方法に移行している。
RNA干渉(RNA interference, RNAi)を利用した遺伝子発現量減少
RNA干渉とは21-23塩基からなる対からなるsiRNA(short interfering RNA)と呼ばれる2本鎖RNAが相補的な配列を持つ生体内のmRNAと結合し、そのmRNAを分解する現象である。 この現象はウイルスなどに対する生体防御機構と考えられているが、近年ではこの現象を利用して特定の遺伝子発現の抑制を誘導する実験系が種々の動植物で確立され、さらにこれらを応用した疾患に対する治療法が開発されている。siRNAそのものを細胞内へ導入し、遺伝子発現抑制を誘導するためには大量のsiRNAが必要であることやその効果が一過性であることから、近年ではこのsiRNAを発現するベクターとして導入する方法が多く用いられている。
機能の減弱
遺伝子変異 機能ドメインに変異
目的のタンパク質が持つアミノ酸がリン酸化されることでその機能が増強する場合、そのアミノ酸をリン酸化を受けない他のアミノ酸に置換した変異体を発現することでリン酸化による機能の増強を抑制することができる。 一般的にはセリン, スレオニンはアラニン、チロシンはフェニルアラニン、のように類似構造のアミノ酸に置換する。