「自己組織化マップ」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| (同じ利用者による、間の17版が非表示) | |||
| 1行目: | 1行目: | ||
古川 徹生 九州工業大学 | |||
九州工業大学 | |||
{{box|text= 自己組織化マップはT. | 英語 Self-Organizing Map, Kohonen map <br> | ||
略称 SOM<br> | |||
同義語 自己組織化写像<br> | |||
{{box|text= 自己組織化マップはT. Kohonenによって提案された教師なしニューラルネットの一種である。自己組織化マップはもともと大脳の機能地図の自己組織化現象を説明する数理モデルに由来する。しかし実データ解析へ応用するため、ニューロン間の結合やダイナミクスが簡約化され、計算の効率化が図られている。自己組織化マップの学習原理はWinner-Take-Allによる競合学習とニューロンの空間的配置に基づく近傍学習の組み合わせである。自己組織化マップはデータの次元削減や可視化を行うニューラルネットであり、高次元データの可視化やデータマイニングなどの目的で幅広い分野で利用されてきた。}} | |||
==脳の数理モデルとしての自己組織化マップ== | ==脳の数理モデルとしての自己組織化マップ== | ||
大脳皮質には機能局在性があり、機能の類似するニューロンは皮質上で隣接して分布することが知られる。また感覚系では入力の空間的トポロジーが保存されるトポグラフィックマッピングが知られる。これらの空間的な機能分化が自己組織的に生じる原理について、形式ニューロンを用いた数理モデルの研究が行われた。たとえばMarsburg<ref name=vonderMalsburg1973><pubmed>4786750</pubmed></ref>[14]やAmari<ref name=Amari1980><pubmed>6246997</pubmed></ref>[1]は外界からの刺激によりニューロンの機能局在と位相的な配置が自己組織化することを示した。 | |||
このような空間的機能分化の自己組織化は2種に分けられる<ref name=Kohonen2006><pubmed>16774731</pubmed></ref> | このような空間的機能分化の自己組織化は2種に分けられる<ref name=Kohonen2006><pubmed>16774731</pubmed></ref>[5]。タイプ1はレチノトピーを典型例とするトポグラフィックマッピングであり、入力信号の類似性に基づく自己組織化である。タイプ2は視覚系における方向選択性ニューロンなどの機能マップであり、出力信号の類似性に基づく自己組織化である。Kohonenはタイプ2の自己組織化の数理モデルを元に、ニューロン間の結合やダイナミクスを簡約化し、実データ解析に応用可能なシンプルなアルゴリズムに帰着した<ref name=Kohonen1982>'''T. Kohonen.(1982).'''<br>Self-organized formation of topologically correct feature maps. Biological Cybernetics, 43(1):59-69 [https://doi.org/10.1007/BF00337288 PDF]</ref>[4]。これが自己組織化マップである。自己組織化マップは初期の提案からいくつかの改良を重ねており、最終形であるバッチ型自己組織化マップにおいては安定した学習が達成できる一方で、形式ニューロンを用いた数理モデルからは大きく様変わりしている<ref name=Kohonen2013><pubmed>23067803</pubmed></ref>[6]。 | ||
==ニューラルネットとしての自己組織化マップ== | ==ニューラルネットとしての自己組織化マップ== | ||
[[ファイル:Furukawa Self organizing map fig1.png|サムネイル| | [[ファイル:Furukawa Self organizing map fig1.png|サムネイル|図1. 自己組織化マップによる動物マップ <br>表 1のデータをバッチ型自己組織化マップで学習した結果.哺乳類/鳥類,肉食/草 食,大型/小型などの動物の特徴に基づき,類似した動物の地図を作っている.このマップは U-matrix 法<ref name=Ultsch1993>'''A. Ultsch. (1993).'''<br>Self-organizing neural networks for visualization and classification. In O. Opitz, B. Lausen, and R. Klar, editors, Information and Classification, pages 307-313. Springer, Berlin. | ||
[https://doi.org/10.1007/978-3-642-50974-2_31 PDF]</ref>で色付けしており、赤い領域がクラスタ境界を表す。]] | [https://doi.org/10.1007/978-3-642-50974-2_31 PDF]</ref>[11]で色付けしており、赤い領域がクラスタ境界を表す。]] | ||
===入力と出力=== | ===入力と出力=== | ||
自己組織化マップの入力は通常、高次元のベクトルデータセットである。一方、出力はデータセットを低次元(通常は2次元) | 自己組織化マップの入力は通常、高次元のベクトルデータセットである。一方、出力はデータセットを低次元(通常は2次元)に射影したものであり、データ分布を地図として可視化して見ることができる。また低次元マップ空間から高次元データ空間への写像も学習によって得られる。 | ||
'''図1'''は自己組織化マップにより得られた「動物マップ」である。入力データ('''表1''')は16種の動物データであり、それぞれ15次元のベクトルで表されている。マップ上で類似する動物(哺乳類や鳥類、肉食や草食)は互いに近くに配置されている。 | |||
学習が終了した後、得られたマップは主に3つの用途で使うことができる。第一は可視化によるデータマイニングである。動物マップの例ならば、どの動物が互いに似ているか、どのような動物クラスタが存在するかを知ることができる。第二は新規データをマップ上へ射影することである。これにより新規データがマップのどこに位置するかを可視化できる。またラベル付き学習データを用いた場合は、新規データのラベルを推定することも可能である。動物マップの例ならば、新規の動物がマップのどこに位置するかを示したり、その動物が哺乳類か鳥類かを推測したりできる。第三は、新規データの予測や生成である。たとえば2種の中間的な性質を持つ動物の特徴を予測することができる。 | |||
===構造と学習原理=== | ===構造と学習原理=== | ||
自己組織化マップではニューロン(ユニット)が低次元(通常は2次元)のマップ空間に格子状に並んだ構造を持つ(図2)。これは皮質上にニューロンが並んでいるものに見立てられる。またマップ空間においてニューロンの位置は固定されている。これらニューロンへの入力はq次元のベクトルx = (x1 , . . . , xq )であり、すべてのニューロンへ等しく入力される。xはq個の感覚ニューロンから皮質ニューロンへの入力に相当する。 | |||
自己組織化マップではニューロン(ユニット)が低次元(通常は2次元)のマップ空間に格子状に並んだ構造を持つ( | |||
一方、各ニューロンは参照ベクトルと呼ばれるq次元のベクトル値mi =(mi1,...,miq)を保持する(iは ニューロンの番号).参照ベクトルは感覚ニューロンから皮質ニューロンへのシナプス強度と解釈できる。ただし自己組織化マップでは各ニューロンが参照ベクトル値を記憶さえしていればよく、必ずしもシナプスという形で実装される必要はない。 | |||
自己組織化マップは競合原理と近傍学習原理という2つの原理で動作する。第一の競合原理では、入力データにもっとも合致するニューロンが勝者(最適ユニット: Best Matching Unitとも呼ばれる)として選ばれる。すなわち入力データxにもっとも近い参照ベクトルmiを持つニューロンが勝者となり、そのデータを学習する権利をすべて獲得する。この競合原理はWinner-Take-Allとも呼ばれる。 | |||
第二の原理である近傍学習は、勝者が獲得した学習の権利を近傍のニューロンに分配するものである。勝者に隣接するニューロンには入力データを学習する権利が分配される一方で、勝者から離れたニューロンには学習の権利が与えられない。近傍学習原理により、勝者およびその近傍ニューロンの参照ベクトルは入力xとの誤差が小さくなるように更新され、次に同じ入力が来たときに再び勝者になりやすくなる。この2つの学習原理が位相的な順序を自己組織化する上で重要な役割を果たす。 | |||
===オンライン型アルゴリズム=== | ===オンライン型アルゴリズム=== | ||
自己組織化マップの学習アルゴリズムは、競合・協調・適合という3プロセスの繰り返し計算である<ref name=Haykin1998>'''Haykin, S. (1998).'''<br>Neural Networks - A Comprehensive Foundation (2nd ed). Prentice Hall.</ref> [2]。時刻 tにおける入力データをx(t)とすれば、それにもっとも近い参照ベクトルを持つニューロンc(t)が時刻tの勝者となる: | |||
c(t) = arg min ∥x(t) − mi (t)∥. i | |||
これが競合プロセスである。 | これが競合プロセスである。 | ||
一方、各ニューロンが学習する量はマップ空間上で勝者c(t)に近いニューロンほど大きい。この配分は近傍関数h(·,·)で決まる。近傍関数は勝者ニューロンcに対してニューロンiがどれくらいデータを学習するかを表し、しばしばガウス関数が用いられる: | |||
hci =h(zc,zi)=exp −2σ2(t)∥zc −zi∥2 | |||
ここでzc,ziはマップ空間上でのニューロンc,iの座標であり、σは近傍の広さを決めるパラメータである。これが協調プロセスである。 | |||
:: | 最後に、入力x(t)との誤差が小さくなるように各ニューロンの参照ベクトルを更新する: mi(t + 1) := mi(t) + εhci (x(t) − mi(t)). | ||
ここでεは正の小さな定数である。これを適合プロセスである。 | |||
このように入力x(t)を変えながら競合・協調・適合プロセスを繰り返すのがオンライン型自己組織化マップのアルゴリズムである。また近傍の広さσは学習の初期に広くしておき、学習が進むに連れて小さくしていく。このオンライン型アルゴリズムは、他の数理モデルや現実の脳との関連性を考えるうえで有用である。しかしオンライン型は学習時間がかかる上に計算結果が不安定であり、実データ解析には次に述べるバッチ型アルゴリズムを用いるべきであるとKohone自身も指摘している<ref name=Kohonen2013><pubmed>23067803</pubmed></ref>[6]。 | |||
===バッチ型アルゴリズム=== | ===バッチ型アルゴリズム=== | ||
入力データすべての集合をX={xn}とする。バッチ型アルゴリズムでは、学習の各ステップですべてのデータを用いる。 | |||
競合プロセスでは、全データに対する勝者をすべて求める。 | 競合プロセスでは、全データに対する勝者をすべて求める。 | ||
∀n, cn(t) = arg min ∥xn − mi(t)∥ i | |||
続く協調プロセスでは、すべてのデータとニューロンの組み合わせについて学習量を決定する。 | |||
∀n, i, hni(t) = h (cn(t), i) 最後の適合プロセスでは、参照ベクトルを学習量の重み付き平均値へ更新する。 | |||
∑ | |||
n hni(t)xn ∀i, mi(t+1):= ∑ | |||
これらを収束するまで繰り返すのがバッチ型アルゴリズムである。オンライン型が数百ないし数千ステップの繰り返し計算が必要なのに対し、バッチ型は数十ステップで収束する。また学習結果の安定性についてもバッチ型が優れている。 | |||
=== 自己組織化マップの学習理論 === | === 自己組織化マップの学習理論 === | ||
自己組織化マップの学習理論に関しては多くの研究が行われてきた。Kohonenの自己組織化マップに直接対応する目的関数は存在しないが、アルゴリズムをわずかに修正することで目的関数を得ることができる<ref name=Heskes2001><pubmed>18249959</pubmed></ref>。また自己組織化マップのアルゴリズムは競合・協調プロセスをEステップ、適合プロセスをMステップとするEMアルゴリズムとして解釈できる<ref name=Heskes2001 /><ref name=Verbeek2005>'''J.J. Verbeek, N. Vlassis, and B.J.A. Kröse. (2005).'''<br>Self-organizing mixture models. Neurocomputing, 63(SPEC. ISS.):99-123 [https://doi.org/10.1016/j.neucom.2004.04.008 PDF]</ref>。 | 自己組織化マップの学習理論に関しては多くの研究が行われてきた。Kohonenの自己組織化マップに直接対応する目的関数は存在しないが、アルゴリズムをわずかに修正することで目的関数を得ることができる<ref name=Heskes2001><pubmed>18249959</pubmed></ref> [3]。また自己組織化マップのアルゴリズムは競合・協調プロセスをEステップ、適合プロセスをMステップとするEMアルゴリズムとして解釈できる<ref name=Heskes2001 /><ref name=Verbeek2005>'''J.J. Verbeek, N. Vlassis, and B.J.A. Kröse. (2005).'''<br>Self-organizing mixture models. Neurocomputing, 63(SPEC. ISS.):99-123 [https://doi.org/10.1016/j.neucom.2004.04.008 PDF]</ref> [3, 13]。 | ||
===自己組織化マップと機械学習=== | ===自己組織化マップと機械学習=== | ||
自己組織化マップは高次元データを低次元に射影して可視化するため、次元削減法の一種とみることができる。したがって高次元データの可視化やデータマイニングのみを目的とする場合は、他の次元削減法、たとえばt-SNE<ref name=VanDerMaaten2008>'''L. Van Der Maaten and G. Hinton. (2008).'''<br>Visualizing data using t-sne. Journal of Machine Learning Research, 9:2579-2625, 2008.</ref>[12], Isomap<ref name=Tenenbaum2000><pubmed>11125149</pubmed></ref> [10], Locally Linear Embedding <ref name=Roweis2000><pubmed>11125150</pubmed></ref>[9]などでも代用できる。これらの手法と自己組織化マップの大きく異る点は、学習終了後、新規の入力データに対してもマップ上へ射影できること、および新規データの予測や生成ができるという点である。 | |||
新規データの射影・予測・生成も含めた自己組織化マップと等価な手法として、ガウス過程潜在変数モデル(Gaussianprocess latent variable model, GPLVM)がある<ref name=Lawrence2004>'''N.D. Lawrence. (2004).''' Gaussian process latent variable models for visualisation of high dimensional data.</ref>[7]。GPLVMはベイズ推論に基づくため柔軟な拡張が可能である。また教師なしカーネル回帰(Unsupervisedkernelregression, UKR) は自己組織化マップと同じ目的関数を用いており、自己組織化マップの直接的な発展形と見ることができる<ref name=Meinicke2005><pubmed>16173183</pubmed></ref>[8]。マップ空間を離散化する自己組織化マップと異なり、GPLVMとUKRは低次元空間を連続空間のまま扱える。また可視化を目的としないのであれば、変分オートエンコーダ(Variational auto-encoder, VAE)も自己組織化マップと同じ機能を持つ。現在の機械学習・AIの分野では自己組織化マップに代わってこれらの手法が広く使われている。 | |||
==参考文献== | ==参考文献== | ||
<references /> | <references /> | ||
2022年1月6日 (木) 00:00時点における版
古川 徹生 九州工業大学
英語 Self-Organizing Map, Kohonen map
略称 SOM
同義語 自己組織化写像
自己組織化マップはT. Kohonenによって提案された教師なしニューラルネットの一種である。自己組織化マップはもともと大脳の機能地図の自己組織化現象を説明する数理モデルに由来する。しかし実データ解析へ応用するため、ニューロン間の結合やダイナミクスが簡約化され、計算の効率化が図られている。自己組織化マップの学習原理はWinner-Take-Allによる競合学習とニューロンの空間的配置に基づく近傍学習の組み合わせである。自己組織化マップはデータの次元削減や可視化を行うニューラルネットであり、高次元データの可視化やデータマイニングなどの目的で幅広い分野で利用されてきた。
脳の数理モデルとしての自己組織化マップ
大脳皮質には機能局在性があり、機能の類似するニューロンは皮質上で隣接して分布することが知られる。また感覚系では入力の空間的トポロジーが保存されるトポグラフィックマッピングが知られる。これらの空間的な機能分化が自己組織的に生じる原理について、形式ニューロンを用いた数理モデルの研究が行われた。たとえばMarsburg[1][14]やAmari[2][1]は外界からの刺激によりニューロンの機能局在と位相的な配置が自己組織化することを示した。
このような空間的機能分化の自己組織化は2種に分けられる[3][5]。タイプ1はレチノトピーを典型例とするトポグラフィックマッピングであり、入力信号の類似性に基づく自己組織化である。タイプ2は視覚系における方向選択性ニューロンなどの機能マップであり、出力信号の類似性に基づく自己組織化である。Kohonenはタイプ2の自己組織化の数理モデルを元に、ニューロン間の結合やダイナミクスを簡約化し、実データ解析に応用可能なシンプルなアルゴリズムに帰着した[4][4]。これが自己組織化マップである。自己組織化マップは初期の提案からいくつかの改良を重ねており、最終形であるバッチ型自己組織化マップにおいては安定した学習が達成できる一方で、形式ニューロンを用いた数理モデルからは大きく様変わりしている[5][6]。
ニューラルネットとしての自己組織化マップ
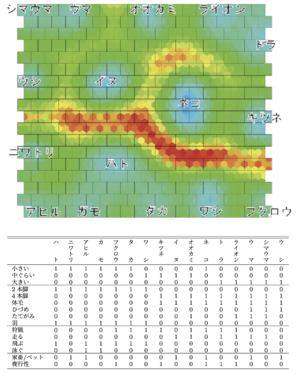
表 1のデータをバッチ型自己組織化マップで学習した結果.哺乳類/鳥類,肉食/草 食,大型/小型などの動物の特徴に基づき,類似した動物の地図を作っている.このマップは U-matrix 法[6][11]で色付けしており、赤い領域がクラスタ境界を表す。
入力と出力
自己組織化マップの入力は通常、高次元のベクトルデータセットである。一方、出力はデータセットを低次元(通常は2次元)に射影したものであり、データ分布を地図として可視化して見ることができる。また低次元マップ空間から高次元データ空間への写像も学習によって得られる。
図1は自己組織化マップにより得られた「動物マップ」である。入力データ(表1)は16種の動物データであり、それぞれ15次元のベクトルで表されている。マップ上で類似する動物(哺乳類や鳥類、肉食や草食)は互いに近くに配置されている。
学習が終了した後、得られたマップは主に3つの用途で使うことができる。第一は可視化によるデータマイニングである。動物マップの例ならば、どの動物が互いに似ているか、どのような動物クラスタが存在するかを知ることができる。第二は新規データをマップ上へ射影することである。これにより新規データがマップのどこに位置するかを可視化できる。またラベル付き学習データを用いた場合は、新規データのラベルを推定することも可能である。動物マップの例ならば、新規の動物がマップのどこに位置するかを示したり、その動物が哺乳類か鳥類かを推測したりできる。第三は、新規データの予測や生成である。たとえば2種の中間的な性質を持つ動物の特徴を予測することができる。
構造と学習原理
自己組織化マップではニューロン(ユニット)が低次元(通常は2次元)のマップ空間に格子状に並んだ構造を持つ(図2)。これは皮質上にニューロンが並んでいるものに見立てられる。またマップ空間においてニューロンの位置は固定されている。これらニューロンへの入力はq次元のベクトルx = (x1 , . . . , xq )であり、すべてのニューロンへ等しく入力される。xはq個の感覚ニューロンから皮質ニューロンへの入力に相当する。
一方、各ニューロンは参照ベクトルと呼ばれるq次元のベクトル値mi =(mi1,...,miq)を保持する(iは ニューロンの番号).参照ベクトルは感覚ニューロンから皮質ニューロンへのシナプス強度と解釈できる。ただし自己組織化マップでは各ニューロンが参照ベクトル値を記憶さえしていればよく、必ずしもシナプスという形で実装される必要はない。
自己組織化マップは競合原理と近傍学習原理という2つの原理で動作する。第一の競合原理では、入力データにもっとも合致するニューロンが勝者(最適ユニット: Best Matching Unitとも呼ばれる)として選ばれる。すなわち入力データxにもっとも近い参照ベクトルmiを持つニューロンが勝者となり、そのデータを学習する権利をすべて獲得する。この競合原理はWinner-Take-Allとも呼ばれる。 第二の原理である近傍学習は、勝者が獲得した学習の権利を近傍のニューロンに分配するものである。勝者に隣接するニューロンには入力データを学習する権利が分配される一方で、勝者から離れたニューロンには学習の権利が与えられない。近傍学習原理により、勝者およびその近傍ニューロンの参照ベクトルは入力xとの誤差が小さくなるように更新され、次に同じ入力が来たときに再び勝者になりやすくなる。この2つの学習原理が位相的な順序を自己組織化する上で重要な役割を果たす。
オンライン型アルゴリズム
自己組織化マップの学習アルゴリズムは、競合・協調・適合という3プロセスの繰り返し計算である[7] [2]。時刻 tにおける入力データをx(t)とすれば、それにもっとも近い参照ベクトルを持つニューロンc(t)が時刻tの勝者となる: c(t) = arg min ∥x(t) − mi (t)∥. i これが競合プロセスである。 一方、各ニューロンが学習する量はマップ空間上で勝者c(t)に近いニューロンほど大きい。この配分は近傍関数h(·,·)で決まる。近傍関数は勝者ニューロンcに対してニューロンiがどれくらいデータを学習するかを表し、しばしばガウス関数が用いられる: hci =h(zc,zi)=exp −2σ2(t)∥zc −zi∥2
ここでzc,ziはマップ空間上でのニューロンc,iの座標であり、σは近傍の広さを決めるパラメータである。これが協調プロセスである。
最後に、入力x(t)との誤差が小さくなるように各ニューロンの参照ベクトルを更新する: mi(t + 1) := mi(t) + εhci (x(t) − mi(t)).
ここでεは正の小さな定数である。これを適合プロセスである。
このように入力x(t)を変えながら競合・協調・適合プロセスを繰り返すのがオンライン型自己組織化マップのアルゴリズムである。また近傍の広さσは学習の初期に広くしておき、学習が進むに連れて小さくしていく。このオンライン型アルゴリズムは、他の数理モデルや現実の脳との関連性を考えるうえで有用である。しかしオンライン型は学習時間がかかる上に計算結果が不安定であり、実データ解析には次に述べるバッチ型アルゴリズムを用いるべきであるとKohone自身も指摘している[5][6]。
バッチ型アルゴリズム
入力データすべての集合をX={xn}とする。バッチ型アルゴリズムでは、学習の各ステップですべてのデータを用いる。 競合プロセスでは、全データに対する勝者をすべて求める。 ∀n, cn(t) = arg min ∥xn − mi(t)∥ i 続く協調プロセスでは、すべてのデータとニューロンの組み合わせについて学習量を決定する。 ∀n, i, hni(t) = h (cn(t), i) 最後の適合プロセスでは、参照ベクトルを学習量の重み付き平均値へ更新する。 ∑ n hni(t)xn ∀i, mi(t+1):= ∑ これらを収束するまで繰り返すのがバッチ型アルゴリズムである。オンライン型が数百ないし数千ステップの繰り返し計算が必要なのに対し、バッチ型は数十ステップで収束する。また学習結果の安定性についてもバッチ型が優れている。
自己組織化マップの学習理論
自己組織化マップの学習理論に関しては多くの研究が行われてきた。Kohonenの自己組織化マップに直接対応する目的関数は存在しないが、アルゴリズムをわずかに修正することで目的関数を得ることができる[8] [3]。また自己組織化マップのアルゴリズムは競合・協調プロセスをEステップ、適合プロセスをMステップとするEMアルゴリズムとして解釈できる[8][9] [3, 13]。
自己組織化マップと機械学習
自己組織化マップは高次元データを低次元に射影して可視化するため、次元削減法の一種とみることができる。したがって高次元データの可視化やデータマイニングのみを目的とする場合は、他の次元削減法、たとえばt-SNE[10][12], Isomap[11] [10], Locally Linear Embedding [12][9]などでも代用できる。これらの手法と自己組織化マップの大きく異る点は、学習終了後、新規の入力データに対してもマップ上へ射影できること、および新規データの予測や生成ができるという点である。
新規データの射影・予測・生成も含めた自己組織化マップと等価な手法として、ガウス過程潜在変数モデル(Gaussianprocess latent variable model, GPLVM)がある[13][7]。GPLVMはベイズ推論に基づくため柔軟な拡張が可能である。また教師なしカーネル回帰(Unsupervisedkernelregression, UKR) は自己組織化マップと同じ目的関数を用いており、自己組織化マップの直接的な発展形と見ることができる[14][8]。マップ空間を離散化する自己組織化マップと異なり、GPLVMとUKRは低次元空間を連続空間のまま扱える。また可視化を目的としないのであれば、変分オートエンコーダ(Variational auto-encoder, VAE)も自己組織化マップと同じ機能を持つ。現在の機械学習・AIの分野では自己組織化マップに代わってこれらの手法が広く使われている。
参考文献
- ↑
von der Malsburg, C. (1973).
Self-organization of orientation sensitive cells in the striate cortex. Kybernetik, 14(2), 85-100. [PubMed:4786750] [WorldCat] [DOI] - ↑
Amari, S. (1980).
Topographic organization of nerve fields. Bulletin of mathematical biology, 42(3), 339-64. [PubMed:6246997] [WorldCat] [DOI] - ↑
Kohonen, T. (2006).
Self-organizing neural projections. Neural networks : the official journal of the International Neural Network Society, 19(6-7), 723-33. [PubMed:16774731] [WorldCat] [DOI] - ↑ T. Kohonen.(1982).
Self-organized formation of topologically correct feature maps. Biological Cybernetics, 43(1):59-69 PDF - ↑ 5.0 5.1
Kohonen, T. (2013).
Essentials of the self-organizing map. Neural networks : the official journal of the International Neural Network Society, 37, 52-65. [PubMed:23067803] [WorldCat] [DOI] - ↑ A. Ultsch. (1993).
Self-organizing neural networks for visualization and classification. In O. Opitz, B. Lausen, and R. Klar, editors, Information and Classification, pages 307-313. Springer, Berlin. PDF - ↑ Haykin, S. (1998).
Neural Networks - A Comprehensive Foundation (2nd ed). Prentice Hall. - ↑ 8.0 8.1
Heskes, T. (2001).
Self-organizing maps, vector quantization, and mixture modeling. IEEE transactions on neural networks, 12(6), 1299-305. [PubMed:18249959] [WorldCat] [DOI] - ↑ J.J. Verbeek, N. Vlassis, and B.J.A. Kröse. (2005).
Self-organizing mixture models. Neurocomputing, 63(SPEC. ISS.):99-123 PDF - ↑ L. Van Der Maaten and G. Hinton. (2008).
Visualizing data using t-sne. Journal of Machine Learning Research, 9:2579-2625, 2008. - ↑
Tenenbaum, J.B., de Silva, V., & Langford, J.C. (2000).
A global geometric framework for nonlinear dimensionality reduction. Science (New York, N.Y.), 290(5500), 2319-23. [PubMed:11125149] [WorldCat] [DOI] - ↑
Roweis, S.T., & Saul, L.K. (2000).
Nonlinear dimensionality reduction by locally linear embedding. Science (New York, N.Y.), 290(5500), 2323-6. [PubMed:11125150] [WorldCat] [DOI] - ↑ N.D. Lawrence. (2004). Gaussian process latent variable models for visualisation of high dimensional data.
- ↑
Meinicke, P., Klanke, S., Memisevic, R., & Ritter, H. (2005).
Principal surfaces from unsupervised kernel regression. IEEE transactions on pattern analysis and machine intelligence, 27(9), 1379-91. [PubMed:16173183] [WorldCat] [DOI]