「In situハイブリダイゼーション法」の版間の差分
細編集の要約なし |
|||
| (3人の利用者による、間の15版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | |||
<font size="+1">[http://researchmap.jp/ohuchihideyo 大内 淑代]</font><[[br]]> | |||
''岡山大学 大学院医歯薬学総合研究科細胞組織学分野''<br> | |||
DOI:<selfdoi /> 原稿受付日:2012年7月12日 原稿完成日:2013年3月25日<br> | |||
担当編集委員:[http://researchmap.jp/noriko1128 大隅 典子](東北大学 大学院医学系研究科 附属創生応用医学研究センター 脳神経科学コアセンター 発生発達神経科学分野)<br> | |||
</div> | |||
[[Image:In situハイブリダイゼーション法1A.png|thumb|300px|'''図1A.ISHの実験例'''<br>組織切片ISH(トリ胚網膜、opsin 5、青い点状のシグナルがmRNA発現細胞、右はセンスプローブによる陰性コントロール)<br>発色基質:NBT, BCIP]] | [[Image:In situハイブリダイゼーション法1A.png|thumb|300px|'''図1A.ISHの実験例'''<br>組織切片ISH(トリ胚網膜、opsin 5、青い点状のシグナルがmRNA発現細胞、右はセンスプローブによる陰性コントロール)<br>発色基質:NBT, BCIP]] | ||
| 10行目: | 17行目: | ||
[[Image:In situハイブリダイゼーション法2.png|thumb|300px|'''図2.組織切片を用いた''in situ''ハイブリダイゼーション法の工程''']] | [[Image:In situハイブリダイゼーション法2.png|thumb|300px|'''図2.組織切片を用いた''in situ''ハイブリダイゼーション法の工程''']] | ||
''In situ''とは”原位置で”という意味で、''in situ''ハイブリダイゼーション(''in situ'' hybridization: ISH)とは原位置でのハイブリダイゼーション(後述)ということである。ISH法には、[[ | 英:''in situ'' hybridization 独:''In situ''-Hybridisierung 仏:hybridation in situ | ||
{{box|text= | |||
''In situ''とは”原位置で”という意味で、''in situ''ハイブリダイゼーション(''in situ'' hybridization: ISH)とは原位置でのハイブリダイゼーション(後述)ということである。ISH法には、[[wj:染色体|染色体]]ISHと組織切片ISH、ホールマウントISH (whole-mount ISH: WISH) がある<ref>'''野地澄晴編'''<br>免疫染色&''in situ''ハイブリダイゼーション<br>''羊土社''、東京、2006</ref><ref>'''D.G. Wilkinson'''<br>In Situ Hybridization, A Practical Approach, 2nd Edition<br>''Oxford University Press'', Oxford, 1999</ref>。染色体ISH法は、染色体における目的遺伝子の[[wj:遺伝子座|遺伝子座]]を明らかにし、染色体異常を検出することができる。組織ISH法は、組織切片を用いて遺伝子発現の第一段階であるmRNAの局在を細胞レベルで明らかにする。[[wj:病理|病理]]組織から[[wj:ウイルス|ウイルス]][[wj:ゲノム|ゲノム]]を検出し、ウイルス感染の診断に用いられることもある。また、胚や器官の一部などを丸ごと用いるISH法を、ホールマウントISH(WISH)という。実験例を図1に示す。遺伝子発現部位の三次元的な情報を得た後で、細胞レベルで遺伝子発現部位を同定しなければならない場合は、WISH後の胚などの組織切片を作製する。図2に、組織切片を用いた''in situ''ハイブリダイゼーション法の工程を模式的に示す。ISHは、細胞内mRNAの局在を明らかにする実験であるので、分解されやすいRNAをいかに分解させずに実験を行うかが重要である。 | |||
}} | |||
== ''In situ''ハイブリダイゼーション法の各工程 == | == ''In situ''ハイブリダイゼーション法の各工程 == | ||
| 17行目: | 28行目: | ||
=== 組織の固定 === | === 組織の固定 === | ||
ISH法では細胞が死んだ状態で[[mRNA]]を検出する。細胞が死んでしまうと、mRNAは急速に分解され消滅してしまう。そのため、''in situ''ハイブリダイゼーション法を用いる場合は、細胞内のmRNAの分解を防ぎ、できるだけ生きていた状態を維持しておく必要がある。細胞や組織などをなるべく自然の状態に保存することを[[wj:固定|固定]]とよんでいるが、''in situ''ハイブリダイゼーション法の場合はmRNAの固定が非常に重要である。''In situ''ハイブリダイゼーション法では、用時調製した4%[[wj:パラホルムアルデヒド|パラホルムアルデヒド]]液を用いて[[wj:還流固定|還流固定]]し、[[wj:臓器|臓器]]等を取り出して短時間、低温でさらに[[wj:浸漬固定|浸漬固定]]することが多い。新鮮迅速凍結切片を用いてISHを行うこともある。組織切片はスライドガラスに貼付けたもの、あるいは浮遊[[切片]]<ref><[[pubmed]]> 21037591</pubmed></ref>を用いる。 | |||
=== 前処理 === | === 前処理 === | ||
組織ISH法、WISH法のいずれにおいても、ハイブリダイゼーションを行う前に固定した組織の前処理を行う。主な目的は、[[ | 組織ISH法、WISH法のいずれにおいても、ハイブリダイゼーションを行う前に固定した組織の前処理を行う。主な目的は、[[wj:界面活性剤|界面活性剤]]で細胞膜の透過性を上げ、タンパク質分解酵素proteinase Kなどを用いて結合組織等を分解しプローブの浸透性をよくすることと、プローブの非特異的な吸着を防ぐことである。特に、[[wj:タンパク質分解酵素|タンパク質分解酵素]]の濃度と処理時間は、組織の種類、固定法とも関連し、''in situ''ハイブリダイゼーション法の結果を左右する重要な因子である。 | ||
=== プローブの調製 === | === プローブの調製 === | ||
目的のmRNAを検出するために、そのmRNAと特異的にハイブリダイズする、つまり、目的のmRNAと[[ | 目的のmRNAを検出するために、そのmRNAと特異的にハイブリダイズする、つまり、目的のmRNAと[[wj:相補的|相補的]]な配列をもったRNAまたはDNAプローブが必要である。プローブとして、 | ||
# 化学合成したDNAを用いる[[ | # 化学合成したDNAを用いる[[wj:オリゴヌクレオチドプローブ|オリゴヌクレオチドプローブ]]<br> | ||
# DNA合成酵素によりin vitroで合成したDNAプローブ<br> | # DNA合成酵素によりin vitroで合成したDNAプローブ<br> | ||
# RNA合成酵素によりin vitroで合成したRNAプローブ<br> | # RNA合成酵素によりin vitroで合成したRNAプローブ<br> | ||
の3種類がよく用いられている。DNAオリゴプローブは、DNA合成装置で合成する。B, Cにおいては、プローブ合成のための[[ | の3種類がよく用いられている。DNAオリゴプローブは、DNA合成装置で合成する。B, Cにおいては、プローブ合成のための[[wj:鋳型|鋳型]]DNAが必要である。RNA-RNAハイブリッドが3者の中で最も安定であり、現在RNAプローブを用いる方法が一般的である。合成したRNAが分解されないように細心の注意を払う。他に[[wj:locked nucleic acid|locked nucleic acid]] (LNA)(後述)や[[wj:ペプチド核酸|ペプチド核酸]]をプローブとして用いる方法がある。プローブを可視化のために標識する方法には主に次の2つの方法がある。 | ||
# [[ | # [[wj:酵素抗体法|酵素抗体法]]または[[wj:蛍光抗体法|蛍光抗体法]]:適当な[[wj:抗原|抗原]]([[wj:ジゴキシゲニン|ジゴキシゲニン]] (digoxigenin, DIG), [[wj:フルオレセイン|フルオレセイン]] (fluorescein), [[wj:ビオチン|ビオチン]] (biotin)など)の結合した[[wj:ヌクレオチド|ヌクレオチド]]を用いてプローブを標識し、その抗原に対する[[wj:抗体|抗体]]を用いて発色または蛍光により可視化する。<br> | ||
# [[ | # [[wj:放射性同位元素|放射性同位元素]](radioisotope)を用いて、[[wj:オートラジオグラフィー|オートラジオグラフィー]]法により可視化する。 | ||
===ハイブリダイゼーション=== | ===ハイブリダイゼーション=== | ||
DNAの[[ | DNAの[[wj:二重らせん|二重らせん]]構造は、塩基対A:Tに形成される2つの[[wj:水素結合|水素結合]]と塩基対G:Cに形成される3つの水素結合により安定に保たれている。この二本鎖を一本鎖にする方法の1つとして、熱変性がある。二重らせんDNA溶液の温度を高くしながら、DNA溶液の260 nmの[[wj:吸光度|吸光度]]A260を測定すると、しだいにA260は高くなる。これはDNAの二重らせんが壊れ、一本鎖になるためで、この温度による吸光度の変化を表す曲線をDNAの[[wj:融解曲線|融解曲線]]とよんでいる。この現象は、らせんが消失して塩基間の相互作用が少なくなるため、塩基の光吸収の効率が変化し(深色効果)、各塩基の[[wj:分子吸光係数|分子吸光係数]]が高くなるために生じる。温度の低い時のDNAをヘリックス100%とし、高温での吸光度が一定になる状態でヘリックス0%と仮定すると、ヘリックス50%になる温度([[wj:融解温度|融解温度]] (melting temperature: Tm))を決定することができる。 | ||
Tmは二重らせんの安定度の目安になる。非常に安定ならせんであれば、Tmは80〜90℃になる。逆に不安定であれば、30〜40℃になる。TmはGC塩基対の含量、核酸の長さ、核酸の塩基対のミスマッチなどに依存し、DNA溶液のイオン強度(塩濃度)や組成により変化する。Tmに関する経験的な式は、例えば、RNA-RNAハイブリッドの場合、 | Tmは二重らせんの安定度の目安になる。非常に安定ならせんであれば、Tmは80〜90℃になる。逆に不安定であれば、30〜40℃になる。TmはGC塩基対の含量、核酸の長さ、核酸の塩基対のミスマッチなどに依存し、DNA溶液のイオン強度(塩濃度)や組成により変化する。Tmに関する経験的な式は、例えば、RNA-RNAハイブリッドの場合、 | ||
| 43行目: | 54行目: | ||
Tm=79.8+18.5(logM)+0.584(%G+C)+0.118 × 10<sup>-2</sup>(%G+C)<sup>2</sup>−0.35(%formamide)− 820/l<br> | Tm=79.8+18.5(logM)+0.584(%G+C)+0.118 × 10<sup>-2</sup>(%G+C)<sup>2</sup>−0.35(%formamide)− 820/l<br> | ||
ここで、Mは1価の正イオンのモル濃度、%G+CはGC含量、%formamideは[[ | ここで、Mは1価の正イオンのモル濃度、%G+CはGC含量、%formamideは[[wj:ホルムアミド濃度|ホルムアミド濃度]]、lはプローブの長さ(bp)である。 | ||
Tmの温度では、全体の50%ほど二重らせんが形成されていることになる。ハイブリダイゼーションの温度Thは、Tmより5〜20℃低い温度で行う。どの程度まで塩基対のマッチを許容するかを[[ | Tmの温度では、全体の50%ほど二重らせんが形成されていることになる。ハイブリダイゼーションの温度Thは、Tmより5〜20℃低い温度で行う。どの程度まで塩基対のマッチを許容するかを[[wj:stringency|stringency]]とよぶ。例えば、(Tm−5)℃でハイブリダイズさせると、stringencyが非常に高くなり、完全マッチした安定なDNA-DNAしかハイブリッドを形成しない。しかし、ハイブリダイズする温度を(Tm−20)℃と低下させ、stringencyを低くすると、ミスマッチをもったハイブリッドが形成される可能性が高くなる。 | ||
DIGを用いた組織''in situ''ハイブリダイゼーション法では、1kb程度のRNAプローブ(GC含量約50%)を使用する場合、50%ホルムアミド中65℃、12〜14時間で、ほぼよい結果が得られる。もし、短いRNAやGC含量の低いRNAをプローブとする場合は、低stringencyの条件でハイブリダイゼーションを行う。プローブの濃度も重要であり、濃度の高いほうが再会合率が高くなる。プローブの長さが大きいほどシグナルが強くなるが、プローブの配列によってはハイブリダイゼーションに交差性を認める結果となる場合もある。2kbをこえると[[ | DIGを用いた組織''in situ''ハイブリダイゼーション法では、1kb程度のRNAプローブ(GC含量約50%)を使用する場合、50%ホルムアミド中65℃、12〜14時間で、ほぼよい結果が得られる。もし、短いRNAやGC含量の低いRNAをプローブとする場合は、低stringencyの条件でハイブリダイゼーションを行う。プローブの濃度も重要であり、濃度の高いほうが再会合率が高くなる。プローブの長さが大きいほどシグナルが強くなるが、プローブの配列によってはハイブリダイゼーションに交差性を認める結果となる場合もある。2kbをこえると[[wj:アルカリ|アルカリ]][[wj:加水分解|加水分解]]によってRNAの長さを小さくし、組織への浸透を高めるようにする。 | ||
=== 洗浄 === | === 洗浄 === | ||
非特異的に結合したプローブを除去するために、組織を洗浄する.洗浄の条件を厳しくするとバックグラウンドが低下するが、同時にシグナルも弱くなる。RNA-RNAハイブリットは通常の[[ | 非特異的に結合したプローブを除去するために、組織を洗浄する.洗浄の条件を厳しくするとバックグラウンドが低下するが、同時にシグナルも弱くなる。RNA-RNAハイブリットは通常の[[wj:RNA分解酵素|RNA分解酵素]] (RNAse)では分解されないので、これ以降の実験工程はRNAseフリーでなくてよい。 | ||
===シグナルの検出=== | ===シグナルの検出=== | ||
| 57行目: | 68行目: | ||
====酵素抗体法と蛍光抗体法==== | ====酵素抗体法と蛍光抗体法==== | ||
[[image:In situハイブリダイゼーション法3.png|thumb| | [[image:In situハイブリダイゼーション法3.png|thumb|500px|'''図3.アルカリフォスファッターゼによる発色反応'''<br>BCIPがAPにより加水分解されてまず中間産物となり、さらに2量体になって青色色素を産生する。2量体化するときに、2個の水素イオンによりNBTは還元されて不溶性のNBTホルマザンを形成する。(tautomerism:ケト・エノール互変異性)<br>一方、Fast Red TRは、ナフトールAS-MXリン酸の存在下でAPにより赤色の沈殿物を生じる。Fast Red TRを蛍光性のAP基質であるHNPP (2-hydroxy-3-naphtoic acid-2’-phenylanilide phosphate)と共に用いると、還元されてできるHNPの組織への沈着が増し、HNPの蛍光がさらに長波長側シフトして (565~620nm) 強い橙蛍光を発する。[http://www.piercenet.com/browse.cfm?fldID=5A423056-5056-8A76-4E25-1E5F9C0596B2 発色反応、発色基質全般についての説明。]]] | ||
標識プローブのみでは発色することができないので、プローブの中に取り込まれている抗原に対する抗体を用いプローブの検出を行う.プローブをDIGで標識した場合、DIGに特異的に結合する一次抗体を用いて検出する。検出に用いる一次抗体には通常の抗体とは異なり、[[ | 標識プローブのみでは発色することができないので、プローブの中に取り込まれている抗原に対する抗体を用いプローブの検出を行う.プローブをDIGで標識した場合、DIGに特異的に結合する一次抗体を用いて検出する。検出に用いる一次抗体には通常の抗体とは異なり、[[wj:アルカリホスファターゼ|アルカリホスファターゼ]](alkaline phosphatase: AP)または[[w:Horseradish_peroxidase|西洋ワサビペルオキシダーゼ]](horseradish peroxidase: HRP)などの酵素が結合(conjugate)している.例えば、抗体に結合されたAPの基質として、5-Bromo-4-Chloro-3'-Indolylphosphate p-Toluidine salt (BCIP)およびnitro-blue tetrazolium chloride (NBT)を用いた場合は、APがBCIPを脱リン酸化し、中間産物が遊離すると発色する(図3)。一方、抗体に蛍光色素が結合している場合には、[[wj:蛍光顕微鏡|蛍光顕微鏡]]や[[共焦点レーザー走査顕微鏡]]で検出する。 | ||
====放射性同位元素を用いる方法==== | ====放射性同位元素を用いる方法==== | ||
放射性同位元素(RI)として<sup>35</sup>S標識された核酸を用いてプローブを合成する。写真用フイルムまたはオートラジオグラフィー用の[[ | 放射性同位元素(RI)として<sup>35</sup>S標識された核酸を用いてプローブを合成する。写真用フイルムまたはオートラジオグラフィー用の[[wj:乳剤|乳剤]]を用いてRIの放出する[[wj:β線|β線]]を検出する。 | ||
===観察と記録=== | ===観察と記録=== | ||
| 69行目: | 80行目: | ||
== 2色ISH法、蛍光ISH法、抗体染色との2重染色 == | == 2色ISH法、蛍光ISH法、抗体染色との2重染色 == | ||
発色法では、アルカリフォスファターゼ (AP) の基質を変えることにより、2つの遺伝子の発現を同時に検出できる。すなわち、調べたい2つの遺伝子のRNAプローブをDIGまたはフルオレセインで標識し、混合プローブでハイブリダイゼーションさせる。まずAP標識抗フルオレセイン抗体で反応させ、洗浄、発色まで行う(基質は[[ | 発色法では、アルカリフォスファターゼ (AP) の基質を変えることにより、2つの遺伝子の発現を同時に検出できる。すなわち、調べたい2つの遺伝子のRNAプローブをDIGまたはフルオレセインで標識し、混合プローブでハイブリダイゼーションさせる。まずAP標識抗フルオレセイン抗体で反応させ、洗浄、発色まで行う(基質は[[wj:Fast Red|Fast Red]])。その後、酸処理(0.1M[[グリシン]]-HCl、pH2.2)および後固定を行って1色めのアルカリフォスファターゼを失活させる。その後、AP標識抗DIG抗体で反応させ、洗浄、発色を行う(基質NBT/BCIP)。または、DIGまたはフルオレセインで標識した2つのRNAプローブでハイブリダイゼーションさせて洗浄、HRP標識抗DIG抗体で反応させ、HRPの基質ジアミノベンジジン(Diaminobenzidine: DAB)を用いてまず発色させる。次に、AP標識抗フルオレセイン抗体で反応させ、その後APの基質(NBT, BCIP)を用いて発色させる。 | ||
蛍光ISH法では、HRP標識の抗体に対して、[[ | 蛍光ISH法では、HRP標識の抗体に対して、[[wj:tyramide|tyramide]]を結合させた低分子(ビオチンやフルオレセインなど)を反応させシグナルを増強させる方法 (tyramide signal amplification: TSA) を用いる。すなわちtyramideは、ペルオキシダーゼ活性によってラジカル化し組織(タンパク質のTyr, Trp残基など)に低分子ごと集積する。集積した低分子を、蛍光物質の場合はそのまま蛍光顕微鏡等で検出でき、そうでなければその低分子に対する蛍光標識抗体または[[wj:ストレプトアビジン|ストレプトアビジン]]を反応させて間接的に検出することもできる。細胞レベルでmRNAの共在性を証明するためには蛍光ISH法は必須であり、AP反応系Fast Red/[[wj:HNPP|HNPP]]と組み合わせることで2重蛍光ISH法を行うことができる。[[抗体染色]]との2重染色では、以上のISH工程をタンパク質分解酵素なしで行ってそのあと通常の間接蛍光抗体染色を行う、あるいは先にビオチン化抗体と蛍光標識アビジンを用いてRNAseフリーで抗体染色を行い、その後AP反応系Fast Red/HNPPによるISH法を行うなどの方法がある。さらなる多重蛍光ISH法については、hybridization chain reaction法がある。 | ||
== マイクロRNAを検出するISH法 == | == マイクロRNAを検出するISH法 == | ||
Locked nucleic acid (LNA)プローブを用いる。LNAは、[[ | Locked nucleic acid (LNA)プローブを用いる。LNAは、[[wj:リボ核酸|リボ核酸]]の2’位の[[wj:酸素|酸素]]原子と4’位の[[wj:炭素|炭素]]原子とが架橋しており、相補的な核酸に対合したときの熱安定性が非常に高く、短いRNAに対して高いTm値でハイブリダイゼーションできる。この性質を利用してLNAプローブによりマイクロRNAのISHが可能になった。 | ||
== ISHデータベース == | == ISHデータベース == | ||
Allen Brain Atlasは、[[ | Allen Brain Atlasは、[[wj:マイクロソフト社|マイクロソフト社]]創設者の一人である[[w:Paul G. Allen|Paul G. Allen]]の出資によって2003年に設立された[[wj:Allen Institute for Brain Science|Allen Institute for Brain Science]]のISHデータベースである。2006年12月発表のNature の記事によると、まず約2万の遺伝子のマウス成体脳における組織切片ISHのデータが公開された<ref><pubmed> 17151600</pubmed></ref>。現在、[[マウス]][[脳]]に加えて、[[wj:ヒト脳|ヒト脳]]、発生期マウス脳、発生期ヒト脳、マウス脳神経回路、ヒト以外の[[wj:霊長類|霊長類]]脳、マウス[[脊髄]]、[[ヒト]][[神経膠芽腫]]に関するISHデータベースが公開されている。 | ||
==関連項目== | |||
*[[免疫組織化学]] | |||
== 外部リンク == | == 外部リンク == | ||
* [http://www.brain-map.org/ Allen Brain Atlas] | * [http://www.brain-map.org/ Allen Brain Atlas] | ||
* [https://e-labdoc.roche.com/LFR_PublicDocs/ras/11209256910_en_15.pdf | * [https://e-labdoc.roche.com/LFR_PublicDocs/ras/11209256910_en_15.pdf Roche社のジゴキシゲニンの説明。] | ||
* [https://e-labdoc.roche.com/LFR_PublicDocs/ras/11758888001_en_07.pdf | * [https://e-labdoc.roche.com/LFR_PublicDocs/ras/11758888001_en_07.pdf Roche社の発色基質(HNPP, Fast Red)の説明。] | ||
* [http://www.perkinelmer.co.jp/products_ls/resrch/pdf/BRO_TSA_TSAPlus.pdf | * [http://www.perkinelmer.co.jp/products_ls/resrch/pdf/BRO_TSA_TSAPlus.pdf Perkin-Elmer社のチラミドを用いたISHとシグナル増強(CARD)の説明。] | ||
* [http://www.cosmobio.co.jp/product/bunshiseibutsu/mirna/mirna_5/exq_20101007.asp?entry_id=6535 | * [http://www.cosmobio.co.jp/product/bunshiseibutsu/mirna/mirna_5/exq_20101007.asp?entry_id=6535 コスモバイオ社のマイクロRNAのISH (locked nucleic acid)の説明。] | ||
* [http://www.nibb.ac.jp/brish/index.html 基礎生物学研究所山森研究室(渡我部昭哉博士作成)ホームページ BraInSitu] - 脳の組織切片ISH法(特に浮遊法)について詳細なプロトコールが紹介されている。 | * [http://www.nibb.ac.jp/brish/index.html 基礎生物学研究所山森研究室(渡我部昭哉博士作成)ホームページ BraInSitu] - 脳の組織切片ISH法(特に浮遊法)について詳細なプロトコールが紹介されている。 | ||
* [http://www.piercenet.com/browse.cfm?fldID=5A423056-5056-8A76-4E25-1E5F9C0596B2 Pierce社の発色反応、発色基質全般についての説明。] | |||
== 参考文献 == | == 参考文献 == | ||
<references /> | <references /> | ||
2021年12月15日 (水) 20:38時点における最新版
大内 淑代<br>
岡山大学 大学院医歯薬学総合研究科細胞組織学分野
DOI:10.14931/bsd.2163 原稿受付日:2012年7月12日 原稿完成日:2013年3月25日
担当編集委員:大隅 典子(東北大学 大学院医学系研究科 附属創生応用医学研究センター 脳神経科学コアセンター 発生発達神経科学分野)
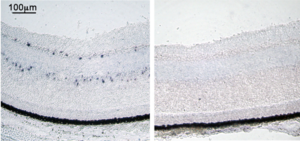
組織切片ISH(トリ胚網膜、opsin 5、青い点状のシグナルがmRNA発現細胞、右はセンスプローブによる陰性コントロール)
発色基質:NBT, BCIP
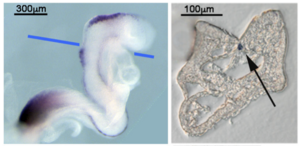
ホールマウントISH (WISH)
マウス胚WISHとWISH後の切片化(Wnt1、青線で切片化、シグナルは矢印[脊索]、凍結切片、未封入)

組織切片蛍光ISH+抗体染色
トリ胚網膜、opsin 5 mRNA (緑)、Islet1タンパク質(マゼンダCy3、上)・GAD65/67タンパク質(マゼンタCy3、下)、核(DAPI)[青])
ISHはFluorescein-tyramide法
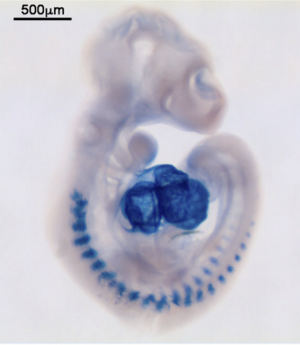
マイクロRNAのWISH(マウス胚、miR-1)
発色基質: NBT, BCIP
英:in situ hybridization 独:In situ-Hybridisierung 仏:hybridation in situ
In situとは”原位置で”という意味で、in situハイブリダイゼーション(in situ hybridization: ISH)とは原位置でのハイブリダイゼーション(後述)ということである。ISH法には、染色体ISHと組織切片ISH、ホールマウントISH (whole-mount ISH: WISH) がある[1][2]。染色体ISH法は、染色体における目的遺伝子の遺伝子座を明らかにし、染色体異常を検出することができる。組織ISH法は、組織切片を用いて遺伝子発現の第一段階であるmRNAの局在を細胞レベルで明らかにする。病理組織からウイルスゲノムを検出し、ウイルス感染の診断に用いられることもある。また、胚や器官の一部などを丸ごと用いるISH法を、ホールマウントISH(WISH)という。実験例を図1に示す。遺伝子発現部位の三次元的な情報を得た後で、細胞レベルで遺伝子発現部位を同定しなければならない場合は、WISH後の胚などの組織切片を作製する。図2に、組織切片を用いたin situハイブリダイゼーション法の工程を模式的に示す。ISHは、細胞内mRNAの局在を明らかにする実験であるので、分解されやすいRNAをいかに分解させずに実験を行うかが重要である。
In situハイブリダイゼーション法の各工程
組織の固定
ISH法では細胞が死んだ状態でmRNAを検出する。細胞が死んでしまうと、mRNAは急速に分解され消滅してしまう。そのため、in situハイブリダイゼーション法を用いる場合は、細胞内のmRNAの分解を防ぎ、できるだけ生きていた状態を維持しておく必要がある。細胞や組織などをなるべく自然の状態に保存することを固定とよんでいるが、in situハイブリダイゼーション法の場合はmRNAの固定が非常に重要である。In situハイブリダイゼーション法では、用時調製した4%パラホルムアルデヒド液を用いて還流固定し、臓器等を取り出して短時間、低温でさらに浸漬固定することが多い。新鮮迅速凍結切片を用いてISHを行うこともある。組織切片はスライドガラスに貼付けたもの、あるいは浮遊切片[3]を用いる。
前処理
組織ISH法、WISH法のいずれにおいても、ハイブリダイゼーションを行う前に固定した組織の前処理を行う。主な目的は、界面活性剤で細胞膜の透過性を上げ、タンパク質分解酵素proteinase Kなどを用いて結合組織等を分解しプローブの浸透性をよくすることと、プローブの非特異的な吸着を防ぐことである。特に、タンパク質分解酵素の濃度と処理時間は、組織の種類、固定法とも関連し、in situハイブリダイゼーション法の結果を左右する重要な因子である。
プローブの調製
目的のmRNAを検出するために、そのmRNAと特異的にハイブリダイズする、つまり、目的のmRNAと相補的な配列をもったRNAまたはDNAプローブが必要である。プローブとして、
- 化学合成したDNAを用いるオリゴヌクレオチドプローブ
- DNA合成酵素によりin vitroで合成したDNAプローブ
- RNA合成酵素によりin vitroで合成したRNAプローブ
の3種類がよく用いられている。DNAオリゴプローブは、DNA合成装置で合成する。B, Cにおいては、プローブ合成のための鋳型DNAが必要である。RNA-RNAハイブリッドが3者の中で最も安定であり、現在RNAプローブを用いる方法が一般的である。合成したRNAが分解されないように細心の注意を払う。他にlocked nucleic acid (LNA)(後述)やペプチド核酸をプローブとして用いる方法がある。プローブを可視化のために標識する方法には主に次の2つの方法がある。
- 酵素抗体法または蛍光抗体法:適当な抗原(ジゴキシゲニン (digoxigenin, DIG), フルオレセイン (fluorescein), ビオチン (biotin)など)の結合したヌクレオチドを用いてプローブを標識し、その抗原に対する抗体を用いて発色または蛍光により可視化する。
- 放射性同位元素(radioisotope)を用いて、オートラジオグラフィー法により可視化する。
ハイブリダイゼーション
DNAの二重らせん構造は、塩基対A:Tに形成される2つの水素結合と塩基対G:Cに形成される3つの水素結合により安定に保たれている。この二本鎖を一本鎖にする方法の1つとして、熱変性がある。二重らせんDNA溶液の温度を高くしながら、DNA溶液の260 nmの吸光度A260を測定すると、しだいにA260は高くなる。これはDNAの二重らせんが壊れ、一本鎖になるためで、この温度による吸光度の変化を表す曲線をDNAの融解曲線とよんでいる。この現象は、らせんが消失して塩基間の相互作用が少なくなるため、塩基の光吸収の効率が変化し(深色効果)、各塩基の分子吸光係数が高くなるために生じる。温度の低い時のDNAをヘリックス100%とし、高温での吸光度が一定になる状態でヘリックス0%と仮定すると、ヘリックス50%になる温度(融解温度 (melting temperature: Tm))を決定することができる。
Tmは二重らせんの安定度の目安になる。非常に安定ならせんであれば、Tmは80〜90℃になる。逆に不安定であれば、30〜40℃になる。TmはGC塩基対の含量、核酸の長さ、核酸の塩基対のミスマッチなどに依存し、DNA溶液のイオン強度(塩濃度)や組成により変化する。Tmに関する経験的な式は、例えば、RNA-RNAハイブリッドの場合、
Tm=79.8+18.5(logM)+0.584(%G+C)+0.118 × 10-2(%G+C)2−0.35(%formamide)− 820/l
ここで、Mは1価の正イオンのモル濃度、%G+CはGC含量、%formamideはホルムアミド濃度、lはプローブの長さ(bp)である。
Tmの温度では、全体の50%ほど二重らせんが形成されていることになる。ハイブリダイゼーションの温度Thは、Tmより5〜20℃低い温度で行う。どの程度まで塩基対のマッチを許容するかをstringencyとよぶ。例えば、(Tm−5)℃でハイブリダイズさせると、stringencyが非常に高くなり、完全マッチした安定なDNA-DNAしかハイブリッドを形成しない。しかし、ハイブリダイズする温度を(Tm−20)℃と低下させ、stringencyを低くすると、ミスマッチをもったハイブリッドが形成される可能性が高くなる。
DIGを用いた組織in situハイブリダイゼーション法では、1kb程度のRNAプローブ(GC含量約50%)を使用する場合、50%ホルムアミド中65℃、12〜14時間で、ほぼよい結果が得られる。もし、短いRNAやGC含量の低いRNAをプローブとする場合は、低stringencyの条件でハイブリダイゼーションを行う。プローブの濃度も重要であり、濃度の高いほうが再会合率が高くなる。プローブの長さが大きいほどシグナルが強くなるが、プローブの配列によってはハイブリダイゼーションに交差性を認める結果となる場合もある。2kbをこえるとアルカリ加水分解によってRNAの長さを小さくし、組織への浸透を高めるようにする。
洗浄
非特異的に結合したプローブを除去するために、組織を洗浄する.洗浄の条件を厳しくするとバックグラウンドが低下するが、同時にシグナルも弱くなる。RNA-RNAハイブリットは通常のRNA分解酵素 (RNAse)では分解されないので、これ以降の実験工程はRNAseフリーでなくてよい。
シグナルの検出
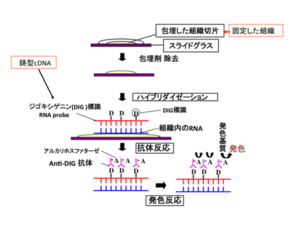
酵素抗体法と蛍光抗体法
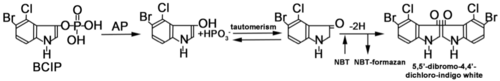
BCIPがAPにより加水分解されてまず中間産物となり、さらに2量体になって青色色素を産生する。2量体化するときに、2個の水素イオンによりNBTは還元されて不溶性のNBTホルマザンを形成する。(tautomerism:ケト・エノール互変異性)
一方、Fast Red TRは、ナフトールAS-MXリン酸の存在下でAPにより赤色の沈殿物を生じる。Fast Red TRを蛍光性のAP基質であるHNPP (2-hydroxy-3-naphtoic acid-2’-phenylanilide phosphate)と共に用いると、還元されてできるHNPの組織への沈着が増し、HNPの蛍光がさらに長波長側シフトして (565~620nm) 強い橙蛍光を発する。発色反応、発色基質全般についての説明。
標識プローブのみでは発色することができないので、プローブの中に取り込まれている抗原に対する抗体を用いプローブの検出を行う.プローブをDIGで標識した場合、DIGに特異的に結合する一次抗体を用いて検出する。検出に用いる一次抗体には通常の抗体とは異なり、アルカリホスファターゼ(alkaline phosphatase: AP)または西洋ワサビペルオキシダーゼ(horseradish peroxidase: HRP)などの酵素が結合(conjugate)している.例えば、抗体に結合されたAPの基質として、5-Bromo-4-Chloro-3'-Indolylphosphate p-Toluidine salt (BCIP)およびnitro-blue tetrazolium chloride (NBT)を用いた場合は、APがBCIPを脱リン酸化し、中間産物が遊離すると発色する(図3)。一方、抗体に蛍光色素が結合している場合には、蛍光顕微鏡や共焦点レーザー走査顕微鏡で検出する。
放射性同位元素を用いる方法
放射性同位元素(RI)として35S標識された核酸を用いてプローブを合成する。写真用フイルムまたはオートラジオグラフィー用の乳剤を用いてRIの放出するβ線を検出する。
観察と記録
組織切片の場合、色素で対比染色し形態学的な観察を容易にする。結果を顕微鏡で観察し、さらに写真や画像として記録する。
2色ISH法、蛍光ISH法、抗体染色との2重染色
発色法では、アルカリフォスファターゼ (AP) の基質を変えることにより、2つの遺伝子の発現を同時に検出できる。すなわち、調べたい2つの遺伝子のRNAプローブをDIGまたはフルオレセインで標識し、混合プローブでハイブリダイゼーションさせる。まずAP標識抗フルオレセイン抗体で反応させ、洗浄、発色まで行う(基質はFast Red)。その後、酸処理(0.1Mグリシン-HCl、pH2.2)および後固定を行って1色めのアルカリフォスファターゼを失活させる。その後、AP標識抗DIG抗体で反応させ、洗浄、発色を行う(基質NBT/BCIP)。または、DIGまたはフルオレセインで標識した2つのRNAプローブでハイブリダイゼーションさせて洗浄、HRP標識抗DIG抗体で反応させ、HRPの基質ジアミノベンジジン(Diaminobenzidine: DAB)を用いてまず発色させる。次に、AP標識抗フルオレセイン抗体で反応させ、その後APの基質(NBT, BCIP)を用いて発色させる。
蛍光ISH法では、HRP標識の抗体に対して、tyramideを結合させた低分子(ビオチンやフルオレセインなど)を反応させシグナルを増強させる方法 (tyramide signal amplification: TSA) を用いる。すなわちtyramideは、ペルオキシダーゼ活性によってラジカル化し組織(タンパク質のTyr, Trp残基など)に低分子ごと集積する。集積した低分子を、蛍光物質の場合はそのまま蛍光顕微鏡等で検出でき、そうでなければその低分子に対する蛍光標識抗体またはストレプトアビジンを反応させて間接的に検出することもできる。細胞レベルでmRNAの共在性を証明するためには蛍光ISH法は必須であり、AP反応系Fast Red/HNPPと組み合わせることで2重蛍光ISH法を行うことができる。抗体染色との2重染色では、以上のISH工程をタンパク質分解酵素なしで行ってそのあと通常の間接蛍光抗体染色を行う、あるいは先にビオチン化抗体と蛍光標識アビジンを用いてRNAseフリーで抗体染色を行い、その後AP反応系Fast Red/HNPPによるISH法を行うなどの方法がある。さらなる多重蛍光ISH法については、hybridization chain reaction法がある。
マイクロRNAを検出するISH法
Locked nucleic acid (LNA)プローブを用いる。LNAは、リボ核酸の2’位の酸素原子と4’位の炭素原子とが架橋しており、相補的な核酸に対合したときの熱安定性が非常に高く、短いRNAに対して高いTm値でハイブリダイゼーションできる。この性質を利用してLNAプローブによりマイクロRNAのISHが可能になった。
ISHデータベース
Allen Brain Atlasは、マイクロソフト社創設者の一人であるPaul G. Allenの出資によって2003年に設立されたAllen Institute for Brain ScienceのISHデータベースである。2006年12月発表のNature の記事によると、まず約2万の遺伝子のマウス成体脳における組織切片ISHのデータが公開された[4]。現在、マウス脳に加えて、ヒト脳、発生期マウス脳、発生期ヒト脳、マウス脳神経回路、ヒト以外の霊長類脳、マウス脊髄、ヒト神経膠芽腫に関するISHデータベースが公開されている。
関連項目
外部リンク
- Allen Brain Atlas
- Roche社のジゴキシゲニンの説明。
- Roche社の発色基質(HNPP, Fast Red)の説明。
- Perkin-Elmer社のチラミドを用いたISHとシグナル増強(CARD)の説明。
- コスモバイオ社のマイクロRNAのISH (locked nucleic acid)の説明。
- 基礎生物学研究所山森研究室(渡我部昭哉博士作成)ホームページ BraInSitu - 脳の組織切片ISH法(特に浮遊法)について詳細なプロトコールが紹介されている。
- Pierce社の発色反応、発色基質全般についての説明。
参考文献
- ↑ 野地澄晴編
免疫染色&in situハイブリダイゼーション
羊土社、東京、2006 - ↑ D.G. Wilkinson
In Situ Hybridization, A Practical Approach, 2nd Edition
Oxford University Press, Oxford, 1999 - ↑ <pubmed> 21037591</pubmed>
- ↑
Lein, E.S., Hawrylycz, M.J., Ao, N., Ayres, M., Bensinger, A., Bernard, A., ..., & Jones, A.R. (2007).
Genome-wide atlas of gene expression in the adult mouse brain. Nature, 445(7124), 168-76. [PubMed:17151600] [WorldCat] [DOI]