「アルゴノート」の版間の差分
細 →関連用語 |
|||
| 3行目: | 3行目: | ||
{{box|text= アルゴノートは、PAZドメインやPIWIドメインなど特徴的なドメインから成るタンパク質で、20-30塩基長の小分子RNAを介して標的とする遺伝子の転写産物(ガイドRNA)に結合しRNA誘導型サイレンシング複合体(RNA-induced silencing complex, RISC)を形成し、遺伝子の発現を抑制する。多くの生物はアルゴノートを複数持ち、各メンバーは発現する組織の違いによってAGOサブファミリーとPIWIサブファミリーに分類される。AGOサブファミリーメンバーは全ての組織で発現する一方、PIWIサブファミリーメンバーは生殖組織特異的に発現する。AGOサブファミリーメンバーと結合して機能する小分子RNAとしては、マイクロRNA(microRNA, miRNA)やsmall interfering RNA(siRNA)がある。PIWIサブファミリーメンバーに結合する小分子RNAは、PIWI-interacting RNA(piRNA)と称される。アルゴノートの機能は生体の恒常性維持に必須で、その機能欠損は、知的障害、がん、不妊などの原因となる。}} | {{box|text= アルゴノートは、PAZドメインやPIWIドメインなど特徴的なドメインから成るタンパク質で、20-30塩基長の小分子RNAを介して標的とする遺伝子の転写産物(ガイドRNA)に結合しRNA誘導型サイレンシング複合体(RNA-induced silencing complex, RISC)を形成し、遺伝子の発現を抑制する。多くの生物はアルゴノートを複数持ち、各メンバーは発現する組織の違いによってAGOサブファミリーとPIWIサブファミリーに分類される。AGOサブファミリーメンバーは全ての組織で発現する一方、PIWIサブファミリーメンバーは生殖組織特異的に発現する。AGOサブファミリーメンバーと結合して機能する小分子RNAとしては、マイクロRNA(microRNA, miRNA)やsmall interfering RNA(siRNA)がある。PIWIサブファミリーメンバーに結合する小分子RNAは、PIWI-interacting RNA(piRNA)と称される。アルゴノートの機能は生体の恒常性維持に必須で、その機能欠損は、知的障害、がん、不妊などの原因となる。}} | ||
[[ファイル:Shiomi Argonaute Fig1.png|サムネイル|'''図1. アルゴノートのドメイン構造'''<br>アルゴノートは、N、PAZ、 MID、PIWIの4つのドメインと、L1(Linker-1)、L2(Linker- 2)の2つのリンカーからなる。]] | |||
[[ファイル:Shiomi Argonaute Fig2.png|サムネイル|'''図2. ショウジョウバエとマウスのmiRNA機構とRNA干渉'''<br>ショウジョウバエAGO1とヒトAGO2はエンドヌクレアーぜ活性を持つが、miRNAの中央部分と標的mRNAの対合性が不完全であるためエンドヌクレアーぜ活性を発揮できない(グレー のハサミ)。ヒトAGO2はsiRNAと結合してRNA干渉において 機能することもある。ショウジョウバエAGO2は、RNA干渉に 特化している。]] | |||
==アルゴノートとは== | ==アルゴノートとは== | ||
アルゴノートとは、PAZドメインやPIWIドメインなど特徴的なドメインを持つ一群のタンパク質である('''図1''')。 | アルゴノートとは、PAZドメインやPIWIドメインなど特徴的なドメインを持つ一群のタンパク質である('''図1''')。 | ||
2023年4月23日 (日) 14:40時点における版
英語:Argonaute
アルゴノートは、PAZドメインやPIWIドメインなど特徴的なドメインから成るタンパク質で、20-30塩基長の小分子RNAを介して標的とする遺伝子の転写産物(ガイドRNA)に結合しRNA誘導型サイレンシング複合体(RNA-induced silencing complex, RISC)を形成し、遺伝子の発現を抑制する。多くの生物はアルゴノートを複数持ち、各メンバーは発現する組織の違いによってAGOサブファミリーとPIWIサブファミリーに分類される。AGOサブファミリーメンバーは全ての組織で発現する一方、PIWIサブファミリーメンバーは生殖組織特異的に発現する。AGOサブファミリーメンバーと結合して機能する小分子RNAとしては、マイクロRNA(microRNA, miRNA)やsmall interfering RNA(siRNA)がある。PIWIサブファミリーメンバーに結合する小分子RNAは、PIWI-interacting RNA(piRNA)と称される。アルゴノートの機能は生体の恒常性維持に必須で、その機能欠損は、知的障害、がん、不妊などの原因となる。
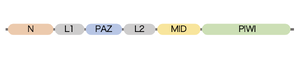
アルゴノートは、N、PAZ、 MID、PIWIの4つのドメインと、L1(Linker-1)、L2(Linker- 2)の2つのリンカーからなる。
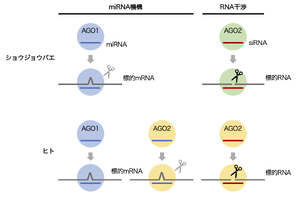
ショウジョウバエAGO1とヒトAGO2はエンドヌクレアーぜ活性を持つが、miRNAの中央部分と標的mRNAの対合性が不完全であるためエンドヌクレアーぜ活性を発揮できない(グレー のハサミ)。ヒトAGO2はsiRNAと結合してRNA干渉において 機能することもある。ショウジョウバエAGO2は、RNA干渉に 特化している。
アルゴノートとは
アルゴノートとは、PAZドメインやPIWIドメインなど特徴的なドメインを持つ一群のタンパク質である(図1)。
シロイヌナズナ(Arabidopsis thaliana)の、ある遺伝子の変異による表現型がアオイガイ(Argonauta argo)に似ていたことから、この遺伝子はアルゴノート1(Argonaute 1, AGO1)と名付けられた[1] [1]。AGO1は植物に限らず多くの生物で保存されている(表)。AGO1は、マイクロRNA(microRNA, miRNA)と特異的に結合し、RNA誘導型サイレンシング複合体(RNA-induced silencing complex, RISC)を形成する[2][3][4] [2-4]。RISC内のmiRNAは、高い配列相補性を示す内在性の伝令RNA(mRNA)と対合することによってAGO1を標的mRNAに運び、mRNAの不安定性や翻訳阻害を促進することで、タンパク質合成を抑制する[4][3][2] [2-4](図2)。
ショウジョウバエのRNA干渉(RNA interference, RNAi)の中核因子として同定されたアルゴノートは、ショウジョウバエのAGO1や線虫(Caenorhabditis elegans)のRDE1(AGO1ホモログ)と高い相同性を示したことから、アルゴノート2(AGO2)と名付けられた[5] [5]。AGO2は、RNA干渉において機能する小分子RNAであるsmall interfering RNA(siRNA)とRISCを形成する[6][7][8][6-8](図2)。RISC内のsiRNAは、高い配列相補性を示すRNAと対合することによってAGO2を標的RNA(mRNAに限らない)に運び[6][7][8][6-8]、標的RNAを切断することで遺伝子発現や機能を抑制する[6][7][8] [6-8]。
ショウジョウバエの雄の生殖幹細胞維持に必須な因子として同定されたP-element wimpy testis(Piwi)は、配列相同性からアルゴノートのパラログであると報告された[9][9]。ショウジョウバエはPiwiパラログを3つ持ち、Piwi以外の2つはAubergine、アルゴノート3(AGO3)と名付けられている[10][11] [10, 11](表)。Piwi同様、Aubergine、AGO3の発現も生殖組織に限られており、恒常的に発現するAGO1やAGO2と区別するために、PIWIサブファミリーメンバーと呼ばれる[10][11] [10, 11]。PIWIサブファミリーメンバーのガイドRNAは、PIWI-interacting RNA(piRNA)と称される[10][11] [10, 11]。piRNAの多くはトランスポゾンと高い配列相補性を示す。PIWIメンバーは、標的トランスポゾンの転写産物(RNA)を細胞質で切断する、あるいは核でトランスポゾンの転写を阻害することによってその発現を抑制する[6][7][8][6-8]。
アルゴノートを持つ細菌も存在する。細菌のアルゴノートの中には、小分子RNAではなく、小分子DNAをガイドとするものがある。また、RNAを標的とするものと、DNAを標的とするものがある[12][12]。
サブファミリー
アルゴノートは、発現する組織の違いによってAGOサブファミリーとPIWIサブファミリーに分類される(表)。AGOサブファミリーメンバーは生殖組織を含む全ての組織で、PIWIサブファミリーメンバーは生殖組織(生殖細胞とそれを取り囲む生殖系体細胞)で特異的に発現する。それぞれのサブファミリーに属するアルゴノートの数は生物によって異なる。
ショウジョウバエは5種類のアルゴノートを持ち、AGO1とAGO2はAGOサブファミリーに、Piwi、Aubergine、AGO3はPIWIサブファミリーに分類される[10][11][13] [10, 11, 13]。ヒトは8種類のアルゴノートを持つが[14][15] [14, 15]、そのうちの4つ(AGO1、AGO2、AGO3、AGO4)はAGOサブファミリーに、残りの4つ(PIWIL1/HIWI、PIWIL2/HILI、PIWIL3、PIWIL4/HIWI2)はPIWIサブファミリーに属する[14][10] [10, 15] 。マウスは、4つのAGOメンバー(AGO1、AGO2、AGO3、AGO4)と、3つのPIWIメンバー(PIWIL1/MIWI、PIWIL2/MILI、PIWIL4/MIWI2)を持つ[14][10] [10, 15]。マウスのPIWIは全て雄特異的であることを特徴とする。
線虫は27種類のアルゴノートを持ち、そのうちの5メンバーはAGOサブファミリーに、3メンバーはPIWIサブファミリーに属する。残りのアルゴノートは線虫特異的でWAGO(Worm-specific Argonaute)と称される[16] [16]。
| AGOサブファミリー | PIWIサブファミリー | |
|---|---|---|
| ショウジョウバエ | AGO1 AGO2 | Piwi Aubergine AGO3 |
| マウス | AGO1 AGO2 AGO3 AGO4 | MIWI(PIWIL1) MILI(PIWIL2) MIWI2(PIWIL4) |
| ヒト | AGO1 AGO2 AGO3 AGO4 | HIWI(PIWIL1) HILI(PIWIL2) PIWIL3 HIWI2(PIWIL4) |
ドメイン構造と立体構造
全てのアルゴノートは、N、PAZ、MID、PIWIという4つの主要ドメインと、2つのリンカー(Linker-1とLinker-2)を持つ[14][17] [15, 17] (図1)。
NドメインはRISC形成に寄与する。PAZドメインはガイド(小分子)RNAの3’末端に、MIDドメインは5’末端に結合する。PIWIドメインはRNaseH様構造をとっており、アルゴノートが標的RNAを切断するためのエンドヌクレアーゼ活性(スライサー活性ともいう)を担う[17][18] [17, 18]。その活性中心に相当するアスパラギン酸-グルタミン酸-アスパラギン酸-ヒスチジン(Asp-Glu-Asp-His)の4つのアミノ酸を変異させると、RNA切断活性を示さなくなる。但し、ヒトのAGO3はこの4つのアミノ酸を持つにもかかわらず、エンドヌクレアーゼ活性を示さない。
最初に立体構造が解明されたアルゴノートは、超高熱古細菌(Pyrococcus furiosus)のAGOである[18] [18]。ヒトの4種類全てのAGOと、ショウジョウバエPiwi、カイコ(Bombyx mori)Piwiホモログ(Siwi)の立体構造も既に解かれている[19][17][20] [17, 19, 20]。
細胞内局在と機能
アルゴノートには、細胞質で機能するものと核で機能するものがある。いずれの場合も、ガイド(小分子)RNAとの結合、つまりRISC形成は細胞質で起こる[2][10] [2, 10]。RISCとなったアルゴノートの細胞内局在は、アルゴノートが核局在シグナル(nuclear localization signal, NLS)を持つか、持たないかによって決まる。NLSを持たないアルゴノートは、細胞質で標的RNAを切断するか、RNAの不安定化や翻訳阻害を促進することによって標的RNAからのタンパク質合成を阻害する。NLSを持つアルゴノートは核で標的遺伝子の転写を阻害する。
AGOサブファミリー
細胞質での機能
AGO1
AGO1は、miRNAを介して標的となる内在性mRNAに結合し、その不安定化や翻訳阻害を促進する(図2)。
ショウジョウバエのAGO1はエンドヌクレアーゼ活性を持つが、miRNAの中央部分(5’末端から9-12番目)の標的RNAへの対合性が不完全なため、標的RNAを切断することができない。よって、GW182などの補因子と協調して目的を達成する[21][22] [21, 22]。GW182はポリA鎖短縮化などに関わる因子をAGO1の反応場にリクルートする。
ヒトやマウスなど哺乳動物のAGO1は、元々エンドヌクレアーゼ活性を持たないため、ショウジョウバエのAGO1と同様に、TNRC6(GW182ホモログ)等と協調してRNA不安定化や翻訳阻害を促進する。哺乳動物のAGO3とAGO4は、AGO1と同様にエンドヌクレアーゼ活性を持たず、miRNAと結合して細胞質で機能する。
AGO2
AGO2は、siRNAと結合してRNA干渉で機能する(図2)。
ショウジョウバエのAGO2はRNA干渉に特化しているが、哺乳動物のAGO2は、通常、miRNAと結合してmiRNA機構で機能する。siRNA(あるいはその前駆体)の導入により哺乳動物細胞でRNA干渉を人工的に誘導すると、siRNAはAGO2と結合し、RNA干渉で機能する様になる。
AGO2はエンドヌクレアーゼ活性を持つが、miRNAと結合した哺乳動物のAGO2は、ショウジョウバエAGO1の場合と同様に標的RNAを切断することができないため、TNRC6等と協調してRNA不安定化や翻訳阻害を促進する[21, 22]。
核での機能
哺乳動物のAGOは、核に局在し、転写後レベルで標的遺伝子の発現を抑制する場合もある[23] [23]。分裂酵母(Schizosaccharomyces pombe)のAGO1は、核内でヘテロクロマチンを誘導することによって標的遺伝子の発現を転写レベルで抑制する[24] [24]。
PIWIサブファミリーメンバー
細胞質での機能
ショウジョウバエAubergineに代表されるNLSを持たないPIWIメンバーは、RISC形成後も細胞質に局在し、piRNAを介して結合したトランスポゾンmRNAをエンドヌクレアーゼ活性依存的に切断することによってトランスポゾンの発現を抑制する。その分子機序はAGO2の分子機序とよく似ている。AGO2によって切断されたRNA断片は細胞質で分解される運命にあるが、Aubergineによって切断されたRNA断片からはpiRNAが生成されAGO3と結合する[25][26][27] [25-27]。これらpiRNAはトランスポゾンmRNAから生成されるため、AGO3はトランスポゾンのアンチセンス方向の転写産物を切断する。アンチセンス転写産物RNA断片から生成されたpiRNAはAubergineと結合する。
このAubergineとAGO3による反応は相互に連続して起こり、piRNAを増幅させるためpiRNA増幅機構と呼ばれる。また、その様相からピンポン機構としても知られる。AGO2と異なり、AubergineとAGO3は切断後もRNA断片を保持し続ける。しかし、このままではpiRNA増幅が停滞してしまうため、VasaなどのRNAヘリカーゼが、頃を見計らってエネルギーを消費しつつPIWIからRNA断片を解離する[28][29] [28, 29]。ショウジョウバエ生殖系体細胞では、脳腫瘍抑制因子L(3)mbtがpiRNA増幅因子の発現を抑制しているためpiRNAを増幅しない[30] [30]。マウスMILIは、トランスポゾンのアンチセンスRNAを切断することによってMIWI2に結合するpiRNAを産生する[10] [10]。これによってMIWI2はトランスポゾンのmRNAを標的とすることが可能になる。
核での機能
マウスMIWI2やショウジョウバエPiwiに代表されるNLSを持つPIWIは、細胞質でRISCとなった後に核に移行し、転写中のRNA、つまり遺伝子ゲノムに結合した状態にあるRNAに結合して、そのゲノム領域のDNAやヒストンのメチル化を介してヘテロクロマチンを誘導する[24] [24]。この機構には、DNAメチル化酵素やヒストンメチル化酵素など多くの補因子が関わる。
生体での機能
AGOサブファミリー
ヒトAGO2やショウジョウバエAGO1は、miRNAに配列相補的な内在性遺伝子を抑制することにより、個体発生や分化、生体恒常性を制御する。線虫のRDE1(AGO1ホモログ)やショウジョウバエのAGO2は、miRNAではなく、ウイルスなど外来性核酸から生成されたsiRNAと結合し、ウイルス由来のRNAなどを切断して感染から身を守る、つまり一種の免疫機構として機能する[2][10] [2, 10]。線虫のRDE1(AGO1ホモログ)やショウジョウバエのAGO2はトランスポゾン由来のsiRNAとも結合する[31][32] [31, 32]。ショウジョウバエや線虫などの下等動物では、miRNA機構とRNA干渉(siRNA機構)が独立して存在するが、ヒトなどの高等動物ではsiRNA機構が存在しない。
PIWIサブファミリー
PIWIは生殖組織においてトランスポゾンの発現を抑制し、トランスポゾン転移によるDNA損傷から生殖ゲノムを保護する[10][11] [10, 11]。PIWIやpiRNAの機能が破綻すると、卵子や精子の形成不全が起こり、最終的には不妊(不稔)へと至る。万が一、不妊を回避できたとしても、高い確率で異常な遺伝子を子孫に受け継ぐことになる。ショウジョウバエのPIWIサブファミリーメンバーであるPiwi、Aubergine、AGO3は、いずれもpiRNAと結合し、トランスポゾンの発現を抑制する。各々のPIWIメンバーに結合するpiRNAは異なる特徴を示す。また各々のPIWIメンバーの作用点(細胞内局在など)が異なるため[10][11] [10, 11]、いずれのPIWIメンバーを欠失しても不妊(不稔)となる[9][33][34] [9, 33, 34]。PIWIメンバー間の機能的な代償がないことは、マウスでも示されている[35][10] [10, 35]。
プラナリアもPIWIを持ち、piRNAとRISCを形成してトランスポゾンの発現を抑制する[36] [36]。その機能は体性幹細胞維持に必須で再生に欠かせない。アメフラシの神経細胞では、piRNA機構が記憶の維持に重要な役割を果たす[37] [37]。脳腫瘍抑制因子L(3)mbtを欠損したショウジョウバエ脳では、Piwiを含むPIWIメンバーやpiRNA増幅因子、piRNAが異所的に発現するが、Piwiの発現を強制的に抑制すると腫瘍化が解除される[38] [38]。
活性調節機構
アルゴノートの機能は、リン酸化などの翻訳後修飾によって調節される[39][40][41] [39-41]。AGOのユビキチン化は、標的RNA依存的miRNA分解(target-directed miRNA degradation, TDMD)に関わる[40] [40]。
機能欠損と疾患
ヒトの4種類のAGOメンバーと結合するmiRNAは高い割合で重複するため、AGOメンバー間の機能に重複があると推察された[42] [42]。しかし、例えばAGO3の機能喪失は、知的障害を発症することが報告されており、AGOメンバー間の機能的代償は部分的であるといえる。ヒトのAGO1およびAGO3遺伝子を含むゲノム領域の欠失は、神経認知障害、発達遅延、知的障害、骨年齢遅延等に関与する可能性が示唆されている[43] [43]。また、ヒトのAGO1遺伝子の変異は、自閉症スペクトラム障害や知的障害に関連することが示唆されている[44] [44]。miRNAの機能と、がんなどの疾患との相関に関しては多くの報告がある[45] [45]。PIWIとpiRNAの機能欠損は不妊症を導く[46] [46]。
関連用語
参考文献
- ↑
Bohmert, K., Camus, I., Bellini, C., Bouchez, D., Caboche, M., & Benning, C. (1998).
AGO1 defines a novel locus of Arabidopsis controlling leaf development. The EMBO journal, 17(1), 170-80. [PubMed:9427751] [PMC] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2 2.3
Kim, V.N., Han, J., & Siomi, M.C. (2009).
Biogenesis of small RNAs in animals. Nature reviews. Molecular cell biology, 10(2), 126-39. [PubMed:19165215] [WorldCat] [DOI] - ↑ 3.0 3.1
Guo, H., Ingolia, N.T., Weissman, J.S., & Bartel, D.P. (2010).
Mammalian microRNAs predominantly act to decrease target mRNA levels. Nature, 466(7308), 835-40. [PubMed:20703300] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1
Bartel, D.P. (2018).
Metazoan MicroRNAs. Cell, 173(1), 20-51. [PubMed:29570994] [PMC] [WorldCat] [DOI] - ↑
Hammond, S.M., Boettcher, S., Caudy, A.A., Kobayashi, R., & Hannon, G.J. (2001).
Argonaute2, a link between genetic and biochemical analyses of RNAi. Science (New York, N.Y.), 293(5532), 1146-50. [PubMed:11498593] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2 6.3
Liu, J., Carmell, M.A., Rivas, F.V., Marsden, C.G., Thomson, J.M., Song, J.J., ..., & Hannon, G.J. (2004).
Argonaute2 is the catalytic engine of mammalian RNAi. Science (New York, N.Y.), 305(5689), 1437-41. [PubMed:15284456] [WorldCat] [DOI] - ↑ 7.0 7.1 7.2 7.3
Meister, G., Landthaler, M., Patkaniowska, A., Dorsett, Y., Teng, G., & Tuschl, T. (2004).
Human Argonaute2 mediates RNA cleavage targeted by miRNAs and siRNAs. Molecular cell, 15(2), 185-97. [PubMed:15260970] [WorldCat] [DOI] - ↑ 8.0 8.1 8.2 8.3
Miyoshi, K., Tsukumo, H., Nagami, T., Siomi, H., & Siomi, M.C. (2005).
Slicer function of Drosophila Argonautes and its involvement in RISC formation. Genes & development, 19(23), 2837-48. [PubMed:16287716] [PMC] [WorldCat] [DOI] - ↑ 9.0 9.1
Cox, D.N., Chao, A., Baker, J., Chang, L., Qiao, D., & Lin, H. (1998).
A novel class of evolutionarily conserved genes defined by piwi are essential for stem cell self-renewal. Genes & development, 12(23), 3715-27. [PubMed:9851978] [PMC] [WorldCat] [DOI] - ↑ 10.00 10.01 10.02 10.03 10.04 10.05 10.06 10.07 10.08 10.09 10.10 10.11
Siomi, M.C., Sato, K., Pezic, D., & Aravin, A.A. (2011).
PIWI-interacting small RNAs: the vanguard of genome defence. Nature reviews. Molecular cell biology, 12(4), 246-58. [PubMed:21427766] [WorldCat] [DOI] - ↑ 11.0 11.1 11.2 11.3 11.4 11.5
Yamashiro, H., & Siomi, M.C. (2018).
PIWI-Interacting RNA in Drosophila: Biogenesis, Transposon Regulation, and Beyond. Chemical reviews, 118(8), 4404-4421. [PubMed:29281264] [WorldCat] [DOI] - ↑
Lisitskaya, L., Aravin, A.A., & Kulbachinskiy, A. (2018).
DNA interference and beyond: structure and functions of prokaryotic Argonaute proteins. Nature communications, 9(1), 5165. [PubMed:30514832] [PMC] [WorldCat] [DOI] - ↑
Williams, R.W., & Rubin, G.M. (2002).
ARGONAUTE1 is required for efficient RNA interference in Drosophila embryos. Proceedings of the National Academy of Sciences of the United States of America, 99(10), 6889-94. [PubMed:12011447] [PMC] [WorldCat] [DOI] - ↑ 14.0 14.1 14.2 14.3
Hutvagner, G., & Simard, M.J. (2008).
Argonaute proteins: key players in RNA silencing. Nature reviews. Molecular cell biology, 9(1), 22-32. [PubMed:18073770] [WorldCat] [DOI] - ↑
Sasaki, T., Shiohama, A., Minoshima, S., & Shimizu, N. (2003).
Identification of eight members of the Argonaute family in the human genome. Genomics, 82(3), 323-30. [PubMed:12906857] [WorldCat] [DOI] - ↑
Ketting, R.F., & Cochella, L. (2021).
Concepts and functions of small RNA pathways in C. elegans. Current topics in developmental biology, 144, 45-89. [PubMed:33992161] [WorldCat] [DOI] - ↑ 17.0 17.1 17.2
Nakanishi, K. (2022).
Anatomy of four human Argonaute proteins. Nucleic acids research, 50(12), 6618-6638. [PubMed:35736234] [PMC] [WorldCat] [DOI] - ↑ 18.0 18.1
Song, J.J., Smith, S.K., Hannon, G.J., & Joshua-Tor, L. (2004).
Crystal structure of Argonaute and its implications for RISC slicer activity. Science (New York, N.Y.), 305(5689), 1434-7. [PubMed:15284453] [WorldCat] [DOI] - ↑
Matsumoto, N., Nishimasu, H., Sakakibara, K., Nishida, K.M., Hirano, T., Ishitani, R., ..., & Nureki, O. (2016).
Crystal Structure of Silkworm PIWI-Clade Argonaute Siwi Bound to piRNA. Cell, 167(2), 484-497.e9. [PubMed:27693359] [WorldCat] [DOI] - ↑
Yamaguchi, M., Chan, J.C.L., Moon, I.J., Yoshida, K., & Mizuta, R. (2020).
Global warming changes tropical cyclone translation speed. Nature communications, 11(1), 47. [PubMed:31913276] [PMC] [WorldCat] [DOI] - ↑
Gregory, R.I., Chendrimada, T.P., Cooch, N., & Shiekhattar, R. (2005).
Human RISC couples microRNA biogenesis and posttranscriptional gene silencing. Cell, 123(4), 631-40. [PubMed:16271387] [WorldCat] [DOI] - ↑
Jonas, S., & Izaurralde, E. (2015).
Towards a molecular understanding of microRNA-mediated gene silencing. Nature reviews. Genetics, 16(7), 421-33. [PubMed:26077373] [WorldCat] [DOI] - ↑
Sarshad, A.A., Juan, A.H., Muler, A.I.C., Anastasakis, D.G., Wang, X., Genzor, P., ..., & Hafner, M. (2018).
Argonaute-miRNA Complexes Silence Target mRNAs in the Nucleus of Mammalian Stem Cells. Molecular cell, 71(6), 1040-1050.e8. [PubMed:30146314] [PMC] [WorldCat] [DOI] - ↑ 24.0 24.1
Onishi, R., Yamanaka, S., & Siomi, M.C. (2021).
piRNA- and siRNA-mediated transcriptional repression in Drosophila, mice, and yeast: new insights and biodiversity. EMBO reports, 22(10), e53062. [PubMed:34347367] [PMC] [WorldCat] [DOI] - ↑
Brennecke, J., Aravin, A.A., Stark, A., Dus, M., Kellis, M., Sachidanandam, R., & Hannon, G.J. (2007).
Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila. Cell, 128(6), 1089-103. [PubMed:17346786] [WorldCat] [DOI] - ↑
De Fazio, S., Bartonicek, N., Di Giacomo, M., Abreu-Goodger, C., Sankar, A., Funaya, C., ..., & O'Carroll, D. (2011).
The endonuclease activity of Mili fuels piRNA amplification that silences LINE1 elements. Nature, 480(7376), 259-63. [PubMed:22020280] [WorldCat] [DOI] - ↑
Gunawardane, L.S., Saito, K., Nishida, K.M., Miyoshi, K., Kawamura, Y., Nagami, T., ..., & Siomi, M.C. (2007).
A slicer-mediated mechanism for repeat-associated siRNA 5' end formation in Drosophila. Science (New York, N.Y.), 315(5818), 1587-90. [PubMed:17322028] [WorldCat] [DOI] - ↑
Nishida, K.M., Iwasaki, Y.W., Murota, Y., Nagao, A., Mannen, T., Kato, Y., ..., & Siomi, M.C. (2015).
Respective functions of two distinct Siwi complexes assembled during PIWI-interacting RNA biogenesis in Bombyx germ cells. Cell reports, 10(2), 193-203. [PubMed:25558067] [WorldCat] [DOI] - ↑
Xiol, J., Spinelli, P., Laussmann, M.A., Homolka, D., Yang, Z., Cora, E., ..., & Pillai, R.S. (2014).
RNA clamping by Vasa assembles a piRNA amplifier complex on transposon transcripts. Cell, 157(7), 1698-711. [PubMed:24910301] [WorldCat] [DOI] - ↑
Sumiyoshi, T., Sato, K., Yamamoto, H., Iwasaki, Y.W., Siomi, H., & Siomi, M.C. (2016).
Loss of l(3)mbt leads to acquisition of the ping-pong cycle in Drosophila ovarian somatic cells. Genes & development, 30(14), 1617-22. [PubMed:27474440] [PMC] [WorldCat] [DOI] - ↑
Kawamura, Y., Saito, K., Kin, T., Ono, Y., Asai, K., Sunohara, T., ..., & Siomi, H. (2008).
Drosophila endogenous small RNAs bind to Argonaute 2 in somatic cells. Nature, 453(7196), 793-7. [PubMed:18463636] [WorldCat] [DOI] - ↑
Tabara, H., Sarkissian, M., Kelly, W.G., Fleenor, J., Grishok, A., Timmons, L., ..., & Mello, C.C. (1999).
The rde-1 gene, RNA interference, and transposon silencing in C. elegans. Cell, 99(2), 123-32. [PubMed:10535731] [WorldCat] [DOI] - ↑
Harris, A.N., & Macdonald, P.M. (2001).
Aubergine encodes a Drosophila polar granule component required for pole cell formation and related to eIF2C. Development (Cambridge, England), 128(14), 2823-32. [PubMed:11526087] [WorldCat] [DOI] - ↑
Li, C., Vagin, V.V., Lee, S., Xu, J., Ma, S., Xi, H., ..., & Zamore, P.D. (2009).
Collapse of germline piRNAs in the absence of Argonaute3 reveals somatic piRNAs in flies. Cell, 137(3), 509-21. [PubMed:19395009] [PMC] [WorldCat] [DOI] - ↑
Siomi, M.C., & Kuramochi-Miyagawa, S. (2009).
RNA silencing in germlines--exquisite collaboration of Argonaute proteins with small RNAs for germline survival. Current opinion in cell biology, 21(3), 426-34. [PubMed:19303759] [WorldCat] [DOI] - ↑
Shibata, N., Kashima, M., Ishiko, T., Nishimura, O., Rouhana, L., Misaki, K., ..., & Agata, K. (2016).
Inheritance of a Nuclear PIWI from Pluripotent Stem Cells by Somatic Descendants Ensures Differentiation by Silencing Transposons in Planarian. Developmental cell, 37(3), 226-37. [PubMed:27165555] [WorldCat] [DOI] - ↑
Rajasethupathy, P., Antonov, I., Sheridan, R., Frey, S., Sander, C., Tuschl, T., & Kandel, E.R. (2012).
A role for neuronal piRNAs in the epigenetic control of memory-related synaptic plasticity. Cell, 149(3), 693-707. [PubMed:22541438] [PMC] [WorldCat] [DOI] - ↑
Janic, A., Mendizabal, L., Llamazares, S., Rossell, D., & Gonzalez, C. (2010).
Ectopic expression of germline genes drives malignant brain tumor growth in Drosophila. Science (New York, N.Y.), 330(6012), 1824-7. [PubMed:21205669] [WorldCat] [DOI] - ↑
Shen, J., Xia, W., Khotskaya, Y.B., Huo, L., Nakanishi, K., Lim, S.O., ..., & Hung, M.C. (2013).
EGFR modulates microRNA maturation in response to hypoxia through phosphorylation of AGO2. Nature, 497(7449), 383-7. [PubMed:23636329] [PMC] [WorldCat] [DOI] - ↑ 40.0 40.1
Han, J., LaVigne, C.A., Jones, B.T., Zhang, H., Gillett, F., & Mendell, J.T. (2020).
A ubiquitin ligase mediates target-directed microRNA decay independently of tailing and trimming. Science (New York, N.Y.), 370(6523). [PubMed:33184234] [PMC] [WorldCat] [DOI] - ↑
Nishida, K.M., Sakakibara, K., Sumiyoshi, T., Yamazaki, H., Mannen, T., Kawamura, T., ..., & Siomi, M.C. (2020).
Siwi levels reversibly regulate secondary piRISC biogenesis by affecting Ago3 body morphology in Bombyx mori. The EMBO journal, 39(20), e105130. [PubMed:32914505] [PMC] [WorldCat] [DOI] - ↑
Hafner, M., Landthaler, M., Burger, L., Khorshid, M., Hausser, J., Berninger, P., ..., & Tuschl, T. (2010).
Transcriptome-wide identification of RNA-binding protein and microRNA target sites by PAR-CLIP. Cell, 141(1), 129-41. [PubMed:20371350] [PMC] [WorldCat] [DOI] - ↑
Tokita, M.J., Chow, P.M., Mirzaa, G., Dikow, N., Maas, B., Isidor, B., ..., & Prontera, P. (2015).
Five children with deletions of 1p34.3 encompassing AGO1 and AGO3. European journal of human genetics : EJHG, 23(6), 761-5. [PubMed:25271087] [PMC] [WorldCat] [DOI] - ↑
Schalk, A., Cousin, M.A., Dsouza, N.R., Challman, T.D., Wain, K.E., Powis, Z., ..., & Gerard, B. (2022).
De novo coding variants in the AGO1 gene cause a neurodevelopmental disorder with intellectual disability. Journal of medical genetics, 59(10), 965-975. [PubMed:34930816] [PMC] [WorldCat] [DOI] - ↑
Iorio, M.V., & Croce, C.M. (2012).
MicroRNA dysregulation in cancer: diagnostics, monitoring and therapeutics. A comprehensive review. EMBO molecular medicine, 4(3), 143-59. [PubMed:22351564] [PMC] [WorldCat] [DOI] - ↑
Wang, X., Gou, L.T., & Liu, M.F. (2022).
Noncanonical functions of PIWIL1/piRNAs in animal male germ cells and human diseases†. Biology of reproduction, 107(1), 101-108. [PubMed:35403682] [WorldCat] [DOI]