Chromosome 9 open reading frame 72
藤野 雄三1,2、永井 義隆1,3
1. 近畿大学医学部 脳神経内科
2. 京都府立医科大学大学院医学研究科 脳神経内科学
3. 近畿大学ライフサイエンス研究所
DOI:10.14931/bsd.11350 原稿受付日:2025年7月7日 原稿完成日:2025年9月1日
担当編集委員:古屋敷 智之(東京科学大学大学院 医歯学総合研究科 薬理学分野)
英: Chromosome 9 open reading frame 72
略語: C9orf72
C9orf72はDENNドメインを有するタンパク質であり、種々の低分子量Gタンパク質の活性制御を介して、小胞輸送やオートファジー、自然免疫といった多岐にわたる生体機能に関与する。2011年に、本遺伝子の非翻訳領域内に存在するGGGGCCリピート配列の異常伸長が、筋萎縮性側索硬化症および前頭側頭型認知症における最も高頻度の原因遺伝子変異として同定され、その病態メカニズムが注目されている。現在、①C9orf72遺伝子の機能喪失、②転写された変異リピートRNAによる毒性、③リピート関連非AUG依存性翻訳によって産生されるリピートペプチドによる毒性が、協調的に神経変性病態に寄与すると考えられている。
歴史的推移
C9orf72遺伝子は、神経変性疾患である筋萎縮性側索硬化症(ALS: amyotrophic lateral sclerosis)および前頭側頭型認知症(FTD: frontotemporal dementia)の原因となる代表的な遺伝子の1つとして、研究が進展してきた。
2006年、家族性ALS/FTD家系を用いた連鎖解析によって、責任遺伝子領域が9番染色体短腕に同定され[1][1][2][2]、その後の遺伝子探索によって、2011年にC9orf72遺伝子非翻訳領域内のGGGGCC 6塩基リピート配列の異常伸長(正常: 2-24回リピート, 疾患: 60-4000回リピート)が、原因遺伝子変異として同定された[3][3][4][4]。このリピート伸長変異は、欧米のALSFTDコホートにおいて最も頻度の高い原因遺伝子変異であることが明らかとなり、同遺伝子の生理的機能やリピート伸長変異による病態メカニズムに注目が集まり、研究が展開されている。
生理的機能に関しては未解明の点が多いものの、分子系統解析では本遺伝子は真核生物の進化初期に出現し、哺乳類を含む広範な生物種で保存されていることから、生物学的に重要な役割を担うと想定されている[5][5]。特に、C9orf72タンパク質はDENN(differentially expressed in normal and neoplastic cells)ドメインを有し、本ドメインを有する一群のタンパク質はRabファミリーを中心とした低分子量Gタンパク質の活性制御に関与することから、小胞輸送やオートファジーとの関連が注目されている。近年では、自然免疫応答の制御機能についても示唆されている(後述)[5][5]。
C9orf72遺伝子連鎖性ALS/FTD(C9-ALS/FTD)の病態メカニズムに関しては、当初、GGGGCCリピート伸長変異による遺伝子機能の喪失や、転写された異常伸長リピート配列を含む変異RNA(リピートRNA)による毒性が想定されていた。しかし2013年に、リピートRNAから非古典的なリピート関連非AUG依存性(RAN: repeat-associated non-AUG)翻訳によって病的なジペプチドリピートペプチドタンパク質(DPR: dipeptide repeat protein)が産生され、実際にDPRが患者脳に蓄積することが明らかになり、現在ではこれら3つの病態メカニズムが協調的に神経変性病態に寄与すると考えられている[6][6]。
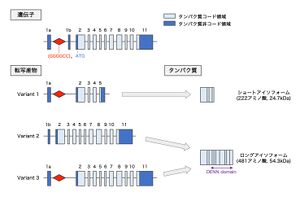
C9orf72遺伝子は12のエクソンから構成され、このうちエクソン2~11がタンパク質をコードする。GGGGCCリピート配列はエクソン1aと1bの間の非翻訳領域に存在する。3種類の転写産物と2種類のタンパク質アイソフォームが存在し、Variant 1はショートアイソフォームに、Variant2および3はロングアイソフォームにそれぞれ翻訳される。Variant 1およびVariant 3はGGGGCCリピート配列を含む。ロングアイソフォームにはDENNドメインが含まれる。
構造
遺伝子
ヒトでは染色体9p21.2に存在する41kbの遺伝子で、12のエクソン(エクソン 1a、1b、2~11)から構成される[5][5][6][6]。翻訳開始に利用されるATGコドンはエクソン2に存在し、エクソン2~11がC9orf72タンパク質をコードする。エクソン1aおよび1bはタンパク質をコードしない非翻訳領域である。ALS, FTDと関連するGGGGCCリピート配列は、エクソン1aと1bの間の非翻訳領域に存在する(図1)[5][5][6][6]。
転写産物
転写開始点および転写終結点を有し、転写産物については3種類のバリアントが報告されている。Variant 1(V1)はエクソン1a、2~5から構成される短鎖転写産物である。Variant 2(V2)はエクソン1b、2~11、Variant 3(V3)はエクソン1a、2~11から構成される長鎖転写産物である[5][5][6][6]。GGGGCCリピート配列は、V1およびV3ではイントロン領域に存在する一方で、V2では転写開始点直上流に存在し、転写産物自体には含まれない(図1)[5][5][6][6]。
タンパク質
222アミノ酸から構成されるショートアイソフォーム(分子量24.7kDa)と、481アミノ酸から構成されるロングアイソフォーム(分子量54.3kDa)の2種類のアイソフォームが存在する。ショートアイソフォームはV1転写産物から、ロングアイソフォームはV2およびV3転写産物から翻訳される(図1)[5][5][6][6]。ロングアイソフォームはDENNドメインを有し、主にGDP/GTP交換因子(GEF: GDP/GTP exchange factor)として低分子量Gタンパク質の活性制御を担うDENNファミリータンパク質の一員と考えられている[7][7][8][8]。
発現
RNAレベル
C9orf72転写産物はほとんどのヒト組織において発現が認められるが、特に脳、血液細胞、脾臓、脾臓、卵巣などにおいて高発現を示す[9][9]。脳内では小脳おける発現が顕著であり、血液細胞ではリンパ球よりも骨髄系細胞において高い発現が報告されている[4][4][10][10]。
タンパク質レベル
C9orf72タンパク質もまた、ヒトのほとんどの組織で発現している。マウスを用いた解析では、脳、脊髄、および脾臓で特に高い発現を認めている[11][11]。
培養細胞では、C9orf72タンパク質はリソソームと共局在することが報告されている[12][12]。ヒト脳組織における免疫組織化学では、ショートアイソフォームが主に神経細胞の核膜に分布する一方で、ロングアイソフォームは細胞質全体に広く分布し、神経突起や細胞体に点状構造を形成することが確認されている[13][13]。また、ヒトiPS細胞由来運動ニューロンを用いた検討により、C9orf72タンパク質がシナプス小胞にも局在することが示唆されている[11][11]。
機能
DENNファミリータンパク質の1つであるC9orf72タンパク質は、同じくDENNドメインを有するSMCR8、およびWDR41と複合体を形成する[14][14][15][15][16][16][17][17]。この複合体はGTPase活性化タンパク質(GAP: GTPase activating protein)として[16][16][17][17]。C9orf72タンパク質が相互作用する代表的な低分子量G タンパク質は、小胞輸送に関与するRabファミリータンパク質であり、これによってオートファジーやシナプス機能などの様々な生体反応を制御することが明らかになっている。また近年では、自然免疫応答の制御においても重要な役割を担うことが示唆されている。
オートファジーの制御
培養神経細胞系やヒト脊髄運動ニューロンにおいて、C9orf72タンパク質はRab1、Rab5、Rab7、Rab11などのRabファミリータンパク質と共局在する[18][18]。また培養細胞を用いた免疫沈降法による解析では、Rab8aやRab39b、FIP200/Ulk1/ATG13/ATG101複合体と相互作用することが報告されている[15][15][16][16]。これらの分子との相互作用を介して、C9orf72タンパク質はオートファジーの開始、基質のリクルートメント、隔離膜の伸長と閉鎖、オートファゴソームとリソソームの融合など、オートファジーの各段階を制御すると考えられている[19][19]。実際に、C9orf72遺伝子をノックダウンした培養細胞では、オートファジーの誘導阻害や異常なオートファゴソームの蓄積が観察されている[16][16][18][18]。
シナプス機能の制御
C9orf72タンパク質が相互作用するRab39bは、AMPA型グルタミン酸受容体の小胞輸送およびリサイクリングに関与し、受容体のサブユニット構成やシナプスの電気生理学的特性を調節する[20][20]。C9orf72ノックアウトマウスでは、シナプス後部においてRab39bの発現が低下し、AMPA型グルタミン酸受容体GluR1サブユニットの発現が亢進することが報告されており、C9orf72タンパク質がRab39bの発現や活性を制御することでシナプス機能を調節している可能性が示唆されている[21][21]。
細胞骨格の制御
C9orf72タンパク質は、Rabファミリータンパク質とは異なる低分子量G タンパク質であるArfファミリータンパク質とも相互作用し、特にArf6との相互作用を介して、運動ニューロンにおけるアクチン細胞骨格の動態を制御することが報告されている[22][22]。培養神経細胞ではC9orf72遺伝子の過剰発現によって軸索が伸長し、ノックダウンによって短縮することが確認されている[22][22]。
自然免疫応答の制御
C9orf72ノックアウトマウスでは、脾腫やリンパ節腫脹といった免疫系組織の腫大が認められ、さらにTypeⅠインターフェロンなどの炎症性サイトカインや自己抗体の産生が亢進していることから、免疫系における役割が注目されている[23][23][24][24]。特に、骨髄系細胞特異的C9orf72ノックアウトマウスにおいても、同様の異常が確認されることから、同細胞での作用が免疫系の制御に重要であると考えられる[25][25]。その詳細な機序は未解明であるものの、C9orf72タンパク質がオートファジー活性を調節することで、STINGやmTORC1といった自然免疫応答に関与する分子の発現を制御する可能性が示唆されている[25][25][26][26]。
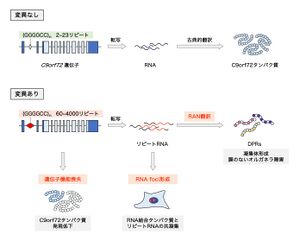
非翻訳領域のGGGGCCリピート配列の異常伸長によって、1) C9orf72タンパク質の発現が低下し、遺伝子の機能喪失をきたす。また、2) リピートRNAはRNA結合タンパク質を巻き込みながらRNA fociとして凝集し、これらのタンパク質の生理的機能を撹乱する。さらに、3) リピートRNAから非古典的なRAN翻訳によって異常なDPRが産生される。これら3つの主要な病態メカニズムが協調的に神経変性に寄与する。
疾患との関連
2006年に、欧米の家族性ALS/FTDコホートの連鎖解析によって、責任遺伝子領域として9番染色体短腕13.2-21.3が同定された[1][1][2][2]。その後5年にわたる原因遺伝子変異探索の結果、2011年にC9orf72遺伝子の非翻訳領域内に存在するGGGGCCリピート配列の異常伸長が、原因遺伝子変異として同定された[3][3][4][4]。この配列のリピート回数は、健常人では2~23回であるが、C9-ALS/FTDでは40~6000回に異常伸長している[27][27]。リピート伸長変異は不完全浸透であり、一般人口においても約0.1~0.4%の頻度で認められる[4][4][28][28]。異常リピート回数の明確な閾値の決定は困難であり、リピート回数が24回から30回程度の中間伸長アレルも疾患発症リスクとなることが知られている[29][29]。本リピート伸長変異は、家族性ALSの約40%、家族性FTDの約25%、孤発性ALSおよびFTDの5~6%で認められており、両疾患における最も頻度の高い遺伝的素因である[30][30]。なお、変異の頻度には地域差があり、欧米では高頻度であるのに対し、日本を含むアジア地域では稀である[30][30]。
臨床的には、ALS、FTD、あるいは両者の合併症状を来たすことが多い[6][6]。病理学的には、RNA結合タンパク質TDP-43の核内からの消失および細胞質での異常凝集が認められる点で、大部分の孤発性ALSや一部のFTD症例と共通している[6][6]。
病態機序については未解明な点が多く残るが、現在までに以下の3つの主要なメカニズムが明らかになり、これらは相互に作用して神経変性病態に寄与すると考えられている(図2)。
C9orf72遺伝子の機能喪失
C9-ALS/FTD患者の脳組織では、C9orf72転写産物およびタンパク質の発現がともに低下しており、ハプロ不全が神経変性病態に関与する可能性が示唆されている[3][3][31][31][32][32][33][33]。先述の通り、C9orf72ノックアウトマウスでは、免疫系の異常を認められるものの、明らかな神経変性は生じない[23][23][24][24]。しかし、C9orf72遺伝子の発現低下はオートファジーの障害を介して、後述するDPRによる神経変性病態を増悪させることが報告されており、ハプロ不全と毒性獲得性のメカニズムが協調的に病態に関与していると考えられている[34][34][35][35]。
リピートRNA毒性
異常伸長したリピート配列は両方向性に転写され、GGGGCCリピートおよびその逆鎖であるCCCCGGリピートを含むリピートRNAが産生される。これらのリピートRNAはRNA結合タンパク質(RBP: RNA-binding protein)と異常な相互作用を獲得して、RBPの生理的な分布や機能を撹乱し得る。特に、リピートRNAは異常な高次構造を形成してRNA fociと呼ばれるRNA凝集体を形成し、種々のRBPと共凝集することが示されている[3][3][36][36]。例えば、GGGGCCリピートRNAが形成するRNA fociは、hnRNPH, hnRNPF, hnRNPA1, ALYREF, SRSF2, nucleolinといった多数のRBPと共局在し[36][36][37][37][38][38][39][39]hnRNPHのRNA fociへの共凝集は標的mRNAのスプライシング異常を来すことが報告されている[37][37]。
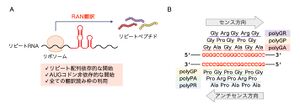
(A) RAN翻訳は、C9orf72連鎖性ALS/FTDなど、遺伝子の非翻訳領域内のリピート伸長変異を原因とするノンコーディングリピート病の病態に関与する非古典的な翻訳機構である。リピートRNAを鋳型として、AUGコドン非依存的に翻訳が開始し、異常なリピートペプチドを産生する。翻訳は全ての読み枠で進行する。
(B) C9orf72連鎖性ALS/FTD においては、GGGGCCおよびCCCCGGリピートRNAを鋳型として、RAN翻訳によって合計5種類のDPRが産生される。
RAN翻訳とDPR毒性
非翻訳領域由来のリピートRNAは、AUG開始コドンを欠くものの、RAN翻訳によって異常なDPRが産生される。RAN翻訳は鋳型となるRNAにリピート配列が含まれる際に、AUG開始コドン非存在下に翻訳が開始し、リピートペプチドが産生される非古典的な翻訳であり(図3A)、元々2011年に、同じく遺伝子非翻訳領域で異常伸長したリピート配列を原因とする脊髄小脳失調症8型(CAGリピート)および筋強直性ジストロフィー1型(CTGリピート)で発見された[40][40]。AUGコドン非依存的な翻訳開始には、リピートRNAが形成する高次構造が重要であると示唆されている[40][40]。3種類全ての読み枠で翻訳が進行するため、GGGGCCリピートRNAからはDPRとしてポリグリシン-アルギニン(polyGR)、ポリグリシン-アラニン(polyGA)、ポリグリシン-プロリン(polyGP)ペプチドが、CCCCGGリピートRNAからはポリグリシン-プロリン(polyGP)、ポリプロリン-アルギニン(polyPR)、ポリプロリン-アラニン(polyPA)ペプチドが産生され、実際に患者脳や髄液で検出される(図3B)[41][41][42][42][43][43][44][44][45][45]。
polyGAは凝集性が高く、病理学的にC9-ALS/FTD患者脳で最も豊富に検出されるDPRである[46][46]。培養細胞や動物モデルではpolyGAは弱い神経毒性を生じ、またプロテアソームと相互作用することからタンパク質分解系を障害する可能性がある[47][47][48][48][49][49]。一方、アルギニンを含有するDPRであるpolyGRやpolyPRは、患者脳での発現はpolyGAと比較して少ないが[46][46]、培養細胞や動物モデルで最も高度な毒性を来す[47][47][50][50]。その機序として、核細胞質輸送の障害[51][51]や、核小体やストレス顆粒といった膜のないオルガネラの動態の障害[52][52][53][53]が実験的に示唆されている。
なお、C9-ALS/FTD以外にも、ゲノム中に存在するリピート配列の異常伸長は60を超える遺伝性疾患の原因となり、多くが運動ニューロン疾患、小脳失調、認知症、末梢神経障害、てんかん、ミオパチーなどを来す神経筋疾患である(リピート病)。その中でも遺伝子の非翻訳領域内のリピート伸長変異を原因とする疾患群はノンコーディングリピート病として総称され、C9-ALS/FTDと共通する病態基盤が想定されている[54][54][55][55]。
関連項目
参考文献
- ↑ 1.0 1.1
Morita, M., Al-Chalabi, A., Andersen, P.M., Hosler, B., Sapp, P., Englund, E., ..., & Brown, R.H. (2006).
A locus on chromosome 9p confers susceptibility to ALS and frontotemporal dementia. Neurology, 66(6), 839-44. [PubMed:16421333] [WorldCat] [DOI] - ↑ 2.0 2.1
Vance, C., Al-Chalabi, A., Ruddy, D., Smith, B.N., Hu, X., Sreedharan, J., ..., & Shaw, C.E. (2006).
Familial amyotrophic lateral sclerosis with frontotemporal dementia is linked to a locus on chromosome 9p13.2-21.3. Brain : a journal of neurology, 129(Pt 4), 868-76. [PubMed:16495328] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2 3.3
DeJesus-Hernandez, M., Mackenzie, I.R., Boeve, B.F., Boxer, A.L., Baker, M., Rutherford, N.J., ..., & Rademakers, R. (2011).
Expanded GGGGCC hexanucleotide repeat in noncoding region of C9ORF72 causes chromosome 9p-linked FTD and ALS. Neuron, 72(2), 245-56. [PubMed:21944778] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1 4.2 4.3
Renton, A.E., Majounie, E., Waite, A., Simón-Sánchez, J., Rollinson, S., Gibbs, J.R., ..., & Traynor, B.J. (2011).
A hexanucleotide repeat expansion in C9ORF72 is the cause of chromosome 9p21-linked ALS-FTD. Neuron, 72(2), 257-68. [PubMed:21944779] [PMC] [WorldCat] [DOI] - ↑ 5.0 5.1 5.2 5.3 5.4 5.5 5.6
Smeyers, J., Banchi, E.G., & Latouche, M. (2021).
C9ORF72: What It Is, What It Does, and Why It Matters. Frontiers in cellular neuroscience, 15, 661447. [PubMed:34025358] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2 6.3 6.4 6.5 6.6 6.7
Balendra, R., & Isaacs, A.M. (2018).
C9orf72-mediated ALS and FTD: multiple pathways to disease. Nature reviews. Neurology, 14(9), 544-558. [PubMed:30120348] [PMC] [WorldCat] [DOI] - ↑
Levine, T.P., Daniels, R.D., Gatta, A.T., Wong, L.H., & Hayes, M.J. (2013).
The product of C9orf72, a gene strongly implicated in neurodegeneration, is structurally related to DENN Rab-GEFs. Bioinformatics (Oxford, England), 29(4), 499-503. [PubMed:23329412] [PMC] [WorldCat] [DOI] - ↑
Zhang, D., Iyer, L.M., He, F., & Aravind, L. (2012).
Discovery of Novel DENN Proteins: Implications for the Evolution of Eukaryotic Intracellular Membrane Structures and Human Disease. Frontiers in genetics, 3, 283. [PubMed:23248642] [PMC] [WorldCat] [DOI] - ↑ GTExPortal [Internet]. Available from: https://www.gtexportal.org/home/gene/C9orf72
- ↑
Rizzu, P., Blauwendraat, C., Heetveld, S., Lynes, E.M., Castillo-Lizardo, M., Dhingra, A., ..., & Heutink, P. (2016).
C9orf72 is differentially expressed in the central nervous system and myeloid cells and consistently reduced in C9orf72, MAPT and GRN mutation carriers. Acta neuropathologica communications, 4(1), 37. [PubMed:27079381] [PMC] [WorldCat] [DOI] - ↑ 11.0 11.1
Frick, P., Sellier, C., Mackenzie, I.R.A., Cheng, C.Y., Tahraoui-Bories, J., Martinat, C., ..., & Neumann, M. (2018).
Novel antibodies reveal presynaptic localization of C9orf72 protein and reduced protein levels in C9orf72 mutation carriers. Acta neuropathologica communications, 6(1), 72. [PubMed:30075745] [PMC] [WorldCat] [DOI] - ↑
Laflamme, C., McKeever, P.M., Kumar, R., Schwartz, J., Kolahdouzan, M., Chen, C.X., ..., & McPherson, P.S. (2019).
Implementation of an antibody characterization procedure and application to the major ALS/FTD disease gene C9ORF72. eLife, 8. [PubMed:31612854] [PMC] [WorldCat] [DOI] - ↑
Xiao, S., MacNair, L., McGoldrick, P., McKeever, P.M., McLean, J.R., Zhang, M., ..., & Robertson, J. (2015).
Isoform-specific antibodies reveal distinct subcellular localizations of C9orf72 in amyotrophic lateral sclerosis. Annals of neurology, 78(4), 568-83. [PubMed:26174152] [WorldCat] [DOI] - ↑
Yang, M., Liang, C., Swaminathan, K., Herrlinger, S., Lai, F., Shiekhattar, R., & Chen, J.F. (2016).
A C9ORF72/SMCR8-containing complex regulates ULK1 and plays a dual role in autophagy. Science advances, 2(9), e1601167. [PubMed:27617292] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1
Sellier, C., Campanari, M.L., Julie Corbier, C., Gaucherot, A., Kolb-Cheynel, I., Oulad-Abdelghani, M., ..., & Charlet-Berguerand, N. (2016).
Loss of C9ORF72 impairs autophagy and synergizes with polyQ Ataxin-2 to induce motor neuron dysfunction and cell death. The EMBO journal, 35(12), 1276-97. [PubMed:27103069] [PMC] [WorldCat] [DOI] - ↑ 16.0 16.1 16.2 16.3
Su, M.Y., Fromm, S.A., Zoncu, R., & Hurley, J.H. (2020).
Structure of the C9orf72 ARF GAP complex that is haploinsufficient in ALS and FTD. Nature, 585(7824), 251-255. [PubMed:32848248] [PMC] [WorldCat] [DOI] - ↑ 17.0 17.1
Tang, D., Sheng, J., Xu, L., Zhan, X., Liu, J., Jiang, H., ..., & Qi, S. (2020).
Cryo-EM structure of C9ORF72-SMCR8-WDR41 reveals the role as a GAP for Rab8a and Rab11a. Proceedings of the National Academy of Sciences of the United States of America, 117(18), 9876-9883. [PubMed:32303654] [PMC] [WorldCat] [DOI] - ↑ 18.0 18.1
Farg, M.A., Sundaramoorthy, V., Sultana, J.M., Yang, S., Atkinson, R.A., Levina, V., ..., & Atkin, J.D. (2014).
C9ORF72, implicated in amytrophic lateral sclerosis and frontotemporal dementia, regulates endosomal trafficking. Human molecular genetics, 23(13), 3579-95. [PubMed:24549040] [PMC] [WorldCat] [DOI] - ↑
Beckers, J., Tharkeshwar, A.K., & Van Damme, P. (2021).
C9orf72 ALS-FTD: recent evidence for dysregulation of the autophagy-lysosome pathway at multiple levels. Autophagy, 17(11), 3306-3322. [PubMed:33632058] [PMC] [WorldCat] [DOI] - ↑
Mignogna, M.L., Giannandrea, M., Gurgone, A., Fanelli, F., Raimondi, F., Mapelli, L., ..., & D'Adamo, P. (2015).
The intellectual disability protein RAB39B selectively regulates GluA2 trafficking to determine synaptic AMPAR composition. Nature communications, 6, 6504. [PubMed:25784538] [PMC] [WorldCat] [DOI] - ↑
Xiao, S., McKeever, P.M., Lau, A., & Robertson, J. (2019).
Synaptic localization of C9orf72 regulates post-synaptic glutamate receptor 1 levels. Acta neuropathologica communications, 7(1), 161. [PubMed:31651360] [PMC] [WorldCat] [DOI] - ↑ 22.0 22.1
Sivadasan, R., Hornburg, D., Drepper, C., Frank, N., Jablonka, S., Hansel, A., ..., & Sendtner, M. (2016).
C9ORF72 interaction with cofilin modulates actin dynamics in motor neurons. Nature neuroscience, 19(12), 1610-1618. [PubMed:27723745] [WorldCat] [DOI] - ↑ 23.0 23.1
O'Rourke, J.G., Bogdanik, L., Yáñez, A., Lall, D., Wolf, A.J., Muhammad, A.K., ..., & Baloh, R.H. (2016).
C9orf72 is required for proper macrophage and microglial function in mice. Science (New York, N.Y.), 351(6279), 1324-9. [PubMed:26989253] [PMC] [WorldCat] [DOI] - ↑ 24.0 24.1
Sudria-Lopez, E., Koppers, M., de Wit, M., van der Meer, C., Westeneng, H.J., Zundel, C.A., ..., & Pasterkamp, R.J. (2016).
Full ablation of C9orf72 in mice causes immune system-related pathology and neoplastic events but no motor neuron defects. Acta neuropathologica, 132(1), 145-7. [PubMed:27206760] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1
McCauley, M.E., O'Rourke, J.G., Yáñez, A., Markman, J.L., Ho, R., Wang, X., ..., & Baloh, R.H. (2020).
C9orf72 in myeloid cells suppresses STING-induced inflammation. Nature, 585(7823), 96-101. [PubMed:32814898] [PMC] [WorldCat] [DOI] - ↑
Shao, Q., Yang, M., Liang, C., Ma, L., Zhang, W., Jiang, Z., ..., & Chen, J.F. (2020).
C9orf72 and smcr8 mutant mice reveal MTORC1 activation due to impaired lysosomal degradation and exocytosis. Autophagy, 16(9), 1635-1650. [PubMed:31847700] [PMC] [WorldCat] [DOI] - ↑ Gossye H., Engelborghs S., Van Broeckhoven C., van der Zee J. (2015). C9orf72 FTD and/or ALS. GeneReviews® [Internet]. Available from: https://www.ncbi.nlm.nih.gov/books/NBK268647/
- ↑
Beck, J., Poulter, M., Hensman, D., Rohrer, J.D., Mahoney, C.J., Adamson, G., ..., & Mead, S. (2013).
Large C9orf72 hexanucleotide repeat expansions are seen in multiple neurodegenerative syndromes and are more frequent than expected in the UK population. American journal of human genetics, 92(3), 345-53. [PubMed:23434116] [PMC] [WorldCat] [DOI] - ↑
Iacoangeli, A., Al Khleifat, A., Jones, A.R., Sproviero, W., Shatunov, A., Opie-Martin, S., ..., & Al-Chalabi, A. (2019).
C9orf72 intermediate expansions of 24-30 repeats are associated with ALS. Acta neuropathologica communications, 7(1), 115. [PubMed:31315673] [PMC] [WorldCat] [DOI] - ↑ 30.0 30.1
Majounie, E., Renton, A.E., Mok, K., Dopper, E.G., Waite, A., Rollinson, S., ..., & Traynor, B.J. (2012).
Frequency of the C9orf72 hexanucleotide repeat expansion in patients with amyotrophic lateral sclerosis and frontotemporal dementia: a cross-sectional study. The Lancet. Neurology, 11(4), 323-30. [PubMed:22406228] [PMC] [WorldCat] [DOI] - ↑
van Blitterswijk, M., Gendron, T.F., Baker, M.C., DeJesus-Hernandez, M., Finch, N.A., Brown, P.H., ..., & Rademakers, R. (2015).
Novel clinical associations with specific C9ORF72 transcripts in patients with repeat expansions in C9ORF72. Acta neuropathologica, 130(6), 863-76. [PubMed:26437865] [PMC] [WorldCat] [DOI] - ↑
Waite, A.J., Bäumer, D., East, S., Neal, J., Morris, H.R., Ansorge, O., & Blake, D.J. (2014).
Reduced C9orf72 protein levels in frontal cortex of amyotrophic lateral sclerosis and frontotemporal degeneration brain with the C9ORF72 hexanucleotide repeat expansion. Neurobiology of aging, 35(7), 1779.e5-1779.e13. [PubMed:24559645] [PMC] [WorldCat] [DOI] - ↑
Saberi, S., Stauffer, J.E., Jiang, J., Garcia, S.D., Taylor, A.E., Schulte, D., ..., & Ravits, J. (2018).
Sense-encoded poly-GR dipeptide repeat proteins correlate to neurodegeneration and uniquely co-localize with TDP-43 in dendrites of repeat-expanded C9orf72 amyotrophic lateral sclerosis. Acta neuropathologica, 135(3), 459-474. [PubMed:29196813] [PMC] [WorldCat] [DOI] - ↑
Zhu, Q., Jiang, J., Gendron, T.F., McAlonis-Downes, M., Jiang, L., Taylor, A., ..., & Cleveland, D.W. (2020).
Reduced C9ORF72 function exacerbates gain of toxicity from ALS/FTD-causing repeat expansion in C9orf72. Nature neuroscience, 23(5), 615-624. [PubMed:32284607] [PMC] [WorldCat] [DOI] - ↑
Boivin, M., Pfister, V., Gaucherot, A., Ruffenach, F., Negroni, L., Sellier, C., & Charlet-Berguerand, N. (2020).
Reduced autophagy upon C9ORF72 loss synergizes with dipeptide repeat protein toxicity in G4C2 repeat expansion disorders. The EMBO journal, 39(4), e100574. [PubMed:31930538] [PMC] [WorldCat] [DOI] - ↑ 36.0 36.1
Haeusler, A.R., Donnelly, C.J., Periz, G., Simko, E.A., Shaw, P.G., Kim, M.S., ..., & Wang, J. (2014).
C9orf72 nucleotide repeat structures initiate molecular cascades of disease. Nature, 507(7491), 195-200. [PubMed:24598541] [PMC] [WorldCat] [DOI] - ↑ 37.0 37.1 Conlon E.G., Lu L., Sharma A., Yamazaki T., Tang T., Shneider N.A., Manley J.L. (2016). eLife, 5:e17820. https://doi.org/10.7554/eLife.17820.030 引用エラー: 無効な
<ref>タグ; name "Conlon2016"が異なる内容で複数回定義されています - ↑
Lee, Y.B., Chen, H.J., Peres, J.N., Gomez-Deza, J., Attig, J., Stalekar, M., ..., & Shaw, C.E. (2013).
Hexanucleotide repeats in ALS/FTD form length-dependent RNA foci, sequester RNA binding proteins, and are neurotoxic. Cell reports, 5(5), 1178-86. [PubMed:24290757] [PMC] [WorldCat] [DOI] - ↑
Cooper-Knock, J., Walsh, M.J., Higginbottom, A., Robin Highley, J., Dickman, M.J., Edbauer, D., ..., & Shaw, P.J. (2014).
Sequestration of multiple RNA recognition motif-containing proteins by C9orf72 repeat expansions. Brain : a journal of neurology, 137(Pt 7), 2040-51. [PubMed:24866055] [PMC] [WorldCat] [DOI] - ↑ 40.0 40.1
Zu, T., Gibbens, B., Doty, N.S., Gomes-Pereira, M., Huguet, A., Stone, M.D., ..., & Ranum, L.P. (2011).
Non-ATG-initiated translation directed by microsatellite expansions. Proceedings of the National Academy of Sciences of the United States of America, 108(1), 260-5. [PubMed:21173221] [PMC] [WorldCat] [DOI] - ↑
Mori, K., Weng, S.M., Arzberger, T., May, S., Rentzsch, K., Kremmer, E., ..., & Edbauer, D. (2013).
The C9orf72 GGGGCC repeat is translated into aggregating dipeptide-repeat proteins in FTLD/ALS. Science (New York, N.Y.), 339(6125), 1335-8. [PubMed:23393093] [WorldCat] [DOI] - ↑
Mori, K., Arzberger, T., Grässer, F.A., Gijselinck, I., May, S., Rentzsch, K., ..., & Edbauer, D. (2013).
Bidirectional transcripts of the expanded C9orf72 hexanucleotide repeat are translated into aggregating dipeptide repeat proteins. Acta neuropathologica, 126(6), 881-93. [PubMed:24132570] [WorldCat] [DOI] - ↑
Ash, P.E., Bieniek, K.F., Gendron, T.F., Caulfield, T., Lin, W.L., Dejesus-Hernandez, M., ..., & Petrucelli, L. (2013).
Unconventional translation of C9ORF72 GGGGCC expansion generates insoluble polypeptides specific to c9FTD/ALS. Neuron, 77(4), 639-46. [PubMed:23415312] [PMC] [WorldCat] [DOI] - ↑
Zu, T., Liu, Y., Bañez-Coronel, M., Reid, T., Pletnikova, O., Lewis, J., ..., & Ranum, L.P. (2013).
RAN proteins and RNA foci from antisense transcripts in C9ORF72 ALS and frontotemporal dementia. Proceedings of the National Academy of Sciences of the United States of America, 110(51), E4968-77. [PubMed:24248382] [PMC] [WorldCat] [DOI] - ↑
Gendron, T.F., Bieniek, K.F., Zhang, Y.J., Jansen-West, K., Ash, P.E., Caulfield, T., ..., & Petrucelli, L. (2013).
Antisense transcripts of the expanded C9ORF72 hexanucleotide repeat form nuclear RNA foci and undergo repeat-associated non-ATG translation in c9FTD/ALS. Acta neuropathologica, 126(6), 829-44. [PubMed:24129584] [PMC] [WorldCat] [DOI] - ↑ 46.0 46.1
Mackenzie, I.R., Frick, P., Grässer, F.A., Gendron, T.F., Petrucelli, L., Cashman, N.R., ..., & Neumann, M. (2015).
Quantitative analysis and clinico-pathological correlations of different dipeptide repeat protein pathologies in C9ORF72 mutation carriers. Acta neuropathologica, 130(6), 845-61. [PubMed:26374446] [WorldCat] [DOI] - ↑ 47.0 47.1
Mizielinska, S., Grönke, S., Niccoli, T., Ridler, C.E., Clayton, E.L., Devoy, A., ..., & Isaacs, A.M. (2014).
C9orf72 repeat expansions cause neurodegeneration in Drosophila through arginine-rich proteins. Science (New York, N.Y.), 345(6201), 1192-1194. [PubMed:25103406] [PMC] [WorldCat] [DOI] - ↑
May, S., Hornburg, D., Schludi, M.H., Arzberger, T., Rentzsch, K., Schwenk, B.M., ..., & Edbauer, D. (2014).
C9orf72 FTLD/ALS-associated Gly-Ala dipeptide repeat proteins cause neuronal toxicity and Unc119 sequestration. Acta neuropathologica, 128(4), 485-503. [PubMed:25120191] [PMC] [WorldCat] [DOI] - ↑
Guo, Q., Lehmer, C., Martínez-Sánchez, A., Rudack, T., Beck, F., Hartmann, H., ..., & Fernández-Busnadiego, R. (2018).
In Situ Structure of Neuronal C9orf72 Poly-GA Aggregates Reveals Proteasome Recruitment. Cell, 172(4), 696-705.e12. [PubMed:29398115] [PMC] [WorldCat] [DOI] - ↑
Wen, X., Tan, W., Westergard, T., Krishnamurthy, K., Markandaiah, S.S., Shi, Y., ..., & Trotti, D. (2014).
Antisense proline-arginine RAN dipeptides linked to C9ORF72-ALS/FTD form toxic nuclear aggregates that initiate in vitro and in vivo neuronal death. Neuron, 84(6), 1213-25. [PubMed:25521377] [PMC] [WorldCat] [DOI] - ↑
Cook, C.N., Wu, Y., Odeh, H.M., Gendron, T.F., Jansen-West, K., Del Rosso, G., ..., & Petrucelli, L. (2020).
C9orf72 poly(GR) aggregation induces TDP-43 proteinopathy. Science translational medicine, 12(559). [PubMed:32878979] [PMC] [WorldCat] [DOI] - ↑
Boeynaems, S., Bogaert, E., Kovacs, D., Konijnenberg, A., Timmerman, E., Volkov, A., ..., & Van Den Bosch, L. (2017).
Phase Separation of C9orf72 Dipeptide Repeats Perturbs Stress Granule Dynamics. Molecular cell, 65(6), 1044-1055.e5. [PubMed:28306503] [PMC] [WorldCat] [DOI] - ↑
Lee, K.H., Zhang, P., Kim, H.J., Mitrea, D.M., Sarkar, M., Freibaum, B.D., ..., & Taylor, J.P. (2016).
C9orf72 Dipeptide Repeats Impair the Assembly, Dynamics, and Function of Membrane-Less Organelles. Cell, 167(3), 774-788.e17. [PubMed:27768896] [PMC] [WorldCat] [DOI] - ↑
Malik, I., Kelley, C.P., Wang, E.T., & Todd, P.K. (2021).
Molecular mechanisms underlying nucleotide repeat expansion disorders. Nature reviews. Molecular cell biology, 22(9), 589-607. [PubMed:34140671] [PMC] [WorldCat] [DOI] - ↑
Fujino, Y., & Nagai, Y. (2022).
The molecular pathogenesis of repeat expansion diseases. Biochemical Society transactions, 50(1), 119-134. [PubMed:34940797] [WorldCat] [DOI]