「プロテアソーム」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| (2人の利用者による、間の47版が非表示) | |||
| 1行目: | 1行目: | ||
生体を構成する主要成分であり、生命現象を支える機能素子であるタンパク質は、細胞内で絶えず合成と分解を繰り返しており、きわめて動的なリサイクル(新陳代謝)システムを構成している。即ち、細胞内の全てのタンパク質は、千差万別の寿命をもってダイナミックにターンオーバー(代謝回転)しており、この新陳代謝の中心はタンパク質分解が担っている。タンパク質分解は多様な生体反応を不可逆的に制御する方法として発生や分化など様々な生命現象に不可欠な役割を果たしている。例えば、タンパク質分解は細胞内に生じた不良品の積極的な除去に深く関与しているほか、良品であっても不要な(細胞活動に支障をきたす)場合、あるいは緊急時の栄養素の確保のために、積極的に作動される。このようにタンパク質は細胞内でリサイクルし, 動的平衡を保つことによって身体の中を浄化(不要品のクリアランス)して新鮮さを保ち、健康を維持しているのである。即ち、細胞内のタンパク質は、恒常的な入れ替わり(ターンオーバー)によって品質を管理して健全な細胞活動に貢献していると言い換えることができる。 | |||
真核生物の細胞内には進化的に高度に保存された二つの大規模なタンパク質分解系、即ちユビキチン・プロテアソームシステム(UPS)とオートファジー・リソソームシステムが存在している。前者のUPSは選択的タンパク質を担う中心的な酵素系であり、後者は一般に非選択的なタンパク質を担っているが、最近、選択的なオートファジーが数多く発見され、注目されている(詳細は、オートファジーの項参照)。そしてこれらのタンパク質分解しステムは、時空間的に厳密に制御されており、その破綻が高齢化社会を迎えた今日急増しているガンや神経変性疾患などの重篤疾患を引き起こすことが相次いで判明している。 | |||
== ユビキチンシステム == | |||
[[image:プロテオソーム1.jpg|thumb|350px|'''図1.ユビキチン・プロテアソームシステム'''<br>Ub:ユビキチン、E1: Ub活性化酵素、E2: Ub結合酵素、E3: Ubリガーゼ、DUB: 脱ユビキチン酵素。基質を選別するE3酵素は、HECT型とRING型に大別される。26Sプロテアソームは不要なユビキチン化タンパク質をエネルギー依存的に分解する巨大で複雑なタンパク質分解酵素複合体である。(その他の詳細は本文及び文献[22]参照)。UPSの作動機構においてエネルギー(ATPの加水分解)は、基質のユビキチン化(E1の作用)と26Sプロテアソームによる分解作用(基質のアンフォールディング)の二つのプロセスに必要である。]] | |||
1977年、米国ハーバード大学のGoldbergのグループは網状赤血球の抽出液が非常に高いエネルギー依存性のタンパク質分解活性を示すという画期的な論文を発表した<ref name=ref><pubmed></pubmed></ref>[1]。その後間もなく、イスラエルのHershkoとCiechanoverは、酵素学のmentorである米国のRoseと共に、この分解系に注目、その機構解明にいち早く取り組み、熱安定性の小さなタンパク質であるユビキチンがその主役であることを見出した。ユビキチンは76個のアミノ酸からなる小さなタンパク質であり、進化的保存性が高くそのアミノ酸配列は全ての真核生物でほとんど同じである。1980年頃までに彼らは、ユビキチンが活性化酵素(E1)・結合酵素(E2)・リガーゼ(E3)から構成された複合酵素系(ユビキチンシステム)によって標的タンパク質に共有結合(ユビキチンのC末端のカルボキシル基とタンパク質中のリジン残基のε-アミノ基が縮合したイソペプチド結合)する翻訳後修飾分子(モディファイヤー)であることを明らかにした(図1)<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[2, 3]。このE1の作用にはATPの加水分解、即ちエネルギーが必要である。そしてタンパク質に結合したユビキチン内の(主として48番目の)リジン残基と新しいユビキチン分子内のC末端のグリシンの間でイソペプチド結合ができ、さらにユビキチン分子間での縮合反応を繰り返すことによって,多数のユビキチン分子が鎖状に伸長したポリユビキチン鎖が形成されることが判明した(現在では、ユビキチン分子内にある7個のリジン残基と N末端のαアミノ基からユビキチンポリマーが伸長し、多彩な生理機能を担っていることが分かっており、最近、この多様なポリユビキチン鎖に刻印された情報を、われわれは“ユビキチンコード”と定義している)。1980年前後、Hershkoら及びVarshavskyらは生じたポリユビキチン鎖が基質タンパク質を分解装置に輸送するためのシグナル(目印)として機能するという“ユビキチンシグナル”仮説を提唱した<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[2-4]。この仮説の秀逸な点は、ポリユビキチン鎖の形成が(オーバーオールの反応としては)分解シグナルの提示反応であるが、実際に起きている化学反応は(イソ)ペプチド結合の形成(タンパク質合成と類似の反応)であり、エネルギー要求性を見事に説明できることであった。実際、ユビキチン研究の勃興から黎明期をへて今日の隆盛期に至る道程は、まさに破竹の勢いでタンパク質分解の世界を席巻し尽くしてきた。その象徴的な出来事として、2004年、ユビキチンシステムの発見者たち3名は、ノーベル化学賞の栄誉に浴した。 | |||
= | ユビキチンは二つの異なったタイプの遺伝子にコードされている。一つは、ユビキチンとリボソームタンパク質の融合遺伝子であり、もう一つは数個〜10数個のユビキチンがタンデムに連なったポリユビキチン遺伝子である。後者のポリユビキチン遺伝子は、1回の転写・翻訳で多数のユビキチンを合成することができる点で秀逸であり、かつ熱ショック応答遺伝子でもあることから、細胞は環境ストレスに曝されたとき、必要とするユビキチンを迅速に生成することができるように合理的に設計されている。このことは、細胞内のユビキチンレベルが外環境の変化に応答して厳格に制御されていることを示唆している<ref name=ref><pubmed></pubmed></ref>[3]。 | ||
興味深いことに細胞内には、ユビキチン化の逆反応を触媒する脱ユビキチン酵素(deubiquitylating enzyme:DUB)が存在し、それらは生物種を問わず大きな遺伝子ファミリーを形成している。ヒトゲノムには、典型的な55種のDUBを含めて80種を越える脱ユビキチン酵素がコードされており、これらは6種のサブファミリー(4 UCH, 52 USP, 7 ULP, 2 Josephin domain-type, 16 OTU, 2 JAMM)に細分される<ref name=ref><pubmed></pubmed></ref>[5]。多数のDUBが存在することは、ユビキチン化による翻訳後修飾が可逆的かつ多面的であることを示唆している。このようにUSPは、リン酸化- 脱リン酸化システムに匹敵する細胞内制御系と考えられる。 | |||
== 分子構造と作動機構 == | |||
[[image:プロテオソーム2.jpg|thumb|350px|'''図2.26S プロテアソームの構造モデル:分子形状とサブユニット構成'''<br>左図:26Sプロテアソーム(CPとRPの複合体)の電子顕微鏡による分子形状(単粒子解析: 独マックスプランク研究所W. Baumeister・S. Nickellから供与)。U:ユビキチン。<br>右図:サブユニットの構成モデル.CP(20Sプロテアソーム)はα/βリングがαββαの順に会合した円柱状粒子。RP(PA700)はlid(蓋部)とbase(基底部)から構成された複合体。RPはRpn(RP non-ATPase)とRpt(RP triple-ATPase)サブユニット群から構成されている。Rpn10、Rpn13:ポリユビキチンリセプター、Rpn11:DUB、 β1 (カスパーゼ様活性), β2(トリプシン様活性), β5(キモトリプシン様活性):触媒サブユニット。図には示していないが、USP14はRpn1に、そして Uch37はRpn13を介してRpn2に会合している(図3参照)。]] | |||
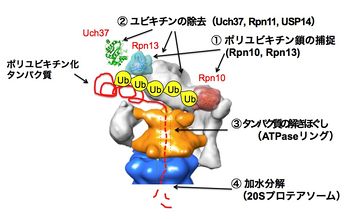
[[image:プロテオソーム3.jpg|thumb|350px|'''図3.26Sプロテアソームの作動機構モデル'''<br>Ub:ユビキチン、Rpn10、Rpn13:ユビキチンリセプター、Rpn11, USP14/(酵母のUbp6), Uch37:脱ユビキチン酵素。詳細は本文及び文献[11][12]参照。]] | |||
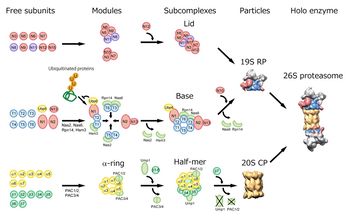
[[image:プロテオソーム4.jpg|thumb|350px|'''図4.26Sプロテアソームの分子集合機構パスウエイ'''<br>詳細は本文及び文献[25]参照。]] | |||
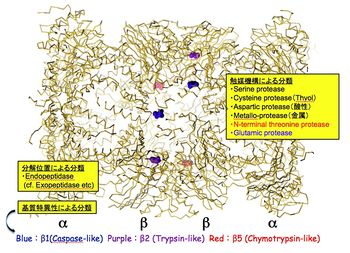
1983年、われわれはユビキチン化タンパク質の分解にもATPのエネルギーが必要であることを見出し“エネルギー依存性タンパク質分解機構の2段階説”を発表した<ref name=ref><pubmed></pubmed></ref>[6]。後に、このATP要求性のタンパク質分解反応を触媒する酵素が、真核生物のATP依存性プロテアーゼであることが判明し、1988年、Proteasome(protease活性を有した巨大粒子〜some)と命名した(厳密には20Sプロテアソームの発見に対する命名:後述)。その後の研究から、このユビキチン化タンパク質を分解する巨大で複雑なタンパク質分解装置は、真核生物のATP依存性プロテアーゼ(26Sプロテアソーム)であり、触媒粒子であるcore particle(CP、別名20Sプロテアソーム)の両端に調節粒子であるregulatory particle (19S RP)が会合した分子量250万、総サブユニット数66個から構成された多成分複合体であることが判明した(図2)<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[7-9]。CPはαリングとβリング(各々7種のサブユニットから構成)がαββαの順で会合した分子量75万の円筒型粒子である。本酵素はカスパーゼ型(β1)、トリプシン型(β2),キモトリプシン型(β5)の触媒活性を有しており、これらの活性中心はβリングの内表面に露出している。CPは、通常、αリングが閉じているため細胞内では不活性型として存在している。2011年、タンパク質合成装置である真核生物リボソームの高次構造がX線結晶解析から解明された(古細菌リボソームの構造と機能に対する研究に対して2009年、ノーベル化学賞が授与された)が、26Sプロテアソームの原子レベルでの構造は不明であり、現在、Cryo-electron microscopy(Cryo-EM:極低温電子顕微鏡)による単粒子解析が進行中である<ref name=ref><pubmed></pubmed></ref>[10]。 | |||
RP(別称:PA700)はlid(蓋部)とbase(基底部)から構成されており,lid複合体とbase複合体は、夫々10個と9個のサブユニットから構成されている。ごく最近、二つのユビキチンリセプターRpn10とRpn13は分子表面の離れた位置に存在してユビキチン化タンパク質を補足していることが判明した<ref name=ref><pubmed></pubmed></ref>[11]。RPにはポリユビキチン鎖を根本から切断して解離するRpn11とそれ以外に末端からユビキチンを1個ずつ解離させる酵素USP14(酵母のUbp6)とUch37(酵母には存在しない)の3つのDUBが存在する。ごく最近、lidサブユニット群の位置情報がCryo-EMよる解析から明らかにされた<ref name=ref><pubmed></pubmed></ref>[12]。またbaseは6種のAAA型ATPaseサブユニット(Rpt1〜Rpt6)を含んでおり、この冠(Crown)型構造のATPaseリングは,CPのαリングと結合してその中央部のゲートを開き,基質タンパク質の通過を可能にさせる機能を有している他、ATPの加水分解エネルギーを利用してタンパク質の3次元構造を破壊(アンフォールディング)し,変性した基質がαリングを通ってβリングの内部に到達できるようにするアンチシャペロン作用を持っている<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[13-15]。このプロテアソームの作動機構を図3に模式化して示した。 | |||
=== | 他方、RP/PA700以外の活性化因子としては、PA28(α,β,γの3種のファミリーを構成)が存在する他<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[16, 17]、20Sプロテアソームの両端にPA700とPA28の両調節ユニットを併せ持った“ハイブリッドプロテアソーム”も存在する<ref name=ref><pubmed></pubmed></ref>[18]。ヘテロ7量体のPA28α/β(細胞質局在)はIFNγによって強く誘導され、内在性抗原のプロセッシングに関与している。ホモ7量体を形成しているPA28γ(核局在)の欠損マウスは成長が遅延する。さらにPA200と名付けられた活性化因子が酵母からヒトまで普遍的に存在するが、その役割は諸説あって確定していない<ref name=ref><pubmed></pubmed></ref>[19]。また20Sプロテアソームが上記の活性化因子の介在なしに単独で、天然変成タンパク質や酸化修飾タンパク質を直接分解することも報告されている<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[20, 21]。このようにプロテアソームの作動機構は、複雑かつ多様である。 | ||
プロテアソームは巨大で複雑な多成分複合体であり、その分子集合には専門的な多数のシャペロン分子が関与している(図4)<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[22, 23]。20Sプロテアソームの形成に特化した分子シャペロンであるPAC(Proteasome Assembling Chaperone)1-4は、生合成された7種のαサブユニットと階層性をもって結合し、αリングの形成を促進する。PAC1/PAC2ヘテロ二量体はαリング同士の凝集体の形成を阻止する働きを示し、PAC3/PAC4ヘテロ二量体はαリング上へのαサブユニットの段階的な会合を促進して、迅速に正確なαリングを形成させる。βサブユニット群は、逐次的にαリング上に会合してβリングを形成する。この段階的な会合にはβ2やβ5のプロペプチド及びβ2のC末端伸長領域などが“分子内シャペロン”として作用する<ref name=ref><pubmed></pubmed></ref>[22]。さらにもう一つのシャペロンUmp1/POMP/Proteassemblinは、βサブユニットの会合やハーフ・プロテアソームの重合プロセスに関与している<ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref> <ref name=ref><pubmed></pubmed></ref>[22, 24]。 | |||
一方、調節部位である 19S RPの分子集合機構についても、最近、baseを構成するATPase リングの分子集合に関与する4種のbaseシャペロン分子が発見された。これら4分子は、当初、プロテアソームと一時的に結合するタンパク質(proteasome-interacting proteins、 PIPs:数十個存在)として同定されていた分子群、即ちNas2/p27, Nas6/gankyrin=p28, Rpn14/PAAF1, and Hsm3/S5b(出芽酵母/ヒト)であった[25](これらは最近、RP assembling chaperones RAC 1-4とも呼ばれている[23])。またUBP6/USP14がユビキチン鎖のbase中間体への偶発的な結合を阻止し、base複合体の形成を促進していることも判明している[26]。 | |||
== | == 生理 == | ||
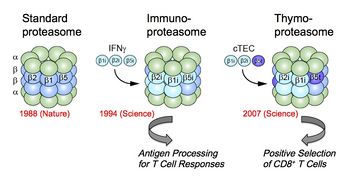
[[image: | [[image:プロテオソーム5.jpg|thumb|350px|'''図5.プロテアソームの多様性:免疫型酵素の発見'''<br>詳細は本文参照。図は文献[39]の図を改変。]] | ||
]] | |||
[[ | UPSをコードする遺伝子の数は、ゲノム総遺伝子数の3〜5 %を占めると推定されており、これが正確であるとすると、このシステムは予想外に多様で重要な役割を担っているのかもしれない[27]。実際UPSは、多様な生体反応を迅速に、順序よく、一過的にかつ一方向に決定する合理的な手段として細胞周期・DNA修復・アポトーシス・シグナル伝達・転写制御・代謝調節・免疫応答・品質管理・ストレス応答・感染応答など生命科学の様々な領域で中心的な役割を果たしている(詳細はユビキチンの項参照)。この多様な生理作用は、細胞内における標的タンパク質の量の厳密な制御を反映しており、とくにユビキチンシステムの多様性に負うところが大きいと考えられている。一方、プロテアソームは単に分解マシーンとしての役割以外に、前駆体タンパク質のプロセシングによる活性型への転換(例えば、NF-κBの成熟プロセス)やその生成ペプチドを抗原エピトープとして利用するなどポジティブな生命応答に貢献していることも知られている。とくに後者は、適応(獲得)免疫の中心的なテーマである自己と非自己の識別において必須な役割を果たすために、プロテアソームのアイソフォーム(免疫型プロテアソーム)(図5)を造成して対処するといった際立った手段を駆使している[28-30]。 | ||
具体的な例を示すと、主要組織適合性遺伝子複合体MHCを獲得した有顎脊椎動物ではプロテアソームはMHCクラスI結合ペプチド産生の必須酵素でもあり、CD8+T細胞を介した細胞性免疫応答に不可欠な役割を果たしている。われわれは、このときウイルスやガン抗原等の内在性抗原のプロセシング酵素として専門的に作用する酵素が存在することを見出し、標準/構成型プロテアソーム(standard/constitutive proteasome)と区別して、1994年、“免疫プロテアソーム(immunoproteasome)”と名付けた[31-33]。この亜型酵素はインターフェロンγ(IFNγ)などのサイトカインにより強く誘導される3種の新しいβ型触媒サブユニット(β1i, β2i, β5i)が優先的分子集合機構によって分子内置換した酵素である。免疫プロテアソームは高いキモトリプシン様活性を有し、MHCクラスIのペプチド収容溝に高い親和性をもつペプチドを効率的に産生することができる(分子レベルでの自己と非自己の識別)。当初免疫プロテアソームは抗原プロセシングに特化した酵素と見られていたが、最近、免疫プロテアソームが有害タンパク質の凝集阻止を通してインターフェロン依存的な酸化ストレスによる細胞死を防御していること[34]やβ5iの特異的な阻害剤PR-957がサイトカインの産生や自己抗体レベルを低下させることから自己免疫疾患に関与していること[35, 36]などの新たな役割を担っていることが示唆されている。 | |||
[[ | さらにわれわれは、脊椎動物の胸腺皮質上皮細胞cTECにはβ5tという新規な触媒サブユニットが特異的に発現していることを見出した。そしてβ5tの組み込まれた(β1iとβ2iをパートナーとする)新種の亜型酵素を、2007年、胸腺プロテアソーム(thymoproteasome)と命名した[37, 38]。胸腺プロテアソームは、MHCクラスIに結合するリガンド(抗原エピトープ)の種類を変化させていると予想された。実際、β5t欠損マウスではCD8+T細胞が著明に減少し、リンパ球分化(様々なTCRを持った有用なCD8+T細胞のレパトア形成)に異常をきたしていることが判明し、胸腺プロテアソームが胸腺における“正の選択”を駆動する抗原ペプチドを生成していることが判明した(細胞レベルでの自己と非自己の識別)[39]。このように免疫型(免疫/胸腺)プロテアソームの遺伝子は、進化的には適応免疫の誕生と同時期に獲得していることは注目される[28, 40]。 | ||
== 病理 == | |||
===エイジングと神経変性疾患=== | |||
[[image:プロテオソーム6.jpg|thumb|350px|'''図6.プロテアソーム分子集合因子PAC1の中枢神経系特異的欠損マウス'''<br>20Sプロテアソーム(αリング)の形成に必須な分子集合因子PAC1(図4参照)の条件的ノックアウトマウスをNestin-Creトランスジェニックマウス を交配させたマウスの生後3週間後の行動動態。詳細は本文及び文献[21]参照。]] | |||
[[image:プロテオソーム7.jpg|thumb|350px|'''図7.PINK1/Parkin依存的な“ミトコンドリア品質管理”仮説のモデル図'''<br>詳細は本文参照。]] | |||
一般にエイジング(老化)と共にプロテアソームの機能が低下するとの報告は、数多くある[41]が、実際にはプロテアソームの機能評価は必ずしも容易でなく、それらの信憑性には疑義がもたれていた。というのは、多くの場合、蛍光合成基質を用いたペプチダーゼ活性を指標とした報告であるが、これらの実験値(活性測定値)が真にこの酵素の細胞内での機能レベルを正確に反映していることの保証はないからである。ところが最近、ハエを用いた遺伝学的スクリーンから老化に依存したニューロンのproteotoxity(異常タンパク質の蓄積による細胞障害)を抑圧する遺伝子としてプロテアソームのRPサブユニット(Rpn11)が分離され、26Sプロテアソームの障害を起因としたプロテアソーム活性の低下が明らかとなった[42]。この結果は、プロテアソームの機能破綻が寿命の短縮に寄与していることを直接的に示しており、エイジングにおけるプロテアソームの役割の重要性が具体的に示唆された。 | |||
老化は、様々な神経変性疾患における最大のリスク要因として挙げられている。通常、活発に分裂している細胞のサイトゾルや核に蓄積した異常タンパク質(アンフォールド/ミスフォールドした変異タンパク質)は、細胞増殖によってクリアランス(浄化)できるが、非分裂細胞であるニューロンにおいては、それらを処理できないために、タンパク質の品質管理(不要タンパク質の処理)が細胞の生存に不可欠である[43, 44]。興味深いことにKopitoらは、細胞内に異常タンパク質を強制発現させると、26Sプロテアソームがそれらを処理できずに活性の急激な低下を引き起こし[45]、蓄積した異常タンパク質が凝集しアグレゾーム(様々な神経変性疾患・患者脳の残存ニューロンに同定されている封入体と類似の凝集構造体)を形成することを示した[46]。この結果は、プロテアソームの機能減弱と神経変性の関連性を強く示唆している。 | |||
他方、McNaughtらはプロテアソーム阻害剤を直接マウスの小脳に注入してパーキンソン病(PD)と類似の症状を引き起こすことを報告し、プロテアソームの抑制とニューロン死の直接的な関係を示唆した[47]。この方法は“McNaughtの方法”として脚光を浴びたが、その後、複数のグループが追試実験を実施したが、成功と失敗が相半ばして再現性が保証されず、この手法に関して決定的な結論が得られていない[48]。しかしごく最近、生後間もないマウスへのプロテアソーム阻害剤の長期間・連続投与によって神経変性が誘導されることが報告され、脚光を浴びている[49]。一方、MayerらはプロテアソームRPを構成するATPaseサブユニットRpt2を脳において条件的にノックアウトすると、ユビキチン陽性のLewy body様の封入体が蓄積すると共に神経変性のトリガーを引くことが出来ることを報告した[50]。われわれも20Sプロテアソームの分子集合因子PAC1をマウス・中枢神経系で欠損させてニューロンのプロテアソームレベルを持続的に低下させると、小脳変性を誘発して神経変性疾患様の症状に陥ることを見出した(図6)。これらの結果は、プロテアソームが神経細胞の恒常性維持に必須であることを遺伝学的に証明したと考えられる[21]。 | |||
一方、多くの神経変性疾患の患者に観察される封入体のほとんどが抗ユビキチン抗体で濃染されること[51]から、UPSの破綻が神経変性疾患の発症原因であるとの主張が華々しく展開された[52]が、UPSの破綻が神経変性疾患の発症に直接的に関わることを示唆する結果は、長い間得られなかった。しかし、1998年、プロテアソームのパートナーであるユビキチン系の酵素であるパーキンが常染色体劣性若年性PD(ARJP)の原因遺伝子であることが同定[53]され、次いで、2000年、パーキンがE3リガーゼをコードしていることが判明したこと[54]から、UPSの破綻と神経変性疾患の関係が全世界で注目されるようになった。即ち、パーキンの標的分子がドーパミンニューロンに蓄積し、細胞死を誘導するという単純な図式が現実味を帯びてきたのである。しかし、爾来10年余、数多くのパーキン基質の同定に関する報告が洪水のように発表されてきたが、ARJPの発症機構を合理的に説明することは、困難を極めた。その後、パーキン研究は意外な展開を見せた。 | |||
不良なミトコンドリア(Mt)の累積は、活性酸素(ROS)を増産させ、DNA・タンパク質・脂質などを修飾して細胞障害を引き起こす。自立的な増殖が可能なMtの品質管理(不良品の処理)は、細胞分裂によって損傷Mtを浄化(クリアランス)できないニューロンなどの非分裂細胞にとっては、健康を維持するために必須である。実際、PDにおけるMtの機能異常(呼吸鎖の低下やMtDNAの欠失など)の報告は、この10年余、集積の一途を辿っている[55]。従ってMtの良・不良をモニター(監視)することは、ニューロンが健全に活動するために不可欠である。これらの知見を受けて最近、Mtの品質管理の研究が、国内外で急速に進展している。Youleらやわれわれは若年性に発症する常染色体劣性遺伝性パーキンソン病の原因遺伝子産物であるPINK1(セリン/スレオニン型タンパク質リン酸化酵素)と Parkin(ユビキチン連結酵素)に着目し,これらのMt品質管理における役割を明らかにすることで,PDの発症機構解明に挑んできた[56, 57]。即ち、通常Mt外膜局在型のPINK1は不安定(健常なMtにおいては、PARL酵素とプロテアソーム系による恒常的な分解を受けている)であるが、膜電位が低下すると、これらの分解系から免れて外膜上に蓄積する。蓄積したPINK1はサイトゾルの不活性型Parkinを損傷Mtに移行・活性型に変換させる。即ちPINK1はParkinの損傷Mtへのリクルート因子として作用する。その結果、複数のMt外膜タンパク質がユビキチン化されると、これが引き金となってプロテアソームによる損傷Mtの消化及び選択的なオートファジーによる分解(Mitophagy)を受け、不良Mtは除去される(詳細は、オートファジーの項参照)[58]。言い換えると、PINK1/Parkin はMtを破壊する“死神”であり、PINK1/Parkinが常に働いているとMtは次々と分解されて細胞は生存できないが、この経路は膜電位が低下した時のみに発動するように巧妙に制御されているので、損傷Mtだけが細胞から除去されることになる。この品質管理が適切に行われずにニューロン内に異常Mtが蓄積すると、ドーパミンニューロンの変性を引き起こしPDが発症すると想定される(図7)。このスキームにおける核心は、不良Mtのモニタリングであり、その機序としてYouleらは、膜電位依存的なPINK1の(PARLが局在する)Mt内膜への輸送仮説を提案しており、その骨子は「膜電位が低下するとPINK1の内膜への輸送が障害されてPINK1が外膜に蓄積する」ことである[59]。一方われわれは膜電位依存的な不活性型PINK1の自己リン酸化による活性化が不良Mtを感知するもう一つのキーメカニズムであることを突き止めた(尾勝ら、論文投稿中)。現在「PINK1 と Parkin が協調して不良Mtの選択的なクリアランスに導く仕組み」の破綻が、PDの発症機構の一翼を担っているメカニズムであることは、確実な情勢となってきつつある。 | |||
===ガン=== | |||
細胞周期制御におけるUPSの重要性は、その基質である サイクリンをHuntらが発見したことを端緒とする(2001年、ノーベル生理学医学賞)。その後、細胞周期に関わる様々な制御因子がリン酸化とUPSに率いられたタンパク質分解で調節されていることが、津波のように生命科学の世界を闊歩した。そしてその破綻がガン化の要因となることの知見は、枚挙に暇が無い状況が今日まで続いている。またガン遺伝子やガン抑制遺伝子の多くが短寿命タンパク質であり、これらがUPSで厳格に調節されている。その代表例は、ゲノムの守護神とも呼ばれているガン抑制遺伝子p53である(詳細は、ユビキチンの項参照)。これらの多くの例は、ユビキチンシステムによる基質の識別が注目されてきており、プロテアソームは調節というよりは寧ろ分解装置としての役割で貢献してきた。 | |||
一方、最近臨床的に注目されているのは、プロテアソーム阻害剤のガン研究における貢献である。スレオニンプロテアーゼであるプロテアソームに対して、これまでに多種多様な(合成・天然)阻害剤が開発・発見されてきた。中でも脚光を浴びているのはPS-341(別名bortezomib、商品名velcade)である[60]。この阻害剤は血液ガンの一種である多発性骨髄腫に際だった効果(ミエローマ細胞のアポトーシス誘導)を示すことが報告され、2003年再発・難治性骨髄腫を対象疾患として米国FDAで認可、現在、欧米を中心に世界の85カ国(日本では2006年)以上で臨床応用されている。さらに副作用の少ない多くのプロテアソーム阻害剤の開発が世界中で凌ぎを削っており、また米国では既存の抗ガン剤との併用を視野に固形ガンを含め多数のガン治療への臨床治験が進行中である。 | |||
===免疫疾患=== | ===免疫疾患=== | ||
最近、免疫プロテアソームのキモトリプシン型活性をコードするβ5i遺伝子(PSMB8)のミスセンス変異が、中条-西村症候群(Nakajo-Nishimura Syndrome NNS:凍瘡様皮疹と限局性脂肪萎縮を伴う遺伝性周期熱症候群)を引き起こすことが判明した[61, 62]。NNSは、1939に発見された遺伝性疾病で、プロテアソームの分子集合異常に起因した機能不全のためにタンパク質の品質管理が破綻し、IL-6が過剰に産生されることによって引き起こされる炎症性疾患である。これはプロテアソームのヒト遺伝性疾患の最初の例であり、国内外で注目されている。 | |||
== おわりに == | |||
本稿ではUPSとくにプロテアソームに焦点を絞って解説してきたが、神経変性疾患に関係しては、近年、協同的に作用するオートファジーの役割が注目されている。オートファジーの最も重要な機能は飢餓適応(低栄養応答)である。即ちオートファジーによる自己成分の分解はアミノ酸供給を通じてエネルギーの恒常的な確保に関与しており、飢餓に対するバックアップシステムと考えられる(誘導的オートファジー)。この意味では、オートファジーは自律的な栄養素確保を可能にする究極の生存戦略となっている。しかしオートファジーは飢餓誘導とは無関係に、日常的に低いレベルでおこっている現象でもある。この基底レベルのオートファジー(恒常的オートファジー)は栄養制御というより、細胞内タンパク質の選択的なクリアランス機構(aggrephagy)として重要であると考えられている。即ちオートファジーは栄養制御と細胞内浄化という大きく二つの役割を果たしているといえる[63]。 | |||
異常タンパク質あるいは変性タンパク質の細胞内蓄積はさまざまな変性疾患や老化過程でも観察されており、プロテアソームは大きな凝集体は処理できないために、これらの病態発症におけるオートファジーの関与が注目されている。実際、多く神経変性疾患においてオートファジーの亢進が報告されている。そして中枢神経系特異的にオートファジーを欠損させたマウスが神経変性疾患の症状を引き起こしたことは、多くの神経病理学者に衝撃を与えた[64, 65]。さらにプロテアソームを阻害すると、オートファジーが亢進することから、この二つの大掛かりなタンパク質分解系には、相互補完的な役割があることも判明している[66]。 | |||
このようにタンパク質分解研究はUPSやオートファジーの発見以来、騎虎の勢いで生命科学の中枢に迫ってきた。しかし現在なお「知っていることよりも知らないことが遙かに多い」という成長期の科学に見られる発展途上の段階にあると言えよう。現在、タンパク質分解システムの阻害剤・活性化剤に関する研究も多方面で行われている。プロテアソームの天然の阻害剤としてlactacystinやepoxomicinが知られているが、前述したようにvelcadeが多発性骨髄腫に対する出色の抗ガン剤として臨床的に多用されていることを背景に、現在、様々な阻害剤が開発され米国で臨床治験が行われている。またプロテアソームのDUBの一つUSP14の阻害剤IU1が、プロテアソームを活性化することが見出され、世間を驚かせた[67]。またプロテアソームに会合している二種のDUB(USP14とUch37/UCHL5)を同時に抑制する阻害薬として開発されたb-AP15が、ガン細胞のアポトーシシスを誘導することが見出され、がん治療への貢献が期待されている[68]。さらにユビキチンリガーゼや脱ユビキチン化酵素の異常で様々な病気が発症することが知られており、これらに対する阻害剤の開発が欧米を中心に活発に行われている。従って近未来にこれらをターゲットとした新薬が開発され、21世紀の長寿社会における健康科学の発展に大きく寄与することが期待されている[69]。 | |||
== 参考文献 == | |||
<references /> | |||
(執筆者:田中啓二、佐伯泰 担当編集委員:林康紀) | |||
2013年3月15日 (金) 11:54時点における版
生体を構成する主要成分であり、生命現象を支える機能素子であるタンパク質は、細胞内で絶えず合成と分解を繰り返しており、きわめて動的なリサイクル(新陳代謝)システムを構成している。即ち、細胞内の全てのタンパク質は、千差万別の寿命をもってダイナミックにターンオーバー(代謝回転)しており、この新陳代謝の中心はタンパク質分解が担っている。タンパク質分解は多様な生体反応を不可逆的に制御する方法として発生や分化など様々な生命現象に不可欠な役割を果たしている。例えば、タンパク質分解は細胞内に生じた不良品の積極的な除去に深く関与しているほか、良品であっても不要な(細胞活動に支障をきたす)場合、あるいは緊急時の栄養素の確保のために、積極的に作動される。このようにタンパク質は細胞内でリサイクルし, 動的平衡を保つことによって身体の中を浄化(不要品のクリアランス)して新鮮さを保ち、健康を維持しているのである。即ち、細胞内のタンパク質は、恒常的な入れ替わり(ターンオーバー)によって品質を管理して健全な細胞活動に貢献していると言い換えることができる。 真核生物の細胞内には進化的に高度に保存された二つの大規模なタンパク質分解系、即ちユビキチン・プロテアソームシステム(UPS)とオートファジー・リソソームシステムが存在している。前者のUPSは選択的タンパク質を担う中心的な酵素系であり、後者は一般に非選択的なタンパク質を担っているが、最近、選択的なオートファジーが数多く発見され、注目されている(詳細は、オートファジーの項参照)。そしてこれらのタンパク質分解しステムは、時空間的に厳密に制御されており、その破綻が高齢化社会を迎えた今日急増しているガンや神経変性疾患などの重篤疾患を引き起こすことが相次いで判明している。
ユビキチンシステム
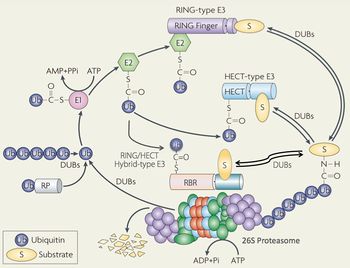
Ub:ユビキチン、E1: Ub活性化酵素、E2: Ub結合酵素、E3: Ubリガーゼ、DUB: 脱ユビキチン酵素。基質を選別するE3酵素は、HECT型とRING型に大別される。26Sプロテアソームは不要なユビキチン化タンパク質をエネルギー依存的に分解する巨大で複雑なタンパク質分解酵素複合体である。(その他の詳細は本文及び文献[22]参照)。UPSの作動機構においてエネルギー(ATPの加水分解)は、基質のユビキチン化(E1の作用)と26Sプロテアソームによる分解作用(基質のアンフォールディング)の二つのプロセスに必要である。
1977年、米国ハーバード大学のGoldbergのグループは網状赤血球の抽出液が非常に高いエネルギー依存性のタンパク質分解活性を示すという画期的な論文を発表した[1][1]。その後間もなく、イスラエルのHershkoとCiechanoverは、酵素学のmentorである米国のRoseと共に、この分解系に注目、その機構解明にいち早く取り組み、熱安定性の小さなタンパク質であるユビキチンがその主役であることを見出した。ユビキチンは76個のアミノ酸からなる小さなタンパク質であり、進化的保存性が高くそのアミノ酸配列は全ての真核生物でほとんど同じである。1980年頃までに彼らは、ユビキチンが活性化酵素(E1)・結合酵素(E2)・リガーゼ(E3)から構成された複合酵素系(ユビキチンシステム)によって標的タンパク質に共有結合(ユビキチンのC末端のカルボキシル基とタンパク質中のリジン残基のε-アミノ基が縮合したイソペプチド結合)する翻訳後修飾分子(モディファイヤー)であることを明らかにした(図1)[1] [1][2, 3]。このE1の作用にはATPの加水分解、即ちエネルギーが必要である。そしてタンパク質に結合したユビキチン内の(主として48番目の)リジン残基と新しいユビキチン分子内のC末端のグリシンの間でイソペプチド結合ができ、さらにユビキチン分子間での縮合反応を繰り返すことによって,多数のユビキチン分子が鎖状に伸長したポリユビキチン鎖が形成されることが判明した(現在では、ユビキチン分子内にある7個のリジン残基と N末端のαアミノ基からユビキチンポリマーが伸長し、多彩な生理機能を担っていることが分かっており、最近、この多様なポリユビキチン鎖に刻印された情報を、われわれは“ユビキチンコード”と定義している)。1980年前後、Hershkoら及びVarshavskyらは生じたポリユビキチン鎖が基質タンパク質を分解装置に輸送するためのシグナル(目印)として機能するという“ユビキチンシグナル”仮説を提唱した[1] [1] [1][2-4]。この仮説の秀逸な点は、ポリユビキチン鎖の形成が(オーバーオールの反応としては)分解シグナルの提示反応であるが、実際に起きている化学反応は(イソ)ペプチド結合の形成(タンパク質合成と類似の反応)であり、エネルギー要求性を見事に説明できることであった。実際、ユビキチン研究の勃興から黎明期をへて今日の隆盛期に至る道程は、まさに破竹の勢いでタンパク質分解の世界を席巻し尽くしてきた。その象徴的な出来事として、2004年、ユビキチンシステムの発見者たち3名は、ノーベル化学賞の栄誉に浴した。
ユビキチンは二つの異なったタイプの遺伝子にコードされている。一つは、ユビキチンとリボソームタンパク質の融合遺伝子であり、もう一つは数個〜10数個のユビキチンがタンデムに連なったポリユビキチン遺伝子である。後者のポリユビキチン遺伝子は、1回の転写・翻訳で多数のユビキチンを合成することができる点で秀逸であり、かつ熱ショック応答遺伝子でもあることから、細胞は環境ストレスに曝されたとき、必要とするユビキチンを迅速に生成することができるように合理的に設計されている。このことは、細胞内のユビキチンレベルが外環境の変化に応答して厳格に制御されていることを示唆している[1][3]。
興味深いことに細胞内には、ユビキチン化の逆反応を触媒する脱ユビキチン酵素(deubiquitylating enzyme:DUB)が存在し、それらは生物種を問わず大きな遺伝子ファミリーを形成している。ヒトゲノムには、典型的な55種のDUBを含めて80種を越える脱ユビキチン酵素がコードされており、これらは6種のサブファミリー(4 UCH, 52 USP, 7 ULP, 2 Josephin domain-type, 16 OTU, 2 JAMM)に細分される[1][5]。多数のDUBが存在することは、ユビキチン化による翻訳後修飾が可逆的かつ多面的であることを示唆している。このようにUSPは、リン酸化- 脱リン酸化システムに匹敵する細胞内制御系と考えられる。
分子構造と作動機構
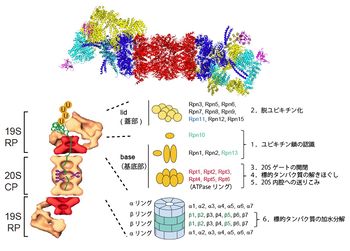
左図:26Sプロテアソーム(CPとRPの複合体)の電子顕微鏡による分子形状(単粒子解析: 独マックスプランク研究所W. Baumeister・S. Nickellから供与)。U:ユビキチン。
右図:サブユニットの構成モデル.CP(20Sプロテアソーム)はα/βリングがαββαの順に会合した円柱状粒子。RP(PA700)はlid(蓋部)とbase(基底部)から構成された複合体。RPはRpn(RP non-ATPase)とRpt(RP triple-ATPase)サブユニット群から構成されている。Rpn10、Rpn13:ポリユビキチンリセプター、Rpn11:DUB、 β1 (カスパーゼ様活性), β2(トリプシン様活性), β5(キモトリプシン様活性):触媒サブユニット。図には示していないが、USP14はRpn1に、そして Uch37はRpn13を介してRpn2に会合している(図3参照)。
Ub:ユビキチン、Rpn10、Rpn13:ユビキチンリセプター、Rpn11, USP14/(酵母のUbp6), Uch37:脱ユビキチン酵素。詳細は本文及び文献[11][12]参照。
詳細は本文及び文献[25]参照。
1983年、われわれはユビキチン化タンパク質の分解にもATPのエネルギーが必要であることを見出し“エネルギー依存性タンパク質分解機構の2段階説”を発表した[1][6]。後に、このATP要求性のタンパク質分解反応を触媒する酵素が、真核生物のATP依存性プロテアーゼであることが判明し、1988年、Proteasome(protease活性を有した巨大粒子〜some)と命名した(厳密には20Sプロテアソームの発見に対する命名:後述)。その後の研究から、このユビキチン化タンパク質を分解する巨大で複雑なタンパク質分解装置は、真核生物のATP依存性プロテアーゼ(26Sプロテアソーム)であり、触媒粒子であるcore particle(CP、別名20Sプロテアソーム)の両端に調節粒子であるregulatory particle (19S RP)が会合した分子量250万、総サブユニット数66個から構成された多成分複合体であることが判明した(図2)[1] [1] [1][7-9]。CPはαリングとβリング(各々7種のサブユニットから構成)がαββαの順で会合した分子量75万の円筒型粒子である。本酵素はカスパーゼ型(β1)、トリプシン型(β2),キモトリプシン型(β5)の触媒活性を有しており、これらの活性中心はβリングの内表面に露出している。CPは、通常、αリングが閉じているため細胞内では不活性型として存在している。2011年、タンパク質合成装置である真核生物リボソームの高次構造がX線結晶解析から解明された(古細菌リボソームの構造と機能に対する研究に対して2009年、ノーベル化学賞が授与された)が、26Sプロテアソームの原子レベルでの構造は不明であり、現在、Cryo-electron microscopy(Cryo-EM:極低温電子顕微鏡)による単粒子解析が進行中である[1][10]。
RP(別称:PA700)はlid(蓋部)とbase(基底部)から構成されており,lid複合体とbase複合体は、夫々10個と9個のサブユニットから構成されている。ごく最近、二つのユビキチンリセプターRpn10とRpn13は分子表面の離れた位置に存在してユビキチン化タンパク質を補足していることが判明した[1][11]。RPにはポリユビキチン鎖を根本から切断して解離するRpn11とそれ以外に末端からユビキチンを1個ずつ解離させる酵素USP14(酵母のUbp6)とUch37(酵母には存在しない)の3つのDUBが存在する。ごく最近、lidサブユニット群の位置情報がCryo-EMよる解析から明らかにされた[1][12]。またbaseは6種のAAA型ATPaseサブユニット(Rpt1〜Rpt6)を含んでおり、この冠(Crown)型構造のATPaseリングは,CPのαリングと結合してその中央部のゲートを開き,基質タンパク質の通過を可能にさせる機能を有している他、ATPの加水分解エネルギーを利用してタンパク質の3次元構造を破壊(アンフォールディング)し,変性した基質がαリングを通ってβリングの内部に到達できるようにするアンチシャペロン作用を持っている[1] [1] [1][13-15]。このプロテアソームの作動機構を図3に模式化して示した。
他方、RP/PA700以外の活性化因子としては、PA28(α,β,γの3種のファミリーを構成)が存在する他[1] [1][16, 17]、20Sプロテアソームの両端にPA700とPA28の両調節ユニットを併せ持った“ハイブリッドプロテアソーム”も存在する[1][18]。ヘテロ7量体のPA28α/β(細胞質局在)はIFNγによって強く誘導され、内在性抗原のプロセッシングに関与している。ホモ7量体を形成しているPA28γ(核局在)の欠損マウスは成長が遅延する。さらにPA200と名付けられた活性化因子が酵母からヒトまで普遍的に存在するが、その役割は諸説あって確定していない[1][19]。また20Sプロテアソームが上記の活性化因子の介在なしに単独で、天然変成タンパク質や酸化修飾タンパク質を直接分解することも報告されている[1] [1][20, 21]。このようにプロテアソームの作動機構は、複雑かつ多様である。 プロテアソームは巨大で複雑な多成分複合体であり、その分子集合には専門的な多数のシャペロン分子が関与している(図4)[1] [1][22, 23]。20Sプロテアソームの形成に特化した分子シャペロンであるPAC(Proteasome Assembling Chaperone)1-4は、生合成された7種のαサブユニットと階層性をもって結合し、αリングの形成を促進する。PAC1/PAC2ヘテロ二量体はαリング同士の凝集体の形成を阻止する働きを示し、PAC3/PAC4ヘテロ二量体はαリング上へのαサブユニットの段階的な会合を促進して、迅速に正確なαリングを形成させる。βサブユニット群は、逐次的にαリング上に会合してβリングを形成する。この段階的な会合にはβ2やβ5のプロペプチド及びβ2のC末端伸長領域などが“分子内シャペロン”として作用する[1][22]。さらにもう一つのシャペロンUmp1/POMP/Proteassemblinは、βサブユニットの会合やハーフ・プロテアソームの重合プロセスに関与している[1] [1] [1][22, 24]。 一方、調節部位である 19S RPの分子集合機構についても、最近、baseを構成するATPase リングの分子集合に関与する4種のbaseシャペロン分子が発見された。これら4分子は、当初、プロテアソームと一時的に結合するタンパク質(proteasome-interacting proteins、 PIPs:数十個存在)として同定されていた分子群、即ちNas2/p27, Nas6/gankyrin=p28, Rpn14/PAAF1, and Hsm3/S5b(出芽酵母/ヒト)であった[25](これらは最近、RP assembling chaperones RAC 1-4とも呼ばれている[23])。またUBP6/USP14がユビキチン鎖のbase中間体への偶発的な結合を阻止し、base複合体の形成を促進していることも判明している[26]。
生理
詳細は本文参照。図は文献[39]の図を改変。
UPSをコードする遺伝子の数は、ゲノム総遺伝子数の3〜5 %を占めると推定されており、これが正確であるとすると、このシステムは予想外に多様で重要な役割を担っているのかもしれない[27]。実際UPSは、多様な生体反応を迅速に、順序よく、一過的にかつ一方向に決定する合理的な手段として細胞周期・DNA修復・アポトーシス・シグナル伝達・転写制御・代謝調節・免疫応答・品質管理・ストレス応答・感染応答など生命科学の様々な領域で中心的な役割を果たしている(詳細はユビキチンの項参照)。この多様な生理作用は、細胞内における標的タンパク質の量の厳密な制御を反映しており、とくにユビキチンシステムの多様性に負うところが大きいと考えられている。一方、プロテアソームは単に分解マシーンとしての役割以外に、前駆体タンパク質のプロセシングによる活性型への転換(例えば、NF-κBの成熟プロセス)やその生成ペプチドを抗原エピトープとして利用するなどポジティブな生命応答に貢献していることも知られている。とくに後者は、適応(獲得)免疫の中心的なテーマである自己と非自己の識別において必須な役割を果たすために、プロテアソームのアイソフォーム(免疫型プロテアソーム)(図5)を造成して対処するといった際立った手段を駆使している[28-30]。 具体的な例を示すと、主要組織適合性遺伝子複合体MHCを獲得した有顎脊椎動物ではプロテアソームはMHCクラスI結合ペプチド産生の必須酵素でもあり、CD8+T細胞を介した細胞性免疫応答に不可欠な役割を果たしている。われわれは、このときウイルスやガン抗原等の内在性抗原のプロセシング酵素として専門的に作用する酵素が存在することを見出し、標準/構成型プロテアソーム(standard/constitutive proteasome)と区別して、1994年、“免疫プロテアソーム(immunoproteasome)”と名付けた[31-33]。この亜型酵素はインターフェロンγ(IFNγ)などのサイトカインにより強く誘導される3種の新しいβ型触媒サブユニット(β1i, β2i, β5i)が優先的分子集合機構によって分子内置換した酵素である。免疫プロテアソームは高いキモトリプシン様活性を有し、MHCクラスIのペプチド収容溝に高い親和性をもつペプチドを効率的に産生することができる(分子レベルでの自己と非自己の識別)。当初免疫プロテアソームは抗原プロセシングに特化した酵素と見られていたが、最近、免疫プロテアソームが有害タンパク質の凝集阻止を通してインターフェロン依存的な酸化ストレスによる細胞死を防御していること[34]やβ5iの特異的な阻害剤PR-957がサイトカインの産生や自己抗体レベルを低下させることから自己免疫疾患に関与していること[35, 36]などの新たな役割を担っていることが示唆されている。
さらにわれわれは、脊椎動物の胸腺皮質上皮細胞cTECにはβ5tという新規な触媒サブユニットが特異的に発現していることを見出した。そしてβ5tの組み込まれた(β1iとβ2iをパートナーとする)新種の亜型酵素を、2007年、胸腺プロテアソーム(thymoproteasome)と命名した[37, 38]。胸腺プロテアソームは、MHCクラスIに結合するリガンド(抗原エピトープ)の種類を変化させていると予想された。実際、β5t欠損マウスではCD8+T細胞が著明に減少し、リンパ球分化(様々なTCRを持った有用なCD8+T細胞のレパトア形成)に異常をきたしていることが判明し、胸腺プロテアソームが胸腺における“正の選択”を駆動する抗原ペプチドを生成していることが判明した(細胞レベルでの自己と非自己の識別)[39]。このように免疫型(免疫/胸腺)プロテアソームの遺伝子は、進化的には適応免疫の誕生と同時期に獲得していることは注目される[28, 40]。
病理
エイジングと神経変性疾患
20Sプロテアソーム(αリング)の形成に必須な分子集合因子PAC1(図4参照)の条件的ノックアウトマウスをNestin-Creトランスジェニックマウス を交配させたマウスの生後3週間後の行動動態。詳細は本文及び文献[21]参照。

詳細は本文参照。
一般にエイジング(老化)と共にプロテアソームの機能が低下するとの報告は、数多くある[41]が、実際にはプロテアソームの機能評価は必ずしも容易でなく、それらの信憑性には疑義がもたれていた。というのは、多くの場合、蛍光合成基質を用いたペプチダーゼ活性を指標とした報告であるが、これらの実験値(活性測定値)が真にこの酵素の細胞内での機能レベルを正確に反映していることの保証はないからである。ところが最近、ハエを用いた遺伝学的スクリーンから老化に依存したニューロンのproteotoxity(異常タンパク質の蓄積による細胞障害)を抑圧する遺伝子としてプロテアソームのRPサブユニット(Rpn11)が分離され、26Sプロテアソームの障害を起因としたプロテアソーム活性の低下が明らかとなった[42]。この結果は、プロテアソームの機能破綻が寿命の短縮に寄与していることを直接的に示しており、エイジングにおけるプロテアソームの役割の重要性が具体的に示唆された。
老化は、様々な神経変性疾患における最大のリスク要因として挙げられている。通常、活発に分裂している細胞のサイトゾルや核に蓄積した異常タンパク質(アンフォールド/ミスフォールドした変異タンパク質)は、細胞増殖によってクリアランス(浄化)できるが、非分裂細胞であるニューロンにおいては、それらを処理できないために、タンパク質の品質管理(不要タンパク質の処理)が細胞の生存に不可欠である[43, 44]。興味深いことにKopitoらは、細胞内に異常タンパク質を強制発現させると、26Sプロテアソームがそれらを処理できずに活性の急激な低下を引き起こし[45]、蓄積した異常タンパク質が凝集しアグレゾーム(様々な神経変性疾患・患者脳の残存ニューロンに同定されている封入体と類似の凝集構造体)を形成することを示した[46]。この結果は、プロテアソームの機能減弱と神経変性の関連性を強く示唆している。
他方、McNaughtらはプロテアソーム阻害剤を直接マウスの小脳に注入してパーキンソン病(PD)と類似の症状を引き起こすことを報告し、プロテアソームの抑制とニューロン死の直接的な関係を示唆した[47]。この方法は“McNaughtの方法”として脚光を浴びたが、その後、複数のグループが追試実験を実施したが、成功と失敗が相半ばして再現性が保証されず、この手法に関して決定的な結論が得られていない[48]。しかしごく最近、生後間もないマウスへのプロテアソーム阻害剤の長期間・連続投与によって神経変性が誘導されることが報告され、脚光を浴びている[49]。一方、MayerらはプロテアソームRPを構成するATPaseサブユニットRpt2を脳において条件的にノックアウトすると、ユビキチン陽性のLewy body様の封入体が蓄積すると共に神経変性のトリガーを引くことが出来ることを報告した[50]。われわれも20Sプロテアソームの分子集合因子PAC1をマウス・中枢神経系で欠損させてニューロンのプロテアソームレベルを持続的に低下させると、小脳変性を誘発して神経変性疾患様の症状に陥ることを見出した(図6)。これらの結果は、プロテアソームが神経細胞の恒常性維持に必須であることを遺伝学的に証明したと考えられる[21]。
一方、多くの神経変性疾患の患者に観察される封入体のほとんどが抗ユビキチン抗体で濃染されること[51]から、UPSの破綻が神経変性疾患の発症原因であるとの主張が華々しく展開された[52]が、UPSの破綻が神経変性疾患の発症に直接的に関わることを示唆する結果は、長い間得られなかった。しかし、1998年、プロテアソームのパートナーであるユビキチン系の酵素であるパーキンが常染色体劣性若年性PD(ARJP)の原因遺伝子であることが同定[53]され、次いで、2000年、パーキンがE3リガーゼをコードしていることが判明したこと[54]から、UPSの破綻と神経変性疾患の関係が全世界で注目されるようになった。即ち、パーキンの標的分子がドーパミンニューロンに蓄積し、細胞死を誘導するという単純な図式が現実味を帯びてきたのである。しかし、爾来10年余、数多くのパーキン基質の同定に関する報告が洪水のように発表されてきたが、ARJPの発症機構を合理的に説明することは、困難を極めた。その後、パーキン研究は意外な展開を見せた。
不良なミトコンドリア(Mt)の累積は、活性酸素(ROS)を増産させ、DNA・タンパク質・脂質などを修飾して細胞障害を引き起こす。自立的な増殖が可能なMtの品質管理(不良品の処理)は、細胞分裂によって損傷Mtを浄化(クリアランス)できないニューロンなどの非分裂細胞にとっては、健康を維持するために必須である。実際、PDにおけるMtの機能異常(呼吸鎖の低下やMtDNAの欠失など)の報告は、この10年余、集積の一途を辿っている[55]。従ってMtの良・不良をモニター(監視)することは、ニューロンが健全に活動するために不可欠である。これらの知見を受けて最近、Mtの品質管理の研究が、国内外で急速に進展している。Youleらやわれわれは若年性に発症する常染色体劣性遺伝性パーキンソン病の原因遺伝子産物であるPINK1(セリン/スレオニン型タンパク質リン酸化酵素)と Parkin(ユビキチン連結酵素)に着目し,これらのMt品質管理における役割を明らかにすることで,PDの発症機構解明に挑んできた[56, 57]。即ち、通常Mt外膜局在型のPINK1は不安定(健常なMtにおいては、PARL酵素とプロテアソーム系による恒常的な分解を受けている)であるが、膜電位が低下すると、これらの分解系から免れて外膜上に蓄積する。蓄積したPINK1はサイトゾルの不活性型Parkinを損傷Mtに移行・活性型に変換させる。即ちPINK1はParkinの損傷Mtへのリクルート因子として作用する。その結果、複数のMt外膜タンパク質がユビキチン化されると、これが引き金となってプロテアソームによる損傷Mtの消化及び選択的なオートファジーによる分解(Mitophagy)を受け、不良Mtは除去される(詳細は、オートファジーの項参照)[58]。言い換えると、PINK1/Parkin はMtを破壊する“死神”であり、PINK1/Parkinが常に働いているとMtは次々と分解されて細胞は生存できないが、この経路は膜電位が低下した時のみに発動するように巧妙に制御されているので、損傷Mtだけが細胞から除去されることになる。この品質管理が適切に行われずにニューロン内に異常Mtが蓄積すると、ドーパミンニューロンの変性を引き起こしPDが発症すると想定される(図7)。このスキームにおける核心は、不良Mtのモニタリングであり、その機序としてYouleらは、膜電位依存的なPINK1の(PARLが局在する)Mt内膜への輸送仮説を提案しており、その骨子は「膜電位が低下するとPINK1の内膜への輸送が障害されてPINK1が外膜に蓄積する」ことである[59]。一方われわれは膜電位依存的な不活性型PINK1の自己リン酸化による活性化が不良Mtを感知するもう一つのキーメカニズムであることを突き止めた(尾勝ら、論文投稿中)。現在「PINK1 と Parkin が協調して不良Mtの選択的なクリアランスに導く仕組み」の破綻が、PDの発症機構の一翼を担っているメカニズムであることは、確実な情勢となってきつつある。
ガン
細胞周期制御におけるUPSの重要性は、その基質である サイクリンをHuntらが発見したことを端緒とする(2001年、ノーベル生理学医学賞)。その後、細胞周期に関わる様々な制御因子がリン酸化とUPSに率いられたタンパク質分解で調節されていることが、津波のように生命科学の世界を闊歩した。そしてその破綻がガン化の要因となることの知見は、枚挙に暇が無い状況が今日まで続いている。またガン遺伝子やガン抑制遺伝子の多くが短寿命タンパク質であり、これらがUPSで厳格に調節されている。その代表例は、ゲノムの守護神とも呼ばれているガン抑制遺伝子p53である(詳細は、ユビキチンの項参照)。これらの多くの例は、ユビキチンシステムによる基質の識別が注目されてきており、プロテアソームは調節というよりは寧ろ分解装置としての役割で貢献してきた。
一方、最近臨床的に注目されているのは、プロテアソーム阻害剤のガン研究における貢献である。スレオニンプロテアーゼであるプロテアソームに対して、これまでに多種多様な(合成・天然)阻害剤が開発・発見されてきた。中でも脚光を浴びているのはPS-341(別名bortezomib、商品名velcade)である[60]。この阻害剤は血液ガンの一種である多発性骨髄腫に際だった効果(ミエローマ細胞のアポトーシス誘導)を示すことが報告され、2003年再発・難治性骨髄腫を対象疾患として米国FDAで認可、現在、欧米を中心に世界の85カ国(日本では2006年)以上で臨床応用されている。さらに副作用の少ない多くのプロテアソーム阻害剤の開発が世界中で凌ぎを削っており、また米国では既存の抗ガン剤との併用を視野に固形ガンを含め多数のガン治療への臨床治験が進行中である。
免疫疾患
最近、免疫プロテアソームのキモトリプシン型活性をコードするβ5i遺伝子(PSMB8)のミスセンス変異が、中条-西村症候群(Nakajo-Nishimura Syndrome NNS:凍瘡様皮疹と限局性脂肪萎縮を伴う遺伝性周期熱症候群)を引き起こすことが判明した[61, 62]。NNSは、1939に発見された遺伝性疾病で、プロテアソームの分子集合異常に起因した機能不全のためにタンパク質の品質管理が破綻し、IL-6が過剰に産生されることによって引き起こされる炎症性疾患である。これはプロテアソームのヒト遺伝性疾患の最初の例であり、国内外で注目されている。
おわりに
本稿ではUPSとくにプロテアソームに焦点を絞って解説してきたが、神経変性疾患に関係しては、近年、協同的に作用するオートファジーの役割が注目されている。オートファジーの最も重要な機能は飢餓適応(低栄養応答)である。即ちオートファジーによる自己成分の分解はアミノ酸供給を通じてエネルギーの恒常的な確保に関与しており、飢餓に対するバックアップシステムと考えられる(誘導的オートファジー)。この意味では、オートファジーは自律的な栄養素確保を可能にする究極の生存戦略となっている。しかしオートファジーは飢餓誘導とは無関係に、日常的に低いレベルでおこっている現象でもある。この基底レベルのオートファジー(恒常的オートファジー)は栄養制御というより、細胞内タンパク質の選択的なクリアランス機構(aggrephagy)として重要であると考えられている。即ちオートファジーは栄養制御と細胞内浄化という大きく二つの役割を果たしているといえる[63]。
異常タンパク質あるいは変性タンパク質の細胞内蓄積はさまざまな変性疾患や老化過程でも観察されており、プロテアソームは大きな凝集体は処理できないために、これらの病態発症におけるオートファジーの関与が注目されている。実際、多く神経変性疾患においてオートファジーの亢進が報告されている。そして中枢神経系特異的にオートファジーを欠損させたマウスが神経変性疾患の症状を引き起こしたことは、多くの神経病理学者に衝撃を与えた[64, 65]。さらにプロテアソームを阻害すると、オートファジーが亢進することから、この二つの大掛かりなタンパク質分解系には、相互補完的な役割があることも判明している[66]。
このようにタンパク質分解研究はUPSやオートファジーの発見以来、騎虎の勢いで生命科学の中枢に迫ってきた。しかし現在なお「知っていることよりも知らないことが遙かに多い」という成長期の科学に見られる発展途上の段階にあると言えよう。現在、タンパク質分解システムの阻害剤・活性化剤に関する研究も多方面で行われている。プロテアソームの天然の阻害剤としてlactacystinやepoxomicinが知られているが、前述したようにvelcadeが多発性骨髄腫に対する出色の抗ガン剤として臨床的に多用されていることを背景に、現在、様々な阻害剤が開発され米国で臨床治験が行われている。またプロテアソームのDUBの一つUSP14の阻害剤IU1が、プロテアソームを活性化することが見出され、世間を驚かせた[67]。またプロテアソームに会合している二種のDUB(USP14とUch37/UCHL5)を同時に抑制する阻害薬として開発されたb-AP15が、ガン細胞のアポトーシシスを誘導することが見出され、がん治療への貢献が期待されている[68]。さらにユビキチンリガーゼや脱ユビキチン化酵素の異常で様々な病気が発症することが知られており、これらに対する阻害剤の開発が欧米を中心に活発に行われている。従って近未来にこれらをターゲットとした新薬が開発され、21世紀の長寿社会における健康科学の発展に大きく寄与することが期待されている[69]。
参考文献
(執筆者:田中啓二、佐伯泰 担当編集委員:林康紀)