「ヒストン脱アセチル化酵素」の版間の差分
細編集の要約なし |
細 (→分類) |
||
| 16行目: | 16行目: | ||
== 分類 == | == 分類 == | ||
現在までに、哺乳動物では18種類のヒストン脱アセチル化酵素が同定されており、構造の違いからクラスIからクラスIVに大別される('''表1''') | 現在までに、哺乳動物では18種類のヒストン脱アセチル化酵素が同定されており、構造の違いからクラスIからクラスIVに大別される('''表1''')。クラスⅠ([[HDAC1]]、[[HDAC2|2]]、[[HDAC3|3]]、[[HDAC8|8]])、クラスIIa([[HDAC4]]、[[HDAC5|5]]、[[HDAC7|7]]、[[HDAC9|9]])、クラスIIb([[HDAC6]]、[[HDAC10|10]])、クラスIII([[SIRT1]]~[[SIRT7|7]])、クラスIV([[HDAC11]])である。ヒストン脱アセチル化酵素は単独の酵素として機能するのではなく、[[転写因子]]や[[コリプレッサー]]などを含む複数の構成要素からなる大きな複合体を形成することで脱アセチル化酵素として機能している。 | ||
{| class="wikitable" style="text-align: center; " | {| class="wikitable" style="text-align: center; " | ||
| 25行目: | 25行目: | ||
| rowspan="4"|I||[[HDAC1]]||1||核||ユビキタス||[[アンドロゲン受容体]]、[[SHP]], [[p53]], [[MyoD]], [[E2F1]], [[STAT3]], [[MEF2D]]|| -||胚性致死、ヒストンアセチル化の増加、[[p21]]と[[p27]]の増加。 | | rowspan="4"|I||[[HDAC1]]||1||核||ユビキタス||[[アンドロゲン受容体]]、[[SHP]], [[p53]], [[MyoD]], [[E2F1]], [[STAT3]], [[MEF2D]]|| -||胚性致死、ヒストンアセチル化の増加、[[p21]]と[[p27]]の増加。 | ||
|- | |- | ||
| [[HDAC2]] || 1||核||ユビキタス||[[グルココルチコイド受容体]]、[[YY1]]、[[BCL6]]、STAT3|| - || 心筋障害 | | [[HDAC2]] || 1||[[核]]||ユビキタス||[[グルココルチコイド受容体]]、[[YY1]]、[[BCL6]]、STAT3|| - || 心筋障害 | ||
|- | |- | ||
| [[HDAC3]] || 1||核||ユビキタス||SHP、YY1、[[GATA1]]、[[RELA]]、STAT3、MEF2D||[[NCOR1]]|| - | | [[HDAC3]] || 1||核||ユビキタス||SHP、YY1、[[GATA1]]、[[RELA]]、STAT3、MEF2D||[[NCOR1]]|| - | ||
|- | |- | ||
| [[HDAC8]] || 1|| | | [[HDAC8]] || 1||核・[[細胞質]]||ユビキタス?|| - ||[[EST1B]]||- | ||
|- | |- | ||
| rowspan="4"| IIA || [[HDAC4]]|| 1||核・細胞質|| | | rowspan="4"| IIA || [[HDAC4]]|| 1||核・細胞質||[[心臓]]・[[骨格筋]]・[[脳]]||[[GCMA]], GATA1, [[HP1]]||[[RFXANK]]||[[軟骨細胞]]分化の欠陥 | ||
|- | |- | ||
| [[HDAC5]] || 1||核・細胞質||心臓・骨格筋・脳||GCMA, [[SMAD7]], HP1|| [[REA]]、[[エストロゲン受容体]]|| 心筋障害 | | [[HDAC5]] || 1||核・細胞質||心臓・骨格筋・脳||GCMA, [[SMAD7]], HP1|| [[REA]]、[[エストロゲン受容体]]|| 心筋障害 | ||
|- | |- | ||
| [[HDAC7]] || 1||核・細胞質・ミトコンドリア|| | | [[HDAC7]] || 1||核・細胞質・ミトコンドリア||心臓・骨格筋・[[膵臓]]・[[胎盤]]||[[PLAG1]], [[PLAG2]]||[[HIF1A]], [[BCL6]], [[エンドセリン受容体]], [[ACTN1]], [[ACTN4]], [[アンドロゲン受容体]], [[KAT5|Tip60]]||血管完全性の維持、[[MMP10]]の増加 | ||
|- | |- | ||
| [[HDAC9]]|| 1||核・細胞質||脳・骨格筋||-||[[FOXP3]]|| 心筋障害 | | [[HDAC9]]|| 1||核・細胞質||脳・骨格筋||-||[[FOXP3]]|| 心筋障害 | ||
|- | |- | ||
| rowspan="2" |IIB || [[HDAC6]] || 2||ほとんどが細胞質|| | | rowspan="2" |IIB || [[HDAC6]] || 2||ほとんどが細胞質||心臓、肝臓、[[腎臓]]、胎盤|| [[α-チューブリン]], [[HSP90]], SHP, SMAD7||[[RUNX2]]|| - | ||
|- | |- | ||
|| [[HDAC10]] ||1||ほとんどが細胞質||肝臓、脾臓、腎臓|| - || - || - | || [[HDAC10]] ||1||ほとんどが細胞質||肝臓、脾臓、腎臓|| - || - || - | ||
2023年6月1日 (木) 18:55時点における版
内田 周作
京都大学大学院医学研究科
DOI:10.14931/bsd.3112 原稿受付日:2023年5月22日 原稿完成日:2023年5月31日
担当編集委員:和田 圭司(国立研究開発法人国立精神・神経医療研究センター)
英:histone deacetylase
英略称:HDAC
ヒストン脱アセチル化酵素はタンパク質のリジン残基のアセチル基を取り除く酵素。脳においては、神経細胞の分化・増殖や神経可塑性などに重要な役割を担っており、神経精神疾患との関連が示唆されている。
ヒストン脱アセチル化酵素とは
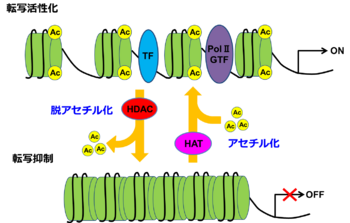
DNAはヒストンに巻き付いてヌクレオソームと呼ばれるユニットにまとめられ、さらにこのユニットを高度に折り畳むことでクロマチンを形成して細胞核にDNAを収納している。ヌクレオソームは、コアヒストンと総称される4種類のヒストン(H2A、H2B、 H3、 H4) の各2分子ずつで形成される8量体の周りに145~147 bp の二本鎖DNAが1.75 回巻き付いた構造を持っている。各々のヒストンには、安定な8量体を形成する際に重要なカルボキシル末端側の「フォールドドメイン」と、DNA が巻き付いた状態でもヌクレオソーム構造から外に突出するような状態で存在し、特定の二次構造を持たないアミノ末端側の「テールドメイン」という二つのドメインを持っている。フォールドドメインはDNA の収納に必須な構造体であるのに対し、テールドメインは細胞内で、アセチル化、メチル化、リン酸化、ユビキチン化など様々な翻訳後修飾を受けることが知られている(図1)。コアヒストンのテールドメインは、正電荷を持つアミノ酸残基に富んでおり、その中の特定の側鎖がアセチル化されると、電荷が変化することでDNA との相互作用が弱まり、ヌクレオソームの構造変化を伴って転写因子などのDNA 結合タンパク質が接近しやすくなる。逆に、ヒストン脱アセチル化酵素の作用によってヒストンが脱アセチル化されると、DNAと強く結合することでクロマチン構造がコンパクトになって標的遺伝子の発現が抑制される。
分類
現在までに、哺乳動物では18種類のヒストン脱アセチル化酵素が同定されており、構造の違いからクラスIからクラスIVに大別される(表1)。クラスⅠ(HDAC1、2、3、8)、クラスIIa(HDAC4、5、7、9)、クラスIIb(HDAC6、10)、クラスIII(SIRT1~7)、クラスIV(HDAC11)である。ヒストン脱アセチル化酵素は単独の酵素として機能するのではなく、転写因子やコリプレッサーなどを含む複数の構成要素からなる大きな複合体を形成することで脱アセチル化酵素として機能している。
| クラス | メンバー | 触媒部位 | 細胞内局在 | 組織分布 | 基質 | 結合タンパク質 | ノックアウト表現型 |
|---|---|---|---|---|---|---|---|
| I | HDAC1 | 1 | 核 | ユビキタス | アンドロゲン受容体、SHP, p53, MyoD, E2F1, STAT3, MEF2D | - | 胚性致死、ヒストンアセチル化の増加、p21とp27の増加。 |
| HDAC2 | 1 | 核 | ユビキタス | グルココルチコイド受容体、YY1、BCL6、STAT3 | - | 心筋障害 | |
| HDAC3 | 1 | 核 | ユビキタス | SHP、YY1、GATA1、RELA、STAT3、MEF2D | NCOR1 | - | |
| HDAC8 | 1 | 核・細胞質 | ユビキタス? | - | EST1B | - | |
| IIA | HDAC4 | 1 | 核・細胞質 | 心臓・骨格筋・脳 | GCMA, GATA1, HP1 | RFXANK | 軟骨細胞分化の欠陥 |
| HDAC5 | 1 | 核・細胞質 | 心臓・骨格筋・脳 | GCMA, SMAD7, HP1 | REA、エストロゲン受容体 | 心筋障害 | |
| HDAC7 | 1 | 核・細胞質・ミトコンドリア | 心臓・骨格筋・膵臓・胎盤 | PLAG1, PLAG2 | HIF1A, BCL6, エンドセリン受容体, ACTN1, ACTN4, アンドロゲン受容体, Tip60 | 血管完全性の維持、MMP10の増加 | |
| HDAC9 | 1 | 核・細胞質 | 脳・骨格筋 | - | FOXP3 | 心筋障害 | |
| IIB | HDAC6 | 2 | ほとんどが細胞質 | 心臓、肝臓、腎臓、胎盤 | α-チューブリン, HSP90, SHP, SMAD7 | RUNX2 | - |
| HDAC10 | 1 | ほとんどが細胞質 | 肝臓、脾臓、腎臓 | - | - | - | |
| III | サーチュイン(SIRT1, SIRT2, SIRT3, SIRT4, SIRT5, SIRT6, SIRT7) | - | - | - | - | - | - |
| IV | HDAC11 | 2 | 核・細胞質 | 脳、心臓、骨格筋、腎臓 | - | - | - |
Wikipediaより、編集部にて翻訳、改変。
発現
クラスⅠは主に核に局在し、クラスⅡ~IVは核と細胞質に局在するが脳部位や細胞種によって異なる可能性がある。また、クラスIIbについては神経活動や環境依存的に細胞内局在が変化することが報告されている[1][2] 。
機能
ヒストン脱アセチル化酵素の神経可塑性に対する役割はまず記憶研究から明らかとなった。マウスへのヒストン脱アセチル化酵素阻害剤投与は神経細胞樹状突起スパイン密度増加と長期記憶形成の増強を引き起こす[3][4][5][6] 。マウスへの文脈学習課題はBdnf遺伝子プロモーター領域のヒストンH3のアセチル化を増加させ、その転写活性を増大させる[3] 。このBdnf遺伝子プロモーターのヒストン脱アセチル化を担う分子としてHDAC2が見出されている[4] 。HDAC2過剰発現マウスは、Bdnf遺伝子上のヒストンアセチル化レベルの低下、海馬神経細胞樹状突起スパイン密度の低下、長期増強の減退、記憶能力の低下といった表現型を示し、逆にHDAC2遺伝子欠損マウスはスパイン密度の増加、長期増強の亢進、記憶能力の促進を示した。
核内ヒストンタンパクの脱アセチル化酵素として働くHDAC4は脳内に広く分布しており、神経活動に伴いリン酸化されることで核から細胞質に移行する。この結果、シナプス造成やシナプス再構築に関わる遺伝子群を抑制するためのスイッチが解除され、結果的に標的遺伝子の発現が活性化される[1] 。脳内HDAC4の活性化はシナプス可塑性を低下させ、記憶・学習能力の低下を引き起こす[1] 。このように、ヒストン脱アセチル化酵素によるエピジェネティック修飾がクロマチンの構造をダイナミックに変化させることで神経可塑性に影響を与え、その結果、記憶・学習や情動・気分の制御に関わっていると想定されている。
上記のヒストン脱アセチル化酵素はヒストン修飾により遺伝子発現を調節する機能を担うが、非ヒストンタンパク質を脱アセチル化するヒストン脱アセチル化酵素も知られている。HDAC6の主な基質はα-チュブリンであり、微小管の安定性を制御することで、様々なタンパク質やミトコンドリアなどのオルガネラの細胞内輸送等に重要な役割を果たしている [7] 。
疾患との関わり
アルツハイマー病患者死後脳海馬におけるHDAC2発現の増加が観察されており、HDAC2のアルツハイマー病態に対する関与が示唆されている[8] 。
大うつ病性障害、双極性障害、統合失調症患者における大規模ゲノム関連解析(GWAS)の結果、いずれの疾患においてもヒストン修飾に関わる遺伝子パスウェイとの強い関連が見出されている [9] 。
慢性ストレスを負荷したうつモデルマウスやうつ病患者死後脳において、様々なクラスのヒストン脱アセチル化酵素の発現・機能異常が観察されている。慢性的ストレスを曝露したうつ状態のマウスにおいてHDAC2(側坐核)やHDAC4/5(海馬)の有意な発現増加が認められている [10][11] 。一方、ヒストン脱アセチル化酵素阻害剤投与マウスにおいては慢性ストレス負荷後のうつ様行動が消失していた。これらの結果は、ストレス負荷によるHDAC2やHDAC4/5の発現・機能亢進が標的遺伝子群の発現を抑制することでうつ様行動を誘発することを示唆している。
気分障害とSIRT1遺伝子との関連を示唆するエビデンスも多数報告されている。2010年、日本人の大うつ病性障害患者におけるSIRT1遺伝子の多型解析から、SIRT1遺伝子rs10997875と大うつ病性障害との有意な関連が報告された [12] 。また、2011年にはSIRT1遺伝子rs12413112と大うつ病性障害との有意な関連が報告された [13] 。さらに、2015年、4509人の大うつ病性障害の女性患者に限定した全ゲノムシークエンシング解析の結果、SIRT1遺伝子が同定された [14] 。遺伝子発現解析においても大うつ病性障害患者におけるSIRT1遺伝子の有意な発現減少が報告されている [15][16] 。非臨床研究においてもうつモデルマウス海馬におけるSIRT1発現の有意な低下とSIRT1活性化剤投与による抗うつ様作用が報告されている [17] 。
コカインの乱用によりひき起こされる異常行動は、薬物のエピジェネティックな作用が原因となっていることが示唆されている。HDAC5はコカインにより誘発されたcAMPシグナル伝達系により核へと移行し,コカインにより誘発される報酬行動に関わることから、依存との関連が示唆されている[2] 。
自閉症とヒストン脱アセチル化酵素との関連も示唆されている。特発性自閉症の原因として、胎児期の造血系細胞のHDAC1異常に起因する脳や腸に見られる免疫異常が指摘されている [18] 。また、自閉症モデルマウスへのヒストン脱アセチル化酵素阻害剤投与は社会性低下を回復させる [19] 。
精神疾患病態に加えて、向精神薬の作用/副作用機序との関連も示唆されている。抗うつ薬による抗うつ効果にはHDAC5阻害活性が必須であること [20] 、抗精神病薬による認知機能低下といった副作用にはHDAC2機能亢進が関わっていることが報告されている [21] 。
ヒストン脱アセチル化酵素阻害剤
ヒストン脱アセチル化酵素阻害剤はがん、感染症、炎症性疾患に対する治療薬としての開発が進められている(表2)が、上述の通り最近では、うつ病、自閉症、アルツハイマー病などの神経精神疾患に対する治療薬としての可能性も期待されている。そのため、脳移行性が高くアイソフォーム選択的なヒストン脱アセチル化酵素阻害剤が必要とされている。
古典的なヒストン脱アセチル化酵素阻害剤はクラスI、II、IVに作用し、ヒストン脱アセチル化酵素の亜鉛含有触媒部位に結合する。クラスIIIのヒストン脱アセチル化酵素であるサーチュインはNAD+に依存しており、ニコチンアミドとその誘導体によって阻害される。
| 名称 | タイプ | 選択性 | ポテンシー |
|---|---|---|---|
| バルプロ酸 (VPA) | 脂肪族化合物 | クラスI, IIa | mM |
| 酪酸ナトリウム(NaB) | 脂肪族化合物 | クラスI, IIa | mM |
| トリコスタチンA (TSA) | ヒドロキサム酸系 | Pan | nM |
| スベロイルアニリドヒドロキサム酸 (SAHA, ボリノスタット) | ヒドロキサム酸系 | Pan | μM |
| APHA 8 | ヒドロキサム酸系 | クラスI | μM |
| MC1568 | ヒドロキサム酸系 | クラスII | nM |
| 1-ナフトルヒドロキサム酸 | ヒドロキサム酸系 | HDAC8 | μM |
| エンチノスタット (MS-275) | ベンズアミド系 | クラスI | μM |
| ツバスタチンA (TBSA) | ベンズアミド系 | HDAC6 | nM |
| RGFP966 | ベンズアミド系 | HDAC3 | nM |
| シルチノ-ル | - | SIRT1, SIRT2 | μM |
| Ex-527 | - | SIRT1 | nM |
| AGK2 | - | SIRT2 | μM |
関連語
参考文献
- ↑ 1.0 1.1 1.2
Sando, R., Gounko, N., Pieraut, S., Liao, L., Yates, J., & Maximov, A. (2012).
HDAC4 governs a transcriptional program essential for synaptic plasticity and memory. Cell, 151(4), 821-834. [PubMed:23141539] [PMC] [WorldCat] [DOI] - ↑ 2.0 2.1
Taniguchi, M., Carreira, M.B., Smith, L.N., Zirlin, B.C., Neve, R.L., & Cowan, C.W. (2012).
Histone deacetylase 5 limits cocaine reward through cAMP-induced nuclear import. Neuron, 73(1), 108-20. [PubMed:22243750] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1
Bredy, T.W., & Barad, M. (2008).
The histone deacetylase inhibitor valproic acid enhances acquisition, extinction, and reconsolidation of conditioned fear. Learning & memory (Cold Spring Harbor, N.Y.), 15(1), 39-45. [PubMed:18174372] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1
Guan, J.S., Haggarty, S.J., Giacometti, E., Dannenberg, J.H., Joseph, N., Gao, J., ..., & Tsai, L.H. (2009).
HDAC2 negatively regulates memory formation and synaptic plasticity. Nature, 459(7243), 55-60. [PubMed:19424149] [PMC] [WorldCat] [DOI] - ↑
Levenson, J.M., O'Riordan, K.J., Brown, K.D., Trinh, M.A., Molfese, D.L., & Sweatt, J.D. (2004).
Regulation of histone acetylation during memory formation in the hippocampus. The Journal of biological chemistry, 279(39), 40545-59. [PubMed:15273246] [WorldCat] [DOI] - ↑
Wood, M.A., Hawk, J.D., & Abel, T. (2006).
Combinatorial chromatin modifications and memory storage: a code for memory? Learning & memory (Cold Spring Harbor, N.Y.), 13(3), 241-4. [PubMed:16741277] [PMC] [WorldCat] [DOI] - ↑
Matsuyama, A., Shimazu, T., Sumida, Y., Saito, A., Yoshimatsu, Y., Seigneurin-Berny, D., ..., & Yoshida, M. (2002).
In vivo destabilization of dynamic microtubules by HDAC6-mediated deacetylation. The EMBO journal, 21(24), 6820-31. [PubMed:12486003] [PMC] [WorldCat] [DOI] - ↑
Gräff, J., Rei, D., Guan, J.S., Wang, W.Y., Seo, J., Hennig, K.M., ..., & Tsai, L.H. (2012).
An epigenetic blockade of cognitive functions in the neurodegenerating brain. Nature, 483(7388), 222-6. [PubMed:22388814] [PMC] [WorldCat] [DOI] - ↑
Network and Pathway Analysis Subgroup of Psychiatric Genomics Consortium (2015).
Psychiatric genome-wide association study analyses implicate neuronal, immune and histone pathways. Nature neuroscience, 18(2), 199-209. [PubMed:25599223] [PMC] [WorldCat] [DOI] - ↑
Higuchi, F., Uchida, S., Yamagata, H., Abe-Higuchi, N., Hobara, T., Hara, K., ..., & Watanabe, Y. (2016).
Hippocampal MicroRNA-124 Enhances Chronic Stress Resilience in Mice. The Journal of neuroscience : the official journal of the Society for Neuroscience, 36(27), 7253-67. [PubMed:27383599] [PMC] [WorldCat] [DOI] - ↑
Uchida, S., Hara, K., Kobayashi, A., Otsuki, K., Yamagata, H., Hobara, T., ..., & Watanabe, Y. (2011).
Epigenetic status of Gdnf in the ventral striatum determines susceptibility and adaptation to daily stressful events. Neuron, 69(2), 359-72. [PubMed:21262472] [WorldCat] [DOI] - ↑
Kishi, T., Yoshimura, R., Kitajima, T., Okochi, T., Okumura, T., Tsunoka, T., ..., & Iwata, N. (2010).
SIRT1 gene is associated with major depressive disorder in the Japanese population. Journal of affective disorders, 126(1-2), 167-73. [PubMed:20451257] [WorldCat] [DOI] - ↑
Libert, S., Pointer, K., Bell, E.L., Das, A., Cohen, D.E., Asara, J.M., ..., & Guarente, L. (2011).
SIRT1 activates MAO-A in the brain to mediate anxiety and exploratory drive. Cell, 147(7), 1459-72. [PubMed:22169038] [PMC] [WorldCat] [DOI] - ↑
CONVERGE consortium (2015).
Sparse whole-genome sequencing identifies two loci for major depressive disorder. Nature, 523(7562), 588-91. [PubMed:26176920] [PMC] [WorldCat] [DOI] - ↑
Abe, N., Uchida, S., Otsuki, K., Hobara, T., Yamagata, H., Higuchi, F., ..., & Watanabe, Y. (2011).
Altered sirtuin deacetylase gene expression in patients with a mood disorder. Journal of psychiatric research, 45(8), 1106-12. [PubMed:21349544] [WorldCat] [DOI] - ↑
Liu, W., Yan, H., Zhou, D., Cai, X., Zhang, Y., Li, S., ..., & Xiao, X. (2019).
The depression GWAS risk allele predicts smaller cerebellar gray matter volume and reduced SIRT1 mRNA expression in Chinese population. Translational psychiatry, 9(1), 333. [PubMed:31819045] [PMC] [WorldCat] [DOI] - ↑
Abe-Higuchi, N., Uchida, S., Yamagata, H., Higuchi, F., Hobara, T., Hara, K., ..., & Watanabe, Y. (2016).
Hippocampal Sirtuin 1 Signaling Mediates Depression-like Behavior. Biological psychiatry, 80(11), 815-826. [PubMed:27016384] [WorldCat] [DOI] - ↑
Lin, C.W., Septyaningtrias, D.E., Chao, H.W., Konda, M., Atarashi, K., Takeshita, K., ..., & Takumi, T. (2022).
A common epigenetic mechanism across different cellular origins underlies systemic immune dysregulation in an idiopathic autism mouse model. Molecular psychiatry, 27(8), 3343-3354. [PubMed:35491410] [WorldCat] [DOI] - ↑
Qin, L., Ma, K., Wang, Z.J., Hu, Z., Matas, E., Wei, J., & Yan, Z. (2018).
Social deficits in Shank3-deficient mouse models of autism are rescued by histone deacetylase (HDAC) inhibition. Nature neuroscience, 21(4), 564-575. [PubMed:29531362] [PMC] [WorldCat] [DOI] - ↑
Tsankova, N.M., Berton, O., Renthal, W., Kumar, A., Neve, R.L., & Nestler, E.J. (2006).
Sustained hippocampal chromatin regulation in a mouse model of depression and antidepressant action. Nature neuroscience, 9(4), 519-25. [PubMed:16501568] [WorldCat] [DOI] - ↑
Ibi, D., de la Fuente Revenga, M., Kezunovic, N., Muguruza, C., Saunders, J.M., Gaitonde, S.A., ..., & González-Maeso, J. (2017).
Antipsychotic-induced Hdac2 transcription via NF-κB leads to synaptic and cognitive side effects. Nature neuroscience, 20(9), 1247-1259. [PubMed:28783139] [PMC] [WorldCat] [DOI]