「核内受容体」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| 67行目: | 67行目: | ||
6塩基RGGTCA(DNAハーフサイト)が、同じ方向あるいは反対方向に反復したDNA配列を認識する。リガンドによって、GRE(前出)などと呼ぶ。モノマーの場合は1つのハーフサイトのみに結合する。 | 6塩基RGGTCA(DNAハーフサイト)が、同じ方向あるいは反対方向に反復したDNA配列を認識する。リガンドによって、GRE(前出)などと呼ぶ。モノマーの場合は1つのハーフサイトのみに結合する。 | ||
== 研究の歴史、背景 == | == 研究の歴史、背景<ref name=ref2><pubmed>16892386</pubmed></ref> == | ||
*1985年 ヒトGRのクローニング | *1985年 ヒトGRのクローニング | ||
*1986年 ヒトERαのクローニング | *1986年 ヒトERαのクローニング | ||
| 96行目: | 95行目: | ||
== リンク(データベース) == | == リンク(データベース) == | ||
*[http://www.nursa.org/index.cfm Nuclear Receptor Signaling Atlas (NURSA): 2002年から米国NIHのサポートで設立されたコンソーシアムで、核内受容体と関連する転写コレギュエーターについてのゲノミクス・プロテオミクス公開データベースとなっている。 | *[http://www.nursa.org/index.cfm Nuclear Receptor Signaling Atlas (NURSA):]<br>2002年から米国NIHのサポートで設立されたコンソーシアムで、核内受容体と関連する転写コレギュエーターについてのゲノミクス・プロテオミクス公開データベースとなっている。 | ||
*[http://en.wikipedia.org/wiki/Nuclear_receptor Nuclear receptor, Wikipedia] | *[http://en.wikipedia.org/wiki/Nuclear_receptor Nuclear receptor, Wikipedia] | ||
2012年8月7日 (火) 10:57時点における版
英語名:nuclear receptor: NR
核内受容体は、ステロイドや甲状腺ホルモン、レチノイド、ビタミンDなどの受容体であり、主に、リガンドが結合すると細胞質から核内へ移行して転写調節因子としてはたらく[1]。リガンドの不明な核内受容体、リガンド結合とは別のしくみで活性が調節される核内受容体もある[2]。ヒトで48の遺伝子にコードされており、代謝、恒常性、分化、成長、発生、老化、生殖などの機能を担う。
リガンド
(表1)[3]
リガンドからNRを分類すると、(1)ホルモンやビタミンをリガンドとする内分泌受容体、(2)配列相同性から発見され、後に生体内でのリガンドが同定されたもの、(3)リガンドの生体での機能が明らかでないもの、(4)リガンドの同定されていないものがある。
カテゴリー(1):内分泌受容体
- ステロイドホルモン [ER, GR, MR, PR, AR](略称は表1を参照)
- ビタミンD(ステロイドから生成)[VDR]
- 昆虫脱皮ホルモン(エクダイソンecdysone)(ステロイド骨格をもつ)
- 甲状腺ホルモン(tyrosineから生成)[TR]
- レチノイド(ビタミンA):レチノイン酸など [RAR]
カテゴリー(2):脂質センサー
- 9-シスレチノイン酸:RXRのリガンド。RXRはVDR, TR, RAR, LXR, PPARなどとヘテロダイマーをつくる。
- oxysterols (コレステロール酸化物):LXRのリガンド。
- 胆汁酸(胆汁中のステロイド誘導体):FXRのリガンド。
- 脂肪酸、細胞内脂質代謝物:PPARs のリガンド。
- 生体異物 (xenobiotics):CAR, PXRのリガンド。薬物代謝物で、肝臓のチトクローム酵素P450によって生成される。
カテゴリー(3)
- androstane(男性ホルモン代謝中間体):CARに結合。
- 脂肪酸:HNF-4a, gに結合。
- リン酸化脂質(ホスファチジルイノシトール類):SF1/LRH1に結合。
- コレステロール、レチノイン酸:RORa,b,gに結合。
カテゴリー(4):オーファン受容体
SHP, DAX1, TLX, PNR, GCNF, TR2,4, NGF1B, Rev-ErbAa,b, COUP-TFI,II,III
構造
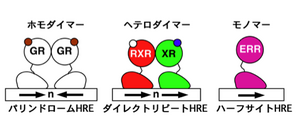
ホモダイマー化したGRなどホルモン受容体は、パリンドローム(回文配列)状に並んだ2つのホルモン応答エレメント(HRE)に結合する。ヘテロダイマー化したRXRと他の核内受容体(XR)は、同方向に並んだ(ダイレクトリピート)2つのHREに結合する。
ERRなどオーファン受容体は、モノマーのまま1つのHRE(ハーフサイト)に結合する。
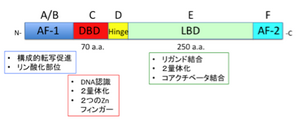
N末端にAF-1領域 (かつてA/Bドメインと呼ばれた)があり、リガンド非依存的に転写活性化作用をもつ。AF-1は、核内受容体間で多様性に富む領域である。中央部にDNA結合領域 (DBD) (C) があり、2つのジンクフィンガーモチーフ(70アミノ酸)から成る。DBDは、受容体間のホモロジーが高い。C末端側にリガンド結合領域 (LBD) (E)(250アミノ酸)をもつ。LBDのC末端 (F領域) にあるαヘリックスをAF-2ヘリックスといい、受容体の活性調節に関係がある。構造の特殊なNRとして、A/B領域を欠くもの(HNF4g)、A/B, C領域を欠くもの(SHP)がある。D領域はヒンジ領域で、DBDとLBDの連結部位である。
核内受容体は通常2量体、ホモダイマー(ステロイド受容体)あるいはヘテロダイマー(RXRとPPARs, LXR, FXRなど)を形成して転写調節を行う(図2)。単量体でDNAに結合するもの(ERR, LRH1, SF1, NGFIB)もある。HNF4sやNGFIBは、リガンド結合とは無関係に活性化されており、これらはオーファン核内受容体と呼ばれる。
作用機序
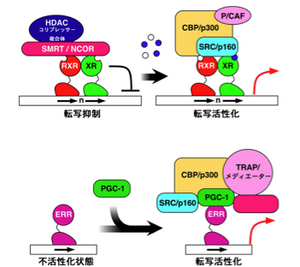
上図:リガンド結合による活性化。リガンドのないとき(左図)、核内受容体はHDAC(ヒストンデアセチラーゼ)やSMRT/NCORなどとリプレッサー複合体を形成しており、転写抑制状態にある。リガンドが結合すると(右図)、コリプレッサーが解離し、HAT(ヒストンアセチルトランスフェラーゼ)やクロマチン再編成複合体から成るコアクチベーターを取り込んで、転写活性化状態になる。
下図:リガンド結合によらない活性化。ERRなどリガンドなしに活性化される核内受容体もある。コアクチベーター(PGC-1など)が結合することで、さらに大きなコアクチベーター複合体を呼び込んで転写活性化状態になる。
核内受容体のリガンドは、輸送蛋白質に結合して血中や体液中を運搬され、標的細胞の中へは単独で入り、細胞質に存在する核内受容体に結合する。例えば、グルココルチコイド受容体 (GR) は、細胞質でシャペロン蛋白質であるhsp90やp23と結合しており、リガンドが結合するとシャペロンから離れて核内に移行し、標的遺伝子の「グルココルチコイド応答エレメント(glucocorticoid response element: GRE)」と呼ばれるDNA配列に結合する[1](後述)。リガンドおよびDNAと結合したNRは、コアクチベーター蛋白質などと結合して、クロマチンの構造を変えて転写を調節する大きな複合体としてはたらく。また、細胞核内でリガンドと結合していないNRはコリプレッサー蛋白質と結合しており、標的遺伝子の転写を抑制している(図3)。NRは、細胞質蛋白質であるSMAD3やJNKとも相互作用する。AF-1領域にはリン酸化部位があり、リン酸化による活性調節を受ける。
核内受容体スーパーファミリー
ヒトで48の遺伝子(表1)、マウスで49の遺伝子にコードされる。分子系統樹から7つのサブファミリー(NR0~6)に分類され、個々の慣用名に対応する正式名がある[4]。サブファミリー0 (NR0)は、DNA結合領域(DBD, C領域)またはリガンド結合領域(LBD, E領域)の一方しか持たないもので、例えばSHPはLBDしか持たずNR0B2と呼ばれる。各サブファミリーはさらにA, B,,,のグループに分けられ、1つのグループはパラログによって構成される。例えば、甲状腺ホルモン受容体 (TR) はサブファミリー1グループA (NR1A)で、TRαはNR1A1, TRβはNR1A2となる。また、例えばNR5A1a (=SF1)とNR5A1b(ELP)とは、同じ遺伝子からスプライシングの違いによってできた異なったアイソフォームである。
他の分類:組織特異的発現パターンや生理的機能から6群(クラスター)に分けられる(表2)[5] [6]。
- クラスターI: ステロイド合成
- クラスターII: 生殖と発生
- クラスターIII: 中枢神経系、概日リズム、基礎代謝機能
- クラスターIV: 胆汁酸と生体異物の代謝
- クラスターVとVI: 脂質代謝とエネルギーの恒常性
認識するDNA配列(ホルモン応答エレメント:hormone response element)
(図2)
6塩基RGGTCA(DNAハーフサイト)が、同じ方向あるいは反対方向に反復したDNA配列を認識する。リガンドによって、GRE(前出)などと呼ぶ。モノマーの場合は1つのハーフサイトのみに結合する。
研究の歴史、背景[2]
- 1985年 ヒトGRのクローニング
- 1986年 ヒトERαのクローニング
- ウイルスガン遺伝子のv-erbAとホルモン受容体とに、相同性のあることがわかった。
- 1986年 TRがv-erbAであることが明らかにされた。
- その後、MR, PR, AR, 脂溶性ビタミンA, Dの受容体のクローニングが相次いだ。配列相同性からオーファン核内受容体が多くクローニングされた。
- PXR(1998年)やPNR (1999年)が、遺伝子情報(ESTデータベース)をもとに発見された最後のNRメンバーとなった。
- 2001年 ヒトゲノムが明らかにされ、核内受容体はヒトでは48遺伝子、マウスでは49遺伝子にコードされることがわかった。
病気、創薬との関連
処方薬上位200のうち34がNRを標的としたものであるというデータがある(2003年)[2] 。Tamoxifenが最初に合成されたNRリガンドで、更年期障害の改善薬として使用されたが、子宮体ガンのリスクを高めることがわかり、現在ではER陽性の乳ガン治療薬として用いられている。その後、NRサブタイプ特異的アゴニスト薬剤の開発が進み、ER beta 特異的なアゴニストは骨粗鬆症に対する効果のみをもち、子宮内膜への増殖作用はないなど、副作用が極力抑えられるようになった。
HNF4a遺伝子変異により、糖尿病の一つ である成人発症型若年性糖尿病(Maturity Onset Diabetes of the Young [MODY1])がおこる[3]。また、HNF4a遺伝子のプロモータ配列の多型性により成人発症2型糖尿病がおこる。SHP遺伝子の変異で肥満症となる。
脳科学との関連
表1のように中枢神経系に存在して機能を担うNRがある。最近、NRは概日リズムを調節することがわかった[6]。概日リズムの形成には、1)転写アクチベーターであるBMAL1とCLOCKのヘテロダイマーが、Period (PER)とCryptochrome (CRY)遺伝子の転写を活性化すること、2)PERとCRYのヘテロダイマーは、逆にBMAL1/CLOCKのリプレッサーとして働くことが重要である。BMAL1/CLOCKはオーファンNRであるREV-ERBsの発現を促し、逆にREV-ERBsはBMAL1の発現を抑制する。
糖質グルココルチコイド(Gc)の血中濃度は、視床下部視交叉上核や副腎のはたらきにより日内変動する。Gcと結合したGRは、GREを介してPER1, PER2遺伝子発現を調節するので、Gcの日内変動もまた概日リズムの強化に関わっている。 甲状腺ホルモンの血中濃度にも日内変動がある。甲状腺を除去するとPER2の周期的な発現が消失する。PPARgのリガンドであるoleoylethanolamide (OEA)も食餌摂取によって昼間に高値を示し、PPARgはBMAL1, REV-ERBaの転写を直接制御している。他にレチノイド受容体もCLOCKと関連がある。
このように、概日リズムと代謝は核内受容体シグナル経路によって連携的にはたらくようになっている。概日時計が、核内受容体の周期的な発現を直接制御して代謝を調節している。核内受容体は、逆に代謝性のシグナルに応答して概日リズムを制御する。
リンク(データベース)
- Nuclear Receptor Signaling Atlas (NURSA):
2002年から米国NIHのサポートで設立されたコンソーシアムで、核内受容体と関連する転写コレギュエーターについてのゲノミクス・プロテオミクス公開データベースとなっている。 - Nuclear receptor, Wikipedia
参考文献
- ↑ 1.0 1.1 Alberts B, Johnson A, Lewis J, Raff M.
Molecular Biology of the Cell, 5th Edition,
pp889-891, Garland Science, New York, 2008. - ↑ 2.0 2.1 2.2 2.3
Moore, J.T., Collins, J.L., & Pearce, K.H. (2006).
The nuclear receptor superfamily and drug discovery. ChemMedChem, 1(5), 504-23. [PubMed:16892386] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2 3.3 3.4
Sonoda, J., Pei, L., & Evans, R.M. (2008).
Nuclear receptors: decoding metabolic disease. FEBS letters, 582(1), 2-9. [PubMed:18023286] [PMC] [WorldCat] [DOI] - ↑
Nuclear Receptors Nomenclature Committee (1999).
A unified nomenclature system for the nuclear receptor superfamily. Cell, 97(2), 161-3. [PubMed:10219237] [WorldCat] [DOI] - ↑
Bookout, A.L., Jeong, Y., Downes, M., Yu, R.T., Evans, R.M., & Mangelsdorf, D.J. (2006).
Anatomical profiling of nuclear receptor expression reveals a hierarchical transcriptional network. Cell, 126(4), 789-99. [PubMed:16923397] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Fan, W., Downes, M., Atkins, A., Yu, R., & Evans, R.M. (2011).
Nuclear receptors and AMPK: resetting metabolism. Cold Spring Harbor symposia on quantitative biology, 76, 17-22. [PubMed:22411605] [PMC] [WorldCat] [DOI]
(執筆者:大内淑代 担当編集委員:大隅典子)