「機能的磁気共鳴画像法」の版間の差分
細 (→fMRIデータの前処理) |
細 (→fMRIデータの前処理) |
||
| 53行目: | 53行目: | ||
またEPI画像法には、生体との干渉により生じる静磁場(B0)の空間的不均一性により画像の歪み・位置ずれが生じる。そこで静磁場不均一性の空間分布を示す画像を別に撮像し、この情報を用いてfMRI画像の歪みを補正することで本来の位置を復元する('''図4''')<ref><pubmed> 14568458</pubmed></ref>。撮像条件にもよるが、この空間的位置ずれは数mm~数10 mm程度発生し、fMRI画像を解剖画像に位置合わせする際に誤差を引き起こす。皮質厚が平均2.6 mm程度しかないことを考えると、正確な機能マッピングを行うためには位置ずれ補正が重要である。 | またEPI画像法には、生体との干渉により生じる静磁場(B0)の空間的不均一性により画像の歪み・位置ずれが生じる。そこで静磁場不均一性の空間分布を示す画像を別に撮像し、この情報を用いてfMRI画像の歪みを補正することで本来の位置を復元する('''図4''')<ref><pubmed> 14568458</pubmed></ref>。撮像条件にもよるが、この空間的位置ずれは数mm~数10 mm程度発生し、fMRI画像を解剖画像に位置合わせする際に誤差を引き起こす。皮質厚が平均2.6 mm程度しかないことを考えると、正確な機能マッピングを行うためには位置ずれ補正が重要である。 | ||
また個人間の脳の形や大きさの違いによらない機能マッピングを行うため、3次元位置合わせ法を適用することで脳形状の標準化が行われる。脳の3次元アトラス空間の国際標準として、モントリオール神経研究所(Montreal Neurological Institute, MNI)が作成した座標系が一般的に使われ、MNI標準I空間内の座標位置(x, y, z)上に脳活動を位置同定(マッピング)することで脳の機能構築の解明が進んできた。これまでの膨大なfMRI研究の成果は、MNI座標での機能マッピングデータベースとして構築され<ref><pubmed> 21706013</pubmed></ref>、web上の視覚ツールも公開されている( | また個人間の脳の形や大きさの違いによらない機能マッピングを行うため、3次元位置合わせ法を適用することで脳形状の標準化が行われる。脳の3次元アトラス空間の国際標準として、モントリオール神経研究所(Montreal Neurological Institute, MNI)が作成した座標系が一般的に使われ、MNI標準I空間内の座標位置(x, y, z)上に脳活動を位置同定(マッピング)することで脳の機能構築の解明が進んできた。これまでの膨大なfMRI研究の成果は、MNI座標での機能マッピングデータベースとして構築され<ref><pubmed> 21706013</pubmed></ref>、web上の視覚ツールも公開されている([https://neurosynth.org neurosynth.org])。 | ||
MNI座標系のような3次元空間での位置合わせ技術も、高次元の非線形法が進歩したことで精度が向上している。しかし複雑な脳回のパターンを保ったまま3次元位置合わせを行うことは原理的に困難である。そこで、大脳皮質が2次元のシートが折れ曲がった構造をしていることに注目した皮質表面解析法が生まれた<ref><pubmed> 9448242</pubmed></ref><ref><pubmed> 22248573</pubmed></ref>。構造MRI画像から皮質を分画化し、その外表面(皮質と脳軟膜境界)と内表面(白質・皮質境界)を抽出し「皮質表面」を得る。そしてこの皮質表面上で脳回のパターン(曲率や深度)を位置合わせする手法(surface registration)が提案され<ref><pubmed> 10619420</pubmed></ref>、個人間の皮質機能構築を2次元座標上で標準化できるようになった。また皮質表面の2次元座標系と、皮質下構造の3次元MNI座標系を組み合わせた新しい座標(灰白質座標)と専用フォーマットCIFTIも設計された。正確な皮質表面の抽出には、高解像度・高画質の脳構造画像が必要であり、得られた表面境界とfMRI画像との正確な位置合わせを行うことで<ref><pubmed> 19573611</pubmed></ref>、fMRI画像を灰白質画像に正確に重ね合わせ、個人間解析を高い位置精度で行うことが可能になってきている。さらに、脳回情報だけでなく機能や構造に関わる値(RSNやミエリンマップ等)を複数組み合わせた、さらに高い精度の表面位置合わせ法も開発され、応用も進んでいる<ref><pubmed> 29100940</pubmed></ref>。 | MNI座標系のような3次元空間での位置合わせ技術も、高次元の非線形法が進歩したことで精度が向上している。しかし複雑な脳回のパターンを保ったまま3次元位置合わせを行うことは原理的に困難である。そこで、大脳皮質が2次元のシートが折れ曲がった構造をしていることに注目した皮質表面解析法が生まれた<ref><pubmed> 9448242</pubmed></ref><ref><pubmed> 22248573</pubmed></ref>。構造MRI画像から皮質を分画化し、その外表面(皮質と脳軟膜境界)と内表面(白質・皮質境界)を抽出し「皮質表面」を得る。そしてこの皮質表面上で脳回のパターン(曲率や深度)を位置合わせする手法(surface registration)が提案され<ref><pubmed> 10619420</pubmed></ref>、個人間の皮質機能構築を2次元座標上で標準化できるようになった。また皮質表面の2次元座標系と、皮質下構造の3次元MNI座標系を組み合わせた新しい座標(灰白質座標)と専用フォーマットCIFTIも設計された。正確な皮質表面の抽出には、高解像度・高画質の脳構造画像が必要であり、得られた表面境界とfMRI画像との正確な位置合わせを行うことで<ref><pubmed> 19573611</pubmed></ref>、fMRI画像を灰白質画像に正確に重ね合わせ、個人間解析を高い位置精度で行うことが可能になってきている。さらに、脳回情報だけでなく機能や構造に関わる値(RSNやミエリンマップ等)を複数組み合わせた、さらに高い精度の表面位置合わせ法も開発され、応用も進んでいる<ref><pubmed> 29100940</pubmed></ref>。 | ||
2020年8月22日 (土) 09:46時点における版
林 拓也、麻生 俊彦
理化学研究所生命機能科学研究センター脳コネクトミクスイメージング研究チーム
藤本晃司
京都大学医学部医学研究科脳機能総合研究センター
花川 隆
京都大学医学部医学研究科脳統合イメージング分野
DOI:10.14931/bsd.9325 原稿受付日:2020年8月21日 原稿完成日:2020年X月XX日
担当編集委員:定藤 規弘(自然科学研究機構生理学研究所 大脳皮質機能研究系)
英:functional magnetic resonance imaging 英略称:fMRI 独:Funktionelle Magnetresonanztomographie 仏:Imagerie par résonance magnétique fonctionnelle
1段落程度の抄録をお願いいたします。
はじめに
機能的磁気共鳴画像とは、磁気共鳴画像 (magnetic resonance imaging; MRI)を用いて生体の脳や脊髄を一定時間連続的に撮像し、脳活動(神経活動とシナプス活動等の総和)と相関するMRI信号の変動を非侵襲的に計測する技術である。
1990年代の初頭に開発されるやいなや、当時ヒト脳機能イメージング研究手法の主流であったポジトロン断層像(PET)による血流・代謝測定を置き換えた。現在では、脳機能イメージング研究の代名詞として、健常脳の機能分離や機能連関の理解、あるいは精神・神経疾患の病態生理の解明のため、欠かすことのできないツールとなっている。ただしfMRIは、PETと同様、脳活動の本態である神経細胞の電気化学的活動そのものを測定しているのではなく、脳活動の代用マーカー(surrogate marker)としての局所酸素代謝・血流動態を画像化していることには留意が必要である。また、脳活動に由来するfMRI信号の変動は、脳活動以外の要因による信号変動と比較して必ずしも大きくないため、興味のある脳活動を抽出するために適切な画像・信号処理を行うことも重要である。本項目では、脳機能を解明するツールとしてのfMRIの原理、解析法とそれらを応用した脳科学研究の潮流を概説する。
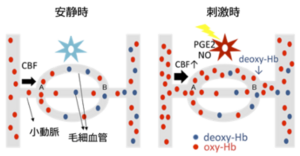
脳血流(CBF)は動脈〜小動脈(arteriole)から流入し、動脈血中では赤血球のヘモグロビンは酸素化(oxy-Hb)されている。刺激がない安静時(左)であっても、酸素は脳の基礎代謝要求により消費される。酸素は毛細血管で脳組織に供給され、酸素化ヘモグロビン(oxy-Hb)は、常磁性体である還元ヘモグロビン(deoxy-Hb)に変わる。外的刺激などによりシナプス入力と神経活動が増加すると(右)、局所の酸素・エネルギー代謝要求は安静時と比較して増加する。酸素消費により還元ヘモグロビン(常磁性体)が増加するはずだから、局所磁場が乱れてT2*が短縮するように思われる(陰性BOLD信号)。しかし、神経血管単位は基礎代謝要求の増加を検知して動脈血の流入を要求量以上に増加させるらしい。この過程にはプロスタグランジン(PG)や一酸化窒素(NO)が関わっているとされる。これらの影響の総和として、脳活動が増加する部分ではdeoxy-Hbが相対的に薄まって局所磁場が安定し、T2*延長が観察される。多くのfMRI法ではこのT2*の延長を陽性BOLD信号として計測している。
原理
BOLD信号の発見
1990年に小川誠二らはグラジエントエコー(GE)法MRIでマウス生体脳内の血管を撮像し、血管近傍のMRI信号が酸素飽和度に依存して変化することを発見した[1]。小川はこの観察について、酸素飽和度が減少する部位では、赤血球内の還元ヘモグロビン(deoxy-Hb)が増加し、近傍の脳実質の信号が変化するためであると考えた。なぜなら還元ヘモグロビンは不対電子による常磁性(paramagnetic)を示し、還元ヘモグロビンの増加は局所磁場を乱し横磁化の減衰を増強するからである。小川はこの原理をblood oxygen-level-dependent (BOLD) コントラストと名付けた。
さらに小川らは、ヒト健常ボランティアに視覚刺激を与えると一次視覚野のMRI信号が増加することを示した。このMRI信号の増加は、後述するように脳活動の増加に続く局所脳血流の過剰流入が酸素飽和度を上昇させ、還元ヘモグロビン濃度が薄まるために生じると説明されている [2]。なお、還元ヘモグロビンによる横磁化減衰の程度は、静磁場強度の2乗に比例して増加する[3]ため、高磁場MRIに測定上の優位性がある。
神経血管連関
脳の神経・シナプス活動に応答する局所血流変化は、神経細胞、微小血管(細動脈~毛細血管)の内皮細胞や周皮細胞、さらに星状膠細胞などからなる神経血管単位(neurovascular unit)により制御され、脳活動に引き続いて局所に過剰な酸素供給(機能的充血、functional hyperemia)をもたらす。つまり、BOLD信号を脳活動の代用マーカーとするfMRIは、神経活動の増加に続いて機能的充血が生じる仕組み(神経血管連関、neurovascular coupling)に依存している(図1)。
2001年のLogothetisらによるサルを対象としたfMRIと神経活動の同時計測は、刺激や課題に伴う脳活動の増加とBOLD信号の関係性の理解に大きく貢献した[4]。ある視覚刺激条件において、マルチユニット神経活動(複数の神経細胞の出力)は一過性にのみ増加を示したが、BOLD信号と局所電場電位(local field potential, LFP)は一過性の増加に引き続く持続性の増加を示した。すなわちBOLD信号変化は、マルチユニット神経活動よりもLFPと良く相関する。LFPはシナプス活動、すなわち神経細胞への情報入力を反映するから、BOLD信号変化は皮質からの出力量よりも皮質への入力量との関係が強いことになる。最近では、超高磁場・高解像度のfMRIにより、皮質層ごとの情報処理を反映するBOLD信号変化の計測技術が精力的に開発されている[5]。
脳活動とBOLD信号の相関関係は、外的刺激や課題の無い安静時にも観察される。そもそも課題遂行では脳の酸素代謝は数%しか増えず、脳のエネルギーは課題の無い(task free)安静時(resting state)の活動に大半が消費されている。これは脳が安静時にも組織的かつ活発な自発活動を示すことによる。1990年代後半のPET研究により、内側前頭前野、後部帯状回や両側外側頭頂葉などは、課題遂行時と比べて安静時にむしろ脳血流が増加することが知られていた[6]。安静時に著明な自発的神経活動を示すこれらの領域は、Raichleによりデフォルトモードネットワーク(default mode network, DMN)と名づけられ、基底状態の脳の統合性に関わる内因性機構として提唱された。一方で、Biswalは1995年に安静状態のfMRIを解析し、両側運動感覚野の信号が主に0.1Hz以下の低い周波数(f)帯域において1/fのパターンで同期していることを見出していた[7]。このようなfMRI信号同期は、安静状態神経ネットワーク(resting-state network, RSN)が有する機能結合(functional connectivity, FC)を反映すると考えられた。さらに興味深いことに、安静時fMRIにより、Raichleの提唱したDMN内の脳領域間には強い機能結合が存在することがわかった[8]。このように2つの独立した研究の潮流が融合したことで、安静時fMRIを用いて局所の自発脳活動と領域間の機能結合状態を評価できる可能性に大きな注目が集まった。その後、安静時fMRIの信号同期性がサル脳における神経連絡性に対応していることも判明した[1]。
現在、安静時fMRIの同期現象は、神経連絡を持つ遠隔領域間で同期して発生する自発性のシナプス・神経活動(及びこれらに伴うBOLD効果)に基づいていると考えられている。領域間の同期の詳細を知るための解析手法の改善、覚醒時の基底状態としての意識との関連、精神疾患や認知症などの病態との関連、神経連絡性との対応などについて研究が進んでいる。
血流動態応答
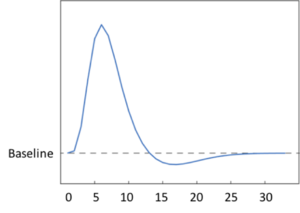
短時間の感覚刺激後に生じるBOLD信号を経時的に計測すると、信号強度は刺激呈示から約2秒後に基準線(baseline)を超え、約5秒後に最大値を示す。この応答を血流動態応答関数(hemodynamic response function, HRF)と呼ぶ(図2)。初期に1-2秒間の負のBOLD反応(initial dip)が観測されることもある。神経活動増加の後、信号強度は基準線より低下し、しばらくその状態が続く(undershoot)。刺激直後の負のBOLD反応(initial dip)に関しては、血管拡張が最も早い皮質最深部の層では見られず、最も遅い最表層の皮質で見られることから、血流動態応答に先行して生じる酸素消費と還元ヘモグロビンの増加を反映する、という仮説が支持されている[9]。Duongらはネコの視覚野を対象とした実験で、刺激直後に観察される陰性BOLD信号(initial dip)のほうが、陽性BOLD反応よりも空間選択性が高いことを示している[10]。なお、血流動態応答は、1)前述の神経血管連関に基づく現象であり、2)後述のようにBOLD信号の変化から課題遂行中の神経活動を推定する逆問題を解く場合に必要な応答関数であることから、その生物学的機構や応答関数の妥当性などについてさらなる解明が望まれる。
fMRIを用いた脳機能解明法
fMRI撮像・実験デザイン
fMRIの撮影には、BOLD信号のコントラスト源であるT2*減衰を鋭敏にとらえ、かつ時間分解能も高いエコープラナー画像(EPI)法を用いることがほとんどである。fMRIの性能を上げるためには、空間と時間両次元の分解能を高めることが理想的である。従って、撮像する際の時・空間解像度としては、MRI装置の性能を最大限に活かした高解像度が望ましいが、一方で解像度を上げすぎると信号雑音(S/N)比が低下することには注意が必要である。空間解像度と時間解像度もトレードオフの関係にあるが、マルチバンドEPIなどの高速撮像法の開発により長足の進歩があった。マルチバンドEPIは、従来のEPIに比べて空間解像度を1.2-1.5倍程度あるいは時間分解能を6-8倍近く向上させることができ、脳科学研究に汎用され始めている。
fMRIは撮像中の研究参加者(あるいは動物)の状態により2種類に大別される。安静状態の脳活動を計測する安静時(resting-stateまたはtask-free)fMRIと、研究参加者(あるいは動物)が指示された課題を遂行中に計測する課題(task) fMRIである。安静時fMRIでは、閉眼条件あるいは眼前の十字を固視した開眼条件で、「何も考えない」ように指示して5-10分程度のfMRI撮像を行うことが多い。課題の練習を行う必要がない楽な検査法であり、健常者のみならず患者や小児の脳研究にも使いやすく、上述のDMNの評価にも応用可能である。一方、課題fMRIでは、数秒から数十秒単位の課題遂行を間歇的に行うよう指示をして実行してもらう。通常、コンピュータを用いて感覚刺激を提示したり応答を記録したりする。そのため、課題fMRIを施行するためには、MRI撮像に影響を与えない感覚刺激・応答記録用のシステムを整備する必要がある。課題fMRIでは、課題遂行の時間情報と前述のHRFを畳み込んだBOLD信号変化モデルを作成し統計解析を行う(後述「fMRIの統計解析」参照)。課題遂行時間がやや長い場合(10-20秒以上)の場合は前述のDMNの周期や呼吸変動ノイズと周波数が近くなり、数秒以内の課題遂行の場合には体動や心拍の周波数帯と近くなる。そのためこうした関心の無い実験要因による解析精度の劣化の可能性を考慮した課題デザインを設計することが望ましいが、課題デザインについての詳細は本項目の範囲を超えるため別項目に譲りたい。
fMRIデータの前処理
3テスラMRI装置でのfMRIにおいて、神経活動による信号変化は全信号変動の約7パーセント程度とされる。生体や装置の熱ノイズが約50%を占め、残りはMRI装置の不完全性による信号ドリフトや、非撮像体の動き等に由来するアーチファクトとされる[11]。頭部の動きは画像位置合わせにより補正(motion correction/realignment)することが通常であるが、この処理では補正しきれないMRI信号変動が残存する。加えて心拍や呼吸などの生理的要因による信号変動も存在するため、これら非神経性の信号を可能な限り分離・除去することが重要である(図3)。古典的には、頭部動き補正の後、安静時・課題fMRIともに一定の周波数帯域の信号を除去するフィルター(high pass filterやlow pass filter)をかけることでノイズ除去が行われてきた。しかしこの方法では、関心ある神経活動信号も除去してしまうため、独立成分分析によるノイズ同定・軽減への期待が高い[12](後述「fMRIの統計解析」参照)。
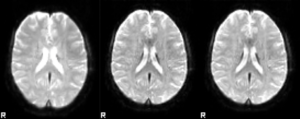
左. 前処理していない元fMRI画像(TR=1.5秒で5分間撮影したデータのうち15秒間のデータのみ提示)
中. 剛体変換による数学的な動き補正(motion correction/realignment)のみを行ったfMRI画像。動きはある程度補正されているものの、撮像中の頭部の動きは大きなMRI信号の変動を引き起こすためにノイズ軽減処理はまだ十分ではないことが見て取れる。
右. 動き補正に続き、独立成分分析によるノイズ軽減処理と脳外組織のマスク除去を行った後の画像。頭部の動きによる画像アーチファクトは目立たない。いずれも脳標準空間への位置合わせを行い、38倍速で表示している。
また上述のようにfMRIデータにおける熱ノイズ量はBOLD信号変動に対して非常に大きい。そのため統計解析の前に、ガウスフィルタを適応して信号ノイズ比を向上させる操作(空間平滑化 spatial smoothing)が行われる。しかしこの方法は位置情報の正確さを低下させるため、位置情報を犠牲にしないノイズ軽減法(例:ウィシャートフィルター)の開発も現在進んでいる([13]の図S7を参照)。
またEPI画像法には、生体との干渉により生じる静磁場(B0)の空間的不均一性により画像の歪み・位置ずれが生じる。そこで静磁場不均一性の空間分布を示す画像を別に撮像し、この情報を用いてfMRI画像の歪みを補正することで本来の位置を復元する(図4)[14]。撮像条件にもよるが、この空間的位置ずれは数mm~数10 mm程度発生し、fMRI画像を解剖画像に位置合わせする際に誤差を引き起こす。皮質厚が平均2.6 mm程度しかないことを考えると、正確な機能マッピングを行うためには位置ずれ補正が重要である。
また個人間の脳の形や大きさの違いによらない機能マッピングを行うため、3次元位置合わせ法を適用することで脳形状の標準化が行われる。脳の3次元アトラス空間の国際標準として、モントリオール神経研究所(Montreal Neurological Institute, MNI)が作成した座標系が一般的に使われ、MNI標準I空間内の座標位置(x, y, z)上に脳活動を位置同定(マッピング)することで脳の機能構築の解明が進んできた。これまでの膨大なfMRI研究の成果は、MNI座標での機能マッピングデータベースとして構築され[15]、web上の視覚ツールも公開されている(neurosynth.org)。
MNI座標系のような3次元空間での位置合わせ技術も、高次元の非線形法が進歩したことで精度が向上している。しかし複雑な脳回のパターンを保ったまま3次元位置合わせを行うことは原理的に困難である。そこで、大脳皮質が2次元のシートが折れ曲がった構造をしていることに注目した皮質表面解析法が生まれた[16][17]。構造MRI画像から皮質を分画化し、その外表面(皮質と脳軟膜境界)と内表面(白質・皮質境界)を抽出し「皮質表面」を得る。そしてこの皮質表面上で脳回のパターン(曲率や深度)を位置合わせする手法(surface registration)が提案され[18]、個人間の皮質機能構築を2次元座標上で標準化できるようになった。また皮質表面の2次元座標系と、皮質下構造の3次元MNI座標系を組み合わせた新しい座標(灰白質座標)と専用フォーマットCIFTIも設計された。正確な皮質表面の抽出には、高解像度・高画質の脳構造画像が必要であり、得られた表面境界とfMRI画像との正確な位置合わせを行うことで[19]、fMRI画像を灰白質画像に正確に重ね合わせ、個人間解析を高い位置精度で行うことが可能になってきている。さらに、脳回情報だけでなく機能や構造に関わる値(RSNやミエリンマップ等)を複数組み合わせた、さらに高い精度の表面位置合わせ法も開発され、応用も進んでいる[20]。

ヒト脳コネクトームプロジェクト(HCP)の課題fMRIデータを使用。
指示に反応して体の各部位(右足・左足・右手・左手・舌)を動かす課題fMRIの結果。A. 運動時の脳活動をモデル化後、血流動態反応(HRF)を畳み込んでBOLD信号変化モデル(緑線)を作成する。データへのあてはめ(GLM)を行い、モデルとデータが有意に相関した部位(体素)の信号変化(赤線)を示している。B. 運動課題のBOLD信号変化モデルを30例の被験者のデータにあてはめた際のt検定の結果(t値)を、大脳皮質表面に投影して表示。左側は平均の皮質表面、右側はその皮質表面を膨らませて表示することで、脳溝内の統計値を見やすくしている。運動野、補足運動野、2次体性感覚野の神経活動が示唆される。
fMRIの統計解析
単変量解析
univariate analysis 課題fMRIの解析には、課題に対応する脳活動とそれに応答するBOLD信号変化を合わせてモデル化し、この理論的な信号変化モデルと実際に測定したBOLD信号との適合度を体積画素/体素(voxel)ごとに計算し、課題に関連する脳活動を反映する統計画像を得る方法が広く用いられている。信号変化モデルは、複数の説明変数(課題遂行と相関する「脳活動」、頭部の動きなど脳活動以外の要因など)や脳活動に対するBOLD反応の時間的遅れやバラつき(血流動態関数、HRF)を考慮して構築する。多重回帰を含む一般線形モデル(general linear model, GLM)により、信号変化モデルとデータとの適合程度を調べ、各説明変数の重み付け係数値画像や誤差値画像を算出する(第1段階解析)(図5A)。
臨床検査としての課題fMRI(術前の言語機能評価など)のように、個人の結果に興味がある場合は、係数値画像を誤差値画像で割って得られるt値画像などを用いた個人内要因の統計検定を行う。例えば、課題Aと課題B遂行時の脳活動の差を見るためには、係数の引き算に相当する重みづけ操作を行い(”コントラストを立てる”)、得られたt値画像を用いて統計検定を行う。
医学研究や神経科学研究では、測定対象とした複数の個人を母集団からランダムに抽出したサンプルと考え、母集団についての推定を行いたいことが一般的である。その際は、研究者が関心のある実験要因に対する重みづけ係数画像について、個人間でのバラつきの違い「変量効果(random effect)」を考慮した統計検定(一般化線形混合モデル[generalized linear mixed model, GLMM])を行い、課題と相関する脳活動とその群間差を示す脳部位を検出する(2段階解析)(図5B)。
ここで問題となるのが統計学的有意性の閾値の決定の方法である。画像統計では数十~百万単位の体素を同時に扱うため、適切な多重比較補正を行わないと、脳科学的に意味のない信号変化を脳活動として検出されてしまう危険(偽陽性)を伴う。そのため、古典的には脳の中に脳活動領域としての塊(cluster)を想定し、そのような塊が偶然形成される確率から閾値を設定する理論が採用されている[21]。しかし、この統計閾値法の妥当性については議論が続いており[22]、課題fMRIの再現性が低い主要因になっているとの見方がある[23]。再現性は画像の平滑化処理や分画化などの前処置法にも依存するため、偽陽性率を適切に制御できる統計法の開発が行われている(例: 順列統計Permutation testなど[24]。
多変量解析
multivariate/multivoxel pattern analysis, MVPA
一般線形モデル(GLM)では体素ごとの統計検定を行うのに対し、MVPAでは複数の体素の信号値の関係性(パターン)が課題の差や、疾患と健常の差を表現すると考えた多変量解析を行う。MVPAは「課題に対する脳活動や疾患に特有の脳活動の時空間的なパターンを判別」するアプローチとも言える。初期にはGLMによる重み付け画像同士の相関性がパターンの類似性指標として使われた[25]。最近ではサポートベクターマシン(support vector machine, SVM)などの機械学習技術が判別法に採用されている。脳活動の空間的なパターンを情報として、与えられた課題(例えば視覚刺激)の逆問題を推定または解読(decoding)する研究も行われている[26]。また安静時fMRIから得られた多変量の機能結合パターンを、機械学習技術を応用して判別し、精神神経疾患の診断補助を目指す先駆的な医療技術の開発も進んでいる[27]。
機能結合解析
functional connectivity analysis
システムとしての脳の仕組みを探るにあたり、機能局在を追求することだけでは限界がある。そこで脳部位間の繋がりが注目され、2005年には脳の網羅的な結線図を意味するコネクトーム(connectome)という概念が提唱された[28]。脳コネクトームには様々な空間スケールがありうる。全脳を観察できるfMRIでは、脳領域間機能結合の正方行列を計算することで、全脳レベル(macroscale)の機能的コネクトーム解析が可能である。fMRIでは各脳領域間の連絡の強さの指標を機能結合(functional connetivity, FC)と呼ぶ。FCの産出法としては領域間の時系列信号のピアソン相関係数が良く使われる[7]。ある特定の脳領域を関心領域(シード)として設定し、相手方の関心領域(または体素)を、全脳に渡って隙間なく設定した相関解析を行えば、特定のシードに対する脳全体の機能結合マップが得られる。例えば、安静時fMRI解析において後部帯状回に関心領域を設定すれば、PETで見られたDMNがRSNとして抽出できる[29][30]。さらにシードを全脳の関心領域すべてに設定することで、多対多の網羅的な機能結合、すなわち機能的コネクトームが算出できる。このような機能的コネクトーム解析を行う際には、通常のピアソンの相関係数ではなく偏相関分析(partial correlation)を用いることで、2領域間に固有性の高いFCを評価できることが示唆されている[31]。また機能的コネクトームを用いた新たな機能結合性モデルの提唱や、より高次な解析への展開(グラフ理論、独立成分分析、機械学習など)、他の指標や測定(脳波や臨床兆候など)との関連付け、疾患バイオマーカーの探索等の様々な研究も行われている。一方でヒトの脳コネクトーム研究単独では結果の妥当性の検証が難しいため、動物脳での検証も重要であり、例えば神経連絡トレーサーと機能的結合の比較は重要な課題である。
独立成分分析
independent component analysis
ICAはデータ駆動型の解析でありブラインド信号分離の技術として多くの分野で活用されている。主成分分析(principle component analysis, PCA)と比べ、データマイニングの手法として柔軟であると考えられている。主成分分析では無相関、つまりベクトルとして直交する成分のセットが解析的に一意に定まる。これに対しICAでは、互いに他の情報を持たないという特性(独立性)が大きくなるよう成分を分離する。無相関であっても独立ではないことがあるから、独立性は相関性よりも柔軟な条件である。確率変数としてデータの空間的な分布を採るか、経時変化を採るかによって、空間的ICAと時間的ICAが可能である。fMRIデータが空間的に高次元であることから、初期のfMRIでの活用は空間的ICAにより数十個の独立成分を求めるものであった。神経解剖学的に解釈可能な皮質ネットワークを得ることができDMNの同定にも使用される(図6)。ICAは従前から脳波など電気生理データのノイズ除去に利用されてきたが、機械学習判別によって個人レベルのfMR元データから構造ノイズ成分を除去する前処理過程にもICAが利用されている(fMRIデータの前処理の項参照)。
また、空間的標準化を行った後、複数の個人の安静時fMRIに空間的ICAを適応することで、個人間に共通のネットワークを同定し(group ICA)、グループとしての安静状態神経ネットワーク(RSN)を検出することも可能である。さらにこのグループRSNの各個人のデータへのあてはめ(GLM)を、時間と空間について2回行うことで、個人毎のRSNが得られる。この個人毎のRSNを用いた集団解析が可能であり[32]、病態解明への応用研究も進んでいる。最近は時間分解能の高いfMRIデータが取得されるようになってきたことで、時間的ICAの応用も進み、空間的に重なり合う成分や神経連絡性とも合致する成分の抽出[33]や、脳全体の広範なノイズの検出に有用であることが分かってきている。ただしICAの結果の解釈には数学的な仮定(例えば次元数や線形性等)と脳のシステム・神経-血管連関・画像ノイズの妥当性や検証に留意する必要がある[34]。
fMRIによる最近の脳科学研究
1990年後半からfMRI研究は飛躍的に世界中に広まり多くの成果をあげてきた。その間に基礎となるMRI技術も大きく進展し、質の高いデータが得られるようになった。初期の研究の興味は脳の機能局在・分離の同定にあったが、2010年代以後はネットワークとしての脳の解明に興味がシフトした[36]。一つの研究が扱うfMRIデータの数も、数10人から数百人、数万人の規模へと拡大し、種としてのヒトの脳機能構築やその複雑性の理解、個体差の理解へと興味が広がった。2010年から2016年まで、米国NIHの支援により大規模研究プロジェクトHuman Connectome Project(HCP)が行われた。このプロジェクトでは、ワシントン大学セントルイス医学校およびオックスフォード大学が中心となり約1200名の健康な若年成人被験者を対象として、安静時fMRI、標準的な課題を用いた課題fMRI、構造MRI(T1・T2強調画像)、拡散強調画像を撮像し、脳内の機能的結合と構造的結合について統合的に解析を進めた。このプロジェクトは(1)質が高い画像データを大量に取得する、(2)空間分解能を犠牲にすることなくデータを処理する手法を開発する、(3)FACT、すなわち皮質機能(Function)・連絡性(Connectivity)、構造(Architecture)、位置(Topography)の情報を統合した脳の領域分割(parcellation)を行う、(4)取得した生データ、解析データ、データ解析に必要なプログラム・コードを無料で公開する、といった多くの野心的な目標を達成し、現在も世界のMRI脳科学研究に大きな影響を与え続けている。特にFACT法は片側半球を180領域に分割することに成功し[35]、100年以上続くヒト脳の機能分画の歴史の中で初めて非侵襲手法により生きた個人の脳地図を作成することに成功した(図7)。こうした技術をさらに発展・拡張させ、脳の個体差の検出や、疾患の診断技術の開発が期待されており、更に規模の大きな研究プロジェクトが各国で推進されている(UKバイオバンク、ABCDなど)。また動物モデルへの技術展開も進めることで動物種を超えた脳のシステムの解明や、適切な動物疾患モデルの開発や評価法検証も期待される。
関連項目
参考文献
- ↑ 1.0 1.1
Ogawa, S., Lee, T.M., Nayak, A.S., & Glynn, P. (1990).
Oxygenation-sensitive contrast in magnetic resonance image of rodent brain at high magnetic fields. Magnetic resonance in medicine, 14(1), 68-78. [PubMed:2161986] [WorldCat] [DOI] - ↑
Ogawa, S., Tank, D.W., Menon, R., Ellermann, J.M., Kim, S.G., Merkle, H., & Ugurbil, K. (1992).
Intrinsic signal changes accompanying sensory stimulation: functional brain mapping with magnetic resonance imaging. Proceedings of the National Academy of Sciences of the United States of America, 89(13), 5951-5. [PubMed:1631079] [PMC] [WorldCat] [DOI] - ↑
Thulborn, K.R., Waterton, J.C., Matthews, P.M., & Radda, G.K. (1982).
Oxygenation dependence of the transverse relaxation time of water protons in whole blood at high field. Biochimica et biophysica acta, 714(2), 265-70. [PubMed:6275909] [WorldCat] [DOI] - ↑
Logothetis, N.K., Pauls, J., Augath, M., Trinath, T., & Oeltermann, A. (2001).
Neurophysiological investigation of the basis of the fMRI signal. Nature, 412(6843), 150-7. [PubMed:11449264] [WorldCat] [DOI] - ↑
Kok, P., Bains, L.J., van Mourik, T., Norris, D.G., & de Lange, F.P. (2016).
Selective Activation of the Deep Layers of the Human Primary Visual Cortex by Top-Down Feedback. Current biology : CB, 26(3), 371-6. [PubMed:26832438] [WorldCat] [DOI] - ↑
Raichle, M.E. (2015).
The brain's default mode network. Annual review of neuroscience, 38, 433-47. [PubMed:25938726] [WorldCat] [DOI] - ↑ 7.0 7.1
Biswal, B., Yetkin, F.Z., Haughton, V.M., & Hyde, J.S. (1995).
Functional connectivity in the motor cortex of resting human brain using echo-planar MRI. Magnetic resonance in medicine, 34(4), 537-41. [PubMed:8524021] [WorldCat] [DOI] - ↑
Greicius, M.D., Krasnow, B., Reiss, A.L., & Menon, V. (2003).
Functional connectivity in the resting brain: a network analysis of the default mode hypothesis. Proceedings of the National Academy of Sciences of the United States of America, 100(1), 253-8. [PubMed:12506194] [PMC] [WorldCat] [DOI] - ↑
Tian, P., Teng, I.C., May, L.D., Kurz, R., Lu, K., Scadeng, M., ..., & Devor, A. (2010).
Cortical depth-specific microvascular dilation underlies laminar differences in blood oxygenation level-dependent functional MRI signal. Proceedings of the National Academy of Sciences of the United States of America, 107(34), 15246-51. [PubMed:20696904] [PMC] [WorldCat] [DOI] - ↑
Duong, T.Q., Kim, D.S., Uğurbil, K., & Kim, S.G. (2001).
Localized cerebral blood flow response at submillimeter columnar resolution. Proceedings of the National Academy of Sciences of the United States of America, 98(19), 10904-9. [PubMed:11526212] [PMC] [WorldCat] [DOI] - ↑
Marcus, D.S., Harms, M.P., Snyder, A.Z., Jenkinson, M., Wilson, J.A., Glasser, M.F., ..., & WU-Minn HCP Consortium (2013).
Human Connectome Project informatics: quality control, database services, and data visualization. NeuroImage, 80, 202-19. [PubMed:23707591] [PMC] [WorldCat] [DOI] - ↑
Salimi-Khorshidi, G., Douaud, G., Beckmann, C.F., Glasser, M.F., Griffanti, L., & Smith, S.M. (2014).
Automatic denoising of functional MRI data: combining independent component analysis and hierarchical fusion of classifiers. NeuroImage, 90, 449-68. [PubMed:24389422] [PMC] [WorldCat] [DOI] - ↑
Glasser, M.F., Smith, S.M., Marcus, D.S., Andersson, J.L., Auerbach, E.J., Behrens, T.E., ..., & Van Essen, D.C. (2016).
The Human Connectome Project's neuroimaging approach. Nature neuroscience, 19(9), 1175-87. [PubMed:27571196] [PMC] [WorldCat] [DOI] - ↑
Andersson, J.L., Skare, S., & Ashburner, J. (2003).
How to correct susceptibility distortions in spin-echo echo-planar images: application to diffusion tensor imaging. NeuroImage, 20(2), 870-88. [PubMed:14568458] [WorldCat] [DOI] - ↑
Yarkoni, T., Poldrack, R.A., Nichols, T.E., Van Essen, D.C., & Wager, T.D. (2011).
Large-scale automated synthesis of human functional neuroimaging data. Nature methods, 8(8), 665-70. [PubMed:21706013] [PMC] [WorldCat] [DOI] - ↑
Van Essen, D.C., Drury, H.A., Joshi, S., & Miller, M.I. (1998).
Functional and structural mapping of human cerebral cortex: solutions are in the surfaces. Proceedings of the National Academy of Sciences of the United States of America, 95(3), 788-95. [PubMed:9448242] [PMC] [WorldCat] [DOI] - ↑
Fischl, B. (2012).
FreeSurfer. NeuroImage, 62(2), 774-81. [PubMed:22248573] [PMC] [WorldCat] [DOI] - ↑
Fischl, B., Sereno, M.I., Tootell, R.B., & Dale, A.M. (1999).
High-resolution intersubject averaging and a coordinate system for the cortical surface. Human brain mapping, 8(4), 272-84. [PubMed:10619420] [PMC] [WorldCat] [DOI] - ↑
Greve, D.N., & Fischl, B. (2009).
Accurate and robust brain image alignment using boundary-based registration. NeuroImage, 48(1), 63-72. [PubMed:19573611] [PMC] [WorldCat] [DOI] - ↑
Robinson, E.C., Garcia, K., Glasser, M.F., Chen, Z., Coalson, T.S., Makropoulos, A., ..., & Rueckert, D. (2018).
Multimodal surface matching with higher-order smoothness constraints. NeuroImage, 167, 453-465. [PubMed:29100940] [PMC] [WorldCat] [DOI] - ↑
Worsley, K.J., Cao, J., Paus, T., Petrides, M., & Evans, A.C. (1998).
Applications of random field theory to functional connectivity. Human brain mapping, 6(5-6), 364-7. [PubMed:9788073] [PMC] [WorldCat] - ↑
Eklund, A., Nichols, T.E., & Knutsson, H. (2016).
Cluster failure: Why fMRI inferences for spatial extent have inflated false-positive rates. Proceedings of the National Academy of Sciences of the United States of America, 113(28), 7900-5. [PubMed:27357684] [PMC] [WorldCat] [DOI] - ↑
Botvinik-Nezer, R., Holzmeister, F., Camerer, C.F., Dreber, A., Huber, J., Johannesson, M., ..., & Schonberg, T. (2020).
Variability in the analysis of a single neuroimaging dataset by many teams. Nature, 582(7810), 84-88. [PubMed:32483374] [WorldCat] [DOI] - ↑
Winkler, A.M., Ridgway, G.R., Webster, M.A., Smith, S.M., & Nichols, T.E. (2014).
Permutation inference for the general linear model. NeuroImage, 92, 381-97. [PubMed:24530839] [PMC] [WorldCat] [DOI] - ↑
Haxby, J.V., Gobbini, M.I., Furey, M.L., Ishai, A., Schouten, J.L., & Pietrini, P. (2001).
Distributed and overlapping representations of faces and objects in ventral temporal cortex. Science (New York, N.Y.), 293(5539), 2425-30. [PubMed:11577229] [WorldCat] [DOI] - ↑
Miyawaki, Y., Uchida, H., Yamashita, O., Sato, M.A., Morito, Y., Tanabe, H.C., ..., & Kamitani, Y. (2008).
Visual image reconstruction from human brain activity using a combination of multiscale local image decoders. Neuron, 60(5), 915-29. [PubMed:19081384] [WorldCat] [DOI] - ↑
Yahata, N., Morimoto, J., Hashimoto, R., Lisi, G., Shibata, K., Kawakubo, Y., ..., & Kawato, M. (2016).
A small number of abnormal brain connections predicts adult autism spectrum disorder. Nature communications, 7, 11254. [PubMed:27075704] [PMC] [WorldCat] [DOI] - ↑
Sporns, O., Tononi, G., & Kötter, R. (2005).
The human connectome: A structural description of the human brain. PLoS computational biology, 1(4), e42. [PubMed:16201007] [PMC] [WorldCat] [DOI] - ↑
Greicius, M.D., Krasnow, B., Reiss, A.L., & Menon, V. (2003).
Functional connectivity in the resting brain: a network analysis of the default mode hypothesis. Proceedings of the National Academy of Sciences of the United States of America, 100(1), 253-8. [PubMed:12506194] [PMC] [WorldCat] [DOI] - ↑
Vincent, J.L., Patel, G.H., Fox, M.D., Snyder, A.Z., Baker, J.T., Van Essen, D.C., ..., & Raichle, M.E. (2007).
Intrinsic functional architecture in the anaesthetized monkey brain. Nature, 447(7140), 83-6. [PubMed:17476267] [WorldCat] [DOI] - ↑
Smith, S.M. (2012).
The future of FMRI connectivity. NeuroImage, 62(2), 1257-66. [PubMed:22248579] [WorldCat] [DOI] - ↑
Nickerson, L.D., Smith, S.M., Öngür, D., & Beckmann, C.F. (2017).
Using Dual Regression to Investigate Network Shape and Amplitude in Functional Connectivity Analyses. Frontiers in neuroscience, 11, 115. [PubMed:28348512] [PMC] [WorldCat] [DOI] - ↑
Smith, S.M., Miller, K.L., Moeller, S., Xu, J., Auerbach, E.J., Woolrich, M.W., ..., & Ugurbil, K. (2012).
Temporally-independent functional modes of spontaneous brain activity. Proceedings of the National Academy of Sciences of the United States of America, 109(8), 3131-6. [PubMed:22323591] [PMC] [WorldCat] [DOI] - ↑
Calhoun, V.D., Adali, T., Pearlson, G.D., & Pekar, J.J. (2001).
A method for making group inferences from functional MRI data using independent component analysis. Human brain mapping, 14(3), 140-51. [PubMed:11559959] [PMC] [WorldCat] [DOI] - ↑ 35.0 35.1
Glasser, M.F., Coalson, T.S., Robinson, E.C., Hacker, C.D., Harwell, J., Yacoub, E., ..., & Van Essen, D.C. (2016).
A multi-modal parcellation of human cerebral cortex. Nature, 536(7615), 171-178. [PubMed:27437579] [PMC] [WorldCat] [DOI] - ↑
Smith, K. (2012).
Brain imaging: fMRI 2.0. Nature, 484(7392), 24-6. [PubMed:22481337] [WorldCat] [DOI]