「膜貫通AMPA受容体調節性タンパク質」の版間の差分
細編集の要約なし |
|||
| 33行目: | 33行目: | ||
===TARP type II (TARPγ-5/7)=== | ===TARP type II (TARPγ-5/7)=== | ||
Type | Type Iと異なり、典型的なPDZドメイン結合配列およびアルギニン酸残基クラスターリング部位を有さない<ref name=ref2 />。 | ||
==分布== | ==分布== | ||
2016年2月4日 (木) 15:52時点における版
富田 進
イェール大学
DOI:10.14931/bsd.6840 原稿受付日:2016年1月27日 原稿完成日:2016年月日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
英語名: Transmembrane AMPA receptor regulatory protein
英語略称:TARP
膜貫通AMPA受容体調節性タンパク質 TARPは、線虫から哺乳類まで保存されているAMPA型グルタミン酸受容体の補助サブユニットである。構造上の類似性から、当初カルシウムチャネルγサブユニットと名付けられたが、 これまでにAMPA型グルタミン酸受容体に特異的に作用することが明らかになっている。TARPはAMPA受容体に特異的に結合し、受容体発現量、シナプス局在、チャネル活性を制御する[1] [2] [3] [4]。
イントロダクション
自然発生変異マウスであるスターゲイザーは運動失調や欠伸発作を示す[5]。その原因遺伝子 はカルシウムチャネルγサブユニットと38%の相同性を示すことから Calcium channel gamma subunit 2 (CACNG2)/Stargazinと名付けられ[6]、その後8つのCACNG類似遺伝子群が報告された[7]。しかしながら、電気生理学的解析により、スターゲイザーマウスの小脳苔状繊維-小脳顆粒細胞シナプスにおけるAMPA受容体活性が特異的に消失していること[8]、カルシムチャネル活性に変化がないことが示され[9]。さらなる解析により、8つのCACNGファミリーのうちCACNG2/3/4/5/7/8の6つがAMPA 受容体に特異的に結合すること示され、これらはTransmembrane AMPA receptor Regulatory Protein (TARP) γ-2/3/4/5/7/8と名付けられた[10] [11] [12]。
ファミリー
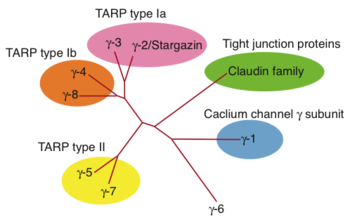
6つのアイソフォームが同定されており、後述のようにAMPA 受容体修飾機能の違いにより、さらにサブクラスに分類されている(図1)。また、線虫において、その機能的相同体STG-1/2が示されている[13] [14]。
構造
4回膜貫通部位を有し、NとCの両末端が細胞内にある(図2)。1つ目の細胞外ドメインに、N結合型糖鎖修飾部位をもつ。この細胞外ドメインは、AMPA 受容体活性の調節に必須である[15] [16] [17]。
TARP type I (TARPγ-2/3/4/8)
C末端にはPDZドメイン結合配列を有し、PSD-95様MAGUKs や他のPDZドメイン含有タンパク質に結合し、シナプス局在を調節することが示されている[9] [18] [19] [20]。
C末端細胞内ドメインには、アルギニン酸残基クラスターリング部位があり、陽性に荷電している。この領域には、少なくとも9つのリン酸化セリン残基(P-Ser)がこれまでに同定されており、タンパク質-脂質結合、タンパク質-タンパク質結合を調整していると考えられている[21] [22] [23] [24]。
TARP type II (TARPγ-5/7)
Type Iと異なり、典型的なPDZドメイン結合配列およびアルギニン酸残基クラスターリング部位を有さない[2]。
分布
組織レベルでは、脳に強い発現を示し、それぞれのアイソフォームは脳内で特徴的な発現パターンを示す[10] [25]。例えば、γ-2は小脳顆粒細胞、γ-8は海馬、γ-3は大脳皮質、γ-4は発生過程において強く発現している。
TARPは、免疫染色、生化学分画によりAMPA受容体同様に、シナプス及び膜表面に強く検出される。 また、TARPはAMPA受容体と安定的複合体を形成することも、AMPA受容体との共局在を支持する[10] [26] [27] [28] [29]。
機能
TARPはAMPA受容体に結合し、AMPA受容体のタンパク質発現量、チャネル活性、および細胞表面とシナプス局在を調節する。
チャネル活性
TARP はAMPA 受容体のチャネル活性を制御する[15] [17] [30]。TARPの細胞外ドメインを介して、AMPA 受容体のチャネルの開口速度を速くすることにより、脱感作、脱活性の速度を遅くする[15]。さらに、脳において、AMPA 受容体由来のシナプス伝達の減衰速度を調節しており[15]、クラス1b型TARPは、クラス1a型TARPに比べて、定量的により効果的に減衰速度を遅延する[31] [32]。このとき、TARP はAMPA 受容体の開口確率を変えずに、サブコンダクタンスの支配率を変える[15]。
シナプス局在
TARP はAMPA 受容体のシナプス局在を制御する9 。小脳顆粒細胞および海馬において、TARPとそのPDZドメイン結合配列はAMPA 受容体のシナプス局在に必須である[9] [33]。PDZドメイン結合配列は、PSD-95を始めとするMAGUKsに結合する[9] [18] [19] [20]。また、TARP細胞質ドメインのリン酸化依存的に脂質結合が変化し、シナプス局在が調節されることが知られている[21] [22] [34]。
AMPA受容体のタンパク質発現量
TARPはAMPA受容体の発現を安定化する。海馬に多く発現するγ-8遺伝子欠損マウスにおいて、海馬におけるAMPA受容体の発現量が10%になる[28] [29]。このとき、N結合型糖鎖が成熟したAMPA受容体のみが減少し、ERに多く局在するN結合型糖鎖の未成熟なAMPA受容体に影響を及ぼさないことから、TARPは成熟したAMPA受容体を安定化していると考えられている[33]。また、小脳顆粒細胞に発現するγ-2の変異マウス、スターゲイザーにおいて、15%程度のAMPA受容体の発現量減少が小脳(全体)で観測される[10]。
細胞表面の発現
TARP はAMPA 受容体の細胞表面への発現を調節する。この機能に、PDZ結合ドメインは関与していない[9] [33]。また、TARP細胞質ドメインのリン酸化にともなって、AP4を介してエンドサイトーシスが調節される[23]。
個体レベルでの機能
TARP γ-2遺伝子欠損マウスでは、運動失調や欠伸発作が観測される[5]。
TARP γ-2を含むトリプルTARP typeI 遺伝子欠損マウスは致死となる[35]。
TARP γ-4遺伝子欠損マウスでは、けいれんが観測される[36]。
TARP γ-2/7の両TARP遺伝子欠損マウスにおいて、小脳プルキンエ細胞中のシナプスAMPA 受容体活性が消失する[37]。
関連項目
参考文献
- ↑ Resource not found in PubMed.
- ↑ 2.0 2.1 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ 5.0 5.1 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ 9.0 9.1 9.2 9.3 9.4 Resource not found in PubMed.
- ↑ 10.0 10.1 10.2 10.3 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ 15.0 15.1 15.2 15.3 15.4 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ 17.0 17.1 Resource not found in PubMed.
- ↑ 18.0 18.1 Resource not found in PubMed.
- ↑ 19.0 19.1 Resource not found in PubMed.
- ↑ 20.0 20.1 Resource not found in PubMed.
- ↑ 21.0 21.1 Resource not found in PubMed.
- ↑ 22.0 22.1 Resource not found in PubMed.
- ↑ 23.0 23.1 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ 28.0 28.1 Resource not found in PubMed.
- ↑ 29.0 29.1 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ 33.0 33.1 33.2 Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.
- ↑ Resource not found in PubMed.