「転写制御因子」の版間の差分
(ページの作成:「転写制御因子 英:transcriptional regulator 同義語:転写調節因子 転写制御因子は、ゲノムDNA上の特定の塩基配列に結合し、RNAポ...」) |
細編集の要約なし |
||
| 1行目: | 1行目: | ||
英:transcriptional regulator, transcription regulatory factor | |||
英:transcriptional regulator | |||
同義語:転写調節因子 | 同義語:転写調節因子 | ||
転写制御因子は、[[wikipedia:ja:ゲノム|ゲノム]] [[wikipedia:ja:DNA|DNA]]上の特定の塩基配列に結合し、[[wikipedia:ja:RNAポリメラーゼ|RNAポリメラーゼ]]による転写を促進あるいは抑制する[[タンパク質]]の一群である<ref name=ref1> Tetsuichiro Saito<br>Transcription factor. Encyclopedia of Neuroscience<br> Springer-Verlag Gmbh Berlin Heidelberg: 2009</ref>。DNAに結合するドメインと他のタンパク質などと相互作用し転写制御に関わるドメインを有する。構造上の特徴により、いくつかのファミリーに分類される<ref name=ref1 />。転写制御因子は[[wikipedia:ja:リン酸化|リン酸化]]などの様々な調節を受け、個体発生から脳高次機能までの多くの過程を制御する<ref name=ref2><pubmed>11823631</pubmed></ref><ref name=ref3><pubmed>23498934</pubmed></ref>。転写制御因子をコードすると推測される遺伝子は、[[wikipedia:ja:ヒトゲノム|ヒトゲノム]]において約2000個存在する<ref><pubmed>15193307</pubmed></ref><ref><pubmed>19274049</pubmed></ref>。 | |||
==転写制御因子とは== | ==転写制御因子とは== | ||
遺伝子の転写は、RNAポリメラーゼがDNAを鋳型として相補的な[[wikipedia:ja: RNA|RNA]]を5’から3’の方向へ合成する反応である。転写においてRNAポリメラーゼの他に必要とされるタンパク質を転写因子 (transcription factor) という。その中で、転写制御因子はDNAに結合し、転写を調節するタンパク質であり、転写を促進するものを転写活性化因子、抑えるものを転写抑制因子という<ref name=ref1 />。一方、DNAに直接結合せず、転写制御因子とRNAポリメラーゼなどとの仲介役として転写を調節するタンパク質複合体はメディエーターである。メディエーターの機能は多岐に渡り、転写を活性化するコアクチベーターや、転写を抑制するコリプレッサーとして働く<ref name=ref4><pubmed>15680973</pubmed></ref><ref name=ref5><pubmed>20940737</pubmed></ref>。メディエーター以外にも、コアクチベーターやコリプレッサーと呼ばれる因子があり、[[wikipedia:ja:ヒストン|ヒストン]]の[[アセチル化]]や[[メチル化]]などを介して、[[wikipedia:ja: クロマチン|クロマチン]]の状態を制御する<ref name=ref5 /><ref name=ref8><pubmed>21321607</pubmed></ref>。DNA上で転写の活性化に働く領域は[[エンハンサー]]、転写を抑制する領域がサイレンサーであり、それぞれ複数の転写制御因子が結合することが多い<ref name=ref15><pubmed>12853946</pubmed></ref><ref name=ref6><pubmed>21358745</pubmed></ref><ref name=ref7><pubmed>22868264</pubmed></ref>。 | |||
==転写制御機構== | ==転写制御機構== | ||
in | in vitroの解析により、転写は[[wikipedia:ja:TATAボックス| TATAボックス]]などのコアプロモーター上で[[wikipedia:ja:TATA結合タンパク質|TATA結合タンパク質]] (TATA-binding protein; TBP) やTAFs (TBP-associated factors) などの[[wikipedia:ja:基本転写因子|基本転写因子]]がRNAポリメラーゼと[[wikipedia:ja:転写開始前複合体|転写開始前複合体]]を形成することにより開始されることが示されている<ref name=ref4 /><ref name=ref15 /><ref name=ref16><pubmed>20628347</pubmed></ref>。これらの因子のみによる転写は基本転写と呼ばれる。生体内では、様々な転写制御因子が働き、その組み合わせにより遺伝子の転写量が組織や発生の時期で異なる<ref name=ref2 /><ref name=ref3 /><ref name=ref7 />。 | ||
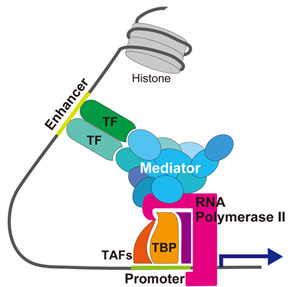
[[ファイル:TF0730.jpg|frame|right|図1 | [[ファイル:TF0730.jpg|frame|right|'''図1 転写活性化機構''' <br> | ||
転写活性化因子(TF)はエンハンサーに結合し、メディエーターを介してプロモーターにおける転写開始前複合体の形成を促進する。]] | |||
===転写活性化の機構=== | ===転写活性化の機構=== | ||
転写の活性化は、転写の開始と伸長で起こる。活性化の例として、エンハンサーに結合した転写活性化因子が基本転写因子と相互作用し、[[wikipedia:ja:プロモーター|プロモーター]]における転写開始前複合体の形成を促進することが知られている<ref name=ref1 /><ref name=ref15 /><ref name=ref16 />。一方、メディエーターは、転写制御因子の転写活性化ドメインとRNAポリメラーゼの双方に結合し、転写開始前複合体の形成を促し、コアクチベーターとして働く<ref name=ref4 /><ref name=ref5 /><ref name=ref17><pubmed>20299225</pubmed></ref> (図1)。 | |||
真核細胞のDNAはヒストンに巻きつき、[[wikipedia:ja: ヌクレオソーム|ヌクレオソーム]]を形成する。ヌクレオソーム構造を弛緩させ、転写制御因子などをDNAに作用しやすくし、転写を活性化する因子もコアクチベーターと呼ばれる。コアクチベーターのCBP (CREB-binding protein) は、ヒストンをアセチル化し、ヒストンとDNA間の親和性を弱める<ref name=ref8 />。ヒストンリモデリング因子の[[wikipedia:SWI/SNF|SWI/SNF]](switch/sucrose nonfermentable) は、[[wikipedia:ja:ATP|ATP]]の[[wikipedia:ja:加水分解|加水分解]]で生じるエネルギーを用いてクロマチン構造を変化させる<ref><pubmed>21358755</pubmed></ref>。 | |||
RNAポリメラーゼは数十塩基程度の短いRNAを転写後、DSIF (DRB-sensitivity inducing factor) やNELF (negative elongation factor) などの伸長阻害因子の働きにより転写を休止する<ref name=ref9><pubmed>22986266</pubmed></ref>。Mycなどの転写制御因子は、[[wikipedia:ja:プロテインキナーゼ|プロテインキナーゼ]]P-TEFb (positive transcription elongation factor b) の作用を介して転写の休止を解除し、RNA伸長を再開させることが知られている<ref name=ref9 />。 | |||
===転写抑制の機構=== | ===転写抑制の機構=== | ||
転写抑制では、サイレンサーに結合した転写抑制因子がTLE/Grg (transducin-like Enhancer of split/groucho-related gene) やNcoR / SMRT (nuclear receptor corepressor/silencing mediator for retinoic and thyroid receptors) などのコリプレッサーを介して[[wikipedia:ja:ヒストンデアセチラーゼ|ヒストンデアセチラーゼ]]を引き寄せ、ヒストンのアセチル基を除去することによりヌクレオソームを安定化させ遺伝子を不活化する<ref><pubmed>18721877</pubmed></ref><ref><pubmed>21049000</pubmed></ref>。また、ヒストンメチルトランスフェラーゼを引き寄せ、ヒストンの特定の部位を[[wikipedia:ja:メチル化|メチル化]]し、転写が起きないクロマチン状態を維持することにより抑制する場合もある<ref name=ref5 /><ref name=ref8 />。 | |||
転写抑制因子のNRSF/REST (neuron-restrictive silencer factor/RE1-silencing transcription factor) は、サイレンサー中の塩基配列NRSE/RE1 (neural restrictive silencer element/repressor element 1) に結合し、コリプレッサーを介したヒストンデアセチラーゼやヒストンメチルトランスフェラーゼの作用により、[[神経細胞]]で働く''SCG10 (superior cervical ganglia 10)'' などの遺伝子の転写を非神経細胞で抑える<ref><pubmed>16150588</pubmed></ref>。 | |||
CDK8 (Cyclin dependent kinase 8) などのコリプレッサーは、転写制御因子をリン酸化し分解を促進したり、基本転写因子をリン酸化し転写開始前複合体の形成を阻害する<ref name=ref5 /><ref name=ref17 />。 | |||
==転写制御配列== | |||
転写制御因子のDNA結合ドメインは、多くの場合、4から10塩基の特定の配列(コンセンサス配列)に結合する。エンハンサーやサイレンサーには、通常、転写制御因子の結合する場所(転写制御因子結合部位)がいくつかあり、複数種の転写制御因子で転写が調節される<ref name=ref6/><ref name=ref7 />。 遺伝子の転写が複数のエンハンサーで調節されることも多く、発生段階に特異的なエンハンサーと組織特異的なエンハンサーなどに分離できることもある。 | |||
DNA配列における転写制御因子結合部位は、MatInspector (http://www.genomatix.de/index.html) や、PAZAR (http://www.pazar.info/) などで予測できる。 | |||
ENCODEプロジェクトなどにより、ゲノムのかなりの領域が転写されることが明らかとなった<ref><pubmed>22955616</pubmed></ref>。エンハンサー部位で合成されるeRNA (enhancer RNA) などの非翻訳RNAが転写に関わることが示唆されている<ref><pubmed>23273978</pubmed></ref><ref><pubmed>20887892</pubmed></ref>。 | |||
==構造== | ==構造== | ||
転写活性化因子の転写活性化ドメインは、コアクチベーターなどのタンパク質と結合し、酸性アミノ酸、もしくは[[wikipedia:ja:グルタミン酸|グルタミン酸]]、[[wikipedia:ja:プロリン|プロリン]]のいずれかに富む領域などに分類される。転写抑制因子の転写抑制ドメインには、[[wikipedia:ja:トリプトファン|トリプトファン]]、[[wikipedia:ja:アルギニン|アルギニン]]、プロリンからなるWRPWドメインや、芳香族アミノ酸と疎水性アミノ酸からなるEH1 (engrailed homology 1) ドメインなどがあり、コリプレッサーとの結合に必須である<ref><pubmed>18254933</pubmed></ref><ref><pubmed>16309560</pubmed></ref>。[[wikipedia:ja:ジンクフィンガー|ジンクフィンガー]]型因子などは、リガンドと結合するドメインも有する。 | |||
転写制御因子の構造については、Protein Data Bank (http://www.rcsb.org/pdb/home/home.do) やPfam (http://pfam.sanger.ac.uk/) などのデータベースで検索できる。 | |||
===DNA結合ドメインによる分類=== | ===DNA結合ドメインによる分類=== | ||
転写制御因子は、DNA結合ドメインの構造モチーフに基づき、ホメオドメイン、ジンクフィンガー、塩基性へリックス・ループ・へリックスなどのファミリーに分けられる。 | |||
====ホメオドメイン(homeodomain)==== | |||
[[wikipedia:ja:ホメオボックス|ホメオボックス]]がコードする約60個のアミノ酸配列である。ショウジョウバエの体節形成の研究で発見され、ヒトを含む高等動物まで[[wikipedia:ja:ホモログ|ホモログ]]間でよく保存されている<ref><pubmed>11470884</pubmed></ref>。へリックス・ターン・へリックス構造をとり、2番目のヘリックスがDNAの主溝に入り込んで結合する。アミノ酸配列の類似性やホメオドメインの外のモチーフから、さらにサブファミリーに分けられる。ホメオドメインが結合するコンセンサス配列の代表としてATTAが知られ、サブファミリーにより認識配列が異なる<ref><pubmed>18585359</pubmed></ref>。 | |||
====ジンクフィンガー(zinc finger)==== | |||
GATAファミリーの因子などに見られる構造で、亜鉛イオンに2個の[[wikipedia:ja:システイン|システイン]]と2個のヒスチジンが配位結合するC2H2タイプや、4個のシステインが配位結合するC4タイプなどがある<ref><pubmed>18253864</pubmed></ref>。亜鉛に配位したシステインあるいはヒスチジン残基に挟まれたアミノ酸領域が指状のループをつくる。[[wikipedia:ja:ステロイドホルモン|ステロイドホルモン]]受容体などの[[wikipedia:ja:核内受容体|核内受容体]]は、[[wikipedia:ja:リガンド|リガンド]]と直接結合すると核内に入り転写制御能を発揮するようになる<ref><pubmed>20813267</pubmed></ref><ref><pubmed>20850017</pubmed></ref>。コンセンサス配列にはGATAファミリーの因子が結合するGATAなどがある。 | |||
====塩基性へリックス・ループ・へリックス(basic helix-loop-helix、bHLH) ==== | |||
Mash1/Ascl1 (mammalian achaete-scute homolog 1/ achaete-scute complex homolog 1)やMath1/ Atoh1 (mammalian atonal homolog 1/ atonal homolog 1) などの神経分化を開始させるプロニューラル因子などに見られる<ref name=ref10><pubmed>12094208</pubmed></ref>。E12/Tcf3 (transcription factor 3) タンパク質などのへリックス・ループ・へリックスドメインとヘテロ[[wikipedia:ja:二量体|二量体]]を形成し、DNAに結合する。コンセンサス配列として、プロニューラル因子などの転写活性化因子が結合するE box (CANNTG) と、Hes1 (hairy, Enhancer of split 1) などの転写抑制因子が結合するN box (CACNAG) などが知られている<ref name=ref11><pubmed>17329370</pubmed></ref>。 | |||
====ロイシンジッパー(leucine zipper)==== | |||
c-FosやMycなどに見られ、7アミノ酸ごとに[[wikipedia:ja:ロイシン|ロイシン]]が配置された[[wikipedia:ja:コイルドコイル|コイルドコイル]]と呼ばれる構造を取る。同様の構造を持つ因子とコイルドコイル間でヘテロ二量体を形成し、コイルドコイルのN端側に存在する塩基性アミノ酸に富む領域でDNAに結合する。コンセンサス配列には[[wikipedia:ja:CREB|CREB]] (cAMP-responsive element-binding protein) が結合するサイクリックAMP応答配列(TGACGTCA)などがある<ref name=ref18><pubmed>20223527</pubmed></ref>。 | |||
==== | ====HMG(high mobility group)ボックス ==== | ||
[[ | クロマチンから0.35M[[wikipedia:ja:塩化ナトリウム|塩化ナトリウム]]によって抽出され、電気泳動で高い移動度を示すタンパク質のDNA結合領域で見つかった。約80アミノ酸からなり、三つのαへリックスを形成する<ref><pubmed>23153957</pubmed></ref>。特に、性決定遺伝子''[[wikipedia:ja:Sry|Sry]] ''がコードするタンパク質と高い相同性を持つ因子はSoxファミリーと呼ばれる。コンセンサス配列にはSoxファミリーの結合するT(A/T)(A/T)CAAGなどがある。 | ||
{| class="wikitable" style="text-align:center" | {| class="wikitable" style="text-align:center" | ||
| 44行目: | 62行目: | ||
! DNA結合ドメイン|| ファミリー || 例 | ! DNA結合ドメイン|| ファミリー || 例 | ||
|- | |- | ||
| rowspan=" | | rowspan="9" | homeodomain | ||
| Hox | | Hox | ||
| HoxA1,HoxC8 | | HoxA1, HoxC8 | ||
|- | |||
| Pax | |||
| Pax2, Pax6 | |||
|- | |- | ||
| | | Dlx | ||
| | | Dlx1, Dlx2 | ||
|- | |- | ||
| Emx | | Emx | ||
| 55行目: | 76行目: | ||
|- | |- | ||
| Nkx | | Nkx | ||
| Nkx2.2,Nkx6.1 | | Nkx2.2, Nkx6.1 | ||
|- | |- | ||
| En | | En | ||
| 61行目: | 82行目: | ||
|- | |- | ||
|Bar | |Bar | ||
| | | Barh1, Barh2 | ||
|- | |- | ||
| | |paired-like | ||
|Phox2a,DRG11 | |Phox2a, DRG11 | ||
|- | |- | ||
|POU | |POU | ||
|Brn3a | |Brn3a | ||
|- | |- | ||
| [[ | |rowspan="4" | [[zinc finger]] | ||
| | |C2H2 | ||
| | |Gli1, NRSF/REST, Zif268, Krox20, Fezl | ||
|- | |||
|kruppel-like | |||
|Klf4 | |||
|- | |||
|Gata | |||
|Gata2, Gata3 | |||
|- | |||
|nuclear hormone receptor | |||
|RARα1, RXR | |||
|- | |- | ||
|rowspan=" | |rowspan="3" | [[basic helix-loop-helix]] | ||
|bHLH | |bHLH | ||
| Mash1, | | Mash1/Ascl1, Math1/Atoh1, Neurogenin1, NeuroD, Ptf1a, Hes1, Olig2, Scl/Tal1, E12/Tcf3 | ||
|- | |- | ||
|bHLH-PAS | |bHLH-PAS | ||
|Per1,Clk | |Per1, Clk | ||
|- | |||
|bHLH-leucine zipper | |||
|Myc, MycN/N-Myc | |||
|- | |- | ||
| [[ | | [[leucine zipper]] | ||
| | | | ||
| | |c-Fos, CREB | ||
|- | |- | ||
|rowspan=" | |rowspan="2" | [[HMG box]] | ||
|Sox | |Sox | ||
|Sox2,Sox9,Sox10 | |Sox2, Sox9, Sox10 | ||
|- | |||
|Tcf/Lef | |||
|Tcf1/Lef1 | |||
|- | |- | ||
| [[Ets]] | |||
| | | | ||
| | |Pea3, Er81 | ||
|- | |- | ||
| | | [[T box]] | ||
| | | | ||
|Tbr1 | |||
|- | |- | ||
| | | [[forkhead]] | ||
| | |Fox | ||
| FoxG1/Bf1, FoxO, FoxP2 | |||
|- | |- | ||
| [[Rel homology]] | |||
| [[Rel homology | |||
| | | | ||
|Rbpj | | Rbpj /CBF1, NF-κB | ||
|- | |- | ||
| [[MH1 | | [[MH1]] | ||
|Smad | |Smad | ||
|Smad2, Smad4 | |Smad2, Smad4 | ||
|- | |- | ||
| [[ | | [[SH2]] | ||
| | |STAT | ||
| | |STAT3 | ||
|} | |} | ||
==転写制御因子の活性制御== | ==転写制御因子の活性制御== | ||
転写制御因子の活性は、細胞の分化段階や細胞外からの刺激などにより制御され、リン酸化やリガンド結合、[[wikipedia:ja:ユビキチン|ユビキチン]]化などの影響を受ける<ref name=ref2 />。また、転写制御因子には、構造的に類似した因子同士で二量体を形成し機能するものも多い。へリックス・ループ・へリックス因子の一つであるIdファミリーは塩基性領域を欠き、塩基性へリックス・ループ・へリックス因子のE12/Tcf3などとへテロ二量体を形成し、E12/Tcf3がDNAに結合することを阻害する<ref><pubmed>11807807</pubmed></ref>。 | |||
TGF-β (transforming growth factor-β) ファミリーの分泌タンパク質が膜受容体に結合すると、MH1 (Mad homology 1) ドメイン型因子のSmad2とSmad3はリン酸化され、Smad4とヘテロ二量体を形成後、核に移行して転写を制御する<ref><pubmed>21565618</pubmed></ref>。 | |||
[[wikipedia:ja:インターフェロン|インターフェロン]]などの[[wikipedia:ja:サイトカイン|サイトカイン]]やホルモンが受容体に結合すると、Jak (Janus kinase)によってSH2 (Src homology 2) ドメイン型因子の[[シグナル伝達因子件転写活性化因子]] (signal transducers and activator of transcription; STAT) がリン酸化される。リン酸化されたSTATはSH2ドメインを介して二量体を形成し、核に移行する<ref><pubmed>24058789</pubmed></ref>。 | |||
Hedgehogが膜上の受容体Patchedに結合すると、膜タンパク質Smoothendを介してジンクフィンガー型因子のGliが活性化される。活性化したGliは核に移行し、標的遺伝子の転写を制御する<ref><pubmed>18621990</pubmed></ref>。 | |||
DeltaやJaggedが受容体[[Notch]]に結合すると、γセクレターゼの作用によりNotch受容体の細胞内ドメインが切り出される。Notchの細胞内ドメインは核内に移行し、Rel homology型因子のRbpj/CBF-1 (recombination signal sequence-binding protein J/C promoter-binding factor 1) やコアクチベーターMaml1 (Mastermind-like 1) と複合体を形成し''Hes1''などの転写を活性化する<ref><pubmed>21505516</pubmed></ref>。 | |||
[[Wnt]]が膜受容体Frizzledと結合するとβ-cateninが遊離され、このβ-cateninはHMGボックス型因子のTcf1/Lef1 (T-cell factor 1/lymphoid enhancer binding factor 1) とヘテロ二量体を形成後、核内に移行し転写を制御する<ref><pubmed>23772206</pubmed></ref><ref><pubmed>18083108</pubmed></ref>。 | |||
ロイシンジッパー型因子のCREBは、サイクリックAMP 濃度の上昇などの様々な刺激によりサイクリックAMP依存性プロテインキナーゼや[[カルシウムカルモジュリン依存性プロテインキナーゼ]]などによるリン酸化を受け、活性化する<ref name=ref18 /><ref name=ref12><pubmed>19126756</pubmed></ref>。 | |||
Pax6 (paired box 6) やMycN/N-mycなどの転写制御因子はユビキチン化され、ユビキチン・[[wikipedia:ja:プロテアソーム|プロテアソーム]]系によって分解されることが明らかとなっている<ref><pubmed>18430159</pubmed></ref>。 | |||
==生体内での役割== | |||
===発生・細胞分化=== | ===発生・細胞分化=== | ||
発生過程では、多数の転写制御因子が働く。神経発生の初期においては、Pax6やEmx2 (empty spiracles homeobox 2)、Nkx6.1 (NK6 homeobox 6.1) などのホメオドメイン型因子が[[神経管]]の前後軸や背腹軸に沿って特異的に発現し、[[終脳]]形成を制御する<ref><pubmed>19143049</pubmed></ref>。Hoxファミリーのタンパク質は、後脳と[[脊髄]]の[[前後軸]]沿いの特定の領域の個性に関わり、[[運動神経細胞]]などの分化を正常に進めるために必須である<ref name=ref13><pubmed>18524570</pubmed></ref>。 | |||
Mash1/Ascl1 やMath1/Atoh1 などのプロニューラル因子は神経分化の開始を司り、神経細胞の分化や個性を制御する遺伝子の転写を活性化する<ref name=ref10 />。[[神経幹細胞]]や[[神経前駆細胞]]ではNotchがHes1やHes5を活性化し、Hes1やHes5タンパク質がプロニューラル遺伝子の上流などに結合し、転写を抑える<ref name=ref11 />。 | |||
Math1はBar型ホメオボックス遺伝子''Barh1 (Bar-class homeobox 1)'' と''Barh2'' を直接活性化し、Barh1とBarh2タンパク質が脊髄の交連神経細胞の個性を決定する<ref><pubmed>15788459</pubmed></ref><ref name=ref14><pubmed>20599893</pubmed></ref>。[[小脳]]ではMath1/Atoh1は、[[グルタミン酸]]作動性の顆粒細胞の分化を促進する一方、GABA (γ-aminobutyric acid) 作動性神経細胞産生に必須な''Ptf1a (pancreas transcription factor 1a)'' の発現を抑える<ref><pubmed>18723012</pubmed></ref>。 | |||
脊髄において、塩基性へリックス・ループ・へリックス型因子のOlig2は運動神経細胞の分化と[[オリゴデンドロサイト]]の産生に必須な一方、Scl/Tal1は[[介在神経細胞]]の分化と[[アストロサイト]]の産生を制御する<ref><pubmed>21068830</pubmed></ref>。 | |||
ホメオボックス遺伝子の''Dlx1 (distal-less homeobox 1)'' と''Dlx2'' は、[[大脳基底核原基]]由来の細胞で発現し、大脳のGABA作動性神経細胞の産生に必須である<ref><pubmed>19428236</pubmed></ref>。 | |||
HMGボックス型因子のSox2は、プロニューラル因子と拮抗的に働き、神経分化を抑制する<ref name=ref15><pubmed>16139372</pubmed></ref>。また、Sox9やSox10はオリゴデンドロサイトの前駆細胞で発現し、オリゴデンドロサイトの分化を促進するとともに、ミエリン塩基性タンパク質などの遺伝子の転写を活性化する<ref name=ref15 />。 | |||
===軸索伸長、細胞極性=== | ===軸索伸長、細胞極性=== | ||
プロニューラル因子のNeurogenin2は、神経細胞の移動や[[軸索]]投射にも関与する<ref><pubmed>16202708</pubmed></ref><ref><pubmed>21864333</pubmed></ref>。 | |||
脊髄交連神経の軸索投射は、Bar型やLIM型 のホメオドメイン型因子によって制御される。Barh1 とBarh2の下流では、''Lhx2 (LIM homeobox 2)'' を介した''Rig1/Robo3 (Rb-inhibiting gene 1/roundabout homolog 3) ''の転写調節とともに、''Lhx2''を介さない''Neuropilin2''の転写調節が行われる<ref name=ref14 />。 | |||
運動神経や感覚神経では、軸索が標的筋肉の近傍に到着すると、''Pea3 (polyomavirus enhancer activator 3)'' や''Er81 (ets-related protein 81)'' などのEtsファミリー遺伝子の転写が誘導され、軸索の枝分かれが制御される<ref name=ref13/>。また、フォークヘッド型因子のFoxO (forkhead box O) は、''Pak1(p21 protein-activated kinase 1)'' などの細胞極性を制御する遺伝子の転写を調節し、神経細胞の形態制御に関わる<ref><pubmed>21982366</pubmed></ref>。 | |||
===高次機能=== | ===高次機能=== | ||
CREBは、神経活動で活性化され[[シナプス]]の構造を制御する遺伝子の転写を調節し、長期記憶の形成に関与する<ref name=ref18 /><ref name=ref12 />。 | |||
Rel homology型因子のNF-κB (nuclear factor-κB) は、発生過程において神経軸索の伸長や樹状突起の枝分かれなど神経突起の発達に重要な役割を果たす一方、成体では[[樹状突起]]のスパイン数やシナプス形成などを介して[[学習]]や記憶に関わることが示唆されている<ref name= ref12 /><ref><pubmed>21459462</pubmed></ref>。 | |||
ロイシンジッパー型因子のc-Fosやジンクフィンガー型因子のZif268は、神経活動などの刺激により一過的に誘導される[[最初期遺伝子]]であり、神経回路の可塑的変化への関与が示唆されている<ref name= ref12 />。 | |||
フォークヘッド型因子のFoxP2 (forkhead box P2)は、発声や言語発達に関わることが示唆されている<ref><pubmed>21663442</pubmed></ref>。 | |||
===環境への応答=== | ===環境への応答=== | ||
低酸素状態では、塩基性へリックス・ループ・へリックス型因子のHIF-1α (hypoxia inducible factor 1 α) が安定化され、HIF-1βとヘテロ二量体を形成して血管新生や解糖系に関わる遺伝子群の転写を活性化する<ref><pubmed>14643885</pubmed></ref>。 | |||
時計遺伝子''Period''は塩基性へリックス・ループ・へリックス型因子をコードし、光刺激により誘導される。''Period''の転写は他の塩基性へリックス・ループ・へリックス型因子のClockとBmal1 (brain and muscle Arnt-like 1) により調節されており、これらの因子がネガティブフィードバックループを形成し、約24時間周期で発現が変動する<ref><pubmed>22483041</pubmed></ref>。 | |||
==病理== | ==病理== | ||
ヒトの疾患の原因となる転写制御因子の変異はTRANSFACやOMIM Morbid Map (http://www.omim.org/search/advanced/geneMap) などのデータベースなどで調べられる。 | |||
==転写制御因子データベース == | |||
転写制御因子のリストはTRANSFACデータベース(http://www.gene-regulation.com) から入手可能である。また、転写制御因子についてのレビューもデータベース化されている (Transcription Factor Encyclopedia、 http://www.cisreg.ca/cgi-bin/tfe/home.pl) <ref><pubmed>22458515</pubmed></ref>。 | |||
== 関連項目 == | |||
*[[エンハンサー]] | |||
<references/> | <references/> | ||
(執筆担当者: 室山優子、斎藤哲一郎 担当編集委員: 大隅典子) | (執筆担当者: 室山優子、斎藤哲一郎 担当編集委員: 大隅典子) | ||
2013年11月8日 (金) 12:56時点における版
英:transcriptional regulator, transcription regulatory factor
同義語:転写調節因子
転写制御因子は、ゲノム DNA上の特定の塩基配列に結合し、RNAポリメラーゼによる転写を促進あるいは抑制するタンパク質の一群である[1]。DNAに結合するドメインと他のタンパク質などと相互作用し転写制御に関わるドメインを有する。構造上の特徴により、いくつかのファミリーに分類される[1]。転写制御因子はリン酸化などの様々な調節を受け、個体発生から脳高次機能までの多くの過程を制御する[2][3]。転写制御因子をコードすると推測される遺伝子は、ヒトゲノムにおいて約2000個存在する[4][5]。
転写制御因子とは
遺伝子の転写は、RNAポリメラーゼがDNAを鋳型として相補的なRNAを5’から3’の方向へ合成する反応である。転写においてRNAポリメラーゼの他に必要とされるタンパク質を転写因子 (transcription factor) という。その中で、転写制御因子はDNAに結合し、転写を調節するタンパク質であり、転写を促進するものを転写活性化因子、抑えるものを転写抑制因子という[1]。一方、DNAに直接結合せず、転写制御因子とRNAポリメラーゼなどとの仲介役として転写を調節するタンパク質複合体はメディエーターである。メディエーターの機能は多岐に渡り、転写を活性化するコアクチベーターや、転写を抑制するコリプレッサーとして働く[6][7]。メディエーター以外にも、コアクチベーターやコリプレッサーと呼ばれる因子があり、ヒストンのアセチル化やメチル化などを介して、クロマチンの状態を制御する[7][8]。DNA上で転写の活性化に働く領域はエンハンサー、転写を抑制する領域がサイレンサーであり、それぞれ複数の転写制御因子が結合することが多い[9][10][11]。
転写制御機構
in vitroの解析により、転写は TATAボックスなどのコアプロモーター上でTATA結合タンパク質 (TATA-binding protein; TBP) やTAFs (TBP-associated factors) などの基本転写因子がRNAポリメラーゼと転写開始前複合体を形成することにより開始されることが示されている[6][9][12]。これらの因子のみによる転写は基本転写と呼ばれる。生体内では、様々な転写制御因子が働き、その組み合わせにより遺伝子の転写量が組織や発生の時期で異なる[2][3][11]。
転写活性化の機構
転写の活性化は、転写の開始と伸長で起こる。活性化の例として、エンハンサーに結合した転写活性化因子が基本転写因子と相互作用し、プロモーターにおける転写開始前複合体の形成を促進することが知られている[1][9][12]。一方、メディエーターは、転写制御因子の転写活性化ドメインとRNAポリメラーゼの双方に結合し、転写開始前複合体の形成を促し、コアクチベーターとして働く[6][7][13] (図1)。
真核細胞のDNAはヒストンに巻きつき、ヌクレオソームを形成する。ヌクレオソーム構造を弛緩させ、転写制御因子などをDNAに作用しやすくし、転写を活性化する因子もコアクチベーターと呼ばれる。コアクチベーターのCBP (CREB-binding protein) は、ヒストンをアセチル化し、ヒストンとDNA間の親和性を弱める[8]。ヒストンリモデリング因子のSWI/SNF(switch/sucrose nonfermentable) は、ATPの加水分解で生じるエネルギーを用いてクロマチン構造を変化させる[14]。
RNAポリメラーゼは数十塩基程度の短いRNAを転写後、DSIF (DRB-sensitivity inducing factor) やNELF (negative elongation factor) などの伸長阻害因子の働きにより転写を休止する[15]。Mycなどの転写制御因子は、プロテインキナーゼP-TEFb (positive transcription elongation factor b) の作用を介して転写の休止を解除し、RNA伸長を再開させることが知られている[15]。
転写抑制の機構
転写抑制では、サイレンサーに結合した転写抑制因子がTLE/Grg (transducin-like Enhancer of split/groucho-related gene) やNcoR / SMRT (nuclear receptor corepressor/silencing mediator for retinoic and thyroid receptors) などのコリプレッサーを介してヒストンデアセチラーゼを引き寄せ、ヒストンのアセチル基を除去することによりヌクレオソームを安定化させ遺伝子を不活化する[16][17]。また、ヒストンメチルトランスフェラーゼを引き寄せ、ヒストンの特定の部位をメチル化し、転写が起きないクロマチン状態を維持することにより抑制する場合もある[7][8]。
転写抑制因子のNRSF/REST (neuron-restrictive silencer factor/RE1-silencing transcription factor) は、サイレンサー中の塩基配列NRSE/RE1 (neural restrictive silencer element/repressor element 1) に結合し、コリプレッサーを介したヒストンデアセチラーゼやヒストンメチルトランスフェラーゼの作用により、神経細胞で働くSCG10 (superior cervical ganglia 10) などの遺伝子の転写を非神経細胞で抑える[18]。
CDK8 (Cyclin dependent kinase 8) などのコリプレッサーは、転写制御因子をリン酸化し分解を促進したり、基本転写因子をリン酸化し転写開始前複合体の形成を阻害する[7][13]。
転写制御配列
転写制御因子のDNA結合ドメインは、多くの場合、4から10塩基の特定の配列(コンセンサス配列)に結合する。エンハンサーやサイレンサーには、通常、転写制御因子の結合する場所(転写制御因子結合部位)がいくつかあり、複数種の転写制御因子で転写が調節される[10][11]。 遺伝子の転写が複数のエンハンサーで調節されることも多く、発生段階に特異的なエンハンサーと組織特異的なエンハンサーなどに分離できることもある。 DNA配列における転写制御因子結合部位は、MatInspector (http://www.genomatix.de/index.html) や、PAZAR (http://www.pazar.info/) などで予測できる。
ENCODEプロジェクトなどにより、ゲノムのかなりの領域が転写されることが明らかとなった[19]。エンハンサー部位で合成されるeRNA (enhancer RNA) などの非翻訳RNAが転写に関わることが示唆されている[20][21]。
構造
転写活性化因子の転写活性化ドメインは、コアクチベーターなどのタンパク質と結合し、酸性アミノ酸、もしくはグルタミン酸、プロリンのいずれかに富む領域などに分類される。転写抑制因子の転写抑制ドメインには、トリプトファン、アルギニン、プロリンからなるWRPWドメインや、芳香族アミノ酸と疎水性アミノ酸からなるEH1 (engrailed homology 1) ドメインなどがあり、コリプレッサーとの結合に必須である[22][23]。ジンクフィンガー型因子などは、リガンドと結合するドメインも有する。
転写制御因子の構造については、Protein Data Bank (http://www.rcsb.org/pdb/home/home.do) やPfam (http://pfam.sanger.ac.uk/) などのデータベースで検索できる。
DNA結合ドメインによる分類
転写制御因子は、DNA結合ドメインの構造モチーフに基づき、ホメオドメイン、ジンクフィンガー、塩基性へリックス・ループ・へリックスなどのファミリーに分けられる。
ホメオドメイン(homeodomain)
ホメオボックスがコードする約60個のアミノ酸配列である。ショウジョウバエの体節形成の研究で発見され、ヒトを含む高等動物までホモログ間でよく保存されている[24]。へリックス・ターン・へリックス構造をとり、2番目のヘリックスがDNAの主溝に入り込んで結合する。アミノ酸配列の類似性やホメオドメインの外のモチーフから、さらにサブファミリーに分けられる。ホメオドメインが結合するコンセンサス配列の代表としてATTAが知られ、サブファミリーにより認識配列が異なる[25]。
ジンクフィンガー(zinc finger)
GATAファミリーの因子などに見られる構造で、亜鉛イオンに2個のシステインと2個のヒスチジンが配位結合するC2H2タイプや、4個のシステインが配位結合するC4タイプなどがある[26]。亜鉛に配位したシステインあるいはヒスチジン残基に挟まれたアミノ酸領域が指状のループをつくる。ステロイドホルモン受容体などの核内受容体は、リガンドと直接結合すると核内に入り転写制御能を発揮するようになる[27][28]。コンセンサス配列にはGATAファミリーの因子が結合するGATAなどがある。
塩基性へリックス・ループ・へリックス(basic helix-loop-helix、bHLH)
Mash1/Ascl1 (mammalian achaete-scute homolog 1/ achaete-scute complex homolog 1)やMath1/ Atoh1 (mammalian atonal homolog 1/ atonal homolog 1) などの神経分化を開始させるプロニューラル因子などに見られる[29]。E12/Tcf3 (transcription factor 3) タンパク質などのへリックス・ループ・へリックスドメインとヘテロ二量体を形成し、DNAに結合する。コンセンサス配列として、プロニューラル因子などの転写活性化因子が結合するE box (CANNTG) と、Hes1 (hairy, Enhancer of split 1) などの転写抑制因子が結合するN box (CACNAG) などが知られている[30]。
ロイシンジッパー(leucine zipper)
c-FosやMycなどに見られ、7アミノ酸ごとにロイシンが配置されたコイルドコイルと呼ばれる構造を取る。同様の構造を持つ因子とコイルドコイル間でヘテロ二量体を形成し、コイルドコイルのN端側に存在する塩基性アミノ酸に富む領域でDNAに結合する。コンセンサス配列にはCREB (cAMP-responsive element-binding protein) が結合するサイクリックAMP応答配列(TGACGTCA)などがある[31]。
HMG(high mobility group)ボックス
クロマチンから0.35M塩化ナトリウムによって抽出され、電気泳動で高い移動度を示すタンパク質のDNA結合領域で見つかった。約80アミノ酸からなり、三つのαへリックスを形成する[32]。特に、性決定遺伝子Sry がコードするタンパク質と高い相同性を持つ因子はSoxファミリーと呼ばれる。コンセンサス配列にはSoxファミリーの結合するT(A/T)(A/T)CAAGなどがある。
| DNA結合ドメイン | ファミリー | 例 |
|---|---|---|
| homeodomain | Hox | HoxA1, HoxC8 |
| Pax | Pax2, Pax6 | |
| Dlx | Dlx1, Dlx2 | |
| Emx | Emx2 | |
| Nkx | Nkx2.2, Nkx6.1 | |
| En | En1 | |
| Bar | Barh1, Barh2 | |
| paired-like | Phox2a, DRG11 | |
| POU | Brn3a | |
| zinc finger | C2H2 | Gli1, NRSF/REST, Zif268, Krox20, Fezl |
| kruppel-like | Klf4 | |
| Gata | Gata2, Gata3 | |
| nuclear hormone receptor | RARα1, RXR | |
| basic helix-loop-helix | bHLH | Mash1/Ascl1, Math1/Atoh1, Neurogenin1, NeuroD, Ptf1a, Hes1, Olig2, Scl/Tal1, E12/Tcf3 |
| bHLH-PAS | Per1, Clk | |
| bHLH-leucine zipper | Myc, MycN/N-Myc | |
| leucine zipper | c-Fos, CREB | |
| HMG box | Sox | Sox2, Sox9, Sox10 |
| Tcf/Lef | Tcf1/Lef1 | |
| Ets | Pea3, Er81 | |
| T box | Tbr1 | |
| forkhead | Fox | FoxG1/Bf1, FoxO, FoxP2 |
| Rel homology | Rbpj /CBF1, NF-κB | |
| MH1 | Smad | Smad2, Smad4 |
| SH2 | STAT | STAT3 |
転写制御因子の活性制御
転写制御因子の活性は、細胞の分化段階や細胞外からの刺激などにより制御され、リン酸化やリガンド結合、ユビキチン化などの影響を受ける[2]。また、転写制御因子には、構造的に類似した因子同士で二量体を形成し機能するものも多い。へリックス・ループ・へリックス因子の一つであるIdファミリーは塩基性領域を欠き、塩基性へリックス・ループ・へリックス因子のE12/Tcf3などとへテロ二量体を形成し、E12/Tcf3がDNAに結合することを阻害する[33]。
TGF-β (transforming growth factor-β) ファミリーの分泌タンパク質が膜受容体に結合すると、MH1 (Mad homology 1) ドメイン型因子のSmad2とSmad3はリン酸化され、Smad4とヘテロ二量体を形成後、核に移行して転写を制御する[34]。
インターフェロンなどのサイトカインやホルモンが受容体に結合すると、Jak (Janus kinase)によってSH2 (Src homology 2) ドメイン型因子のシグナル伝達因子件転写活性化因子 (signal transducers and activator of transcription; STAT) がリン酸化される。リン酸化されたSTATはSH2ドメインを介して二量体を形成し、核に移行する[35]。
Hedgehogが膜上の受容体Patchedに結合すると、膜タンパク質Smoothendを介してジンクフィンガー型因子のGliが活性化される。活性化したGliは核に移行し、標的遺伝子の転写を制御する[36]。
DeltaやJaggedが受容体Notchに結合すると、γセクレターゼの作用によりNotch受容体の細胞内ドメインが切り出される。Notchの細胞内ドメインは核内に移行し、Rel homology型因子のRbpj/CBF-1 (recombination signal sequence-binding protein J/C promoter-binding factor 1) やコアクチベーターMaml1 (Mastermind-like 1) と複合体を形成しHes1などの転写を活性化する[37]。
Wntが膜受容体Frizzledと結合するとβ-cateninが遊離され、このβ-cateninはHMGボックス型因子のTcf1/Lef1 (T-cell factor 1/lymphoid enhancer binding factor 1) とヘテロ二量体を形成後、核内に移行し転写を制御する[38][39]。
ロイシンジッパー型因子のCREBは、サイクリックAMP 濃度の上昇などの様々な刺激によりサイクリックAMP依存性プロテインキナーゼやカルシウムカルモジュリン依存性プロテインキナーゼなどによるリン酸化を受け、活性化する[31][40]。
Pax6 (paired box 6) やMycN/N-mycなどの転写制御因子はユビキチン化され、ユビキチン・プロテアソーム系によって分解されることが明らかとなっている[41]。
生体内での役割
発生・細胞分化
発生過程では、多数の転写制御因子が働く。神経発生の初期においては、Pax6やEmx2 (empty spiracles homeobox 2)、Nkx6.1 (NK6 homeobox 6.1) などのホメオドメイン型因子が神経管の前後軸や背腹軸に沿って特異的に発現し、終脳形成を制御する[42]。Hoxファミリーのタンパク質は、後脳と脊髄の前後軸沿いの特定の領域の個性に関わり、運動神経細胞などの分化を正常に進めるために必須である[43]。
Mash1/Ascl1 やMath1/Atoh1 などのプロニューラル因子は神経分化の開始を司り、神経細胞の分化や個性を制御する遺伝子の転写を活性化する[29]。神経幹細胞や神経前駆細胞ではNotchがHes1やHes5を活性化し、Hes1やHes5タンパク質がプロニューラル遺伝子の上流などに結合し、転写を抑える[30]。
Math1はBar型ホメオボックス遺伝子Barh1 (Bar-class homeobox 1) とBarh2 を直接活性化し、Barh1とBarh2タンパク質が脊髄の交連神経細胞の個性を決定する[44][45]。小脳ではMath1/Atoh1は、グルタミン酸作動性の顆粒細胞の分化を促進する一方、GABA (γ-aminobutyric acid) 作動性神経細胞産生に必須なPtf1a (pancreas transcription factor 1a) の発現を抑える[46]。
脊髄において、塩基性へリックス・ループ・へリックス型因子のOlig2は運動神経細胞の分化とオリゴデンドロサイトの産生に必須な一方、Scl/Tal1は介在神経細胞の分化とアストロサイトの産生を制御する[47]。
ホメオボックス遺伝子のDlx1 (distal-less homeobox 1) とDlx2 は、大脳基底核原基由来の細胞で発現し、大脳のGABA作動性神経細胞の産生に必須である[48]。
HMGボックス型因子のSox2は、プロニューラル因子と拮抗的に働き、神経分化を抑制する[9]。また、Sox9やSox10はオリゴデンドロサイトの前駆細胞で発現し、オリゴデンドロサイトの分化を促進するとともに、ミエリン塩基性タンパク質などの遺伝子の転写を活性化する[9]。
軸索伸長、細胞極性
プロニューラル因子のNeurogenin2は、神経細胞の移動や軸索投射にも関与する[49][50]。
脊髄交連神経の軸索投射は、Bar型やLIM型 のホメオドメイン型因子によって制御される。Barh1 とBarh2の下流では、Lhx2 (LIM homeobox 2) を介したRig1/Robo3 (Rb-inhibiting gene 1/roundabout homolog 3) の転写調節とともに、Lhx2を介さないNeuropilin2の転写調節が行われる[45]。
運動神経や感覚神経では、軸索が標的筋肉の近傍に到着すると、Pea3 (polyomavirus enhancer activator 3) やEr81 (ets-related protein 81) などのEtsファミリー遺伝子の転写が誘導され、軸索の枝分かれが制御される[43]。また、フォークヘッド型因子のFoxO (forkhead box O) は、Pak1(p21 protein-activated kinase 1) などの細胞極性を制御する遺伝子の転写を調節し、神経細胞の形態制御に関わる[51]。
高次機能
CREBは、神経活動で活性化されシナプスの構造を制御する遺伝子の転写を調節し、長期記憶の形成に関与する[31][40]。
Rel homology型因子のNF-κB (nuclear factor-κB) は、発生過程において神経軸索の伸長や樹状突起の枝分かれなど神経突起の発達に重要な役割を果たす一方、成体では樹状突起のスパイン数やシナプス形成などを介して学習や記憶に関わることが示唆されている[40][52]。
ロイシンジッパー型因子のc-Fosやジンクフィンガー型因子のZif268は、神経活動などの刺激により一過的に誘導される最初期遺伝子であり、神経回路の可塑的変化への関与が示唆されている[40]。
フォークヘッド型因子のFoxP2 (forkhead box P2)は、発声や言語発達に関わることが示唆されている[53]。
環境への応答
低酸素状態では、塩基性へリックス・ループ・へリックス型因子のHIF-1α (hypoxia inducible factor 1 α) が安定化され、HIF-1βとヘテロ二量体を形成して血管新生や解糖系に関わる遺伝子群の転写を活性化する[54]。
時計遺伝子Periodは塩基性へリックス・ループ・へリックス型因子をコードし、光刺激により誘導される。Periodの転写は他の塩基性へリックス・ループ・へリックス型因子のClockとBmal1 (brain and muscle Arnt-like 1) により調節されており、これらの因子がネガティブフィードバックループを形成し、約24時間周期で発現が変動する[55]。
病理
ヒトの疾患の原因となる転写制御因子の変異はTRANSFACやOMIM Morbid Map (http://www.omim.org/search/advanced/geneMap) などのデータベースなどで調べられる。
転写制御因子データベース
転写制御因子のリストはTRANSFACデータベース(http://www.gene-regulation.com) から入手可能である。また、転写制御因子についてのレビューもデータベース化されている (Transcription Factor Encyclopedia、 http://www.cisreg.ca/cgi-bin/tfe/home.pl) [56]。
関連項目
- ↑ 1.0 1.1 1.2 1.3 Tetsuichiro Saito
Transcription factor. Encyclopedia of Neuroscience
Springer-Verlag Gmbh Berlin Heidelberg: 2009 - ↑ 2.0 2.1 2.2
Brivanlou, A.H., & Darnell, J.E. (2002).
Signal transduction and the control of gene expression. Science (New York, N.Y.), 295(5556), 813-8. [PubMed:11823631] [WorldCat] [DOI] - ↑ 3.0 3.1
Lee, T.I., & Young, R.A. (2013).
Transcriptional regulation and its misregulation in disease. Cell, 152(6), 1237-51. [PubMed:23498934] [PMC] [WorldCat] [DOI] - ↑
Babu, M.M., Luscombe, N.M., Aravind, L., Gerstein, M., & Teichmann, S.A. (2004).
Structure and evolution of transcriptional regulatory networks. Current opinion in structural biology, 14(3), 283-91. [PubMed:15193307] [WorldCat] [DOI] - ↑
Vaquerizas, J.M., Kummerfeld, S.K., Teichmann, S.A., & Luscombe, N.M. (2009).
A census of human transcription factors: function, expression and evolution. Nature reviews. Genetics, 10(4), 252-63. [PubMed:19274049] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2
Roeder, R.G. (2005).
Transcriptional regulation and the role of diverse coactivators in animal cells. FEBS letters, 579(4), 909-15. [PubMed:15680973] [WorldCat] [DOI] - ↑ 7.0 7.1 7.2 7.3 7.4
Malik, S., & Roeder, R.G. (2010).
The metazoan Mediator co-activator complex as an integrative hub for transcriptional regulation. Nature reviews. Genetics, 11(11), 761-72. [PubMed:20940737] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1 8.2
Bannister, A.J., & Kouzarides, T. (2011).
Regulation of chromatin by histone modifications. Cell research, 21(3), 381-95. [PubMed:21321607] [PMC] [WorldCat] [DOI] - ↑ 9.0 9.1 9.2 9.3 9.4
Levine, M., & Tjian, R. (2003).
Transcription regulation and animal diversity. Nature, 424(6945), 147-51. [PubMed:12853946] [WorldCat] [DOI] 引用エラー: 無効な<ref>タグ; name "ref15"が異なる内容で複数回定義されています - ↑ 10.0 10.1
Ong, C.T., & Corces, V.G. (2011).
Enhancer function: new insights into the regulation of tissue-specific gene expression. Nature reviews. Genetics, 12(4), 283-93. [PubMed:21358745] [PMC] [WorldCat] [DOI] - ↑ 11.0 11.1 11.2
Spitz, F., & Furlong, E.E. (2012).
Transcription factors: from enhancer binding to developmental control. Nature reviews. Genetics, 13(9), 613-26. [PubMed:22868264] [WorldCat] [DOI] - ↑ 12.0 12.1
Goodrich, J.A., & Tjian, R. (2010).
Unexpected roles for core promoter recognition factors in cell-type-specific transcription and gene regulation. Nature reviews. Genetics, 11(8), 549-58. [PubMed:20628347] [PMC] [WorldCat] [DOI] - ↑ 13.0 13.1
Taatjes, D.J. (2010).
The human Mediator complex: a versatile, genome-wide regulator of transcription. Trends in biochemical sciences, 35(6), 315-22. [PubMed:20299225] [PMC] [WorldCat] [DOI] - ↑
Hargreaves, D.C., & Crabtree, G.R. (2011).
ATP-dependent chromatin remodeling: genetics, genomics and mechanisms. Cell research, 21(3), 396-420. [PubMed:21358755] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1
Adelman, K., & Lis, J.T. (2012).
Promoter-proximal pausing of RNA polymerase II: emerging roles in metazoans. Nature reviews. Genetics, 13(10), 720-31. [PubMed:22986266] [PMC] [WorldCat] [DOI] - ↑
Cinnamon, E., & Paroush, Z. (2008).
Context-dependent regulation of Groucho/TLE-mediated repression. Current opinion in genetics & development, 18(5), 435-40. [PubMed:18721877] [WorldCat] [DOI] - ↑
Hayakawa, T., & Nakayama, J. (2011).
Physiological roles of class I HDAC complex and histone demethylase. Journal of biomedicine & biotechnology, 2011, 129383. [PubMed:21049000] [PMC] [WorldCat] [DOI] - ↑
Ballas, N., & Mandel, G. (2005).
The many faces of REST oversee epigenetic programming of neuronal genes. Current opinion in neurobiology, 15(5), 500-6. [PubMed:16150588] [WorldCat] [DOI] - ↑
ENCODE Project Consortium (2012).
An integrated encyclopedia of DNA elements in the human genome. Nature, 489(7414), 57-74. [PubMed:22955616] [PMC] [WorldCat] [DOI] - ↑
Melo, C.A., Drost, J., Wijchers, P.J., van de Werken, H., de Wit, E., Oude Vrielink, J.A., ..., & Agami, R. (2013).
eRNAs are required for p53-dependent enhancer activity and gene transcription. Molecular cell, 49(3), 524-35. [PubMed:23273978] [WorldCat] [DOI] - ↑
Ørom, U.A., Derrien, T., Beringer, M., Gumireddy, K., Gardini, A., Bussotti, G., ..., & Shiekhattar, R. (2010).
Long noncoding RNAs with enhancer-like function in human cells. Cell, 143(1), 46-58. [PubMed:20887892] [PMC] [WorldCat] [DOI] - ↑
Jennings, B.H., & Ish-Horowicz, D. (2008).
The Groucho/TLE/Grg family of transcriptional co-repressors. Genome biology, 9(1), 205. [PubMed:18254933] [PMC] [WorldCat] [DOI] - ↑
Copley, R.R. (2005).
The EH1 motif in metazoan transcription factors. BMC genomics, 6, 169. [PubMed:16309560] [PMC] [WorldCat] [DOI] - ↑
Banerjee-Basu, S., & Baxevanis, A.D. (2001).
Molecular evolution of the homeodomain family of transcription factors. Nucleic acids research, 29(15), 3258-69. [PubMed:11470884] [PMC] [WorldCat] [DOI] - ↑
Berger, M.F., Badis, G., Gehrke, A.R., Talukder, S., Philippakis, A.A., Peña-Castillo, L., ..., & Hughes, T.R. (2008).
Variation in homeodomain DNA binding revealed by high-resolution analysis of sequence preferences. Cell, 133(7), 1266-76. [PubMed:18585359] [PMC] [WorldCat] [DOI] - ↑
Brayer, K.J., & Segal, D.J. (2008).
Keep your fingers off my DNA: protein-protein interactions mediated by C2H2 zinc finger domains. Cell biochemistry and biophysics, 50(3), 111-31. [PubMed:18253864] [WorldCat] [DOI] - ↑
McKenna, N.J., & O'Malley, B.W. (2010).
SnapShot: Nuclear receptors I. Cell, 142(5), 822-822.e1. [PubMed:20813267] [WorldCat] [DOI] - ↑
McKenna, N.J., & O'Malley, B.W. (2010).
SnapShot: Nuclear receptors II. Cell, 142(6), 986.e1. [PubMed:20850017] [WorldCat] [DOI] - ↑ 29.0 29.1
Bertrand, N., Castro, D.S., & Guillemot, F. (2002).
Proneural genes and the specification of neural cell types. Nature reviews. Neuroscience, 3(7), 517-30. [PubMed:12094208] [WorldCat] [DOI] - ↑ 30.0 30.1
Kageyama, R., Ohtsuka, T., & Kobayashi, T. (2007).
The Hes gene family: repressors and oscillators that orchestrate embryogenesis. Development (Cambridge, England), 134(7), 1243-51. [PubMed:17329370] [WorldCat] [DOI] - ↑ 31.0 31.1 31.2
Benito, E., & Barco, A. (2010).
CREB's control of intrinsic and synaptic plasticity: implications for CREB-dependent memory models. Trends in neurosciences, 33(5), 230-40. [PubMed:20223527] [WorldCat] [DOI] - ↑
Malarkey, C.S., & Churchill, M.E. (2012).
The high mobility group box: the ultimate utility player of a cell. Trends in biochemical sciences, 37(12), 553-62. [PubMed:23153957] [PMC] [WorldCat] [DOI] - ↑
Yokota, Y., & Mori, S. (2002).
Role of Id family proteins in growth control. Journal of cellular physiology, 190(1), 21-8. [PubMed:11807807] [WorldCat] [DOI] - ↑
Dutko, J.A., & Mullins, M.C. (2011).
SnapShot: BMP signaling in development. Cell, 145(4), 636, 636.e1-2. [PubMed:21565618] [WorldCat] [DOI] - ↑
Nicolas, C.S., Amici, M., Bortolotto, Z.A., Doherty, A., Csaba, Z., Fafouri, A., ..., & Peineau, S. (2013).
The role of JAK-STAT signaling within the CNS. JAK-STAT, 2(1), e22925. [PubMed:24058789] [PMC] [WorldCat] [DOI] - ↑
Dessaud, E., McMahon, A.P., & Briscoe, J. (2008).
Pattern formation in the vertebrate neural tube: a sonic hedgehog morphogen-regulated transcriptional network. Development (Cambridge, England), 135(15), 2489-503. [PubMed:18621990] [WorldCat] [DOI] - ↑
Ables, J.L., Breunig, J.J., Eisch, A.J., & Rakic, P. (2011).
Not(ch) just development: Notch signalling in the adult brain. Nature reviews. Neuroscience, 12(5), 269-83. [PubMed:21505516] [PMC] [WorldCat] [DOI] - ↑
Avilés, E.C., Wilson, N.H., & Stoeckli, E.T. (2013).
Sonic hedgehog and Wnt: antagonists in morphogenesis but collaborators in axon guidance. Frontiers in cellular neuroscience, 7, 86. [PubMed:23772206] [PMC] [WorldCat] [DOI] - ↑
Macdonald, B.T., Semenov, M.V., & He, X. (2007).
SnapShot: Wnt/beta-catenin signaling. Cell, 131(6), 1204. [PubMed:18083108] [WorldCat] [DOI] - ↑ 40.0 40.1 40.2 40.3
Alberini, C.M. (2009).
Transcription factors in long-term memory and synaptic plasticity. Physiological reviews, 89(1), 121-45. [PubMed:19126756] [PMC] [WorldCat] [DOI] - ↑
Kageyama, R., Ohtsuka, T., & Kobayashi, T. (2008).
Roles of Hes genes in neural development. Development, growth & differentiation, 50 Suppl 1, S97-103. [PubMed:18430159] [WorldCat] [DOI] - ↑
Hébert, J.M., & Fishell, G. (2008).
The genetics of early telencephalon patterning: some assembly required. Nature reviews. Neuroscience, 9(9), 678-85. [PubMed:19143049] [PMC] [WorldCat] [DOI] - ↑ 43.0 43.1
Dalla Torre di Sanguinetto, S.A., Dasen, J.S., & Arber, S. (2008).
Transcriptional mechanisms controlling motor neuron diversity and connectivity. Current opinion in neurobiology, 18(1), 36-43. [PubMed:18524570] [WorldCat] [DOI] - ↑
Saba, R., Johnson, J.E., & Saito, T. (2005).
Commissural neuron identity is specified by a homeodomain protein, Mbh1, that is directly downstream of Math1. Development (Cambridge, England), 132(9), 2147-55. [PubMed:15788459] [WorldCat] [DOI] - ↑ 45.0 45.1
Kawauchi, D., Muroyama, Y., Sato, T., & Saito, T. (2010).
Expression of major guidance receptors is differentially regulated in spinal commissural neurons transfated by mammalian Barh genes. Developmental biology, 344(2), 1026-34. [PubMed:20599893] [WorldCat] [DOI] - ↑
Kawauchi, D., & Saito, T. (2008).
Transcriptional cascade from Math1 to Mbh1 and Mbh2 is required for cerebellar granule cell differentiation. Developmental biology, 322(2), 345-54. [PubMed:18723012] [WorldCat] [DOI] - ↑
Rowitch, D.H., & Kriegstein, A.R. (2010).
Developmental genetics of vertebrate glial-cell specification. Nature, 468(7321), 214-22. [PubMed:21068830] [WorldCat] [DOI] - ↑
Chédotal, A., & Rijli, F.M. (2009).
Transcriptional regulation of tangential neuronal migration in the developing forebrain. Current opinion in neurobiology, 19(2), 139-45. [PubMed:19428236] [WorldCat] [DOI] - ↑
Hand, R., Bortone, D., Mattar, P., Nguyen, L., Heng, J.I., Guerrier, S., ..., & Polleux, F. (2005).
Phosphorylation of Neurogenin2 specifies the migration properties and the dendritic morphology of pyramidal neurons in the neocortex. Neuron, 48(1), 45-62. [PubMed:16202708] [WorldCat] [DOI] - ↑
Hand, R., & Polleux, F. (2011).
Neurogenin2 regulates the initial axon guidance of cortical pyramidal neurons projecting medially to the corpus callosum. Neural development, 6, 30. [PubMed:21864333] [PMC] [WorldCat] [DOI] - ↑
de la Torre-Ubieta, L., & Bonni, A. (2011).
Transcriptional regulation of neuronal polarity and morphogenesis in the mammalian brain. Neuron, 72(1), 22-40. [PubMed:21982366] [PMC] [WorldCat] [DOI] - ↑
Gutierrez, H., & Davies, A.M. (2011).
Regulation of neural process growth, elaboration and structural plasticity by NF-κB. Trends in neurosciences, 34(6), 316-25. [PubMed:21459462] [PMC] [WorldCat] [DOI] - ↑
Kang, C., & Drayna, D. (2011).
Genetics of speech and language disorders. Annual review of genomics and human genetics, 12, 145-64. [PubMed:21663442] [WorldCat] [DOI] - ↑
Kewley, R.J., Whitelaw, M.L., & Chapman-Smith, A. (2004).
The mammalian basic helix-loop-helix/PAS family of transcriptional regulators. The international journal of biochemistry & cell biology, 36(2), 189-204. [PubMed:14643885] [WorldCat] [DOI] - ↑
Mohawk, J.A., Green, C.B., & Takahashi, J.S. (2012).
Central and peripheral circadian clocks in mammals. Annual review of neuroscience, 35, 445-62. [PubMed:22483041] [PMC] [WorldCat] [DOI] - ↑
Yusuf, D., Butland, S.L., Swanson, M.I., Bolotin, E., Ticoll, A., Cheung, W.A., ..., & Wasserman, W.W. (2012).
The transcription factor encyclopedia. Genome biology, 13(3), R24. [PubMed:22458515] [PMC] [WorldCat] [DOI]
(執筆担当者: 室山優子、斎藤哲一郎 担当編集委員: 大隅典子)