Activity-regulated cytoskeleton-associated protein
比嘉なつみ、奥野 浩行
鹿児島大学 大学院医歯学総合研究科
DOI:10.14931/bsd.10448 原稿受付日:2023年6月28日 原稿完成日:2023年7月19日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
同義語:Activity-regulated gene 3.1 protein homolog
英略称:Arc
Activity-regulated cytoskeleton-associated protein、別名Activity-regulated gene 3.1 protein homolog (Arc/Arg3.1)は神経細胞に特異的に発現する最初期遺伝子のひとつである。特に、大脳皮質や海馬の興奮性細胞において神経活動による一過的な発現誘導がみられるため、c-fos等と同様に神経活動履歴の分子マーカーとして汎用されている。Arcの遺伝子産物はAMPA型グルタミン酸受容体の膜表面発現の制御に関わり、シナプス可塑性や学習・記憶への関与が示唆されている。
発見の経緯
Arc遺伝子は1995年、米国・ジョンズホプキンス大のPaul Worley博士らの研究グループによって、電気痙攣刺激を行ったラット海馬cDNAから単離された[1] 。mRNA配列から予想されるアミノ酸配列は細胞骨格タンパク質であるαスペクトリンと部分的に相同性があり、また、リコンビナントタンパク質はF-アクチンと共沈降したことから、単離された遺伝子産物はactivity-regulated cytoskeleton-associated protein (Arc)と命名された。また、同じく1995年、Worley研とは独立してドイツ・ハンブルグ大のDietmar Kuhl博士らの研究グループは、痙攣誘発剤ペンチレンテトラゾール (Pentylenetetrazole, PTZ)刺激を行ったラットから調製した海馬cDNAを用いたライブラリーから活動依存的に発現上昇する全長約3 kbのmRNAを同定し、この遺伝子をarg3.1と命名した[2] 。Arcとarg3.1は同一の最初期遺伝子である。
Arc遺伝子は活動依存的遺伝子の中でも特に長期増強現象 (long-term potentiation, LTP)誘発刺激による発現上昇率が高いことなどから長期シナプス可塑性との関連が示唆されていたが、mRNAの一部は樹状突起に輸送され局所翻訳されることが明らかになり、さらなる注目を集めた[3] 。大脳皮質や海馬の神経細胞では、感覚刺激や新規環境暴露などの生理的刺激によって素早く一過的なmRNA発現誘導がみられ、c-fosやnpas4、egr1などと並んで神経活動分子マーカーとして広く使用されている。
構造
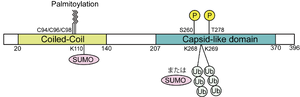
哺乳類のArcタンパク質は約400個のアミノ酸からなる(図1)。酵素活性領域などの既知の機能ドメインを持たず、他のタンパク質との直接的あるいは非直接的な相互作用の場を提供する“Hubタンパク質”であることが提唱されている[4] 。N末側にはArcタンパク質の多量体化や他のタンパク質との相互作用に関わるコイルド・コイル構造をもつ[5][6] 。また、近年、結晶構造解析の結果から、Arcタンパク質のC末側(従来、スペクトリン相同領域と報告されていた領域付近)には、レトロウイルスhuman immunodeficiency virus (HIV)やレトロトランスポゾンTy3/gypsyのgagタンパク質の一部(カプシド部)と高い構造上の相同性があることが明らかになった[7][8] 。
神経細胞で活動依存的に発現誘導されたArcタンパク質は細胞内で速やかにユビキチンプロテアソーム系により分解される[9] 。Arcタンパク質はユビキチン化以外にもSUMO化、リン酸化、パルミトイル化などの多様な翻訳後修飾を受けることが示されており、これらの修飾により細胞内局在や機能が調節されていると考えられるが詳細は不明である。
Arc遺伝子の起源とウイルス様性質
進化的な観点からみると、脊椎動物において四肢動物(両生類、爬虫類、鳥類、哺乳類)では上記のN末側コイルド・コイルドメインとc末側GAGカプシドドメインの両構造を持つArc遺伝子オーソログが確認できるが、魚類では同様の構造をもつ遺伝子は確認できない。一方、魚類のゲノムにはTy3/Gypsyレトロトランスポゾンファミリー遺伝子が検出されるが、哺乳類ではこのレトロトランスポゾンは検出されない。Ty3/Gypsyレトロトランスポゾンはレトロウイルスと同じ祖先をもつと考えられており、gag(カプシド、ヌクレオカプシド)とpol(プロテアーゼ、逆転写酵素、インテグラーゼ)をコードする。一部の魚類のTy3/Gypsyレトロトランスポゾン遺伝子ではN末側にコイルド・コイルドメインが挿入されていることから、脊椎動物のArc遺伝子はTy3/Gypsyレトロトランスポゾンに遺伝子の構造変化が起こり、内在性遺伝子への”順化(domestication)’の結果、生じたものであると考えられる[8] 。
興味深いことに、昆虫類でもTy3/Gypsyレトロトランスポゾン由来遺伝子は存在し、一部の種においてはpol領域を欠損してgag領域のみが遺伝子として存在している。ショウジョウバエのArcホモローグ(dArc1, dArc2)はそのような遺伝子の例であり[10] 、これらは脊椎動物のArcとは独立して進化してきたものであると考えられる。
哺乳類のArcタンパク質は多量体を形成することが生化学的に示されており、さらに電子顕微鏡観察からカプシド多量体様の球形構造を形成することが明らかになっている[6][7] 。また、ショウジョウバエdArcも多量体化し、球形カプシド様構造を作る[10] 。レトロウイルスの球状カプシド構造がウイルスゲノムを内包するのと同様、Arcタンパク質によって形成されるカプシド様構造はArc mRNAを内包する[10][7] 。また、レトロウイルスのカプシド構造は細胞膜に包まれた後、細胞外小胞として放出されるが、Arcカプシド様構造体も細胞外に放出される[7] 。これらの知見はArcがレトロトランスポゾンを起源とする“内在性レトロウイルス遺伝子”であるという仮説を支持し、“Arc含有カプシド様構造を含む細胞外小胞は細胞間でArc mRNAを伝達する”という新たな可能性が提唱されている[11][7] 。
サブファミリー
哺乳動物においてはゲノム中にArcと相同性の高い遺伝子は他に存在しないユニークな遺伝子である。魚類においてはTy3/Gypsyトランスポゾン遺伝子群のgag-capsid領域と哺乳類ArcのC末側カプシド様領域との間には相同性があり、広義のファミリー遺伝子と考えることができる。ショウジョウバエではdArc1とdArc2の2種のファミリー遺伝子が存在する。
発現
組織分布
成熟マウスにおける組織毎の網羅的遺伝子発現解析では、Arc mRNAは主に脳と下垂体において高発現しているが、副腎や前立腺、精巣や脂肪組織にもある程度のレベルで発現している。脳におけるArc発現は大脳皮質や海馬、線条体で高く、視床や間脳、小脳においては相対的に低い(Allen Brain Atlas)。大脳皮質や海馬におけるArcは主に興奮性神経細胞に発現しており、抑制性神経細胞での発現は低い。一方、線条体においては抑制性神経細胞である中型有棘神経細胞がArcを発現している。アストロサイトにおける発現も報告されている。Arc発現は発達過程によっても変化することが報告されており、大脳皮質においてArc発現は、生後から成熟するにつれて増加し、その後加齢に伴い減少する[12][1] 。
神経系以外の組織におけるArc発現の発達にともなう変化や細胞種に関する情報は少ない。
細胞内分布
神経細胞で発現したArc mRNAの大部分は細胞体にてタンパク質翻訳に用いられた後に速やかに分解されると考えられるが、一部は樹状突起へと輸送される。樹状突起におけるArc mRNAは強いシナプス入力を受けた領域に集積し、樹状突起局所でタンパク質に翻訳される[3] 。Arcタンパク質の細胞内局在に関しては、細胞分画実験、細胞免疫染色および免疫電顕の結果などから後シナプス肥厚部に集積していることが示されている。Arcタンパク質は主要な後シナプス肥厚部タンパク質であるPSD-95やCaMKIIαおよびCaMKIIβと直接結合することが示されており、これらの相互作用はシナプス活動依存的に制御されている可能性が示唆されている[13][14] 。Arcタンパク質は細胞体や核においても存在し、これらの細胞区分においても何らかの機能を担っている可能性がある。核局在は神経活動依存的に制御されており転写制御に関わる可能性も示唆されている[15] 。
発現調節機構
神経細胞においてArc mRNAの発現はシナプス入力によって巧妙に調節されている。シナプス後部へのシナプス刺激によりNMDA型グルタミン酸受容体と電位依存性カルシウムチャネルの活性化によって細胞内へのカルシウム流入が生じ、カルシウムカルモジュリン依存性タンパク質キナーゼ(CaMK)経路およびMEK-MAPキナーゼ経路の活性化を引き起こす[16] 。また、細胞内カルシウムイオン上昇はカルシニューリン(CN)を活性化し、CN依存的経路を活性化する。さらにBDNFを含む多くの神経調節ペプチドはArc mRNAの発現誘導作用を持つ。これら複数の細胞内シグナルは核へと伝えられ、各シグナル経路に対応する転写因子および転写コファクターの活性化を引き起こす(図2)。Arc遺伝子の転写調節領域には複数のプロモーター・エンハンサーが同定されているが、中でも、マウスArc遺伝子の転写開始部位7 kb上流に存在するエンハンサーであるSARE (synaptic activity-responsive element)はArcの神経活動依存的な転写誘導に重要な機能を有する[16] 。SAREにはCREB、SRF、MEF2などの主要なシナプス活動依存的な転写因子の結合配列が含まれており、これらの転写因子およびコアクチベーターが協調して、Arcの高いシナプス刺激依存性を実現していると考えられる[17] 。
一過的なシナプス刺激が与えられると核において数分以内にArc mRNAの新規転写が開始される。この核内でのmRNA合成は一過的であり、刺激停止後には速やかに完了する。一方、新規合成されたmRNAはその後、細胞質に移動しタンパク合成に供される。このような細胞内mRNA局在の時間的変化を利用して、異なる2時点における神経活動状態を1細胞レベルで可視化する方法が考案されている[18] 。
機能
分子機能
Arcタンパク質は様々なシナプス調節に関わる。Arcはエンドフィリンやダイナミンなどと複合体を形成し、シナプス後部においてAMPA型グルタミン酸受容体のエンドサイトーシスを促進する[5] 。この性質と活動依存的発現によりArcはシナプス恒常性の分子機構を担う[19] 。すなわち、神経活動の亢進により発現上昇したArcは細胞全体のシナプス強度を下げる方向に働き、逆に活動抑制された神経細胞ではArc発現が抑えられるため細胞全体のシナプス強度は強められる。シナプス可塑性に関しては、Arcを欠損した海馬神経細胞では長期抑圧 (LTD)が阻害される[20] 。一方、LTPに関しては当初Arc欠損による障害が報告されていたが、最近の研究ではArcが欠損されても海馬のLTPの成立は正常である(刺激条件によってはむしろ亢進している)ことが示されている[21] 。これらの結果と一致して、Arcタンパク質はLTPが誘導されて強化されたシナプス後部には局在せず、活動の低いシナプスに集積するという”逆シナプスタグ“として働くことが提唱されている[14] 。小脳におけるArcは発達期の登上線維の刈り込みやプルキニエ細胞のLTDに関与していることが報告されている[22][23] 。Arcタンパク質は核にも局在しうるが、その役割はほとんど明らかになっていない[24][15] 。
個体での機能
Arc欠損マウスは正常に成長し、交配・繁殖可能である。脳や神経細胞の形態的な異常および運動や感覚障害も認められない。一方、神経可塑性が関わる神経機構には広範な異常が報告されている。Arc欠損マウスでは視覚野発達期の眼優位可塑性に障害がみられる[25] 。また、モリス水迷路などの空間学習や恐怖条件づけによる長期恐怖記憶の形成や維持が障害されるが、短期記憶への影響は少ない[20] 。大脳発達初期から前脳のみでArcを欠損させた条件的欠損マウスにおいても空間学習や記憶の障害が認められる[26] 。これらのArc欠損の表現型からは1) 神経回路の成熟過程におけるArcの重要性や2)長期記憶維持におけるLTPとLTDの協調の重要性などが考察される。なお、成熟後に海馬のみでArcを欠損させたマウスでは空間学習は正常であるが、長期記憶は障害されている[26] [22]。
恐怖条件づけ訓練時にArc陽性となる海馬神経細胞を記憶想起テスト時に光遺伝学を用いて活動抑制すると恐怖記憶想起が障害されることから、Arc陽性細胞もc-fos陽性細胞と同様に記憶痕跡(エングラム)細胞を構成すると考えられる[27] 。
疾患とのかかわり
ヒトにおいてはArc遺伝子の変異と特定の疾患との直接の関連は示されていない。大規模な全エクソームシーケンス解析の結果からはArcを含むタンパク質複合体 (Arc signaling complex)に関わる遺伝子変異と統合失調症との関連が示されている[28][29][30] 。
関連語
参考文献
- ↑ 1.0 1.1
Lyford, G.L., Yamagata, K., Kaufmann, W.E., Barnes, C.A., Sanders, L.K., Copeland, N.G., ..., & Worley, P.F. (1995).
Arc, a growth factor and activity-regulated gene, encodes a novel cytoskeleton-associated protein that is enriched in neuronal dendrites. Neuron, 14(2), 433-45. [PubMed:7857651] [WorldCat] [DOI] - ↑
Link, W., Konietzko, U., Kauselmann, G., Krug, M., Schwanke, B., Frey, U., & Kuhl, D. (1995).
Somatodendritic expression of an immediate early gene is regulated by synaptic activity. Proceedings of the National Academy of Sciences of the United States of America, 92(12), 5734-8. [PubMed:7777577] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1
Steward, O., Wallace, C.S., Lyford, G.L., & Worley, P.F. (1998).
Synaptic activation causes the mRNA for the IEG Arc to localize selectively near activated postsynaptic sites on dendrites. Neuron, 21(4), 741-51. [PubMed:9808461] [WorldCat] [DOI] - ↑
Nikolaienko, O., Patil, S., Eriksen, M.S., & Bramham, C.R. (2018).
Arc protein: a flexible hub for synaptic plasticity and cognition. Seminars in cell & developmental biology, 77, 33-42. [PubMed:28890419] [WorldCat] [DOI] - ↑ 5.0 5.1
Chowdhury, S., Shepherd, J.D., Okuno, H., Lyford, G., Petralia, R.S., Plath, N., ..., & Worley, P.F. (2006).
Arc/Arg3.1 interacts with the endocytic machinery to regulate AMPA receptor trafficking. Neuron, 52(3), 445-59. [PubMed:17088211] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Eriksen, M.S., Nikolaienko, O., Hallin, E.I., Grødem, S., Bustad, H.J., Flydal, M.I., ..., & Bramham, C.R. (2021).
Arc self-association and formation of virus-like capsids are mediated by an N-terminal helical coil motif. The FEBS journal, 288(9), 2930-2955. [PubMed:33175445] [WorldCat] [DOI] - ↑ 7.0 7.1 7.2 7.3 7.4
Pastuzyn, E.D., Day, C.E., Kearns, R.B., Kyrke-Smith, M., Taibi, A.V., McCormick, J., ..., & Shepherd, J.D. (2018).
The Neuronal Gene Arc Encodes a Repurposed Retrotransposon Gag Protein that Mediates Intercellular RNA Transfer. Cell, 173(1), 275. [PubMed:29570995] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1
Zhang, W., Wu, J., Ward, M.D., Yang, S., Chuang, Y.A., Xiao, M., ..., & Worley, P.F. (2015).
Structural basis of arc binding to synaptic proteins: implications for cognitive disease. Neuron, 86(2), 490-500. [PubMed:25864631] [PMC] [WorldCat] [DOI] - ↑
Mabb, A.M., Je, H.S., Wall, M.J., Robinson, C.G., Larsen, R.S., Qiang, Y., ..., & Ehlers, M.D. (2014).
Triad3A regulates synaptic strength by ubiquitination of Arc. Neuron, 82(6), 1299-316. [PubMed:24945773] [PMC] [WorldCat] [DOI] - ↑ 10.0 10.1 10.2
Ashley, J., Cordy, B., Lucia, D., Fradkin, L.G., Budnik, V., & Thomson, T. (2018).
Retrovirus-like Gag Protein Arc1 Binds RNA and Traffics across Synaptic Boutons. Cell, 172(1-2), 262-274.e11. [PubMed:29328915] [PMC] [WorldCat] [DOI] - ↑
Erlendsson, S., Morado, D.R., Cullen, H.B., Feschotte, C., Shepherd, J.D., & Briggs, J.A.G. (2020).
Structures of virus-like capsids formed by the Drosophila neuronal Arc proteins. Nature neuroscience, 23(2), 172-175. [PubMed:31907439] [PMC] [WorldCat] [DOI] - ↑
Jenks, K.R., Kim, T., Pastuzyn, E.D., Okuno, H., Taibi, A.V., Bito, H., ..., & Shepherd, J.D. (2017).
Arc restores juvenile plasticity in adult mouse visual cortex. Proceedings of the National Academy of Sciences of the United States of America, 114(34), 9182-9187. [PubMed:28790183] [PMC] [WorldCat] [DOI] - ↑
Fernández, E., Collins, M.O., Frank, R.A.W., Zhu, F., Kopanitsa, M.V., Nithianantharajah, J., ..., & Grant, S.G.N. (2017).
Arc Requires PSD95 for Assembly into Postsynaptic Complexes Involved with Neural Dysfunction and Intelligence. Cell reports, 21(3), 679-691. [PubMed:29045836] [PMC] [WorldCat] [DOI] - ↑ 14.0 14.1
Okuno, H., Akashi, K., Ishii, Y., Yagishita-Kyo, N., Suzuki, K., Nonaka, M., ..., & Bito, H. (2012).
Inverse synaptic tagging of inactive synapses via dynamic interaction of Arc/Arg3.1 with CaMKIIβ. Cell, 149(4), 886-98. [PubMed:22579289] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1
Korb, E., Wilkinson, C.L., Delgado, R.N., Lovero, K.L., & Finkbeiner, S. (2013).
Arc in the nucleus regulates PML-dependent GluA1 transcription and homeostatic plasticity. Nature neuroscience, 16(7), 874-83. [PubMed:23749147] [PMC] [WorldCat] [DOI] - ↑ 16.0 16.1
Kawashima, T., Okuno, H., Nonaka, M., Adachi-Morishima, A., Kyo, N., Okamura, M., ..., & Bito, H. (2009).
Synaptic activity-responsive element in the Arc/Arg3.1 promoter essential for synapse-to-nucleus signaling in activated neurons. Proceedings of the National Academy of Sciences of the United States of America, 106(1), 316-21. [PubMed:19116276] [PMC] [WorldCat] [DOI] - ↑
Okuno, H. (2011).
Regulation and function of immediate-early genes in the brain: beyond neuronal activity markers. Neuroscience research, 69(3), 175-86. [PubMed:21163309] [WorldCat] [DOI] - ↑
Guzowski, J.F., McNaughton, B.L., Barnes, C.A., & Worley, P.F. (1999).
Environment-specific expression of the immediate-early gene Arc in hippocampal neuronal ensembles. Nature neuroscience, 2(12), 1120-4. [PubMed:10570490] [WorldCat] [DOI] - ↑
Shepherd, J.D., Rumbaugh, G., Wu, J., Chowdhury, S., Plath, N., Kuhl, D., ..., & Worley, P.F. (2006).
Arc/Arg3.1 mediates homeostatic synaptic scaling of AMPA receptors. Neuron, 52(3), 475-84. [PubMed:17088213] [PMC] [WorldCat] [DOI] - ↑ 20.0 20.1
Plath, N., Ohana, O., Dammermann, B., Errington, M.L., Schmitz, D., Gross, C., ..., & Kuhl, D. (2006).
Arc/Arg3.1 is essential for the consolidation of synaptic plasticity and memories. Neuron, 52(3), 437-44. [PubMed:17088210] [WorldCat] [DOI] - ↑
Kyrke-Smith, M., Volk, L.J., Cooke, S.F., Bear, M.F., Huganir, R.L., & Shepherd, J.D. (2021).
The Immediate Early Gene Arc Is Not Required for Hippocampal Long-Term Potentiation. The Journal of neuroscience : the official journal of the Society for Neuroscience, 41(19), 4202-4211. [PubMed:33833081] [PMC] [WorldCat] [DOI] - ↑
Mikuni, T., Uesaka, N., Okuno, H., Hirai, H., Deisseroth, K., Bito, H., & Kano, M. (2013).
Arc/Arg3.1 is a postsynaptic mediator of activity-dependent synapse elimination in the developing cerebellum. Neuron, 78(6), 1024-35. [PubMed:23791196] [PMC] [WorldCat] [DOI] - ↑
Smith-Hicks, C., Xiao, B., Deng, R., Ji, Y., Zhao, X., Shepherd, J.D., ..., & Linden, D.J. (2010).
SRF binding to SRE 6.9 in the Arc promoter is essential for LTD in cultured Purkinje cells. Nature neuroscience, 13(9), 1082-9. [PubMed:20694003] [PMC] [WorldCat] [DOI] - ↑
Bloomer, W.A., VanDongen, H.M., & VanDongen, A.M. (2007).
Activity-regulated cytoskeleton-associated protein Arc/Arg3.1 binds to spectrin and associates with nuclear promyelocytic leukemia (PML) bodies. Brain research, 1153, 20-33. [PubMed:17466953] [WorldCat] [DOI] - ↑
McCurry, C.L., Shepherd, J.D., Tropea, D., Wang, K.H., Bear, M.F., & Sur, M. (2010).
Loss of Arc renders the visual cortex impervious to the effects of sensory experience or deprivation. Nature neuroscience, 13(4), 450-7. [PubMed:20228806] [PMC] [WorldCat] [DOI] - ↑ 26.0 26.1
Gao, X., Castro-Gomez, S., Grendel, J., Graf, S., Süsens, U., Binkle, L., ..., & Ohana, O. (2018).
Arc/Arg3.1 mediates a critical period for spatial learning and hippocampal networks. Proceedings of the National Academy of Sciences of the United States of America, 115(49), 12531-12536. [PubMed:30442670] [PMC] [WorldCat] [DOI] - ↑
Denny, C.A., Kheirbek, M.A., Alba, E.L., Tanaka, K.F., Brachman, R.A., Laughman, K.B., ..., & Hen, R. (2014).
Hippocampal memory traces are differentially modulated by experience, time, and adult neurogenesis. Neuron, 83(1), 189-201. [PubMed:24991962] [PMC] [WorldCat] [DOI] - ↑
Fromer, M., Pocklington, A.J., Kavanagh, D.H., Williams, H.J., Dwyer, S., Gormley, P., ..., & O'Donovan, M.C. (2014).
De novo mutations in schizophrenia implicate synaptic networks. Nature, 506(7487), 179-84. [PubMed:24463507] [PMC] [WorldCat] [DOI] - ↑
Kirov, G., Pocklington, A.J., Holmans, P., Ivanov, D., Ikeda, M., Ruderfer, D., ..., & Owen, M.J. (2012).
De novo CNV analysis implicates specific abnormalities of postsynaptic signalling complexes in the pathogenesis of schizophrenia. Molecular psychiatry, 17(2), 142-53. [PubMed:22083728] [PMC] [WorldCat] [DOI] - ↑
Purcell, S.M., Moran, J.L., Fromer, M., Ruderfer, D., Solovieff, N., Roussos, P., ..., & Sklar, P. (2014).
A polygenic burden of rare disruptive mutations in schizophrenia. Nature, 506(7487), 185-90. [PubMed:24463508] [PMC] [WorldCat] [DOI]