「Notch」の版間の差分
細 (→疾患との関連) |
細 (→ファミリー) |
||
| 31行目: | 31行目: | ||
==ファミリー== | ==ファミリー== | ||
ショウジョウバエ(Drosophila melanogaster)では1種類のNotchレセプター(Notch)があるのに対し、[[線虫]]([[Caenorhabditis elegans]])では2種類のNotchレセプター([[LIN12]], [[GLP1]])、[[哺乳類]]では4種類のNotchパラログ([[Notch1]]-[[Notch4|4]])が報告されている。 | ショウジョウバエ(Drosophila melanogaster)では1種類のNotchレセプター(Notch)があるのに対し、[[線虫]]([[Caenorhabditis elegans]])では2種類のNotchレセプター([[LIN12]], [[GLP1]])、[[哺乳類]]では4種類のNotchパラログ([[Notch1]]-[[Notch4|4]])が報告されている。 | ||
(<u>編集部コメント:タンパク質発現(組織発現、細胞内発現)についても出来れば段落を設けた上で、ご解説頂ければと思います。</u>) | |||
==機能== | ==機能== | ||
2016年6月1日 (水) 02:39時点における版
下條 博美、影山 龍一郎
京都大学
DOI:10.14931/bsd.7111 原稿受付日:2016年5月6日 原稿完成日:2016年月日
担当編集委員:村上 富士夫(大阪大学 大学院生命機能研究科)
一段落程度の抄録をお願い致します。
Notchとは
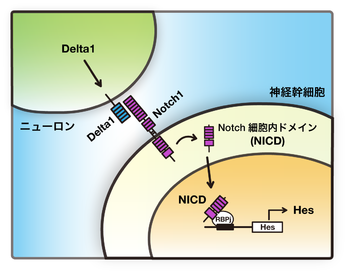
Notchシグナルは、ショウジョウバエから哺乳動物にまで広く保存されたシグナル伝達経路の一つである。このシグナル伝達の特徴は、細胞間の物理的な接触を必要とする近距離間でのシグナル伝達経路である。
細胞表面上で起こるNotchリガンド(DeltaやJagged)とNotchとの相互作用によって、Notchタンパク質が細胞膜から切り出される。Notchの細胞内ドメイン(Notch intra-cellular domain; NICD)は、核内へと輸送され、RBPj/CSLなどのDNA結合タンパク質と複合体を作り下流の標的遺伝子の発現を誘導する。この一連のシグナル伝達経路を「カノニカル(canonical)」信号伝達と呼ぶ(図1)[1] [2] [3]。
構造
Notchは1回膜貫通型タンパク質である。
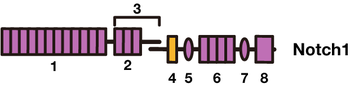
巨大な分子構造を持つNotchタンパク質には生物種間で保存された幾つかのモチーフがある。細胞外領域(N末端側)から列記する(図2)[1][3]。
- EGFリピート (epidermal growth factor (EGF)-like repeats) Notchレセプターの細胞外ドメインには、29〜36個のタンデムにつながったEGFリピートがあり、この領域でリガンドと相互作用する。隣接細胞間とのシグナル伝達(trans-interactions)は、11番目と12番目のEGFリピートによってなされる。また、同一細胞内におけるリガンド分子との相互作用(cis-inhibition)においては、24〜29番目のEGFリピートを介して起こる。EGFリピートの多くはカルシウムイオンと結合することが知られているが、これらのカルシウムイオンはNotchレセプターの構造やリガンド分子との親和性を制御することによって、シグナル伝達効率を制御していると考えられている[4][5]。
- LNR(Lin12-Notch repeat) EGFリピートの後に続くリピート配列。NRRに含まれる。
- NRR (negative regulatory region) LNR(Lin12-Notch repeat)およびHDドメイン(heterodimerization domain)より成る。リガンドがない状態でレセプターが活性化するのを抑制している。
- 膜貫通ドメイン (transmembrane domain; TMD)
- RAMドメイン (RBPjκ association module domain) 核内へ運ばれた後、DNA結合タンパク質であるCSL(CBF1/RBPjκ/Su(H)/Lag-1)に強く結合する領域。
- アンキリンリピート (ankyrin repeats, ANK domain) 7つのアンキリン配列が連なった領域。CSLと結合しコアクチベーターであるMastermind/Lag-3をリクルートする。
- 核移行配列 (nuclear localization sequences, NLS)
- PEST配列 (proline/glutamic acid/serine/threonine-rich motives, PEST) 分解促進配列。NICDの安定性を制御する。
ファミリー
ショウジョウバエ(Drosophila melanogaster)では1種類のNotchレセプター(Notch)があるのに対し、線虫(Caenorhabditis elegans)では2種類のNotchレセプター(LIN12, GLP1)、哺乳類では4種類のNotchパラログ(Notch1-4)が報告されている。
(編集部コメント:タンパク質発現(組織発現、細胞内発現)についても出来れば段落を設けた上で、ご解説頂ければと思います。)
機能
活性化とプロセシング
Notchレセプターの活性化にはタンパク質分解過程が連続的に起こることが重要である[1] [2]。
- S1 cleavage: Furin cleavage Notchレセプタータンパク質が翻訳され細胞膜へと輸送される間に、Notchタンパク質はゴルジ装置へと運ばれ、Furinによって最初の切断をうける。この過程によってNotchレセプターはS1(site1)部分で半分に切断される。切り出された二つの領域は、HDドメインにて非共有結合によって結合したヘテロ二量体を形成する。ヘテロ二量体となったNotchレセプターは細胞表面へと輸送され、細胞外ドメインを外に出してリガンド分子との相互作用に備える。
- S2 cleavage: ADAM cleavage Notchレセプターと隣接細胞上のリガンドとの相互作用は、ADAM(a disintegrin and metaloprotease)タンパク質分解酵素による切断を誘導する。ADAMタンパク質分解酵素による切断は、S2領域で起こる。この領域はNRRドメインによって普段は奥まった場所に位置しており保護されている。この切断のプロセスはNotchシグナル伝達において重要なステップである。
- S3/S4 cleavage: γ-secretase cleavage ADAMタンパク質分解酵素によるNotchレセプターの細胞外領域における切断は、次にγセクレターゼによる分解を誘導する。これによって生じた膜に繋がれたNotchの細胞内ドメインは、Notch extracellular truncation(NEXT)と呼ばれる。γセクレターゼは膜内タンパク質分解酵素であり[6]、NEXTを膜貫通ドメイン(TMD)のS3およびS4の領域で切断する。γセクレターゼの切断が起こらないと、NICDの核内への輸送は起こらない。NICDは核内へと運ばれた後、RBPj/CSLと結合し、コアクチベーターと複合体を形成して、転写因子群の転写を活性化する。
標的遺伝子
哺乳動物の神経系におけるNotchシグナルの標的遺伝子は、bHLH型抑制性転写因子をコードするHesファミリー遺伝子(Hes1, Hes5)[7]およびHes-related遺伝子(Hey1, Hey2)[8]が報告されている。Hesファミリー遺伝子は神経分化を誘導するプロニューラル遺伝子(Ascl1やNeurogenin)の発現や機能を抑制することによって、幹細胞がニューロンへと分化しないように機能している。また、BLBP(brain lipid-binding protein)もNotchシグナルによって直接制御されていることが報告されている[9]。
神経発生における役割
哺乳動物の神経発生においてNotchシグナルは、神経幹細胞の維持に重要な機能を果たしている。また発生過程の網膜においてミュラーグリアへの運命決定に寄与するなど、グリア細胞の運命決定にもNotchが寄与している。
さらに中枢神経系において、Zli、Isthmus(編集部コメント:日本語はあるでしょう。)、底板、蓋板といった領域の境界を形成する構造の形成にも機能していることが報告されている[10] [11]。
神経幹細胞維持
隣接細胞が発現するDeltaによって活性化されたNotchレセプターは、細胞膜上でレセプターのタンパク質が切り出され、細胞内ドメイン(NICD: Notch intracellular domain)が核内へと移行しNotchシグナルが活性化される。NICDの過剰発現により神経幹細胞からのニューロン分化は抑制される。また、Notchシグナルが活性化された細胞においては、bHLH型抑制性転写因子Hes1、Hes5が発現し、これらの因子がプロニューラル遺伝子の発現や機能を抑制することによって、幹細胞の未分化性を維持している[12] [13] [8]。
グリア細胞分化
神経幹細胞から神経細胞が分化した後、Notchシグナルはグリア細胞の分化に寄与していることが報告されている。発生過程の網膜においてHes1の過剰発現によりミュラーグリアの産生が亢進されるのに対して、Hes1のドミナントネガティブによる機能阻害によって、産生されるグリアの数が減少する[14]。またHes5の過剰発現、欠損による実験においても同様の結果が報告されている[15]。
疾患との関連
Notchシグナル伝達は発生過程および成体の幹細胞維持において重要な機能を果たしているため、このシグナル伝達における欠陥は様々な疾患を引き起こすことが知られている。
(編集部コメント:箇条書きになっていたものを表に致しました。ご確認ください。また、それぞれ簡単にどのように関連しているかをいただければと思います。)
| 発生過程における疾患[16] [17] |
成体における疾患[18]
|
参考文献
- ↑ 1.0 1.1 1.2
Bray, S.J. (2006).
Notch signalling: a simple pathway becomes complex. Nature reviews. Molecular cell biology, 7(9), 678-89. [PubMed:16921404] [WorldCat] [DOI] - ↑ 2.0 2.1
Kopan, R., & Ilagan, M.X. (2009).
The canonical Notch signaling pathway: unfolding the activation mechanism. Cell, 137(2), 216-33. [PubMed:19379690] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1
Louvi, A., & Artavanis-Tsakonas, S. (2006).
Notch signalling in vertebrate neural development. Nature reviews. Neuroscience, 7(2), 93-102. [PubMed:16429119] [WorldCat] [DOI] - ↑
Cordle, J., Redfieldz, C., Stacey, M., van der Merwe, P.A., Willis, A.C., Champion, B.R., ..., & Handford, P.A. (2008).
Localization of the delta-like-1-binding site in human Notch-1 and its modulation by calcium affinity. The Journal of biological chemistry, 283(17), 11785-93. [PubMed:18296446] [WorldCat] [DOI] - ↑
Raya, A., Kawakami, Y., Rodríguez-Esteban, C., Ibañes, M., Rasskin-Gutman, D., Rodríguez-León, J., ..., & Izpisúa Belmonte, J.C. (2004).
Notch activity acts as a sensor for extracellular calcium during vertebrate left-right determination. Nature, 427(6970), 121-8. [PubMed:14712268] [WorldCat] [DOI] - ↑
Wolfe, M.S., & Kopan, R. (2004).
Intramembrane proteolysis: theme and variations. Science (New York, N.Y.), 305(5687), 1119-23. [PubMed:15326347] [WorldCat] [DOI] - ↑
Ohtsuka, T., Ishibashi, M., Gradwohl, G., Nakanishi, S., Guillemot, F., & Kageyama, R. (1999).
Hes1 and Hes5 as notch effectors in mammalian neuronal differentiation. The EMBO journal, 18(8), 2196-207. [PubMed:10205173] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1
Sakamoto, M., Hirata, H., Ohtsuka, T., Bessho, Y., & Kageyama, R. (2003).
The basic helix-loop-helix genes Hesr1/Hey1 and Hesr2/Hey2 regulate maintenance of neural precursor cells in the brain. The Journal of biological chemistry, 278(45), 44808-15. [PubMed:12947105] [WorldCat] [DOI] - ↑
Anthony, T.E., Mason, H.A., Gridley, T., Fishell, G., & Heintz, N. (2005).
Brain lipid-binding protein is a direct target of Notch signaling in radial glial cells. Genes & development, 19(9), 1028-33. [PubMed:15879553] [PMC] [WorldCat] [DOI] - ↑
Baek, J.H., Hatakeyama, J., Sakamoto, S., Ohtsuka, T., & Kageyama, R. (2006).
Persistent and high levels of Hes1 expression regulate boundary formation in the developing central nervous system. Development (Cambridge, England), 133(13), 2467-76. [PubMed:16728479] [WorldCat] [DOI] - ↑
Cheng, Y.C., Amoyel, M., Qiu, X., Jiang, Y.J., Xu, Q., & Wilkinson, D.G. (2004).
Notch activation regulates the segregation and differentiation of rhombomere boundary cells in the zebrafish hindbrain. Developmental cell, 6(4), 539-50. [PubMed:15068793] [WorldCat] - ↑
Ishibashi, M., Moriyoshi, K., Sasai, Y., Shiota, K., Nakanishi, S., & Kageyama, R. (1994).
Persistent expression of helix-loop-helix factor HES-1 prevents mammalian neural differentiation in the central nervous system. The EMBO journal, 13(8), 1799-805. [PubMed:7909512] [PMC] [WorldCat] - ↑
Ohtsuka, T., Sakamoto, M., Guillemot, F., & Kageyama, R. (2001).
Roles of the basic helix-loop-helix genes Hes1 and Hes5 in expansion of neural stem cells of the developing brain. The Journal of biological chemistry, 276(32), 30467-74. [PubMed:11399758] [WorldCat] [DOI] - ↑
Furukawa, T., Mukherjee, S., Bao, Z.Z., Morrow, E.M., & Cepko, C.L. (2000).
rax, Hes1, and notch1 promote the formation of Müller glia by postnatal retinal progenitor cells. Neuron, 26(2), 383-94. [PubMed:10839357] [WorldCat] [DOI] - ↑
Hojo, M., Ohtsuka, T., Hashimoto, N., Gradwohl, G., Guillemot, F., & Kageyama, R. (2000).
Glial cell fate specification modulated by the bHLH gene Hes5 in mouse retina. Development (Cambridge, England), 127(12), 2515-22. [PubMed:10821751] [WorldCat] - ↑
Garg, V., Muth, A.N., Ransom, J.F., Schluterman, M.K., Barnes, R., King, I.N., ..., & Srivastava, D. (2005).
Mutations in NOTCH1 cause aortic valve disease. Nature, 437(7056), 270-4. [PubMed:16025100] [WorldCat] [DOI] - ↑
Gridley, T. (2003).
Notch signaling and inherited disease syndromes. Human molecular genetics, 12 Spec No 1, R9-13. [PubMed:12668592] [WorldCat] [DOI] - ↑
Louvi, A., Arboleda-Velasquez, J.F., & Artavanis-Tsakonas, S. (2006).
CADASIL: a critical look at a Notch disease. Developmental neuroscience, 28(1-2), 5-12. [PubMed:16508299] [WorldCat] [DOI] - ↑
Noguera-Troise, I., Daly, C., Papadopoulos, N.J., Coetzee, S., Boland, P., Gale, N.W., ..., & Thurston, G. (2006).
Blockade of Dll4 inhibits tumour growth by promoting non-productive angiogenesis. Nature, 444(7122), 1032-7. [PubMed:17183313] [WorldCat] [DOI] - ↑
Ridgway, J., Zhang, G., Wu, Y., Stawicki, S., Liang, W.C., Chanthery, Y., ..., & Yan, M. (2006).
Inhibition of Dll4 signalling inhibits tumour growth by deregulating angiogenesis. Nature, 444(7122), 1083-7. [PubMed:17183323] [WorldCat] [DOI] - ↑
van Es, J.H., van Gijn, M.E., Riccio, O., van den Born, M., Vooijs, M., Begthel, H., ..., & Clevers, H. (2005).
Notch/gamma-secretase inhibition turns proliferative cells in intestinal crypts and adenomas into goblet cells. Nature, 435(7044), 959-63. [PubMed:15959515] [WorldCat] [DOI] - ↑
Wolfe, M.S., & Kopan, R. (2004).
Intramembrane proteolysis: theme and variations. Science (New York, N.Y.), 305(5687), 1119-23. [PubMed:15326347] [WorldCat] [DOI]