「サザンブロット」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| (4人の利用者による、間の36版が非表示) | |||
| 1行目: | 1行目: | ||
== 概要 == | |||
< | |||
[[Image:サザンブロット図1.jpg|thumb|right|400px]] | |||
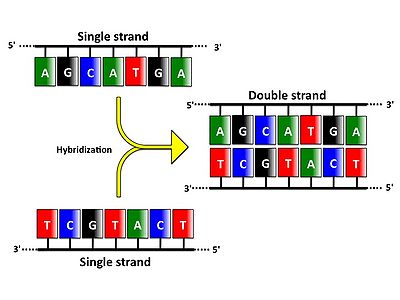
<div>相補性のある2本のDNA1本鎖は一対の2本鎖を形成するというDNAの性質(図1)を利用して、ある特定の塩基配列をもつDNA断片を検出する方法である。E. M. Southernが考案したためにこの名が付けられた。 </div><div></div><div></div><div></div><div></div><div></div><div></div><div></div><div></div><div></div><div></div><div></div> | |||
<br> | |||
</div> | |||
== 手順 == | |||
1) 制限酵素によるDNAの消化 この際にメチル化感受性制限酵素と非メチル化感受性制限酵素を組み合わせて使用することで後述に示すDNAメチル化状態の解析が出来る。 | |||
2) アガロースゲル電気泳動 | |||
3) ゲル内DNAの変性 電気泳動により分離されたDNAをアガロースゲル内で1本鎖にする(変性)ために電気泳動後のアガロースゲルをNaOHなどの塩基性溶液で処理をする。 | |||
4) メンブランへの転写 アガロースゲル内のDNAを泳動パターンを維持したままを毛細管現象を利用してナイロン製やニトロセルロース製のメンブランへ転写する。この作業には数時間から一晩要するため、近年は電気的に転写するエレクトロブロット、吸引によるバキュームブロットなど短時間で転写できる装置も市販されている。 | |||
5) プローブとの反応 特定の塩基配列を検出するための標識(ラベリング)された核酸をプローブと呼び、 | |||
[[Image:サザンブロット図2.jpg|thumb|right|400px]] | |||
<div><br> | |||
<br> | |||
標識には32Pなどの放射性同位体の他、アルカリフォスファターゼなどの酵素が利用される。プローブに用いる核酸はDNA, RNAいずれも使用でき、それぞれDNAプローブ、RNAプローブと呼ばれる。RNAプローブはDNAプローブに比してメンブラン上のDNAとの結合力が高いが扱いにくいためDNAプローブが頻用される。ただしDNAプローブは使用前に1本鎖にする必要がある。 これらのプローブをDNAを転写したメンブラン上で反応させることで相補的な配列をもつDNAとプローブが2本鎖を形成する。 なお、このときに塩濃度を高くすることでプローブの配列と一致しないまでも相同性の高い配列を持つDNAは2本鎖を形成する。これを利用すれば異なる動物種間の遺伝子、あるいは相同遺伝子を検出することが出来る場合もある。 | |||
6) メンブランの洗浄 メンブラン上のDNAに結合しなかったプローブを洗浄する。この際にも洗浄液の塩濃度を高くすることでプローブと多少配列が異なるDNA間の2本鎖は結合したままにすることが出来る。 | |||
7) 検出 プローブ標識に放射性同位体を用いた場合はX線フィルムをを用いたオートラジオグラフィーでプローブの位置を検出する。また、プローブをアルカリホスファターゼ標識した場合は発光基質を反応させその発光をフィルムで検出するほか、CCDカメラなどの検出器なども用いられる。 | |||
== ノーザンブロットとの相違点 == | == ノーザンブロットとの相違点 == | ||
基本原理、手順ともに両者に大きな相違はないが、ノーザンブロットは検出する対象がDNAではなくRNAであること、さらにRNAは1本鎖であるために泳動後のアガロースを塩基性 溶液で処理する必要がないことである。なお、サザンブロットは考案者の名前から命名されているため、英文では大文字から表記される (Southern blot)が、ノーザンブロットはサザンブロットに対する「洒落」で命名されているため小文字から表記 (northern blot)される。これは抗体を用いてSDS-PAGE後のタンパク質を検出するウェスタンブロット (western blot)においても同様である。 | |||
== 使用例 == | |||
=== 遺伝子変異の検出 === | === 遺伝子変異の検出 === | ||
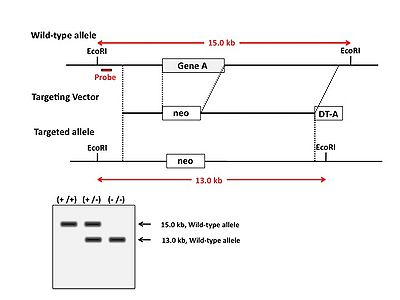
サザンブロットにより検出されるバンドの大きさの変化で遺伝子の変異を検出することが出来る。例えば遺伝子改変動物をジーンターゲティング法で作製した場合には、そのバンドの大きさで遺伝子型(野生型、ヘテロ、ホモ)の判別に用いられる(図)。 | |||
=== DNAメチル化状態の解析 === | === DNAメチル化状態の解析 === | ||
ゲノムDNAメチル化は遺伝子発現における調節因子であるため、その解析は遺伝子発現制御の分野では重要である。 制限酵素HpaIIとMspIは同じ5'- CCGG - 3'を認識し切断するが、メチル化感受性制限酵素であるHpaIIは2番目のCがメチル化された配列 (5'- CmCGG - 3')は切断することが出来ない。一方MspIはメチル化状態にかかわらず切断することが出来る。そこで、これらの制限酵素を利用したサザンブロットで特定のCpG配列のメチル化状態を調べることが出来る(図)。[[Image:サザンブロット図3.jpg|thumb|left|400px]] | |||
</div> | |||
< | |||
2012年5月12日 (土) 17:10時点における版
概要
手順
1) 制限酵素によるDNAの消化 この際にメチル化感受性制限酵素と非メチル化感受性制限酵素を組み合わせて使用することで後述に示すDNAメチル化状態の解析が出来る。
2) アガロースゲル電気泳動
3) ゲル内DNAの変性 電気泳動により分離されたDNAをアガロースゲル内で1本鎖にする(変性)ために電気泳動後のアガロースゲルをNaOHなどの塩基性溶液で処理をする。
4) メンブランへの転写 アガロースゲル内のDNAを泳動パターンを維持したままを毛細管現象を利用してナイロン製やニトロセルロース製のメンブランへ転写する。この作業には数時間から一晩要するため、近年は電気的に転写するエレクトロブロット、吸引によるバキュームブロットなど短時間で転写できる装置も市販されている。
5) プローブとの反応 特定の塩基配列を検出するための標識(ラベリング)された核酸をプローブと呼び、
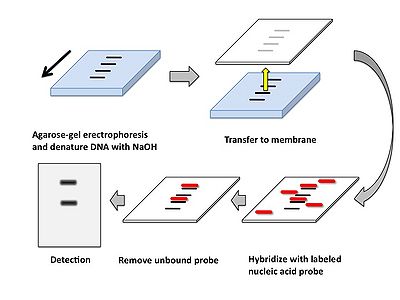
標識には32Pなどの放射性同位体の他、アルカリフォスファターゼなどの酵素が利用される。プローブに用いる核酸はDNA, RNAいずれも使用でき、それぞれDNAプローブ、RNAプローブと呼ばれる。RNAプローブはDNAプローブに比してメンブラン上のDNAとの結合力が高いが扱いにくいためDNAプローブが頻用される。ただしDNAプローブは使用前に1本鎖にする必要がある。 これらのプローブをDNAを転写したメンブラン上で反応させることで相補的な配列をもつDNAとプローブが2本鎖を形成する。 なお、このときに塩濃度を高くすることでプローブの配列と一致しないまでも相同性の高い配列を持つDNAは2本鎖を形成する。これを利用すれば異なる動物種間の遺伝子、あるいは相同遺伝子を検出することが出来る場合もある。
6) メンブランの洗浄 メンブラン上のDNAに結合しなかったプローブを洗浄する。この際にも洗浄液の塩濃度を高くすることでプローブと多少配列が異なるDNA間の2本鎖は結合したままにすることが出来る。
7) 検出 プローブ標識に放射性同位体を用いた場合はX線フィルムをを用いたオートラジオグラフィーでプローブの位置を検出する。また、プローブをアルカリホスファターゼ標識した場合は発光基質を反応させその発光をフィルムで検出するほか、CCDカメラなどの検出器なども用いられる。
ノーザンブロットとの相違点
基本原理、手順ともに両者に大きな相違はないが、ノーザンブロットは検出する対象がDNAではなくRNAであること、さらにRNAは1本鎖であるために泳動後のアガロースを塩基性 溶液で処理する必要がないことである。なお、サザンブロットは考案者の名前から命名されているため、英文では大文字から表記される (Southern blot)が、ノーザンブロットはサザンブロットに対する「洒落」で命名されているため小文字から表記 (northern blot)される。これは抗体を用いてSDS-PAGE後のタンパク質を検出するウェスタンブロット (western blot)においても同様である。
使用例
遺伝子変異の検出
サザンブロットにより検出されるバンドの大きさの変化で遺伝子の変異を検出することが出来る。例えば遺伝子改変動物をジーンターゲティング法で作製した場合には、そのバンドの大きさで遺伝子型(野生型、ヘテロ、ホモ)の判別に用いられる(図)。
DNAメチル化状態の解析
ゲノムDNAメチル化は遺伝子発現における調節因子であるため、その解析は遺伝子発現制御の分野では重要である。 制限酵素HpaIIとMspIは同じ5'- CCGG - 3'を認識し切断するが、メチル化感受性制限酵素であるHpaIIは2番目のCがメチル化された配列 (5'- CmCGG - 3')は切断することが出来ない。一方MspIはメチル化状態にかかわらず切断することが出来る。そこで、これらの制限酵素を利用したサザンブロットで特定のCpG配列のメチル化状態を調べることが出来る(図)。