「Chromosome 9 open reading frame 72」の版間の差分
細 →歴史的推移 |
細編集の要約なし |
||
| (同じ利用者による、間の14版が非表示) | |||
| 7行目: | 7行目: | ||
担当編集委員:[http://researchmap.jp/read0192882 古屋敷 智之](東京科学大学大学院 医歯学総合研究科 薬理学分野)<br> | 担当編集委員:[http://researchmap.jp/read0192882 古屋敷 智之](東京科学大学大学院 医歯学総合研究科 薬理学分野)<br> | ||
</div> | </div> | ||
英: Chromosome 9 open reading frame 72<br> | |||
略語: C9orf72 | 略語: C9orf72 | ||
{{box|text= | {{box|text= C9orf72はDENNドメインを有するタンパク質であり、種々の低分子量Gタンパク質の活性制御を介して、小胞輸送やオートファジー、自然免疫といった多岐にわたる生体機能に関与する。2011年に、本遺伝子の非翻訳領域内に存在するGGGGCCリピート配列の異常伸長が、筋萎縮性側索硬化症および前頭側頭型認知症における最も高頻度の原因遺伝子変異として同定され、その病態メカニズムが注目されている。現在、①C9orf72遺伝子の機能喪失、②転写された変異リピートRNAによる毒性、③リピート関連非AUG依存性翻訳によって産生されるリピートペプチドによる毒性が、協調的に神経変性病態に寄与すると考えられている。}} | ||
== 歴史的推移 == | == 歴史的推移 == | ||
C9orf72遺伝子は、神経変性疾患である筋萎縮性側索硬化症(ALS: amyotrophic lateral sclerosis)および前頭側頭型認知症(FTD: frontotemporal dementia)の原因となる代表的な遺伝子の1つとして、研究が進展してきた。 | |||
2006年、家族性ALS/FTD家系を用いた連鎖解析によって、責任遺伝子領域が9番染色体短腕に同定され<ref name=Morita2006><pubmed>16421333</pubmed></ref><ref name=Vance2006><pubmed>16495328</pubmed></ref> | 2006年、家族性ALS/FTD家系を用いた連鎖解析によって、責任遺伝子領域が9番染色体短腕に同定され<ref name=Morita2006><pubmed>16421333</pubmed></ref>[1]<ref name=Vance2006><pubmed>16495328</pubmed></ref>[2]、その後の遺伝子探索によって、2011年にC9orf72遺伝子非翻訳領域内のGGGGCC 6塩基リピート配列の異常伸長(正常: 2-24回リピート, 疾患: 60-4000回リピート)が、原因遺伝子変異として同定された<ref name=DeJesusHernandez2011><pubmed>21944778</pubmed></ref>[3]<ref name=Renton2011><pubmed>21944779</pubmed></ref>[4]。このリピート伸長変異は、欧米のALSFTDコホートにおいて最も頻度の高い原因遺伝子変異であることが明らかとなり、同遺伝子の生理的機能やリピート伸長変異による病態メカニズムに注目が集まり、研究が展開されている。 | ||
生理的機能に関しては未解明の点が多いものの、分子系統解析では本遺伝子は真核生物の進化初期に出現し、哺乳類を含む広範な生物種で保存されていることから、生物学的に重要な役割を担うと想定されている<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]。特に、C9orf72タンパク質はDENN(differentially expressed in normal and neoplastic cells)ドメインを有し、本ドメインを有する一群のタンパク質はRabファミリーを中心とした低分子量Gタンパク質の活性制御に関与することから、小胞輸送やオートファジーとの関連が注目されている。近年では、自然免疫応答の制御機能についても示唆されている(後述)<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]。 | |||
C9orf72遺伝子連鎖性ALS/ | C9orf72遺伝子連鎖性ALS/FTD(C9-ALS/FTD)の病態メカニズムに関しては、当初、GGGGCCリピート伸長変異による遺伝子機能の喪失や、転写された異常伸長リピート配列を含む変異RNA(リピートRNA)による毒性が想定されていた。しかし2013年に、リピートRNAから非古典的なリピート関連非AUG依存性(RAN: repeat-associated non-AUG)翻訳によって病的なジペプチドリピートペプチドタンパク質(DPR: dipeptide repeat protein)が産生され、実際にDPRが患者脳に蓄積することが明らかになり、現在ではこれら3つの病態メカニズムが協調的に神経変性病態に寄与すると考えられている<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。 | ||
[[ファイル:Nagai C9orf72 Fig1.jpg|サムネイル|'''図1. C9orf72遺伝子, 転写産物, タンパク質'''<br>C9orf72遺伝子は12のエクソンから構成され、このうちエクソン2~11がタンパク質をコードする。GGGGCCリピート配列はエクソン1aと1bの間の非翻訳領域に存在する。3種類の転写産物と2種類のタンパク質アイソフォームが存在し、Variant 1はショートアイソフォームに、Variant2および3はロングアイソフォームにそれぞれ翻訳される。Variant 1およびVariant 3はGGGGCCリピート配列を含む。ロングアイソフォームにはDENNドメインが含まれる。]] | [[ファイル:Nagai C9orf72 Fig1.jpg|サムネイル|'''図1. C9orf72遺伝子, 転写産物, タンパク質'''<br>C9orf72遺伝子は12のエクソンから構成され、このうちエクソン2~11がタンパク質をコードする。GGGGCCリピート配列はエクソン1aと1bの間の非翻訳領域に存在する。3種類の転写産物と2種類のタンパク質アイソフォームが存在し、Variant 1はショートアイソフォームに、Variant2および3はロングアイソフォームにそれぞれ翻訳される。Variant 1およびVariant 3はGGGGCCリピート配列を含む。ロングアイソフォームにはDENNドメインが含まれる。]] | ||
== 構造 == | == 構造 == | ||
=== 遺伝子 === | === 遺伝子 === | ||
ヒトでは染色体9p21. | ヒトでは染色体9p21.2に存在する41kbの遺伝子で、12のエクソン(エクソン 1a、1b、2~11)から構成される<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。翻訳開始に利用されるATGコドンはエクソン2に存在し、エクソン2~11がC9orf72タンパク質をコードする。エクソン1aおよび1bはタンパク質をコードしない非翻訳領域である。ALS, FTDと関連するGGGGCCリピート配列は、エクソン1aと1bの間の非翻訳領域に存在する('''図1''')<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。 | ||
=== 転写産物 === | === 転写産物 === | ||
転写開始点および転写終結点を有し、転写産物については3種類のバリアントが報告されている。Variant 1(V1)はエクソン1a、2~5から構成される短鎖転写産物である。Variant 2(V2)はエクソン1b、2~11、Variant 3(V3)はエクソン1a、2~11から構成される長鎖転写産物である<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。GGGGCCリピート配列は、V1およびV3ではイントロン領域に存在する一方で、V2では転写開始点直上流に存在し、転写産物自体には含まれない('''図1''')<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。 | |||
=== タンパク質 === | === タンパク質 === | ||
222アミノ酸から構成されるショートアイソフォーム(分子量24.7kDa)と、481アミノ酸から構成されるロングアイソフォーム(分子量54.3kDa)の2種類のアイソフォームが存在する。ショートアイソフォームはV1転写産物から、ロングアイソフォームはV2およびV3転写産物から翻訳される('''図1''')<ref name=Smeyers2021><pubmed>34025358</pubmed></ref><ref name=Balendra2018><pubmed>30120348</pubmed></ref> | 222アミノ酸から構成されるショートアイソフォーム(分子量24.7kDa)と、481アミノ酸から構成されるロングアイソフォーム(分子量54.3kDa)の2種類のアイソフォームが存在する。ショートアイソフォームはV1転写産物から、ロングアイソフォームはV2およびV3転写産物から翻訳される('''図1''')<ref name=Smeyers2021><pubmed>34025358</pubmed></ref>[5]<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。ロングアイソフォームはDENNドメインを有し、主にGDP/GTP交換因子(GEF: GDP/GTP exchange factor)として低分子量Gタンパク質の活性制御を担うDENNファミリータンパク質の一員と考えられている<ref name=Levine2019><pubmed>31175302</pubmed></ref>[7]<ref name=Burberry2016><pubmed>27135602</pubmed></ref>[8]。 | ||
== 発現 == | == 発現 == | ||
=== RNAレベル === | === RNAレベル === | ||
C9orf72転写産物はほとんどのヒト組織において発現が認められるが、特に脳、血液細胞、脾臓、脾臓、卵巣などにおいて高発現を示す<ref name=Zhang2015><pubmed>26637796</pubmed></ref>[9]。脳内では小脳おける発現が顕著であり、血液細胞ではリンパ球よりも骨髄系細胞において高い発現が報告されている <ref name=Renton2011><pubmed>21944779</pubmed></ref>[4]<ref name=Zhang2018><pubmed>29379212</pubmed></ref>[10]。 | |||
=== タンパク質レベル === | === タンパク質レベル === | ||
C9orf72タンパク質もまた、ヒトのほとんどの組織で発現している。マウスを用いた解析では、脳、脊髄、および脾臓で特に高い発現を認めている<ref name=Shi2018><pubmed>30388411</pubmed></ref>[11]。 | |||
培養細胞では、C9orf72タンパク質はリソソームと共局在することが報告されている<ref name=Amick2016><pubmed>27488133</pubmed></ref>[12]。ヒト脳組織における免疫組織化学では、ショートアイソフォームが主に神経細胞の核膜に分布する一方で、ロングアイソフォームは細胞質全体に広く分布し、神経突起や細胞体に点状構造を形成することが確認されている<ref name=Yang2016><pubmed>27281217</pubmed></ref>[13]。また、ヒトiPS細胞由来運動ニューロンを用いた検討により、C9orf72タンパク質がシナプス小胞にも局在することが示唆されている<ref name=Shi2018><pubmed>30388411</pubmed></ref>[11]。 | |||
== 機能 == | == 機能 == | ||
DENNファミリータンパク質の1つであるC9orf72タンパク質は、同じくDENNドメインを有するSMCR8、およびWDR41と複合体を形成する<ref name=Webster2016><pubmed>27539327</pubmed></ref>[14]<ref name=Lall2019><pubmed>31029540</pubmed></ref>[15]<ref name=Koppers2015><pubmed>26012240</pubmed></ref>[16]<ref name=Farg2014><pubmed>24367275</pubmed></ref>[17]。この複合体はGTPase活性化タンパク質(GAP: GTPase activating protein)として<ref name=Koppers2015><pubmed>26012240</pubmed></ref>[16]<ref name=Farg2014><pubmed>24367275</pubmed></ref>[17]。C9orf72タンパク質が相互作用する代表的な低分子量G タンパク質は、小胞輸送に関与するRabファミリータンパク質であり、これによってオートファジーやシナプス機能などの様々な生体反応を制御することが明らかになっている。また近年では、自然免疫応答の制御においても重要な役割を担うことが示唆されている。 | |||
=== オートファジーの制御 === | === オートファジーの制御 === | ||
培養神経細胞系やヒト脊髄運動ニューロンにおいて、C9orf72タンパク質はRab1、Rab5、Rab7、Rab11などのRabファミリータンパク質と共局在する<ref name=PrparMihevc2016><pubmed>27669407</pubmed></ref>[18]。また培養細胞を用いた免疫沈降法による解析では、Rab8aやRab39b、FIP200/Ulk1/ATG13/ATG101複合体と相互作用することが報告されている<ref name=Lall2019><pubmed>31029540</pubmed></ref>[15]<ref name=Koppers2015><pubmed>26012240</pubmed></ref>[16]。これらの分子との相互作用を介して、C9orf72タンパク質はオートファジーの開始、基質のリクルートメント、隔離膜の伸長と閉鎖、オートファゴソームとリソソームの融合など、オートファジーの各段階を制御すると考えられている<ref name=LopezGonzalez2016><pubmed>27112497</pubmed></ref>[19]。実際に、C9orf72遺伝子をノックダウンした培養細胞では、オートファジーの誘導阻害や異常なオートファゴソームの蓄積が観察されている<ref name=Koppers2015><pubmed>26012240</pubmed></ref>[16]<ref name=PrparMihevc2016><pubmed>27669407</pubmed></ref>[18]。 | |||
=== シナプス機能の制御 === | === シナプス機能の制御 === | ||
C9orf72タンパク質が相互作用するRab39bは、AMPA型グルタミン酸受容体の小胞輸送およびリサイクリングに関与し、受容体のサブユニット構成やシナプスの電気生理学的特性を調節する<ref name=Westergard2016><pubmed>27073919</pubmed></ref>[20]。C9orf72ノックアウトマウスでは、シナプス後部においてRab39bの発現が低下し、AMPA型グルタミン酸受容体GluR1サブユニットの発現が亢進することが報告されており、C9orf72タンパク質がRab39bの発現や活性を制御することでシナプス機能を調節している可能性が示唆されている<ref name=Jiang2016><pubmed>27022092</pubmed></ref>[21]。 | |||
=== 細胞骨格の制御 === | === 細胞骨格の制御 === | ||
C9orf72タンパク質は、Rabファミリータンパク質とは異なる低分子量G タンパク質であるArfファミリータンパク質とも相互作用し、特にArf6との相互作用を介して、運動ニューロンにおけるアクチン細胞骨格の動態を制御することが報告されている[]<ref name=Kramer2016><pubmed>27377358</pubmed></ref>[22]。培養神経細胞ではC9orf72遺伝子の過剰発現によって軸索が伸長し、ノックダウンによって短縮することが確認されている<ref name=Kramer2016><pubmed>27377358</pubmed></ref>[22]。 | |||
=== 自然免疫応答の制御 === | === 自然免疫応答の制御 === | ||
C9orf72ノックアウトマウスでは、脾腫やリンパ節腫脹といった免疫系組織の腫大が認められ、さらにTypeⅠインターフェロンなどの炎症性サイトカインや自己抗体の産生が亢進していることから、免疫系における役割が注目されている<ref name=Haeusler2014><pubmed>24731978</pubmed></ref>[23]<ref name=CooperKnock2014><pubmed>24559645</pubmed></ref>[24]。特に、骨髄系細胞特異的C9orf72ノックアウトマウスにおいても、同様の異常が確認されることから、同細胞での作用が免疫系の制御に重要であると考えられる<ref name=Conlon2016><pubmed>27041224</pubmed></ref>[25]。その詳細な機序は未解明であるものの、C9orf72タンパク質がオートファジー活性を調節することで、STINGやmTORC1といった自然免疫応答に関与する分子の発現を制御する可能性が示唆されている<ref name=Conlon2016><pubmed>27041224</pubmed></ref>[25]<ref name=Lee2013><pubmed>23911329</pubmed></ref>[26]。 | |||
[[ファイル:Nagai C9orf72 Fig2.jpg|サムネイル|'''図2. C9orf72連鎖性ALS/FTDの病態機序'''<br>非翻訳領域のGGGGCCリピート配列の異常伸長によって、1) C9orf72タンパク質の発現が低下し、遺伝子の機能喪失をきたす。また、2) リピートRNAはRNA結合タンパク質を巻き込みながらRNA fociとして凝集し、これらのタンパク質の生理的機能を撹乱する。さらに、3) | [[ファイル:Nagai C9orf72 Fig2.jpg|サムネイル|'''図2. C9orf72連鎖性ALS/FTDの病態機序'''<br>非翻訳領域のGGGGCCリピート配列の異常伸長によって、1) C9orf72タンパク質の発現が低下し、遺伝子の機能喪失をきたす。また、2) リピートRNAはRNA結合タンパク質を巻き込みながらRNA fociとして凝集し、これらのタンパク質の生理的機能を撹乱する。さらに、3) リピートRNAから非古典的なRAN翻訳によって異常なDPRが産生される。これら3つの主要な病態メカニズムが協調的に神経変性に寄与する。]] | ||
== 疾患との関連 == | == 疾患との関連 == | ||
2006年に、欧米の家族性ALS/FTDコホートの連鎖解析によって、責任遺伝子領域として9番染色体短腕13.2-21.3が同定された<ref name=Morita2006><pubmed>16421333</pubmed></ref>[1]<ref name=Vance2006><pubmed>16495328</pubmed></ref>[2]。その後5年にわたる原因遺伝子変異探索の結果、2011年にC9orf72遺伝子の非翻訳領域内に存在するGGGGCCリピート配列の異常伸長が、原因遺伝子変異として同定された<ref name=DeJesusHernandez2011><pubmed>21944778</pubmed></ref>[3]<ref name=Renton2011><pubmed>21944779</pubmed></ref>[4]。この配列のリピート回数は、健常人では2~23回であるが、C9-ALS/FTDでは40~6000回に異常伸長している<ref name=Zhang2019><pubmed>30602785</pubmed></ref>[27]。リピート伸長変異は不完全浸透であり、一般人口においても約0.1~0.4%の頻度で認められる<ref name=Renton2011><pubmed>21944779</pubmed></ref>[4]<ref name=Cheng2019><pubmed>31209379</pubmed></ref>[28]。異常リピート回数の明確な閾値の決定は困難であり、リピート回数が24回から30回程度の中間伸長アレルも疾患発症リスクとなることが知られている<ref name=Zhu2020><pubmed>32234463</pubmed></ref>[29]。本リピート伸長変異は、家族性ALSの約40%、家族性FTDの約25%、孤発性ALSおよびFTDの5~6%で認められており、両疾患における最も頻度の高い遺伝的素因である<ref name=Meier2018><pubmed>29899452</pubmed></ref>[30]。なお、変異の頻度には地域差があり、欧米では高頻度であるのに対し、日本を含むアジア地域では稀である<ref name=Meier2018><pubmed>29899452</pubmed></ref>[30]。 | |||
臨床的には、ALS、FTD、あるいは両者の合併症状を来たすことが多い<ref name=Balendra2018><pubmed>30120348</pubmed></ref> | 臨床的には、ALS、FTD、あるいは両者の合併症状を来たすことが多い<ref name=Balendra2018><pubmed>30120348</pubmed></ref> | ||
[6]。病理学的には、RNA結合タンパク質TDP-43の核内からの消失および細胞質での異常凝集が認められる点で、大部分の孤発性ALSや一部のFTD症例と共通している<ref name=Balendra2018><pubmed>30120348</pubmed></ref>[6]。 | |||
病態機序については未解明な点が多く残るが、現在までに以下の3つの主要なメカニズムが明らかになり、これらは相互に作用して神経変性病態に寄与すると考えられている('''図2''')。 | 病態機序については未解明な点が多く残るが、現在までに以下の3つの主要なメカニズムが明らかになり、これらは相互に作用して神経変性病態に寄与すると考えられている('''図2''')。 | ||
=== C9orf72遺伝子の機能喪失 === | === C9orf72遺伝子の機能喪失 === | ||
C9-ALS/FTD患者の脳組織では、C9orf72転写産物およびタンパク質の発現がともに低下しており、ハプロ不全が神経変性病態に関与する可能性が示唆されている<ref name=DeJesusHernandez2011><pubmed>21944778</pubmed></ref>[3]<ref name=Liu2016><pubmed>27230221</pubmed></ref>[31]<ref name=Cheng2018><pubmed>29572487</pubmed></ref>[32]<ref name=Sellier2016><pubmed>27112498</pubmed></ref>[33]。先述の通り、C9orf72ノックアウトマウスでは、免疫系の異常を認められるものの、明らかな神経変性は生じない<ref name=Haeusler2014><pubmed>24731978</pubmed></ref>[23]<ref name=CooperKnock2014><pubmed>24559645</pubmed></ref>[24]。しかし、C9orf72遺伝子の発現低下はオートファジーの障害を介して、後述するDPRによる神経変性病態を増悪させることが報告されており、ハプロ不全と毒性獲得性のメカニズムが協調的に病態に関与していると考えられている<ref name=Boivin2020><pubmed>32042195</pubmed></ref>[34]<ref name=Moens2019><pubmed>30952804</pubmed></ref>[35]。 | |||
=== リピートRNA毒性 === | === リピートRNA毒性 === | ||
異常伸長したリピート配列は両方向性に転写され、GGGGCCリピートおよびその逆鎖であるCCCCGGリピートを含むリピートRNAが産生される。これらのリピートRNAはRNA結合タンパク質(RBP: RNA-binding protein)と異常な相互作用を獲得して、RBPの生理的な分布や機能を撹乱し得る。特に、リピートRNAは異常な高次構造を形成してRNA fociと呼ばれるRNA凝集体を形成し、種々のRBPと共凝集することが示されている<ref name=DeJesusHernandez2011><pubmed>21944778</pubmed></ref>[3]<ref name=Kramer2018><pubmed>29472620</pubmed></ref>[36]。例えば、GGGGCCリピートRNAが形成するRNA fociは、hnRNPH, hnRNPF, hnRNPA1, ALYREF, SRSF2, nucleolinといった多数のRBPと共局在し<ref name=Kramer2018><pubmed>29472620</pubmed></ref>[36]<ref name=Gendron2017><pubmed>28794246</pubmed></ref>[37]<ref name=Chew2019><pubmed>31358870</pubmed></ref> | |||
[38]<ref name=LopezGonzalez2019><pubmed>30804510</pubmed></ref>[39]hnRNPHのRNA fociへの共凝集は標的mRNAのスプライシング異常を来すことが報告されている<ref name=Gendron2017><pubmed>28794246</pubmed></ref>[37]。 | |||
[[ファイル:Nagai C9orf72 Fig3.jpg|サムネイル|'''図3. | [[ファイル:Nagai C9orf72 Fig3.jpg|サムネイル|'''図3. RAN翻訳'''<br>'''(A)''' RAN翻訳は、C9orf72連鎖性ALS/FTDなど、遺伝子の非翻訳領域内のリピート伸長変異を原因とするノンコーディングリピート病の病態に関与する非古典的な翻訳機構である。リピートRNAを鋳型として、AUGコドン非依存的に翻訳が開始し、異常なリピートペプチドを産生する。翻訳は全ての読み枠で進行する。<br>'''(B)''' C9orf72連鎖性ALS/FTD においては、GGGGCCおよびCCCCGGリピートRNAを鋳型として、RAN翻訳によって合計5種類のDPRが産生される。]] | ||
=== RAN翻訳とDPR毒性 === | |||
=== | 非翻訳領域由来のリピートRNAは、AUG開始コドンを欠くものの、RAN翻訳によって異常なDPRが産生される。RAN翻訳は鋳型となるRNAにリピート配列が含まれる際に、AUG開始コドン非存在下に翻訳が開始し、リピートペプチドが産生される非古典的な翻訳であり('''図3A''')、元々2011年に、同じく遺伝子非翻訳領域で異常伸長したリピート配列を原因とする脊髄小脳失調症8型(CAGリピート)および筋強直性ジストロフィー1型(CTGリピート)で発見された<ref name=Stopford2017><pubmed>28792425</pubmed></ref>[40]。AUGコドン非依存的な翻訳開始には、リピートRNAが形成する高次構造が重要であると示唆されている<ref name=Stopford2017><pubmed>28792425</pubmed></ref>[40]。3種類全ての読み枠で翻訳が進行するため、GGGGCCリピートRNAからはDPRとしてポリグリシン-アルギニン(polyGR)、ポリグリシン-アラニン(polyGA)、ポリグリシン-プロリン(polyGP)ペプチドが、CCCCGGリピートRNAからはポリグリシン-プロリン(polyGP)、ポリプロリン-アルギニン(polyPR)、ポリプロリン-アラニン(polyPA)ペプチドが産生され、実際に患者脳や髄液で検出される('''図3B''')<ref name=Mori2013><pubmed> 23393093 </pubmed></ref>[41]<ref name=GeneReviews></ref>[42]<ref name=DeJesusHernandez2017><pubmed>28863209</pubmed></ref>[43]<ref name=Mehta2013><pubmed>23280836</pubmed></ref>[44]<ref name=Alonso2015><pubmed>26268563</pubmed></ref>[45]。 | ||
polyGAは凝集性が高く、病理学的にC9-ALS/FTD患者脳で最も豊富に検出されるDPRである<ref name=McEachin2018><pubmed>29305472</pubmed></ref>[46]。培養細胞や動物モデルではpolyGAは弱い神経毒性を生じ、またプロテアソームと相互作用することからタンパク質分解系を障害する可能性がある<ref name=Mizielinska2014><pubmed>25023859</pubmed></ref>[47]<ref name=May2014><pubmed>24793942</pubmed></ref>[48]<ref name=Wen2014><pubmed>24909709</pubmed></ref>[49]。一方、アルギニンを含有するDPRであるpolyGRやpolyPRは、患者脳での発現はpolyGAと比較して少ないが<ref name=McEachin2018><pubmed>29305472</pubmed></ref>[46]、培養細胞や動物モデルで最も高度な毒性を来す<ref name=Mizielinska2014><pubmed>25023859</pubmed></ref>[47]<ref name=Ash2013><pubmed>23390130</pubmed></ref>[50]。その機序として、核細胞質輸送の障害<ref name=Zu2013><pubmed>23393093</pubmed></ref>[51]や、核小体やストレス顆粒といった膜のないオルガネラの動態の障害<ref name=Freibaum2015><pubmed>26168013</pubmed></ref>[52]<ref name=Kwon2014><pubmed>24842906</pubmed></ref>[53]が実験的に示唆されている。 | |||
なお、C9-ALS/FTD以外にも、ゲノム中に存在するリピート配列の異常伸長は60を超える遺伝性疾患の原因となり、多くが運動ニューロン疾患、小脳失調、認知症、末梢神経障害、てんかん、ミオパチーなどを来す神経筋疾患である(リピート病)。その中でも遺伝子の非翻訳領域内のリピート伸長変異を原因とする疾患群はノンコーディングリピート病として総称され、C9-ALS/FTDと共通する病態基盤が想定されている<ref name=Patel2015><pubmed>26412304</pubmed></ref>[54]<ref name=Molliex2015><pubmed>26317470</pubmed></ref>[55]。 | |||
==参考文献== | ==参考文献== | ||
2025年9月1日 (月) 23:21時点における版
藤野 雄三1,2、永井 義隆1,3
1. 近畿大学医学部 脳神経内科
2. 京都府立医科大学大学院医学研究科 脳神経内科学
3. 近畿大学ライフサイエンス研究所
DOI:10.14931/bsd.11350 原稿受付日:2025年7月7日 原稿完成日:2025年9月1日
担当編集委員:古屋敷 智之(東京科学大学大学院 医歯学総合研究科 薬理学分野)
英: Chromosome 9 open reading frame 72
略語: C9orf72
C9orf72はDENNドメインを有するタンパク質であり、種々の低分子量Gタンパク質の活性制御を介して、小胞輸送やオートファジー、自然免疫といった多岐にわたる生体機能に関与する。2011年に、本遺伝子の非翻訳領域内に存在するGGGGCCリピート配列の異常伸長が、筋萎縮性側索硬化症および前頭側頭型認知症における最も高頻度の原因遺伝子変異として同定され、その病態メカニズムが注目されている。現在、①C9orf72遺伝子の機能喪失、②転写された変異リピートRNAによる毒性、③リピート関連非AUG依存性翻訳によって産生されるリピートペプチドによる毒性が、協調的に神経変性病態に寄与すると考えられている。
歴史的推移
C9orf72遺伝子は、神経変性疾患である筋萎縮性側索硬化症(ALS: amyotrophic lateral sclerosis)および前頭側頭型認知症(FTD: frontotemporal dementia)の原因となる代表的な遺伝子の1つとして、研究が進展してきた。
2006年、家族性ALS/FTD家系を用いた連鎖解析によって、責任遺伝子領域が9番染色体短腕に同定され[1][1][2][2]、その後の遺伝子探索によって、2011年にC9orf72遺伝子非翻訳領域内のGGGGCC 6塩基リピート配列の異常伸長(正常: 2-24回リピート, 疾患: 60-4000回リピート)が、原因遺伝子変異として同定された[3][3][4][4]。このリピート伸長変異は、欧米のALSFTDコホートにおいて最も頻度の高い原因遺伝子変異であることが明らかとなり、同遺伝子の生理的機能やリピート伸長変異による病態メカニズムに注目が集まり、研究が展開されている。
生理的機能に関しては未解明の点が多いものの、分子系統解析では本遺伝子は真核生物の進化初期に出現し、哺乳類を含む広範な生物種で保存されていることから、生物学的に重要な役割を担うと想定されている[5][5]。特に、C9orf72タンパク質はDENN(differentially expressed in normal and neoplastic cells)ドメインを有し、本ドメインを有する一群のタンパク質はRabファミリーを中心とした低分子量Gタンパク質の活性制御に関与することから、小胞輸送やオートファジーとの関連が注目されている。近年では、自然免疫応答の制御機能についても示唆されている(後述)[5][5]。
C9orf72遺伝子連鎖性ALS/FTD(C9-ALS/FTD)の病態メカニズムに関しては、当初、GGGGCCリピート伸長変異による遺伝子機能の喪失や、転写された異常伸長リピート配列を含む変異RNA(リピートRNA)による毒性が想定されていた。しかし2013年に、リピートRNAから非古典的なリピート関連非AUG依存性(RAN: repeat-associated non-AUG)翻訳によって病的なジペプチドリピートペプチドタンパク質(DPR: dipeptide repeat protein)が産生され、実際にDPRが患者脳に蓄積することが明らかになり、現在ではこれら3つの病態メカニズムが協調的に神経変性病態に寄与すると考えられている[6][6]。
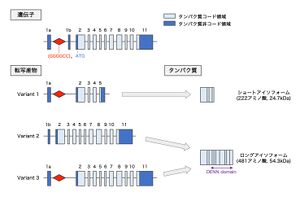
C9orf72遺伝子は12のエクソンから構成され、このうちエクソン2~11がタンパク質をコードする。GGGGCCリピート配列はエクソン1aと1bの間の非翻訳領域に存在する。3種類の転写産物と2種類のタンパク質アイソフォームが存在し、Variant 1はショートアイソフォームに、Variant2および3はロングアイソフォームにそれぞれ翻訳される。Variant 1およびVariant 3はGGGGCCリピート配列を含む。ロングアイソフォームにはDENNドメインが含まれる。
構造
遺伝子
ヒトでは染色体9p21.2に存在する41kbの遺伝子で、12のエクソン(エクソン 1a、1b、2~11)から構成される[5][5][6][6]。翻訳開始に利用されるATGコドンはエクソン2に存在し、エクソン2~11がC9orf72タンパク質をコードする。エクソン1aおよび1bはタンパク質をコードしない非翻訳領域である。ALS, FTDと関連するGGGGCCリピート配列は、エクソン1aと1bの間の非翻訳領域に存在する(図1)[5][5][6][6]。
転写産物
転写開始点および転写終結点を有し、転写産物については3種類のバリアントが報告されている。Variant 1(V1)はエクソン1a、2~5から構成される短鎖転写産物である。Variant 2(V2)はエクソン1b、2~11、Variant 3(V3)はエクソン1a、2~11から構成される長鎖転写産物である[5][5][6][6]。GGGGCCリピート配列は、V1およびV3ではイントロン領域に存在する一方で、V2では転写開始点直上流に存在し、転写産物自体には含まれない(図1)[5][5][6][6]。
タンパク質
222アミノ酸から構成されるショートアイソフォーム(分子量24.7kDa)と、481アミノ酸から構成されるロングアイソフォーム(分子量54.3kDa)の2種類のアイソフォームが存在する。ショートアイソフォームはV1転写産物から、ロングアイソフォームはV2およびV3転写産物から翻訳される(図1)[5][5][6][6]。ロングアイソフォームはDENNドメインを有し、主にGDP/GTP交換因子(GEF: GDP/GTP exchange factor)として低分子量Gタンパク質の活性制御を担うDENNファミリータンパク質の一員と考えられている[7][7][8][8]。
発現
RNAレベル
C9orf72転写産物はほとんどのヒト組織において発現が認められるが、特に脳、血液細胞、脾臓、脾臓、卵巣などにおいて高発現を示す[9][9]。脳内では小脳おける発現が顕著であり、血液細胞ではリンパ球よりも骨髄系細胞において高い発現が報告されている [4][4][10][10]。
タンパク質レベル
C9orf72タンパク質もまた、ヒトのほとんどの組織で発現している。マウスを用いた解析では、脳、脊髄、および脾臓で特に高い発現を認めている[11][11]。
培養細胞では、C9orf72タンパク質はリソソームと共局在することが報告されている[12][12]。ヒト脳組織における免疫組織化学では、ショートアイソフォームが主に神経細胞の核膜に分布する一方で、ロングアイソフォームは細胞質全体に広く分布し、神経突起や細胞体に点状構造を形成することが確認されている[13][13]。また、ヒトiPS細胞由来運動ニューロンを用いた検討により、C9orf72タンパク質がシナプス小胞にも局在することが示唆されている[11][11]。
機能
DENNファミリータンパク質の1つであるC9orf72タンパク質は、同じくDENNドメインを有するSMCR8、およびWDR41と複合体を形成する[14][14][15][15][16][16][17][17]。この複合体はGTPase活性化タンパク質(GAP: GTPase activating protein)として[16][16][17][17]。C9orf72タンパク質が相互作用する代表的な低分子量G タンパク質は、小胞輸送に関与するRabファミリータンパク質であり、これによってオートファジーやシナプス機能などの様々な生体反応を制御することが明らかになっている。また近年では、自然免疫応答の制御においても重要な役割を担うことが示唆されている。
オートファジーの制御
培養神経細胞系やヒト脊髄運動ニューロンにおいて、C9orf72タンパク質はRab1、Rab5、Rab7、Rab11などのRabファミリータンパク質と共局在する[18][18]。また培養細胞を用いた免疫沈降法による解析では、Rab8aやRab39b、FIP200/Ulk1/ATG13/ATG101複合体と相互作用することが報告されている[15][15][16][16]。これらの分子との相互作用を介して、C9orf72タンパク質はオートファジーの開始、基質のリクルートメント、隔離膜の伸長と閉鎖、オートファゴソームとリソソームの融合など、オートファジーの各段階を制御すると考えられている[19][19]。実際に、C9orf72遺伝子をノックダウンした培養細胞では、オートファジーの誘導阻害や異常なオートファゴソームの蓄積が観察されている[16][16][18][18]。
シナプス機能の制御
C9orf72タンパク質が相互作用するRab39bは、AMPA型グルタミン酸受容体の小胞輸送およびリサイクリングに関与し、受容体のサブユニット構成やシナプスの電気生理学的特性を調節する[20][20]。C9orf72ノックアウトマウスでは、シナプス後部においてRab39bの発現が低下し、AMPA型グルタミン酸受容体GluR1サブユニットの発現が亢進することが報告されており、C9orf72タンパク質がRab39bの発現や活性を制御することでシナプス機能を調節している可能性が示唆されている[21][21]。
細胞骨格の制御
C9orf72タンパク質は、Rabファミリータンパク質とは異なる低分子量G タンパク質であるArfファミリータンパク質とも相互作用し、特にArf6との相互作用を介して、運動ニューロンにおけるアクチン細胞骨格の動態を制御することが報告されている[][22][22]。培養神経細胞ではC9orf72遺伝子の過剰発現によって軸索が伸長し、ノックダウンによって短縮することが確認されている[22][22]。
自然免疫応答の制御
C9orf72ノックアウトマウスでは、脾腫やリンパ節腫脹といった免疫系組織の腫大が認められ、さらにTypeⅠインターフェロンなどの炎症性サイトカインや自己抗体の産生が亢進していることから、免疫系における役割が注目されている[23][23][24][24]。特に、骨髄系細胞特異的C9orf72ノックアウトマウスにおいても、同様の異常が確認されることから、同細胞での作用が免疫系の制御に重要であると考えられる[25][25]。その詳細な機序は未解明であるものの、C9orf72タンパク質がオートファジー活性を調節することで、STINGやmTORC1といった自然免疫応答に関与する分子の発現を制御する可能性が示唆されている[25][25][26][26]。
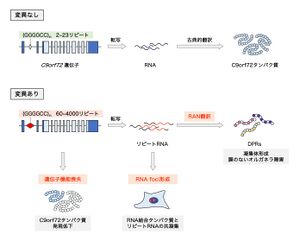
非翻訳領域のGGGGCCリピート配列の異常伸長によって、1) C9orf72タンパク質の発現が低下し、遺伝子の機能喪失をきたす。また、2) リピートRNAはRNA結合タンパク質を巻き込みながらRNA fociとして凝集し、これらのタンパク質の生理的機能を撹乱する。さらに、3) リピートRNAから非古典的なRAN翻訳によって異常なDPRが産生される。これら3つの主要な病態メカニズムが協調的に神経変性に寄与する。
疾患との関連
2006年に、欧米の家族性ALS/FTDコホートの連鎖解析によって、責任遺伝子領域として9番染色体短腕13.2-21.3が同定された[1][1][2][2]。その後5年にわたる原因遺伝子変異探索の結果、2011年にC9orf72遺伝子の非翻訳領域内に存在するGGGGCCリピート配列の異常伸長が、原因遺伝子変異として同定された[3][3][4][4]。この配列のリピート回数は、健常人では2~23回であるが、C9-ALS/FTDでは40~6000回に異常伸長している[27][27]。リピート伸長変異は不完全浸透であり、一般人口においても約0.1~0.4%の頻度で認められる[4][4][28][28]。異常リピート回数の明確な閾値の決定は困難であり、リピート回数が24回から30回程度の中間伸長アレルも疾患発症リスクとなることが知られている[29][29]。本リピート伸長変異は、家族性ALSの約40%、家族性FTDの約25%、孤発性ALSおよびFTDの5~6%で認められており、両疾患における最も頻度の高い遺伝的素因である[30][30]。なお、変異の頻度には地域差があり、欧米では高頻度であるのに対し、日本を含むアジア地域では稀である[30][30]。
臨床的には、ALS、FTD、あるいは両者の合併症状を来たすことが多い[6] [6]。病理学的には、RNA結合タンパク質TDP-43の核内からの消失および細胞質での異常凝集が認められる点で、大部分の孤発性ALSや一部のFTD症例と共通している[6][6]。
病態機序については未解明な点が多く残るが、現在までに以下の3つの主要なメカニズムが明らかになり、これらは相互に作用して神経変性病態に寄与すると考えられている(図2)。
C9orf72遺伝子の機能喪失
C9-ALS/FTD患者の脳組織では、C9orf72転写産物およびタンパク質の発現がともに低下しており、ハプロ不全が神経変性病態に関与する可能性が示唆されている[3][3][31][31][32][32][33][33]。先述の通り、C9orf72ノックアウトマウスでは、免疫系の異常を認められるものの、明らかな神経変性は生じない[23][23][24][24]。しかし、C9orf72遺伝子の発現低下はオートファジーの障害を介して、後述するDPRによる神経変性病態を増悪させることが報告されており、ハプロ不全と毒性獲得性のメカニズムが協調的に病態に関与していると考えられている[34][34][35][35]。
リピートRNA毒性
異常伸長したリピート配列は両方向性に転写され、GGGGCCリピートおよびその逆鎖であるCCCCGGリピートを含むリピートRNAが産生される。これらのリピートRNAはRNA結合タンパク質(RBP: RNA-binding protein)と異常な相互作用を獲得して、RBPの生理的な分布や機能を撹乱し得る。特に、リピートRNAは異常な高次構造を形成してRNA fociと呼ばれるRNA凝集体を形成し、種々のRBPと共凝集することが示されている[3][3][36][36]。例えば、GGGGCCリピートRNAが形成するRNA fociは、hnRNPH, hnRNPF, hnRNPA1, ALYREF, SRSF2, nucleolinといった多数のRBPと共局在し[36][36][37][37][38] [38][39][39]hnRNPHのRNA fociへの共凝集は標的mRNAのスプライシング異常を来すことが報告されている[37][37]。
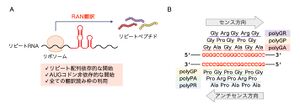
(A) RAN翻訳は、C9orf72連鎖性ALS/FTDなど、遺伝子の非翻訳領域内のリピート伸長変異を原因とするノンコーディングリピート病の病態に関与する非古典的な翻訳機構である。リピートRNAを鋳型として、AUGコドン非依存的に翻訳が開始し、異常なリピートペプチドを産生する。翻訳は全ての読み枠で進行する。
(B) C9orf72連鎖性ALS/FTD においては、GGGGCCおよびCCCCGGリピートRNAを鋳型として、RAN翻訳によって合計5種類のDPRが産生される。
RAN翻訳とDPR毒性
非翻訳領域由来のリピートRNAは、AUG開始コドンを欠くものの、RAN翻訳によって異常なDPRが産生される。RAN翻訳は鋳型となるRNAにリピート配列が含まれる際に、AUG開始コドン非存在下に翻訳が開始し、リピートペプチドが産生される非古典的な翻訳であり(図3A)、元々2011年に、同じく遺伝子非翻訳領域で異常伸長したリピート配列を原因とする脊髄小脳失調症8型(CAGリピート)および筋強直性ジストロフィー1型(CTGリピート)で発見された[40][40]。AUGコドン非依存的な翻訳開始には、リピートRNAが形成する高次構造が重要であると示唆されている[40][40]。3種類全ての読み枠で翻訳が進行するため、GGGGCCリピートRNAからはDPRとしてポリグリシン-アルギニン(polyGR)、ポリグリシン-アラニン(polyGA)、ポリグリシン-プロリン(polyGP)ペプチドが、CCCCGGリピートRNAからはポリグリシン-プロリン(polyGP)、ポリプロリン-アルギニン(polyPR)、ポリプロリン-アラニン(polyPA)ペプチドが産生され、実際に患者脳や髄液で検出される(図3B)[41][41][42][42][43][43][44][44][45][45]。
polyGAは凝集性が高く、病理学的にC9-ALS/FTD患者脳で最も豊富に検出されるDPRである[46][46]。培養細胞や動物モデルではpolyGAは弱い神経毒性を生じ、またプロテアソームと相互作用することからタンパク質分解系を障害する可能性がある[47][47][48][48][49][49]。一方、アルギニンを含有するDPRであるpolyGRやpolyPRは、患者脳での発現はpolyGAと比較して少ないが[46][46]、培養細胞や動物モデルで最も高度な毒性を来す[47][47][50][50]。その機序として、核細胞質輸送の障害[51][51]や、核小体やストレス顆粒といった膜のないオルガネラの動態の障害[52][52][53][53]が実験的に示唆されている。
なお、C9-ALS/FTD以外にも、ゲノム中に存在するリピート配列の異常伸長は60を超える遺伝性疾患の原因となり、多くが運動ニューロン疾患、小脳失調、認知症、末梢神経障害、てんかん、ミオパチーなどを来す神経筋疾患である(リピート病)。その中でも遺伝子の非翻訳領域内のリピート伸長変異を原因とする疾患群はノンコーディングリピート病として総称され、C9-ALS/FTDと共通する病態基盤が想定されている[54][54][55][55]。
参考文献
- ↑ 1.0 1.1
Morita, M., Al-Chalabi, A., Andersen, P.M., Hosler, B., Sapp, P., Englund, E., ..., & Brown, R.H. (2006).
A locus on chromosome 9p confers susceptibility to ALS and frontotemporal dementia. Neurology, 66(6), 839-44. [PubMed:16421333] [WorldCat] [DOI] - ↑ 2.0 2.1
Vance, C., Al-Chalabi, A., Ruddy, D., Smith, B.N., Hu, X., Sreedharan, J., ..., & Shaw, C.E. (2006).
Familial amyotrophic lateral sclerosis with frontotemporal dementia is linked to a locus on chromosome 9p13.2-21.3. Brain : a journal of neurology, 129(Pt 4), 868-76. [PubMed:16495328] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2 3.3
DeJesus-Hernandez, M., Mackenzie, I.R., Boeve, B.F., Boxer, A.L., Baker, M., Rutherford, N.J., ..., & Rademakers, R. (2011).
Expanded GGGGCC hexanucleotide repeat in noncoding region of C9ORF72 causes chromosome 9p-linked FTD and ALS. Neuron, 72(2), 245-56. [PubMed:21944778] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1 4.2 4.3
Renton, A.E., Majounie, E., Waite, A., Simón-Sánchez, J., Rollinson, S., Gibbs, J.R., ..., & Traynor, B.J. (2011).
A hexanucleotide repeat expansion in C9ORF72 is the cause of chromosome 9p21-linked ALS-FTD. Neuron, 72(2), 257-68. [PubMed:21944779] [PMC] [WorldCat] [DOI] - ↑ 5.0 5.1 5.2 5.3 5.4 5.5 5.6
Smeyers, J., Banchi, E.G., & Latouche, M. (2021).
C9ORF72: What It Is, What It Does, and Why It Matters. Frontiers in cellular neuroscience, 15, 661447. [PubMed:34025358] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2 6.3 6.4 6.5 6.6 6.7
Balendra, R., & Isaacs, A.M. (2018).
C9orf72-mediated ALS and FTD: multiple pathways to disease. Nature reviews. Neurology, 14(9), 544-558. [PubMed:30120348] [PMC] [WorldCat] [DOI] - ↑
Xie, Y., Tang, J., Xie, X., Li, X., Huang, J., Fei, Y., ..., & Chen, L. (2019).
An asymmetric allelic interaction drives allele transmission bias in interspecific rice hybrids. Nature communications, 10(1), 2501. [PubMed:31175302] [PMC] [WorldCat] [DOI] - ↑
Goldmann, T., Wieghofer, P., Jordão, M.J., Prutek, F., Hagemeyer, N., Frenzel, K., ..., & Prinz, M. (2016).
Origin, fate and dynamics of macrophages at central nervous system interfaces. Nature immunology, 17(7), 797-805. [PubMed:27135602] [PMC] [WorldCat] [DOI] - ↑
O'Rourke, J.G., Bogdanik, L., Muhammad, A.K.M.G., Gendron, T.F., Kim, K.J., Austin, A., ..., & Baloh, R.H. (2015).
C9orf72 BAC Transgenic Mice Display Typical Pathologic Features of ALS/FTD. Neuron, 88(5), 892-901. [PubMed:26637796] [PMC] [WorldCat] [DOI] - ↑
Taniguchi, K., & Karin, M. (2018).
NF-κB, inflammation, immunity and cancer: coming of age. Nature reviews. Immunology, 18(5), 309-324. [PubMed:29379212] [WorldCat] [DOI] - ↑ 11.0 11.1
Blazquez, L., Emmett, W., Faraway, R., Pineda, J.M.B., Bajew, S., Gohr, A., ..., & Ule, J. (2018).
Exon Junction Complex Shapes the Transcriptome by Repressing Recursive Splicing. Molecular cell, 72(3), 496-509.e9. [PubMed:30388411] [PMC] [WorldCat] [DOI] - ↑
Tsuzuki, S., Park, S.H., Eber, M.R., Peters, C.M., & Shiozawa, Y. (2016).
Skeletal complications in cancer patients with bone metastases. International journal of urology : official journal of the Japanese Urological Association, 23(10), 825-832. [PubMed:27488133] [PMC] [WorldCat] [DOI] - ↑
Hyslop, L.A., Blakeley, P., Craven, L., Richardson, J., Fogarty, N.M., Fragouli, E., ..., & Herbert, M. (2016).
Towards clinical application of pronuclear transfer to prevent mitochondrial DNA disease. Nature, 534(7607), 383-6. [PubMed:27281217] [PMC] [WorldCat] [DOI] - ↑
Schweitzer, P., Husky, M., Allard, M., Amieva, H., Pérès, K., Foubert-Samier, A., ..., & Swendsen, J. (2017).
Feasibility and validity of mobile cognitive testing in the investigation of age-related cognitive decline. International journal of methods in psychiatric research, 26(3). [PubMed:27539327] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1
Lu, Z., Zhou, Y., & Liu, B. (2019).
Preparation of chitosan microcarriers by high voltage electrostatic field and freeze drying. Journal of bioscience and bioengineering, 128(4), 504-509. [PubMed:31029540] [WorldCat] [DOI] - ↑ 16.0 16.1 16.2 16.3
Kovacev, N., Antić, J., Gvozdenović, N., Obradović, M., Vranješ, M., & Milankov, M. (2015).
Patellar tendon rupture--treatment results. Medicinski pregled, 68(1-2), 22-8. [PubMed:26012240] [WorldCat] [DOI] - ↑ 17.0 17.1
Ruiz de los Mozos, I., Vergara-Irigaray, M., Segura, V., Villanueva, M., Bitarte, N., Saramago, M., ..., & Toledo-Arana, A. (2013).
Base pairing interaction between 5'- and 3'-UTRs controls icaR mRNA translation in Staphylococcus aureus. PLoS genetics, 9(12), e1004001. [PubMed:24367275] [PMC] [WorldCat] [DOI] - ↑ 18.0 18.1
Ahmed-Leitao, F., Spies, G., van den Heuvel, L., & Seedat, S. (2016).
Hippocampal and amygdala volumes in adults with posttraumatic stress disorder secondary to childhood abuse or maltreatment: A systematic review. Psychiatry research. Neuroimaging, 256, 33-43. [PubMed:27669407] [WorldCat] [DOI] - ↑
Jiang, J., Zhu, Q., Gendron, T.F., Saberi, S., McAlonis-Downes, M., Seelman, A., ..., & Lagier-Tourenne, C. (2016).
Gain of Toxicity from ALS/FTD-Linked Repeat Expansions in C9ORF72 Is Alleviated by Antisense Oligonucleotides Targeting GGGGCC-Containing RNAs. Neuron, 90(3), 535-50. [PubMed:27112497] [PMC] [WorldCat] [DOI] - ↑
Xu, D., Wang, L., Wang, G., & Zheng, S. (2016).
Domibacillus antri sp. nov., isolated from the soil of a cave. International journal of systematic and evolutionary microbiology, 66(7), 2502-2508. [PubMed:27073919] [WorldCat] [DOI] - ↑
Kedersha, N., Panas, M.D., Achorn, C.A., Lyons, S., Tisdale, S., Hickman, T., ..., & Anderson, P. (2016).
G3BP-Caprin1-USP10 complexes mediate stress granule condensation and associate with 40S subunits. The Journal of cell biology, 212(7), 845-60. [PubMed:27022092] [PMC] [WorldCat] [DOI] - ↑ 22.0 22.1
Hudlikar, R.R., Venkadakrishnan, V.B., Kumar, R., Thorat, R.A., Kannan, S., Ingle, A.D., ..., & Mahimkar, M.B. (2017).
Polymeric black tea polyphenols (PBPs) inhibit benzo(a)pyrene and 4-(methylnitrosamino)-1-(3-pyridyl)-1- butanone-induced lung carcinogenesis potentially through down-regulation of p38 and Akt phosphorylation in A/J mice. Molecular carcinogenesis, 56(2), 625-640. [PubMed:27377358] [WorldCat] [DOI] - ↑ 23.0 23.1
Ali, S., & Garcia, J.M. (2014).
Sarcopenia, cachexia and aging: diagnosis, mechanisms and therapeutic options - a mini-review. Gerontology, 60(4), 294-305. [PubMed:24731978] [PMC] [WorldCat] [DOI] - ↑ 24.0 24.1
Waite, A.J., Bäumer, D., East, S., Neal, J., Morris, H.R., Ansorge, O., & Blake, D.J. (2014).
Reduced C9orf72 protein levels in frontal cortex of amyotrophic lateral sclerosis and frontotemporal degeneration brain with the C9ORF72 hexanucleotide repeat expansion. Neurobiology of aging, 35(7), 1779.e5-1779.e13. [PubMed:24559645] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1
Leenay, R.T., Maksimchuk, K.R., Slotkowski, R.A., Agrawal, R.N., Gomaa, A.A., Briner, A.E., ..., & Beisel, C.L. (2016).
Identifying and Visualizing Functional PAM Diversity across CRISPR-Cas Systems. Molecular cell, 62(1), 137-47. [PubMed:27041224] [PMC] [WorldCat] [DOI] - ↑
Tepper, R.G., Ashraf, J., Kaletsky, R., Kleemann, G., Murphy, C.T., & Bussemaker, H.J. (2013).
PQM-1 complements DAF-16 as a key transcriptional regulator of DAF-2-mediated development and longevity. Cell, 154(3), 676-690. [PubMed:23911329] [PMC] [WorldCat] [DOI] - ↑
Bainbridge, W.A., Hall, E.H., & Baker, C.I. (2019).
Drawings of real-world scenes during free recall reveal detailed object and spatial information in memory. Nature communications, 10(1), 5. [PubMed:30602785] [PMC] [WorldCat] [DOI] - ↑
Gunner, G., Cheadle, L., Johnson, K.M., Ayata, P., Badimon, A., Mondo, E., ..., & Schafer, D.P. (2019).
Sensory lesioning induces microglial synapse elimination via ADAM10 and fractalkine signaling. Nature neuroscience, 22(7), 1075-1088. [PubMed:31209379] [PMC] [WorldCat] [DOI] - ↑
Blanke, P., Leipsic, J.A., Popma, J.J., Yakubov, S.J., Deeb, G.M., Gada, H., ..., & Evolut Low Risk LTI Substudy Investigators (2020).
Bioprosthetic Aortic Valve Leaflet Thickening in the Evolut Low Risk Sub-Study. Journal of the American College of Cardiology, 75(19), 2430-2442. [PubMed:32234463] [WorldCat] [DOI] - ↑ 30.0 30.1
Venot, Q., Blanc, T., Rabia, S.H., Berteloot, L., Ladraa, S., Duong, J.P., ..., & Canaud, G. (2018).
Targeted therapy in patients with PIK3CA-related overgrowth syndrome. Nature, 558(7711), 540-546. [PubMed:29899452] [PMC] [WorldCat] [DOI] - ↑
Chen, X.L., Yu, R., Wu, X.Y., Liang, D., Jia, J.H., & Lu, C.Z. (2016).
Correction: A strongly greenish-blue-emitting Cu4Cl4 cluster with an efficient spin-orbit coupling (SOC): fast phosphorescence versus thermally activated delayed fluorescence. Chemical communications (Cambridge, England), 52(49), 7738. [PubMed:27230221] [WorldCat] [DOI] - ↑
Wang, S., Peng, Z., Wang, S., Yang, L., Chen, Y., Kong, X., ..., & Zhang, L. (2018).
Author Correction: KRAB-type zinc-finger proteins PITA and PISA specifically regulate p53-dependent glycolysis and mitochondrial respiration. Cell research, 28(5), 605. [PubMed:29572487] [PMC] [WorldCat] [DOI] - ↑
Oettl, L.L., Ravi, N., Schneider, M., Scheller, M.F., Schneider, P., Mitre, M., ..., & Kelsch, W. (2016).
Oxytocin Enhances Social Recognition by Modulating Cortical Control of Early Olfactory Processing. Neuron, 90(3), 609-21. [PubMed:27112498] [PMC] [WorldCat] [DOI] - ↑
Staniszewka, S. (2020).
A patient-researcher partnership for rare cancer research. Nature medicine, 26(2), 164-165. [PubMed:32042195] [WorldCat] [DOI] - ↑
Szabo, P.A., Miron, M., & Farber, D.L. (2019).
Location, location, location: Tissue resident memory T cells in mice and humans. Science immunology, 4(34). [PubMed:30952804] [PMC] [WorldCat] [DOI] - ↑ 36.0 36.1
Huang, Y., Xu, Z., Xiong, S., Sun, F., Qin, G., Hu, G., ..., & Peng, B. (2018).
Repopulated microglia are solely derived from the proliferation of residual microglia after acute depletion. Nature neuroscience, 21(4), 530-540. [PubMed:29472620] [WorldCat] [DOI] - ↑ 37.0 37.1
Kokubo, Y., Banack, S.A., Morimoto, S., Murayama, S., Togashi, T., Metcalf, J.S., ..., & Kuzuhara, S. (2017).
β-N-methylamino-l-alanine analysis in the brains of patients with Kii ALS/PDC. Neurology, 89(10), 1091-1092. [PubMed:28794246] [WorldCat] [DOI] - ↑
Wang, T.J., Chang, H.W., Chen, J.S., & Chiang, H.P. (2019).
Nanotip-assisted photoreduction of silver nanostructures on chemically patterned ferroelectric crystals for surface enhanced Raman scattering. Scientific reports, 9(1), 10962. [PubMed:31358870] [PMC] [WorldCat] [DOI] - ↑
Kang, K., Li, T., Sohn, E., Shan, J., & Mak, K.F. (2019).
Nonlinear anomalous Hall effect in few-layer WTe2. Nature materials, 18(4), 324-328. [PubMed:30804510] [WorldCat] [DOI] - ↑ 40.0 40.1
Lee, H.J., & Kim, J.Y. (2017).
Correspondence. Retina (Philadelphia, Pa.), 37(9), e100-e101. [PubMed:28792425] [WorldCat] [DOI] - ↑
Mori, K., Weng, S.M., Arzberger, T., May, S., Rentzsch, K., Kremmer, E., ..., & Edbauer, D. (2013).
The C9orf72 GGGGCC repeat is translated into aggregating dipeptide-repeat proteins in FTLD/ALS. Science (New York, N.Y.), 339(6125), 1335-8. [PubMed:23393093] [WorldCat] [DOI] - ↑ 引用エラー: 無効な
<ref>タグです。「GeneReviews」という名前の注釈に対するテキストが指定されていません - ↑
Ianella, P., Albuquerque, M.S.M., Paiva, S.R., Egito, A.A., Almeida, L.D., Sereno, F.T.P.S., ..., & McManus, C.M. (2017).
D-loop haplotype diversity in Brazilian horse breeds. Genetics and molecular biology, 40(3), 604-609. [PubMed:28863209] [PMC] [WorldCat] [DOI] - ↑
Hills, N.K., Johnston, S.C., Sidney, S., Zielinski, B.A., & Fullerton, H.J. (2012).
Recent trauma and acute infection as risk factors for childhood arterial ischemic stroke. Annals of neurology, 72(6), 850-8. [PubMed:23280836] [PMC] [WorldCat] [DOI] - ↑
Wang, C., Yu, C., Liu, J., Sun, F., Teng, L., & Li, Y. (2015).
Stabilization of Human Immunoglobulin G Encapsulated within Biodegradable Poly (Cyclohexane-1, 4-diyl Acetone Dimethylene Ketal) (PCADK)/ Poly (Lactic-co-Glycolic Acid) (PLGA) Blend Microspheres. Protein and peptide letters, 22(11), 963-71. [PubMed:26268563] [WorldCat] [DOI] - ↑ 46.0 46.1
(2018).
Disease surveillance in England and Wales, December 2017. The Veterinary record, 182(1), 11-15. [PubMed:29305472] [WorldCat] [DOI] - ↑ 47.0 47.1
Meyer, G. (2014).
Effective diagnosis and treatment of pulmonary embolism: Improving patient outcomes. Archives of cardiovascular diseases, 107(6-7), 406-14. [PubMed:25023859] [WorldCat] [DOI] - ↑
Jain, S., Vashistt, J., & Changotra, H. (2014).
Rotaviruses: is their surveillance needed? Vaccine, 32(27), 3367-78. [PubMed:24793942] [WorldCat] [DOI] - ↑
Sujada, N., Sungthong, R., & Lumyong, S. (2014).
Termite nests as an abundant source of cultivable actinobacteria for biotechnological purposes. Microbes and environments, 29(2), 211-9. [PubMed:24909709] [PMC] [WorldCat] [DOI] - ↑
Joubert, R., Vignaud, A., Le, M., Moal, C., Messaddeq, N., & Buj-Bello, A. (2013).
Site-specific Mtm1 mutagenesis by an AAV-Cre vector reveals that myotubularin is essential in adult muscle. Human molecular genetics, 22(9), 1856-66. [PubMed:23390130] [WorldCat] [DOI] - ↑
Mori, K., Weng, S.M., Arzberger, T., May, S., Rentzsch, K., Kremmer, E., ..., & Edbauer, D. (2013).
The C9orf72 GGGGCC repeat is translated into aggregating dipeptide-repeat proteins in FTLD/ALS. Science (New York, N.Y.), 339(6125), 1335-8. [PubMed:23393093] [WorldCat] [DOI] - ↑
Hu, X., Deutsch, A.J., Lenz, T.L., Onengut-Gumuscu, S., Han, B., Chen, W.M., ..., & Raychaudhuri, S. (2015).
Additive and interaction effects at three amino acid positions in HLA-DQ and HLA-DR molecules drive type 1 diabetes risk. Nature genetics, 47(8), 898-905. [PubMed:26168013] [PMC] [WorldCat] [DOI] - ↑
Young, S.G., Jung, H.J., Lee, J.M., & Fong, L.G. (2014).
Nuclear lamins and neurobiology. Molecular and cellular biology, 34(15), 2776-85. [PubMed:24842906] [PMC] [WorldCat] [DOI] - ↑
Pontel, L.B., Rosado, I.V., Burgos-Barragan, G., Garaycoechea, J.I., Yu, R., Arends, M.J., ..., & Patel, K.J. (2015).
Endogenous Formaldehyde Is a Hematopoietic Stem Cell Genotoxin and Metabolic Carcinogen. Molecular cell, 60(1), 177-88. [PubMed:26412304] [PMC] [WorldCat] [DOI] - ↑
Patel, A., Lee, H.O., Jawerth, L., Maharana, S., Jahnel, M., Hein, M.Y., ..., & Alberti, S. (2015).
A Liquid-to-Solid Phase Transition of the ALS Protein FUS Accelerated by Disease Mutation. Cell, 162(5), 1066-77. [PubMed:26317470] [WorldCat] [DOI]