「ロドプシン」の版間の差分
細編集の要約なし |
細 →翻訳後修飾 |
||
| (3人の利用者による、間の41版が非表示) | |||
| 1行目: | 1行目: | ||
{{PBB|geneid=6010}}別名:桿体視物質、視紅 英:Rhodopsin, Visual purple | |||
(概要をここに御願い致します。元からあった文章を短くして書いてみましたので、物性などを付け加えて頂けば幸いです) 視細胞には光を受容するために特別に分化したタンパク質(光受容タンパク質)が含まれ、視物質と呼ぶ<ref>'''Dowling J'''<br>The Retina: An approachable part of the brain<br>''The Belknap Press of Harvard Univ. Press'':1987</ref><ref name><pubmed> 9893707 </pubmed></ref>。そのうち桿体に含まれる視物質(桿体視物質)をロドプシンと呼ぶ。桿体の外節にはパンケーキ状の円盤膜(disk membrane)が何層にも重なっており、ロドプシンはこの円盤膜に埋め込まれて存在している。微弱光でも効率よく受容できるように、ロドプシンは桿体の円盤膜に大量に発現している(円盤膜面積の50%以上がロドプシン分子である)。光を受容したロドプシンは構造変化を起こし、[[Gタンパク質]]を介して[[細胞内シグナル伝達系]]を駆動する。この際にロドプシンの1分子は数百のGタンパク質を活性化し、光情報が増幅される。シグナル伝達系の下流でもさらに増幅機構が働き、その結果として、桿体はわずか1個の光子を受容しただけで応答することができる。 | |||
== ロドプシン分子 == | |||
[[wikipedia:JA:脊椎動物|脊椎動物]]の眼には2種類の[[視細胞]]、[[桿体]]と[[錐体]]が存在し、それぞれ、[[暗所視]]、[[明所視]]を司る。両視細胞には光を受容するために特別に分化したタンパク質(光受容タンパク質)が含まれ、それらを視物質と呼ぶ<ref>'''Dowling J'''<br>The Retina: An approachable part of the brain<br>''The Belknap Press of Harvard Univ. Press'':1987</ref><ref name><pubmed> 9893707 </pubmed></ref>。桿体に含まれる視物質(桿体視物質)をロドプシンと呼び、ロドプシンは視物質の代表として多くの研究に利用されている。錐体には複数のサブタイプがあり、それぞれに波長感受性の異なる錐体視物質が含まれている。ヒトの錐体には、赤、緑、青に感受性の高い3種類の錐体視物質がそれぞれ含まれている。そして、これら錐体の応答が統合されることにより、色覚が生じる<ref><pubmed> 1385866 </pubmed></ref>。 | |||
「ロドプシン」という名前は、もともとは脊椎動物の桿体視細胞に含まれる視物質につけられた名前であった。しかし、最近では錐体視物質をはじめ無脊椎動物の視物質や視覚以外の機能に関わる光受容体などロドプシンとアミノ酸配列の相同性をもつ多くの光受容タンパク質が発見されるようになってきた。そこで、これらの光受容タンパク質をまとめてロドプシン類(またはオプシン類)と呼ぶことが多い。 | |||
視細胞には[[wikipedia:JA:繊毛|繊毛]]が分化した[[外節]]と呼ばれる特別の部位がある。桿体の外節にはパンケーキ状の円盤膜(disk membrane)が何層にも重なっている。そして、ロドプシンはこの円盤膜に埋め込まれて存在している。錐体の外節はひだ状の層構造になっており、この構造の中に錐体視物質が埋め込まれている(図1)。 微弱光でも効率よく受容できるように、ロドプシンは桿体の円盤膜に大量に発現している(円盤膜面積の50%以上がロドプシン分子である)。光を受容したロドプシンは構造変化を起こし、[[GTP結合タンパク質]](Gタンパク質)を介して[[細胞内シグナル伝達系]]を駆動する。この際にロドプシンの1分子は数百のGタンパク質を活性化し、光情報が増幅される。シグナル伝達系の下流でもさらに増幅機構が働き、その結果として、桿体はわずか1個の光子を受容しただけで応答することができる。 | |||
円盤膜は定常的にリニューアルされている。外節の根元から新しい円盤膜が作られ、先端の円盤膜は[[網膜色素上皮細胞]]に取り込まれる。[[マウス]]ではおよそ10日で円盤膜が根元から網膜色素上皮細胞層に達する。 | 円盤膜は定常的にリニューアルされている。外節の根元から新しい円盤膜が作られ、先端の円盤膜は[[網膜色素上皮細胞]]に取り込まれる。[[マウス]]ではおよそ10日で円盤膜が根元から網膜色素上皮細胞層に達する。 | ||
[[ | ロドプシンについて初めて報告があったのは1876〜77年頃である。ドイツの[[wikipedia:Franz Christian Boll|Franz Boll]] (1849-1879)、続いて[[wikipedia:ja:Wilhelm Kühne|Friedrich Wilhelm (通称Willy) Kühne]](1837−1900)がカエル網膜の桿体視細胞の外節にある赤い物質の感光性を報告した。 Kühneはこの色を“Sehpurpur”と呼び(英:Visual Purple, 日:視紅)その基となる化学物質をRhodopsinと名付けた。初期の視物質研究では視物質のことをVisual Purpleと呼んでいたが、しだいにRhodopsinが多く使われるようになり現在ではRhodopsinというのが一般的である。 | ||
[[Image:Mammal eye.png|thumb|right|300px|'''図1:ほ乳類の眼'''<br />眼に入った光は、[[角膜]]、[[レンズ]]、[[ガラス体]]を通過し、光受容に特化した視細胞に受容される。[[網膜]]中の視細胞は光が入射する方向と反対側にあり、そのため、光は視細胞に達するまでに神経節細胞や双極細胞などが含まれる神経層を通過することになる。 脊椎動物の眼には形態的に異なる2種類の視細胞、桿体(Rod)と錐体(Cone)があり、それぞれ、暗所、明所での視覚を分担している。そのため、それぞれ異なる応答特性を持っている。 桿体は感度が高いが応答が遅く、錐体は桿体よりも感度は低いが応答が速い。 また、錐体には複数のサブタイプがあり、それぞれ、赤、緑、青の光を吸収しやすい視物質が含まれており、色識別を可能にしている。桿体にはロドプシンが大量に含まれる円盤膜がパンケーキ状に重なっている。暗所での光受容に特化した桿体は単一光子を検出するほどの感度を有している。]] | |||
== 基本構造 | ==基本構造 == | ||
=== 発色団レチナール | === 発色団レチナール === | ||
ロドプシンの大きな特徴の一つは光を受容する[[wikipedia:JA:発色団|発色団]]として[[wikipedia:JA:レチナール|レチナール]]を含むことである。つまり、ロドプシンはアポタンパク質と発色団レチナールからなる。[[wikipedia:JA:アポタンパク質|アポタンパク質]]のことをオプシン(opsin) | ロドプシンの大きな特徴の一つは光を受容する[[wikipedia:JA:発色団|発色団]]として[[wikipedia:JA:レチナール|レチナール]]を含むことである。つまり、ロドプシンはアポタンパク質と発色団レチナールからなる。[[wikipedia:JA:アポタンパク質|アポタンパク質]]のことをオプシン(opsin)と呼ぶ(桿体視物質と錐体視物質のオプシンを区別する場合、scotopsin、photopsinと呼ぶ場合がある)。「オプシン」や「ロドプシン」という言葉は、広義には、ロドプシンに相同なタンパク質という意味でも使われている。 | ||
レチナールは[[wikipedia:JA:ビタミンA|ビタミンA]]の[[wikipedia:JA:アルデヒド|アルデヒド]]型であり、ロドプシン中ではその11- | レチナールは[[wikipedia:JA:ビタミンA|ビタミンA]]の[[wikipedia:JA:アルデヒド|アルデヒド]]型であり、ロドプシン中ではその11-シス型がオプシンのH7(ヘリックス7)に位置するN末端から296番目の[[wikipedia:JA:リジン|リジン]]残基と[[wikipedia:JA:シッフ塩基結合|シッフ塩基結合]]を介して結合している。光を受容したロドプシンでは、レチナールが11-シス型から全トランス型に光異性化される。その後、ロドプシンのタンパク質部分の構造変化がおこり、Gタンパク質を活性化する状態に変化する。この過程を「ロドプシンの光反応過程」と呼ぶ。生成した全トランスレチナールは、その後タンパク質部分から遊離し、タンパク質部分は新たに11-シス型のレチナールと結合してロドプシンになる。この過程を「ロドプシンの再生」と呼ぶ。タンパク質から遊離したレチナール(全トランス型に異性化)は視細胞の外に運ばれ、網膜色素上皮細胞で11-シス型に再[[wikipedia:JA:異性化|異性化]]され視細胞へ戻り、再びオプシンと結合してロドプシンになる。レチナールがどのような経路を経てオプシンと結合するのかは知られておらず、ロドプシン研究の一つの課題となっている。 | ||
[[wikipedia:JA:11-シスレチナール|11-シスレチナール]]はロドプシンが光を受容するために必須の分子である。また、11- | [[wikipedia:JA:11-シスレチナール|11-シスレチナール]]はロドプシンが光を受容するために必須の分子である。また、11-シスレチナールがオプシンと結合すると(ロドプシンになると)、オプシンの暗状態でのGタンパク質活性化能が強く抑制される。一方、光を受容して全トランス型に異性化すると、ロドプシンを高効率でGタンパク質を活性化する状態にする。つまり、薬理学的には、11-シスレチナールはinverse agonist(活性を抑制するリガンド、[[逆作動薬]])、全トランス型レチナールはagonist( 活性を促進するリガンド、[[作動薬]])と考えることができる。 | ||
=== 7回膜貫通構造 === | === 7回膜貫通構造 === | ||
ロドプシンのタンパク質部分(オプシン)は膜を貫通する7本の[[wikipedia:JA:α-ヘリックス|α-ヘリックス]]構造を持つ単一ペプチドである。これらα-ヘリックスは、その間にある細胞質ループ(Cytoplasmic/Intracellular loop: CL/IL)と細胞外ループ(Extracellular loop: EL)でつながれている。N末端が円盤膜の内側(トポロジー的には細胞外)に位置し、C末端が[[wikipedia:JA:細胞質|細胞質]]側にある。ヘリックス領域は膜を貫通するため、レチナールや[[wikipedia:JA:構造水|構造水]]と相互作用する少数の[[wikipedia:JA:親水性|親水性]]残基をのぞいて、ほとんどが疎水性残基で構成されている。一方、それ以外の領域には親水性残基が多く見られる。 | ロドプシンのタンパク質部分(オプシン)は膜を貫通する7本の[[wikipedia:JA:α-ヘリックス|α-ヘリックス]]構造を持つ単一ペプチドである。これらα-ヘリックスは、その間にある細胞質ループ(Cytoplasmic/Intracellular loop: CL/IL)と細胞外ループ(Extracellular loop: EL)でつながれている。N末端が円盤膜の内側(トポロジー的には細胞外)に位置し、C末端が[[wikipedia:JA:細胞質|細胞質]]側にある。ヘリックス領域は膜を貫通するため、レチナールや[[wikipedia:JA:構造水|構造水]]と相互作用する少数の[[wikipedia:JA:親水性|親水性]]残基をのぞいて、ほとんどが疎水性残基で構成されている。一方、それ以外の領域には親水性残基が多く見られる。 | ||
ヘリックス領域はフレキシブルなループ領域とは異なり、[[wikipedia:JA:剛体運動|剛体運動]](rigid body motion)によってヘッリクス間の配置が変わるような構造変化を起こす<ref><pubmed> 8864113 </pubmed></ref> | ヘリックス領域はフレキシブルなループ領域とは異なり、[[wikipedia:JA:剛体運動|剛体運動]](rigid body motion)によってヘッリクス間の配置が変わるような構造変化を起こす<ref><pubmed> 8864113 </pubmed></ref>。この変化により、ロドプシンの活性状態が生成することが知られている。また、細胞質側の第2、第3ループ(CL2, 3)はGタンパク質と結合するサイトとして重要である。 | ||
=== ヘリックス8 | === ヘリックス8 === | ||
ロドプシンにはヘリックス7とC末端の間に[[wikipedia:JA:翻訳後修飾|翻訳後修飾]]([[パルミトイル化]])を受ける[[wikipedia:JA:システイン|システイン]]残基が存在し、結合したパルミチン酸は[[wikipedia:JA:脂質二重膜|脂質二重膜]]に挿入されると考えられている。そのため、ヘリックス7とシステイン残基との間が細胞質側のもう一つのループとなり、この領域はさらにヘリックス構造を形成している。このヘリックスはヘリックス8と呼ばれている。膜表面に存在するH8は[[wikipedia:JA:両親媒性|両親媒性]]のヘリックスで膜側に疎水性の残基を含んでいる(図2参照)。 | |||
== 翻訳後修飾 == | |||
視細胞の[[wikipedia:JA:ER|ER]]で生合成されたオプシンは外節につながる繊毛部分に輸送され、外節の根元から生成する新たな円盤膜に取り込まれていく。前述したように、光受容体であるロドプシンは翻訳後にレチナールを取り込む必要があるが、それ以外にも円盤膜に運ばれるまでにいくつかの翻訳後修飾を受ける。ロドプシンの大きな特徴の一つが[[システイン]]110(この配列の動物種は?)とシステイン187の間に形成される[[wikipedia:JA:S−S結合|S−S結合]]である(図2参照)。このジスフィルド結合は多くの[[Gタンパク質共役型受容体]](GPCR)でも保存されておりECL2とH3を架橋することによって構造安定化に寄与している。 | |||
また、ロドプシンのアスパラギン2/アスパラギン15は[[wikipedia:JA:糖鎖修飾|糖鎖修飾]]を受ける。このためロドプシンのアスパラギン2/アスパラギン15は生物種を超えて良く保存されている。このようなタンパク質の[[wikipedia:JA:N結合型糖鎖付加|N結合型糖鎖付加]]は、修飾されるアミノ酸残基の位置は異なるが、オプシン類そしてファミリー1のGPCRにも見られる。一般に、糖鎖修飾はタンパク質の輸送やフォールディングに関わると考えられている。これ以外にもN末端の[[wikipedia:JA:メチル基|メチル基]](メチオニンの間違い?)は[[wikipedia:JA:アセチル化|アセチル化]]され、前述のC末端のシステイン322/システイン322(御確認下さい)の残基はパルミトイル化(脂質修飾)されている。 | |||
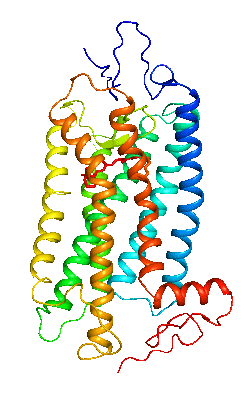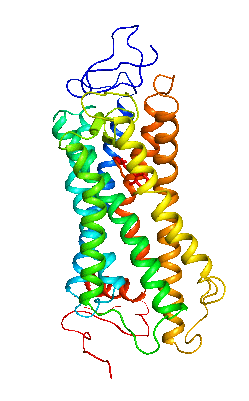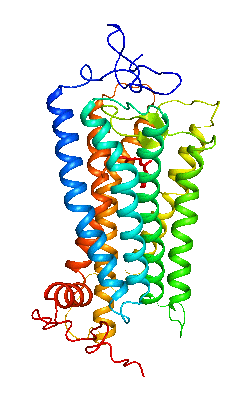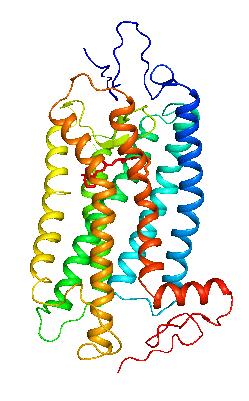
[[Image:Rhodopsin structure.png|thumb|right|400px|'''図2:ロドプシンの立体構造モデル'''<br />'''a''':基底状態のロドプシンの立体構造(PDBID:1U19)。ヘリックス1を青色で示しH8をオレンジ色で示している。7本の膜貫通ヘリックスに加えて膜面に平行なヘリックス8が特徴的である。ヘリックス3は大きく傾いていて細胞質側はヘリックス4とヘリックス5の間に入り込んでいる。上が円板膜内側、下がGタンパク質と相互作用する細胞質側である。手前のヘリックス7にレチナール(11−シス)とその結合部位であるリジン296、そしてシッフ塩基の対イオンとして機能するヘリックス3のグルタミン酸113のアミノ酸、システイン110-システイン187のジスフィルド結合、細胞質側にはヘリックス3にERYモチーフ、ヘリックス7にはNPXXYモチーフのアミノ酸を示している。<br />'''b''':活性化に伴う構造変化。[[wikipedia:JA:基底状態|基底状態]](緑色PDBID:1U19)と較べて[[wikipedia:JA:活性状態|活性状態]]は(オレンジ色PDBID:3PQR)ヘリックス6が大きく外側に動きヘリックス5も細胞質側に伸びるている。また基底状態ではヘリックス3とヘリックス6間のイオニックロックの相互作用が活性状態では解除されアルギニン135はNPXXYモチーフやチロシン223等と新たな相互作用を形成する。]] | |||
== 膜環境 == | |||
[[ | 膜タンパク質であるロドプシンの分子特性はその膜環境に大きく依存する。ただし、ヘリックス領域に囲まれている発色団の光化学的な特性([[wikipedia:JA:分子吸光係数|分子吸光係数]]、[[wikipedia:JA:量子収率|量子収率]]、[[wikipedia:JA:光感受性|光感受性]]など)は膜環境による影響を受けにくい。一方で、中間体や活性状態の平衡、寿命や生成速度等は膜環境の影響を顕著に受ける。 | ||
桿体外節の円盤膜は[[wikipedia:phosphatidylcholine|ホスファチジルコリン]]や[[wikipedia:phosphatidylethanolamine|ホスファチジルエタノールアミン]]を主成分とし、他にも[[wikipedia:phosphatidylserine|ホスファチジールセリン]]や[[ホスファチジルイノシトール]]を含むことが知られている。また膜の[[wikipedia:JA:コレステロール|コレステロール]]含有量によってロドプシンの活性状態とその前駆体の平衡が変化することが知られている。 | |||
== シッフ塩基プロトン・対イオン == | |||
ロドプシン中でのレチナールはリシン残基とシッフ塩基結合をしている。分子内でレチナールが共有結合しているのは、いつでも光受容出来るように発色団をタンパク質内に留めておく働きがある。さらにシッフ塩基を介したこの共有結合はロドプシンの機能発現にも重要な役割を果たしている。 | |||
ロドプシンが[[wikipedia:JA:可視光|可視光]](最大吸収波長500nm)を受容できるのは、このシッフ塩基の[[wikipedia:JA:窒素|窒素]]原子が[[wikipedia:JA:プロトン|プロトン]]化しているからである。レチナールやレチナールシッフ塩基は吸収極大波長が[[wikipedia:JA:紫外部|紫外部]]にあり、紫外光しか吸収することができない。一方、レチナールシッフ塩基がプロトン化すると、分子内の[[wikipedia:JA:二重結合|二重結合]]系が非局在化され、その結果、吸収極大波長が可視部に移動する。 | |||
レチナールはオプシンの内部に埋め込まれており、また、そのプロトン化シッフ塩基は疎水的な環境に位置している。そのためそのままでは非常に不安定である。オプシン内にはこの[[wikipedia:JA:正電荷|正電荷]]を安定化する対イオン(counterion)が存在する。ロドプシンではグルタミン酸113が対イオンとして働き<ref><pubmed> 2573063 </pubmed></ref>、ヘリックス7のシッフ塩基プロトンの正電荷とH3の[[グルタミン酸]]の負電荷の間に塩橋(salt bridge)が形成される<ref><pubmed> 1356370 </pubmed></ref>。また対イオンはシッフ塩基の[[wikipedia:JA:pKa|pKa]]を上げシッフ塩基の[[wikipedia:JA:加水分解|加水分解]]を防いでいる。対イオンは単独で働いているのではなく、構造水を含む[[wikipedia:JA:水素結合|水素結合]]ネットワークを形成して働いていると考えられている。 | |||
[[Image:Central Ionic Lock.png|thumb|right|300px|'''図3:シッフ塩基・対イオン・塩橋'''<br />ヘッリックス7の296番目のリシン残基の正電荷とヘリックス3の対イオンの負電荷は塩橋を形成し、リガンド非結合状態の受容体で不活性状態を安定化する。11-シス-レチナールが結合した状態でもシッフ塩基プロトンと対イオンの間で塩橋が生じ不活性状態を安定化する。]] | |||
[[Image:Central Ionic Lock.png|thumb|right| | |||
== 構造モチーフ == | |||
ロドプシン類あるいはGタンパク質共役型受容体(GPCR)のファミリー間で良く保存されている構造モチーフが幾つか知られており、これらは受容体の機能発現に重要である<ref><pubmed> 19836958 </pubmed></ref>。 | ロドプシン類あるいはGタンパク質共役型受容体(GPCR)のファミリー間で良く保存されている構造モチーフが幾つか知られており、これらは受容体の機能発現に重要である<ref><pubmed> 19836958 </pubmed></ref>。 | ||
(D/E)R(Y/W) | (D/E)R(Y/W)モチーフはファミリーAのGPCR間でよく保存されている構造モチーフで、ロドプシンではH3の細胞質側末端のE134/R135/Y136に相当する。また、H7とH8の先端にある302番目から306番目の残基はNPXXYモチーフと呼ばれ、このモチーフもファミリー1のGPCRの間でよく保存されている。ロドプシンの暗状態ではR135とE134の間に塩橋がある。また、R135はH6に存在するE247とT251との間で静電的な相互作用をしている(これらの相互作用を通常Ionic Lockと呼ぶ)。ロドプシンが光を受容することによりタンパク質部分の構造変化がおこると、E134は溶液中のプロトンと結合して中性になる。その結果、E134とR135の塩橋がなくなり、R135はNPXXYモチーフ中のY306やその他の残基(M257やY223)と新たな相互作用ネットワークを形成し、ロドプシンの活性構造の形成に寄与していると考えられている(図2参照)。 | ||
== 吸収スペクトル == | |||
ロドプシンは可視部に吸収極大を示す光受容タンパク質である。すでに述べたように、ロドプシンの可視部の吸収スペクトルは分子内に含まれているレチナールに由来する。有機溶媒中に溶かしたレチナールの吸収スペクトルは380 nm付近に吸収極大を示すが、レチナールがオプシン中のリシン残基とプロトン化したシッフ塩基を形成すると、500 nm付近に吸収極大がシフトする。有機溶媒中のプロトン化シッフ塩基は約440 nmに吸収極大を示す。そこで、440 nmからタンパク質の作用によって変化する差分を「オプシンシフト(Opsin shift)」と呼ぶ(図4a参照)。 このように、ロドプシンの吸収極大はプロトン化したレチナールシッフ塩基の吸収極大がまわりのアミノ酸残基によって調節されたものである<ref>''K Nakanishi, V Baloghair, M Arnaboli, K Tsujimoto, and B Honig'''<br>An External Point-Charge Model for Bacteriorhodopsin to Account for Its Purple Color<br>''J Am Chem Soc'':1980</ref>。実際、多くの動物のロドプシンは500nm付近に吸収極大を示すが、深海など極端な光環境下で生息する生物はそれぞれの光環境に適した吸収極大を示す。 | |||
[[ | オプシンシフト以外にもロドプシンはレチナールの種類を変えることによって吸収スペクトルを変えることができる。多くの脊椎動物は通常ビタミンA1(retinal)を用いるが、[[wikipedia:JA:魚類|魚類]]、[[wikipedia:JA:両生類|両生類]]や[[wikipedia:JA:爬虫類|爬虫類]]のなかには[[wikipedia:JA:A2 retinal|A2 retinal]] (3,4-dehydroretinal) を用いるものもいる。 共役二重結合系が長いのでA2レチナールはA1に比べてより長波長に吸収を持つ(図4b参照)。従ってA1/A2の視物質は同じタンパク質でもそれぞれ違う色をもつ。Opsin+A1 retinalの視物質がRhodopsin(rhod=紅)と呼ばれるのに対してOpsin+A2 retinalはPorphyropsin(porphyr=紫)と呼ばれる。(無脊椎動物の視物質ではA1, A2 retinalの他にA3([[wikipedia:JA:3-hydroxyretina|3-hydroxyretina]])やA4([[wikipedia:JA:4-hydroxyretinal|4-hydroxyretinal]]) retinalが用いられる。図4c参照)カエル幼生([[wikipedia:JA:オタマジャクシ|オタマジャクシ]])のオプシンがA2レチナールを発色団とし、成体(カエル)になるとA1レチナールを発色団とするのは有名な話である。つまり、オタマジャクシは、濁った淡水でより透過に優れた長波長の光を利用するためにA2レチナールを利用していると言われている。また、魚類(特に淡水魚)などは2種類のレチナールを持ち、季節変動などの環境要因によってA1/A2レチナールを使い分けていると考えられている。 | ||
[[Image:Rhodopsin spectrum and retinal.png|thumb|right|300px|'''図4:ロドプシンの吸収スペクトル'''<br />a:[[wikipedia:JA:有機溶媒|有機溶媒]]中のRetinalは380nmに吸収極大(λmax)を示すが、アミノ基を持つ化合物(例えば[[wikipedia:JA:プロピルアミン|プロピルアミン]])とシッフ塩基を形成してプロトン化されると、λmaxが440nmまでシフトする。さらにロドプシン中ではまわりのアミノ酸残基との相互作用によって、λmaxが約500 nmまでシフトする。この440nmからの差分をオプシンシフトと呼ぶ。つまり、オプシンシフトが大きいロドプシン類はより長波長側にλmaxを示す。ロドプシンの吸収スペクトルは可視部の吸収(αバンド)の他に、紫外領域に小さな吸収(βバンド)、280nm付近にタンパク質の[[wikipedia:JA:芳香族|芳香族]]アミノ酸残基に由来する吸収(γバンド)を示すのが特徴である。<br />b:桿体視物質、ロドプシンには2種類のレチナールが知られている。A2レチナールより構成するものを特にポルフィロプシンとよび、A1レチナールよりも共役二重結合系が長いのでより長波長の光を吸収することができる。一般的に多くの動物がA1レチナールを用いるが魚類、両生類や爬虫類等ではA2レチナールを用いるものが知られている。<br />c:無脊椎動物の視物質にはA1、A2レチナールの他にA3やA4レチナールを用いるものもある。発色団の種類に関わらずこれらの光受容タンパク質を総称してロドプシン類と呼ぶのが慣習である。]] | |||
== | ==光反応過程 == | ||
光を受容したロドプシンが活性状態に変化する過程を通常「ロドプシンの光反応過程」と呼ぶ。しかし、厳密には光が関与するのは発色団であるレチナールの光吸収と[[wikipedia:JA:光異性化|光異性化]]反応だけであり、活性状態に変化するタンパク質の構造変化は熱反応である<ref><pubmed> 11743865 </pubmed></ref>。ロドプシンの研究でノーベル賞を受賞したGeorge Wald博士は、この反応過程を写真を撮る過程になぞらえている。ロドプシンはカメラのフィルムのように光によって何らかの変化が生じるが、この変化は「現像」する過程によって初めて目に見えるものになるのである。ロドプシンでも同じように、光によって生じた変化が熱反応を経て活性状態の生成へとつながる<ref><pubmed> 4877437 </pubmed></ref>。 | |||
ロドプシンの最初のステップはレチナールの光吸収と光異性化反応である。暗状態で結合している11- | === 光反応 === | ||
ロドプシンの最初のステップはレチナールの光吸収と光異性化反応である。暗状態で結合している11-cis-retinalは光を受容するとall-trans-retinalに異性化する。レチナールの光異性化反応は溶液中でも起こるが、ロドプシン中での異性化反応は非常に高効率、高速で起こることが特徴である。溶液中のレチナールは20%程度の異性化効率(量子収率)しか示さないが、ロドプシン中のレチナールは65%の異性化効率を示す。そして200フェムト秒(200×10<sup>−15</sup>秒)<ref><pubmed> 1925597 </pubmed></ref>で起こるレチナールの光異性化反応は現在知られている最も速い[[wikipedia:JA:化学反応|化学反応]]の一つである。ロドプシン中でのレチナールの構造やその光反応性は近傍のアミノ酸残基によって調節されている。 | |||
=== 熱反応 === | |||
ロドプシン中でのレチナールの異性化反応は超高速で起こる。そのため、まわりのタンパク質部分はレチナールの異性化による構造変化についていけず、異性化直後のレチナールは非常にねじれた構造をとる。その結果、レチナールの吸収スペクトルは大幅に長波長シフトする。また、光子のエネルギーの約70%はレチナールの[[wikipedia:JA:構造ポテンシャルエネルギー|構造ポテンシャルエネルギー]]として蓄えられ、このエネルギーを使ってレチナール近傍のアミノ酸残基との相互作用が変化し、最終的にタンパク質全体の構造変化が誘起され、活性状態が生成する。 | ロドプシン中でのレチナールの異性化反応は超高速で起こる。そのため、まわりのタンパク質部分はレチナールの異性化による構造変化についていけず、異性化直後のレチナールは非常にねじれた構造をとる。その結果、レチナールの吸収スペクトルは大幅に長波長シフトする。また、光子のエネルギーの約70%はレチナールの[[wikipedia:JA:構造ポテンシャルエネルギー|構造ポテンシャルエネルギー]]として蓄えられ、このエネルギーを使ってレチナール近傍のアミノ酸残基との相互作用が変化し、最終的にタンパク質全体の構造変化が誘起され、活性状態が生成する。 | ||
レチナールの「ねじれ」の解消およびタンパク質部分の構造変化に伴って、光を吸収したロドプシンは種々の準安定な状態を経て変化していく。この準安定な状態を中間体と呼び、光吸収後から、フォト(photo)、バソ(batho)、ルミ(lumi)、メタ(meta)と名付けられた中間体が同定されている。これらの中間体は異なる吸収スペクトルを持つので、スペクトル変化によって熱反応を観測することができる<ref><pubmed> 13613341 </pubmed></ref>。反応の最終段階ではシッフ塩基のプロトンが対イオンに移動し、380 nmに吸収極大を示すメタロドプシンⅡ(Metarhodopsin II, Meta II)が生成する。Meta | レチナールの「ねじれ」の解消およびタンパク質部分の構造変化に伴って、光を吸収したロドプシンは種々の準安定な状態を経て変化していく。この準安定な状態を中間体と呼び、光吸収後から、フォト(photo)、バソ(batho)、ルミ(lumi)、メタ(meta)と名付けられた中間体が同定されている。これらの中間体は異なる吸収スペクトルを持つので、スペクトル変化によって熱反応を観測することができる<ref><pubmed> 13613341 </pubmed></ref>。反応の最終段階ではシッフ塩基のプロトンが対イオンに移動し、380 nmに吸収極大を示すメタロドプシンⅡ(Metarhodopsin II, Meta II)が生成する。Meta IIはGタンパク質活性化能をもつロドプシンの活性状態である<ref><pubmed> 6288450 </pubmed></ref>。 | ||
Meta IIはその前駆体Meta Iとの間でpH平衡にある(MetaI/IIの平衡はpH以外にも温度や膜の組成等で変化することが知られている)。 興味深いことに、平衡中の両者の量比は、シッフ塩基が脱プロトン化しているMeta IIが低pH(外液のプロトンが多い条件)で多くなり、プロトン化シッフ塩基を持つ Meta Iが高pHで多くなる。つまり、Meta II(活性状態)の生成には、シッフ塩基の脱プロトン化に伴う外界からのプロトンの取り込みが必要なことを示している。最近の研究によると、シッフ塩基の脱プロトン化がヘリックスの再配置(剛体運動)を誘起し、その結果、ERYモチーフが主となって形成するIonic | Meta IIはその前駆体Meta Iとの間でpH平衡にある(MetaI/IIの平衡はpH以外にも温度や膜の組成等で変化することが知られている)。 興味深いことに、平衡中の両者の量比は、シッフ塩基が脱プロトン化しているMeta IIが低pH(外液のプロトンが多い条件)で多くなり、プロトン化シッフ塩基を持つ Meta Iが高pHで多くなる。つまり、Meta II(活性状態)の生成には、シッフ塩基の脱プロトン化に伴う外界からのプロトンの取り込みが必要なことを示している。最近の研究によると、シッフ塩基の脱プロトン化がヘリックスの再配置(剛体運動)を誘起し、その結果、ERYモチーフが主となって形成するIonic lockが解除(E134がプロトン化)されることが知られている。ロドプシンの活性状態はこのような逐次的な構造変化によって生成するのである。 | ||
== | ==光シグナル伝達 == | ||
=== Gタンパク質のシグナル === | |||
光を受容したロドプシンは数ミリ秒の間にGタンパク質を活性化する状態に変化する。ロドプシンと共役するGタンパク質はαβγのサブユットからなる3量体Gタンパク質である。Gタンパク質(guanine nucleotide-binding proteins: G-proteins)はGTPを結合すると「on」、GDPを結合すると「off」になる分子スイッチとして機能する。一般にoff状態では3量体として存在し、Gα中でGDP-GTP交換反応が起こると、GαはGβγと解離して活性状態になる。活性化したロドプシンは1秒間に数百のGタンパク質を活性化することができるため、大きなシグナル増幅作用がある。 | |||
活性化したGαはcGMPを5’-GMPに加水分解する酵素である[[ホスホジエステラーゼ]](Phosphodiesterase、PDE)に作用する。PDEは酵素活性部位であるαβサブユニットとこれらと特異的に結合しその活性を抑制する2つのγサブユニットからなる。GαはこのPDEγに結合することによってγサブユニットの抑制効果を解除し、PDEを活性化する。 | |||
PDEが活性化すると細胞内のcGMPの濃度が急減し、cGMP依存性陽イオンチャネル(cyclic nucleotide-gated ion channels: CNG channels) が閉じる。光を受容したロドプシンからのシグナルがこない状態では、CNGチャネルは開いた状態であり、細胞内にNa<sup>+</sup>やCa2<sup>+</sup>が流入している。シグナルがくると上記の反応が起こるため、CNGチャネルが閉じ、細胞が過分極する。 | |||
= | 視細胞は暗状態では少し[[脱分極]]しており、その[[シナプス]]末端から[[神経伝達物質]]であるグルタミン酸が放出されている。光を受容して上記のシグナル伝達系が働くと過分極し、グルタミン酸の放出量が減少する。この変化が[[双極細胞]]などの下流の神経細胞に伝えられ、出力ニューロンである[[神経節細胞]]を経て[[脳]]にその情報が伝えられる。 (シグナル伝達についての参考文献<ref><pubmed> 19837030 </pubmed></ref><ref name=ref_shichida><pubmed> 19720651 </pubmed></ref>) | ||
=== シグナルのシャットダウンと視細胞の回復 === | |||
光を受容して応答した視細胞は、次の光を受容するために速やかにもとの静止状態に戻る必要がある。素早く戻ることが光受容の[[wikipedia:JA:時間分解能|時間分解能]]に関わるので、能動的に応答をシャットダウンし、もとの状態に戻ることが重要となる。 | 光を受容して応答した視細胞は、次の光を受容するために速やかにもとの静止状態に戻る必要がある。素早く戻ることが光受容の[[wikipedia:JA:時間分解能|時間分解能]]に関わるので、能動的に応答をシャットダウンし、もとの状態に戻ることが重要となる。 | ||
視細胞における光情報伝達はロドプシンの光受容によって始まり、また、ロドプシンからGタンパク質へのシグナル伝達の際に大きく増幅される。したがって、ロドプシンの活性状態を素早くシャットダウンすることは非常に重要である。ロドプシンの活性状態はメタロドプシンⅡ(Metarhodopsin II)と名付けられており、その名のとおり準安定(metastable)である。しかし、数十秒間は安定に存在する。視細胞の単一光子応答は1秒以内で終結するので、メタロドプシンⅡはこの時間よりも速くシャットダウン(不活性化)されている。メタロドプシンⅡの不活性化にはSer/Thrキナーゼである[[ロドプシンキナーゼ]](Rhodopsin Kinase: RK)が関与する。メタロドプシンⅡはこの酵素によって[[リン酸化]] | 視細胞における光情報伝達はロドプシンの光受容によって始まり、また、ロドプシンからGタンパク質へのシグナル伝達の際に大きく増幅される。したがって、ロドプシンの活性状態を素早くシャットダウンすることは非常に重要である。ロドプシンの活性状態はメタロドプシンⅡ(Metarhodopsin II)と名付けられており、その名のとおり準安定(metastable)である。しかし、数十秒間は安定に存在する。視細胞の単一光子応答は1秒以内で終結するので、メタロドプシンⅡはこの時間よりも速くシャットダウン(不活性化)されている。メタロドプシンⅡの不活性化にはSer/Thrキナーゼである[[ロドプシンキナーゼ]](Rhodopsin Kinase: RK)が関与する。メタロドプシンⅡはこの酵素によって[[リン酸化]]され、そのGタンパク質活性化能が減少する。メタロドプシンⅡのC末端領域にはリン酸化される部位として複数のS/Tが同定されている。生体内では主にS334, S338, S343がリン酸化される。さらにリン酸化されたメタロドプシンⅡにアレスチン(Arrestin)が結合することによってGタンパク質との結合が完全に阻害される。 | ||
桿体視細胞の応答プロファイルは非常に再現性が良い。これを実現するために、応答を複数のステップで制御する(Multi step shutoff)機構があると考えられている。ロドプシンの不活性化が単一ステップだけで起こる場合、分子間の相互作用は確率論的に起こるので、速くシャッタダウンする場合や遅くシャットダウンする場合がでてくる。このようにシャットダウンがランダムだと単一光子応答がばらつき、ノイズと区別できなくなる。そのため、ロドプシンは複数ステップのシャットダウン機構を用いることによってシャットダウン時間の可変性を平均化し応答の再現性を保証していると考えられている。 | |||
Gαは内在的な[[GTPase]]活性をもつ。そのため、GTPと結合して活性状態になったGαは自発的にGTPをGDPに加水分解し、再びGβγと結合して不活性状態に戻る。しかし、Gαの自発的な酵素活性は低く、視細胞内ではPDEγに結合したGαにGAP([[GTPase Activating Protein]])が作用することによってGTPの加水分解を加速している。Gαが不活性化しPDEから遊離するとPDEγは再び酵素活性部位であるPDEαβを阻害するためPDEの活性が抑えられる。 | |||
細胞が完全にもとの状態に戻るには、PDEの作用によって急減した細胞内cGMP濃度ももとに戻す必要がある。cGMPはGC([[Guanyl Cyclase]])によって定常的に合成されているが、GCの活性はGCAP ([[Guanyl Cyclase Activating Protein]])によって調節されている。光応答により細胞膜のCNGチャネルが閉じ細胞内のCa2+濃度が下がるとGCAPはGCの活性を促進するようになる。細胞内カルシウムの増減によるこの制御機構をカルシウムフィードバック機構と呼ぶ。この機構により細胞内のcGMP濃度が速やかに上昇すると[[CNGチャネル]]も開き、視細胞が元の状態に戻る。なお、細胞内カルシウム濃度の減少により、ロドプシンキナーゼを制御する因子([[Sモジュリン]]あるいはリカバリンと呼ばれている)も知られており<ref><pubmed> 8386803 </pubmed></ref>、GCAPとあわせて視細胞の[[明順応]]を説明する一つの機構と考えられている。 | |||
== | == ロドプシン類 == | ||
本来「ロドプシン」とは桿体視物質をあらわす言葉であった。しかし、生化学・分子生物学の進展により、桿体視細胞以外の光受容細胞や脊椎動物以外の生物種から相同性のある光受容タンパク質が続々と報告されるようになり<ref><pubmed> 15774036 </pubmed></ref><ref name=ref_shichida />、これらの光受容タンパク質も「ロドプシン」あるいは「オプシン」と呼ばれるようになった。これら多くの発見の中でも、ニワトリの[[松果体]]に存在する[[ピノプシン]]の発見は特筆される<ref><pubmed> 7969427 </pubmed></ref>。つまり、それまでに発見されていたロドプシン類はいわゆる「視覚」に関与する受容体であったが、ピノプシンは視覚以外の機能に関与する受容体であったからである。この発見以降、「視覚オプシン(visual opsin)」、「非視覚オプシン(non-visual opsin)」という言葉が使われるようになった。最近では1000種類以上のロドプシン遺伝子が報告されており、これらはGタンパク質共役型受容体(G Protein Coupled Receptor: GPCR)の一員であることが知られている。 | |||
GPCRは[[ペプチド]]、[[ホルモン]]、[[匂い]]物質などのさまざまな[[wikipedia:JA:化学物質|化学物質]]を受容し、Gタンパク質を介する細胞内シグナル伝達機構を駆動する受容体である。GPCRによる外界からのシグナル受容はほとんどの細胞で観測され、細胞間のコミュニケーションを担う上でも非常に重要な受容体である。また、マウスやヒトではゲノム中で最も大きなタンパク質ファミリーであることが知られている。ロドプシン類はGPCRのメンバーであるが、分子内に内在性のリガンド(11-シス型のレチナール)を含んでいることが特徴である。 | GPCRは[[ペプチド]]、[[ホルモン]]、[[匂い]][[リンクの名前]]物質などのさまざまな[[wikipedia:JA:化学物質|化学物質]]を受容し、Gタンパク質を介する細胞内シグナル伝達機構を駆動する受容体である。GPCRによる外界からのシグナル受容はほとんどの細胞で観測され、細胞間のコミュニケーションを担う上でも非常に重要な受容体である。また、マウスやヒトではゲノム中で最も大きなタンパク質ファミリーであることが知られている。ロドプシン類はGPCRのメンバーであるが、分子内に内在性のリガンド(11-シス型のレチナール)を含んでいることが特徴である。 | ||
=== GPCRとロドプシン === | |||
ロドプシンはGPCRファミリー1の代表的な受容体として知られている。 実際ファミリー1のGPCRはrhodopsin-like GPCRとも呼ばれている。近年の[[wikipedia:JA:結晶構造解析|結晶構造解析]]の結果、ロドプシンの立体構造、特に膜貫通領域の構造は、他のGPCRのそれらと酷似していることが証明された。しかし、非常に多様化しているGPCRのなかでロドプシンは必ずしも典型的なGPCRというわけではない。ロドプシンは11-シスレチナールを内在性のリガンドとしてもともと結合している。11-シスレチナールは、発色団として、また、インバースアゴニストとして働き、これはロドプシンのみの特徴である。 | |||
ウシロドプシンの一次配列は1982年に決定され<ref><pubmed> 6759163 </pubmed></ref>、その翌年にはクローニングされている<ref><pubmed> 6194890 </pubmed></ref>。そして2000年にはX線結晶解析により3次元立体構造モデルが提出された<ref><pubmed> 10926528 </pubmed></ref><ref><pubmed> 11972040 </pubmed></ref>。また、現在ではさまざまな中間状態や活性状態<ref><pubmed> 21389988 </pubmed></ref>、変異体などの立体構造も発表されている。一次構造の決定、クローニング、結晶構造決定などについては、種々のGPCRの中ではロドプシンで最初に行われた。ウシロドプシンのように大量の試料を比較的簡単に調製できるGPCRは珍しく、また内在性のリガンドを持つロドプシンは他のGPCRに較べて非常に安定でそのためロドプシンの研究は他の受容体よりも先に進んだ。こうしてロドプシンはGPCR研究のトップランナーとして研究されてきた経歴があり、GPCRファミリー1の代表的な受容体とされている。 | |||
ロドプシンがGPCRであると認知されるようになったのは数十年前からである。1986年にGPCRの一つ[[βアドレナリン受容体]]の一次配列が決定されるとすでに解析されていたロドプシンの配列そしてその配列から予想される7回膜貫通構造が非常に似ていることが発見された。その後も次々に様々なGPCRの配列が決定され、これらは一大タンパク質ファミリーを形成することが明らかになった。 | |||
=== 動物のロドプシンと菌のロドプシン === | |||
様々な動物で見つかっているロドプシン(オプシン)の他に[[wikipedia:JA:バクテリア|バクテリア]]にも光感受性を持つレチナールタンパク質が含まれていることが知られている。1971年にOesterheltとStoeckniusは[[wikipedia:JA:好塩菌|好塩菌]]の一種[[wikipedia:JA:ハロバクテリウム・ハロビウム|ハロバクテリウム・ハロビウム]](最近ではハロバクテリウム・サリナラムという)にレチナールを発色団とする光受容タンパク質が存在することを発見し、このタンパク質をバクテリオロドプシン(bR)と命名した<ref><pubmed> 4940442 </pubmed></ref>。その後の研究により、bRは光駆動の[[wikipedia:JA:プロトンポンプ|プロトンポンプ]]活性を示すことがわかり、また、バクテリアにはbRを含めて4種類のレチナールタンパク質が存在することがわかった。bR以外にはハロロドプシン(hR)、センソリーロドプシン(sR)、センソリーロドプシンII(sRII、フォボロドプシン(pR)ともいう)である。hRは光駆動のクロライドポンプ、sRとsRIIはそれぞれ正・負の光走性に関与するロドプシンである。最近、[[wikipedia:JA:緑藻類|緑藻類]]から光駆動の[[wikipedia:JA:チャネル|チャネル]]活性を示すロドプシン(チャネルロドプシン)が発見され、hRとともに、神経細胞のlight-manipulationに応用されている。さらに最近では、海洋のバクテリアにもbR様のロドプシンが含まれていることが発見され、地球上のエネルギー生産の半分程度がbR様のロドプシン類で担われていることが注目されている。また、遺伝子発現を調節するロドプシン類も[[wikipedia:JA:アナベナ|アナベナ]]から発見されるなど、バクテリアが持つロドプシン類の機能解析は最近の一つのトピックスになっている。 | |||
これらのバクテリアのロドプシン類も、動物のロドプシン類と同様に7回膜貫通領域をもち、発色団としてレチナールを用い、さらにその発色団はレチナールシッフ塩基結合を介してH7に結合している。ただし、動物のロドプシンは主に11-シス型のレチナールを発色団として持ち、光を受容して全トランスに異性化されて活性状態になるが、バクテリアのロドプシンは全トランス型のレチナールを発色団とし、光を吸収して13-シス型に異性化し、機能を発揮することがわかっている。また、バクテリアのロドプシンは活性状態になったあと熱反応で元の状態に戻る光反応サイクルを描く。7本膜貫通α-ヘリックス構造を持つことから、両タンパク質は進化的に系統関係があると考えられていたが、アミノ酸配列からは相同性の無いことが明らかにされている。しかしロドプシン類の中でも20%程度の相同性しか示さないものもあるので、たとえ共通の祖先タンパク質から進化しても遠縁な生物種間では変異が蓄積し有意な相同性がなくなっている可能性もある。 | |||
== 参考文献 | == 参考文献 == | ||
<references/> | |||
(執筆者:松山オジョス武、七田芳則 担当編集委員:林康紀) | |||
2012年2月18日 (土) 00:54時点における版
別名:桿体視物質、視紅 英:Rhodopsin, Visual purple
(概要をここに御願い致します。元からあった文章を短くして書いてみましたので、物性などを付け加えて頂けば幸いです) 視細胞には光を受容するために特別に分化したタンパク質(光受容タンパク質)が含まれ、視物質と呼ぶ[1][2]。そのうち桿体に含まれる視物質(桿体視物質)をロドプシンと呼ぶ。桿体の外節にはパンケーキ状の円盤膜(disk membrane)が何層にも重なっており、ロドプシンはこの円盤膜に埋め込まれて存在している。微弱光でも効率よく受容できるように、ロドプシンは桿体の円盤膜に大量に発現している(円盤膜面積の50%以上がロドプシン分子である)。光を受容したロドプシンは構造変化を起こし、Gタンパク質を介して細胞内シグナル伝達系を駆動する。この際にロドプシンの1分子は数百のGタンパク質を活性化し、光情報が増幅される。シグナル伝達系の下流でもさらに増幅機構が働き、その結果として、桿体はわずか1個の光子を受容しただけで応答することができる。
ロドプシン分子
脊椎動物の眼には2種類の視細胞、桿体と錐体が存在し、それぞれ、暗所視、明所視を司る。両視細胞には光を受容するために特別に分化したタンパク質(光受容タンパク質)が含まれ、それらを視物質と呼ぶ[3][4]。桿体に含まれる視物質(桿体視物質)をロドプシンと呼び、ロドプシンは視物質の代表として多くの研究に利用されている。錐体には複数のサブタイプがあり、それぞれに波長感受性の異なる錐体視物質が含まれている。ヒトの錐体には、赤、緑、青に感受性の高い3種類の錐体視物質がそれぞれ含まれている。そして、これら錐体の応答が統合されることにより、色覚が生じる[5]。
「ロドプシン」という名前は、もともとは脊椎動物の桿体視細胞に含まれる視物質につけられた名前であった。しかし、最近では錐体視物質をはじめ無脊椎動物の視物質や視覚以外の機能に関わる光受容体などロドプシンとアミノ酸配列の相同性をもつ多くの光受容タンパク質が発見されるようになってきた。そこで、これらの光受容タンパク質をまとめてロドプシン類(またはオプシン類)と呼ぶことが多い。
視細胞には繊毛が分化した外節と呼ばれる特別の部位がある。桿体の外節にはパンケーキ状の円盤膜(disk membrane)が何層にも重なっている。そして、ロドプシンはこの円盤膜に埋め込まれて存在している。錐体の外節はひだ状の層構造になっており、この構造の中に錐体視物質が埋め込まれている(図1)。 微弱光でも効率よく受容できるように、ロドプシンは桿体の円盤膜に大量に発現している(円盤膜面積の50%以上がロドプシン分子である)。光を受容したロドプシンは構造変化を起こし、GTP結合タンパク質(Gタンパク質)を介して細胞内シグナル伝達系を駆動する。この際にロドプシンの1分子は数百のGタンパク質を活性化し、光情報が増幅される。シグナル伝達系の下流でもさらに増幅機構が働き、その結果として、桿体はわずか1個の光子を受容しただけで応答することができる。
円盤膜は定常的にリニューアルされている。外節の根元から新しい円盤膜が作られ、先端の円盤膜は網膜色素上皮細胞に取り込まれる。マウスではおよそ10日で円盤膜が根元から網膜色素上皮細胞層に達する。
ロドプシンについて初めて報告があったのは1876〜77年頃である。ドイツのFranz Boll (1849-1879)、続いてFriedrich Wilhelm (通称Willy) Kühne(1837−1900)がカエル網膜の桿体視細胞の外節にある赤い物質の感光性を報告した。 Kühneはこの色を“Sehpurpur”と呼び(英:Visual Purple, 日:視紅)その基となる化学物質をRhodopsinと名付けた。初期の視物質研究では視物質のことをVisual Purpleと呼んでいたが、しだいにRhodopsinが多く使われるようになり現在ではRhodopsinというのが一般的である。

眼に入った光は、角膜、レンズ、ガラス体を通過し、光受容に特化した視細胞に受容される。網膜中の視細胞は光が入射する方向と反対側にあり、そのため、光は視細胞に達するまでに神経節細胞や双極細胞などが含まれる神経層を通過することになる。 脊椎動物の眼には形態的に異なる2種類の視細胞、桿体(Rod)と錐体(Cone)があり、それぞれ、暗所、明所での視覚を分担している。そのため、それぞれ異なる応答特性を持っている。 桿体は感度が高いが応答が遅く、錐体は桿体よりも感度は低いが応答が速い。 また、錐体には複数のサブタイプがあり、それぞれ、赤、緑、青の光を吸収しやすい視物質が含まれており、色識別を可能にしている。桿体にはロドプシンが大量に含まれる円盤膜がパンケーキ状に重なっている。暗所での光受容に特化した桿体は単一光子を検出するほどの感度を有している。
基本構造
発色団レチナール
ロドプシンの大きな特徴の一つは光を受容する発色団としてレチナールを含むことである。つまり、ロドプシンはアポタンパク質と発色団レチナールからなる。アポタンパク質のことをオプシン(opsin)と呼ぶ(桿体視物質と錐体視物質のオプシンを区別する場合、scotopsin、photopsinと呼ぶ場合がある)。「オプシン」や「ロドプシン」という言葉は、広義には、ロドプシンに相同なタンパク質という意味でも使われている。
レチナールはビタミンAのアルデヒド型であり、ロドプシン中ではその11-シス型がオプシンのH7(ヘリックス7)に位置するN末端から296番目のリジン残基とシッフ塩基結合を介して結合している。光を受容したロドプシンでは、レチナールが11-シス型から全トランス型に光異性化される。その後、ロドプシンのタンパク質部分の構造変化がおこり、Gタンパク質を活性化する状態に変化する。この過程を「ロドプシンの光反応過程」と呼ぶ。生成した全トランスレチナールは、その後タンパク質部分から遊離し、タンパク質部分は新たに11-シス型のレチナールと結合してロドプシンになる。この過程を「ロドプシンの再生」と呼ぶ。タンパク質から遊離したレチナール(全トランス型に異性化)は視細胞の外に運ばれ、網膜色素上皮細胞で11-シス型に再異性化され視細胞へ戻り、再びオプシンと結合してロドプシンになる。レチナールがどのような経路を経てオプシンと結合するのかは知られておらず、ロドプシン研究の一つの課題となっている。
11-シスレチナールはロドプシンが光を受容するために必須の分子である。また、11-シスレチナールがオプシンと結合すると(ロドプシンになると)、オプシンの暗状態でのGタンパク質活性化能が強く抑制される。一方、光を受容して全トランス型に異性化すると、ロドプシンを高効率でGタンパク質を活性化する状態にする。つまり、薬理学的には、11-シスレチナールはinverse agonist(活性を抑制するリガンド、逆作動薬)、全トランス型レチナールはagonist( 活性を促進するリガンド、作動薬)と考えることができる。
7回膜貫通構造
ロドプシンのタンパク質部分(オプシン)は膜を貫通する7本のα-ヘリックス構造を持つ単一ペプチドである。これらα-ヘリックスは、その間にある細胞質ループ(Cytoplasmic/Intracellular loop: CL/IL)と細胞外ループ(Extracellular loop: EL)でつながれている。N末端が円盤膜の内側(トポロジー的には細胞外)に位置し、C末端が細胞質側にある。ヘリックス領域は膜を貫通するため、レチナールや構造水と相互作用する少数の親水性残基をのぞいて、ほとんどが疎水性残基で構成されている。一方、それ以外の領域には親水性残基が多く見られる。
ヘリックス領域はフレキシブルなループ領域とは異なり、剛体運動(rigid body motion)によってヘッリクス間の配置が変わるような構造変化を起こす[6]。この変化により、ロドプシンの活性状態が生成することが知られている。また、細胞質側の第2、第3ループ(CL2, 3)はGタンパク質と結合するサイトとして重要である。
ヘリックス8
ロドプシンにはヘリックス7とC末端の間に翻訳後修飾(パルミトイル化)を受けるシステイン残基が存在し、結合したパルミチン酸は脂質二重膜に挿入されると考えられている。そのため、ヘリックス7とシステイン残基との間が細胞質側のもう一つのループとなり、この領域はさらにヘリックス構造を形成している。このヘリックスはヘリックス8と呼ばれている。膜表面に存在するH8は両親媒性のヘリックスで膜側に疎水性の残基を含んでいる(図2参照)。
翻訳後修飾
視細胞のERで生合成されたオプシンは外節につながる繊毛部分に輸送され、外節の根元から生成する新たな円盤膜に取り込まれていく。前述したように、光受容体であるロドプシンは翻訳後にレチナールを取り込む必要があるが、それ以外にも円盤膜に運ばれるまでにいくつかの翻訳後修飾を受ける。ロドプシンの大きな特徴の一つがシステイン110(この配列の動物種は?)とシステイン187の間に形成されるS−S結合である(図2参照)。このジスフィルド結合は多くのGタンパク質共役型受容体(GPCR)でも保存されておりECL2とH3を架橋することによって構造安定化に寄与している。
また、ロドプシンのアスパラギン2/アスパラギン15は糖鎖修飾を受ける。このためロドプシンのアスパラギン2/アスパラギン15は生物種を超えて良く保存されている。このようなタンパク質のN結合型糖鎖付加は、修飾されるアミノ酸残基の位置は異なるが、オプシン類そしてファミリー1のGPCRにも見られる。一般に、糖鎖修飾はタンパク質の輸送やフォールディングに関わると考えられている。これ以外にもN末端のメチル基(メチオニンの間違い?)はアセチル化され、前述のC末端のシステイン322/システイン322(御確認下さい)の残基はパルミトイル化(脂質修飾)されている。
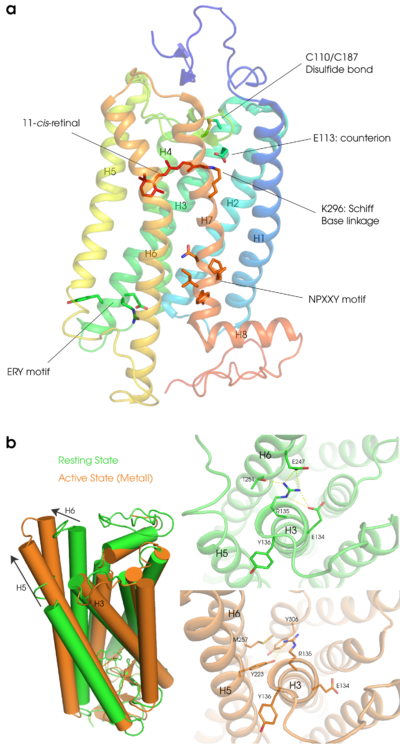
a:基底状態のロドプシンの立体構造(PDBID:1U19)。ヘリックス1を青色で示しH8をオレンジ色で示している。7本の膜貫通ヘリックスに加えて膜面に平行なヘリックス8が特徴的である。ヘリックス3は大きく傾いていて細胞質側はヘリックス4とヘリックス5の間に入り込んでいる。上が円板膜内側、下がGタンパク質と相互作用する細胞質側である。手前のヘリックス7にレチナール(11−シス)とその結合部位であるリジン296、そしてシッフ塩基の対イオンとして機能するヘリックス3のグルタミン酸113のアミノ酸、システイン110-システイン187のジスフィルド結合、細胞質側にはヘリックス3にERYモチーフ、ヘリックス7にはNPXXYモチーフのアミノ酸を示している。
b:活性化に伴う構造変化。基底状態(緑色PDBID:1U19)と較べて活性状態は(オレンジ色PDBID:3PQR)ヘリックス6が大きく外側に動きヘリックス5も細胞質側に伸びるている。また基底状態ではヘリックス3とヘリックス6間のイオニックロックの相互作用が活性状態では解除されアルギニン135はNPXXYモチーフやチロシン223等と新たな相互作用を形成する。
膜環境
膜タンパク質であるロドプシンの分子特性はその膜環境に大きく依存する。ただし、ヘリックス領域に囲まれている発色団の光化学的な特性(分子吸光係数、量子収率、光感受性など)は膜環境による影響を受けにくい。一方で、中間体や活性状態の平衡、寿命や生成速度等は膜環境の影響を顕著に受ける。
桿体外節の円盤膜はホスファチジルコリンやホスファチジルエタノールアミンを主成分とし、他にもホスファチジールセリンやホスファチジルイノシトールを含むことが知られている。また膜のコレステロール含有量によってロドプシンの活性状態とその前駆体の平衡が変化することが知られている。
シッフ塩基プロトン・対イオン
ロドプシン中でのレチナールはリシン残基とシッフ塩基結合をしている。分子内でレチナールが共有結合しているのは、いつでも光受容出来るように発色団をタンパク質内に留めておく働きがある。さらにシッフ塩基を介したこの共有結合はロドプシンの機能発現にも重要な役割を果たしている。
ロドプシンが可視光(最大吸収波長500nm)を受容できるのは、このシッフ塩基の窒素原子がプロトン化しているからである。レチナールやレチナールシッフ塩基は吸収極大波長が紫外部にあり、紫外光しか吸収することができない。一方、レチナールシッフ塩基がプロトン化すると、分子内の二重結合系が非局在化され、その結果、吸収極大波長が可視部に移動する。
レチナールはオプシンの内部に埋め込まれており、また、そのプロトン化シッフ塩基は疎水的な環境に位置している。そのためそのままでは非常に不安定である。オプシン内にはこの正電荷を安定化する対イオン(counterion)が存在する。ロドプシンではグルタミン酸113が対イオンとして働き[7]、ヘリックス7のシッフ塩基プロトンの正電荷とH3のグルタミン酸の負電荷の間に塩橋(salt bridge)が形成される[8]。また対イオンはシッフ塩基のpKaを上げシッフ塩基の加水分解を防いでいる。対イオンは単独で働いているのではなく、構造水を含む水素結合ネットワークを形成して働いていると考えられている。

ヘッリックス7の296番目のリシン残基の正電荷とヘリックス3の対イオンの負電荷は塩橋を形成し、リガンド非結合状態の受容体で不活性状態を安定化する。11-シス-レチナールが結合した状態でもシッフ塩基プロトンと対イオンの間で塩橋が生じ不活性状態を安定化する。
構造モチーフ
ロドプシン類あるいはGタンパク質共役型受容体(GPCR)のファミリー間で良く保存されている構造モチーフが幾つか知られており、これらは受容体の機能発現に重要である[9]。
(D/E)R(Y/W)モチーフはファミリーAのGPCR間でよく保存されている構造モチーフで、ロドプシンではH3の細胞質側末端のE134/R135/Y136に相当する。また、H7とH8の先端にある302番目から306番目の残基はNPXXYモチーフと呼ばれ、このモチーフもファミリー1のGPCRの間でよく保存されている。ロドプシンの暗状態ではR135とE134の間に塩橋がある。また、R135はH6に存在するE247とT251との間で静電的な相互作用をしている(これらの相互作用を通常Ionic Lockと呼ぶ)。ロドプシンが光を受容することによりタンパク質部分の構造変化がおこると、E134は溶液中のプロトンと結合して中性になる。その結果、E134とR135の塩橋がなくなり、R135はNPXXYモチーフ中のY306やその他の残基(M257やY223)と新たな相互作用ネットワークを形成し、ロドプシンの活性構造の形成に寄与していると考えられている(図2参照)。
吸収スペクトル
ロドプシンは可視部に吸収極大を示す光受容タンパク質である。すでに述べたように、ロドプシンの可視部の吸収スペクトルは分子内に含まれているレチナールに由来する。有機溶媒中に溶かしたレチナールの吸収スペクトルは380 nm付近に吸収極大を示すが、レチナールがオプシン中のリシン残基とプロトン化したシッフ塩基を形成すると、500 nm付近に吸収極大がシフトする。有機溶媒中のプロトン化シッフ塩基は約440 nmに吸収極大を示す。そこで、440 nmからタンパク質の作用によって変化する差分を「オプシンシフト(Opsin shift)」と呼ぶ(図4a参照)。 このように、ロドプシンの吸収極大はプロトン化したレチナールシッフ塩基の吸収極大がまわりのアミノ酸残基によって調節されたものである[10]。実際、多くの動物のロドプシンは500nm付近に吸収極大を示すが、深海など極端な光環境下で生息する生物はそれぞれの光環境に適した吸収極大を示す。
オプシンシフト以外にもロドプシンはレチナールの種類を変えることによって吸収スペクトルを変えることができる。多くの脊椎動物は通常ビタミンA1(retinal)を用いるが、魚類、両生類や爬虫類のなかにはA2 retinal (3,4-dehydroretinal) を用いるものもいる。 共役二重結合系が長いのでA2レチナールはA1に比べてより長波長に吸収を持つ(図4b参照)。従ってA1/A2の視物質は同じタンパク質でもそれぞれ違う色をもつ。Opsin+A1 retinalの視物質がRhodopsin(rhod=紅)と呼ばれるのに対してOpsin+A2 retinalはPorphyropsin(porphyr=紫)と呼ばれる。(無脊椎動物の視物質ではA1, A2 retinalの他にA3(3-hydroxyretina)やA4(4-hydroxyretinal) retinalが用いられる。図4c参照)カエル幼生(オタマジャクシ)のオプシンがA2レチナールを発色団とし、成体(カエル)になるとA1レチナールを発色団とするのは有名な話である。つまり、オタマジャクシは、濁った淡水でより透過に優れた長波長の光を利用するためにA2レチナールを利用していると言われている。また、魚類(特に淡水魚)などは2種類のレチナールを持ち、季節変動などの環境要因によってA1/A2レチナールを使い分けていると考えられている。
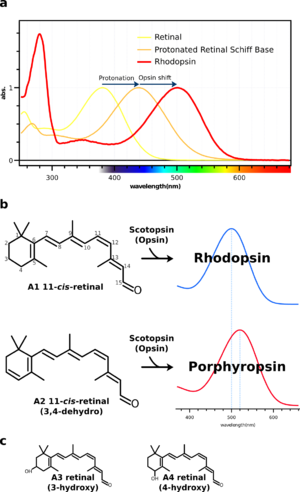
a:有機溶媒中のRetinalは380nmに吸収極大(λmax)を示すが、アミノ基を持つ化合物(例えばプロピルアミン)とシッフ塩基を形成してプロトン化されると、λmaxが440nmまでシフトする。さらにロドプシン中ではまわりのアミノ酸残基との相互作用によって、λmaxが約500 nmまでシフトする。この440nmからの差分をオプシンシフトと呼ぶ。つまり、オプシンシフトが大きいロドプシン類はより長波長側にλmaxを示す。ロドプシンの吸収スペクトルは可視部の吸収(αバンド)の他に、紫外領域に小さな吸収(βバンド)、280nm付近にタンパク質の芳香族アミノ酸残基に由来する吸収(γバンド)を示すのが特徴である。
b:桿体視物質、ロドプシンには2種類のレチナールが知られている。A2レチナールより構成するものを特にポルフィロプシンとよび、A1レチナールよりも共役二重結合系が長いのでより長波長の光を吸収することができる。一般的に多くの動物がA1レチナールを用いるが魚類、両生類や爬虫類等ではA2レチナールを用いるものが知られている。
c:無脊椎動物の視物質にはA1、A2レチナールの他にA3やA4レチナールを用いるものもある。発色団の種類に関わらずこれらの光受容タンパク質を総称してロドプシン類と呼ぶのが慣習である。
光反応過程
光を受容したロドプシンが活性状態に変化する過程を通常「ロドプシンの光反応過程」と呼ぶ。しかし、厳密には光が関与するのは発色団であるレチナールの光吸収と光異性化反応だけであり、活性状態に変化するタンパク質の構造変化は熱反応である[11]。ロドプシンの研究でノーベル賞を受賞したGeorge Wald博士は、この反応過程を写真を撮る過程になぞらえている。ロドプシンはカメラのフィルムのように光によって何らかの変化が生じるが、この変化は「現像」する過程によって初めて目に見えるものになるのである。ロドプシンでも同じように、光によって生じた変化が熱反応を経て活性状態の生成へとつながる[12]。
光反応
ロドプシンの最初のステップはレチナールの光吸収と光異性化反応である。暗状態で結合している11-cis-retinalは光を受容するとall-trans-retinalに異性化する。レチナールの光異性化反応は溶液中でも起こるが、ロドプシン中での異性化反応は非常に高効率、高速で起こることが特徴である。溶液中のレチナールは20%程度の異性化効率(量子収率)しか示さないが、ロドプシン中のレチナールは65%の異性化効率を示す。そして200フェムト秒(200×10−15秒)[13]で起こるレチナールの光異性化反応は現在知られている最も速い化学反応の一つである。ロドプシン中でのレチナールの構造やその光反応性は近傍のアミノ酸残基によって調節されている。
熱反応
ロドプシン中でのレチナールの異性化反応は超高速で起こる。そのため、まわりのタンパク質部分はレチナールの異性化による構造変化についていけず、異性化直後のレチナールは非常にねじれた構造をとる。その結果、レチナールの吸収スペクトルは大幅に長波長シフトする。また、光子のエネルギーの約70%はレチナールの構造ポテンシャルエネルギーとして蓄えられ、このエネルギーを使ってレチナール近傍のアミノ酸残基との相互作用が変化し、最終的にタンパク質全体の構造変化が誘起され、活性状態が生成する。
レチナールの「ねじれ」の解消およびタンパク質部分の構造変化に伴って、光を吸収したロドプシンは種々の準安定な状態を経て変化していく。この準安定な状態を中間体と呼び、光吸収後から、フォト(photo)、バソ(batho)、ルミ(lumi)、メタ(meta)と名付けられた中間体が同定されている。これらの中間体は異なる吸収スペクトルを持つので、スペクトル変化によって熱反応を観測することができる[14]。反応の最終段階ではシッフ塩基のプロトンが対イオンに移動し、380 nmに吸収極大を示すメタロドプシンⅡ(Metarhodopsin II, Meta II)が生成する。Meta IIはGタンパク質活性化能をもつロドプシンの活性状態である[15]。
Meta IIはその前駆体Meta Iとの間でpH平衡にある(MetaI/IIの平衡はpH以外にも温度や膜の組成等で変化することが知られている)。 興味深いことに、平衡中の両者の量比は、シッフ塩基が脱プロトン化しているMeta IIが低pH(外液のプロトンが多い条件)で多くなり、プロトン化シッフ塩基を持つ Meta Iが高pHで多くなる。つまり、Meta II(活性状態)の生成には、シッフ塩基の脱プロトン化に伴う外界からのプロトンの取り込みが必要なことを示している。最近の研究によると、シッフ塩基の脱プロトン化がヘリックスの再配置(剛体運動)を誘起し、その結果、ERYモチーフが主となって形成するIonic lockが解除(E134がプロトン化)されることが知られている。ロドプシンの活性状態はこのような逐次的な構造変化によって生成するのである。
光シグナル伝達
Gタンパク質のシグナル
光を受容したロドプシンは数ミリ秒の間にGタンパク質を活性化する状態に変化する。ロドプシンと共役するGタンパク質はαβγのサブユットからなる3量体Gタンパク質である。Gタンパク質(guanine nucleotide-binding proteins: G-proteins)はGTPを結合すると「on」、GDPを結合すると「off」になる分子スイッチとして機能する。一般にoff状態では3量体として存在し、Gα中でGDP-GTP交換反応が起こると、GαはGβγと解離して活性状態になる。活性化したロドプシンは1秒間に数百のGタンパク質を活性化することができるため、大きなシグナル増幅作用がある。
活性化したGαはcGMPを5’-GMPに加水分解する酵素であるホスホジエステラーゼ(Phosphodiesterase、PDE)に作用する。PDEは酵素活性部位であるαβサブユニットとこれらと特異的に結合しその活性を抑制する2つのγサブユニットからなる。GαはこのPDEγに結合することによってγサブユニットの抑制効果を解除し、PDEを活性化する。
PDEが活性化すると細胞内のcGMPの濃度が急減し、cGMP依存性陽イオンチャネル(cyclic nucleotide-gated ion channels: CNG channels) が閉じる。光を受容したロドプシンからのシグナルがこない状態では、CNGチャネルは開いた状態であり、細胞内にNa+やCa2+が流入している。シグナルがくると上記の反応が起こるため、CNGチャネルが閉じ、細胞が過分極する。
視細胞は暗状態では少し脱分極しており、そのシナプス末端から神経伝達物質であるグルタミン酸が放出されている。光を受容して上記のシグナル伝達系が働くと過分極し、グルタミン酸の放出量が減少する。この変化が双極細胞などの下流の神経細胞に伝えられ、出力ニューロンである神経節細胞を経て脳にその情報が伝えられる。 (シグナル伝達についての参考文献[16][17])
シグナルのシャットダウンと視細胞の回復
光を受容して応答した視細胞は、次の光を受容するために速やかにもとの静止状態に戻る必要がある。素早く戻ることが光受容の時間分解能に関わるので、能動的に応答をシャットダウンし、もとの状態に戻ることが重要となる。
視細胞における光情報伝達はロドプシンの光受容によって始まり、また、ロドプシンからGタンパク質へのシグナル伝達の際に大きく増幅される。したがって、ロドプシンの活性状態を素早くシャットダウンすることは非常に重要である。ロドプシンの活性状態はメタロドプシンⅡ(Metarhodopsin II)と名付けられており、その名のとおり準安定(metastable)である。しかし、数十秒間は安定に存在する。視細胞の単一光子応答は1秒以内で終結するので、メタロドプシンⅡはこの時間よりも速くシャットダウン(不活性化)されている。メタロドプシンⅡの不活性化にはSer/Thrキナーゼであるロドプシンキナーゼ(Rhodopsin Kinase: RK)が関与する。メタロドプシンⅡはこの酵素によってリン酸化され、そのGタンパク質活性化能が減少する。メタロドプシンⅡのC末端領域にはリン酸化される部位として複数のS/Tが同定されている。生体内では主にS334, S338, S343がリン酸化される。さらにリン酸化されたメタロドプシンⅡにアレスチン(Arrestin)が結合することによってGタンパク質との結合が完全に阻害される。
桿体視細胞の応答プロファイルは非常に再現性が良い。これを実現するために、応答を複数のステップで制御する(Multi step shutoff)機構があると考えられている。ロドプシンの不活性化が単一ステップだけで起こる場合、分子間の相互作用は確率論的に起こるので、速くシャッタダウンする場合や遅くシャットダウンする場合がでてくる。このようにシャットダウンがランダムだと単一光子応答がばらつき、ノイズと区別できなくなる。そのため、ロドプシンは複数ステップのシャットダウン機構を用いることによってシャットダウン時間の可変性を平均化し応答の再現性を保証していると考えられている。
Gαは内在的なGTPase活性をもつ。そのため、GTPと結合して活性状態になったGαは自発的にGTPをGDPに加水分解し、再びGβγと結合して不活性状態に戻る。しかし、Gαの自発的な酵素活性は低く、視細胞内ではPDEγに結合したGαにGAP(GTPase Activating Protein)が作用することによってGTPの加水分解を加速している。Gαが不活性化しPDEから遊離するとPDEγは再び酵素活性部位であるPDEαβを阻害するためPDEの活性が抑えられる。
細胞が完全にもとの状態に戻るには、PDEの作用によって急減した細胞内cGMP濃度ももとに戻す必要がある。cGMPはGC(Guanyl Cyclase)によって定常的に合成されているが、GCの活性はGCAP (Guanyl Cyclase Activating Protein)によって調節されている。光応答により細胞膜のCNGチャネルが閉じ細胞内のCa2+濃度が下がるとGCAPはGCの活性を促進するようになる。細胞内カルシウムの増減によるこの制御機構をカルシウムフィードバック機構と呼ぶ。この機構により細胞内のcGMP濃度が速やかに上昇するとCNGチャネルも開き、視細胞が元の状態に戻る。なお、細胞内カルシウム濃度の減少により、ロドプシンキナーゼを制御する因子(Sモジュリンあるいはリカバリンと呼ばれている)も知られており[18]、GCAPとあわせて視細胞の明順応を説明する一つの機構と考えられている。
ロドプシン類
本来「ロドプシン」とは桿体視物質をあらわす言葉であった。しかし、生化学・分子生物学の進展により、桿体視細胞以外の光受容細胞や脊椎動物以外の生物種から相同性のある光受容タンパク質が続々と報告されるようになり[19][17]、これらの光受容タンパク質も「ロドプシン」あるいは「オプシン」と呼ばれるようになった。これら多くの発見の中でも、ニワトリの松果体に存在するピノプシンの発見は特筆される[20]。つまり、それまでに発見されていたロドプシン類はいわゆる「視覚」に関与する受容体であったが、ピノプシンは視覚以外の機能に関与する受容体であったからである。この発見以降、「視覚オプシン(visual opsin)」、「非視覚オプシン(non-visual opsin)」という言葉が使われるようになった。最近では1000種類以上のロドプシン遺伝子が報告されており、これらはGタンパク質共役型受容体(G Protein Coupled Receptor: GPCR)の一員であることが知られている。
GPCRはペプチド、ホルモン、匂いリンクの名前物質などのさまざまな化学物質を受容し、Gタンパク質を介する細胞内シグナル伝達機構を駆動する受容体である。GPCRによる外界からのシグナル受容はほとんどの細胞で観測され、細胞間のコミュニケーションを担う上でも非常に重要な受容体である。また、マウスやヒトではゲノム中で最も大きなタンパク質ファミリーであることが知られている。ロドプシン類はGPCRのメンバーであるが、分子内に内在性のリガンド(11-シス型のレチナール)を含んでいることが特徴である。
GPCRとロドプシン
ロドプシンはGPCRファミリー1の代表的な受容体として知られている。 実際ファミリー1のGPCRはrhodopsin-like GPCRとも呼ばれている。近年の結晶構造解析の結果、ロドプシンの立体構造、特に膜貫通領域の構造は、他のGPCRのそれらと酷似していることが証明された。しかし、非常に多様化しているGPCRのなかでロドプシンは必ずしも典型的なGPCRというわけではない。ロドプシンは11-シスレチナールを内在性のリガンドとしてもともと結合している。11-シスレチナールは、発色団として、また、インバースアゴニストとして働き、これはロドプシンのみの特徴である。
ウシロドプシンの一次配列は1982年に決定され[21]、その翌年にはクローニングされている[22]。そして2000年にはX線結晶解析により3次元立体構造モデルが提出された[23][24]。また、現在ではさまざまな中間状態や活性状態[25]、変異体などの立体構造も発表されている。一次構造の決定、クローニング、結晶構造決定などについては、種々のGPCRの中ではロドプシンで最初に行われた。ウシロドプシンのように大量の試料を比較的簡単に調製できるGPCRは珍しく、また内在性のリガンドを持つロドプシンは他のGPCRに較べて非常に安定でそのためロドプシンの研究は他の受容体よりも先に進んだ。こうしてロドプシンはGPCR研究のトップランナーとして研究されてきた経歴があり、GPCRファミリー1の代表的な受容体とされている。
ロドプシンがGPCRであると認知されるようになったのは数十年前からである。1986年にGPCRの一つβアドレナリン受容体の一次配列が決定されるとすでに解析されていたロドプシンの配列そしてその配列から予想される7回膜貫通構造が非常に似ていることが発見された。その後も次々に様々なGPCRの配列が決定され、これらは一大タンパク質ファミリーを形成することが明らかになった。
動物のロドプシンと菌のロドプシン
様々な動物で見つかっているロドプシン(オプシン)の他にバクテリアにも光感受性を持つレチナールタンパク質が含まれていることが知られている。1971年にOesterheltとStoeckniusは好塩菌の一種ハロバクテリウム・ハロビウム(最近ではハロバクテリウム・サリナラムという)にレチナールを発色団とする光受容タンパク質が存在することを発見し、このタンパク質をバクテリオロドプシン(bR)と命名した[26]。その後の研究により、bRは光駆動のプロトンポンプ活性を示すことがわかり、また、バクテリアにはbRを含めて4種類のレチナールタンパク質が存在することがわかった。bR以外にはハロロドプシン(hR)、センソリーロドプシン(sR)、センソリーロドプシンII(sRII、フォボロドプシン(pR)ともいう)である。hRは光駆動のクロライドポンプ、sRとsRIIはそれぞれ正・負の光走性に関与するロドプシンである。最近、緑藻類から光駆動のチャネル活性を示すロドプシン(チャネルロドプシン)が発見され、hRとともに、神経細胞のlight-manipulationに応用されている。さらに最近では、海洋のバクテリアにもbR様のロドプシンが含まれていることが発見され、地球上のエネルギー生産の半分程度がbR様のロドプシン類で担われていることが注目されている。また、遺伝子発現を調節するロドプシン類もアナベナから発見されるなど、バクテリアが持つロドプシン類の機能解析は最近の一つのトピックスになっている。
これらのバクテリアのロドプシン類も、動物のロドプシン類と同様に7回膜貫通領域をもち、発色団としてレチナールを用い、さらにその発色団はレチナールシッフ塩基結合を介してH7に結合している。ただし、動物のロドプシンは主に11-シス型のレチナールを発色団として持ち、光を受容して全トランスに異性化されて活性状態になるが、バクテリアのロドプシンは全トランス型のレチナールを発色団とし、光を吸収して13-シス型に異性化し、機能を発揮することがわかっている。また、バクテリアのロドプシンは活性状態になったあと熱反応で元の状態に戻る光反応サイクルを描く。7本膜貫通α-ヘリックス構造を持つことから、両タンパク質は進化的に系統関係があると考えられていたが、アミノ酸配列からは相同性の無いことが明らかにされている。しかしロドプシン類の中でも20%程度の相同性しか示さないものもあるので、たとえ共通の祖先タンパク質から進化しても遠縁な生物種間では変異が蓄積し有意な相同性がなくなっている可能性もある。
参考文献
- ↑ Dowling J
The Retina: An approachable part of the brain
The Belknap Press of Harvard Univ. Press:1987 - ↑
Shichida, Y., & Imai, H. (1998).
Visual pigment: G-protein-coupled receptor for light signals. Cellular and molecular life sciences : CMLS, 54(12), 1299-315. [PubMed:9893707] [WorldCat] [DOI] - ↑ Dowling J
The Retina: An approachable part of the brain
The Belknap Press of Harvard Univ. Press:1987 - ↑
Shichida, Y., & Imai, H. (1998).
Visual pigment: G-protein-coupled receptor for light signals. Cellular and molecular life sciences : CMLS, 54(12), 1299-315. [PubMed:9893707] [WorldCat] [DOI] - ↑
Okano, T., Kojima, D., Fukada, Y., Shichida, Y., & Yoshizawa, T. (1992).
Primary structures of chicken cone visual pigments: vertebrate rhodopsins have evolved out of cone visual pigments. Proceedings of the National Academy of Sciences of the United States of America, 89(13), 5932-6. [PubMed:1385866] [PMC] [WorldCat] [DOI] - ↑
Farrens, D.L., Altenbach, C., Yang, K., Hubbell, W.L., & Khorana, H.G. (1996).
Requirement of rigid-body motion of transmembrane helices for light activation of rhodopsin. Science (New York, N.Y.), 274(5288), 768-70. [PubMed:8864113] [WorldCat] [DOI] - ↑
Sakmar, T.P., Franke, R.R., & Khorana, H.G. (1989).
Glutamic acid-113 serves as the retinylidene Schiff base counterion in bovine rhodopsin. Proceedings of the National Academy of Sciences of the United States of America, 86(21), 8309-13. [PubMed:2573063] [PMC] [WorldCat] [DOI] - ↑
Robinson, P.R., Cohen, G.B., Zhukovsky, E.A., & Oprian, D.D. (1992).
Constitutively active mutants of rhodopsin. Neuron, 9(4), 719-25. [PubMed:1356370] [WorldCat] [DOI] - ↑
Hofmann, K.P., Scheerer, P., Hildebrand, P.W., Choe, H.W., Park, J.H., Heck, M., & Ernst, O.P. (2009).
A G protein-coupled receptor at work: the rhodopsin model. Trends in biochemical sciences, 34(11), 540-52. [PubMed:19836958] [WorldCat] [DOI] - ↑ K Nakanishi, V Baloghair, M Arnaboli, K Tsujimoto, and B Honig'
An External Point-Charge Model for Bacteriorhodopsin to Account for Its Purple Color
J Am Chem Soc:1980 - ↑
Kandori, H., Shichida, Y., & Yoshizawa, T. (2001).
Photoisomerization in rhodopsin. Biochemistry. Biokhimiia, 66(11), 1197-209. [PubMed:11743865] [WorldCat] [DOI] - ↑
Wald, G. (1968).
Molecular basis of visual excitation. Science (New York, N.Y.), 162(3850), 230-9. [PubMed:4877437] [WorldCat] [DOI] - ↑
Schoenlein, R.W., Peteanu, L.A., Mathies, R.A., & Shank, C.V. (1991).
The first step in vision: femtosecond isomerization of rhodopsin. Science (New York, N.Y.), 254(5030), 412-5. [PubMed:1925597] [WorldCat] [DOI] - ↑
YOSHIZAWA, T., & KITO, Y. (1958).
Chemistry of the rhodopsin cycle. Nature, 182(4649), 1604-5. [PubMed:13613341] [WorldCat] [DOI] - ↑
Emeis, D., Kühn, H., Reichert, J., & Hofmann, K.P. (1982).
Complex formation between metarhodopsin II and GTP-binding protein in bovine photoreceptor membranes leads to a shift of the photoproduct equilibrium. FEBS letters, 143(1), 29-34. [PubMed:6288450] [WorldCat] [DOI] - ↑
Yau, K.W., & Hardie, R.C. (2009).
Phototransduction motifs and variations. Cell, 139(2), 246-64. [PubMed:19837030] [PMC] [WorldCat] [DOI] - ↑ 17.0 17.1
Shichida, Y., & Matsuyama, T. (2009).
Evolution of opsins and phototransduction. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 364(1531), 2881-95. [PubMed:19720651] [PMC] [WorldCat] [DOI] - ↑
Kawamura, S. (1993).
Rhodopsin phosphorylation as a mechanism of cyclic GMP phosphodiesterase regulation by S-modulin. Nature, 362(6423), 855-7. [PubMed:8386803] [WorldCat] [DOI] - ↑
Terakita, A. (2005).
The opsins. Genome biology, 6(3), 213. [PubMed:15774036] [PMC] [WorldCat] [DOI] - ↑
Okano, T., Yoshizawa, T., & Fukada, Y. (1994).
Pinopsin is a chicken pineal photoreceptive molecule. Nature, 372(6501), 94-7. [PubMed:7969427] [WorldCat] [DOI] - ↑
Ovchinnikov YuA (1982).
Rhodopsin and bacteriorhodopsin: structure-function relationships. FEBS letters, 148(2), 179-91. [PubMed:6759163] [WorldCat] [DOI] - ↑
Nathans, J., & Hogness, D.S. (1983).
Isolation, sequence analysis, and intron-exon arrangement of the gene encoding bovine rhodopsin. Cell, 34(3), 807-14. [PubMed:6194890] [WorldCat] [DOI] - ↑
Palczewski, K., Kumasaka, T., Hori, T., Behnke, C.A., Motoshima, H., Fox, B.A., ..., & Miyano, M. (2000).
Crystal structure of rhodopsin: A G protein-coupled receptor. Science (New York, N.Y.), 289(5480), 739-45. [PubMed:10926528] [WorldCat] [DOI] - ↑
Okada, T., Fujiyoshi, Y., Silow, M., Navarro, J., Landau, E.M., & Shichida, Y. (2002).
Functional role of internal water molecules in rhodopsin revealed by X-ray crystallography. Proceedings of the National Academy of Sciences of the United States of America, 99(9), 5982-7. [PubMed:11972040] [PMC] [WorldCat] [DOI] - ↑
Choe, H.W., Kim, Y.J., Park, J.H., Morizumi, T., Pai, E.F., Krauss, N., ..., & Ernst, O.P. (2011).
Crystal structure of metarhodopsin II. Nature, 471(7340), 651-5. [PubMed:21389988] [WorldCat] [DOI] - ↑
Oesterhelt, D., & Stoeckenius, W. (1971).
Rhodopsin-like protein from the purple membrane of Halobacterium halobium. Nature: New biology, 233(39), 149-52. [PubMed:4940442] [WorldCat] [DOI]
(執筆者:松山オジョス武、七田芳則 担当編集委員:林康紀)