血清応答因子
英語名:serum response factor 英略称:SRF 独:Serum-Response-Faktor
SRFは、MADSボックス(MADS-box)ファミリーに属する転写因子である[1]。遺伝子のCC(A/T)6GG (CArG) ボックス[2]に二量体で結合し[3] [4]、c-fosなどの最初期遺伝子やβ-アクチンなど細胞骨格系遺伝子の発現を制御することが知られている[5]。SRFは、中胚葉形成などの胚発生[6]、筋分化[7]、心機能[7]、免疫系細胞の成熟[7]など多彩な生命現象に関与するとの指摘がある。中枢神経系においては、海馬の神経回路形成[8][9]、樹状突起や軸索形態[8][9][10][11]、シナプス機能[5][8][12]への関与、海馬や大脳皮質の層構造形成[10][11]、神経細胞移動[8][13]、末梢神経系においては後根神経節の軸索分岐形成や伸長への関与[14]が指摘されている。
歴史
血清刺激によって最初期遺伝子c-fosの発現誘導が起こる。c-fos遺伝子の転写開始点より上流に存在し、血清に応答して転写を制御する働きをもつ塩基配列を血清応答要素(serum response element, SRE)とよび、SREに結合する分子は血清応答因子(serum response factor, SRF)と名付けられた[15]。1988年にはSRF cDNAが単離され、ホモ二量体を形成してDNAに結合することが指摘された[3]。SRE配列には、CC(A/T)6GG、いわゆるCArGボックス[2]が含まれており、SRFが結合する配列はこのCArGボックスである。CArGボックスは、c-fosやegr-1などの最初期遺伝子だけではなく、アクチンなどの細胞骨格系遺伝子の上流にも存在していることが判明し、SRFの標的遺伝子候補となっている[5][16][17][18]。SRFを含む多くの転写因子群に共通して保存されている領域は、4つの転写因子(Minichromosome maintenance 1 protein, Agamous, Deficiens, SRF)の頭文字をとってMADSボックス(MADS-box)とよばれている[1]。したがってSRFはMADSボックスファミリーの一員である[1]。
構造
SRFは、N末端側に約56アミノ酸残基で構成されるMADSボックス[1]、それに続くSRFコファクターとの相互作用部位、C末端側に転写活性化ドメイン[19][20]を有する。MADSボックス内にDNA結合ドメイン、二量体形成ドメインが存在する[4]。
SRFコアホモ二量体とDNAとの複合体の構造がX線構造解析により明らかにされた[4]。SRFコアドメインは、約90アミノ酸残基から成り、DNA結合領域、二量体形成、SRFコファクターとの相互作用部位を持つ。SRFコアの各サブユニットから伸びる両親媒性α-ヘリックス(αI)が逆平行コイルドコイルを形成し、CArGボックスの小溝内に平行に配置している[4]。αIから伸びる塩基性N末端はDNAの主溝にはまり込んでいる[4]。また、βI、βIIから成る4本の逆平行βシートが二量体形成のための中心的要素となっている[4]。C末端は変則的なコイル構造と短いα-へリックス(αII)をとっている[4]。したがって、SRF コアドメインはDNA結合領域であるコイルドコイルの下層、βシートから成る中間層、C末端領域からなる上層という3層から成る。SRFコアホモ二量体と結合しているDNAは折れ曲がり、通常とは異なる構造をとっている[4]。
脳内発現
SRFは、あらゆる組織で発現が認められている。中枢神経系においては、梨状皮質、大脳皮質、線条体、海馬、扁桃体に比較的強い発現が認められており、特に梨状皮質、海馬歯状回、CA1で強く発現する[21]。一方、淡蒼球でほとんど認められず[21][22] 、中脳、視床下部では、弱いかほとんど認められない[21][22]。小脳においても顆粒細胞やプルキンエ細胞にも発現している[21][22]。また、発達過程に伴って発現は変化し、大脳皮質、海馬歯状回とCA1においては生後28日までに発現量が上昇し、大脳皮質では成体時まで発現が維持される[21]。また、末梢神経系においては、後根神経節での発現が報告されている[14]。
細胞内制御機構
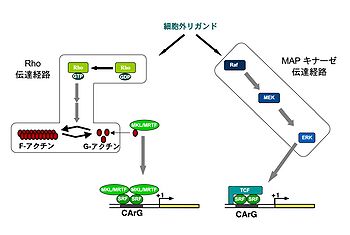
SRFは、血清[15]、神経成長因子(nerve growth factor, NGF)[14], 脳由来神経栄養因子(brain-derived neurotrophic factor, BDNF)[23]などの神経栄養因子、 (transforming growth factor- β, TGF-β)スーパーファミリー[24]、リゾホスファチジン酸(lysophosphatidic acid, LPA)[25]などの細胞外リガンドによって制御される。SRF制御のための細胞内情報伝達は、MAPキナーゼ伝達経路[25]と低分子量Gタンパク質Rho伝達経路の大きく2つがよく知られている(図)[25]。情報の最終到達点は、SRFによる標的遺伝子の制御であるが、SRFに結合して転写を制御するSRFコファクター(後述)が細胞内情報の統合や標的遺伝子の決定を行う可能性が指摘されている[25]。
コファクター
Ternary complex factor
Ternary complex factor(TCF)は、Ets-like transcription factor (Elk-1)、SRF accessory protein 1 (SAP-1)/Elk-4、New ets transcription factor (Net)/Ets-related protein(ERP)/SAP-2/Elk-3の3つが知られている[26]。TCFはE-twenty six (ETS)転写因子ファミリー(ETS transcription factor family)に属し、DNA結合ドメインであるETSドメインを持つ[26]。
TCFは、CArGボックス近傍のDNA配列(GGAA/T)とSRFに結合して三量体を形成し、下流遺伝子の発現を制御する[26]。またMAPキナーゼによりリン酸化されて活性調節される[26]。c-fos遺伝子の転写調節に重要な因子として同定され、解析が進んだ[27]。しかし、TCFによる転写の正負制御はシグナル、遺伝子、細胞の種類によって異なると考えられる。
MyocardinとMKL/MRTF
Myocardinとmegakaryoblastic leukemia (MKL)/myocardin-related transcription factor (MRTF)もSRFに結合するコファクターである[18]。MyocardinとMKL/MRTFは、ドメイン構造が類似しているが、アクチン動態のシグナルに対しては応答性が異なる[28]。主にNIH3T3細胞等の非神経細胞において、MKL/MRTFはRhoシグナル活性化によるアクチン細胞骨格の再編成によってG-アクチンから解離し、核移行して下流遺伝子の発現を制御するモデルが提唱されている(図)[29]。一方、myocardinはRhoシグナルに対する応答性は低いとされている[28]。MKL/MRTFは、異なる遺伝子にコードされるMKL1/MRTF-A (別名megakaryocytic acute leukemia (MAL), basic, SAP, and coiled-coil domain (BSAC))とMKL2/MRTF-B (別名MAL16)の2種類が知られている[25]。Myocardinは、心臓、骨格筋に高発現し、平滑筋関連遺伝子の発現を制御する[30]が、MKL1/MRTF-Aは、精巣と脳、MKL2/MRTF-Bは脳に高い発現が認められる[24]。MKL/MRTFによる転写の正負制御もシグナル、遺伝子、細胞の種類によって異なっていると考えられる。
標的遺伝子
SRFが結合して転写を制御しているかどうかは、遺伝子上流のCArGボックスの有無だけでは判断できないが、最初期遺伝子や多くの細胞骨格関連遺伝子にCArGボックスが存在している[16][17][18]。代表的なSRF標的遺伝子としてc-fos、egr-1などの最初期遺伝子[5]のほかにβ-アクチン遺伝子が知られている[5]。近年、神経系でのSRF標的遺伝子としてactivity-regulated cytoskeleton-associated protein (Arc)遺伝子が報告された[5][31]。
生理機能
SRFの標的遺伝子は、最初期遺伝子と細胞骨格関連遺伝子に分類されるものが多く、細胞レベルでも、これらに関連した細胞移動、細胞形態などの機能が示されている[32]。個体レベルでは、SRFノックアウト(KO)マウスは、胚発生時の中胚葉形成不全で胎生致死であるため[6]、現在では主にCre-loxPシステムを用いて作製した30種類近い組織•細胞特異的SRF KOマウスについて検討が進んでいる[7]。筋分化[7]、心機能[7]、免疫系細胞の成熟[7]など多彩な生命現象に関与すると報告されている。
中枢神経系を標的としたSRFKOマウスは、これまでに少なくとも8系統が報告されている。
CaMKIIαプロモーターでCreレコンビナーゼを発現させ、前脳特異的にSRFをKOしたマウスが3系統あるが[5][9][10][12][13]、それぞれCreレコンビナーゼの発現時期が異なるため、表現型は異なっている。周産期や出生前においてSRFをKOさせたマウス系統の解析では、脳室下帯から嗅球への細胞移動[13]、海馬層形成と樹状突起形態[10]、海馬における軸索ガイダンス[9]への重要性が示されている。また、神経細胞におけるSRF依存性遺伝子発現が細胞非自律的なオリゴデンドロサイトの分化に重要であるとの指摘がある[33]。また、生後8週目からSRFがKOされるマウス系統の解析では、海馬依存性即時記憶や長期抑圧現象に重要な役割を果たすこと指摘されている[12]。
一方、生後数ヶ月でSRFがKOされるマウス系統では、脳の構造上の変化や細胞移動の異常はなく、β-アクチンやc-fos, egr-1, Arc遺伝子というSRFの標的遺伝子の神経活動依存的発現が障害された。通常よりも豊かな環境(enriched environment, EE)においてもこのような異常は認められた[5]。さらに長期増強現象も障害されたことが報告されている[5]。
また、シナプシンI(synapsin I)プロモーターでCreレコンビナーゼを発現させ、SRFをKOしたマウス系統もあり、CaMKIIプロモーターの系統と行動学的な違いが比較検討されている[34]。
近年、ネスチン(nestin)プロモーターでCreレコンビナーゼを発現させた神経前駆細胞特異的SRF KOマウスとneuronal helix-loop-helix protein-1 (NEX) プロモーターを用いた大脳皮質と海馬のグルタミン酸作動性神経特異的SRF KOマウスが報告され、皮質軸索投射へのSRFの関与が指摘された[35]。
ドーパミンD1受容体発現細胞やドーパミンアクティブトランスポーター(dopamine active transporter, DAT)発現細胞を標的としたSRF KOマウスも作製されており、ドーパミンD1受容体発現細胞におけるSRF KOで多動症状を呈することが報告されている[36]。
末梢神経系においては、Wnt1プロモーターでCreレコンビナーゼを発現させた後根神経節特異的SRF KOマウスの解析により、軸索投射におけるSRFの重要性が指摘された[14]。
参考文献
- ↑ 1.0 1.1 1.2 1.3
Shore, P., & Sharrocks, A.D. (1995).
The MADS-box family of transcription factors. European journal of biochemistry, 229(1), 1-13. [PubMed:7744019] [WorldCat] [DOI] - ↑ 2.0 2.1
Miwa, T., & Kedes, L. (1987).
Duplicated CArG box domains have positive and mutually dependent regulatory roles in expression of the human alpha-cardiac actin gene. Molecular and cellular biology, 7(8), 2803-13. [PubMed:2823106] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1
Norman, C., Runswick, M., Pollock, R., & Treisman, R. (1988).
Isolation and properties of cDNA clones encoding SRF, a transcription factor that binds to the c-fos serum response element. Cell, 55(6), 989-1003. [PubMed:3203386] [WorldCat] [DOI] - ↑ 4.0 4.1 4.2 4.3 4.4 4.5 4.6 4.7
Pellegrini, L., Tan, S., & Richmond, T.J. (1995).
Structure of serum response factor core bound to DNA. Nature, 376(6540), 490-8. [PubMed:7637780] [WorldCat] [DOI] - ↑ 5.0 5.1 5.2 5.3 5.4 5.5 5.6 5.7 5.8
Ramanan, N., Shen, Y., Sarsfield, S., Lemberger, T., Schütz, G., Linden, D.J., & Ginty, D.D. (2005).
SRF mediates activity-induced gene expression and synaptic plasticity but not neuronal viability. Nature neuroscience, 8(6), 759-67. [PubMed:15880109] [WorldCat] [DOI] - ↑ 6.0 6.1
Arsenian, S., Weinhold, B., Oelgeschläger, M., Rüther, U., & Nordheim, A. (1998).
Serum response factor is essential for mesoderm formation during mouse embryogenesis. The EMBO journal, 17(21), 6289-99. [PubMed:9799237] [PMC] [WorldCat] [DOI] - ↑ 7.0 7.1 7.2 7.3 7.4 7.5 7.6
Miano, J.M. (2010).
Role of serum response factor in the pathogenesis of disease. Laboratory investigation; a journal of technical methods and pathology, 90(9), 1274-84. [PubMed:20498652] [WorldCat] [DOI] - ↑ 8.0 8.1 8.2 8.3
Knöll, B., & Nordheim, A. (2009).
Functional versatility of transcription factors in the nervous system: the SRF paradigm. Trends in neurosciences, 32(8), 432-42. [PubMed:19643506] [WorldCat] [DOI] - ↑ 9.0 9.1 9.2 9.3
Knöll, B., Kretz, O., Fiedler, C., Alberti, S., Schütz, G., Frotscher, M., & Nordheim, A. (2006).
Serum response factor controls neuronal circuit assembly in the hippocampus. Nature neuroscience, 9(2), 195-204. [PubMed:16415869] [WorldCat] [DOI] - ↑ 10.0 10.1 10.2 10.3
Stritt, C., & Knöll, B. (2010).
Serum response factor regulates hippocampal lamination and dendrite development and is connected with reelin signaling. Molecular and cellular biology, 30(7), 1828-37. [PubMed:20123976] [PMC] [WorldCat] [DOI] - ↑ 11.0 11.1
Lu, P.P., & Ramanan, N. (2011).
Serum response factor is required for cortical axon growth but is dispensable for neurogenesis and neocortical lamination. The Journal of neuroscience : the official journal of the Society for Neuroscience, 31(46), 16651-64. [PubMed:22090492] [PMC] [WorldCat] [DOI] - ↑ 12.0 12.1 12.2
Etkin, A., Alarcón, J.M., Weisberg, S.P., Touzani, K., Huang, Y.Y., Nordheim, A., & Kandel, E.R. (2006).
A role in learning for SRF: deletion in the adult forebrain disrupts LTD and the formation of an immediate memory of a novel context. Neuron, 50(1), 127-43. [PubMed:16600861] [WorldCat] [DOI] - ↑ 13.0 13.1 13.2
Alberti, S., Krause, S.M., Kretz, O., Philippar, U., Lemberger, T., Casanova, E., ..., & Nordheim, A. (2005).
Neuronal migration in the murine rostral migratory stream requires serum response factor. Proceedings of the National Academy of Sciences of the United States of America, 102(17), 6148-53. [PubMed:15837932] [PMC] [WorldCat] [DOI] - ↑ 14.0 14.1 14.2 14.3
Wickramasinghe, S.R., Alvania, R.S., Ramanan, N., Wood, J.N., Mandai, K., & Ginty, D.D. (2008).
Serum response factor mediates NGF-dependent target innervation by embryonic DRG sensory neurons. Neuron, 58(4), 532-45. [PubMed:18498735] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1
Treisman, R. (1986).
Identification of a protein-binding site that mediates transcriptional response of the c-fos gene to serum factors. Cell, 46(4), 567-74. [PubMed:3524858] [WorldCat] [DOI] - ↑ 16.0 16.1
Minty, A., & Kedes, L. (1986).
Upstream regions of the human cardiac actin gene that modulate its transcription in muscle cells: presence of an evolutionarily conserved repeated motif. Molecular and cellular biology, 6(6), 2125-36. [PubMed:3785189] [PMC] [WorldCat] [DOI] - ↑ 17.0 17.1
Miano, J.M. (2003).
Serum response factor: toggling between disparate programs of gene expression. Journal of molecular and cellular cardiology, 35(6), 577-93. [PubMed:12788374] [WorldCat] [DOI] - ↑ 18.0 18.1 18.2
Olson, E.N., & Nordheim, A. (2010).
Linking actin dynamics and gene transcription to drive cellular motile functions. Nature reviews. Molecular cell biology, 11(5), 353-65. [PubMed:20414257] [PMC] [WorldCat] [DOI] - ↑
Sharrocks, A.D., Gille, H., & Shaw, P.E. (1993).
Identification of amino acids essential for DNA binding and dimerization in p67SRF: implications for a novel DNA-binding motif. Molecular and cellular biology, 13(1), 123-32. [PubMed:8417320] [PMC] [WorldCat] [DOI] - ↑
Liu, S.H., Ma, J.T., Yueh, A.Y., Lees-Miller, S.P., Anderson, C.W., & Ng, S.Y. (1993).
The carboxyl-terminal transactivation domain of human serum response factor contains DNA-activated protein kinase phosphorylation sites. The Journal of biological chemistry, 268(28), 21147-54. [PubMed:8407951] [WorldCat] - ↑ 21.0 21.1 21.2 21.3 21.4
Stringer, J.L., Belaguli, N.S., Iyer, D., Schwartz, R.J., & Balasubramanyam, A. (2002).
Developmental expression of serum response factor in the rat central nervous system. Brain research. Developmental brain research, 138(1), 81-6. [PubMed:12234660] [WorldCat] [DOI] - ↑ 22.0 22.1 22.2
Herdegen, T., Blume, A., Buschmann, T., Georgakopoulos, E., Winter, C., Schmid, W., ..., & Gass, P. (1997).
Expression of activating transcription factor-2, serum response factor and cAMP/Ca response element binding protein in the adult rat brain following generalized seizures, nerve fibre lesion and ultraviolet irradiation. Neuroscience, 81(1), 199-212. [PubMed:9300412] [WorldCat] [DOI] - ↑
Kalita, K., Kharebava, G., Zheng, J.J., & Hetman, M. (2006).
Role of megakaryoblastic acute leukemia-1 in ERK1/2-dependent stimulation of serum response factor-driven transcription by BDNF or increased synaptic activity. The Journal of neuroscience : the official journal of the Society for Neuroscience, 26(39), 10020-32. [PubMed:17005865] [PMC] [WorldCat] [DOI] - ↑ 24.0 24.1
Ishikawa, M., Nishijima, N., Shiota, J., Sakagami, H., Tsuchida, K., Mizukoshi, M., ..., & Tabuchi, A. (2010).
Involvement of the serum response factor coactivator megakaryoblastic leukemia (MKL) in the activin-regulated dendritic complexity of rat cortical neurons. The Journal of biological chemistry, 285(43), 32734-43. [PubMed:20709749] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1 25.2 25.3 25.4
Posern, G., & Treisman, R. (2006).
Actin' together: serum response factor, its cofactors and the link to signal transduction. Trends in cell biology, 16(11), 588-96. [PubMed:17035020] [WorldCat] [DOI] - ↑ 26.0 26.1 26.2 26.3
Buchwalter, G., Gross, C., & Wasylyk, B. (2004).
Ets ternary complex transcription factors. Gene, 324, 1-14. [PubMed:14693367] [WorldCat] [DOI] - ↑
Shaw, P.E., Schröter, H., & Nordheim, A. (1989).
The ability of a ternary complex to form over the serum response element correlates with serum inducibility of the human c-fos promoter. Cell, 56(4), 563-72. [PubMed:2492906] [WorldCat] [DOI] - ↑ 28.0 28.1
Guettler, S., Vartiainen, M.K., Miralles, F., Larijani, B., & Treisman, R. (2008).
RPEL motifs link the serum response factor cofactor MAL but not myocardin to Rho signaling via actin binding. Molecular and cellular biology, 28(2), 732-42. [PubMed:18025109] [PMC] [WorldCat] [DOI] - ↑
Miralles, F., Posern, G., Zaromytidou, A.I., & Treisman, R. (2003).
Actin dynamics control SRF activity by regulation of its coactivator MAL. Cell, 113(3), 329-42. [PubMed:12732141] [WorldCat] [DOI] - ↑
Wang, Z., Wang, D.Z., Pipes, G.C., & Olson, E.N. (2003).
Myocardin is a master regulator of smooth muscle gene expression. Proceedings of the National Academy of Sciences of the United States of America, 100(12), 7129-34. [PubMed:12756293] [PMC] [WorldCat] [DOI] - ↑
Kawashima, T., Okuno, H., Nonaka, M., Adachi-Morishima, A., Kyo, N., Okamura, M., ..., & Bito, H. (2009).
Synaptic activity-responsive element in the Arc/Arg3.1 promoter essential for synapse-to-nucleus signaling in activated neurons. Proceedings of the National Academy of Sciences of the United States of America, 106(1), 316-21. [PubMed:19116276] [PMC] [WorldCat] [DOI] - ↑
Schratt, G., Philippar, U., Berger, J., Schwarz, H., Heidenreich, O., & Nordheim, A. (2002).
Serum response factor is crucial for actin cytoskeletal organization and focal adhesion assembly in embryonic stem cells. The Journal of cell biology, 156(4), 737-50. [PubMed:11839767] [PMC] [WorldCat] [DOI] - ↑
Stritt, C., Stern, S., Harting, K., Manke, T., Sinske, D., Schwarz, H., ..., & Knöll, B. (2009).
Paracrine control of oligodendrocyte differentiation by SRF-directed neuronal gene expression. Nature neuroscience, 12(4), 418-27. [PubMed:19270689] [WorldCat] [DOI] - ↑
Johnson, A.W., Crombag, H.S., Smith, D.R., & Ramanan, N. (2011).
Effects of serum response factor (SRF) deletion on conditioned reinforcement. Behavioural brain research, 220(2), 312-8. [PubMed:21329726] [WorldCat] [DOI] - ↑
Lu, P.P., & Ramanan, N. (2011).
Serum response factor is required for cortical axon growth but is dispensable for neurogenesis and neocortical lamination. The Journal of neuroscience : the official journal of the Society for Neuroscience, 31(46), 16651-64. [PubMed:22090492] [PMC] [WorldCat] [DOI] - ↑
Parkitna, J.R., Bilbao, A., Rieker, C., Engblom, D., Piechota, M., Nordheim, A., ..., & Schütz, G. (2010).
Loss of the serum response factor in the dopamine system leads to hyperactivity. FASEB journal : official publication of the Federation of American Societies for Experimental Biology, 24(7), 2427-35. [PubMed:20223941] [WorldCat] [DOI]