「Cre/loxPシステム」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| 1行目: | 1行目: | ||
Cre/loxPシステムとはloxP配列と呼ばれる[[wikipedia:ja:DNA|DNA]]配列に対しDNA組換え酵素Creが働くことにより生じる部位特異的組換え反応を利用した[[wikipedia:ja:遺伝子組換え実験|遺伝子組換え実験]]系である。本来、[[wikipedia:ja:バクテリオファージ|バクテリオファージ]]P1が[[wikipedia:ja:宿主|宿主]]である[[wikipedia:ja:大腸菌|大腸菌]]内で複製される際に自身のゲノムを環状化するための組換えシステムである。遺伝子工学、発生工学に分野で主にloxP配列間に存在する遺伝子をCre発現により欠失させるために使用される<ref><pubmed> 11340053 </pubmed></ref>。 | Cre/loxPシステムとはloxP配列と呼ばれる[[wikipedia:ja:DNA|DNA]]配列に対しDNA組換え酵素Creが働くことにより生じる部位特異的組換え反応を利用した[[wikipedia:ja:遺伝子組換え実験|遺伝子組換え実験]]系である。本来、[[wikipedia:ja:バクテリオファージ|バクテリオファージ]]P1が[[wikipedia:ja:宿主|宿主]]である[[wikipedia:ja:大腸菌|大腸菌]]内で複製される際に自身のゲノムを環状化するための組換えシステムである。遺伝子工学、発生工学に分野で主にloxP配列間に存在する遺伝子をCre発現により欠失させるために使用される<ref><pubmed> 11340053 </pubmed></ref>。 | ||
== Cre/loxPシステムとは == | == Cre/loxPシステムとは == | ||
Cre/loxPシステムとはloxP配列と呼ばれる[[wikipedia:ja:DNA|DNA]]配列に対しDNA組換え酵素Creが働くことにより生じる部位特異的組換え反応を利用した[[wikipedia:ja:遺伝子組換え実験|遺伝子組換え実験]]系である。この組換え反応は主に特定のDNA配列の除去に用いられるが、現在ではこの技術を応用して後述のCre発現依存的遺伝子欠損やCre発現依存的遺伝子発現など遺伝子発現実験などにも利用されている。最初は1987年にBrian Sauerにより酵母を用いた実験系で報告された<ref><pubmed> 3037344 </pubmed></ref>が、現在では多くの動物種で使用されている。 | Cre/loxPシステムとはloxP配列と呼ばれる[[wikipedia:ja:DNA|DNA]]配列に対しDNA組換え酵素Creが働くことにより生じる部位特異的組換え反応を利用した[[wikipedia:ja:遺伝子組換え実験|遺伝子組換え実験]]系である。この組換え反応は主に特定のDNA配列の除去に用いられるが、現在ではこの技術を応用して後述のCre発現依存的遺伝子欠損やCre発現依存的遺伝子発現など遺伝子発現実験などにも利用されている。最初は1987年にBrian Sauerにより酵母を用いた実験系で報告された<ref><pubmed> 3037344 </pubmed></ref>が、現在では多くの動物種で使用されている。 | ||
==基本要素== | ==基本要素== | ||
[[Image:Crelox図1.jpg|thumb|right|300px|''' | [[Image:Crelox図1.jpg|thumb|right|300px|'''図1 loxP配列が同方向の場合のCreによる組換え''']] | ||
[[Image:Crelox図2.jpg|thumb|right|300px|''' | [[Image:Crelox図2.jpg|thumb|right|300px|'''図2 loxP配列が異なる方向の場合のCreによる組換え''']] | ||
=== Creレコンビナーゼ=== | === Creレコンビナーゼ=== | ||
| 21行目: | 17行目: | ||
バクテリオファージP1ゲノムに由来する34bpのDNA配列で両末端に存在するCre結合部位13bpは対称に、中心部は非対称な配列になっている。 | バクテリオファージP1ゲノムに由来する34bpのDNA配列で両末端に存在するCre結合部位13bpは対称に、中心部は非対称な配列になっている。 | ||
5'-ATAACTTCGTATAGCATACATTATACGAAGTTAT-3' | <tt>5'-ATAACTTCGTATAGCATACATTATACGAAGTTAT-3' </tt> | ||
=== CreとloxP配列の反応 === | === CreとloxP配列の反応 === | ||
| 35行目: | 31行目: | ||
Creと変異[[エストロゲン]]受容体の融合タンパク質であるCre-ERタンパク質は通常[[wikipedia:ja:細胞質|細胞質]]に存在するが、エストロゲン誘導体である[[wikipedia:ja:タモキシフェン|タモキシフェン]]と結合することにより核内に移行し、loxP配列に対して組換えを起こす。これを利用してCre-loxPシステムの働く時期をタモキシフェン依存的に調節することが可能である(図3)<ref><pubmed> 7624356 </pubmed></ref>。 | Creと変異[[エストロゲン]]受容体の融合タンパク質であるCre-ERタンパク質は通常[[wikipedia:ja:細胞質|細胞質]]に存在するが、エストロゲン誘導体である[[wikipedia:ja:タモキシフェン|タモキシフェン]]と結合することにより核内に移行し、loxP配列に対して組換えを起こす。これを利用してCre-loxPシステムの働く時期をタモキシフェン依存的に調節することが可能である(図3)<ref><pubmed> 7624356 </pubmed></ref>。 | ||
[[Image:CreloxP図3.jpg|thumb|right|300px|''' | [[Image:CreloxP図3.jpg|thumb|right|300px|'''図3 タモキシフェンによるCre-ERの制御''']] | ||
=== loxP配列のバリエーション === | === loxP配列のバリエーション === | ||
| 41行目: | 37行目: | ||
Creによって組換わるものの、loxP配列とは組換わらないlox511, lox2272, loxFASなど種々の変異lox配列<ref><pubmed> 11418130 </pubmed></ref> | Creによって組換わるものの、loxP配列とは組換わらないlox511, lox2272, loxFASなど種々の変異lox配列<ref><pubmed> 11418130 </pubmed></ref> | ||
lox511: 5'-ATAACTTCGTATAGtATACATTATACGAAGTTAT-3' | <tt>lox511: 5'-ATAACTTCGTATAGtATACATTATACGAAGTTAT-3' | ||
lox2272: 5'-ATAACTTCGTATAGgATACtTTATACGAAGTTAT-3' | lox2272: 5'-ATAACTTCGTATAGgATACtTTATACGAAGTTAT-3' | ||
loxFAS: 5'-ATAACTTCGTATAtacctttcTATACGAAGTTAT-3' | loxFAS: 5'-ATAACTTCGTATAtacctttcTATACGAAGTTAT-3' </tt> | ||
(小文字はloxP配列と異なる塩基) | (小文字はloxP配列と異なる塩基) | ||
| 51行目: | 47行目: | ||
や染色体への遺伝子挿入が可能なlox RE, loxLE配列<ref><pubmed> 9016639 </pubmed></ref> | や染色体への遺伝子挿入が可能なlox RE, loxLE配列<ref><pubmed> 9016639 </pubmed></ref> | ||
lox RE: 5'-ATAACTTCGTATAGCATACATTATACGAAcggta-3' | <tt>lox RE: 5'-ATAACTTCGTATAGCATACATTATACGAAcggta-3' | ||
lox LE: 5'-taccgTTCGTATAGCATACATTATACGAAGTTAT-3' | lox LE: 5'-taccgTTCGTATAGCATACATTATACGAAGTTAT-3' </tt> | ||
(小文字はloxP配列と異なる塩基) | (小文字はloxP配列と異なる塩基) | ||
などが存在する。 | などが存在する。 | ||
=== Cre/loxPと類似のシステム === | === Cre/loxPと類似のシステム === | ||
| 64行目: | 59行目: | ||
[[wikipedia:ja:出芽酵母|出芽酵母]](''Saccharomyces cerevisiae'')由来の組換え酵素FLPとFRT配列 <ref><pubmed> 6286142 </pubmed></ref> | [[wikipedia:ja:出芽酵母|出芽酵母]](''Saccharomyces cerevisiae'')由来の組換え酵素FLPとFRT配列 <ref><pubmed> 6286142 </pubmed></ref> | ||
5'-GAAGTTCCTATTCTCTAGAAAGTATAGGAACTTC-3' | <tt>5'-GAAGTTCCTATTCTCTAGAAAGTATAGGAACTTC-3' </tt> | ||
もCre-loxPシステムと同様の目的で頻用される。 また、[[wikipedia:ja:醤油酵母|醤油酵母]](''Zygosaccharomyces rouxii'')由来の組換え酵素RとRS配列<ref><pubmed> 3889347 </pubmed></ref>、バクテリオファージMu由来の組換え酵素Ginとgix配列<ref><pubmed> 1836050 </pubmed></ref>も例は少ないものの使用されている。 | もCre-loxPシステムと同様の目的で頻用される。 また、[[wikipedia:ja:醤油酵母|醤油酵母]](''Zygosaccharomyces rouxii'')由来の組換え酵素RとRS配列<ref><pubmed> 3889347 </pubmed></ref>、バクテリオファージMu由来の組換え酵素Ginとgix配列<ref><pubmed> 1836050 </pubmed></ref>も例は少ないものの使用されている。 | ||
2012年7月24日 (火) 20:15時点における版
Cre/loxPシステムとはloxP配列と呼ばれるDNA配列に対しDNA組換え酵素Creが働くことにより生じる部位特異的組換え反応を利用した遺伝子組換え実験系である。本来、バクテリオファージP1が宿主である大腸菌内で複製される際に自身のゲノムを環状化するための組換えシステムである。遺伝子工学、発生工学に分野で主にloxP配列間に存在する遺伝子をCre発現により欠失させるために使用される[1]。
Cre/loxPシステムとは
Cre/loxPシステムとはloxP配列と呼ばれるDNA配列に対しDNA組換え酵素Creが働くことにより生じる部位特異的組換え反応を利用した遺伝子組換え実験系である。この組換え反応は主に特定のDNA配列の除去に用いられるが、現在ではこの技術を応用して後述のCre発現依存的遺伝子欠損やCre発現依存的遺伝子発現など遺伝子発現実験などにも利用されている。最初は1987年にBrian Sauerにより酵母を用いた実験系で報告された[2]が、現在では多くの動物種で使用されている。
基本要素
Creレコンビナーゼ
バクテリオファージP1に由来し、TypeIトポイソメラーゼに分類される約38kDaのDNA組換え酵素でloxP配列とよばれる塩基配列を認識し組換えを起こす。
loxP配列
バクテリオファージP1ゲノムに由来する34bpのDNA配列で両末端に存在するCre結合部位13bpは対称に、中心部は非対称な配列になっている。
5'-ATAACTTCGTATAGCATACATTATACGAAGTTAT-3'
CreとloxP配列の反応
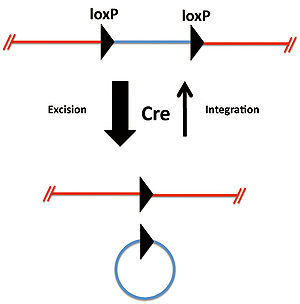
2つのloxP配列が同方向に位置している場合、CreによりloxP配列間の遺伝子は切り出され環状化する(図1)。この反応は平衡反応であるが切り出され環状化した遺伝子断片が元の状態に戻ることは極めて稀である。
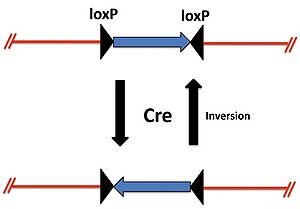
また、2つのloxP配列が異なる方向に位置しているときはCreによりloxP間の遺伝子は反転する(図2)。
バリエーション
Creのバリエーション
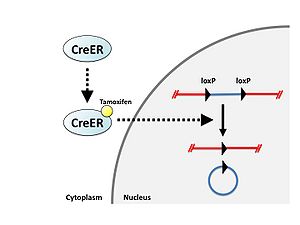
Creと変異エストロゲン受容体の融合タンパク質であるCre-ERタンパク質は通常細胞質に存在するが、エストロゲン誘導体であるタモキシフェンと結合することにより核内に移行し、loxP配列に対して組換えを起こす。これを利用してCre-loxPシステムの働く時期をタモキシフェン依存的に調節することが可能である(図3)[3]。
loxP配列のバリエーション
Creによって組換わるものの、loxP配列とは組換わらないlox511, lox2272, loxFASなど種々の変異lox配列[4]
lox511: 5'-ATAACTTCGTATAGtATACATTATACGAAGTTAT-3'
lox2272: 5'-ATAACTTCGTATAGgATACtTTATACGAAGTTAT-3'
loxFAS: 5'-ATAACTTCGTATAtacctttcTATACGAAGTTAT-3'
(小文字はloxP配列と異なる塩基)
や染色体への遺伝子挿入が可能なlox RE, loxLE配列[5]
lox RE: 5'-ATAACTTCGTATAGCATACATTATACGAAcggta-3'
lox LE: 5'-taccgTTCGTATAGCATACATTATACGAAGTTAT-3'
(小文字はloxP配列と異なる塩基)
などが存在する。
Cre/loxPと類似のシステム
出芽酵母(Saccharomyces cerevisiae)由来の組換え酵素FLPとFRT配列 [6]
5'-GAAGTTCCTATTCTCTAGAAAGTATAGGAACTTC-3'
もCre-loxPシステムと同様の目的で頻用される。 また、醤油酵母(Zygosaccharomyces rouxii)由来の組換え酵素RとRS配列[7]、バクテリオファージMu由来の組換え酵素Ginとgix配列[8]も例は少ないものの使用されている。
使用例
Cre発現依存的遺伝子欠損(コンディショナルノックアウト)
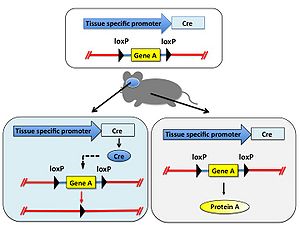
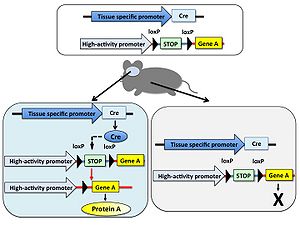
特定の遺伝子の機能をマウスなど動物個体で解析することを目的として、その遺伝子を欠損させた遺伝子ノックアウト動物が用いられる。しかし対象とする遺伝子の欠損により致死となるため目的の解析が出来ないことがある。そのような場合はある特定の組織だけで遺伝子を欠損することでこの問題を回避できる。 まず、欠損させたい遺伝子の両端にloxP配列をジーンターゲティング法により挿入した遺伝子改変動物を作製し、さらに組織特異的プロモーターの制御下でCreを発現するトランスジェニック動物を交配する。得られた動物個体内では組織特異的プロモーターが働く部位のみ遺伝子が欠損する。また、Creを発現するためのプロモーターを選択することにより、組織特異的な遺伝子ノックアウトだけではなく時期特異的な遺伝子ノックアウトも可能である(図4)。
Cre発現依存的遺伝子発現
ある遺伝子を細胞あるいは動物個体の組織で高発現させるためには組織特異的プロモーターの下流に目的の遺伝子をつなげた外来遺伝子を構築し導入する方法が用いられる。 しかし、そのプロモーターによる発現量が低かった場合には以下の方法を用いることでプロモーターの組織特性を保ちながら目的遺伝子の高発現が期待できる。 まず、組織特異的プロモーターの下流にCre遺伝子をつけた外来遺伝子およびサイトメガロウイルス(CMV)プロモーターなどの高発現が期待できるプロモーター下流に2つのloxP配列に挟まれたpolyA付加シグナルなどの転写停止配列、さらにその下流に目的の遺伝子をつなげた外来遺伝子をそれぞれ構築する。これらの外来遺伝子を同時に導入すると、組織特異的プロモーターが働く部位ではloxP配列間の転写停止配列が欠損し、その下流に位置する目的遺伝子が高発現する。一方、組織特異的プロモーターが働かない部位では転写停止配列が存在するため目的の遺伝子は発現しない(図5)。
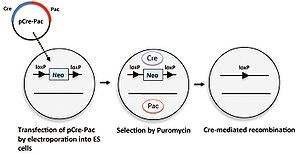
培養細胞における選択マーカーの除去
培養細胞に外来遺伝子を導入する際、細胞の遺伝子が導入された細胞を選択をするためにネオマイシン耐性遺伝子など薬剤耐性遺伝子を選択マーカーとして用いる。しかし、目的の細胞が得られた後にはこれらの選択マーカー遺伝子を発現するためのプロモーターが内在性のプロモーターに干渉する例があるために、選択後は除くことが望ましい。この場合にもCre/loxPシステムが利用される。細胞に導入する外来遺伝子の選択マーカーは2つのloxP配列で挟まれた形にしておく。この選択マーカーを除くためにCreとともにピューロマイシン耐性遺伝子を発現するプラスミドpCrePacを一過性に導入し、ピューロマイシン存在下で培養するとCreにより選択マーカーが除去された細胞のみが生存する(図6)[9]。
参考文献
- ↑
Van Duyne, G.D. (2001).
A structural view of cre-loxp site-specific recombination. Annual review of biophysics and biomolecular structure, 30, 87-104. [PubMed:11340053] [WorldCat] [DOI] - ↑
Sauer, B. (1987).
Functional expression of the cre-lox site-specific recombination system in the yeast Saccharomyces cerevisiae. Molecular and cellular biology, 7(6), 2087-96. [PubMed:3037344] [PMC] [WorldCat] [DOI] - ↑
Metzger, D., Clifford, J., Chiba, H., & Chambon, P. (1995).
Conditional site-specific recombination in mammalian cells using a ligand-dependent chimeric Cre recombinase. Proceedings of the National Academy of Sciences of the United States of America, 92(15), 6991-5. [PubMed:7624356] [PMC] [WorldCat] [DOI] - ↑
Siegel, R.W., Jain, R., & Bradbury, A. (2001).
Using an in vivo phagemid system to identify non-compatible loxP sequences. FEBS letters, 499(1-2), 147-53. [PubMed:11418130] [WorldCat] [DOI] - ↑
Araki, K., Araki, M., & Yamamura, K. (1997).
Targeted integration of DNA using mutant lox sites in embryonic stem cells. Nucleic acids research, 25(4), 868-72. [PubMed:9016639] [PMC] [WorldCat] [DOI] - ↑
Broach, J.R., Guarascio, V.R., & Jayaram, M. (1982).
Recombination within the yeast plasmid 2mu circle is site-specific. Cell, 29(1), 227-34. [PubMed:6286142] [WorldCat] [DOI] - ↑
Araki, H., Jearnpipatkul, A., Tatsumi, H., Sakurai, T., Ushio, K., Muta, T., & Oshima, Y. (1985).
Molecular and functional organization of yeast plasmid pSR1. Journal of molecular biology, 182(2), 191-203. [PubMed:3889347] [WorldCat] [DOI] - ↑
Maeser, S., & Kahmann, R. (1991).
The Gin recombinase of phage Mu can catalyse site-specific recombination in plant protoplasts. Molecular & general genetics : MGG, 230(1-2), 170-6. [PubMed:1836050] [WorldCat] [DOI] - ↑
Taniguchi, M., Sanbo, M., Watanabe, S., Naruse, I., Mishina, M., & Yagi, T. (1998).
Efficient production of Cre-mediated site-directed recombinants through the utilization of the puromycin resistance gene, pac: a transient gene-integration marker for ES cells. Nucleic acids research, 26(2), 679-80. [PubMed:9421534] [PMC] [WorldCat] [DOI]
(執筆者:平林敬浩、八木健 担当編集委員:大隅典子)