プロモーター
喜田 聡、芹田龍郎
東京農業大学 応用生物科学部バイオサイエンス学科
DOI:10.14931/bsd.4580 原稿受付日:2013年12月9日 原稿完成日:2014年2月8日
担当編集委員:林 康紀(独立行政法人理化学研究所 脳科学総合研究センター)
英語名:promoter 独:Promotor, Promoter 仏:promoteur
プロモーターとはDNAにおける転写制御を担う領域である。転写調節因子群の働きにより、転写基本因子群とRNAポリメラーゼがプロモーター上に転写開始複合体を形成し、転写が開始される。特に、mRNAの転写は、それぞれの細胞において、発現させる遺伝子を選択する、また、遺伝子の発現量を規定する重要なステップであるため、プロモーターにおける転写制御は生命現象の中心的な反応であるとも言える。
プロモーターとは
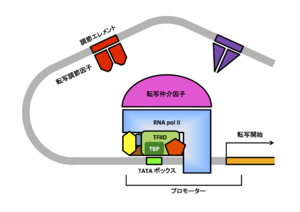
転写調節因子、転写仲介因子の働きにより、TFIID中心とする基本転写因子群とRNA pol IIがTATAやイニシエーターを標的としてプロモーター上に誘引され、転写開始複合体を形成し、転写反応が開始される。
プロモーターとはDNAにおける遺伝子の転写制御を行う領域である(図1)。
真核生物において、狭義では、転写基本因子群とRNAポリメラーゼが結合するDNA領域をプロモーターと呼ぶ[1]。
一方、広義では、この領域をコアプロモーターと呼び、転写調節因子群が結合する調節エレメントも含めてプロモーターと呼ぶことも多い。コアプロモーターの上流あるいは下流には様々な転写調節因子が結合して正負の転写制御が行われる調節エレメントが存在している。
コアプロモーター
転写基本因子とRNAポリメラーゼが結合して転写開始複合体が形成される、転写開始に必要最小限のDNA領域であり、転写開始点の前後の数十base pair (bp)程度の大きさである[1]。
分類
真核生物の場合、RNA合成酵素であるRNAポリメラーゼはPolI、PolII、PolIIの3種類存在し、3つのクラスのプロモーターが存在している。PolIが働くクラスIのプロモーターからはrRNA、PolIIが働くクラスIIのプロモーターからはmRNA、PolIIIが働くクラスIIIのプロモーターからはtRNAを中心とする低分子RNAが転写される[1]。特に、mRNAの転写制御を担うクラスIIのプロモーターに関しては、各遺伝子がそれぞれ異なる転写制御を受けるため、多様な転写調節機構が存在している。
クラスIIのプロモーターには、転写開始点を含むイニシエーター、また、転写開始点上流にTATAボックスやTFIIB認識領域などが存在する[1]。
転写基本因子
TFIIA, TFIIB, TFIID, TFIIE, TFIIF, TFIIHなどが存在し、RNA polIIと共にコアプロモーター上に転写開始複合体を形成することで、RNApolIIに正確な位置(転写開始点)から転写を開始させる役割を果たしている1)。転写基本因子群の多くはサブユニット構造をとる複合体であり、特に、TFIIDはTATAボックス結合タンパク質(TATA-binding protein; TBP)とそのサブユニット群TBP結合因子(TBP-associated factor; TAF)から構成され、転写開始複合体を形成する上で中心的な役割を果たす[2][3][4]。興味深いことに、TBPはTATAボックス認識に貢献するばかりではなく、以下に紹介するTATAレスプロモーターからの転写、さらには、クラスI及びクラスIIIのプロモーターからの転写にも必要であることが明らかにされており、真核細胞における転写制御において必須な役割を果たしている[5][6]。
TATAボックスとTATAレスプロモーター
TATAボックスとは転写開始点の約25bp程度上流に存在する塩基配列(TATAA)であり、TFIIDのサブユニットの一つTBPによって認識される[1][7]。しかし、クラスIIプロモーターにおいて、TATAボックスを有するプロモーターは意外に少なく(ヒトでは約30%)、これ以外はTATAボックスを持たずに、イニシエーターと下流プロモーター配列 (downstream promoter element; DPE)を有するTATAレスプロモーターであり、GCボックスを有することも多い[1][7]。TATAレスプロモーターはハウスキーピング遺伝子に多く見られ、複数の転写開始点を持つことも多い[7]。TATAボックスを有するプロモーターでは転写制御が厳格かつ劇的に行われる傾向が高いのに対して、TATAレスプロモーターには恒常的な活性を示すものが多い。
調節エレメント
転写調節因子が認識して結合する特異的配列を示すDNA領域のことを調節エレメントと呼び、この調節エレメントを介してプロモーターからの転写がコントロールされている[1][7])。調節エレメントはコアプロモーター近傍に存在するのみではなく、プロモーター領域の上流や下流に存在しており、転写開始点から離れた調節エレメントからの遠隔制御も行われている[1][7])。プロモーターから距離があり、アクティベーターが結合する領域をエンハンサー、一方、リプレッサーが結合する領域をサイレンサーと呼ぶ[1][7]。また、情報伝達経路の下流に存在する転写調節因子が結合する調節配列を応答エレメントと呼ぶ。Activity-regulated cytoskeleton-associated protein (Arc) 遺伝子では、転写開始点から約7kpbも上流に強力な応答エレメントを有するエンハンサーが存在することが示されている[8]。
例えば、cAMP情報伝達経路の下流に存在する応答エレメントはcAMP応答配列(cAMP responsive element; CRE)である[9][10]。細胞内cAMPの濃度上昇に伴い活性化されたAキナーゼ(Protein kinase A; PKA)により転写調節因子CRE結合タンパク質(CREB)がリン酸化を受け、リン酸化型CREBがCREを介して転写活性化を誘導する[9][10]。また、この転写活性化には、リン酸化型CREBのみが相互作用するコアクティベーターCREB結合タンパク質(CBP)が必須である[11]。また、応答エレメントには、カルシウムイオン情報伝達経路下流に存在するものや[12]、エストロゲン、アンドロゲン、糖質コルチコイド、ビタミンD、レチノイン酸などをそれぞれ特異的リガンドとする核内受容体群が結合するものなどが存在する[13]。
さらに、組織・細胞特異的な転写調節因子がおのおのの調節エレメントに結合することで、組織・細胞特異的遺伝子発現も制御されている。αカルシウムカルモジュリン依存性キナーゼII遺伝子の転写開始点より上流約8kbのプロモーター領域(αCaMKIIプロモーター)は前脳領域の興奮性ニューロン特異的に働く強力なプロモーターとして神経科学研究によく用いられており、この8kb内に組織特異的を示すための必要十分な構成要素が含まれているものと考えられる[14]。
転写調節機構
転写調節因子
特異的DNA配列を認識して結合する転写調節因子群によって転写が正負に制御される。転写調節因子には、転写を活性化させるアクティベーターと、逆に、転写を抑制するリプレッサーが存在する[1][7]。アクティベーターは、プロモーター上に転写基本因子群とRNApolIIとによる転写開始複合体の形成を促す[1][7]。アクティベーターの多くは、転写基本因子やPolIIに直接相互作用するわけではなく、DNAに結合しない転写仲介(媒介)因子、コファクター群を介して転写開始複合体の形成を促進する[1][7]。特に、自身はDNAに結合しないものの、転写調節因子群と相互作用し、転写を活性化する仲介因子をコアクティベーター、逆に、転写を抑制する仲介因子をコリプレッサーと呼ぶ[1][7]。転写仲介因子やコファクターも多数のタンパク質からなる複合体を形成しており、しかも、転写基本因子群やpolIIもサブユニットにより構成されることから、プロモーター上には多種多様なタンパク質群が結集することで転写活性化が誘導されることが明らかにされている[1][7]。
エピジェネティクス制御
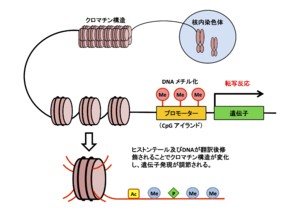
ヒストンテールがメチル化、アセチル化、リン酸化などの翻訳後修飾を受けて、クロマチン構造のリモデリングが引き起こされる。一方、プロモーター領域DNAのCpGアイランドがメチル化修飾を受けると、そのプロモーターからの転写が抑制される。このようなクロマチンリモデリングは転写制御の重要なステップとなっている。以上のような、クロマチンリモデリングやDNAメチル化はエピジェネティクス転写制御と呼ばれる。
転写制御は転写調節因子、転写基本因子、転写仲介因子やコファクターの間でのタンパク質間相互作用によってのみ制御されるわけではない。プロモーター周辺領域のエピジェネティクス制御も転写制御に必須であることが1990年代の中頃から明らかにされた[1][7]。具体的には、DNAと結合してクロマチン構造を形成するヒストン群の翻訳後修飾、すなわち、アセチル化(脱アセチル化)、メチル化(脱メチル化)、リン酸化(脱リン酸化)を介して、プロモーター付近のクロマチン構造の変換(クロマチンリモデリンング)が行われる(図2)。このクロマチンリモデリングがプロモーターからの転写開始の効率、すなわち、転写を制御するタンパク質群のプロモーター領域への集合に大きく影響を及ぼしていると考えられている[1][7]。以上のようなヒストンの翻訳後修飾にも多様性が観察されることから、ヒストンの修飾状態は「ヒストンコード」と呼ばれ、DNAの塩基配列情報の如く、ヒストンの修飾パターンが何らかの暗号的意味を持つのではないかと考えられている[15]。また、CLOCKなどのアクティベーター[16]、CBP/p300、PCAFなどのコアクティベーターはヒストンアセチル化酵素活性を有すること[17][18]、一方、Sin3などのコリプレッサーはヒストン脱アセチル化酵素活性を示すことが明らかにされており[19]、このような転写因子群がプロモーター付近のヒストンリモデリングを直接制御する。
一方、このエピジェネティクス制御はヒストンばかりではなく、DNAにおいても観察される。DNAのメチル化はCpGアイランドのシトシンに多く観察されており、メチル化に富んだプロモーターは不活性化状態となり、その遺伝子からの転写が抑制される[1][7]。DNAメチル化による転写不活性化はインプリンティング、X染色体不活性化を代表として、恒常的な転写制御に関与するものと考えられている。
エピジェネティクス制御は、より大規模なクロマチン構造の変化にも関与しており、ユークロマチンとヘテロクロマチンの変換といった、よりマクロなレベルのクロマチンリモデリングも含んでおり、細胞特異的遺伝子発現を制御するマスターレギュレーター的な働きを担っている[1][7]。また、遺伝子の境界を決定するDNA領域であるインシュレーターなどもこのようなクロマチン構造に大きな影響を及ぼすものと考えられる[1][7]。
脳科学研究領域におけるプロモーターの応用
αCaMKIIプロモーターは遺伝子操作マウス作製に広く利用されている[14]。これ以外には、外来遺伝子の発現をGABAニューロンに限定されるためのGAD67プロモーター[20]、ドーパミン産生ニューロンに限定させるためのチロシン水酸化酵素プロモーター[21]、ドーパミンD1受容体あるいはD2受容体発現ニューロンに限定させるためのD1及びD2受容体プロモーターなどが利用されている[22]。一方、神経活動依存的遺伝子発現の活性化をモニターする、あるいは、遺伝子発現が誘導されたニューロンを標識するためのツールとして、c−fos遺伝子あるいはArc遺伝子のプロモーターが利用されている[18][23]。以上のプロモーター群は脳科学領域における強力な遺伝学的手法のツールとなっている。
関連項目
参考文献
- ↑ 1.00 1.01 1.02 1.03 1.04 1.05 1.06 1.07 1.08 1.09 1.10 1.11 1.12 1.13 1.14 1.15 1.16 1.17 1.18 Jocelyn E Krebs, Elliott S Goldstein, Stephen T Kilpatrick
Genes XI, 11 edition
Jones & Bartlett Learning, Burlington, 2013
- ↑
Goodrich, J.A., & Tjian, R. (1994).
TBP-TAF complexes: selectivity factors for eukaryotic transcription. Current opinion in cell biology, 6(3), 403-9. [PubMed:7917332] [WorldCat] [DOI] - ↑
Burley, S.K., & Roeder, R.G. (1996).
Biochemistry and structural biology of transcription factor IID (TFIID). Annual review of biochemistry, 65, 769-99. [PubMed:8811195] [WorldCat] [DOI] - ↑
Papai, G., Weil, P.A., & Schultz, P. (2011).
New insights into the function of transcription factor TFIID from recent structural studies. Current opinion in genetics & development, 21(2), 219-24. [PubMed:21420851] [PMC] [WorldCat] [DOI] - ↑
Rigby, P.W. (1993).
Three in one and one in three: it all depends on TBP. Cell, 72(1), 7-10. [PubMed:8422684] [WorldCat] [DOI] - ↑
Pugh, B.F., & Tjian, R. (1991).
Transcription from a TATA-less promoter requires a multisubunit TFIID complex. Genes & development, 5(11), 1935-45. [PubMed:1657708] [WorldCat] [DOI] - ↑ 7.00 7.01 7.02 7.03 7.04 7.05 7.06 7.07 7.08 7.09 7.10 7.11 7.12 7.13 7.14 7.15 Tom Strachan, Andrew Read
Human Molecular Genetics, Fourth Edition
Garland Science, New York, 2010 - ↑
Kawashima, T., Okuno, H., Nonaka, M., Adachi-Morishima, A., Kyo, N., Okamura, M., ..., & Bito, H. (2009).
Synaptic activity-responsive element in the Arc/Arg3.1 promoter essential for synapse-to-nucleus signaling in activated neurons. Proceedings of the National Academy of Sciences of the United States of America, 106(1), 316-21. [PubMed:19116276] [PMC] [WorldCat] [DOI] - ↑ 9.0 9.1
Silva, A.J., Kogan, J.H., Frankland, P.W., & Kida, S. (1998).
CREB and memory. Annual review of neuroscience, 21, 127-48. [PubMed:9530494] [WorldCat] [DOI] - ↑ 10.0 10.1
Mayr, B., & Montminy, M. (2001).
Transcriptional regulation by the phosphorylation-dependent factor CREB. Nature reviews. Molecular cell biology, 2(8), 599-609. [PubMed:11483993] [WorldCat] [DOI] - ↑
Chrivia, J.C., Kwok, R.P., Lamb, N., Hagiwara, M., Montminy, M.R., & Goodman, R.H. (1993).
Phosphorylated CREB binds specifically to the nuclear protein CBP. Nature, 365(6449), 855-9. [PubMed:8413673] [WorldCat] [DOI] - ↑
Tao, X., Finkbeiner, S., Arnold, D.B., Shaywitz, A.J., & Greenberg, M.E. (1998).
Ca2+ influx regulates BDNF transcription by a CREB family transcription factor-dependent mechanism. Neuron, 20(4), 709-26. [PubMed:9581763] [WorldCat] [DOI] - ↑
Bookout, A.L., Jeong, Y., Downes, M., Yu, R.T., Evans, R.M., & Mangelsdorf, D.J. (2006).
Anatomical profiling of nuclear receptor expression reveals a hierarchical transcriptional network. Cell, 126(4), 789-99. [PubMed:16923397] [PMC] [WorldCat] [DOI] - ↑ 14.0 14.1
Mayford, M., Wang, J., Kandel, E.R., & O'Dell, T.J. (1995).
CaMKII regulates the frequency-response function of hippocampal synapses for the production of both LTD and LTP. Cell, 81(6), 891-904. [PubMed:7781066] [WorldCat] [DOI] - ↑
Jenuwein, T., & Allis, C.D. (2001).
Translating the histone code. Science (New York, N.Y.), 293(5532), 1074-80. [PubMed:11498575] [WorldCat] [DOI] - ↑
Doi, M., Hirayama, J., & Sassone-Corsi, P. (2006).
Circadian regulator CLOCK is a histone acetyltransferase. Cell, 125(3), 497-508. [PubMed:16678094] [WorldCat] [DOI] - ↑
Spencer, T.E., Jenster, G., Burcin, M.M., Allis, C.D., Zhou, J., Mizzen, C.A., ..., & O'Malley, B.W. (1997).
Steroid receptor coactivator-1 is a histone acetyltransferase. Nature, 389(6647), 194-8. [PubMed:9296499] [WorldCat] [DOI] - ↑ 18.0 18.1
Chan, H.M., & La Thangue, N.B. (2001).
p300/CBP proteins: HATs for transcriptional bridges and scaffolds. Journal of cell science, 114(Pt 13), 2363-73. [PubMed:11559745] [WorldCat] - ↑
Heinzel, T., Lavinsky, R.M., Mullen, T.M., Söderstrom, M., Laherty, C.D., Torchia, J., ..., & Rosenfeld, M.G. (1997).
A complex containing N-CoR, mSin3 and histone deacetylase mediates transcriptional repression. Nature, 387(6628), 43-8. [PubMed:9139820] [WorldCat] [DOI] - ↑
Abe, H., Yanagawa, Y., Kanbara, K., Maemura, K., Hayasaki, H., Azuma, H., ..., & Watanabe, M. (2005).
Epithelial localization of green fluorescent protein-positive cells in epididymis of the GAD67-GFP knock-in mouse. Journal of andrology, 26(5), 568-77. [PubMed:16088032] [WorldCat] [DOI] - ↑
Witten, I.B., Steinberg, E.E., Lee, S.Y., Davidson, T.J., Zalocusky, K.A., Brodsky, M., ..., & Deisseroth, K. (2011).
Recombinase-driver rat lines: tools, techniques, and optogenetic application to dopamine-mediated reinforcement. Neuron, 72(5), 721-33. [PubMed:22153370] [PMC] [WorldCat] [DOI] - ↑
Kravitz, A.V., Freeze, B.S., Parker, P.R., Kay, K., Thwin, M.T., Deisseroth, K., & Kreitzer, A.C. (2010).
Regulation of parkinsonian motor behaviours by optogenetic control of basal ganglia circuitry. Nature, 466(7306), 622-6. [PubMed:20613723] [PMC] [WorldCat] [DOI] - ↑
Reijmers, L.G., Perkins, B.L., Matsuo, N., & Mayford, M. (2007).
Localization of a stable neural correlate of associative memory. Science (New York, N.Y.), 317(5842), 1230-3. [PubMed:17761885] [WorldCat] [DOI]