「GKAP」の版間の差分
細 →発現 |
細編集の要約なし |
||
| (2人の利用者による、間の2版が非表示) | |||
| 25行目: | 25行目: | ||
4つのアイソフォームはアミノ酸レベルで40-50%の相同性があり、いずれも、中央部に14アミノ酸からなる5回の繰り返し配列を持ち、その部位でPSD-95、[[MAGI2]]/S-SCAMのguanylate kinase領域に結合する。繰り返し配列よりN末端側には特徴的配列がないが、ニューロフィラメントなどの中間径フィラメントに結合する<ref><pubmed>10759891</pubmed></ref>。繰り返し配列のC末端側のプロリンリッチ配列を介して、チロシンキナーゼAbl/Arg結合タンパク質であるNArgBP2のSH3領域に結合する<ref><pubmed>10521485</pubmed></ref>。さらにそれよりもC末端側でダイニン軽鎖LC1、LC8に結合するが、その部分には特徴的配列を欠く<ref><pubmed>10844022</pubmed></ref><ref name="Haraguchi"><pubmed>11122378</pubmed></ref>。C末端にPDZ結合モチーフをもち、ShankのPDZ領域に結合する<ref><pubmed>10433268</pubmed></ref><ref><pubmed>10527873</pubmed></ref>。その他、[[神経型一酸化窒素合成酵素]](nNOS)とも結合する<ref name="Haraguchi"><pubmed>11122378</pubmed></ref>。DLGAP3は、[[FAK]]、[[PYK2]]と相互作用すると報告されている<ref><pubmed>16202977</pubmed></ref>。[[カルシウム/カルモジュリン依存性蛋白質キナーゼII|カルシウム/カルモジュリン依存性タンパク質キナーゼII]](CaMKII)、p38 [[MAPK]]により[[リン酸化]]されることも報告されている<ref><pubmed>19896464</pubmed></ref><ref><pubmed>15729360</pubmed></ref>。相互作用分子の研究には、DLGAP1のラット、マウスホモログが用いられていることが多いが、アミノ酸配列の相同性に照らすと、報告されている相互作用の多くは、種とアイソフォームを超えて保存されていると推測される。 | 4つのアイソフォームはアミノ酸レベルで40-50%の相同性があり、いずれも、中央部に14アミノ酸からなる5回の繰り返し配列を持ち、その部位でPSD-95、[[MAGI2]]/S-SCAMのguanylate kinase領域に結合する。繰り返し配列よりN末端側には特徴的配列がないが、ニューロフィラメントなどの中間径フィラメントに結合する<ref><pubmed>10759891</pubmed></ref>。繰り返し配列のC末端側のプロリンリッチ配列を介して、チロシンキナーゼAbl/Arg結合タンパク質であるNArgBP2のSH3領域に結合する<ref><pubmed>10521485</pubmed></ref>。さらにそれよりもC末端側でダイニン軽鎖LC1、LC8に結合するが、その部分には特徴的配列を欠く<ref><pubmed>10844022</pubmed></ref><ref name="Haraguchi"><pubmed>11122378</pubmed></ref>。C末端にPDZ結合モチーフをもち、ShankのPDZ領域に結合する<ref><pubmed>10433268</pubmed></ref><ref><pubmed>10527873</pubmed></ref>。その他、[[神経型一酸化窒素合成酵素]](nNOS)とも結合する<ref name="Haraguchi"><pubmed>11122378</pubmed></ref>。DLGAP3は、[[FAK]]、[[PYK2]]と相互作用すると報告されている<ref><pubmed>16202977</pubmed></ref>。[[カルシウム/カルモジュリン依存性蛋白質キナーゼII|カルシウム/カルモジュリン依存性タンパク質キナーゼII]](CaMKII)、p38 [[MAPK]]により[[リン酸化]]されることも報告されている<ref><pubmed>19896464</pubmed></ref><ref><pubmed>15729360</pubmed></ref>。相互作用分子の研究には、DLGAP1のラット、マウスホモログが用いられていることが多いが、アミノ酸配列の相同性に照らすと、報告されている相互作用の多くは、種とアイソフォームを超えて保存されていると推測される。 | ||
== 発現 == | == 発現 == | ||
神経細胞以外にもmRNAはあり、タンパク質も発現していると考えられるが、神経細胞における発現が極めて高い。脳内での各種アイソフォームの発現については、mRNAレベルでは4つのアイソフォームについて、タンパク質レベルではDLGAP2以外の3つのアイソフォームについて、ラット、マウス脳を用いた解析結果が報告されている<ref><pubmed>15207911</pubmed></ref><ref><pubmed>15024750</pubmed></ref>。ちなみに、「異なるアイソフォームの分布は、オーバーラップしているが、それぞれに特異的である」と表現される。例えば、小脳を例にとると、[http://mouse.brain-map.org/experiment/show/70528599 DLGAP1]は[[顆粒細胞]]、[[プルキンエ細胞]]の両方に等しく、[http://mouse.brain-map.org/experiment/show/69626909 DLGAP2]は小脳になく、[http://mouse.brain-map.org/experiment/show/70528605 DLGAP3]は顆粒細胞に強く、[http://mouse.brain-map.org/experiment/show/68499267 DLGAP4]はプルキンエ細胞に強く発現する。海馬では、DLGAP1は[[CA1]]、[[CA3]]の[[錐体細胞]]と[[歯状回]]の顆粒細胞に発現するが、DLGAP4は歯状回の顆粒細胞にはない。DLGAP3は[[線条体]]に強く発現している。DLGAP3ノックアウトマウスの表現型が、線条体局所へのDLGAP3の強制発現により回復する観察から、DLGAP3は線条体において、他のアイソフォームによっては代償されない機能を果たしていると推論される<ref name="ref2"><pubmed>17713528</pubmed></ref>。さらに、DLGAP3のmRNAは、神経細胞の[[細胞体]]でなく[[樹状突起]]に分布する点でも注目されている。 | |||
== 機能、性状 == | == 機能、性状 == | ||
2015年9月2日 (水) 11:16時点における版
畑 裕
東京医科歯科大学 大学院医歯学総合研究科
DOI:10.14931/bsd.658 原稿受付日:2012年2月15日 原稿完成日:2012年2月19日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
| Discs, large (Drosophila) homolog-associated protein 1 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | DLGAP1; DAP-1; DAP-1-ALPHA; DAP-1-BETA; FLJ38442; GKAP; MGC88156; SAPAP1; hGKAP | ||||||||||||
| External IDs | OMIM: 605445 MGI: 1346065 HomoloGene: 31258 GeneCards: DLGAP1 Gene | ||||||||||||
| |||||||||||||
| RNA expression pattern | |||||||||||||
 | |||||||||||||
 | |||||||||||||
| More reference expression data | |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 9229 | 224997 | |||||||||||
| Ensembl | ENSG00000170579 | ENSMUSG00000003279 | |||||||||||
| UniProt | O14490 | Q3TSN1 | |||||||||||
| RefSeq (mRNA) | NM_001003809.2 | NM_027712 | |||||||||||
| RefSeq (protein) | NP_001003809.1 | NP_081988 | |||||||||||
| Location (UCSC) |
Chr 18: 3.5 – 4.15 Mb |
Chr 17: 70.32 – 71.17 Mb | |||||||||||
| PubMed search | [1] | [2] | |||||||||||
| Discs, large (Drosophila) homolog-associated protein 2 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | DLGAP2; DAP2; SAPAP2 | ||||||||||||
| External IDs | OMIM: 605438 MGI: 2443181 HomoloGene: 3484 GeneCards: DLGAP2 Gene | ||||||||||||
| |||||||||||||
| RNA expression pattern | |||||||||||||
 | |||||||||||||
| More reference expression data | |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 9228 | 244310 | |||||||||||
| Ensembl | ENSG00000198010 | ENSMUSG00000047495 | |||||||||||
| UniProt | Q9P1A6 | Q0VF59 | |||||||||||
| RefSeq (mRNA) | NM_004745 | NM_172910 | |||||||||||
| RefSeq (protein) | NP_004736 | NP_766498 | |||||||||||
| Location (UCSC) |
Chr 8: 1.45 – 1.66 Mb |
Chr 8: 14.1 – 14.85 Mb | |||||||||||
| PubMed search | [3] | [4] | |||||||||||
| Discs, large (Drosophila) homolog-associated protein 3 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | DLGAP3; DAP3; SAPAP3 | ||||||||||||
| External IDs | OMIM: 611413 HomoloGene: 18276 GeneCards: DLGAP3 Gene | ||||||||||||
| |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 58512 | 242667 | |||||||||||
| Ensembl | ENSG00000116544 | ENSMUSG00000042388 | |||||||||||
| UniProt | O95886 | Q6PFD5 | |||||||||||
| RefSeq (mRNA) | NM_001080418.1 | NM_198618.4 | |||||||||||
| RefSeq (protein) | NP_001073887.1 | NP_941020.1 | |||||||||||
| Location (UCSC) |
Chr 1: 35.33 – 35.4 Mb |
Chr 4: 126.85 – 126.91 Mb | |||||||||||
| PubMed search | [5] | [6] | |||||||||||
| Discs, large (Drosophila) homolog-associated protein 4 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | DLGAP4; DAP4; DLP4; FLJ20588; KIAA0964; MGC131862; RP5-977B1.6; SAPAP4 | ||||||||||||
| External IDs | MGI: 2138865 HomoloGene: 8935 GeneCards: DLGAP4 Gene | ||||||||||||
| |||||||||||||
| RNA expression pattern | |||||||||||||
 | |||||||||||||
 | |||||||||||||
 | |||||||||||||
| More reference expression data | |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 22839 | 228836 | |||||||||||
| Ensembl | ENSG00000080845 | ENSMUSG00000061689 | |||||||||||
| UniProt | Q9Y2H0 | n/a | |||||||||||
| RefSeq (mRNA) | NM_001042486.2 | NM_001042487 | |||||||||||
| RefSeq (protein) | NP_001035951.1 | NP_001035952 | |||||||||||
| Location (UCSC) |
Chr 20: 34.89 – 35.16 Mb |
Chr 2: 156.44 – 156.59 Mb | |||||||||||
| PubMed search | [7] | [8] | |||||||||||
英略称:DLGAP
同義語:Discs, large homolog-associated protein, GKAP, SAPAP
興奮性シナプスの裏打ちタンパク質の一つ。Postsynaptic density (PSD)-95のグアニル酸キナーゼ領域に結合するタンパク質として同定された。14アミノ酸からなる5回の繰り返し配列を持ち、その部位でPSD-95、MAGI2/S-SCAMのguanylate kinase領域に結合する。N末端側で、ニューロフィラメント (neurofilament)などの中間径フィラメント、C末端側のプロリンリッチ配列を介して、NArgBP2のSH3領域、さらにそれよりもC末端側でダイニン軽鎖 (dynein light chain) LC1、LC8、C末端にPDZ結合モチーフでShankのPDZ領域に結合する。その他、神経型一酸化窒素合成酵素(nNOS)とも結合する。DLGAP3ノックアウトマウスは、ヒトにおける強迫性障害 (obsessive-compulsive disorder(OCD))を連想させる行動異常を示す。
名称
GKAPあるいはSAPAPともよばれる。GKAPという名称はguanylate kinase-associated proteinに由来する[1]。興奮性シナプスの裏打ちタンパク質PSD-95のグアニル酸キナーゼ領域に結合するタンパク質としてラットとヒトのタンパク質が最初に報告された。なおcGMP-dependent protein kinase-anchoring proteinとglucokinase-associated phosphataseもGKAPと表記されるので、注意が必要である。
PSD-95は別名でsynapse-associated protein (SAP)90ともよばれるが、GKAPの報告とほぼ同時期に、SAP90に結合するタンパク質として、4種類のラットSAP-associated protein (SAPAP)アイソフォームがSAPAP1、-2、-3、-4として報告された[2]。ラットGKAPは666アミノ酸からなり、992アミノ酸からなるラットSAPAP1のN末端を欠損するalternative splicing variantにあたる。
PSD-95と同じMAGUKファミリータンパク質のひとつDlg1(SAP97とも呼ばれ、神経細胞以外にも発現し、細胞極性に関係する)に結合するタンパク質として、やはりほぼ同時期に報告されたDLG-associated protein(DAP)-1はヒトのSAPAP1に相当する[3]。SAPAP1はGKAP/SAPAP1/DAP-1と、それ以外のアイソフォームは、SAPAP2、-3、-4と表記されることが多い。
Hugo Gene Nomenclature Committeeによるオフィシャルネームでは、ヒトの遺伝子は、DLGAP1、-2、-3、-4と表記され、SAPAP1、-2、-3、-4に対応している。それぞれの遺伝子は、18番、8番、1番、20番染色体上にある。DLGAP1には8個のvariantsが登録されている。以下、本稿においては、DLGAPとして表記する。
構造と相互作用分子
4つのアイソフォームはアミノ酸レベルで40-50%の相同性があり、いずれも、中央部に14アミノ酸からなる5回の繰り返し配列を持ち、その部位でPSD-95、MAGI2/S-SCAMのguanylate kinase領域に結合する。繰り返し配列よりN末端側には特徴的配列がないが、ニューロフィラメントなどの中間径フィラメントに結合する[4]。繰り返し配列のC末端側のプロリンリッチ配列を介して、チロシンキナーゼAbl/Arg結合タンパク質であるNArgBP2のSH3領域に結合する[5]。さらにそれよりもC末端側でダイニン軽鎖LC1、LC8に結合するが、その部分には特徴的配列を欠く[6][7]。C末端にPDZ結合モチーフをもち、ShankのPDZ領域に結合する[8][9]。その他、神経型一酸化窒素合成酵素(nNOS)とも結合する[7]。DLGAP3は、FAK、PYK2と相互作用すると報告されている[10]。カルシウム/カルモジュリン依存性タンパク質キナーゼII(CaMKII)、p38 MAPKによりリン酸化されることも報告されている[11][12]。相互作用分子の研究には、DLGAP1のラット、マウスホモログが用いられていることが多いが、アミノ酸配列の相同性に照らすと、報告されている相互作用の多くは、種とアイソフォームを超えて保存されていると推測される。
発現
神経細胞以外にもmRNAはあり、タンパク質も発現していると考えられるが、神経細胞における発現が極めて高い。脳内での各種アイソフォームの発現については、mRNAレベルでは4つのアイソフォームについて、タンパク質レベルではDLGAP2以外の3つのアイソフォームについて、ラット、マウス脳を用いた解析結果が報告されている[13][14]。ちなみに、「異なるアイソフォームの分布は、オーバーラップしているが、それぞれに特異的である」と表現される。例えば、小脳を例にとると、DLGAP1は顆粒細胞、プルキンエ細胞の両方に等しく、DLGAP2は小脳になく、DLGAP3は顆粒細胞に強く、DLGAP4はプルキンエ細胞に強く発現する。海馬では、DLGAP1はCA1、CA3の錐体細胞と歯状回の顆粒細胞に発現するが、DLGAP4は歯状回の顆粒細胞にはない。DLGAP3は線条体に強く発現している。DLGAP3ノックアウトマウスの表現型が、線条体局所へのDLGAP3の強制発現により回復する観察から、DLGAP3は線条体において、他のアイソフォームによっては代償されない機能を果たしていると推論される[15]。さらに、DLGAP3のmRNAは、神経細胞の細胞体でなく樹状突起に分布する点でも注目されている。
機能、性状
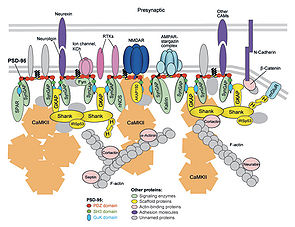
Reprinted, with permission, from the Annual Review of Biochemistry, Volume 76 © 2007 by Annual Reviews www.annualreviews.org
DLGAPはNMDA型グルタミン酸受容体の裏打ちタンパク質PSD-95に結合する分子として同定され、Shankにも結合するため、興奮性シナプスの足場タンパク質と見なされている(図)。上述のように、多くの相互作用分子が同定されているものの、個々の相互作用の生理的意義は必ずしも明快に示されているとはいえない。ダイニン軽鎖との結合からは、PSD-95を含む複合体の輸送を担う機能が措定されている。DLGAP3ノックアウトマウスでは、DLGAP3が本来、強く発現する線条体の神経細胞において、代謝活性型グルタミン酸受容体5型(mGluR5)が細胞膜表面に留まり、そのシグナル活性が上昇し、その結果としてAMPA型グルタミン酸受容体のシグナルの抑制、エンドカンナビノイド (endocannabinoid)による異常なシナプス抑制が観察される[16][17]。この知見をもとに、DLGAP3にはmGluR5のエンドサイトーシスを制御する機能があると推論されている。グリア細胞では、Dlg1をダイニンにつなぐ作用を通じて、中心体の細胞内の位置決めに関与すると報告されている[18]。なお、いずれのSAPAPも界面活性剤に不溶性であるが、その不溶性はN末端に依存し、N末端を欠くGKAP (DLGAP1 variant2)は比較的可溶化されやすい。神経細胞に発現させた場合のSAPAP1 (DLGAP1 variant1)とGKAPの挙動にも、差異があると予測される。したがって、それぞれの研究で、GKAPが論じられているのか、SAPAP1が論じられているのかについて、注意を払う必要がある。
ヒト疾病との関係
DLGAP3ノックアウトマウスは、ヒトにおける強迫性障害を連想させる行動異常を示す[15]。この観察から、DLGAP3遺伝子は、ヒトのOCD、及び、OCDに関連するgrooming disorderやトゥレット症候群 (Tourette syndrome)の原因遺伝子であるという仮説が立てられ解析がなされている。しかし、現時点では、機能的な候補遺伝子の一つという評価に止まり、決定的な連関は示されていない[19][20]。このほか、統合失調症患者において、DLGAP1の発現が高まっているという報告もある[21]。
関連項目
参考文献
- ↑
Kim, E., Naisbitt, S., Hsueh, Y.P., Rao, A., Rothschild, A., Craig, A.M., & Sheng, M. (1997).
GKAP, a novel synaptic protein that interacts with the guanylate kinase-like domain of the PSD-95/SAP90 family of channel clustering molecules. The Journal of cell biology, 136(3), 669-78. [PubMed:9024696] [PMC] [WorldCat] [DOI] - ↑
Takeuchi, M., Hata, Y., Hirao, K., Toyoda, A., Irie, M., & Takai, Y. (1997).
SAPAPs. A family of PSD-95/SAP90-associated proteins localized at postsynaptic density. The Journal of biological chemistry, 272(18), 11943-51. [PubMed:9115257] [WorldCat] [DOI] - ↑
Satoh, K., Yanai, H., Senda, T., Kohu, K., Nakamura, T., Okumura, N., ..., & Akiyama, T. (1997).
DAP-1, a novel protein that interacts with the guanylate kinase-like domains of hDLG and PSD-95. Genes to cells : devoted to molecular & cellular mechanisms, 2(6), 415-24. [PubMed:9286858] [WorldCat] - ↑
Hirao, K., Hata, Y., Deguchi, M., Yao, I., Ogura, M., Rokukawa, C., ..., & Takai, Y. (2000).
Association of synapse-associated protein 90/ postsynaptic density-95-associated protein (SAPAP) with neurofilaments. Genes to cells : devoted to molecular & cellular mechanisms, 5(3), 203-10. [PubMed:10759891] [WorldCat] - ↑
Kawabe, H., Hata, Y., Takeuchi, M., Ide, N., Mizoguchi, A., & Takai, Y. (1999).
nArgBP2, a novel neural member of ponsin/ArgBP2/vinexin family that interacts with synapse-associated protein 90/postsynaptic density-95-associated protein (SAPAP). The Journal of biological chemistry, 274(43), 30914-8. [PubMed:10521485] [WorldCat] [DOI] - ↑
Naisbitt, S., Valtschanoff, J., Allison, D.W., Sala, C., Kim, E., Craig, A.M., ..., & Sheng, M. (2000).
Interaction of the postsynaptic density-95/guanylate kinase domain-associated protein complex with a light chain of myosin-V and dynein. The Journal of neuroscience : the official journal of the Society for Neuroscience, 20(12), 4524-34. [PubMed:10844022] [PMC] [WorldCat] - ↑ 7.0 7.1
Haraguchi, K., Satoh, K., Yanai, H., Hamada, F., Kawabuchi, M., & Akiyama, T. (2000).
The hDLG-associated protein DAP interacts with dynein light chain and neuronal nitric oxide synthase. Genes to cells : devoted to molecular & cellular mechanisms, 5(11), 905-911. [PubMed:11122378] [WorldCat] - ↑
Naisbitt, S., Kim, E., Tu, J.C., Xiao, B., Sala, C., Valtschanoff, J., ..., & Sheng, M. (1999).
Shank, a novel family of postsynaptic density proteins that binds to the NMDA receptor/PSD-95/GKAP complex and cortactin. Neuron, 23(3), 569-82. [PubMed:10433268] [WorldCat] [DOI] - ↑
Boeckers, T.M., Winter, C., Smalla, K.H., Kreutz, M.R., Bockmann, J., Seidenbecher, C., ..., & Gundelfinger, E.D. (1999).
Proline-rich synapse-associated proteins ProSAP1 and ProSAP2 interact with synaptic proteins of the SAPAP/GKAP family. Biochemical and biophysical research communications, 264(1), 247-52. [PubMed:10527873] [WorldCat] [DOI] - ↑
Bongiorno-Borbone, L., Kadaré, G., Benfenati, F., & Girault, J.A. (2005).
FAK and PYK2 interact with SAP90/PSD-95-Associated Protein-3. Biochemical and biophysical research communications, 337(2), 641-6. [PubMed:16202977] [WorldCat] [DOI] - ↑
Dosemeci, A., & Jaffe, H. (2010).
Regulation of phosphorylation at the postsynaptic density during different activity states of Ca2+/calmodulin-dependent protein kinase II. Biochemical and biophysical research communications, 391(1), 78-84. [PubMed:19896464] [PMC] [WorldCat] [DOI] - ↑
Sabio, G., Arthur, J.S., Kuma, Y., Peggie, M., Carr, J., Murray-Tait, V., ..., & Cuenda, A. (2005).
p38gamma regulates the localisation of SAP97 in the cytoskeleton by modulating its interaction with GKAP. The EMBO journal, 24(6), 1134-45. [PubMed:15729360] [PMC] [WorldCat] [DOI] - ↑
Kindler, S., Rehbein, M., Classen, B., Richter, D., & Böckers, T.M. (2004).
Distinct spatiotemporal expression of SAPAP transcripts in the developing rat brain: a novel dendritically localized mRNA. Brain research. Molecular brain research, 126(1), 14-21. [PubMed:15207911] [WorldCat] [DOI] - ↑
Welch, J.M., Wang, D., & Feng, G. (2004).
Differential mRNA expression and protein localization of the SAP90/PSD-95-associated proteins (SAPAPs) in the nervous system of the mouse. The Journal of comparative neurology, 472(1), 24-39. [PubMed:15024750] [WorldCat] [DOI] - ↑ 15.0 15.1
Welch, J.M., Lu, J., Rodriguiz, R.M., Trotta, N.C., Peca, J., Ding, J.D., ..., & Feng, G. (2007).
Cortico-striatal synaptic defects and OCD-like behaviours in Sapap3-mutant mice. Nature, 448(7156), 894-900. [PubMed:17713528] [PMC] [WorldCat] [DOI] - ↑
Chen, M., Wan, Y., Ade, K., Ting, J., Feng, G., & Calakos, N. (2011).
Sapap3 deletion anomalously activates short-term endocannabinoid-mediated synaptic plasticity. The Journal of neuroscience : the official journal of the Society for Neuroscience, 31(26), 9563-73. [PubMed:21715621] [PMC] [WorldCat] [DOI] - ↑
Wan, Y., Feng, G., & Calakos, N. (2011).
Sapap3 deletion causes mGluR5-dependent silencing of AMPAR synapses. The Journal of neuroscience : the official journal of the Society for Neuroscience, 31(46), 16685-91. [PubMed:22090495] [PMC] [WorldCat] [DOI] - ↑
Manneville, J.B., Jehanno, M., & Etienne-Manneville, S. (2010).
Dlg1 binds GKAP to control dynein association with microtubules, centrosome positioning, and cell polarity. The Journal of cell biology, 191(3), 585-98. [PubMed:21041448] [PMC] [WorldCat] [DOI] - ↑
Bienvenu, O.J., Wang, Y., Shugart, Y.Y., Welch, J.M., Grados, M.A., Fyer, A.J., ..., & Nestadt, G. (2009).
Sapap3 and pathological grooming in humans: Results from the OCD collaborative genetics study. American journal of medical genetics. Part B, Neuropsychiatric genetics : the official publication of the International Society of Psychiatric Genetics, 150B(5), 710-20. [PubMed:19051237] [WorldCat] [DOI] - ↑
Crane, J., Fagerness, J., Osiecki, L., Gunnell, B., Stewart, S.E., Pauls, D.L., ..., & Tourette Syndrome International Consortium for Genetics (TSAICG) (2011).
Family-based genetic association study of DLGAP3 in Tourette Syndrome. American journal of medical genetics. Part B, Neuropsychiatric genetics : the official publication of the International Society of Psychiatric Genetics, 156B(1), 108-14. [PubMed:21184590] [PMC] [WorldCat] [DOI] - ↑
Aoyama, S., Shirakawa, O., Ono, H., Hashimoto, T., Kajimoto, Y., & Maeda, K. (2003).
Mutation and association analysis of the DAP-1 gene with schizophrenia. Psychiatry and clinical neurosciences, 57(5), 545-7. [PubMed:12950712] [WorldCat] [DOI]