「トランスポゾン」の版間の差分
Hidenorinishihara (トーク | 投稿記録) 細編集の要約なし |
|||
| (2人の利用者による、間の6版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | |||
<font size="+1">[http://researchmap.jp/nishihara 西原 秀典]</font><br> | |||
''東京工業大学 生命理工学研究科''<br> | |||
<font size="+1">[http://researchmap.jp/read0008506 岡田 典弘]</font><br> | |||
''公益財団法人国際科学振興財団''<br> | |||
DOI:<selfdoi /> 原稿受付日:2013年8月12日 原稿完成日:2013年9月2日<br> | |||
担当編集委員:[http://researchmap.jp/noriko1128 大隅 典子](東北大学 大学院医学系研究科 附属創生応用医学研究センター [[脳神経]]科学コアセンター 発生発達神経科学分野)<br> | |||
</div> | |||
英:Transposable elements、英略語:TEs | 英:Transposable elements、英略語:TEs | ||
同義語:転移因子、Jumping genes | |||
{{box|text= トランスポゾンとは、広義には細胞内で[[wikipedia:jaゲノム|ゲノム]]上の位置を移動する(転移する)[[wikipedia:ja:DNA|DNA]]配列を指し、狭義にはその一種であるDNA(型)トランスポゾンを指す。前者は一般的に転移因子と呼ばれ、時にJumping genesとも呼ばれる。転移因子の中でも特に[[wikipedia:ja:レトロポゾン|レトロポゾン]]はゲノム中で膨大なコピー数が存在し、例えば[[wikipedia:ja:ヒト|ヒト]]ゲノムの46%を占めるなど、ゲノム構造の多様性およびゲノムサイズに大きな影響を与えている。また、転移という特徴を利用し、遺伝学的ツールとして用いることができる。例えばP elementとよばれる転移因子を生体内で転移させることで変異体を多数作成し、特定の表現型を示す個体を選別してその原因となる転移因子の挿入サイトを特定することができる。ゲノムへの遺伝子導入への応用も広くおこなわれている。}} | |||
==発見の歴史== | ==発見の歴史== | ||
転移因子の存在は、1950年に[[wikipedia:ja:バーバラ・マクリントック|バーバラ・マクリントック]](Barbara McClintock, 1902 - 1992年)によって提唱された<ref><pubmed> 15430309 </pubmed></ref>。マクリントックは斑入りの[[wikipedia:ja:トウモロコシ|トウモロコシ]]の[[wikipedia:ja:染色体|染色体]]を調べることで、斑の形成に関わる遺伝子座が染色体の異なる位置に移動することを発見し、この現象を[[wikipedia:ja:トランスポジション|トランスポジション]]と名付けた。しかし、遺伝子が染色体内を移動するといった概念は当時の常識をはるかに越えており、マクリントックの学説はほとんど受け入れられることは無かった。その後1960年代後半から70年代前半にかけて、[[wikipedia:ja:バクテリア|バクテリア]]や[[wikipedia:ja:酵母|酵母]]で「動く遺伝子」が発見されるようになり、それらはトランスポゾンと名付けられた。またマクリントックが発見したトウモロコシの斑の形成に関わる遺伝子座もDNAトランスポゾンの一種であることが明らかにされた。こうしてマクリントックが提唱した学説は認められるようになり、1983年には単独女性として初めて[[wikipedia:ja:ノーベル医学生理学賞|ノーベル医学生理学賞]]を受賞した。 | |||
==分類== | |||
[[ファイル:TE.gif|350px|thumb|right|'''図.転移因子の構造''']] | |||
転移因子は、その転移機序に応じて2つのクラスに分類される<ref><pubmed> 18421312 </pubmed></ref>。1つはカット&ペースト型の転移をするDNA(型)トランスポゾンであり、その転移機構をトランスポジションと呼ぶ。もう1つは[[wikipedia:ja:レトロトランスポジション|レトロトランスポジション]](またはレトロポジション)と呼ばれる転移機序を持つレトロトランスポゾンであり、いわゆるコピー&ペースト型の転移をおこなう。 | |||
またそれぞれの転移因子は、その転移様式によって自律型と非自律型に分類できる。自律型の転移因子は、その配列自身がコードする[[wikipedia:ja:転移酵素|転移酵素]](レトロトランスポゾンの場合は[[wikipedia:ja:逆転写酵素|逆転写酵素]])を利用して転移する。一方で非自律型の転移因子は転移のための酵素コードしておらず、自律型の転移因子から発現した酵素を利用して転移する。そのため、自律型のみならず非自律型の転移因子も、転移酵素や逆転写酵素の認識配列を持つのが一般的である。 | |||
DNAトランスポゾンやレトロトランスポゾンは、その転移機序と進化的起源によって細かく分類されている。特に最近は膨大なゲノム情報の蓄積によって、新規の転移因子ファミリーが次々と発見されており、中にはこれまでの分類に当てはまらない転移因子が発見されることもある。 | DNAトランスポゾンやレトロトランスポゾンは、その転移機序と進化的起源によって細かく分類されている。特に最近は膨大なゲノム情報の蓄積によって、新規の転移因子ファミリーが次々と発見されており、中にはこれまでの分類に当てはまらない転移因子が発見されることもある。 | ||
===DNA(型)トランスポゾン=== | |||
クラスIIトランスポゾンとも呼ばれ、[[wikipedia:ja:トランスポザーゼ|トランスポザーゼ]](transposase)と呼ばれる転移酵素をコードしている。このトランスポザーゼがトランスポゾンの両末端にある逆向き配列(inverted repeat)を認識してゲノムから切り出し、切り出されたトランスポゾンDNAをゲノム上の別の位置に再び挿入させる。DNAトランスポゾンの種類によって、挿入のターゲットとなるゲノム配列の選好性は異なる。 | |||
DNAトランスポゾンは自律性と非自律性の2種類に分けられる。自律性トランスポゾンは上記のように内部にコードするトランスポザーゼを用いて転移する。一方で非自律性の因子は、内部にタンパク質をコードしておらず、自律性因子と類似した逆向き配列を持つ数百塩基対程度の短い因子である。一般にこうした短い非自律性トランスポゾンは[[wikipedia:miniature inverted-repeat transposable element|miniature inverted-repeat transposable element]](MITE)と呼ばれている。非自律性因子は自律性因子がコードするトランスポザーゼを利用して転移する。 | |||
===レトロトランスポゾン=== | ===レトロトランスポゾン=== | ||
クラスIトランスポゾンとも呼ばれる。転移の際、まずレトロトランスポゾン配列が内部[[プロモーター]]によって転写され、自身がコードする逆転写酵素が翻訳される。逆転写酵素はレトロトランスポゾンRNAを鋳型として[[wikipedia:ja:cDNA|cDNA]]を合成し、それをゲノム上の別の位置に挿入させる。したがってすべてのレトロポジションはレトロポゾン配列のコピー数の増加を伴い、ゲノムサイズに大きな影響を与えることが知られている。レトロトランスポゾンは[[wikipedia:long terminal repeat|long terminal repeat]](LTR)型レトロトランスポゾン、およびLINE[[wikipedia:long interspersed element|long interspersed element]](LINE)(非LTR型レトロトランスポゾンとも呼ばれる)に大別され、それぞれ次のように異なる転移機序を持つ。 | |||
LTR型レトロトランスポゾンは両末端に数百~数千塩基対の反復配列(LTR)を持ち、内部に逆転写酵素と[[wikipedia:ja:インテグラーゼ|インテグラーゼ]]をコードしている。レトロウイルスはLTRに加えて内部にenv遺伝子を持つが、分類上LTR型レトロトランスポゾンに含まれることが多い。LTR型レトロトランスポゾンにもLTRのみを持つ非自律性因子が知られており、それらは長さに応じて[[wikipedia:LARD|LARD]]や[[wikipedia:TRIM|TRIM]]と呼ばれることもある。 | |||
LINEは内部に逆転写酵素とエンドヌクレアーゼをコードする。LINEタンパク質はLINE RNAの3’末端配列を認識して結合し、逆転写およびゲノムへの挿入をおこなう。LINEには様々な種類が知られており、生物群ごとにそれぞれ異なる種類のLINEがゲノムの大きな割合を占める。例えば哺乳類では[[L1]]と呼ばれるLINEが主要であり、例えばヒトゲノムの約17%はL1が占めている。一方、[[wikipedia:ja:ニワトリゲノム|ニワトリゲノム]]ではCR1、[[ゼブラフィッシュ]]ではL2がレトロトランスポゾンの中で最も大きな割合を占める。 | |||
さらにLINEに関連する非自律性因子として、[[wikipedia:short interspersed elements|short interspersed elements]](SINE)が知られている。多くのSINEは[[tRNA]]に起源を持ち、[[wikipedia:RNA polymerase III|RNA polymerase III]]によって転写されたSINE RNAが、LINEタンパク質を利用して転移する。そのためSINEの3’末端配列がLINEの3’末端配列と類似している例が数多く報告されている。SINEは進化的に異なる系統で独立に生成する場合が多く見られる。例えば[[wikipedia:ja:霊長類|霊長類]]の[[wikipedia:Alu|Alu]]、[[wikipedia:ja:齧歯類|齧歯類]]の[[wikipedia:B1|B1]]や[[wikipedia:B2|B2]]など、[[wikipedia:ja:哺乳類|哺乳類]]では目あるいは科レベルで独自のSINEを持つ。Aluはヒトゲノムの10%を占めており、そのコピー数は100万を超える。 | |||
==ゲノムにおける影響== | ==ゲノムにおける影響== | ||
一般的に転移因子は、ゲノム中を移動するだけの利己的DNAあるいは寄生因子にすぎないと見なされる場合が多い。それでも、膨大なコピー数が存在し転移を繰り返していることから、その影響は無視できないものとなっている。例えば、転移因子が遺伝子内部に挿入されることで[[wikipedia:ja:血友病|血友病]]や[[wikipedia:ja:癌|癌]]などの疾患をもたらす例が知られている<ref><pubmed> 18256243 </pubmed></ref>。また複数の転移因子の間で非相同組換えが起こり、ゲノム構造を改変することもある<ref><pubmed> 14638329 </pubmed></ref><ref><pubmed> 15016989 </pubmed></ref>。 | |||
また進化的視点から考えると、ゲノムサイズの増大、およびゲノム構造の多様化に大きな影響を与えており、ゲノムの重要な構成要素として認識されている。ゲノム中で転移因子の占める割合は、ヒトでは46%、トウモロコシでは80%以上であり、ゲノムサイズを決定する主要因となっている。一方で転移因子の[[wikipedia:ja:水平伝播|水平伝播]]が起こっていることも知られているが、その詳細なメカニズムはほとんど明らかになっていない<ref><pubmed> 20591532 </pubmed></ref>。 | |||
転移因子は上述のように有害な影響を及ぼす可能性があることから、一般の細胞内では転移が抑制されていることが多い。例外として、[[wikipedia:ja:生殖細胞|生殖細胞]]および[[脳神経細胞]]では転移が観察されており、特にニューロンの[[wikipedia:ja:モザイク|モザイク]]性を生み出している可能性が指摘されている<ref><pubmed> 15959507 </pubmed></ref>。しかしなぜこれらの細胞内のみで転移可能なのかは明らかになっていない。[[wikipedia:ja:体細胞|体細胞]]における抑制機構としては、転移因子配列の[[メチル化]]や[[wikipedia:ja:ヘテロクロマチン化|ヘテロクロマチン化]]などの[[エピジェネティック]]な制御を受けていることが知られている。また、[[wikipedia:small RNA|small RNA]]による抑制も受けていることが近年明らかになってきている<ref><pubmed> 19165215 </pubmed></ref>。 | |||
転移因子の挿入は進化的に中立の変異であり、一般にその配列は進化の過程で塩基置換が蓄積し、やがて転移因子であることの検出が困難になると考えられる。しかし例外的に、その過程で転移因子由来の配列が生物の生存に有利な何らかの機能を獲得する場合が知られている(exaptationまたはco-optionとも呼ばれる)。それらの配列は進化的に保存されることが多く<ref><pubmed> 16717141 </pubmed></ref><ref><pubmed> 16625209 </pubmed></ref>、特に[[wikipedia:ja:有胎盤類|有胎盤類]]では、保存領域の16%が転移因子に由来することが分かっている<ref><pubmed> 17495919 </pubmed></ref>(編集 林 コメント:この文献はmarsupialとありますので、有袋類ではないでしょうか)。転移因子が獲得した機能としては、遺伝子の[[wikipedia:ja:エキソン|エキソン]]化や[[wikipedia:ja:選択的スプライシング|選択的スプライシング]]の生成といったタンパク質[[wikipedia:ja:コード|コード]]配列の改変に加え、[[wikipedia:ja:エンハンサー|エンハンサー]]や[[wikipedia:ja:インシュレーター|インシュレーター]]などの調節配列となることが知られている。特に哺乳類の[[脳]]において複数のレトロポゾンがエンハンサー機能を有することが報告されており、哺乳類の脳の形成に関与したexaptationの好例として知られている<ref><pubmed> 18334644 </pubmed></ref>。 | |||
==生物学的利用== | ==生物学的利用== | ||
また、転移という特徴を利用し、転移因子を遺伝学的ツールとして用いることができる。具体的な利用方法の1つとして、転移因子を用いた順遺伝学的スクリーニングがある。これは転移因子を生体内で転移させることで変異体を多数作成し、特定の表現型を示す個体を選別してその原因となる転移因子の挿入サイトを特定する方法である。代表的なものとして、[[wikipedia:P element|P element]]、[[wikipedia:Sleeping beauty|Sleeping beauty]]、[[wikipedia:piggyBac|piggyBac]]、[[wikipedia:Tol2|Tol2]]などが用いられている<ref><pubmed> 19478801 </pubmed></ref>。この手法は、[[ショウジョウバエ]]をはじめ、ゼブラフィッシュや[[wikipedia:ja:マウス|マウス]]などの脊椎動物でも幅広く用いられている。 | |||
また転移因子を利用したゲノムへの遺伝子導入、すなわち遺伝子組み換え生物の作成も広くおこなわれている。目的遺伝子の両端にDNAトランスポゾンの末端配列を付加したコンストラクトを胚へ導入し、トランスポゼースを利用して転移させることで、効率良くゲノム中に導入することができる。この方法では、piggyBacやTol2がよく利用されている<ref><pubmed> 18047699 </pubmed></ref>。 | また転移因子を利用したゲノムへの遺伝子導入、すなわち遺伝子組み換え生物の作成も広くおこなわれている。目的遺伝子の両端にDNAトランスポゾンの末端配列を付加したコンストラクトを胚へ導入し、トランスポゼースを利用して転移させることで、効率良くゲノム中に導入することができる。この方法では、piggyBacやTol2がよく利用されている<ref><pubmed> 18047699 </pubmed></ref>。 | ||
== 参考文献 == | |||
<references/> | <references/> | ||
2016年2月2日 (火) 20:01時点における最新版
西原 秀典
東京工業大学 生命理工学研究科
岡田 典弘
公益財団法人国際科学振興財団
DOI:10.14931/bsd.4127 原稿受付日:2013年8月12日 原稿完成日:2013年9月2日
担当編集委員:大隅 典子(東北大学 大学院医学系研究科 附属創生応用医学研究センター 脳神経科学コアセンター 発生発達神経科学分野)
英:Transposable elements、英略語:TEs
同義語:転移因子、Jumping genes
トランスポゾンとは、広義には細胞内でゲノム上の位置を移動する(転移する)DNA配列を指し、狭義にはその一種であるDNA(型)トランスポゾンを指す。前者は一般的に転移因子と呼ばれ、時にJumping genesとも呼ばれる。転移因子の中でも特にレトロポゾンはゲノム中で膨大なコピー数が存在し、例えばヒトゲノムの46%を占めるなど、ゲノム構造の多様性およびゲノムサイズに大きな影響を与えている。また、転移という特徴を利用し、遺伝学的ツールとして用いることができる。例えばP elementとよばれる転移因子を生体内で転移させることで変異体を多数作成し、特定の表現型を示す個体を選別してその原因となる転移因子の挿入サイトを特定することができる。ゲノムへの遺伝子導入への応用も広くおこなわれている。
発見の歴史
転移因子の存在は、1950年にバーバラ・マクリントック(Barbara McClintock, 1902 - 1992年)によって提唱された[1]。マクリントックは斑入りのトウモロコシの染色体を調べることで、斑の形成に関わる遺伝子座が染色体の異なる位置に移動することを発見し、この現象をトランスポジションと名付けた。しかし、遺伝子が染色体内を移動するといった概念は当時の常識をはるかに越えており、マクリントックの学説はほとんど受け入れられることは無かった。その後1960年代後半から70年代前半にかけて、バクテリアや酵母で「動く遺伝子」が発見されるようになり、それらはトランスポゾンと名付けられた。またマクリントックが発見したトウモロコシの斑の形成に関わる遺伝子座もDNAトランスポゾンの一種であることが明らかにされた。こうしてマクリントックが提唱した学説は認められるようになり、1983年には単独女性として初めてノーベル医学生理学賞を受賞した。
分類
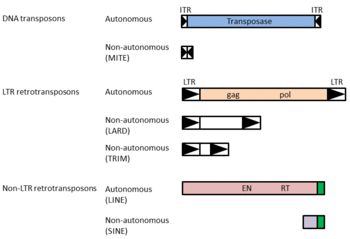
転移因子は、その転移機序に応じて2つのクラスに分類される[2]。1つはカット&ペースト型の転移をするDNA(型)トランスポゾンであり、その転移機構をトランスポジションと呼ぶ。もう1つはレトロトランスポジション(またはレトロポジション)と呼ばれる転移機序を持つレトロトランスポゾンであり、いわゆるコピー&ペースト型の転移をおこなう。
またそれぞれの転移因子は、その転移様式によって自律型と非自律型に分類できる。自律型の転移因子は、その配列自身がコードする転移酵素(レトロトランスポゾンの場合は逆転写酵素)を利用して転移する。一方で非自律型の転移因子は転移のための酵素コードしておらず、自律型の転移因子から発現した酵素を利用して転移する。そのため、自律型のみならず非自律型の転移因子も、転移酵素や逆転写酵素の認識配列を持つのが一般的である。
DNAトランスポゾンやレトロトランスポゾンは、その転移機序と進化的起源によって細かく分類されている。特に最近は膨大なゲノム情報の蓄積によって、新規の転移因子ファミリーが次々と発見されており、中にはこれまでの分類に当てはまらない転移因子が発見されることもある。
DNA(型)トランスポゾン
クラスIIトランスポゾンとも呼ばれ、トランスポザーゼ(transposase)と呼ばれる転移酵素をコードしている。このトランスポザーゼがトランスポゾンの両末端にある逆向き配列(inverted repeat)を認識してゲノムから切り出し、切り出されたトランスポゾンDNAをゲノム上の別の位置に再び挿入させる。DNAトランスポゾンの種類によって、挿入のターゲットとなるゲノム配列の選好性は異なる。
DNAトランスポゾンは自律性と非自律性の2種類に分けられる。自律性トランスポゾンは上記のように内部にコードするトランスポザーゼを用いて転移する。一方で非自律性の因子は、内部にタンパク質をコードしておらず、自律性因子と類似した逆向き配列を持つ数百塩基対程度の短い因子である。一般にこうした短い非自律性トランスポゾンはminiature inverted-repeat transposable element(MITE)と呼ばれている。非自律性因子は自律性因子がコードするトランスポザーゼを利用して転移する。
レトロトランスポゾン
クラスIトランスポゾンとも呼ばれる。転移の際、まずレトロトランスポゾン配列が内部プロモーターによって転写され、自身がコードする逆転写酵素が翻訳される。逆転写酵素はレトロトランスポゾンRNAを鋳型としてcDNAを合成し、それをゲノム上の別の位置に挿入させる。したがってすべてのレトロポジションはレトロポゾン配列のコピー数の増加を伴い、ゲノムサイズに大きな影響を与えることが知られている。レトロトランスポゾンはlong terminal repeat(LTR)型レトロトランスポゾン、およびLINElong interspersed element(LINE)(非LTR型レトロトランスポゾンとも呼ばれる)に大別され、それぞれ次のように異なる転移機序を持つ。
LTR型レトロトランスポゾンは両末端に数百~数千塩基対の反復配列(LTR)を持ち、内部に逆転写酵素とインテグラーゼをコードしている。レトロウイルスはLTRに加えて内部にenv遺伝子を持つが、分類上LTR型レトロトランスポゾンに含まれることが多い。LTR型レトロトランスポゾンにもLTRのみを持つ非自律性因子が知られており、それらは長さに応じてLARDやTRIMと呼ばれることもある。
LINEは内部に逆転写酵素とエンドヌクレアーゼをコードする。LINEタンパク質はLINE RNAの3’末端配列を認識して結合し、逆転写およびゲノムへの挿入をおこなう。LINEには様々な種類が知られており、生物群ごとにそれぞれ異なる種類のLINEがゲノムの大きな割合を占める。例えば哺乳類ではL1と呼ばれるLINEが主要であり、例えばヒトゲノムの約17%はL1が占めている。一方、ニワトリゲノムではCR1、ゼブラフィッシュではL2がレトロトランスポゾンの中で最も大きな割合を占める。
さらにLINEに関連する非自律性因子として、short interspersed elements(SINE)が知られている。多くのSINEはtRNAに起源を持ち、RNA polymerase IIIによって転写されたSINE RNAが、LINEタンパク質を利用して転移する。そのためSINEの3’末端配列がLINEの3’末端配列と類似している例が数多く報告されている。SINEは進化的に異なる系統で独立に生成する場合が多く見られる。例えば霊長類のAlu、齧歯類のB1やB2など、哺乳類では目あるいは科レベルで独自のSINEを持つ。Aluはヒトゲノムの10%を占めており、そのコピー数は100万を超える。
ゲノムにおける影響
一般的に転移因子は、ゲノム中を移動するだけの利己的DNAあるいは寄生因子にすぎないと見なされる場合が多い。それでも、膨大なコピー数が存在し転移を繰り返していることから、その影響は無視できないものとなっている。例えば、転移因子が遺伝子内部に挿入されることで血友病や癌などの疾患をもたらす例が知られている[3]。また複数の転移因子の間で非相同組換えが起こり、ゲノム構造を改変することもある[4][5]。
また進化的視点から考えると、ゲノムサイズの増大、およびゲノム構造の多様化に大きな影響を与えており、ゲノムの重要な構成要素として認識されている。ゲノム中で転移因子の占める割合は、ヒトでは46%、トウモロコシでは80%以上であり、ゲノムサイズを決定する主要因となっている。一方で転移因子の水平伝播が起こっていることも知られているが、その詳細なメカニズムはほとんど明らかになっていない[6]。
転移因子は上述のように有害な影響を及ぼす可能性があることから、一般の細胞内では転移が抑制されていることが多い。例外として、生殖細胞および脳神経細胞では転移が観察されており、特にニューロンのモザイク性を生み出している可能性が指摘されている[7]。しかしなぜこれらの細胞内のみで転移可能なのかは明らかになっていない。体細胞における抑制機構としては、転移因子配列のメチル化やヘテロクロマチン化などのエピジェネティックな制御を受けていることが知られている。また、small RNAによる抑制も受けていることが近年明らかになってきている[8]。
転移因子の挿入は進化的に中立の変異であり、一般にその配列は進化の過程で塩基置換が蓄積し、やがて転移因子であることの検出が困難になると考えられる。しかし例外的に、その過程で転移因子由来の配列が生物の生存に有利な何らかの機能を獲得する場合が知られている(exaptationまたはco-optionとも呼ばれる)。それらの配列は進化的に保存されることが多く[9][10]、特に有胎盤類では、保存領域の16%が転移因子に由来することが分かっている[11](編集 林 コメント:この文献はmarsupialとありますので、有袋類ではないでしょうか)。転移因子が獲得した機能としては、遺伝子のエキソン化や選択的スプライシングの生成といったタンパク質コード配列の改変に加え、エンハンサーやインシュレーターなどの調節配列となることが知られている。特に哺乳類の脳において複数のレトロポゾンがエンハンサー機能を有することが報告されており、哺乳類の脳の形成に関与したexaptationの好例として知られている[12]。
生物学的利用
また、転移という特徴を利用し、転移因子を遺伝学的ツールとして用いることができる。具体的な利用方法の1つとして、転移因子を用いた順遺伝学的スクリーニングがある。これは転移因子を生体内で転移させることで変異体を多数作成し、特定の表現型を示す個体を選別してその原因となる転移因子の挿入サイトを特定する方法である。代表的なものとして、P element、Sleeping beauty、piggyBac、Tol2などが用いられている[13]。この手法は、ショウジョウバエをはじめ、ゼブラフィッシュやマウスなどの脊椎動物でも幅広く用いられている。
また転移因子を利用したゲノムへの遺伝子導入、すなわち遺伝子組み換え生物の作成も広くおこなわれている。目的遺伝子の両端にDNAトランスポゾンの末端配列を付加したコンストラクトを胚へ導入し、トランスポゼースを利用して転移させることで、効率良くゲノム中に導入することができる。この方法では、piggyBacやTol2がよく利用されている[14]。
参考文献
- ↑
McCLINTOCK, B. (1950).
The origin and behavior of mutable loci in maize. Proceedings of the National Academy of Sciences of the United States of America, 36(6), 344-55. [PubMed:15430309] [PMC] [WorldCat] [DOI] - ↑
Kapitonov, V.V., & Jurka, J. (2008).
A universal classification of eukaryotic transposable elements implemented in Repbase. Nature reviews. Genetics, 9(5), 411-2; author reply 414. [PubMed:18421312] [WorldCat] [DOI] - ↑
Belancio, V.P., Hedges, D.J., & Deininger, P. (2008).
Mammalian non-LTR retrotransposons: for better or worse, in sickness and in health. Genome research, 18(3), 343-58. [PubMed:18256243] [WorldCat] [DOI] - ↑
Deininger, P.L., Moran, J.V., Batzer, M.A., & Kazazian, H.H. (2003).
Mobile elements and mammalian genome evolution. Current opinion in genetics & development, 13(6), 651-8. [PubMed:14638329] [WorldCat] [DOI] - ↑
Kazazian, H.H. (2004).
Mobile elements: drivers of genome evolution. Science (New York, N.Y.), 303(5664), 1626-32. [PubMed:15016989] [WorldCat] [DOI] - ↑
Schaack, S., Gilbert, C., & Feschotte, C. (2010).
Promiscuous DNA: horizontal transfer of transposable elements and why it matters for eukaryotic evolution. Trends in ecology & evolution, 25(9), 537-46. [PubMed:20591532] [PMC] [WorldCat] [DOI] - ↑
Muotri, A.R., Chu, V.T., Marchetto, M.C., Deng, W., Moran, J.V., & Gage, F.H. (2005).
Somatic mosaicism in neuronal precursor cells mediated by L1 retrotransposition. Nature, 435(7044), 903-10. [PubMed:15959507] [WorldCat] [DOI] - ↑
Kim, V.N., Han, J., & Siomi, M.C. (2009).
Biogenesis of small RNAs in animals. Nature reviews. Molecular cell biology, 10(2), 126-39. [PubMed:19165215] [WorldCat] [DOI] - ↑
Nishihara, H., Smit, A.F., & Okada, N. (2006).
Functional noncoding sequences derived from SINEs in the mammalian genome. Genome research, 16(7), 864-74. [PubMed:16717141] [PMC] [WorldCat] [DOI] - ↑
Bejerano, G., Lowe, C.B., Ahituv, N., King, B., Siepel, A., Salama, S.R., ..., & Haussler, D. (2006).
A distal enhancer and an ultraconserved exon are derived from a novel retroposon. Nature, 441(7089), 87-90. [PubMed:16625209] [WorldCat] [DOI] - ↑
Mikkelsen, T.S., Wakefield, M.J., Aken, B., Amemiya, C.T., Chang, J.L., Duke, S., ..., & Lindblad-Toh, K. (2007).
Genome of the marsupial Monodelphis domestica reveals innovation in non-coding sequences. Nature, 447(7141), 167-77. [PubMed:17495919] [WorldCat] [DOI] - ↑
Sasaki, T., Nishihara, H., Hirakawa, M., Fujimura, K., Tanaka, M., Kokubo, N., ..., & Okada, N. (2008).
Possible involvement of SINEs in mammalian-specific brain formation. Proceedings of the National Academy of Sciences of the United States of America, 105(11), 4220-5. [PubMed:18334644] [PMC] [WorldCat] [DOI] - ↑
Ivics, Z., Li, M.A., Mátés, L., Boeke, J.D., Nagy, A., Bradley, A., & Izsvák, Z. (2009).
Transposon-mediated genome manipulation in vertebrates. Nature methods, 6(6), 415-22. [PubMed:19478801] [PMC] [WorldCat] [DOI] - ↑
Kawakami, K. (2007).
Tol2: a versatile gene transfer vector in vertebrates. Genome biology, 8 Suppl 1, S7. [PubMed:18047699] [PMC] [WorldCat] [DOI]