ミカエリス・メンテンの式
石田 敦彦
広島大学 大学院総合科学研究科
DOI:10.14931/bsd.2353 原稿受付日:2012年8月29日 原稿完成日:2012年9月10日
担当編集委員:林 康紀(独立行政法人理化学研究所 脳科学総合研究センター)
英語名:Michaelis-Menten equation 独:Michaelis-Menten-Gleichnung 仏:Équation de Michaelis-Menten
同義語: ミハエリス・メンテンの式、ミヒャエリス・メンテンの式
酵素は生体内の各種の化学反応を円滑に行わせるための生体触媒であり、脳内においても情報伝達や物質代謝など、あらゆる生化学反応に関わっている。酵素の生化学的研究をおこなうにあたっては、酵素の性質を定量的に扱うことが大前提となるが、そのような場合の理論的基盤となるものが、以下に述べるミカエリス・メンテンの式である。ここでは更に、それを拡張したブリッグス・ホールデンの式の誘導について述べた後、これらの式による解析から得られる各種速度論的パラメータの求め方や意味について概説する。また、酵素阻害剤の理論的取り扱いについても代表的な例について簡単に解説し、最後に脳科学に関連の深い酵素に関する若干の研究例を紹介する。
背景
脳神経系を理解する上で、個々の酵素の性質を明らかにすることは極めて重要である。1992年にジーンターゲティングの手法を用いて、空間記憶に関わる酵素としてCaMキナーゼⅡが初めて特定された[1][2]が、この輝かしい研究成果も、それを遡ること十数年に渡る本酵素に関する地道で精力的な研究の積み重ね[3]があったればこそのものであろう。そのような酵素研究に必須の理論であるミカエリス・メンテンの式とそれを用いた各種の解析について以下に概説する。
誘導法
L. Michaelis とM. L. Mentenはインベルターゼに関する研究において、酵素の反応速度と基質濃度の関係を明らかにするため、酵素と基質が結合した酵素基質複合体(ES complex)を形成することにより酵素反応が進行するとの概念に基づいて、次のような反応スキームを考えた。
(1)
ここにEは酵素、Sは基質、Pは生成物を表す。この時、k1、k2はk3に比べて十分に大きく、ES、E、Sは平衡状態にあって、k3を速度定数とする過程が全体の酵素反応の律速段階であると仮定すれば、ES complexの解離平衡定数Kdは
(2)
酵素反応の初速度vは
(3)
ここで酵素の全濃度[E0]は
(4)
(2)(4)より[E]を消去して整理すると
(5)
これを(3)に代入すれば
(6)
ここでv = k3[E0] = Vmax、Kd = Kmとおくと
(7)
この(7)式をミカエリス・メンテンの式と呼び、1913年にドイツの学術雑誌に発表された[4]。ちなみにM. L. Mentenは当時としては珍しい女性研究者である[5]。ミカエリス定数Kmは基質濃度無限大の時の最大反応速度Vmaxの1/2の速度を与える時の基質濃度に一致する。KmはES complexの解離平衡定数Kdであるから、酵素と基質の親和性の尺度となり、値が小さいほど酵素と基質の親和性が強い。
ブリッグス・ホールデンの式
しかしながら、上記、Michaelis とMentenの考えではいくつかの仮定を設けており、常にこれらの仮定が成立するとは限らない。そこで1925年にG. E. BriggsとJ. B. S. Haldaneは、ミカエリス・メンテンの式の、より一般化された誘導法を示した[6]。上記(1)の反応スキームにおいて、彼らは酵素反応が直線的に進行する定常状態ではES complexの形成速度と分解速度が釣り合っていて、見かけ上[ES]が一定になると仮定した(定常状態近似)。すなわち、
(8)
ここで上記と同様に酵素の全濃度[E0]は
(9)
(8)(9)より[E]を消去すると
(10)
酵素反応の初速度vは
(11)
(10)(11)より
(12)
ここで (k2 + k3) / k1 = Km、k3[E0] = Vmaxとおくと
(13)
となり、(7)式と同じ式が得られる。 (13)式は厳密にはブリッグス・ホールデンの式と言うが、 (7)式と同じ形であるので実際にはミカエリス・メンテンの式と言うことが多い。また、(13)式のKmもミカエリス定数と言うが、(7)式の場合と異なり、ES complexの解離平衡定数Kdとは一致しない。k2 > > k3の場合にのみ、Km ≈ k2/k1となってKdと一致するのであるが、多くの場合、(13)式のKmも酵素と基質の親和性の尺度を表すと考えてよい。実験的には、(13)式のKmも(7)式の場合と同様、基質濃度無限大の時の最大反応速度の1/2の速度を与える基質濃度として定義される。
ミカエリス・メンテンプロット
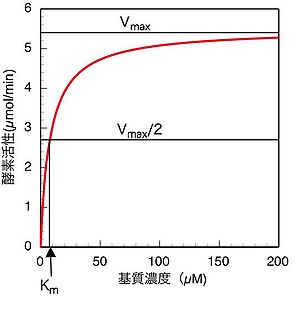
(7)式も(13)式も、酵素反応速度(すなわち酵素活性)と基質濃度の関係を定量的に表した式である。実験的には様々な基質濃度で酵素活性を測定し、横軸に基質濃度、縦軸に酵素活性をとってプロットした場合、図1に示すように、数学的には直角双曲線の形となる。このようなプロットをミカエリス・メンテンプロット(S-vプロット)と呼ぶ。図1から明らかなように、基質濃度がKm値(Vmaxの1/2の速度を与える時の基質濃度)付近或いはそれ以下の場合には酵素活性は基質濃度に大きく依存し、基質濃度の少しの変化でも酵素活性は大きく影響を受けるが、Km値より十分大きい基質濃度の場合、酵素活性はVmaxの値に近づき、濃度が大きくなるにつれて基質濃度依存性が殆どなくなる。従って、一般に酵素活性を測定する場合は、基質初濃度の誤差や、反応の進行に伴う基質濃度減少の影響を避けるため、できるだけ高濃度の基質(Km値の5〜10倍、或いはそれ以上)を用いて活性測定を行うことが望ましい。しかしながら基質阻害により、高濃度では逆に活性が低下する場合もあるので、基質濃度を予め低濃度から高濃度まで振ってみて基質阻害がないことを確認するなどの注意も必要である。
速度論的パラメータの求め方
図1のようなミカエリス・メンテンプロットより、上記KmやVmaxなどの速度論的パラメータを求めることが出来る。(7)式または(13)式の両辺の逆数をとって
(14)
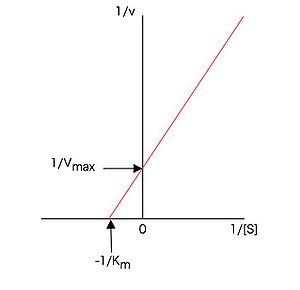
とすれば、1 / [S]に対する1 / vのプロットが直線となる。従ってミカエリス・メンテンの式に従う酵素では、基質濃度の逆数に対して、酵素活性の逆数をプロットすれば図2に示すような直線プロット(ラインウィーバー・バークプロットまたは二重逆数プロット)となり、このプロットのx切片が − 1 / Km,y切片が1 / Vmaxを与える。この方法はグラフ用紙さえあれば簡単にできるので以前はよく行われたが、低基質濃度のデータの誤差が大きく出るなどの欠点もあり、パソコンが普及した現在では、ミカエリス・メンテンプロットを適当なソフトウェアを用いて双曲線にフィッティングして、直接(7)式または(13)式の各パラメータを求めるdirect fitting法によることが多くなった。
(7)式または(13)式(ミカエリス・メンテンの式またはブリッグス・ホールデンの式)は多くの酵素にあてはまる便利な式であるが、(1)の反応スキームに従うことを前提にしているので、当然これにあてはまらない場合も存在する。そのような場合に(7)式または(13)式を無理にあてはめて解析することは、誤った結論を導く可能性があるので注意が必要である。そのような場合の扱いに関しては、例えば以下の文献を参照されたい[7]。
速度論的パラメータの意味
前にも述べたように、KmはVmaxの1/2の速度を与える時の基質濃度として定義され、酵素と基質の親和性の尺度となる。また、Vmaxは基質濃度無限大、つまり酵素分子全てが基質で飽和された時の反応速度である。定義により、Vmax = k3[E0]であるが、このk3を触媒定数、或いはターンオーバー・ナンバーと呼び、通常kcatで表す。すなわち
(15)
である([E0]は全酵素濃度)。kcatは酵素が基質で飽和された状態において、1モルの酵素(或いは活性部位)が1秒間に生成物へ変換できる基質のモル数を表し、単位はs − 1である。すなわちkcatは酵素の触媒効率を表す指標である。また、(7)または(13)式のVmaxを(15)式により、kcat[E0]で置き換えると
(16)
ここで基質濃度が非常に希薄な[S] < < Kmの濃度領域を考えると
(17)
基質が非常に薄い条件下では、基質は殆ど酵素に結合していないと考えられるから
[E0]≈[E]
従って
(18)
この式はEとSの衝突が反応全体の速度を支配していると考えた場合の二次反応速度定数がkcat / Kmであることを示している。kcat / Kmの値は、異なる酵素の触媒効率を比較する際のパラメータとして用いられる。また、同一の酵素に対して、異なる基質の特異性を議論する場合にもkcat / Kmの値が用いられ、特異性定数と呼ばれることがある。この場合、kcat / Kmの値が大きいほど、その酵素に対してよい基質であるということになる。
阻害剤存在下の酵素反応速度論
酵素の特異的阻害剤は、その酵素の生理的意義を明らかにする分子プローブとして重要であるのみならず、医薬品として臨床上、重要な役割を担うものも多い。ミカエリス・メンテンの式を応用して様々な濃度の酵素阻害剤存在下での酵素活性を詳細に調べることにより、阻害剤と酵素の結合様式や結合親和性について重要な情報を得ることが出来る。
競合阻害
(1)の反応スキームにおいて阻害剤が酵素の基質結合部位に結合し、基質Sと阻害剤が結合部位を奪い合うような場合を競合阻害(拮抗阻害:competitive inhibition)と呼ぶ。この場合、(1)の反応スキームに加えて
(19)
という結合解離平衡を仮定し、その解離定数をKiとおくと
(20)
この場合、酵素の全濃度[E0]は(4)式に代わって
(21)
(2)(20)(21)より[E]と[EI]を消去してについて整理すると
(22)
これを(3)に代入すれば
(23)
ここで k3[E0] = Vmax, Kd = Kmであるから
(24)
この式を元のミカエリス・メンテンの式 (7)式と比較すると、競合阻害剤の存在下ではVmaxは変化しないが、Km値が1 + [I] / Ki倍だけ増加していることが分かる。 また、(14)式と同様に(24)式の逆数をとって整理すると
(25)
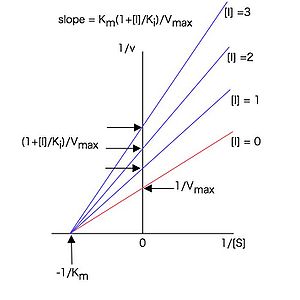
従って1 / [S]に対して1 / vをプロット(ラインウィーバー・バークプロットまたは二重逆数プロット)すると図3のような直線プロットとなり、様々な濃度の阻害剤の存在下で実験すると、y軸上の一点(y切片 = 1 / Vmax)で交わる直線群が得られる。
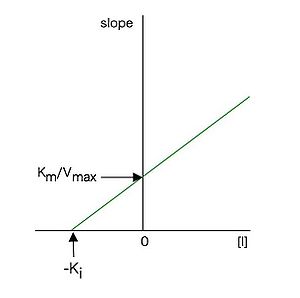
これらの直線の傾きは
(26)
と表せるので、各阻害剤濃度[I]に対して、図3のラインウィーバー・バークプロットの傾きをプロットした図4のような2次プロットを作成すると、(26)式に従った直線となり、その直線のx切片(に相当)の値からKi値を求めることが出来る。Kiは阻害定数と呼ばれ、この場合、酵素—阻害剤複合体の解離定数に相当する。Kiは酵素と阻害剤の親和性の尺度であり、値が小さいほど酵素に対する親和性が強いことを示す。
非競合阻害
(1)の反応スキームにおいて阻害剤と基質Sが互いに異なる部位に独立に結合し、互いの結合に影響を及ぼさないような場合、これを非競合阻害(非拮抗阻害:noncompetitive inhibition)と呼ぶ。この場合、(1)(19)の反応スキームに加えて
(27)
(28)
という結合解離平衡の存在を仮定することになるが、(27)(28)の解離平衡定数は、互いの結合に影響を及ぼさないという定義により、それぞれKi, Kdと等しくなる。すなわち、
(29)
(30)
また酵素の全濃度[E0]は
[E0] = [E] + [ES] + [EI] + [ESI] (31)
となる。上記と同様に(29)(30)(31)より[E], [EI], [ESI]を消去し、得られた[ES]を(3)に代入して、k3[E0] = Vmax, Kd = Kmとおくと、
(32)
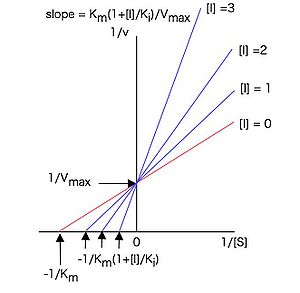
この式を元のミカエリス・メンテンの式 (7)式と比較すると、非競合阻害剤の存在下ではKmは変化しないがVmaxが1 / (1 + [I] / Ki)だけ減少していることが分かる。また、(14)や(24)と同様に(32)式の逆数をとって整理すると
(33)
従って1 / [S]に対して1 / vをプロットすると図5のような直線プロットとなり、様々な濃度の阻害剤の存在下で実験すると、x軸上の一点(x切片 = − 1 / Km)で交わる直線群が得られる。これらの直線の傾きは (26)式で表せるので、競合阻害の場合と同様、各阻害剤濃度[I]に対して、図5のラインウィーバー・バークプロットの傾きをプロットした2次プロットは図4のようになり、その直線のx切片の値からKi値を求めることが出来る。この場合も阻害定数Kiは値が小さいほど酵素に対する親和性が強いことを示す。
以上のように、阻害剤濃度や基質濃度を様々に変えて酵素活性を測定し、図3や図5のようなラインウィーバー・バークプロットのパターンを調べることにより、その阻害剤と酵素の親和性や阻害剤の結合部位に関する情報を簡便に得ることが出来る。
酵素反応速度論的解析の実例
以上述べてきたような各種の速度論的パラメータは、酵素の反応特異性や反応機構に関して、しばしば重要な知見を与える。神経科学分野で重要な役割を担ういくつかの酵素においてもこのような解析がなされている。例えばカテコールアミンの生合成に重要な役割を果たすチロシン水酸化酵素では、cAMP依存性タンパク質リン酸化酵素 (Aキナーゼ)[8]やCa2+/リン脂質依存性タンパク質リン酸化酵素 (Cキナーゼ)[9]によってリン酸化されると、補酵素アナログである6-メチルテトラヒドロビオプテリンに対するが著明に減少し、補酵素との親和性が高まって活性化されることが示されている。
記憶学習に深く関係することが明らかとなっているCaMキナーゼⅡに関しても、詳細な速度論的解析がなされている。CaMキナーゼⅡはThr286が自己リン酸化されるとCa2+/CaM (カルモジュリン)に非依存的な活性が出現し、この活性が記憶やその素過程と考えられる長期増強現象の成立に重要な役割を果たすと考えられているが、様々な基質を用いて速度論的解析を行った結果、このCa2+/CaM非依存性活性ではCa2+/CaM存在下の活性に比べて、には変化がないものの、調べた全ての基質に関してが増大しており、基質との親和性が低下していることが判明した[10]。また、本酵素の活性制御に重要な役割を果たす自己阻害ドメインの合成ペプチドを用いて図3や図5のような阻害実験を行うことにより、活性制御機構に関する重要な知見が得られている[11]。同様に自己リン酸化部位Thr286周辺の配列を模した合成阻害ペプチドを用いて阻害実験を行うことにより、少なくとも2種類の異なる基質結合部位が存在することが初めて示唆されたが[12]、この結果は、後に本酵素の活性制御機構やNMDA型グルタミン酸受容体との相互作用を解明する上で不可欠となるT-site、S-siteという2種類の基質結合部位に関する概念[13]を確立する上で、先駆的な役割を果たしている。
関連項目
参考文献
- ↑
Silva, A.J., Stevens, C.F., Tonegawa, S., & Wang, Y. (1992).
Deficient hippocampal long-term potentiation in alpha-calcium-calmodulin kinase II mutant mice. Science (New York, N.Y.), 257(5067), 201-6. [PubMed:1378648] [WorldCat] [DOI] - ↑
Silva, A.J., Paylor, R., Wehner, J.M., & Tonegawa, S. (1992).
Impaired spatial learning in alpha-calcium-calmodulin kinase II mutant mice. Science (New York, N.Y.), 257(5067), 206-11. [PubMed:1321493] [WorldCat] [DOI] - ↑
Hudmon, A., & Schulman, H. (2002).
Neuronal CA2+/calmodulin-dependent protein kinase II: the role of structure and autoregulation in cellular function. Annual review of biochemistry, 71, 473-510. [PubMed:12045104] [WorldCat] [DOI] - ↑
Michaelis, L., Menten, M.L., Johnson, K.A., & Goody, R.S. (2011).
The original Michaelis constant: translation of the 1913 Michaelis-Menten paper. Biochemistry, 50(39), 8264-9. [PubMed:21888353] [PMC] [WorldCat] [DOI] - ↑ 鈴木紘一、笠井献一、宗川吉汪 監訳
ホートン生化学 第4版
東京化学同人 (東京):2008 - ↑
Briggs, G.E., & Haldane, J.B. (1925).
A Note on the Kinetics of Enzyme Action. The Biochemical journal, 19(2), 338-9. [PubMed:16743508] [PMC] [WorldCat] [DOI] - ↑ 堀尾武一、山下仁平
蛋白質・酵素の基礎実験法
南江堂 (東京):1981 - ↑
Vulliet, P.R., Langan, T.A., & Weiner, N. (1980).
Tyrosine hydroxylase: a substrate of cyclic AMP-dependent protein kinase. Proceedings of the National Academy of Sciences of the United States of America, 77(1), 92-6. [PubMed:6102382] [PMC] [WorldCat] [DOI] - ↑
Albert, K.A., Helmer-Matyjek, E., Nairn, A.C., Müller, T.H., Haycock, J.W., Greene, L.A., ..., & Greengard, P. (1984).
Calcium/phospholipid-dependent protein kinase (protein kinase C) phosphorylates and activates tyrosine hydroxylase. Proceedings of the National Academy of Sciences of the United States of America, 81(24), 7713-7. [PubMed:6151178] [PMC] [WorldCat] [DOI] - ↑
Ikeda, A., Okuno, S., & Fujisawa, H. (1991).
Studies on the generation of Ca2+/calmodulin-independent activity of calmodulin-dependent protein kinase II by autophosphorylation. Autothiophosphorylation of the enzyme. The Journal of biological chemistry, 266(18), 11582-8. [PubMed:1646810] [WorldCat] - ↑
Colbran, R.J., Smith, M.K., Schworer, C.M., Fong, Y.L., & Soderling, T.R. (1989).
Regulatory domain of calcium/calmodulin-dependent protein kinase II. Mechanism of inhibition and regulation by phosphorylation. The Journal of biological chemistry, 264(9), 4800-4. [PubMed:2538462] [WorldCat] - ↑
Ishida, A., & Fujisawa, H. (1995).
Stabilization of calmodulin-dependent protein kinase II through the autoinhibitory domain. The Journal of biological chemistry, 270(5), 2163-70. [PubMed:7836445] [WorldCat] [DOI] - ↑
Bayer, K.U., De Koninck, P., Leonard, A.S., Hell, J.W., & Schulman, H. (2001).
Interaction with the NMDA receptor locks CaMKII in an active conformation. Nature, 411(6839), 801-5. [PubMed:11459059] [WorldCat] [DOI]

![{\displaystyle K_{d}={\frac {[E][S]}{[ES]}}={\frac {k_{2}}{k_{1}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/9d8d8b8bb255d6f7a9055904d2663dda5f1cc03c)
![{\displaystyle v=k_{3}[ES]\,}](https://wikimedia.org/api/rest_v1/media/math/render/svg/470759c2df3dffd24de90535c86cbf0dd33523e6)
![{\displaystyle [E_{0}]=[E]+[ES]\,}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d4472f0c3b9cc7fb11de0e1928582ff1399995d8)
![{\displaystyle [ES]={\frac {[E_{0}][S]}{K_{d}+[S]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/10c46040ce2c698072965fa482e386ff6cc14e4c)
![{\displaystyle v=k_{3}[ES]={\frac {k_{3}[E_{0}][S]}{K_{d}+[S]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/71941aacda4fcaa7289a38338a2eb868ecab95dd)
![{\displaystyle v=k_{3}[ES]={\frac {V_{max}[S]}{K_{m}+[S]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/ea0a12c43b721dbdc43f2f9809852669700b865b)
![{\displaystyle {\frac {d[ES]}{dt}}=0=k_{1}[E][S]-k_{2}[ES]-k_{3}[ES]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/173dd9b63c2d5d73be56e3551c85551ea710c086)
![{\displaystyle [ES]={\frac {k_{1}[E_{0}][S]}{k_{1}[S]+(k_{2}+k_{3})}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/80e18836e284675af4355e804f36f0063e9a3e38)
![{\displaystyle v={\frac {k_{1}k_{3}[E_{0}][S]}{k_{1}[S]+(k_{2}+k_{3})}}={\frac {k_{3}[E_{0}][S]}{[S]+{\frac {k_{2}+k_{3}}{k_{1}}}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/d788fd0d7b1fabccebdb6165429540da26faf812)

![{\displaystyle {\frac {1}{v}}={\frac {K_{m}}{V_{max}}}{\frac {1}{[S]}}+{\frac {1}{V_{max}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e01b24fd3322236f17dabe08f4392bc4374f2a5b)
![{\displaystyle k_{cat}={\frac {V_{max}}{[E_{0}]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/e301ceb37495b61b840ff15980200ad4c80785c5)
![{\displaystyle v=k_{3}[ES]={\frac {k_{cat}[E_{0}][S]}{K_{m}+[S]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a5df3c53399d91bbf92ce17b8bdeeecc6c793ce7)
![{\displaystyle v={\frac {k_{cat}[E_{0}][S]}{K_{m}+[S]}}\approx {\frac {k_{cat}[E_{0}][S]}{K_{m}}}={\frac {k_{cat}}{K_{m}}}[E_{0}][S]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/5894644eea717d60f02a131086387def6be05290)
![{\displaystyle v={\frac {k_{cat}}{K_{m}}}[E][S]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/07bb6087713ffefcf5d71c2ade0031561aa3d135)


![{\displaystyle K_{i}={\frac {[E][I]}{[EI]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/a51fc36b6995e70b7530bc07bfe94e2caf91483b)
![{\displaystyle [E_{0}]=[E]+[ES]+[EI]\,}](https://wikimedia.org/api/rest_v1/media/math/render/svg/05f123186824b7487ce5983a76174ca26476feb3)
![{\displaystyle [ES]}](https://wikimedia.org/api/rest_v1/media/math/render/svg/31639ab6b9c7c728139b5f8ce03991d800ac7741)
![{\displaystyle [ES]={\frac {[E_{0}][S]}{[S]+K_{d}(1+{\frac {[I]}{K_{i}}})}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/1d601b8a9a06243f9a39fddf68ee4ab32416207f)
![{\displaystyle v={\frac {k_{3}[E_{0}][S]}{[S]+K_{d}(1+{\frac {[I]}{K_{i}}})}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/857e0468ca980ecd422b6e6377836dbf632d9cc5)
![{\displaystyle v={\frac {V_{max}[S]}{[S]+K_{m}(1+{\frac {[I]}{K_{i}}})}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/8f17994b28a0d5e3bf69fd8e4299ecbc1ffb4d6b)
![{\displaystyle {\frac {1}{v}}={\frac {K_{m}(1+{\frac {[I]}{K_{i}}})}{V_{max}}}{\frac {1}{[S]}}+{\frac {1}{V_{max}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/f4e61e1e33e0cb5913e910aa266a0b85f1a8f6c7)
![{\displaystyle {\frac {K_{m}(1+{\frac {[I]}{K_{i}}})}{V_{max}}}={\frac {K_{m}}{V_{max}}}{\frac {1}{K_{i}}}[I]+{\frac {K_{m}}{V_{max}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/4e64602c8ce36d6f98e304f5c54cba26babb5b66)



![{\displaystyle K_{i}={\frac {[E][I]}{[EI]}}={\frac {[ES][I]}{[ESI]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/5bb01236ca1bb5872b5262fd0dc4cbf16b4f765e)
![{\displaystyle K_{d}={\frac {[E][S]}{[ES]}}={\frac {[EI][S]}{[ESI]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/82aabf0e73c0c977b2417d4874856d57533c54fd)
![{\displaystyle v={\frac {1}{1+{\frac {[I]}{K_{i}}}}}{\frac {V_{max}[S]}{K_{m}+[S]}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/224f8fd5bbc687d586f2264c0a7e261bea1206aa)
![{\displaystyle {\frac {1}{v}}={\frac {K_{m}(1+{\frac {[I]}{K_{i}}})}{V_{max}}}{\frac {1}{[S]}}+{\frac {1+{\frac {[I]}{K_{i}}}}{V_{max}}}}](https://wikimedia.org/api/rest_v1/media/math/render/svg/2127b8568071e85616e974aea6fb0779db5f384a)