嗅覚受容体
伊原 さよ子、東原 和成
東京大学大学院 農学生命科学研究科
DOI:10.14931/bsd.10533 原稿受付日:2023年11月10日 原稿完成日:2023年X月XX日
担当編集委員:古屋敷 智之(神戸大学大学院医学研究科・医学部 薬理学分野)
英:olfactory receptor、odorant receptor 独:Geruchsrezeptor, olfaktorisch Rezeptor 仏:récepteur olfactif
英略称:OR
嗅覚受容体は、嗅覚器官に存在する嗅神経細胞に発現し、匂い物質との結合を引き金として神経細胞の興奮をもたらし、脳に信号を伝える。ほとんどの生物種において、嗅覚受容体遺伝子はスーパーファミリーを形成し、数多くの嗅覚受容体の存在が多様なシグナルの識別を可能としている。脊椎動物と昆虫では、嗅覚受容体の構造と分子機能が本質的に異なり、各々の生存環境、生活様式を反映していると考えられている。
嗅覚受容体とは
生物は外界からの様々な刺激を感知し、適切に応答することで生存を維持している。様々な感覚のうち、嗅覚は化学物質を媒体として外界の情報を感知するシステムであり、食物や配偶者の探索、敵からの逃避、といった生存に必須な行動に重要な役割を果たす。極めて多様なシグナルを感度良く識別できるのが嗅覚システムの特徴であり、嗅覚受容体はその識別の出発点としての役目を果たす。
日本語ではいずれも嗅覚受容体であるが、英語でolfactory receptorは、広義に嗅覚組織に発現している嗅覚受容体全般を指す一方、odorant receptorは狭義に揮発性匂い物質の受容体を指す。脊椎動物では通常olfactory receptorが用いられるが、昆虫では嗅覚受容体ファミリー名がodorant receptorと定められている。また、げっ歯類において、嗅覚受容体は広義に、鋤鼻器官に発現する”フェロモン受容体”も含める場合もあるが、本項目では含めない。フェロモン受容体の項目を参照。
脊椎動物
発見、歴史的経緯など
我々が匂いを感知する仕組みについては、古くから複数の学説が唱えられていたが、そのうちのひとつが、Amooreによる立体化学説であった。匂い分子の化学構造、形とサイズが鼻腔上皮の受容部位の構造に適合すると匂いが感知されるとの説である[1]。この学説で概念に過ぎなかった”受容体”の存在は、1991年、BuckとAxelによる、ラット嗅覚受容体(遺伝子ファミリーの歴史的な発見により明らかとなった[2]。
その後、嗅覚受容体遺伝子によりコードされるタンパク質が匂い物質に応答し、嗅神経細胞の活性化をもたらすことが実証された[3][4]。嗅覚受容体遺伝子は脊椎動物全般において、最大の遺伝子ファミリーとして存在し、多重遺伝子ファミリーを形成するが、その数は生物種により大きく異なり、例えばマウスでは約1100、ヒトでは約400存在する[5]。嗅覚受容体遺伝子ファミリーは他の遺伝子ファミリーに比べると偽遺伝子の割合が高く、進化の過程での重複、欠失が多いことも特徴である。さらに、ヒト個人間においても数多くの遺伝子多型が存在し、特定の匂いへの知覚感度に影響する例も報告されている [6][7][8][9]。
嗅覚受容体に加え、2006年、嗅上皮で発現する微量アミン関連受容体 (trace amine-associated receptor, TAAR)ファミリーも嗅覚受容体として機能することが報告された[10]。その後、げっ歯類嗅上皮で発現するグアニル酸シクラーゼ D (guanylyl cyclase D, GCD) が呼気中のCO2、CS2の受容体としてはたらくことが示された[11][12]。
さらに2016年、嗅上皮のくぼみに存在する嗅神経細胞に発現する嗅覚受容体として、membrane-spanning 4A receptor (MS4A)が発見されている[13]。
構造
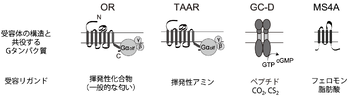
嗅覚受容体はGタンパク質共役型受容体ファミリーのうち、ロドプシンファミリーとよばれるサブファミリーに属し、ヘリックス構造から成る7回膜貫通構造を有する(図1)。全てのGタンパク質共役型受容体に共通な配列の他、3番目の膜貫通領域細胞質側のMAYDRYVAICモチーフをはじめ、嗅覚受容体を特徴づける複数の配列をもつ。
哺乳類嗅覚受容体は、アミノ酸配列の相同性からクラスI、クラスIIに分類され、哺乳類嗅覚受容体の約10~20%がクラスIである [14]。認識するリガンドが、クラスIは親水性、クラスIIは疎水性といった傾向を示す[15]。
嗅覚受容体の立体構造については、2023年3月、脊椎動物で初めてクライオ電子顕微鏡による結果が報告された。嗅覚組織以外の様々な組織でも発現する嗅覚受容体のうち、クラスIに属するOR51E2について、匂い分子としてはやや例外的な親水性リガンド、プロピオン酸との結合状態を示したものであり[16]、画期的な進展であるが、疎水性匂い分子と嗅覚受容体の一般的な結合様式を反映しているかについては、今後の展開が待たれる。
その他の嗅覚受容体については、構造が既知のGタンパク質共役型受容体の情報をもとにした、in silico解析、変異体解析により、匂い物質との相互作用モデルが複数例報告されており、いずれの場合もリガンド結合には、3, 5, 6番目の膜貫通領域のアミノ酸が重要とされている[17][18][19][20]。
微量アミン関連受容体ファミリーもGタンパク質共役型受容体に分類され、ロドプシンファミリーに属するが、嗅覚受容体より生体アミン受容体に高い相同性を示す[21]。
グアニル酸シクラーゼDは1回膜貫通型受容体であり、細胞外にリガンド結合領域を、細胞内にプロテインキナーゼ様ドメインを、C末端に触媒ドメインをもつ。
Membrane-spanning 4A receptorは4回膜貫通型タンパク質で、N末端、C末端をともに細胞質側に配置するトポロジーを示す[13](図1)。
発現部位
嗅覚受容体は、嗅上皮に存在する嗅神経細胞に発現する。嗅上皮は、ヒトの場合、鼻腔天井部の5 cm2程度の領域に存在し、嗅粘液層に覆われている。嗅神経細胞は嗅粘液層にむかって10本程度の線毛を伸ばしており、この上に発現する嗅覚受容体が嗅粘液層に溶け込んだ匂い物質を受容する。嗅覚受容体の発現様式には、1つの嗅神経細胞には1種類の受容体しか発現しない、「1神経細胞1受容体ルール」が存在する。同じ嗅覚受容体を発現する嗅神経細胞は嗅上皮上ではそれぞれ特定の領域に分布するが[22]、投射部位である脳の嗅球と呼ばれる領域では同じ部位に収束する。
1991年嗅覚受容体遺伝子ファミリー発見当時、嗅覚受容体は嗅上皮に限定して発現すると考えられていたが、次第に多くの嗅覚受容体が精巣、腎臓、肺、筋、腸、といった他の様々な組織でも発現することが明らかになった。ヒトの場合、約400種類の嗅覚受容体のうち、約100種類が非嗅覚組織でも発現している[23]。非嗅覚組織で発現する嗅覚受容体は、精子の走化性[24][25]、筋再生[26]、炎症反応[27]、エネルギー代謝調節[28][29][30]といった様々な現象に関わることが示唆されており、創薬の標的としても着目され始めている[31]。
その他、嗅覚受容体として機能する微量アミン関連受容体、グアニル酸シクラーゼD、membrane-spanning 4A receptorのいずれも嗅上皮の嗅神経細胞に発現するが、微量アミン関連受容体, グアニル酸シクラーゼDが嗅上皮全域にわたって発現が見られる一方、membrane-spanning 4A receptorは嗅上皮のくぼみの部分に限局して発現する。
機能
嗅覚受容体は、匂い物質がもつ化学情報を電気信号に変換し、神経細胞の興奮をもたらし、脳に伝達する役目をもつ。
リガンドである匂い分子が結合すると、受容体と共役している3量体Gタンパク質のαサブユニット、Gαolfがβ、γサブユットと解離し、GDP型からGTP型への変換を受け、活性化される。活性化Gαolfがアデニル酸シクラーゼの活性化を引き起こし、細胞内cAMP濃度の上昇をもたらすと環状ヌクレオチド作動性チャネル(cyclic nucleotide-gated channel, CNG)が開口し、Na+イオン、Ca2+イオンの流入による細胞膜の脱分極がおきる。細胞内Ca2+イオン濃度の上昇は、Ca2+作動性Cl-チャネル, transmembrane protein16B (TMEM16B, 別名 anoctamin 2, calcium-activated chloride channel, ANO2)の活性化をもたらし、Cl-イオンが細胞外へ流出することでより大きな脱分極が起きる。これにより、神経細胞の活動電位が生じる。シグナルを終結させる機構として、CNGのcAMPによるチャネルの開口がCa2+濃度依存的なフィードバック制御をうけること、細胞内濃度が上昇したCa2+は、Na+/Ca2+交換体であるNa+/Ca2+-exchanger isoform 4 (NCKX4)によって細胞外へ排出されることが明らかになっている。
嗅覚受容体とリガンドである匂い分子との対応関係は、一部の例外を除いては、「多対多」の関係にある。すなわち、一つの受容体は、複数の匂い分子に応答し、一つの匂い分子は複数の受容体応答を生み出すため、異なる匂いは、応答受容体の組み合わせパターンの違いによって識別される。この仕組みは“combinatorial coding”と呼ばれ[32]、受容体数をはるかに超える膨大な種類の匂いの嗅ぎ分けを可能にする。
嗅覚受容体が多様な構造の匂い物質を広く認識するのに対し、微量アミン関連受容体は、揮発性アミン化合物をリガンドとして認識する。揮発性アミン化合物は、尿中や腐った食物に存在しており、げっ歯類では、微量アミン関連受容体は異性、天敵、食物の質の区別の検知に関わるとされている[33]。微量アミン関連受容体も嗅覚受容体と同様、Gαolfと共役し、cAMP産生を通じて嗅神経細胞の活動を起こすとされている[21]。
グアニル酸シクラーゼDは、糞尿中に存在するペプチドの他、呼気中に存在するCO2, CS2をリガンドとして認識する[11][12]。匂いリガンドとしてはたらくCO2, CS2は嗅神経細胞膜を通過し、細胞内で炭酸水素イオンに変換されるが、この炭酸水素イオンがGCDの細胞内触媒ドメインに作用し、cGMP 産生がおきる。cGMPはcGMP依存性イオンチャネルを開口させることにより、神経細胞の脱分極を引き起こす。
Membrane-spanning 4A receptorはリガンドとして、動物行動に関連のある脂肪酸やフェロモン様物質、2,5-dimethylpyrazine (2,5-DMP)を認識するが、シグナル伝達は明らかになっていない[13]。
疾患との関わり
一部の嗅覚受容体は嗅上皮以外の正常組織でも発現がみられる他、各種がん細胞でも発現が確認されている。正常細胞と比較して、がん細胞で発現が亢進している嗅覚受容体も複数報告されており、腫瘍マーカーや治療標的候補としても着目されているが、がん細胞における具体的な機能については明らかになっていない[31][34]。
昆虫
発見、歴史的経緯など
昆虫の嗅覚受容体については、1991年の脊椎動物での嗅覚受容体遺伝子ファミリーの発見により、昆虫でも同様のGタンパク質共役型受容体ファミリーが存在するとの予想のもと探索が進められたが、試みは失敗に終わった。その後、ショウジョウバエ遺伝子を対象とした発現解析などの研究をもとに、異なる3グループにより、1999年、昆虫嗅覚受容体遺伝子ファミリーの発見が報告された[35][36][37]。構成遺伝子数は、数10~300程度と種により大きく異なり、ショウジョウバエでは、62遺伝子存在する。
その10年後、昆虫の第2の嗅覚受容体遺伝子ファミリーとして、イオノトロピック型嗅覚受容体ファミリーが発見された[38]。構成遺伝子数は嗅覚受容体と同様、種により異なり、10〜100程度存在し、ショウジョウバエでは66遺伝子存在する。イオノトロピック型嗅覚受容体は、匂い物質の他に味物質、湿気、温度も感知する。
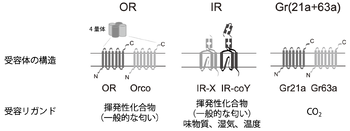
上記嗅覚受容体, イオノトロピック型嗅覚受容体ファミリータンパク質以外に、味覚受容体(gustatory receptor, GR)ファミリータンパク質のメンバー、Gr21a、Gr63aが嗅神経細胞に発現し、CO2を匂い物質として受容することが明らかになっている[39][40](図2)。
構造
昆虫嗅覚受容体
脊椎動物嗅覚受容体と同様、7回膜貫通構造を有するが、その膜トポロジーは逆であり、N末端が細胞質に、C末端が細胞外領域に位置する[41][42](図2)。脊椎動物嗅覚受容体と異なり、Gタンパク質共役型受容体との相同性はない。全般的に種間での配列保存性は低いが、唯一、種を超えて保存性の高い共通の嗅覚受容体が存在し、olfactory receptor co-receptor (Orco)と呼ばれる。Olfactory receptor co-receptorは、リガンド選択性を有する嗅覚受容体とヘテロ多量体を形成して機能すると考えられている。
近年、クライオ電子顕微鏡解析により、イチジク寄生バチの一種、Apocrypta bakerのolfactory receptor co-receptor、および、イシノミ類の昆虫Machilis hrabeiの嗅覚受容体, MhOR5について、立体構造が明らかになった[43][44]。Olfactory receptor co-receptorは単独ではホモ4量体構造を形成することが示され、チャネルの開閉制御に重要な領域が明らかになった[43]。MhOR5については、s種類の匂いリガンドとの共構造からリガンド結合によるチャネルの構造変化が示されるとともに、単一の受容体が多様な構造のリガンドを認識し得る構造基盤として、リガンド受容が複数の疎水的相互作用に基づくことも示された[44]。
イオノトロピック型嗅覚受容体
イオンチャネル型グルタミン酸受容体 (iGluR)と相同性が高く、3回膜貫通構造を持つ。イオノトロピック型受容体においてもリガンド選択性を有するIR-Xと、olfactory receptor co-receptor同様、リガンドに関わらず共通なIR-coY(ショウジョウバエでは、IR8a, IR25a, IR76b)が存在する。チャネルとしての機能ユニットは、2つのIR-Xと2つのIR-coYから構成されるヘテロ4量体と考えられている[45][46]。IR-coYはアミノ末端ドメイン(amino-terminal domain, ATD), リガンド結合ドメイン(ligand-binding domain, LBD)、イオンチャネルドメインから構成され、iGluRと高度な保存性を有する一方、IR-XはATDを持たず、iGluRとの相同性が低く、特にLBDの保存度が低い。イオノトロピック型受容体の立体構造は明らかになっていない。
味覚受容体
嗅覚受容体と同様、7回膜貫通構造を持ち、N末端が細胞質側、C末端が細胞外側のトポロジーを示す。Gr21a, Gr63aそのものの構造は示されていないが、他のGrファミリーメンバーである、カイコBmGr9のホモロジーモデリングと変異体解析において、GRも嗅覚受容体と同様のチャネル構造をもつことが示唆されている[47]。
発現部位
昆虫嗅覚受容体嗅覚受容体, イオノトロピック型嗅覚受容体は、嗅覚器である、触角(antenna)、小顎髭(maxillary palp)に発現する。嗅覚受容体として機能するGr21a, Gr63aも触角で発現する。これらの嗅覚器は感覚子とよばれる匂い物質を受容するための特殊な構造に覆われ、それぞれの感覚子には、1〜4の嗅神経細胞が格納されている。嗅覚受容体, IRはそれぞれ異なるタイプの感覚子に存在する嗅神経細胞樹状突起上に発現し、感覚子の穴から取り込まれた匂い物質を受容する。多くの場合、単一の嗅神経細胞は、リガンド選択性をもつ嗅覚受容体またはイオノトロピック型嗅覚受容体を1種類のみ発現するが、例外も報告されている[48]。
機能
昆虫嗅覚受容体
匂い物質をリガンドとするリガンド作動性非選択性陽イオンチャネルとして機能し、リガンド結合によりNa+, K+, Ca2+を透過させる[49][50]。脊椎動物の嗅覚受容体が、Gタンパク質を介したシグナルの活性化を通じて別分子であるイオンチャネルを開口させ、神経細胞に脱分極を引き起こすのに対し、昆虫嗅覚受容体は自身がイオンチャネルとしてはたらき、直接膜電位変化を引き起こせるため、匂い物質に対してより迅速な応答が可能となる。
認識するリガンドと嗅覚受容体の対応関係は、多くの場合、「多対多」を示すが[51]、特定の行動を引き起こす匂い物質やフェロモンに対しては、選択的に応答する嗅覚受容体が存在する。食物の有害性の指標としてショウジョウバエの忌避行動を引き起こすカビ臭、geosminにOr56a [52], 産卵を促進するシトラス系の果皮の香りにOr19a[53]、カイコガの性フェロモン、bombykolとbombykalにそれぞれBmOR1, BmOR3 [54][55]、ショウジョウバエの性フェロモン11-cis-vaccenyl acetate (cVA)にOr67d[56]が選択的に応答する。
昆虫嗅覚受容体はリガンド非存在下でも自発的なチャネル開口活性をもち[49][50]、嗅覚受容体発現嗅神経細胞の自発発火に寄与する。匂い物質には嗅神経細胞の活性化をもたらすもの以外に、自発発火を抑制するものも多数存在する[57]。
イオノトロピック型嗅覚受容体
匂い物質をリガンドとするリガンド作動性イオンチャネルとして機能し、Na+, K+, Ca2+を透過させる非選択性陽イオンチャネルを構成する[45]。匂い物質のうち、主に酸、アミン、アルデヒドを受容する点で、エステルやアルコールを中心に受容する嗅覚受容体と相補的なはたらきをすると考えられている[58]。嗅覚受容体と同様、リガンド認識は「多対多」が基本であるが、選択的な認識が特定の行動に結びつく場合もあり、ショウジョウバエIr92aによるアミンやアンモニアの受容が誘引行動に、Ir84aによる食物由来の匂いの受容が雄のショウジョウバエの交尾行動促進に繋がる報告例がある[59][60]。イオノトロピック型嗅覚受容体発現神経細胞は、昆虫嗅覚受容体発現神経細胞に比べ、活性化に、より高濃度のリガンドあるいは、長時間のリガンド刺激が必要であり、順応がおきにくい[61]。
関連項目
参考文献
- ↑
AMOORE, J.E. (1963).
Stereochemical theory of olfaction. Nature, 198, 271-2. [PubMed:14012641] [WorldCat] [DOI] - ↑
Buck, L., & Axel, R. (1991).
A novel multigene family may encode odorant receptors: a molecular basis for odor recognition. Cell, 65(1), 175-87. [PubMed:1840504] [WorldCat] [DOI] - ↑
Touhara, K., Sengoku, S., Inaki, K., Tsuboi, A., Hirono, J., Sato, T., ..., & Haga, T. (1999).
Functional identification and reconstitution of an odorant receptor in single olfactory neurons. Proceedings of the National Academy of Sciences of the United States of America, 96(7), 4040-5. [PubMed:10097159] [PMC] [WorldCat] [DOI] - ↑
Zhao, H., Ivic, L., Otaki, J.M., Hashimoto, M., Mikoshiba, K., & Firestein, S. (1998).
Functional expression of a mammalian odorant receptor. Science (New York, N.Y.), 279(5348), 237-42. [PubMed:9422698] [WorldCat] [DOI] - ↑
Niimura, Y., Matsui, A., & Touhara, K. (2014).
Extreme expansion of the olfactory receptor gene repertoire in African elephants and evolutionary dynamics of orthologous gene groups in 13 placental mammals. Genome research, 24(9), 1485-96. [PubMed:25053675] [PMC] [WorldCat] [DOI] - ↑
Li, B., Kamarck, M.L., Peng, Q., Lim, F.L., Keller, A., Smeets, M.A.M., ..., & Wang, S. (2022).
From musk to body odor: Decoding olfaction through genetic variation. PLoS genetics, 18(2), e1009564. [PubMed:35113854] [PMC] [WorldCat] [DOI] - ↑ Niimura Y, Ihara S, Touhara K (2020).
3.25 - Mammalian Olfactory and Vomeronasal Receptor Families. In The Senses: A Comprehensive Reference (Second Edition). Edited by Fritzsch B: Elsevier; pp 516-535. - ↑
Sato-Akuhara, N., Trimmer, C., Keller, A., Niimura, Y., Shirasu, M., Mainland, J.D., & Touhara, K. (2023).
Genetic variation in the human olfactory receptor OR5AN1 associates with the perception of musks. Chemical senses, 48. [PubMed:36625229] [PMC] [WorldCat] [DOI] - ↑
Trimmer, C., Keller, A., Murphy, N.R., Snyder, L.L., Willer, J.R., Nagai, M.H., ..., & Mainland, J.D. (2019).
Genetic variation across the human olfactory receptor repertoire alters odor perception. Proceedings of the National Academy of Sciences of the United States of America, 116(19), 9475-9480. [PubMed:31040214] [PMC] [WorldCat] [DOI] - ↑
Liberles, S.D., & Buck, L.B. (2006).
A second class of chemosensory receptors in the olfactory epithelium. Nature, 442(7103), 645-50. [PubMed:16878137] [WorldCat] [DOI] - ↑ 11.0 11.1
Hu, J., Zhong, C., Ding, C., Chi, Q., Walz, A., Mombaerts, P., ..., & Luo, M. (2007).
Detection of near-atmospheric concentrations of CO2 by an olfactory subsystem in the mouse. Science (New York, N.Y.), 317(5840), 953-7. [PubMed:17702944] [WorldCat] [DOI] - ↑ 12.0 12.1
Munger, S.D., Leinders-Zufall, T., McDougall, L.M., Cockerham, R.E., Schmid, A., Wandernoth, P., ..., & Kelliher, K.R. (2010).
An olfactory subsystem that detects carbon disulfide and mediates food-related social learning. Current biology : CB, 20(16), 1438-44. [PubMed:20637621] [PMC] [WorldCat] [DOI] - ↑ 13.0 13.1 13.2
Greer, P.L., Bear, D.M., Lassance, J.M., Bloom, M.L., Tsukahara, T., Pashkovski, S.L., ..., & Datta, S.R. (2016).
A Family of non-GPCR Chemosensors Defines an Alternative Logic for Mammalian Olfaction. Cell, 165(7), 1734-1748. [PubMed:27238024] [PMC] [WorldCat] [DOI] - ↑
Niimura, Y., & Nei, M. (2007).
Extensive gains and losses of olfactory receptor genes in mammalian evolution. PloS one, 2(8), e708. [PubMed:17684554] [PMC] [WorldCat] [DOI] - ↑
Freitag, J., Ludwig, G., Andreini, I., Rössler, P., & Breer, H. (1998).
Olfactory receptors in aquatic and terrestrial vertebrates. Journal of comparative physiology. A, Sensory, neural, and behavioral physiology, 183(5), 635-50. [PubMed:9839455] [DOI] - ↑
Billesbølle, C.B., de March, C.A., van der Velden, W.J.C., Ma, N., Tewari, J., Del Torrent, C.L., ..., & Manglik, A. (2023).
Structural basis of odorant recognition by a human odorant receptor. Nature, 615(7953), 742-749. [PubMed:36922591] [PMC] [WorldCat] [DOI] - ↑
Ahmed, L., Zhang, Y., Block, E., Buehl, M., Corr, M.J., Cormanich, R.A., ..., & Zhuang, H. (2018).
Molecular mechanism of activation of human musk receptors OR5AN1 and OR1A1 by (R)-muscone and diverse other musk-smelling compounds. Proceedings of the National Academy of Sciences of the United States of America, 115(17), E3950-E3958. [PubMed:29632183] [PMC] [WorldCat] [DOI] - ↑
Bushdid, C., de March, C.A., Fiorucci, S., Matsunami, H., & Golebiowski, J. (2018).
Agonists of G-Protein-Coupled Odorant Receptors Are Predicted from Chemical Features. The journal of physical chemistry letters, 9(9), 2235-2240. [PubMed:29648835] [PMC] [WorldCat] [DOI] - ↑
Geithe, C., Protze, J., Kreuchwig, F., Krause, G., & Krautwurst, D. (2017).
Structural determinants of a conserved enantiomer-selective carvone binding pocket in the human odorant receptor OR1A1. Cellular and molecular life sciences : CMLS, 74(22), 4209-4229. [PubMed:28656349] [WorldCat] [DOI] - ↑
Katada, S., Hirokawa, T., Oka, Y., Suwa, M., & Touhara, K. (2005).
Structural basis for a broad but selective ligand spectrum of a mouse olfactory receptor: mapping the odorant-binding site. The Journal of neuroscience : the official journal of the Society for Neuroscience, 25(7), 1806-15. [PubMed:15716417] [PMC] [WorldCat] [DOI] - ↑ 21.0 21.1
Liberles, S.D. (2015).
Trace amine-associated receptors: ligands, neural circuits, and behaviors. Current opinion in neurobiology, 34, 1-7. [PubMed:25616211] [PMC] [WorldCat] [DOI] - ↑
Ruiz Tejada Segura, M.L., Abou Moussa, E., Garabello, E., Nakahara, T.S., Makhlouf, M., Mathew, L.S., ..., & Saraiva, L.R. (2022).
A 3D transcriptomics atlas of the mouse nose sheds light on the anatomical logic of smell. Cell reports, 38(12), 110547. [PubMed:35320714] [PMC] [WorldCat] [DOI] - ↑
Flegel, C., Manteniotis, S., Osthold, S., Hatt, H., & Gisselmann, G. (2013).
Expression profile of ectopic olfactory receptors determined by deep sequencing. PloS one, 8(2), e55368. [PubMed:23405139] [PMC] [WorldCat] [DOI] - ↑
Fukuda, N., Yomogida, K., Okabe, M., & Touhara, K. (2004).
Functional characterization of a mouse testicular olfactory receptor and its role in chemosensing and in regulation of sperm motility. Journal of cell science, 117(Pt 24), 5835-45. [PubMed:15522887] [WorldCat] [DOI] - ↑
Spehr, M., Kelliher, K.R., Li, X.H., Boehm, T., Leinders-Zufall, T., & Zufall, F. (2006).
Essential role of the main olfactory system in social recognition of major histocompatibility complex peptide ligands. The Journal of neuroscience : the official journal of the Society for Neuroscience, 26(7), 1961-70. [PubMed:16481428] [PMC] [WorldCat] [DOI] - ↑
Griffin, C.A., Kafadar, K.A., & Pavlath, G.K. (2009).
MOR23 promotes muscle regeneration and regulates cell adhesion and migration. Developmental cell, 17(5), 649-61. [PubMed:19922870] [PMC] [WorldCat] [DOI] - ↑
Orecchioni, M., Kobiyama, K., Winkels, H., Ghosheh, Y., McArdle, S., Mikulski, Z., ..., & Ley, K. (2022).
Olfactory receptor 2 in vascular macrophages drives atherosclerosis by NLRP3-dependent IL-1 production. Science (New York, N.Y.), 375(6577), 214-221. [PubMed:35025664] [PMC] [WorldCat] [DOI] - ↑
Cheng, J., Yang, Z., Ge, X.Y., Gao, M.X., Meng, R., Xu, X., ..., & Yu, X. (2022).
Autonomous sensing of the insulin peptide by an olfactory G protein-coupled receptor modulates glucose metabolism. Cell metabolism, 34(2), 240-255.e10. [PubMed:35108512] [WorldCat] [DOI] - ↑
Li, E., Shan, H., Chen, L., Long, A., Zhang, Y., Liu, Y., ..., & Wang, Y. (2019).
OLFR734 Mediates Glucose Metabolism as a Receptor of Asprosin. Cell metabolism, 30(2), 319-328.e8. [PubMed:31230984] [WorldCat] [DOI] - ↑
Wu, C., Hwang, S.H., Jia, Y., Choi, J., Kim, Y.J., Choi, D., ..., & Lee, S.J. (2017).
Olfactory receptor 544 reduces adiposity by steering fuel preference toward fats. The Journal of clinical investigation, 127(11), 4118-4123. [PubMed:28990936] [PMC] [WorldCat] [DOI] - ↑ 31.0 31.1
Lee, S.J., Depoortere, I., & Hatt, H. (2019).
Therapeutic potential of ectopic olfactory and taste receptors. Nature reviews. Drug discovery, 18(2), 116-138. [PubMed:30504792] [WorldCat] [DOI] - ↑
Malnic, B., Hirono, J., Sato, T., & Buck, L.B. (1999).
Combinatorial receptor codes for odors. Cell, 96(5), 713-23. [PubMed:10089886] [WorldCat] [DOI] - ↑
Dewan, A. (2021).
Olfactory signaling via trace amine-associated receptors. Cell and tissue research, 383(1), 395-407. [PubMed:33237477] [PMC] [WorldCat] [DOI] - ↑
Li, M., Schweiger, M.W., Ryan, D.J., Nakano, I., Carvalho, L.A., & Tannous, B.A. (2021).
Olfactory receptor 5B21 drives breast cancer metastasis. iScience, 24(12), 103519. [PubMed:34917897] [PMC] [WorldCat] [DOI] - ↑
Clyne, P.J., Warr, C.G., Freeman, M.R., Lessing, D., Kim, J., & Carlson, J.R. (1999).
A novel family of divergent seven-transmembrane proteins: candidate odorant receptors in Drosophila. Neuron, 22(2), 327-38. [PubMed:10069338] [WorldCat] [DOI] - ↑
Gao, Q., & Chess, A. (1999).
Identification of candidate Drosophila olfactory receptors from genomic DNA sequence. Genomics, 60(1), 31-9. [PubMed:10458908] [WorldCat] [DOI] - ↑
Vosshall, L.B., Amrein, H., Morozov, P.S., Rzhetsky, A., & Axel, R. (1999).
A spatial map of olfactory receptor expression in the Drosophila antenna. Cell, 96(5), 725-36. [PubMed:10089887] [WorldCat] [DOI] - ↑
Benton, R., Vannice, K.S., Gomez-Diaz, C., & Vosshall, L.B. (2009).
Variant ionotropic glutamate receptors as chemosensory receptors in Drosophila. Cell, 136(1), 149-62. [PubMed:19135896] [PMC] [WorldCat] [DOI] - ↑
Jones, W.D., Cayirlioglu, P., Kadow, I.G., & Vosshall, L.B. (2007).
Two chemosensory receptors together mediate carbon dioxide detection in Drosophila. Nature, 445(7123), 86-90. [PubMed:17167414] [WorldCat] [DOI] - ↑
Kwon, J.Y., Dahanukar, A., Weiss, L.A., & Carlson, J.R. (2007).
The molecular basis of CO2 reception in Drosophila. Proceedings of the National Academy of Sciences of the United States of America, 104(9), 3574-8. [PubMed:17360684] [PMC] [WorldCat] [DOI] - ↑
Benton, R., Sachse, S., Michnick, S.W., & Vosshall, L.B. (2006).
Atypical membrane topology and heteromeric function of Drosophila odorant receptors in vivo. PLoS biology, 4(2), e20. [PubMed:16402857] [PMC] [WorldCat] [DOI] - ↑
Hopf, T.A., Morinaga, S., Ihara, S., Touhara, K., Marks, D.S., & Benton, R. (2015).
Amino acid coevolution reveals three-dimensional structure and functional domains of insect odorant receptors. Nature communications, 6, 6077. [PubMed:25584517] [PMC] [WorldCat] [DOI] - ↑ 43.0 43.1
Butterwick, J.A., Del Mármol, J., Kim, K.H., Kahlson, M.A., Rogow, J.A., Walz, T., & Ruta, V. (2018).
Cryo-EM structure of the insect olfactory receptor Orco. Nature, 560(7719), 447-452. [PubMed:30111839] [PMC] [WorldCat] [DOI] - ↑ 44.0 44.1
Del Mármol, J., Yedlin, M.A., & Ruta, V. (2021).
The structural basis of odorant recognition in insect olfactory receptors. Nature, 597(7874), 126-131. [PubMed:34349260] [PMC] [WorldCat] [DOI] - ↑ 45.0 45.1
Abuin, L., Bargeton, B., Ulbrich, M.H., Isacoff, E.Y., Kellenberger, S., & Benton, R. (2011).
Functional architecture of olfactory ionotropic glutamate receptors. Neuron, 69(1), 44-60. [PubMed:21220098] [PMC] [WorldCat] [DOI] - ↑
Abuin, L., Prieto-Godino, L.L., Pan, H., Gutierrez, C., Huang, L., Jin, R., & Benton, R. (2019).
In vivo assembly and trafficking of olfactory Ionotropic Receptors. BMC biology, 17(1), 34. [PubMed:30995910] [PMC] [WorldCat] [DOI] - ↑
Morinaga, S., Nagata, K., Ihara, S., Yumita, T., Niimura, Y., Sato, K., & Touhara, K. (2022).
Structural model for ligand binding and channel opening of an insect gustatory receptor. The Journal of biological chemistry, 298(11), 102573. [PubMed:36209821] [PMC] [WorldCat] [DOI] - ↑
Herre, M., Goldman, O.V., Lu, T.C., Caballero-Vidal, G., Qi, Y., Gilbert, Z.N., ..., & Younger, M.A. (2022).
Non-canonical odor coding in the mosquito. Cell, 185(17), 3104-3123.e28. [PubMed:35985288] [PMC] [WorldCat] [DOI] - ↑ 49.0 49.1
Sato, K., Pellegrino, M., Nakagawa, T., Nakagawa, T., Vosshall, L.B., & Touhara, K. (2008).
Insect olfactory receptors are heteromeric ligand-gated ion channels. Nature, 452(7190), 1002-6. [PubMed:18408712] [WorldCat] [DOI] - ↑ 50.0 50.1
Wicher, D., Schäfer, R., Bauernfeind, R., Stensmyr, M.C., Heller, R., Heinemann, S.H., & Hansson, B.S. (2008).
Drosophila odorant receptors are both ligand-gated and cyclic-nucleotide-activated cation channels. Nature, 452(7190), 1007-11. [PubMed:18408711] [WorldCat] [DOI] - ↑
Hallem, E.A., Ho, M.G., & Carlson, J.R. (2004).
The molecular basis of odor coding in the Drosophila antenna. Cell, 117(7), 965-79. [PubMed:15210116] [WorldCat] [DOI] - ↑
Stensmyr, M.C., Dweck, H.K., Farhan, A., Ibba, I., Strutz, A., Mukunda, L., ..., & Hansson, B.S. (2012).
A conserved dedicated olfactory circuit for detecting harmful microbes in Drosophila. Cell, 151(6), 1345-57. [PubMed:23217715] [WorldCat] [DOI] - ↑
Dweck, H.K., Ebrahim, S.A., Kromann, S., Bown, D., Hillbur, Y., Sachse, S., ..., & Stensmyr, M.C. (2013).
Olfactory preference for egg laying on citrus substrates in Drosophila. Current biology : CB, 23(24), 2472-80. [PubMed:24316206] [WorldCat] [DOI] - ↑
Nakagawa, T., Sakurai, T., Nishioka, T., & Touhara, K. (2005).
Insect sex-pheromone signals mediated by specific combinations of olfactory receptors. Science (New York, N.Y.), 307(5715), 1638-42. [PubMed:15692016] [WorldCat] [DOI] - ↑
Sakurai, T., Nakagawa, T., Mitsuno, H., Mori, H., Endo, Y., Tanoue, S., ..., & Nishioka, T. (2004).
Identification and functional characterization of a sex pheromone receptor in the silkmoth Bombyx mori. Proceedings of the National Academy of Sciences of the United States of America, 101(47), 16653-8. [PubMed:15545611] [PMC] [WorldCat] [DOI] - ↑
Kurtovic, A., Widmer, A., & Dickson, B.J. (2007).
A single class of olfactory neurons mediates behavioural responses to a Drosophila sex pheromone. Nature, 446(7135), 542-6. [PubMed:17392786] [WorldCat] [DOI] - ↑
Hallem, E.A., & Carlson, J.R. (2006).
Coding of odors by a receptor repertoire. Cell, 125(1), 143-60. [PubMed:16615896] [WorldCat] [DOI] - ↑
Silbering, A.F., Rytz, R., Grosjean, Y., Abuin, L., Ramdya, P., Jefferis, G.S., & Benton, R. (2011).
Complementary function and integrated wiring of the evolutionarily distinct Drosophila olfactory subsystems. The Journal of neuroscience : the official journal of the Society for Neuroscience, 31(38), 13357-75. [PubMed:21940430] [PMC] [WorldCat] [DOI] - ↑
Grosjean, Y., Rytz, R., Farine, J.P., Abuin, L., Cortot, J., Jefferis, G.S., & Benton, R. (2011).
An olfactory receptor for food-derived odours promotes male courtship in Drosophila. Nature, 478(7368), 236-40. [PubMed:21964331] [WorldCat] [DOI] - ↑
Min, S., Ai, M., Shin, S.A., & Suh, G.S. (2013).
Dedicated olfactory neurons mediating attraction behavior to ammonia and amines in Drosophila. Proceedings of the National Academy of Sciences of the United States of America, 110(14), E1321-9. [PubMed:23509267] [PMC] [WorldCat] [DOI] - ↑
Getahun, M.N., Wicher, D., Hansson, B.S., & Olsson, S.B. (2012).
Temporal response dynamics of Drosophila olfactory sensory neurons depends on receptor type and response polarity. Frontiers in cellular neuroscience, 6, 54. [PubMed:23162431] [PMC] [WorldCat] [DOI]