「Shank」の版間の差分
Marikohayashi (トーク | 投稿記録) 細編集の要約なし |
細 (→受容体・膜タンパク質) |
||
| (4人の利用者による、間の60版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | |||
<font size="+1">林 真理子</font><br> | |||
''慶應義塾大学 医学部''<br> | |||
DOI:<selfdoi /> 原稿受付日:2012年5月28日 原稿完成日:2012年6月6日<br> | |||
担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | |||
</div> | |||
{{PBB|geneid=50944}} {{PBB|geneid=22941}} {{PBB|geneid=85358}} | |||
英:Shank (SH3 and multiple ankyrin repeat domains protein) | |||
同義語:ProSAP (Proline-rich synapse-associated protein), CortBP (Cortactin-binding protein), Somatostatin receptor-interacting protein (SSTRIP), GKAP/SAPAP-interacting protein, SPANK, Synamon | |||
{{box|text= | |||
Shankは、2000個以上の[[wikipedia:ja:アミノ酸|アミノ酸]]からなる巨大な[[足場タンパク質]]である。[[wikipedia:ja:選択的スプライシング|選択的スプライシング]]によりさまざまな遺伝子産物が得られるが、最も長いものは[[アンキリンリピート]]、[[SH3ドメイン|SH3 (Src homology 3)ドメイン]]、[[PDZドメイン|PDZ (<u>P</u>SD-95, <u>D</u>lg, <u>Z</u>o-1)ドメイン]]、プロリンリッチ配列、[[SAMドメイン|SAM (Sterile alpha motif)ドメイン]]からなる。それぞれのドメインが相互作用するタンパク質を持つので、結合タンパク質は多岐にわたる。[[自閉症]]との関連が指摘されている。 | |||
}} | |||
== 構造 == | |||
Shankには異なった遺伝子にコードされるShank1、2、3がある。Shankのドメイン構造はアミノ端から、アンキリンリピート、SH3ドメイン、PDZドメイン、1000残基以上に及ぶプロリン、セリン、グリシンに富む配列、SAMからなる(図1)。選択的スプライシングにより、アンキリンリピートやSH3ドメイン、SAMドメインを欠くものもある。 | |||
サブタイプ毎の別名は、 | |||
*Shank1 = GKAP/SAPAP-interacting protein = SPANK-1 = Somatostatin receptor-interacting protein (SSTRIP) = Synamon | |||
*Shank2 = CortBP1 (Cortactin-binding protein 1) = ProSAP1 (Proline-rich synapse-associated protein 1) = SPANK-3 | |||
*Shank3 = ProSAP2 = SPANK-2 | |||
[[Image:Shank.png|thumb|center|250px|<b>図1.Shank ドメイン構造と選択的スプライシング産物</b><br />Ank:アンキリンリピート<br />SH3:SH3ドメイン<br />PDZ:PDZドメイン<br />Pro rich:プロリンリッチ配列<br />SAM:SAMドメイン]] | |||
== 発現 == | |||
Shank1, [http://mouse.brain-map.org/gene/show/84192 Shank2], [http://mouse.brain-map.org/gene/show/37264 Shank3]いずれのサブタイプも脳に広範に発現している。細胞レベルでは、[[シナプス後部]]、特に[[シナプス後肥厚部]]に多く分布している。 | |||
< | 脳以外では、Shank2は[[wikipedia:ja:腎臓|腎臓]]、[[wikipedia:ja:肝臓|肝臓]]に、Shank3は[[wikipedia:ja:心臓|心臓]]、[[wikipedia:ja:精巣|精巣]]にも発現している<ref><pubmed>10506216</pubmed></ref> 。 | ||
== 機能 == | |||
< | Shankを神経細胞に過剰発現すると[[スパイン]]の肥大化が起こり、特にShank結合タンパク質である[[Homer]]との共発現はスパインのを更なる肥大化を引き起こす<ref><pubmed>11498055</pubmed></ref>。 さらに、本来、スパインを持たない[[小脳]][[顆粒細胞]]にShankを導入すると、 [[NMDA型グルタミン酸受容体]]や[[AMPA型グルタミン酸受容体]]をもつスパインを形成するようになる<ref><pubmed>15814786</pubmed></ref>。 | ||
Shank分子間のドメイン間相互作用によりオリゴマーを形成するShankと、両端に2つずつのリガンド結合部位をもつ逆平行4量体を形成するHomerは互いに架橋して、高次のネットワーク構造を形成する。このShankとHomerの高次複合体がシナプス後肥厚部の骨格となると考えられる<ref><pubmed>19345194</pubmed></ref> 。 | |||
== | == 分子内・分子間相互作用 == | ||
= | Shank分子内、あるいはShank分子間の相互作用としては、アンキリンリピートとSH3ドメインが相互作用する<ref><pubmed>15496675</pubmed></ref>ほか、PDZドメインはホモ二量体を<ref name="ref2"><pubmed>12954649</pubmed></ref>、SAMドメインは多量体を<ref name="ref3"><pubmed>16439662</pubmed></ref> 形成する。このPDZドメインによるホモ二量体形成には、PDZドメイン本来のタンパク質結合部位は関与しないので、二量体を形成しても、他のPDZリガンドは結合できる(図2)。一方、結晶化されたSAMドメインは一周6分子の螺旋状ポリマーを形成しており(図3)、更にこの螺旋が側面で会合して、Zn<sup>2+</sup>イオンに依存性の二次元の広がりをもつシートを形成する。 | ||
但し、SAMドメインの大きさはShank全長の3%にしか相当しないので、上流の長い配列も含めて大きなポリマーを形成できるかどうかは不明である。 <gallery widths="250px" heights="250px"> | |||
Image:1Q3P.jpg|'''図2 Shank PDZ ドメインによるダイマー形成とGKAPとの相互作用 PDB 1q3p'''<ref name="ref2" /> | |||
Image:Shank-SAM 2F44.png|'''図3 Shank SAM ドメインの結晶構造 PDB 2f44'''<ref name="ref3" /> | |||
</gallery> | |||
== | == Shankと相互作用するタンパク質 == | ||
[[Image:3L4F.jpg|thumb|250px|図4 Shank PDZ ドメインとβPIXとの相互作用 PDB 3l4f <ref><pubmed>20117114</pubmed></ref> ]] | |||
== | === 受容体・膜タンパク質 === | ||
いずれも、C末端(C-terminus)がShankのPDZドメインに結合する。 | |||
*[[G protein-coupled α-latrotoxin receptor]] CL1<ref><pubmed>10958799</pubmed></ref> | |||
<br>< | *[[ソマトスタチン受容体]]サブタイプ2<ref><pubmed>10551867</pubmed></ref> | ||
*[[Na+/H+交換輸送体|Na<sup>+</sup>/H<sup>+</sup>交換輸送体]] 3<ref><pubmed>16293618</pubmed></ref> | |||
*[[嚢胞性線維症膜コンダクタンス制御因子]] (Cystic fibrosis transmembrane conductance regulator, CFTR)<ref><pubmed>14679199</pubmed></ref> | |||
*[[AMPA型グルタミン酸受容体]][[GluA1]]サブユニット<ref><pubmed>16606358</pubmed></ref> | |||
*[[代謝活性型グルタミン酸受容体]]サブユニット[[MGluR1|mGluR1]]、[[MGluR5|mGluR5]]<ref><pubmed>10433269</pubmed></ref> これらは、Shankに結合するHomerとも相互作用する。 | |||
*[[デルタ2型グルタミン酸受容体]][[GluD2]]<ref><pubmed>15207857</pubmed></ref> | |||
=== シナプス足場タンパク質 === | |||
*[[DLGAP]]1/GKAP<ref><pubmed>10488079</pubmed></ref> | |||
*Homer<ref><pubmed>10433269</pubmed></ref> | |||
*[[IRSp53]]<ref><pubmed>12504591</pubmed></ref> | |||
*[[Sharpin]]<ref><pubmed>11178875</pubmed></ref> | |||
=== 低分子量GTP結合タンパク質を制御するタンパク質 === | |||
*IRSp53 | |||
*[[ΒPIX|ΒetaPIX]]<ref><pubmed>12626503</pubmed></ref> | |||
*[[ProSAPiP1]]<ref><pubmed>16522626</pubmed></ref> | |||
=== [[アクチン]]結合タンパク質 === | |||
*IRSp53 | |||
*[[コータクチン]]<ref><pubmed>10433268</pubmed></ref> <ref><pubmed>9742101</pubmed></ref> | |||
*[[Abp1]]<ref><pubmed>15014124</pubmed></ref> | |||
*[[スペクトリンα]]<ref><pubmed>11509555</pubmed></ref> | |||
=== その他のタンパク質 === | |||
*[[ホスホリパーゼC]]β3<ref><pubmed>15632121</pubmed></ref> | |||
*[[ダイナミン]]2<ref><pubmed>11583995</pubmed></ref><br> | |||
*[[Dendrite arborization and synapse maturation 1]] (Dasm1)<ref><pubmed>15340156</pubmed></ref> | |||
== Shank遺伝子の異常 == | |||
自閉症との関連については、ヒトにおけるShank3の変異が最初に報告されたが<ref><pubmed>17173049</pubmed></ref>、その後、Shank2<ref><pubmed>20531469</pubmed></ref>, Shank1<ref><pubmed>22503632</pubmed></ref>についても報告されている。[[Phelan–McDermid 症候群]]は染色体22q13部位の欠失によるもので、Shank3 の異常が原因の一つと考えられている。また、Shank2、Shank3の[[ノックアウトマウス]]は社会的相互作用の欠如や過剰な毛繕いなど自閉症様の表現型を呈する<ref><pubmed>21423165</pubmed></ref> <ref><pubmed>22699620</pubmed></ref> <ref><pubmed>22699619</pubmed></ref>。さらに、自閉症患者にみられるShank3のC末の欠失変異体は、優性にShank3の[[プロテアーゼ]]分解を促進して、シナプスの形成を阻害する<ref><pubmed>21565394</pubmed></ref>。 | |||
== 関連項目 == | |||
*[[シナプス後肥厚部]] | |||
*[[自閉症]] | |||
== 参考文献 == | |||
<references /> | |||
2018年11月17日 (土) 15:32時点における最新版
林 真理子
慶應義塾大学 医学部
DOI:10.14931/bsd.1089 原稿受付日:2012年5月28日 原稿完成日:2012年6月6日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
| SH3 and multiple ankyrin repeat domains 2 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | SHANK2; AUTS17; CORTBP1; CTTNBP1; ProSAP1; SHANK; SPANK-3 | ||||||||||||
| External IDs | OMIM: 603290 MGI: 2671987 HomoloGene: 105965 GeneCards: SHANK2 Gene | ||||||||||||
| |||||||||||||
| RNA expression pattern | |||||||||||||
 | |||||||||||||
 | |||||||||||||
 | |||||||||||||
| More reference expression data | |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 22941 | 210274 | |||||||||||
| Ensembl | ENSG00000162105 | ENSMUSG00000037541 | |||||||||||
| UniProt | Q9UPX8 | Q80Z38 | |||||||||||
| RefSeq (mRNA) | NM_012309.3 | XM_001002370 | |||||||||||
| RefSeq (protein) | NP_036441.2 | XP_001002370 | |||||||||||
| Location (UCSC) |
Chr 11: 70.31 – 70.96 Mb |
Chr 7: 143.99 – 144.23 Mb | |||||||||||
| PubMed search | [3] | [4] | |||||||||||
| SH3 and multiple ankyrin repeat domains 3 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | SHANK3; DEL22q13.3; PROSAP2; PSAP2; SCZD15; SPANK-2 | ||||||||||||
| External IDs | OMIM: 606230 MGI: 1930016 HomoloGene: 75163 GeneCards: SHANK3 Gene | ||||||||||||
| |||||||||||||
| RNA expression pattern | |||||||||||||
 | |||||||||||||
 | |||||||||||||
 | |||||||||||||
| More reference expression data | |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 85358 | 58234 | |||||||||||
| Ensembl | ENSG00000251322 | ENSMUSG00000022623 | |||||||||||
| UniProt | F2Z3L0 | Q4ACU6 | |||||||||||
| RefSeq (mRNA) | NM_001080420.1 | NM_021423.3 | |||||||||||
| RefSeq (protein) | NP_001073889.1 | NP_067398.2 | |||||||||||
| Location (UCSC) |
Chr 22: 51.11 – 51.17 Mb |
Chr 15: 89.33 – 89.39 Mb | |||||||||||
| PubMed search | [5] | [6] | |||||||||||
英:Shank (SH3 and multiple ankyrin repeat domains protein)
同義語:ProSAP (Proline-rich synapse-associated protein), CortBP (Cortactin-binding protein), Somatostatin receptor-interacting protein (SSTRIP), GKAP/SAPAP-interacting protein, SPANK, Synamon
Shankは、2000個以上のアミノ酸からなる巨大な足場タンパク質である。選択的スプライシングによりさまざまな遺伝子産物が得られるが、最も長いものはアンキリンリピート、SH3 (Src homology 3)ドメイン、PDZ (PSD-95, Dlg, Zo-1)ドメイン、プロリンリッチ配列、SAM (Sterile alpha motif)ドメインからなる。それぞれのドメインが相互作用するタンパク質を持つので、結合タンパク質は多岐にわたる。自閉症との関連が指摘されている。
構造
Shankには異なった遺伝子にコードされるShank1、2、3がある。Shankのドメイン構造はアミノ端から、アンキリンリピート、SH3ドメイン、PDZドメイン、1000残基以上に及ぶプロリン、セリン、グリシンに富む配列、SAMからなる(図1)。選択的スプライシングにより、アンキリンリピートやSH3ドメイン、SAMドメインを欠くものもある。
サブタイプ毎の別名は、
- Shank1 = GKAP/SAPAP-interacting protein = SPANK-1 = Somatostatin receptor-interacting protein (SSTRIP) = Synamon
- Shank2 = CortBP1 (Cortactin-binding protein 1) = ProSAP1 (Proline-rich synapse-associated protein 1) = SPANK-3
- Shank3 = ProSAP2 = SPANK-2
発現
Shank1, Shank2, Shank3いずれのサブタイプも脳に広範に発現している。細胞レベルでは、シナプス後部、特にシナプス後肥厚部に多く分布している。
脳以外では、Shank2は腎臓、肝臓に、Shank3は心臓、精巣にも発現している[1] 。
機能
Shankを神経細胞に過剰発現するとスパインの肥大化が起こり、特にShank結合タンパク質であるHomerとの共発現はスパインのを更なる肥大化を引き起こす[2]。 さらに、本来、スパインを持たない小脳顆粒細胞にShankを導入すると、 NMDA型グルタミン酸受容体やAMPA型グルタミン酸受容体をもつスパインを形成するようになる[3]。
Shank分子間のドメイン間相互作用によりオリゴマーを形成するShankと、両端に2つずつのリガンド結合部位をもつ逆平行4量体を形成するHomerは互いに架橋して、高次のネットワーク構造を形成する。このShankとHomerの高次複合体がシナプス後肥厚部の骨格となると考えられる[4] 。
分子内・分子間相互作用
Shank分子内、あるいはShank分子間の相互作用としては、アンキリンリピートとSH3ドメインが相互作用する[5]ほか、PDZドメインはホモ二量体を[6]、SAMドメインは多量体を[7] 形成する。このPDZドメインによるホモ二量体形成には、PDZドメイン本来のタンパク質結合部位は関与しないので、二量体を形成しても、他のPDZリガンドは結合できる(図2)。一方、結晶化されたSAMドメインは一周6分子の螺旋状ポリマーを形成しており(図3)、更にこの螺旋が側面で会合して、Zn2+イオンに依存性の二次元の広がりをもつシートを形成する。
但し、SAMドメインの大きさはShank全長の3%にしか相当しないので、上流の長い配列も含めて大きなポリマーを形成できるかどうかは不明である。
Shankと相互作用するタンパク質
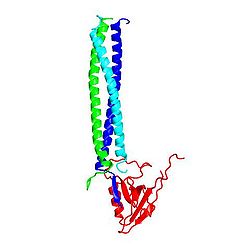
受容体・膜タンパク質
いずれも、C末端(C-terminus)がShankのPDZドメインに結合する。
- ソマトスタチン受容体サブタイプ2[10]
- 嚢胞性線維症膜コンダクタンス制御因子 (Cystic fibrosis transmembrane conductance regulator, CFTR)[12]
- AMPA型グルタミン酸受容体GluA1サブユニット[13]
- 代謝活性型グルタミン酸受容体サブユニットmGluR1、mGluR5[14] これらは、Shankに結合するHomerとも相互作用する。
シナプス足場タンパク質
- Homer[17]
低分子量GTP結合タンパク質を制御するタンパク質
- IRSp53
アクチン結合タンパク質
- IRSp53
その他のタンパク質
Shank遺伝子の異常
自閉症との関連については、ヒトにおけるShank3の変異が最初に報告されたが[29]、その後、Shank2[30], Shank1[31]についても報告されている。Phelan–McDermid 症候群は染色体22q13部位の欠失によるもので、Shank3 の異常が原因の一つと考えられている。また、Shank2、Shank3のノックアウトマウスは社会的相互作用の欠如や過剰な毛繕いなど自閉症様の表現型を呈する[32] [33] [34]。さらに、自閉症患者にみられるShank3のC末の欠失変異体は、優性にShank3のプロテアーゼ分解を促進して、シナプスの形成を阻害する[35]。
関連項目
参考文献
- ↑
Lim, S., Naisbitt, S., Yoon, J., Hwang, J.I., Suh, P.G., Sheng, M., & Kim, E. (1999).
Characterization of the Shank family of synaptic proteins. Multiple genes, alternative splicing, and differential expression in brain and development. The Journal of biological chemistry, 274(41), 29510-8. [PubMed:10506216] [WorldCat] [DOI] - ↑
Sala, C., Piëch, V., Wilson, N.R., Passafaro, M., Liu, G., & Sheng, M. (2001).
Regulation of dendritic spine morphology and synaptic function by Shank and Homer. Neuron, 31(1), 115-30. [PubMed:11498055] [WorldCat] [DOI] - ↑
Roussignol, G., Ango, F., Romorini, S., Tu, J.C., Sala, C., Worley, P.F., ..., & Fagni, L. (2005).
Shank expression is sufficient to induce functional dendritic spine synapses in aspiny neurons. The Journal of neuroscience : the official journal of the Society for Neuroscience, 25(14), 3560-70. [PubMed:15814786] [PMC] [WorldCat] [DOI] - ↑
Hayashi, M.K., Tang, C., Verpelli, C., Narayanan, R., Stearns, M.H., Xu, R.M., ..., & Hayashi, Y. (2009).
The postsynaptic density proteins Homer and Shank form a polymeric network structure. Cell, 137(1), 159-71. [PubMed:19345194] [PMC] [WorldCat] [DOI] - ↑
Romorini, S., Piccoli, G., Jiang, M., Grossano, P., Tonna, N., Passafaro, M., ..., & Sala, C. (2004).
A functional role of postsynaptic density-95-guanylate kinase-associated protein complex in regulating Shank assembly and stability to synapses. The Journal of neuroscience : the official journal of the Society for Neuroscience, 24(42), 9391-404. [PubMed:15496675] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Im, Y.J., Lee, J.H., Park, S.H., Park, S.J., Rho, S.H., Kang, G.B., ..., & Eom, S.H. (2003).
Crystal structure of the Shank PDZ-ligand complex reveals a class I PDZ interaction and a novel PDZ-PDZ dimerization. The Journal of biological chemistry, 278(48), 48099-104. [PubMed:12954649] [WorldCat] [DOI] - ↑ 7.0 7.1
Baron, M.K., Boeckers, T.M., Vaida, B., Faham, S., Gingery, M., Sawaya, M.R., ..., & Bowie, J.U. (2006).
An architectural framework that may lie at the core of the postsynaptic density. Science (New York, N.Y.), 311(5760), 531-5. [PubMed:16439662] [WorldCat] [DOI] - ↑
Im, Y.J., Kang, G.B., Lee, J.H., Park, K.R., Song, H.E., Kim, E., ..., & Eom, S.H. (2010).
Structural basis for asymmetric association of the betaPIX coiled coil and shank PDZ. Journal of molecular biology, 397(2), 457-66. [PubMed:20117114] [WorldCat] [DOI] - ↑
Tobaben, S., Südhof, T.C., & Stahl, B. (2000).
The G protein-coupled receptor CL1 interacts directly with proteins of the Shank family. The Journal of biological chemistry, 275(46), 36204-10. [PubMed:10958799] [WorldCat] [DOI] - ↑
Zitzer, H., Hönck, H.H., Bächner, D., Richter, D., & Kreienkamp, H.J. (1999).
Somatostatin receptor interacting protein defines a novel family of multidomain proteins present in human and rodent brain. The Journal of biological chemistry, 274(46), 32997-3001. [PubMed:10551867] [WorldCat] [DOI] - ↑
Han, W., Kim, K.H., Jo, M.J., Lee, J.H., Yang, J., Doctor, R.B., ..., & Lee, M.G. (2006).
Shank2 associates with and regulates Na+/H+ exchanger 3. The Journal of biological chemistry, 281(3), 1461-9. [PubMed:16293618] [WorldCat] [DOI] - ↑
Kim, J.Y., Han, W., Namkung, W., Lee, J.H., Kim, K.H., Shin, H., ..., & Lee, M.G. (2004).
Inhibitory regulation of cystic fibrosis transmembrane conductance regulator anion-transporting activities by Shank2. The Journal of biological chemistry, 279(11), 10389-96. [PubMed:14679199] [WorldCat] [DOI] - ↑
Uchino, S., Wada, H., Honda, S., Nakamura, Y., Ondo, Y., Uchiyama, T., ..., & Kohsaka, S. (2006).
Direct interaction of post-synaptic density-95/Dlg/ZO-1 domain-containing synaptic molecule Shank3 with GluR1 alpha-amino-3-hydroxy-5-methyl-4-isoxazole propionic acid receptor. Journal of neurochemistry, 97(4), 1203-14. [PubMed:16606358] [WorldCat] [DOI] - ↑
Tu, J.C., Xiao, B., Naisbitt, S., Yuan, J.P., Petralia, R.S., Brakeman, P., ..., & Worley, P.F. (1999).
Coupling of mGluR/Homer and PSD-95 complexes by the Shank family of postsynaptic density proteins. Neuron, 23(3), 583-92. [PubMed:10433269] [WorldCat] [DOI] - ↑
Uemura, T., Mori, H., & Mishina, M. (2004).
Direct interaction of GluRdelta2 with Shank scaffold proteins in cerebellar Purkinje cells. Molecular and cellular neurosciences, 26(2), 330-41. [PubMed:15207857] [WorldCat] [DOI] - ↑
Yao, I., Hata, Y., Hirao, K., Deguchi, M., Ide, N., Takeuchi, M., & Takai, Y. (1999).
Synamon, a novel neuronal protein interacting with synapse-associated protein 90/postsynaptic density-95-associated protein. The Journal of biological chemistry, 274(39), 27463-6. [PubMed:10488079] [WorldCat] [DOI] - ↑
Tu, J.C., Xiao, B., Naisbitt, S., Yuan, J.P., Petralia, R.S., Brakeman, P., ..., & Worley, P.F. (1999).
Coupling of mGluR/Homer and PSD-95 complexes by the Shank family of postsynaptic density proteins. Neuron, 23(3), 583-92. [PubMed:10433269] [WorldCat] [DOI] - ↑
Soltau, M., Richter, D., & Kreienkamp, H.J. (2002).
The insulin receptor substrate IRSp53 links postsynaptic shank1 to the small G-protein cdc42. Molecular and cellular neurosciences, 21(4), 575-83. [PubMed:12504591] [WorldCat] - ↑
Lim, S., Sala, C., Yoon, J., Park, S., Kuroda, S., Sheng, M., & Kim, E. (2001).
Sharpin, a novel postsynaptic density protein that directly interacts with the shank family of proteins. Molecular and cellular neurosciences, 17(2), 385-97. [PubMed:11178875] [WorldCat] [DOI] - ↑
Park, E., Na, M., Choi, J., Kim, S., Lee, J.R., Yoon, J., ..., & Kim, E. (2003).
The Shank family of postsynaptic density proteins interacts with and promotes synaptic accumulation of the beta PIX guanine nucleotide exchange factor for Rac1 and Cdc42. The Journal of biological chemistry, 278(21), 19220-9. [PubMed:12626503] [WorldCat] [DOI] - ↑
Wendholt, D., Spilker, C., Schmitt, A., Dolnik, A., Smalla, K.H., Proepper, C., ..., & Boeckers, T.M. (2006).
ProSAP-interacting protein 1 (ProSAPiP1), a novel protein of the postsynaptic density that links the spine-associated Rap-Gap (SPAR) to the scaffolding protein ProSAP2/Shank3. The Journal of biological chemistry, 281(19), 13805-16. [PubMed:16522626] [WorldCat] [DOI] - ↑
Naisbitt, S., Kim, E., Tu, J.C., Xiao, B., Sala, C., Valtschanoff, J., ..., & Sheng, M. (1999).
Shank, a novel family of postsynaptic density proteins that binds to the NMDA receptor/PSD-95/GKAP complex and cortactin. Neuron, 23(3), 569-82. [PubMed:10433268] [WorldCat] [DOI] - ↑
Du, Y., Weed, S.A., Xiong, W.C., Marshall, T.D., & Parsons, J.T. (1998).
Identification of a novel cortactin SH3 domain-binding protein and its localization to growth cones of cultured neurons. Molecular and cellular biology, 18(10), 5838-51. [PubMed:9742101] [PMC] [WorldCat] [DOI] - ↑
Qualmann, B., Boeckers, T.M., Jeromin, M., Gundelfinger, E.D., & Kessels, M.M. (2004).
Linkage of the actin cytoskeleton to the postsynaptic density via direct interactions of Abp1 with the ProSAP/Shank family. The Journal of neuroscience : the official journal of the Society for Neuroscience, 24(10), 2481-95. [PubMed:15014124] [PMC] [WorldCat] [DOI] - ↑
Böckers, T.M., Mameza, M.G., Kreutz, M.R., Bockmann, J., Weise, C., Buck, F., ..., & Kreienkamp, H.J. (2001).
Synaptic scaffolding proteins in rat brain. Ankyrin repeats of the multidomain Shank protein family interact with the cytoskeletal protein alpha-fodrin. The Journal of biological chemistry, 276(43), 40104-12. [PubMed:11509555] [WorldCat] [DOI] - ↑
Hwang, J.I., Kim, H.S., Lee, J.R., Kim, E., Ryu, S.H., & Suh, P.G. (2005).
The interaction of phospholipase C-beta3 with Shank2 regulates mGluR-mediated calcium signal. The Journal of biological chemistry, 280(13), 12467-73. [PubMed:15632121] [WorldCat] [DOI] - ↑
Okamoto, P.M., Gamby, C., Wells, D., Fallon, J., & Vallee, R.B. (2001).
Dynamin isoform-specific interaction with the shank/ProSAP scaffolding proteins of the postsynaptic density and actin cytoskeleton. The Journal of biological chemistry, 276(51), 48458-65. [PubMed:11583995] [PMC] [WorldCat] [DOI] - ↑
Shi, S.H., Cheng, T., Jan, L.Y., & Jan, Y.N. (2004).
The immunoglobulin family member dendrite arborization and synapse maturation 1 (Dasm1) controls excitatory synapse maturation. Proceedings of the National Academy of Sciences of the United States of America, 101(36), 13346-51. [PubMed:15340156] [PMC] [WorldCat] [DOI] - ↑
Durand, C.M., Betancur, C., Boeckers, T.M., Bockmann, J., Chaste, P., Fauchereau, F., ..., & Bourgeron, T. (2007).
Mutations in the gene encoding the synaptic scaffolding protein SHANK3 are associated with autism spectrum disorders. Nature genetics, 39(1), 25-7. [PubMed:17173049] [PMC] [WorldCat] [DOI] - ↑
Pinto, D., Pagnamenta, A.T., Klei, L., Anney, R., Merico, D., Regan, R., ..., & Betancur, C. (2010).
Functional impact of global rare copy number variation in autism spectrum disorders. Nature, 466(7304), 368-72. [PubMed:20531469] [PMC] [WorldCat] [DOI] - ↑
Sato, D., Lionel, A.C., Leblond, C.S., Prasad, A., Pinto, D., Walker, S., ..., & Scherer, S.W. (2012).
SHANK1 Deletions in Males with Autism Spectrum Disorder. American journal of human genetics, 90(5), 879-87. [PubMed:22503632] [PMC] [WorldCat] [DOI] - ↑
Peça, J., Feliciano, C., Ting, J.T., Wang, W., Wells, M.F., Venkatraman, T.N., ..., & Feng, G. (2011).
Shank3 mutant mice display autistic-like behaviours and striatal dysfunction. Nature, 472(7344), 437-42. [PubMed:21423165] [PMC] [WorldCat] [DOI] - ↑
Won, H., Lee, H.R., Gee, H.Y., Mah, W., Kim, J.I., Lee, J., ..., & Kim, E. (2012).
Autistic-like social behaviour in Shank2-mutant mice improved by restoring NMDA receptor function. Nature, 486(7402), 261-5. [PubMed:22699620] [WorldCat] [DOI] - ↑
Schmeisser, M.J., Ey, E., Wegener, S., Bockmann, J., Stempel, A.V., Kuebler, A., ..., & Boeckers, T.M. (2012).
Autistic-like behaviours and hyperactivity in mice lacking ProSAP1/Shank2. Nature, 486(7402), 256-60. [PubMed:22699619] [WorldCat] [DOI] - ↑
Bangash, M.A., Park, J.M., Melnikova, T., Wang, D., Jeon, S.K., Lee, D., ..., & Worley, P.F. (2011).
Enhanced polyubiquitination of Shank3 and NMDA receptor in a mouse model of autism. Cell, 145(5), 758-72. [PubMed:21565394] [PMC] [WorldCat] [DOI]

![図2 Shank PDZ ドメインによるダイマー形成とGKAPとの相互作用 PDB 1q3p[6]](/w/images/thumb/7/70/1Q3P.jpg/250px-1Q3P.jpg)
![図3 Shank SAM ドメインの結晶構造 PDB 2f44[7]](/w/images/thumb/3/38/Shank-SAM_2F44.png/250px-Shank-SAM_2F44.png)