「ゲノム編集」の版間の差分
細 →エピゲノム制御 |
細編集の要約なし |
||
| (2人の利用者による、間の25版が非表示) | |||
| 2行目: | 2行目: | ||
<font size="+1">[https://researchmap.jp/koichitanaka 田中光一]</font><br> | <font size="+1">[https://researchmap.jp/koichitanaka 田中光一]</font><br> | ||
''東京医科歯科大学難治疾患研究所''<br> | ''東京医科歯科大学難治疾患研究所''<br> | ||
DOI:<selfdoi /> | DOI:<selfdoi /> 原稿受付日:2018年5月25日 原稿完成日:<br> | ||
担当編集委員:[http://researchmap.jp/2rikenbsi 林 康紀] | 担当編集委員:[http://researchmap.jp/2rikenbsi 林 康紀](独立行政法人理化学研究所) | ||
<br> | <br> | ||
</div> | </div> | ||
{{box|text= 遺伝子は、生物の機能を規定する大きな要因の一つである。次世代シークエンサーの開発により、様々な生物種における遺伝子配列の情報や疾患に関与する遺伝子変異が明らかにされている。しかし、塩基配列を比較するだけでは、遺伝子の機能や疾患の病態を明らかにすることは難しい。ゲノム配列を自由に改変し、その影響を解析できて、初めて生命現象や疾患における遺伝子の役割を理解できる。ゲノム編集は、全ての生物・細胞の、全てのゲノム配列を自在に改変することである。この技術は、神経科学のみならず、多くの生命科学への応用が期待されている<ref><pubmed>24444057</pubmed></ref>[1]。}} | |||
</u>(編集部コメント:この文章はむしろイントロにふさわしいものと思います。抄録では、全体の内容に関するサマリーを願い致します。)</u> | |||
{{box|text= | |||
== ゲノム編集とは == | == ゲノム編集とは == | ||
=== 原理 === | === 原理 === | ||
[[Image:ゲノム図1.png|thumb|right|400px|'''図1. DNA二本鎖切断の修復機構を利用したゲノム編集''']] | [[Image:ゲノム図1.png|thumb|right|400px|'''図1. DNA二本鎖切断の修復機構を利用したゲノム編集''']] | ||
| 26行目: | 24行目: | ||
=== ツール === | === ツール === | ||
[[Image:ゲノム図2.png|thumb|right|400px|'''図2. ゲノム編集に用いられる部位特異的ヌクレアーゼの構造'''<br>'''A.''' ZFN<br />'''B.''' TALEN<br />'''C.''' CRISPR/ | [[Image:ゲノム図2.png|thumb|right|400px|'''図2. ゲノム編集に用いられる部位特異的ヌクレアーゼの構造'''<br>'''A.''' ZFN<br />'''B.''' TALEN<br />'''C.'''CRISPR/Cas8]] | ||
ゲノム編集にとって最も重要なステップは、ゲノム上の狙った塩基配列にDNA二本鎖切断を導入することである。そのために、[[zinc-finger nuclease]] ([[ZFN]])、[[transcription activator-like effector nuclease]] ([[TALEN]])、[[clustered regularly interspaced short palindromic repeats]] ( | ゲノム編集にとって最も重要なステップは、ゲノム上の狙った塩基配列にDNA二本鎖切断を導入することである。そのために、[[zinc-finger nuclease]] ([[ZFN]])、[[transcription activator-like effector nuclease]] ([[TALEN]])、[[clustered regularly interspaced short palindromic repeats]] (CRISPR)/[[CRISPR-associated proteins]] (Cas) [[CRISPR]]/[[Cas9]]、以下CRISPR/Casと略)などの部位特異的[[wj:ヌクレアーゼ|ヌクレアーゼ]]を用いる('''図2''')。 | ||
1996年に報告されたZFNと2010年に報告されたTALENは、DNA二本鎖切断活性を持つ[[FokI]]ヌクレアーゼにDNA結合タンパク質のDNA結合ドメインを融合した一対の人工ヌクレアーゼを用い、狙った標的部位にDNA二本鎖切断を導入する。 | 1996年に報告されたZFNと2010年に報告されたTALENは、DNA二本鎖切断活性を持つ[[FokI]]ヌクレアーゼにDNA結合タンパク質のDNA結合ドメインを融合した一対の人工ヌクレアーゼを用い、狙った標的部位にDNA二本鎖切断を導入する。 | ||
第一世代のZFNは、DNA結合ドメインとしてzinc fingerを持つ人工ヌクレアーゼで、1つのzinc fingerは3塩基を認識するので、3〜6個のzinc fingerを持つZFNは9〜18 base pair (bp)に特異的に結合し、一対で18〜36 bpの特異性でDNA二本鎖切断を導入する。 | 第一世代のZFNは、DNA結合ドメインとしてzinc fingerを持つ人工ヌクレアーゼで、1つのzinc fingerは3塩基を認識するので、3〜6個のzinc fingerを持つZFNは9〜18 base pair (bp)に特異的に結合し、一対で18〜36 bpの特異性でDNA二本鎖切断を導入する。 | ||
第二世代のTALENは、DNA結合ドメインとして植物病原細菌の[[wj:キサントモナス属|''Xanthomonas''属]]が有するTALEを持つ人工ヌクレアーゼである。TALEのDNA結合ドメインは、1塩基を認識する34個のアミノ酸が一単位となり、それを15〜20単位持つTALENをセンス鎖、アンチセンス鎖それぞれに作製し、狙った標的部位にDNA二本鎖切断を導入する。 | 第二世代のTALENは、DNA結合ドメインとして植物病原細菌の[[wj:キサントモナス属|''Xanthomonas''属]]が有するTALEを持つ人工ヌクレアーゼである。TALEのDNA結合ドメインは、1塩基を認識する34個のアミノ酸が一単位となり、それを15〜20単位持つTALENをセンス鎖、アンチセンス鎖それぞれに作製し、狙った標的部位にDNA二本鎖切断を導入する。 | ||
第三世代のCRISPR/Casは、単独でDNA二本鎖切断活性を持つCasヌクレアーゼと標的配列特異的一本鎖ガイドRNAとの複合体を用い、狙った塩基配列にDNA二本鎖切断を導入する。 | 第三世代のCRISPR/Casは、単独でDNA二本鎖切断活性を持つCasヌクレアーゼと標的配列特異的一本鎖ガイドRNAとの複合体を用い、狙った塩基配列にDNA二本鎖切断を導入する。 | ||
この中で、2012年に発表されたCRISPR/Cas9は、その利便性、高効率、汎用性から、わずか1年の間に世界中で使われるゲノム編集の標準技術となった<ref><pubmed>24665839</pubmed></ref>。 | この中で、2012年に発表されたCRISPR/Cas9は、その利便性、高効率、汎用性から、わずか1年の間に世界中で使われるゲノム編集の標準技術となった<ref><pubmed>24665839</pubmed></ref>[2]。 | ||
== CRISPR/ | == CRISPR/Casシステム == | ||
CRISPR/Casシステムは、[[wj:真正細菌|真正細菌]]や[[wj:古細菌|古細菌]]の[[wj:獲得免疫系|獲得免疫系]]として発見された。この獲得免疫システムの標的は、[[wj:細菌|細菌]]に感染する[[wj:ファージ|ファージ]]のDNAやRNAであり、異物として認識されたファージ由来のDNAやRNAは分解され除去される。CRISPR/Casシステムによる異物除去の過程は3つのステップ(adaptation, expression, interference)により行われる。侵入した外来DNAは、細菌内で断片化され、その一部が細菌のゲノム中のCRISPR領域に挿入される(adaptation)。次に外来DNAが侵入した際に、CRISPR領域が転写されてpre-CRISPR RNAが生じ、プロセシングを受けCRISPR RNA (crRNA:外来DNA断片と相補的配列を持つ)が生成される(expression)。プロセシングを受けたcrRNAはCasタンパク質と複合体を形成し、外来DNAやRNAと相補的に結合し、それらを切断する(interference)。 | CRISPR/Casシステムは、[[wj:真正細菌|真正細菌]]や[[wj:古細菌|古細菌]]の[[wj:獲得免疫系|獲得免疫系]]として発見された。この獲得免疫システムの標的は、[[wj:細菌|細菌]]に感染する[[wj:ファージ|ファージ]]のDNAやRNAであり、異物として認識されたファージ由来のDNAやRNAは分解され除去される。CRISPR/Casシステムによる異物除去の過程は3つのステップ(adaptation, expression, interference)により行われる。侵入した外来DNAは、細菌内で断片化され、その一部が細菌のゲノム中のCRISPR領域に挿入される(adaptation)。次に外来DNAが侵入した際に、CRISPR領域が転写されてpre-CRISPR RNAが生じ、プロセシングを受けCRISPR RNA (crRNA:外来DNA断片と相補的配列を持つ)が生成される(expression)。プロセシングを受けたcrRNAはCasタンパク質と複合体を形成し、外来DNAやRNAと相補的に結合し、それらを切断する(interference)。 | ||
| 45行目: | 43行目: | ||
=== DNAの編集 === | === DNAの編集 === | ||
==== CRISPR/ | ==== CRISPR/Cas9システム ==== | ||
CRISPR/Cas9システムは、クラス2のⅡ型に分類されるCRISPR/Casシステムであり、CRISPR RNA (crRNA:外来DNA断片と相補的配列を持つ)、trans-activating crRNA (tracrRNA:crRNAの外来DNAと相補的配列以外の部分に結合し、Cas9とcrRNAの複合体形成に必要である)、Cas9タンパク質の3種類の要素から成っている('''図2c''')。[[wj:化膿レンサ球菌|''Streptococcus pyogenes'']]株由来のCas9タンパク質は、標的ゲノム配列の下流にある3つの塩基;N(G, A, T, or C)GGをPMA配列(Proto-spacer Adjacent Motif)として認識し、その3塩期上流を切断する。現在普及しているシステムは、標的DNAに対して相補的配列を持つcrRNAの3’末端にtracrRNAを連結させたsingle guide RNA (sgRNA)とCas9を発現させることにより、ゲノムDNA上の狙った部位にDNA二本鎖切断を導入する。 | CRISPR/Cas9システムは、クラス2のⅡ型に分類されるCRISPR/Casシステムであり、CRISPR RNA (crRNA:外来DNA断片と相補的配列を持つ)、trans-activating crRNA (tracrRNA:crRNAの外来DNAと相補的配列以外の部分に結合し、Cas9とcrRNAの複合体形成に必要である)、Cas9タンパク質の3種類の要素から成っている('''図2c''')。[[wj:化膿レンサ球菌|''Streptococcus pyogenes'']]株由来のCas9タンパク質は、標的ゲノム配列の下流にある3つの塩基;N(G, A, T, or C)GGをPMA配列(Proto-spacer Adjacent Motif)として認識し、その3塩期上流を切断する。現在普及しているシステムは、標的DNAに対して相補的配列を持つcrRNAの3’末端にtracrRNAを連結させたsingle guide RNA (sgRNA)とCas9を発現させることにより、ゲノムDNA上の狙った部位にDNA二本鎖切断を導入する。 | ||
約100塩基のsgRNAのうち、DNA二本鎖切断の標的部位を規定するのは標的部位と相補的配列を持つ20塩基のみである。従って、CRISPR/Cas9システムをゲノム編集ツールとして利用する場合、標的ごとに変える必要があるのはわずか20塩基のみであり、それ以外の塩基配列およびCas9はすべて共通である。CRISPR/Cas9システムは、guide RNAの作製の簡便さ、guide RNAを増やすことにより複数遺伝子の同時編集が可能なことから、誰もが使うことのできるゲノム編集ツールとして急速に普及した。2012年の最初の発表以来、[[wj:大腸菌|大腸菌]]、[[ヒト]]細胞から[[ゼブラフィッシュ]]に至る多くの細胞・生物種への応用が報告されている<ref><pubmed> 25430774</pubmed></ref>。いまやヒトや[[サル]]を含むあらゆる動物個体、植物、微生物への利用が急速に広がっている。 | 約100塩基のsgRNAのうち、DNA二本鎖切断の標的部位を規定するのは標的部位と相補的配列を持つ20塩基のみである。従って、CRISPR/Cas9システムをゲノム編集ツールとして利用する場合、標的ごとに変える必要があるのはわずか20塩基のみであり、それ以外の塩基配列およびCas9はすべて共通である。CRISPR/Cas9システムは、guide RNAの作製の簡便さ、guide RNAを増やすことにより複数遺伝子の同時編集が可能なことから、誰もが使うことのできるゲノム編集ツールとして急速に普及した。2012年の最初の発表以来、[[wj:大腸菌|大腸菌]]、[[ヒト]]細胞から[[ゼブラフィッシュ]]に至る多くの細胞・生物種への応用が報告されている<ref><pubmed> 25430774</pubmed></ref>[3]。いまやヒトや[[サル]]を含むあらゆる動物個体、植物、微生物への利用が急速に広がっている。 | ||
ゲノム編集ツールとしてのCRISPR/ | ゲノム編集ツールとしてのCRISPR/Cas9システムの大きな問題点は、「オフターゲット」と「PAM配列の制約」である。オフターゲットとは、標的でないゲノム部位のDNA配列を変えてしまうことである。オフターゲットの起こる頻度は、細胞種・標的遺伝子座・guide RNAなどにより大きく変化する。オフターゲットを回避する方法として、ダブルニッキング法が考案されている。天然型のCas9は2つのヌクレアーゼドメインを持っているが、その一方をアミノ酸置換により不活性化した一本鎖切断型Cas9(Cas9 nickase)を用いる方法が考案されている<ref><pubmed>23992846</pubmed></ref><ref><pubmed>27208701</pubmed></ref>[4][5]。標的部位に近接したセンス鎖、アンチセンス鎖に1対のCRISPR/Cas9 nickaseが結合した際にのみDNA二本鎖切断が誘導されるので、オフターゲットの起こる頻度は少なくなる。最近、Cas9 nickaseを用いた標的部位でのゲノム編集効率は、天然型のCas9編集効率と同等かそれ以上であることが報告されている<ref><pubmed> 29584876</pubmed></ref>[6]。また、CRISPR/Cas9を用いて作製された遺伝子改変マウスにおけるオフターゲットの頻度は、全ゲノムレベルで解析した例が少なく確定的ではないが、当初報告されたよりは少ないと考えられている<ref>CRISPR off-targets: a reassessment.<br> | ||
Nature Methods. 2018, 15(4):229-30. | Nature Methods. 2018, 15(4):229-30. doi:10.1038/nmeth.4664</ref> <ref>'''Schaefer KA, Darbo BW, Colgan DF, Tsang SH, Bassuk AG, Mahajan VB.'''<br>Corrigendum and follow-up: Whole genome sequencing of multiple CRISPR-edited mouse lines suggests no excess mutations.<br>bioRxiv. 2017, Posted Jun. 23. Doi: http://dx.org/10.1101/154450</ref> [7][8]。 | ||
在ゲノム編集で最もよく使われているSpCas9は''Streptococcus pyogenes''由来であり、DNA二本鎖切断の部位を決めるには標的DNA配列の下流に隣接するNGGというPAM配列が必要である。このPAM配列の制約により、ゲノムの全ての場所を編集できないという制限があった。David Liuのグループは、[[phage-assisted continuous evolution]] ([[PACE]])を利用して、NG、GAAおよびGATをPAMとするSpCas9変異体 (xCas9)の作成に成功した<ref><pubmed>29512652</pubmed></ref> | 在ゲノム編集で最もよく使われているSpCas9は''Streptococcus pyogenes''由来であり、DNA二本鎖切断の部位を決めるには標的DNA配列の下流に隣接するNGGというPAM配列が必要である。このPAM配列の制約により、ゲノムの全ての場所を編集できないという制限があった。David Liuのグループは、[[phage-assisted continuous evolution]] ([[PACE]])を利用して、NG、GAAおよびGATをPAMとするSpCas9変異体 (xCas9)の作成に成功した<ref><pubmed>29512652</pubmed></ref>[9]。xCas9は哺乳類細胞において、最も広範なPAM配列を認識する制約の少ないCasである。さらに機序は不明であるが、xCas9はオフターゲットの頻度も抑制し、Cas9の主要な欠点であるオフターゲットとPAM配列の制約の2つを回避できる理想的なゲノム編集ツールである。 | ||
==== CRISPR/Cpf1システム ==== | |||
[[w:Cpf1|Cpf1]] ([[w:Cas12a|Cas12a]])は、クラス2Ⅴ型のCRISPR/Casシステムに関わるDNA[[wj:エンドヌクレアーゼ|エンドヌクレアーゼ]]であり、新たなゲノム編集ツールとして注目されている<ref><pubmed>26422227</pubmed></ref>[10]。Cpf1はCas9とは異なる以下のような特徴を持っている。 | |||
==== CRISPR/ | |||
[[w:Cpf1|Cpf1]] ([[w:Cas12a|Cas12a]])は、クラス2Ⅴ型のCRISPR/Casシステムに関わるDNA[[wj:エンドヌクレアーゼ|エンドヌクレアーゼ]]であり、新たなゲノム編集ツールとして注目されている<ref><pubmed>26422227</pubmed></ref>。Cpf1はCas9とは異なる以下のような特徴を持っている。 | |||
#Cpf1はgRNAとしてcrRNAのみを必要とし、tracrRNAは必要ない。 | #Cpf1はgRNAとしてcrRNAのみを必要とし、tracrRNAは必要ない。 | ||
#Cpf1はCas9と異なり、PAM配列としてTTTV (VはA, C, G)、TTCV, TCTV, CTTVを認識する。 | #Cpf1はCas9と異なり、PAM配列としてTTTV (VはA, C, G)、TTCV, TCTV, CTTVを認識する。 | ||
#Cas9はDNA二本鎖を切断し平滑末端を形成するが、Cpf1は突出末端を形成する。 | #Cas9はDNA二本鎖を切断し平滑末端を形成するが、Cpf1は突出末端を形成する。 | ||
CRISPR/Cpf1システムは、ヒト細胞株やマウス受精卵のゲノム編集に応用され、CRISPR/Cas9システムよりオフターゲットの頻度が少ないことが報告されている<ref><pubmed>27347757</pubmed></ref><ref><pubmed>27272387</pubmed></ref>。 | CRISPR/Cpf1システムは、ヒト細胞株やマウス受精卵のゲノム編集に応用され、CRISPR/Cas9システムよりオフターゲットの頻度が少ないことが報告されている<ref><pubmed>27347757</pubmed></ref><ref><pubmed>27272387</pubmed></ref>[11][12]。 | ||
==== CRISPR/dCAS9- | ==== CRISPR/dCAS9-BEシステム ==== | ||
従来のゲノム編集は、標的のゲノム部位にDNAの二本鎖切断を起こし、その後に誘導されるDNAの修復機構を利用し、標的DNAを編集する。 | 従来のゲノム編集は、標的のゲノム部位にDNAの二本鎖切断を起こし、その後に誘導されるDNAの修復機構を利用し、標的DNAを編集する。 | ||
一方CRISPR/dCAS9-BEシステムは、DNAを切断することなく標的DNAの塩基を編集する方法である。ヌクレアーゼ活性を失活させたCas9(dCas9)に、脱アミノ化酵素である[[シチジンデアミナーゼ]]を融合させた塩基エディター(BE)を作成し、guide RNAにより狙ったゲノム部位に塩基エディターを働かせ、標的部位の[[wj:シトシン|シトシン]](C)を[[wj:チミン|チミン]](T)(あるいは[[wj:グアニン|グアニン]](G)を[[wj:アデニン|アデニン]](A))に置換する<ref><pubmed>27096365</pubmed></ref><ref><pubmed>27492474</pubmed></ref>。さらにDavid Liuのグループは、PACEを利用してDNAのAをG(あるいはTをC)に置換できる転移RNAのアデノシンデアミナーゼ変異体(アデニン塩基エディター(ABE))の作成に成功した<ref><pubmed>29160308</pubmed></ref> | 一方CRISPR/dCAS9-BEシステムは、DNAを切断することなく標的DNAの塩基を編集する方法である。ヌクレアーゼ活性を失活させたCas9(dCas9)に、脱アミノ化酵素である[[シチジンデアミナーゼ]]を融合させた塩基エディター(BE)を作成し、guide RNAにより狙ったゲノム部位に塩基エディターを働かせ、標的部位の[[wj:シトシン|シトシン]](C)を[[wj:チミン|チミン]](T)(あるいは[[wj:グアニン|グアニン]](G)を[[wj:アデニン|アデニン]](A))に置換する<ref><pubmed>27096365</pubmed></ref><ref><pubmed>27492474</pubmed></ref>[13][14]。さらにDavid Liuのグループは、PACEを利用してDNAのAをG(あるいはTをC)に置換できる転移RNAのアデノシンデアミナーゼ変異体(アデニン塩基エディター(ABE))の作成に成功した<ref><pubmed>29160308</pubmed></ref>[15]。dCas9と融合したBEあるいはABEを用いることにより、DNAの二本鎖切断を起こさずにDNAの4塩基全てを個別に置き換えられる。既知の遺伝性疾患の原因となる一塩基変異の約50%は、G-C塩基対からA-T塩基対への転移なので、CRISPR/dCas-ABEシステムは遺伝性疾患を根本的に治す可能性を持っている。 | ||
===RNAの編集=== | ===RNAの編集=== | ||
CRISPR/Cas9システムやCRISPR/Cpf1システムの標的はDNAである。DNAと同様にRNAは生命現象において重要な役割を担っており、RNAを標的にした編集技術は、生命科学に大きな貢献をする。 | CRISPR/Cas9システムやCRISPR/Cpf1システムの標的はDNAである。DNAと同様にRNAは生命現象において重要な役割を担っており、RNAを標的にした編集技術は、生命科学に大きな貢献をする。 | ||
==== CRISPR/Cas13(C2c2)==== | ==== CRISPR/Cas13(C2c2)システム ==== | ||
[[w:Feng Zhang|Feng Zhang]]のグループは微生物ゲノムデータベースを探索し、クラス2タイプⅥ型CRISPR/Casシステムの[[Cas13]]([[C2c2]])が、標的RNAに相補的なRNA依存性に一本鎖RNAを切断する酵素であることを見つけた<ref><pubmed>27256883</pubmed></ref>。''Leptotrichia wadei''由来の[[Cas13a]] ([[LwaCas13a]])をCRISPRシステムに組み込んだ系は、標的RNAを高効率かつ高い特異性でノックダウンすることができる<ref name=Abudayyeh2017><pubmed>28976959</pubmed></ref>。 | [[w:Feng Zhang|Feng Zhang]]のグループは微生物ゲノムデータベースを探索し、クラス2タイプⅥ型CRISPR/Casシステムの[[Cas13]]([[C2c2]])が、標的RNAに相補的なRNA依存性に一本鎖RNAを切断する酵素であることを見つけた<ref><pubmed>27256883</pubmed></ref>[16]。''Leptotrichia wadei''由来の[[Cas13a]] ([[LwaCas13a]])をCRISPRシステムに組み込んだ系は、標的RNAを高効率かつ高い特異性でノックダウンすることができる<ref name=Abudayyeh2017><pubmed>28976959</pubmed></ref>[17]。 | ||
CRISPR/Cas13aを用いた標的RNAのノックダウンはRNA干渉法(RNAi)に比べ、 | CRISPR/Cas13aを用いた標的RNAのノックダウンはRNA干渉法(RNAi)に比べ、 | ||
| 85行目: | 81行目: | ||
最近、Cas13aより高効率かつ高い特異性で標的RNAをノックダウンすることができる[[Cas13b]]が同定された<ref name=Cox2017><pubmed>29070703</pubmed></ref>[18]。Cas13は、pre-CRISPR RNAをプロセッシングしcrRNAを生成できる活性を持っており、多数の標的RNAを含んだpre-CRISPR RNAをguide RNAとして用いることにより、一度に多くのRNAをノックダウンできる<ref name=Abudayyeh2017/>[17]。 | 最近、Cas13aより高効率かつ高い特異性で標的RNAをノックダウンすることができる[[Cas13b]]が同定された<ref name=Cox2017><pubmed>29070703</pubmed></ref>[18]。Cas13は、pre-CRISPR RNAをプロセッシングしcrRNAを生成できる活性を持っており、多数の標的RNAを含んだpre-CRISPR RNAをguide RNAとして用いることにより、一度に多くのRNAをノックダウンできる<ref name=Abudayyeh2017/>[17]。 | ||
==== CRISPR/dCas13- | ==== CRISPR/dCas13-ADARシステム ==== | ||
[[Image:ゲノム図3.png|thumb|right|400px|'''図3. CRISPR/dCas13-ADARシステム''']] | [[Image:ゲノム図3.png|thumb|right|400px|'''図3. CRISPR/dCas13-ADARシステム''']] | ||
[[w:Feng Zhang|Feng Zhang]]のグループは、失活させたCas13b (dCas13b)にRNAデアミナーゼ(RNAのアデニン(A)を[[wj:イノシン|イノシン]](I)に変化させる酵素,ADAR)を融合させ、標的RNAを編集できる系を確立した(RNA Editing for Programmable A to I Replacement (REPAIR)'''図3''')<ref name=Cox2017/>[18]。この系は、RNAの変異を正常に戻すことができる。さらに、RNAを標的にした編集はオフターゲットが起きてもその影響は一過性であり、遺伝子治療としては安全性が高い。 | |||
== CRISPR/ | == CRISPR/Casシステムの神経科学への応用 == | ||
=== 細胞への応用=== | === 細胞への応用[19] === | ||
CRISPR/ | CRISPR/Casシステムを細胞に適用することにより、遺伝子の機能を欠損させたり亢進させたりすることが簡単にでき、様々な細胞機能に関与する遺伝子群をゲノムワイドに検索できる。<ref><pubmed>27606440</pubmed></ref> | ||
====ゲノム編集 ==== | ====ゲノム編集 ==== | ||
神経細胞などの[[初代培養細胞]]は、一般的にCRISPR/Casシステムによるゲノム編集効率は低く、それぞれの細胞種による最適化が必要である。[[ES細胞]]や[[iPS細胞]]におけるCRISPR/Casシステムを用いたゲノム編集の効率化は、多くの実績がある。従って、ゲノム編集したES細胞やiPS細胞を用い、それらから分化させた神経細胞の機能を解析する方法も有効である。 | 神経細胞などの[[初代培養細胞]]は、一般的にCRISPR/Casシステムによるゲノム編集効率は低く、それぞれの細胞種による最適化が必要である。[[ES細胞]]や[[iPS細胞]]におけるCRISPR/Casシステムを用いたゲノム編集の効率化は、多くの実績がある。従って、ゲノム編集したES細胞やiPS細胞を用い、それらから分化させた神経細胞の機能を解析する方法も有効である。 | ||
また、CRISPR/Casシステムの開発により、疾患原因遺伝子の変異以外は遺伝的に同一(isogenic)なiPS細胞を作成することが効率化された。iPS細胞におけるゲノム編集は、従来の相同組換えを用いた方法では変異導入効率が低く、クローン化に多大な労力が必要とされた。しかし、CRISPR/Casシステムの開発により、iPS細胞のゲノム編集がより効率化され、単純な遺伝子欠損に加え、疾患の原因と考えられる様々な変異([[1塩基置換]]や大きな欠失など)を導入することが可能になった。従って、樹立された患者iPS細胞の疾患原因遺伝子の変異を正常に戻したり、健常人から樹立したiPS細胞に疾患の原因遺伝子変異を導入することによりisogenicな疾患モデル細胞を作成することが可能になった<ref><pubmed>26059412</pubmed></ref>。今後、isogenicなiPS細胞を用いることにより、疾患研究が加速すると期待される。 | また、CRISPR/Casシステムの開発により、疾患原因遺伝子の変異以外は遺伝的に同一(isogenic)なiPS細胞を作成することが効率化された。iPS細胞におけるゲノム編集は、従来の相同組換えを用いた方法では変異導入効率が低く、クローン化に多大な労力が必要とされた。しかし、CRISPR/Casシステムの開発により、iPS細胞のゲノム編集がより効率化され、単純な遺伝子欠損に加え、疾患の原因と考えられる様々な変異([[1塩基置換]]や大きな欠失など)を導入することが可能になった。従って、樹立された患者iPS細胞の疾患原因遺伝子の変異を正常に戻したり、健常人から樹立したiPS細胞に疾患の原因遺伝子変異を導入することによりisogenicな疾患モデル細胞を作成することが可能になった<ref><pubmed>26059412</pubmed></ref>[20]。今後、isogenicなiPS細胞を用いることにより、疾患研究が加速すると期待される。 | ||
==== 転写制御 ==== | ==== 転写制御 ==== | ||
CRISPR/Casシステムは、RNA誘導型ヌクレアーゼであり、ガイドRNAが標的ゲノム部位にCasを誘導する。Casに点変異を入れ、ガイドRNAとは複合体を形成できるがヌクレアーゼ活性を持たないCas を作成することができる。この不活性型Cas(dCas9やdCpf1) | CRISPR/Casシステムは、RNA誘導型ヌクレアーゼであり、ガイドRNAが標的ゲノム部位にCasを誘導する。Casに点変異を入れ、ガイドRNAとは複合体を形成できるがヌクレアーゼ活性を持たないCas を作成することができる。この不活性型Cas(dCas9やdCpf1)に、様々な活性を持つ蛋白質を融合することにより、融合蛋白質を標的ゲノム部位に誘導することができる。転写活性化因子Vp64あるいは転写抑制因子KRABをdCas9やdCpf1と融合し、標的ゲノム部位と相補的配列を持つガイドRNAと伴に細胞に導入すると、標的遺伝子の転写を活性化あるいは抑制することができ、遺伝子の機能を解析することができる<ref><pubmed>23849981</pubmed></ref> <ref><pubmed>29235474</pubmed></ref>[21][22]。しかし、一つのdCasに対し一つの転写調節因子を結合させても遺伝子転写制御は不十分であり、通常は、dCasまたはガイドRNAに複数の転写調節因子を付加する系が使われている<ref><pubmed>27248712</pubmed></ref>[23]。この系は、一つの細胞内の複数の遺伝子の転写を同時に制御することができる。 | ||
==== エピゲノム制御 ==== | ==== エピゲノム制御 ==== | ||
ヒストンの修飾やDNAのメチル化などのエピゲノミックな修飾は、脳形成や神経可塑性において重要な役割をもち、さらに、精神神経疾患の発症に関与することが報告されている。CRISPR/Casシステムを用いることにより、従来困難であった特定のゲノム領域のエピゲノミックな修飾状態を改変する「エピゲノム編集」が可能になった<ref><pubmed>28985525</pubmed></ref>[24]。ヒストンはメチル化やアセチル化などの翻訳後修飾を受け、転写の制御やクロマチン濃縮などに関与する。dCas9にヒストン修飾を導入する酵素(ヒストンアセチル基転移酵素、ヒストン脱アセチル化酵素、リジンメチル基転移酵素、リジン脱メチル化酵素など)を融合させた人工タンパク質と標的ゲノム部位と相補的配列を持つガイドRNAを細胞に導入すると、標的遺伝子のエピゲノミックな修飾状態を改変できる。また、DNAを構成する4種類の塩基のなかでシトシンのみがメチル基の付加・除去を受け、転写を制御している。dCas9にDNAメチル化を制御する酵素(DNAメチル化酵素やDNA脱メチル化酵素)を融合させた人工タンパク質と標的ゲノム部位と相補的配列を持つガイドRNAを細胞に導入すると、標的ゲノム領域のDNAのメチル化状態を改変できる。しかし、一つのdCas9に対し一つのエピゲノム修飾因子を結合させてもエピゲノム制御は不十分であり、通常は、dCas9またはガイドRNAに複数のエピゲノム修飾因子を付加する系が使われている<ref><pubmed> 27571369</pubmed></ref>[25]。Rudolf Jaenischのグループは、dCas9にTet1(シトシンのメチル基を除去する酵素)を融合させた人工タンパク質を用い、Fragile X症候群のiPS細胞モデルの治療に成功している<ref><pubmed>29456084</pubmed></ref>[26]。Fragile X症候群では、FMR1遺伝子の転写開始より少し上流にあるCGGの繰り返し配列が増加しメチル化が促進され、FMR1遺伝子の発現が抑制されている。Fragile X症候群の患者さんから作成したiPS細胞に、FMR1遺伝子の転写開始より少し上流を標的にしたガイドRNAとdCas9-tet1を導入し、CGGの繰り返し配列のメチル基を外し、FMR1遺伝子の発現を回復させることに成功した。FMR1遺伝子の発現が回復した患者iPS細胞を神経細胞に分化させると、神経細胞の過活動が正常に戻った。 | |||
==== RNA制御 ==== | ==== RNA制御 ==== | ||
| 115行目: | 105行目: | ||
==== 受精卵におけるゲノム編集 ==== | ==== 受精卵におけるゲノム編集 ==== | ||
[[Image:ゲノム図4.png|thumb|right|400px|'''図4. CRISPR/Cas9を用いた疾患モデルマウスの作成''']] | [[Image:ゲノム図4.png|thumb|right|400px|'''図4. CRISPR/Cas9を用いた疾患モデルマウスの作成''']] | ||
従来、遺伝子改変動物は、ES細胞の相同組換えによる遺伝子改変を基盤として作成されてきた。しかし、その作成には長い時間、多大な労力、多額の費用が必要である。さらに、遺伝子改変動物の作成は、マウス等、ES細胞が樹立されているごく一部の動物種に限られていた。CRISPR/Cas9システムは、この状況を一変させた。CRISPR/Cas9システムを用いた遺伝子改変動物の作成は、標的配列に対するガイドRNA、Cas9をコードするmRNAおよびノックインの場合にはドナーDNAを受精卵に注入するだけで、受精卵内で標的遺伝子が改変され、短時間にノックアウト・ノックイン動物を得ることができる('''図4''')。さらに最近、従来の「顕微注入法」で必要とされる受精卵の単離、顕微注入、移植といった一連の作業を省略できるGONAD(Genome-editing via Oviductal Nucleic Acids Delivery)法が報告された<ref><pubmed>29482575</pubmed></ref>[27]。GONAD法では、0.7日胚(着床前)を有する妊娠雌マウスの卵管内に標的配列に対するガイドRNA、Cas9をコードするmRNAを注入し、卵管全体に対し直接電気穿孔法を行う。CRISPR/Cas9システムを用いた遺伝子改変動物の作製法は、そのきわめて迅速で簡便で高い効率性が特徴である。以下に、遺伝子欠損・塩基置換・外来遺伝子ノックイン動物の作成法を、マウスを中心に概説する。 | |||
===== 遺伝子欠損マウスの作製 ===== | ===== 遺伝子欠損マウスの作製 ===== | ||
標的配列に対するガイドRNA、Cas9をコードするmRNAを野生型受精卵に注入する('''図4''')。注意点としては、標的遺伝子に3の倍数の塩基欠損や挿入が起こることがあり、その際にはフレームシフトが起こらず、標的遺伝子の機能破壊の有無を何らかの方法で確認する必要がある。CRISPR/Cas9を用いた遺伝子欠損マウスの作成は効率が高く(通常50〜100%)、複数の遺伝子を同時に欠損させることもできる<ref name=Wang2013><pubmed>23643243</pubmed></ref>。 | 標的配列に対するガイドRNA、Cas9をコードするmRNAを野生型受精卵に注入する('''図4''')。注意点としては、標的遺伝子に3の倍数の塩基欠損や挿入が起こることがあり、その際にはフレームシフトが起こらず、標的遺伝子の機能破壊の有無を何らかの方法で確認する必要がある。CRISPR/Cas9を用いた遺伝子欠損マウスの作成は効率が高く(通常50〜100%)、複数の遺伝子を同時に欠損させることもできる<ref name=Wang2013><pubmed>23643243</pubmed></ref>[28]。 | ||
===== 塩基置換マウスの作製 ===== | ===== 塩基置換マウスの作製 ===== | ||
塩基置換する配列が数十塩基以内であれば、一本鎖のオリゴDNAを標的配列に対するガイドRNA、Cas9をコードするmRNAと伴に野生型受精卵に注入する('''図4''')。CRISPR/Cas9を用いた塩基置換マウスの作成は効率が高く、複数の遺伝子に同時に変異を導入することもできる<ref name=Wang2013/>。 | 塩基置換する配列が数十塩基以内であれば、一本鎖のオリゴDNAを標的配列に対するガイドRNA、Cas9をコードするmRNAと伴に野生型受精卵に注入する('''図4''')。CRISPR/Cas9を用いた塩基置換マウスの作成は効率が高く、複数の遺伝子に同時に変異を導入することもできる<ref name=Wang2013/>[28]。 | ||
===== 外来遺伝子ノックインマウスの作製 ===== | ===== 外来遺伝子ノックインマウスの作製 ===== | ||
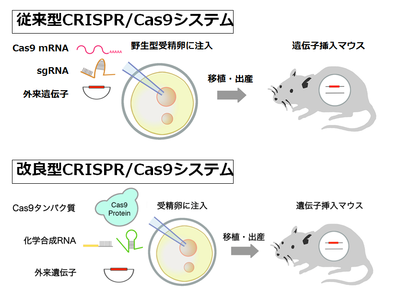
[[Image:ゲノム図5.png|thumb|right|400px|'''図5. クローニングフリーCRISPR/Cas9システム''']] | [[Image:ゲノム図5.png|thumb|right|400px|'''図5. クローニングフリーCRISPR/Cas9システム''']] | ||
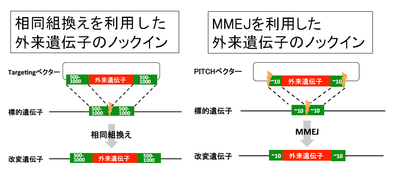
[[Image:ゲノム図6.png|thumb|right|400px|'''図6. PITCh法を用いた外来遺伝子のノックイン''']] | [[Image:ゲノム図6.png|thumb|right|400px|'''図6. PITCh法を用いた外来遺伝子のノックイン''']] | ||
遺伝子改変マウスの作成を容易にしたCRISPR/Cas9であるが、数kbの長い外来遺伝子のノックインマウスの作製は困難である。Rudolf Jaenischらにより外来遺伝子のノックインマウス作製の成功が報告されているが、その作成効率は10%程度であり、遺伝子欠損や塩基置換ノックインと比べて低い<ref><pubmed>23992847</pubmed></ref> | 遺伝子改変マウスの作成を容易にしたCRISPR/Cas9であるが、数kbの長い外来遺伝子のノックインマウスの作製は困難である。Rudolf Jaenischらにより外来遺伝子のノックインマウス作製の成功が報告されているが、その作成効率は10%程度であり、遺伝子欠損や塩基置換ノックインと比べて低い<ref><pubmed>23992847</pubmed></ref>[29]。数kbの外来遺伝子のノックインは、蛍光タンパク質やCreリコンビナーゼ等の機能カセットを標的部位に挿入したり、floxedマウスを作成するのに必要であり、生命科学にとって欠かすことのできない重要な技術である。このため、ノックインマウス作製の効率化は、CRISPR/Cas9を用いたゲノム編集にとって大きな課題の一つであった。 | ||
従来、CRISPR/ | 従来、CRISPR/Cas9を用いたゲノム編集では、Cas9およびガイドRNAをともにRNAの形で受精卵の前核に顕微注入している('''図5''')。従来法でCas9がヌクレアーゼとして前核内で働くためには、まず注入されたCas9 mRNAが前核から細胞質へ輸送され、蛋白質に翻訳され、さらに翻訳された蛋白質が再度、前核に移行し、標的部位に到達、切断を行う必要がある。この過程は時間を要する。外来遺伝子のノックインに必要な相同組換えは、細胞周期のS期からG2期にのみ起こることから、その効率を上げるためには、Cas9は受精卵に注入後できるだけ早くDNA二本鎖切断活性を持つ必要がある。 | ||
そこで筆者らは、Cas9をmRNAではなく蛋白質として受精卵前核に注入することで、ノックイン効率を向上できるのではないかと考えた。実際、培養細胞ではCas9をmRNAではなく蛋白質として導入することにより、DNA二本鎖切断が迅速に起こることが示されている。さらに、外来遺伝子のノックイン効率を向上させるため、Cas9の標的部位切断効率を上げる工夫をした。細菌の獲得免疫系としてのCRISPR/Cas9システムは、Cas9と2種類のガイドRNA(標的部位と相補的な配列を持つをcrRNAとCas9およびcrRNAの橋渡しを担うtracrRNA)からなる3要素のシステムである('''図2C''')。 | そこで筆者らは、Cas9をmRNAではなく蛋白質として受精卵前核に注入することで、ノックイン効率を向上できるのではないかと考えた。実際、培養細胞ではCas9をmRNAではなく蛋白質として導入することにより、DNA二本鎖切断が迅速に起こることが示されている。さらに、外来遺伝子のノックイン効率を向上させるため、Cas9の標的部位切断効率を上げる工夫をした。細菌の獲得免疫系としてのCRISPR/Cas9システムは、Cas9と2種類のガイドRNA(標的部位と相補的な配列を持つをcrRNAとCas9およびcrRNAの橋渡しを担うtracrRNA)からなる3要素のシステムである('''図2C''')。 | ||
しかし従来のCRISPR/Cas9システムは、実験操作を簡便にするためcrRNAとtracrRNAを連結した一本鎖ガイドRNA(sgRNA)とCas9の2要素からなるシステムである('''図5''')。自然界の3要素システムの方が、2要素システムより、高い標的配列切断活性を持つことが報告されている。そこで筆者らは、sgRNAのかわりにcrRNAとtracrRNAを用い、Cas9蛋白質と組み合わせることで、ノックイン効率を向上できるのではないかと考えた。crRNAとtracrRNAを用いるもう一つの利点は、化学合成が可能になり、sgRNAの作製に必要な大腸菌での遺伝子組換え実験を省略できる点である。sgRNAの長さは約100塩基であるが、crRNA、tracrRNAは各々50塩基程度であり、化学合成が可能である。筆者らの改良したCRISPR/Cas9システムは、Cas9蛋白質と化学合成したcrRNAとtracrRNAの3要素からなる('''図5''')。このシステムは、大腸菌での遺伝子組換え実験を行うことなくゲノム編集が可能で、クローニングフリーCRISPR/Cas9システムと名づけた<ref name=Aida2015><pubmed>25924609</pubmed></ref> | しかし従来のCRISPR/Cas9システムは、実験操作を簡便にするためcrRNAとtracrRNAを連結した一本鎖ガイドRNA(sgRNA)とCas9の2要素からなるシステムである('''図5''')。自然界の3要素システムの方が、2要素システムより、高い標的配列切断活性を持つことが報告されている。そこで筆者らは、sgRNAのかわりにcrRNAとtracrRNAを用い、Cas9蛋白質と組み合わせることで、ノックイン効率を向上できるのではないかと考えた。crRNAとtracrRNAを用いるもう一つの利点は、化学合成が可能になり、sgRNAの作製に必要な大腸菌での遺伝子組換え実験を省略できる点である。sgRNAの長さは約100塩基であるが、crRNA、tracrRNAは各々50塩基程度であり、化学合成が可能である。筆者らの改良したCRISPR/Cas9システムは、Cas9蛋白質と化学合成したcrRNAとtracrRNAの3要素からなる('''図5''')。このシステムは、大腸菌での遺伝子組換え実験を行うことなくゲノム編集が可能で、クローニングフリーCRISPR/Cas9システムと名づけた<ref name=Aida2015><pubmed>25924609</pubmed></ref>[30]。 | ||
このクローニングフリーCRISPR/Cas9システムは、ガイドRNAの活性評価も簡便である。従来のような培養細胞を用いた実験は必要なく、試験管内で標的配列を含むPCR産物、Cas9蛋白質、crRNA、tracrRNAをインキュベートして電気泳動するだけで(in vitro digestion assay:IDA)、その切断活性を調べることができる。クローニングフリーCRISPR/Cas9システムを用い、Act(actin beta)遺伝子座にEGFP(enhanced green fluorescent protein)を含む2.5 kbの外来遺伝子をノックインするマウスを作成したところ、およそ50%の新生仔マウスにEGFPが目的部位にノックインされていた。従来用いられてきたCas9 mRNAとsgRNAからなる2要素システムを用い対照実験を行ったところ、その効率は10%程度だった。このことからクローニングフリーCRISPR/Cas9システムは、外来遺伝子のノックイン効率を大幅に向上させることが明らかになった<ref name=Aida2015/>[30]。さらに作製したノックインマウスを野生型マウスと交配し、次世代への伝達効率を調べたところ、すべての系統から、50%の効率で次世代のノックインマウスが得られた。このことは、従来型CRISPR/Cas9システムで問題となるモザイク(同一個体内の一部の細胞のみに遺伝子改変が起こっている)の頻度が低いことを示している。つまり、クローニングフリーCRISPR/Cas9システムでは、標的部位のDNA二本鎖切断が迅速に起こり、受精卵の第一卵割までに片アリルに外来遺伝子がノックインされたことを示している。またオフターゲット変異の候補となる部位を解析したところ、いずれのノックインマウスでも変異は検出されなかった。このことは、クローニングフリーCRISPR/Cas9システムでは、Cas9を蛋白質として注入したことによりCas9の半減期が短くなり、標的部位を切断した後迅速に分解されるため、従来型CRISPR/Cas9システムの大きな課題であるオフターゲット変異(ガイドRNA配列に類似した配列の非特異的切断)が大幅に減少することを示している。 | |||
以上の結果は、クローニングフリーCRISPR/Cas9システムが簡便で高効率、そしてモザイクとオフターゲットの少ないノックインマウス作成法であることを示している。さらに、最近、ノックインする鋳型としてプラスミドDNAより長鎖一本鎖DNAの方が効率が高いことが報告された<ref><pubmed>28511701</pubmed></ref>[31]。従って、現時点で最も高効率な外来遺伝子のノックインマウス作成法は、一本鎖DNAを鋳型として用いるクローニングフリーCRISPR/Cas9システムである。筆者らの研究室では、この方法を用いて約50%の効率でfloxedマウスやCreノックインマウスなどを作成している。 | |||
クローニングフリーCRISPR/Cas9システムを用いた外来遺伝子ノックインマウス作成法の欠点は、煩雑なターゲッティングベクターの作成が必要な点である。その点を改良したのが、PITCh(Precise Integration into Target Chromosomes)法である('''図6''')<ref><pubmed> 25410609</pubmed></ref>[32]。PITCh法は、相同組換やNHEJと異なるDNA二本鎖切断の修復機構であるマイクロホモロジー媒介末端結合(microhomology mediated end-joining; MMEJ)を利用した外来遺伝子のノックイン法である。MMEJは、DNA二本鎖切断の際に生じた切断末端間で、相補的配列(5〜25塩基対)同士で結合し、DNA二本鎖切断を修復する機構である。従来の相同組換えを利用した遺伝子挿入法では、挿入効率を上げるため外来遺伝子の両側に500〜1000塩基対の相同配列を付加したターゲッティングベクターを作成する必要があった。しかし、PITCh法では相同配列の長さが約20塩基対でよく、ターゲッティングベクターの作成が簡便化される。PITCh法では、ターゲッティングベクターにCRISPR/Cas9の認識配列とDNA二本鎖切断時に標的部位とベクターの切断末端で相補結合するような短い配列を外来遺伝子の両端に付加している。筆者らは最近、PITCh法、クローニングフリーCRISPR/Cas9システム、MMEJの効率を上げるexonuclease 1を組み合わせることにより、より簡便で高効率な外来遺伝子ノックインマウス作成法を開発した<ref><pubmed> 27894274</pubmed></ref>[33]。従来のCRISPR/Cas9システムを用いた標的部位への外来遺伝子のノックインは、相同組換えの活性に依存しおり、限られた生物種や細胞種にしか応用できなかった。しかし、PITCh法は、相同組換え活性の低い生物種や細胞種にも応用可能で、昆虫から哺乳類まで幅広く適応可能である。 | |||
===== 遺伝子改変非ヒト霊長類の作製 ===== | ===== 遺伝子改変非ヒト霊長類の作製 ===== | ||
ヒトの疾患、特に精神神経疾患のモデル動物としてマウスよりヒトと解剖学的、生理学的、遺伝学的に類似している非ヒト霊長類の疾患モデルが重要である。ゲノム編集技術を用いた標的遺伝子改変非ヒト霊長類の作成には、2014年に3つのグループが成功した<ref><pubmed>24486104</pubmed></ref> <ref><pubmed>24529597</pubmed></ref> <ref><pubmed>24838303</pubmed></ref>[34][35][36]。TALENを用いたアカゲザルとカニクイザルのMECP2遺伝子の破壊とCRISPR/Cas9システムを用いたカニクイザルの3つの遺伝子(Nr0b1, Ppar-, Rag1)の破壊が報告された。2015年には、CRISPR/Cas9システムを用い一本鎖のオリゴDNAによるp53遺伝子への塩基置換が報告された<ref><pubmed>25430965</pubmed></ref>[37]。2018年には、mCherryやGFPなどの標的部位へのノックインカニクイザルの作成が報告された<ref><pubmed> 29327726</pubmed></ref> <ref><pubmed>29327727</pubmed></ref>[38][39]。現在までに作成された精神疾患の非ヒト霊長類モデルは、レット症候群のモデルであるMecP2欠損カニクイザルと自閉症スペクトラム障害のモデルであるSHANK3欠損カニクイザルがあり、いずれも疾患の症状を再現している<ref><pubmed>28741620</pubmed></ref> <ref><pubmed>28525759</pubmed></ref>[40][41]。CRISPR/Cas9システムを用いることにより、忠実に疾患病態を再現した非ヒト霊長類の作製が可能になった。しかし、ゲノム編集の効率化、モザイクの抑制、オフターゲットの抑制など、まだ技術の改良が必要である。また、非ヒト霊長類をモデル動物として用いる場合、個体間のゲノム多様性も重要な問題になる。最近、カニクイザルで体細胞からのクローン作成が報告されたので<ref><pubmed>29395327</pubmed></ref>[42]、クローン技術とCRISPR/Casシステムを組み合わせることにより、より優れた精神神経疾患モデルを作出できると期待される。 | |||
==== 生体におけるゲノム編集 ==== | |||
生体における遺伝子ノックアウトは、標的遺伝子に対するガイドRNAとCas9のcDNAを、様々な臓器に導入し、非相同末端結合(NHEJ)を利用した修復により可能である。しかし、標的部位への遺伝子ノックインには、相同組換えを利用した修復が必要である。神経細胞などの非分裂細胞は、相同組換え活性が低く、従来の遺伝子ノックイン技術を適用することは難しい。最近、非分裂細胞に効率よく遺伝子をノックインできる技術が開発されたので、以下に2つの新技術を概説する。ゲノム編集に必要な核酸を生体に導入する方法として、アデノ随伴ウイルスベクターを用いる方法、電気穿孔法、ハイドロダイナッミク法などがある。ハイドロダイナッミク法は肝臓でのゲノム編集に用いられるが<ref>'''Ibraheim E, Song CQ, Mir A, Amrani N, Xue W, Sontheimer EJ.'''<br>All-in-One adeno-associated virus delivery and genome editing. <br>bioRxiv Posted April 4, 2018</ref>[43]、脳でのゲノム編集は主にアデノ随伴ウイルスベクターを用いる方法と電気穿孔法が用いられる。 | |||
生体におけるゲノム編集は、疾患の悪化に関与する遺伝子の破壊あるいは変異遺伝子の修復により、いままで治療法のなかった精神神経疾患に新しい治療法を提供する可能性を持っている。筋ジストロフィーの新しい治療法として、アンチセンスオリゴヌクレオチドを用いたexon skipping(変異のあるエクソンを飛ばして、多少短くなるがある程度機能のある原因遺伝子ジストロフィンを産生させる方法)が注目されている。しかし、exon skippingの効率や副作用など、多くの克服すべき課題が多い。そこで、アンチセンスオリゴヌクレオチドの代わりにCRISPR/Cas9を用いてexon skippingを起こさせる試みがデュシャンヌ型筋ジストロフィー(DMD)のモデルマウスを用いて行われた。米国の3つの研究グループは、ほぼ同時に、CRISPR/Cas9を使ってDMDモデルマウスのジストロフィン遺伝子の変異部分のみを切除し、機能的なジストロフィンを生成させ、筋力を回復させることに成功した<ref><pubmed>26721683</pubmed></ref> <ref><pubmed>26721684</pubmed></ref> <ref><pubmed>26721686</pubmed></ref>[44][45][46]。患者を対象とした臨床治験を始めるには、CRISPR/Cas9の効率向上・骨格筋へのデリバリー法の開発・免疫原性など克服すべき課題も多いが、in vivoでのゲノム編集が有効なことを示した成果である。 | |||
===== 非相同末端結合を利用した遺伝子ノックイン===== | ===== 非相同末端結合を利用した遺伝子ノックイン[47] ===== | ||
DNA二本鎖切断の主要な修復機構の一つであるNHEJによる修復は、細胞周期を通して効率が高いので、神経細胞などの分裂を行っていない細胞にも応用可能である。HITI(homology-independent targeted integration)法は、NHEJを利用した外来遺伝子のノックイン法である。ターゲッティングベクターを作成する際、挿入したい外来遺伝子の隣にノックインしたい標的部位と相同な20塩基対程度の配列を逆向きに付加する。この工夫により、標的部位に外来遺伝子が正しくノックインされない場合、Cas9によるDNA二本鎖切断が繰り返され、結果的にノックイン効率が向上する。HITI法を利用することにより、成体マウスの様々な臓器に外来遺伝子をノックインすることが可能になった。この方法を用い、網膜色素変性症モデルラットの疾患原因遺伝子が修復され、視覚障害の部分的な回復が見られた<ref><pubmed>27851729</pubmed></ref>[47]。この方法の欠点は、挿入部位周辺に塩基欠損や挿入を伴うこととターゲッティングベクターの余分な配列が挿入されことである。 | |||
===== 相同組換えを利用した遺伝子ノックイン ===== | ===== 相同組換えを利用した遺伝子ノックイン ===== | ||
最近、挿入する外来遺伝子をアデノ随伴ウイルスベクターを用い脳に導入することにより、相同組換えによるノックイン効率が高くなることが報告された<ref><pubmed>29056297</pubmed></ref>[48]。標的部位のDNA二本鎖切断に必要なガイドRNA、Cas9のcDNA、相同性を持つ外来遺伝子をアデノ随伴ウイルスベクターに組み込み、脳へ局所注入することにより、HITI法と同等な効率で狙った部位に遺伝子をノックインできる。 | |||
== ゲノム編集研究の今後 == | == ゲノム編集研究の今後 == | ||
CRISPR/Casシステムによるゲノム編集技術は、急速に進歩しており、従来困難であった変異遺伝子の修復や外来遺伝子の標的部位へのノックインが可能になりつつある。遺伝子異常が原因で起こる疾患の根本治療は、異常な遺伝子を正常に戻すことであり、ゲノム編集技術はそれを可能にする技術である。最近、CRISPR/Casシステムを用い、ヒト受精卵の遺伝子改変を行った論文が報告され、世界的に大きな問題となっている<ref><pubmed>25894090</pubmed></ref><ref><pubmed> 28251317</pubmed></ref><ref><pubmed>27052831</pubmed></ref><ref><pubmed>28783728</pubmed></ref><ref><pubmed>28875305</pubmed></ref><ref><pubmed>28942539</pubmed></ref>。CRISPR/Casシステムの技術開発および応用に関しては、環境(生物多様性)、安全性、倫理などの面からの議論が必要である。しかし、重要なことは、この技術が人類にもたらすリスクとメリットを客観的に分析し、感情に流されることなく、社会的コンセンサスを得ながら、応用の方向性を決めることである。さらに、この技術の開発スピードは早いので、社会的コンセンサス自体の不断な改変が必要不可欠である。 | CRISPR/Casシステムによるゲノム編集技術は、急速に進歩しており、従来困難であった変異遺伝子の修復や外来遺伝子の標的部位へのノックインが可能になりつつある。遺伝子異常が原因で起こる疾患の根本治療は、異常な遺伝子を正常に戻すことであり、ゲノム編集技術はそれを可能にする技術である。最近、CRISPR/Casシステムを用い、ヒト受精卵の遺伝子改変を行った論文が報告され、世界的に大きな問題となっている<ref><pubmed>25894090</pubmed></ref> <ref><pubmed> 28251317</pubmed></ref> <ref><pubmed>27052831</pubmed></ref> <ref><pubmed>28783728</pubmed></ref> <ref><pubmed>28875305</pubmed></ref> <ref><pubmed>28942539</pubmed></ref>[49][50][51][52][53][54]。CRISPR/Casシステムの技術開発および応用に関しては、環境(生物多様性)、安全性、倫理などの面からの議論が必要である。しかし、重要なことは、この技術が人類にもたらすリスクとメリットを客観的に分析し、感情に流されることなく、社会的コンセンサスを得ながら、応用の方向性を決めることである。さらに、この技術の開発スピードは早いので、社会的コンセンサス自体の不断な改変が必要不可欠である。 | ||
== 参考文献 == | == 参考文献 == | ||
<references/> | <references/> | ||
2018年7月22日 (日) 07:52時点における版
遺伝子は、生物の機能を規定する大きな要因の一つである。次世代シークエンサーの開発により、様々な生物種における遺伝子配列の情報や疾患に関与する遺伝子変異が明らかにされている。しかし、塩基配列を比較するだけでは、遺伝子の機能や疾患の病態を明らかにすることは難しい。ゲノム配列を自由に改変し、その影響を解析できて、初めて生命現象や疾患における遺伝子の役割を理解できる。ゲノム編集は、全ての生物・細胞の、全てのゲノム配列を自在に改変することである。この技術は、神経科学のみならず、多くの生命科学への応用が期待されている[1][1]。
(編集部コメント:この文章はむしろイントロにふさわしいものと思います。抄録では、全体の内容に関するサマリーを願い致します。)
ゲノム編集とは
原理
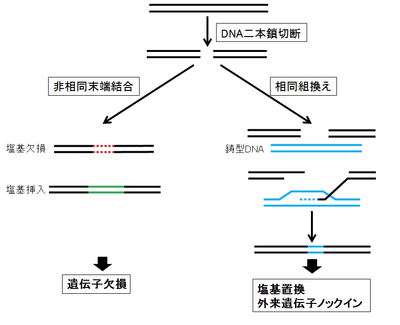
ゲノム編集は、狙ったゲノム部位にDNAの二本鎖切断を起こし、その後に誘導されるDNAの修復機構を利用し、標的ゲノムの破壊・塩基置換、標的ゲノム部位への外来遺伝子の挿入(ノックイン)などを可能にする技術である(図1)。
細胞にはDNA二本鎖切断に対する2つの主要な修復機構が存在する。
一つは、非相同末端結合(non-homologous end joining, NHEJ)であり、切断された末端同士を直接連結する。NHEJによる修復は、再連結の際、ヌクレオチドの欠失(数塩基から数百塩基)・挿入(数塩基から数十塩基)を高頻度で起こすため、修復の正確性は低い。従って、DNA二本鎖切断をタンパク質のコード領域に起こし、NHEJを利用しフレームシフトを起こすことにより遺伝子機能を破壊することができる。
もう一つの修復機構である相同組換えは、外部から導入した鋳型DNAを利用して正確な修復を行う。鋳型DNAに塩基置換や他の遺伝子を挿入することにより、標的ゲノムの塩基置換や外来遺伝子のノックインをすることができる。
さらに、ゲノム編集技術を用いると、同一染色体上の2箇所を切断することにより、大きな欠失や逆位、異なる染色体を切断することにより、染色体転座を起こすことができ、染色体の編集も可能である。NHEJによる修復は、細胞周期を通して作動するが、相同組換えによる修復はS期からG2期にしか起こらず頻度は低い。
ツール
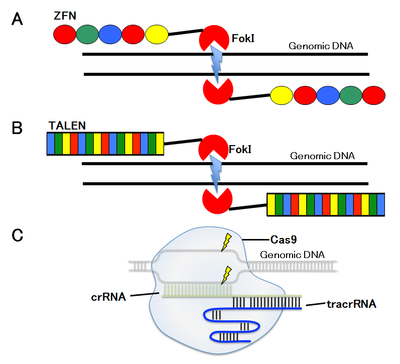
A. ZFN
B. TALEN
C.CRISPR/Cas8
ゲノム編集にとって最も重要なステップは、ゲノム上の狙った塩基配列にDNA二本鎖切断を導入することである。そのために、zinc-finger nuclease (ZFN)、transcription activator-like effector nuclease (TALEN)、clustered regularly interspaced short palindromic repeats (CRISPR)/CRISPR-associated proteins (Cas) CRISPR/Cas9、以下CRISPR/Casと略)などの部位特異的ヌクレアーゼを用いる(図2)。
1996年に報告されたZFNと2010年に報告されたTALENは、DNA二本鎖切断活性を持つFokIヌクレアーゼにDNA結合タンパク質のDNA結合ドメインを融合した一対の人工ヌクレアーゼを用い、狙った標的部位にDNA二本鎖切断を導入する。
第一世代のZFNは、DNA結合ドメインとしてzinc fingerを持つ人工ヌクレアーゼで、1つのzinc fingerは3塩基を認識するので、3〜6個のzinc fingerを持つZFNは9〜18 base pair (bp)に特異的に結合し、一対で18〜36 bpの特異性でDNA二本鎖切断を導入する。
第二世代のTALENは、DNA結合ドメインとして植物病原細菌のXanthomonas属が有するTALEを持つ人工ヌクレアーゼである。TALEのDNA結合ドメインは、1塩基を認識する34個のアミノ酸が一単位となり、それを15〜20単位持つTALENをセンス鎖、アンチセンス鎖それぞれに作製し、狙った標的部位にDNA二本鎖切断を導入する。
第三世代のCRISPR/Casは、単独でDNA二本鎖切断活性を持つCasヌクレアーゼと標的配列特異的一本鎖ガイドRNAとの複合体を用い、狙った塩基配列にDNA二本鎖切断を導入する。
この中で、2012年に発表されたCRISPR/Cas9は、その利便性、高効率、汎用性から、わずか1年の間に世界中で使われるゲノム編集の標準技術となった[2][2]。
CRISPR/Casシステム
CRISPR/Casシステムは、真正細菌や古細菌の獲得免疫系として発見された。この獲得免疫システムの標的は、細菌に感染するファージのDNAやRNAであり、異物として認識されたファージ由来のDNAやRNAは分解され除去される。CRISPR/Casシステムによる異物除去の過程は3つのステップ(adaptation, expression, interference)により行われる。侵入した外来DNAは、細菌内で断片化され、その一部が細菌のゲノム中のCRISPR領域に挿入される(adaptation)。次に外来DNAが侵入した際に、CRISPR領域が転写されてpre-CRISPR RNAが生じ、プロセシングを受けCRISPR RNA (crRNA:外来DNA断片と相補的配列を持つ)が生成される(expression)。プロセシングを受けたcrRNAはCasタンパク質と複合体を形成し、外来DNAやRNAと相補的に結合し、それらを切断する(interference)。
CRISPR/Casシステムは、システムを構成しているCasタンパク質群の違いにより2つのクラスに分類される。クラス1のCRISPR/Casシステムには複数のCasが、クラス2のCRISPR/Casシステムには単一のCasが関与する。さらに作用機序の違いにより、クラス1はⅠ型、Ⅲ型、Ⅳ型に分類され、クラス2はⅡ型、Ⅴ型、Ⅵ型に分類される。Casタンパク質—crRNA複合体は、DNAだけではなくRNAも標的にし、DNAおよびRNAの編集が可能である。
DNAの編集
CRISPR/Cas9システム
CRISPR/Cas9システムは、クラス2のⅡ型に分類されるCRISPR/Casシステムであり、CRISPR RNA (crRNA:外来DNA断片と相補的配列を持つ)、trans-activating crRNA (tracrRNA:crRNAの外来DNAと相補的配列以外の部分に結合し、Cas9とcrRNAの複合体形成に必要である)、Cas9タンパク質の3種類の要素から成っている(図2c)。Streptococcus pyogenes株由来のCas9タンパク質は、標的ゲノム配列の下流にある3つの塩基;N(G, A, T, or C)GGをPMA配列(Proto-spacer Adjacent Motif)として認識し、その3塩期上流を切断する。現在普及しているシステムは、標的DNAに対して相補的配列を持つcrRNAの3’末端にtracrRNAを連結させたsingle guide RNA (sgRNA)とCas9を発現させることにより、ゲノムDNA上の狙った部位にDNA二本鎖切断を導入する。
約100塩基のsgRNAのうち、DNA二本鎖切断の標的部位を規定するのは標的部位と相補的配列を持つ20塩基のみである。従って、CRISPR/Cas9システムをゲノム編集ツールとして利用する場合、標的ごとに変える必要があるのはわずか20塩基のみであり、それ以外の塩基配列およびCas9はすべて共通である。CRISPR/Cas9システムは、guide RNAの作製の簡便さ、guide RNAを増やすことにより複数遺伝子の同時編集が可能なことから、誰もが使うことのできるゲノム編集ツールとして急速に普及した。2012年の最初の発表以来、大腸菌、ヒト細胞からゼブラフィッシュに至る多くの細胞・生物種への応用が報告されている[3][3]。いまやヒトやサルを含むあらゆる動物個体、植物、微生物への利用が急速に広がっている。
ゲノム編集ツールとしてのCRISPR/Cas9システムの大きな問題点は、「オフターゲット」と「PAM配列の制約」である。オフターゲットとは、標的でないゲノム部位のDNA配列を変えてしまうことである。オフターゲットの起こる頻度は、細胞種・標的遺伝子座・guide RNAなどにより大きく変化する。オフターゲットを回避する方法として、ダブルニッキング法が考案されている。天然型のCas9は2つのヌクレアーゼドメインを持っているが、その一方をアミノ酸置換により不活性化した一本鎖切断型Cas9(Cas9 nickase)を用いる方法が考案されている[4][5][4][5]。標的部位に近接したセンス鎖、アンチセンス鎖に1対のCRISPR/Cas9 nickaseが結合した際にのみDNA二本鎖切断が誘導されるので、オフターゲットの起こる頻度は少なくなる。最近、Cas9 nickaseを用いた標的部位でのゲノム編集効率は、天然型のCas9編集効率と同等かそれ以上であることが報告されている[6][6]。また、CRISPR/Cas9を用いて作製された遺伝子改変マウスにおけるオフターゲットの頻度は、全ゲノムレベルで解析した例が少なく確定的ではないが、当初報告されたよりは少ないと考えられている[7] [8] [7][8]。
在ゲノム編集で最もよく使われているSpCas9はStreptococcus pyogenes由来であり、DNA二本鎖切断の部位を決めるには標的DNA配列の下流に隣接するNGGというPAM配列が必要である。このPAM配列の制約により、ゲノムの全ての場所を編集できないという制限があった。David Liuのグループは、phage-assisted continuous evolution (PACE)を利用して、NG、GAAおよびGATをPAMとするSpCas9変異体 (xCas9)の作成に成功した[9][9]。xCas9は哺乳類細胞において、最も広範なPAM配列を認識する制約の少ないCasである。さらに機序は不明であるが、xCas9はオフターゲットの頻度も抑制し、Cas9の主要な欠点であるオフターゲットとPAM配列の制約の2つを回避できる理想的なゲノム編集ツールである。
CRISPR/Cpf1システム
Cpf1 (Cas12a)は、クラス2Ⅴ型のCRISPR/Casシステムに関わるDNAエンドヌクレアーゼであり、新たなゲノム編集ツールとして注目されている[10][10]。Cpf1はCas9とは異なる以下のような特徴を持っている。
- Cpf1はgRNAとしてcrRNAのみを必要とし、tracrRNAは必要ない。
- Cpf1はCas9と異なり、PAM配列としてTTTV (VはA, C, G)、TTCV, TCTV, CTTVを認識する。
- Cas9はDNA二本鎖を切断し平滑末端を形成するが、Cpf1は突出末端を形成する。
CRISPR/Cpf1システムは、ヒト細胞株やマウス受精卵のゲノム編集に応用され、CRISPR/Cas9システムよりオフターゲットの頻度が少ないことが報告されている[11][12][11][12]。
CRISPR/dCAS9-BEシステム
従来のゲノム編集は、標的のゲノム部位にDNAの二本鎖切断を起こし、その後に誘導されるDNAの修復機構を利用し、標的DNAを編集する。
一方CRISPR/dCAS9-BEシステムは、DNAを切断することなく標的DNAの塩基を編集する方法である。ヌクレアーゼ活性を失活させたCas9(dCas9)に、脱アミノ化酵素であるシチジンデアミナーゼを融合させた塩基エディター(BE)を作成し、guide RNAにより狙ったゲノム部位に塩基エディターを働かせ、標的部位のシトシン(C)をチミン(T)(あるいはグアニン(G)をアデニン(A))に置換する[13][14][13][14]。さらにDavid Liuのグループは、PACEを利用してDNAのAをG(あるいはTをC)に置換できる転移RNAのアデノシンデアミナーゼ変異体(アデニン塩基エディター(ABE))の作成に成功した[15][15]。dCas9と融合したBEあるいはABEを用いることにより、DNAの二本鎖切断を起こさずにDNAの4塩基全てを個別に置き換えられる。既知の遺伝性疾患の原因となる一塩基変異の約50%は、G-C塩基対からA-T塩基対への転移なので、CRISPR/dCas-ABEシステムは遺伝性疾患を根本的に治す可能性を持っている。
RNAの編集
CRISPR/Cas9システムやCRISPR/Cpf1システムの標的はDNAである。DNAと同様にRNAは生命現象において重要な役割を担っており、RNAを標的にした編集技術は、生命科学に大きな貢献をする。
CRISPR/Cas13(C2c2)システム
Feng Zhangのグループは微生物ゲノムデータベースを探索し、クラス2タイプⅥ型CRISPR/CasシステムのCas13(C2c2)が、標的RNAに相補的なRNA依存性に一本鎖RNAを切断する酵素であることを見つけた[16][16]。Leptotrichia wadei由来のCas13a (LwaCas13a)をCRISPRシステムに組み込んだ系は、標的RNAを高効率かつ高い特異性でノックダウンすることができる[17][17]。
CRISPR/Cas13aを用いた標的RNAのノックダウンはRNA干渉法(RNAi)に比べ、
- オフターゲットが少ない
- 長鎖ノンコーディングRNAの発現を抑制できる
などの利点がある。
さらに、失活させたCas13a (dCas13a)に蛍光タンパク質を融合させることにより、目的のRNAを可視化することができる[17][17]。
最近、Cas13aより高効率かつ高い特異性で標的RNAをノックダウンすることができるCas13bが同定された[18][18]。Cas13は、pre-CRISPR RNAをプロセッシングしcrRNAを生成できる活性を持っており、多数の標的RNAを含んだpre-CRISPR RNAをguide RNAとして用いることにより、一度に多くのRNAをノックダウンできる[17][17]。
CRISPR/dCas13-ADARシステム
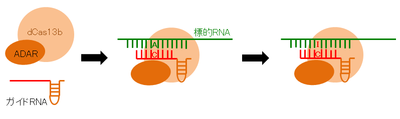
Feng Zhangのグループは、失活させたCas13b (dCas13b)にRNAデアミナーゼ(RNAのアデニン(A)をイノシン(I)に変化させる酵素,ADAR)を融合させ、標的RNAを編集できる系を確立した(RNA Editing for Programmable A to I Replacement (REPAIR)図3)[18][18]。この系は、RNAの変異を正常に戻すことができる。さらに、RNAを標的にした編集はオフターゲットが起きてもその影響は一過性であり、遺伝子治療としては安全性が高い。
CRISPR/Casシステムの神経科学への応用
細胞への応用[19]
CRISPR/Casシステムを細胞に適用することにより、遺伝子の機能を欠損させたり亢進させたりすることが簡単にでき、様々な細胞機能に関与する遺伝子群をゲノムワイドに検索できる。[19]
ゲノム編集
神経細胞などの初代培養細胞は、一般的にCRISPR/Casシステムによるゲノム編集効率は低く、それぞれの細胞種による最適化が必要である。ES細胞やiPS細胞におけるCRISPR/Casシステムを用いたゲノム編集の効率化は、多くの実績がある。従って、ゲノム編集したES細胞やiPS細胞を用い、それらから分化させた神経細胞の機能を解析する方法も有効である。
また、CRISPR/Casシステムの開発により、疾患原因遺伝子の変異以外は遺伝的に同一(isogenic)なiPS細胞を作成することが効率化された。iPS細胞におけるゲノム編集は、従来の相同組換えを用いた方法では変異導入効率が低く、クローン化に多大な労力が必要とされた。しかし、CRISPR/Casシステムの開発により、iPS細胞のゲノム編集がより効率化され、単純な遺伝子欠損に加え、疾患の原因と考えられる様々な変異(1塩基置換や大きな欠失など)を導入することが可能になった。従って、樹立された患者iPS細胞の疾患原因遺伝子の変異を正常に戻したり、健常人から樹立したiPS細胞に疾患の原因遺伝子変異を導入することによりisogenicな疾患モデル細胞を作成することが可能になった[20][20]。今後、isogenicなiPS細胞を用いることにより、疾患研究が加速すると期待される。
転写制御
CRISPR/Casシステムは、RNA誘導型ヌクレアーゼであり、ガイドRNAが標的ゲノム部位にCasを誘導する。Casに点変異を入れ、ガイドRNAとは複合体を形成できるがヌクレアーゼ活性を持たないCas を作成することができる。この不活性型Cas(dCas9やdCpf1)に、様々な活性を持つ蛋白質を融合することにより、融合蛋白質を標的ゲノム部位に誘導することができる。転写活性化因子Vp64あるいは転写抑制因子KRABをdCas9やdCpf1と融合し、標的ゲノム部位と相補的配列を持つガイドRNAと伴に細胞に導入すると、標的遺伝子の転写を活性化あるいは抑制することができ、遺伝子の機能を解析することができる[21] [22][21][22]。しかし、一つのdCasに対し一つの転写調節因子を結合させても遺伝子転写制御は不十分であり、通常は、dCasまたはガイドRNAに複数の転写調節因子を付加する系が使われている[23][23]。この系は、一つの細胞内の複数の遺伝子の転写を同時に制御することができる。
エピゲノム制御
ヒストンの修飾やDNAのメチル化などのエピゲノミックな修飾は、脳形成や神経可塑性において重要な役割をもち、さらに、精神神経疾患の発症に関与することが報告されている。CRISPR/Casシステムを用いることにより、従来困難であった特定のゲノム領域のエピゲノミックな修飾状態を改変する「エピゲノム編集」が可能になった[24][24]。ヒストンはメチル化やアセチル化などの翻訳後修飾を受け、転写の制御やクロマチン濃縮などに関与する。dCas9にヒストン修飾を導入する酵素(ヒストンアセチル基転移酵素、ヒストン脱アセチル化酵素、リジンメチル基転移酵素、リジン脱メチル化酵素など)を融合させた人工タンパク質と標的ゲノム部位と相補的配列を持つガイドRNAを細胞に導入すると、標的遺伝子のエピゲノミックな修飾状態を改変できる。また、DNAを構成する4種類の塩基のなかでシトシンのみがメチル基の付加・除去を受け、転写を制御している。dCas9にDNAメチル化を制御する酵素(DNAメチル化酵素やDNA脱メチル化酵素)を融合させた人工タンパク質と標的ゲノム部位と相補的配列を持つガイドRNAを細胞に導入すると、標的ゲノム領域のDNAのメチル化状態を改変できる。しかし、一つのdCas9に対し一つのエピゲノム修飾因子を結合させてもエピゲノム制御は不十分であり、通常は、dCas9またはガイドRNAに複数のエピゲノム修飾因子を付加する系が使われている[25][25]。Rudolf Jaenischのグループは、dCas9にTet1(シトシンのメチル基を除去する酵素)を融合させた人工タンパク質を用い、Fragile X症候群のiPS細胞モデルの治療に成功している[26][26]。Fragile X症候群では、FMR1遺伝子の転写開始より少し上流にあるCGGの繰り返し配列が増加しメチル化が促進され、FMR1遺伝子の発現が抑制されている。Fragile X症候群の患者さんから作成したiPS細胞に、FMR1遺伝子の転写開始より少し上流を標的にしたガイドRNAとdCas9-tet1を導入し、CGGの繰り返し配列のメチル基を外し、FMR1遺伝子の発現を回復させることに成功した。FMR1遺伝子の発現が回復した患者iPS細胞を神経細胞に分化させると、神経細胞の過活動が正常に戻った。
RNA制御
CRISPR/Cas13bシステムを用いることにより、標的RNAを高効率かつ高い特異性でノックダウンすることができる。全ての遺伝子を標的とする大規模なCRISPR sgRNAライブラリーが構築されている。このライブラリーを用い、細胞を利用してゲノムワイドなRNAのノックダウンが可能なため、良いアッセイ系さえ組めば、様々な細胞内プロセスに関与する遺伝子群を同定することができる。このシステムは特異性が高いので、特定のスプライシングアイソフォームのみをノックダウンすることも可能である。さらに、CRISPR/dCas13-ADARシステムを用いることにより、遺伝子変異をRNAレベルで正常に戻すことも可能である。
個体への応用
受精卵におけるゲノム編集
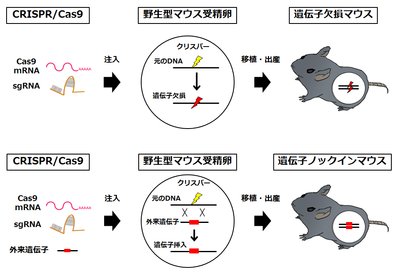
従来、遺伝子改変動物は、ES細胞の相同組換えによる遺伝子改変を基盤として作成されてきた。しかし、その作成には長い時間、多大な労力、多額の費用が必要である。さらに、遺伝子改変動物の作成は、マウス等、ES細胞が樹立されているごく一部の動物種に限られていた。CRISPR/Cas9システムは、この状況を一変させた。CRISPR/Cas9システムを用いた遺伝子改変動物の作成は、標的配列に対するガイドRNA、Cas9をコードするmRNAおよびノックインの場合にはドナーDNAを受精卵に注入するだけで、受精卵内で標的遺伝子が改変され、短時間にノックアウト・ノックイン動物を得ることができる(図4)。さらに最近、従来の「顕微注入法」で必要とされる受精卵の単離、顕微注入、移植といった一連の作業を省略できるGONAD(Genome-editing via Oviductal Nucleic Acids Delivery)法が報告された[27][27]。GONAD法では、0.7日胚(着床前)を有する妊娠雌マウスの卵管内に標的配列に対するガイドRNA、Cas9をコードするmRNAを注入し、卵管全体に対し直接電気穿孔法を行う。CRISPR/Cas9システムを用いた遺伝子改変動物の作製法は、そのきわめて迅速で簡便で高い効率性が特徴である。以下に、遺伝子欠損・塩基置換・外来遺伝子ノックイン動物の作成法を、マウスを中心に概説する。
遺伝子欠損マウスの作製
標的配列に対するガイドRNA、Cas9をコードするmRNAを野生型受精卵に注入する(図4)。注意点としては、標的遺伝子に3の倍数の塩基欠損や挿入が起こることがあり、その際にはフレームシフトが起こらず、標的遺伝子の機能破壊の有無を何らかの方法で確認する必要がある。CRISPR/Cas9を用いた遺伝子欠損マウスの作成は効率が高く(通常50〜100%)、複数の遺伝子を同時に欠損させることもできる[28][28]。
塩基置換マウスの作製
塩基置換する配列が数十塩基以内であれば、一本鎖のオリゴDNAを標的配列に対するガイドRNA、Cas9をコードするmRNAと伴に野生型受精卵に注入する(図4)。CRISPR/Cas9を用いた塩基置換マウスの作成は効率が高く、複数の遺伝子に同時に変異を導入することもできる[28][28]。
外来遺伝子ノックインマウスの作製
遺伝子改変マウスの作成を容易にしたCRISPR/Cas9であるが、数kbの長い外来遺伝子のノックインマウスの作製は困難である。Rudolf Jaenischらにより外来遺伝子のノックインマウス作製の成功が報告されているが、その作成効率は10%程度であり、遺伝子欠損や塩基置換ノックインと比べて低い[29][29]。数kbの外来遺伝子のノックインは、蛍光タンパク質やCreリコンビナーゼ等の機能カセットを標的部位に挿入したり、floxedマウスを作成するのに必要であり、生命科学にとって欠かすことのできない重要な技術である。このため、ノックインマウス作製の効率化は、CRISPR/Cas9を用いたゲノム編集にとって大きな課題の一つであった。
従来、CRISPR/Cas9を用いたゲノム編集では、Cas9およびガイドRNAをともにRNAの形で受精卵の前核に顕微注入している(図5)。従来法でCas9がヌクレアーゼとして前核内で働くためには、まず注入されたCas9 mRNAが前核から細胞質へ輸送され、蛋白質に翻訳され、さらに翻訳された蛋白質が再度、前核に移行し、標的部位に到達、切断を行う必要がある。この過程は時間を要する。外来遺伝子のノックインに必要な相同組換えは、細胞周期のS期からG2期にのみ起こることから、その効率を上げるためには、Cas9は受精卵に注入後できるだけ早くDNA二本鎖切断活性を持つ必要がある。
そこで筆者らは、Cas9をmRNAではなく蛋白質として受精卵前核に注入することで、ノックイン効率を向上できるのではないかと考えた。実際、培養細胞ではCas9をmRNAではなく蛋白質として導入することにより、DNA二本鎖切断が迅速に起こることが示されている。さらに、外来遺伝子のノックイン効率を向上させるため、Cas9の標的部位切断効率を上げる工夫をした。細菌の獲得免疫系としてのCRISPR/Cas9システムは、Cas9と2種類のガイドRNA(標的部位と相補的な配列を持つをcrRNAとCas9およびcrRNAの橋渡しを担うtracrRNA)からなる3要素のシステムである(図2C)。
しかし従来のCRISPR/Cas9システムは、実験操作を簡便にするためcrRNAとtracrRNAを連結した一本鎖ガイドRNA(sgRNA)とCas9の2要素からなるシステムである(図5)。自然界の3要素システムの方が、2要素システムより、高い標的配列切断活性を持つことが報告されている。そこで筆者らは、sgRNAのかわりにcrRNAとtracrRNAを用い、Cas9蛋白質と組み合わせることで、ノックイン効率を向上できるのではないかと考えた。crRNAとtracrRNAを用いるもう一つの利点は、化学合成が可能になり、sgRNAの作製に必要な大腸菌での遺伝子組換え実験を省略できる点である。sgRNAの長さは約100塩基であるが、crRNA、tracrRNAは各々50塩基程度であり、化学合成が可能である。筆者らの改良したCRISPR/Cas9システムは、Cas9蛋白質と化学合成したcrRNAとtracrRNAの3要素からなる(図5)。このシステムは、大腸菌での遺伝子組換え実験を行うことなくゲノム編集が可能で、クローニングフリーCRISPR/Cas9システムと名づけた[30][30]。
このクローニングフリーCRISPR/Cas9システムは、ガイドRNAの活性評価も簡便である。従来のような培養細胞を用いた実験は必要なく、試験管内で標的配列を含むPCR産物、Cas9蛋白質、crRNA、tracrRNAをインキュベートして電気泳動するだけで(in vitro digestion assay:IDA)、その切断活性を調べることができる。クローニングフリーCRISPR/Cas9システムを用い、Act(actin beta)遺伝子座にEGFP(enhanced green fluorescent protein)を含む2.5 kbの外来遺伝子をノックインするマウスを作成したところ、およそ50%の新生仔マウスにEGFPが目的部位にノックインされていた。従来用いられてきたCas9 mRNAとsgRNAからなる2要素システムを用い対照実験を行ったところ、その効率は10%程度だった。このことからクローニングフリーCRISPR/Cas9システムは、外来遺伝子のノックイン効率を大幅に向上させることが明らかになった[30][30]。さらに作製したノックインマウスを野生型マウスと交配し、次世代への伝達効率を調べたところ、すべての系統から、50%の効率で次世代のノックインマウスが得られた。このことは、従来型CRISPR/Cas9システムで問題となるモザイク(同一個体内の一部の細胞のみに遺伝子改変が起こっている)の頻度が低いことを示している。つまり、クローニングフリーCRISPR/Cas9システムでは、標的部位のDNA二本鎖切断が迅速に起こり、受精卵の第一卵割までに片アリルに外来遺伝子がノックインされたことを示している。またオフターゲット変異の候補となる部位を解析したところ、いずれのノックインマウスでも変異は検出されなかった。このことは、クローニングフリーCRISPR/Cas9システムでは、Cas9を蛋白質として注入したことによりCas9の半減期が短くなり、標的部位を切断した後迅速に分解されるため、従来型CRISPR/Cas9システムの大きな課題であるオフターゲット変異(ガイドRNA配列に類似した配列の非特異的切断)が大幅に減少することを示している。
以上の結果は、クローニングフリーCRISPR/Cas9システムが簡便で高効率、そしてモザイクとオフターゲットの少ないノックインマウス作成法であることを示している。さらに、最近、ノックインする鋳型としてプラスミドDNAより長鎖一本鎖DNAの方が効率が高いことが報告された[31][31]。従って、現時点で最も高効率な外来遺伝子のノックインマウス作成法は、一本鎖DNAを鋳型として用いるクローニングフリーCRISPR/Cas9システムである。筆者らの研究室では、この方法を用いて約50%の効率でfloxedマウスやCreノックインマウスなどを作成している。
クローニングフリーCRISPR/Cas9システムを用いた外来遺伝子ノックインマウス作成法の欠点は、煩雑なターゲッティングベクターの作成が必要な点である。その点を改良したのが、PITCh(Precise Integration into Target Chromosomes)法である(図6)[32][32]。PITCh法は、相同組換やNHEJと異なるDNA二本鎖切断の修復機構であるマイクロホモロジー媒介末端結合(microhomology mediated end-joining; MMEJ)を利用した外来遺伝子のノックイン法である。MMEJは、DNA二本鎖切断の際に生じた切断末端間で、相補的配列(5〜25塩基対)同士で結合し、DNA二本鎖切断を修復する機構である。従来の相同組換えを利用した遺伝子挿入法では、挿入効率を上げるため外来遺伝子の両側に500〜1000塩基対の相同配列を付加したターゲッティングベクターを作成する必要があった。しかし、PITCh法では相同配列の長さが約20塩基対でよく、ターゲッティングベクターの作成が簡便化される。PITCh法では、ターゲッティングベクターにCRISPR/Cas9の認識配列とDNA二本鎖切断時に標的部位とベクターの切断末端で相補結合するような短い配列を外来遺伝子の両端に付加している。筆者らは最近、PITCh法、クローニングフリーCRISPR/Cas9システム、MMEJの効率を上げるexonuclease 1を組み合わせることにより、より簡便で高効率な外来遺伝子ノックインマウス作成法を開発した[33][33]。従来のCRISPR/Cas9システムを用いた標的部位への外来遺伝子のノックインは、相同組換えの活性に依存しおり、限られた生物種や細胞種にしか応用できなかった。しかし、PITCh法は、相同組換え活性の低い生物種や細胞種にも応用可能で、昆虫から哺乳類まで幅広く適応可能である。
遺伝子改変非ヒト霊長類の作製
ヒトの疾患、特に精神神経疾患のモデル動物としてマウスよりヒトと解剖学的、生理学的、遺伝学的に類似している非ヒト霊長類の疾患モデルが重要である。ゲノム編集技術を用いた標的遺伝子改変非ヒト霊長類の作成には、2014年に3つのグループが成功した[34] [35] [36][34][35][36]。TALENを用いたアカゲザルとカニクイザルのMECP2遺伝子の破壊とCRISPR/Cas9システムを用いたカニクイザルの3つの遺伝子(Nr0b1, Ppar-, Rag1)の破壊が報告された。2015年には、CRISPR/Cas9システムを用い一本鎖のオリゴDNAによるp53遺伝子への塩基置換が報告された[37][37]。2018年には、mCherryやGFPなどの標的部位へのノックインカニクイザルの作成が報告された[38] [39][38][39]。現在までに作成された精神疾患の非ヒト霊長類モデルは、レット症候群のモデルであるMecP2欠損カニクイザルと自閉症スペクトラム障害のモデルであるSHANK3欠損カニクイザルがあり、いずれも疾患の症状を再現している[40] [41][40][41]。CRISPR/Cas9システムを用いることにより、忠実に疾患病態を再現した非ヒト霊長類の作製が可能になった。しかし、ゲノム編集の効率化、モザイクの抑制、オフターゲットの抑制など、まだ技術の改良が必要である。また、非ヒト霊長類をモデル動物として用いる場合、個体間のゲノム多様性も重要な問題になる。最近、カニクイザルで体細胞からのクローン作成が報告されたので[42][42]、クローン技術とCRISPR/Casシステムを組み合わせることにより、より優れた精神神経疾患モデルを作出できると期待される。
生体におけるゲノム編集
生体における遺伝子ノックアウトは、標的遺伝子に対するガイドRNAとCas9のcDNAを、様々な臓器に導入し、非相同末端結合(NHEJ)を利用した修復により可能である。しかし、標的部位への遺伝子ノックインには、相同組換えを利用した修復が必要である。神経細胞などの非分裂細胞は、相同組換え活性が低く、従来の遺伝子ノックイン技術を適用することは難しい。最近、非分裂細胞に効率よく遺伝子をノックインできる技術が開発されたので、以下に2つの新技術を概説する。ゲノム編集に必要な核酸を生体に導入する方法として、アデノ随伴ウイルスベクターを用いる方法、電気穿孔法、ハイドロダイナッミク法などがある。ハイドロダイナッミク法は肝臓でのゲノム編集に用いられるが[43][43]、脳でのゲノム編集は主にアデノ随伴ウイルスベクターを用いる方法と電気穿孔法が用いられる。
生体におけるゲノム編集は、疾患の悪化に関与する遺伝子の破壊あるいは変異遺伝子の修復により、いままで治療法のなかった精神神経疾患に新しい治療法を提供する可能性を持っている。筋ジストロフィーの新しい治療法として、アンチセンスオリゴヌクレオチドを用いたexon skipping(変異のあるエクソンを飛ばして、多少短くなるがある程度機能のある原因遺伝子ジストロフィンを産生させる方法)が注目されている。しかし、exon skippingの効率や副作用など、多くの克服すべき課題が多い。そこで、アンチセンスオリゴヌクレオチドの代わりにCRISPR/Cas9を用いてexon skippingを起こさせる試みがデュシャンヌ型筋ジストロフィー(DMD)のモデルマウスを用いて行われた。米国の3つの研究グループは、ほぼ同時に、CRISPR/Cas9を使ってDMDモデルマウスのジストロフィン遺伝子の変異部分のみを切除し、機能的なジストロフィンを生成させ、筋力を回復させることに成功した[44] [45] [46][44][45][46]。患者を対象とした臨床治験を始めるには、CRISPR/Cas9の効率向上・骨格筋へのデリバリー法の開発・免疫原性など克服すべき課題も多いが、in vivoでのゲノム編集が有効なことを示した成果である。
非相同末端結合を利用した遺伝子ノックイン[47]
DNA二本鎖切断の主要な修復機構の一つであるNHEJによる修復は、細胞周期を通して効率が高いので、神経細胞などの分裂を行っていない細胞にも応用可能である。HITI(homology-independent targeted integration)法は、NHEJを利用した外来遺伝子のノックイン法である。ターゲッティングベクターを作成する際、挿入したい外来遺伝子の隣にノックインしたい標的部位と相同な20塩基対程度の配列を逆向きに付加する。この工夫により、標的部位に外来遺伝子が正しくノックインされない場合、Cas9によるDNA二本鎖切断が繰り返され、結果的にノックイン効率が向上する。HITI法を利用することにより、成体マウスの様々な臓器に外来遺伝子をノックインすることが可能になった。この方法を用い、網膜色素変性症モデルラットの疾患原因遺伝子が修復され、視覚障害の部分的な回復が見られた[47][47]。この方法の欠点は、挿入部位周辺に塩基欠損や挿入を伴うこととターゲッティングベクターの余分な配列が挿入されことである。
相同組換えを利用した遺伝子ノックイン
最近、挿入する外来遺伝子をアデノ随伴ウイルスベクターを用い脳に導入することにより、相同組換えによるノックイン効率が高くなることが報告された[48][48]。標的部位のDNA二本鎖切断に必要なガイドRNA、Cas9のcDNA、相同性を持つ外来遺伝子をアデノ随伴ウイルスベクターに組み込み、脳へ局所注入することにより、HITI法と同等な効率で狙った部位に遺伝子をノックインできる。
ゲノム編集研究の今後
CRISPR/Casシステムによるゲノム編集技術は、急速に進歩しており、従来困難であった変異遺伝子の修復や外来遺伝子の標的部位へのノックインが可能になりつつある。遺伝子異常が原因で起こる疾患の根本治療は、異常な遺伝子を正常に戻すことであり、ゲノム編集技術はそれを可能にする技術である。最近、CRISPR/Casシステムを用い、ヒト受精卵の遺伝子改変を行った論文が報告され、世界的に大きな問題となっている[49] [50] [51] [52] [53] [54][49][50][51][52][53][54]。CRISPR/Casシステムの技術開発および応用に関しては、環境(生物多様性)、安全性、倫理などの面からの議論が必要である。しかし、重要なことは、この技術が人類にもたらすリスクとメリットを客観的に分析し、感情に流されることなく、社会的コンセンサスを得ながら、応用の方向性を決めることである。さらに、この技術の開発スピードは早いので、社会的コンセンサス自体の不断な改変が必要不可欠である。
参考文献
- ↑
Aida, T., Imahashi, R., & Tanaka, K. (2014).
Translating human genetics into mouse: the impact of ultra-rapid in vivo genome editing. Development, growth & differentiation, 56(1), 34-45. [PubMed:24444057] [WorldCat] [DOI] - ↑
Cai, M., & Yang, Y. (2014).
Targeted genome editing tools for disease modeling and gene therapy. Current gene therapy, 14(1), 2-9. [PubMed:24665839] [WorldCat] - ↑
Doudna, J.A., & Charpentier, E. (2014).
Genome editing. The new frontier of genome engineering with CRISPR-Cas9. Science (New York, N.Y.), 346(6213), 1258096. [PubMed:25430774] [WorldCat] [DOI] - ↑
Ran, F.A., Hsu, P.D., Lin, C.Y., Gootenberg, J.S., Konermann, S., Trevino, A.E., ..., & Zhang, F. (2013).
Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell, 154(6), 1380-9. [PubMed:23992846] [PMC] [WorldCat] [DOI] - ↑
Yee, J.K. (2016).
Off-target effects of engineered nucleases. The FEBS journal, 283(17), 3239-48. [PubMed:27208701] [WorldCat] [DOI] - ↑
Gopalappa, R., Suresh, B., Ramakrishna, S., & Kim, H.H. (2018).
Paired D10A Cas9 nickases are sometimes more efficient than individual nucleases for gene disruption. Nucleic acids research, 46(12), e71. [PubMed:29584876] [PMC] [WorldCat] [DOI] - ↑ CRISPR off-targets: a reassessment.
Nature Methods. 2018, 15(4):229-30. doi:10.1038/nmeth.4664 - ↑ Schaefer KA, Darbo BW, Colgan DF, Tsang SH, Bassuk AG, Mahajan VB.
Corrigendum and follow-up: Whole genome sequencing of multiple CRISPR-edited mouse lines suggests no excess mutations.
bioRxiv. 2017, Posted Jun. 23. Doi: http://dx.org/10.1101/154450 - ↑
Hu, J.H., Miller, S.M., Geurts, M.H., Tang, W., Chen, L., Sun, N., ..., & Liu, D.R. (2018).
Evolved Cas9 variants with broad PAM compatibility and high DNA specificity. Nature, 556(7699), 57-63. [PubMed:29512652] [PMC] [WorldCat] [DOI] - ↑
Zetsche, B., Gootenberg, J.S., Abudayyeh, O.O., Slaymaker, I.M., Makarova, K.S., Essletzbichler, P., ..., & Zhang, F. (2015).
Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 163(3), 759-71. [PubMed:26422227] [PMC] [WorldCat] [DOI] - ↑
Kleinstiver, B.P., Tsai, S.Q., Prew, M.S., Nguyen, N.T., Welch, M.M., Lopez, J.M., ..., & Joung, J.K. (2016).
Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells. Nature biotechnology, 34(8), 869-74. [PubMed:27347757] [PMC] [WorldCat] [DOI] - ↑
Kim, Y., Cheong, S.A., Lee, J.G., Lee, S.W., Lee, M.S., Baek, I.J., & Sung, Y.H. (2016).
Generation of knockout mice by Cpf1-mediated gene targeting. Nature biotechnology, 34(8), 808-10. [PubMed:27272387] [WorldCat] [DOI] - ↑
Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A., & Liu, D.R. (2016).
Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 533(7603), 420-4. [PubMed:27096365] [PMC] [WorldCat] [DOI] - ↑
Nishida, K., Arazoe, T., Yachie, N., Banno, S., Kakimoto, M., Tabata, M., ..., & Kondo, A. (2016).
Targeted nucleotide editing using hybrid prokaryotic and vertebrate adaptive immune systems. Science (New York, N.Y.), 353(6305). [PubMed:27492474] [WorldCat] [DOI] - ↑
Gaudelli, N.M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson, D.I., & Liu, D.R. (2017).
Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature, 551(7681), 464-471. [PubMed:29160308] [PMC] [WorldCat] [DOI] - ↑
Abudayyeh, O.O., Gootenberg, J.S., Konermann, S., Joung, J., Slaymaker, I.M., Cox, D.B., ..., & Zhang, F. (2016).
C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science (New York, N.Y.), 353(6299), aaf5573. [PubMed:27256883] [PMC] [WorldCat] [DOI] - ↑ 17.0 17.1 17.2
Abudayyeh, O.O., Gootenberg, J.S., Essletzbichler, P., Han, S., Joung, J., Belanto, J.J., ..., & Zhang, F. (2017).
RNA targeting with CRISPR-Cas13. Nature, 550(7675), 280-284. [PubMed:28976959] [PMC] [WorldCat] [DOI] - ↑ 18.0 18.1
Cox, D.B.T., Gootenberg, J.S., Abudayyeh, O.O., Franklin, B., Kellner, M.J., Joung, J., & Zhang, F. (2017).
RNA editing with CRISPR-Cas13. Science (New York, N.Y.), 358(6366), 1019-1027. [PubMed:29070703] [PMC] [WorldCat] [DOI] - ↑
Barrangou, R., & Doudna, J.A. (2016).
Applications of CRISPR technologies in research and beyond. Nature biotechnology, 34(9), 933-941. [PubMed:27606440] [WorldCat] [DOI] - ↑
Grobarczyk, B., Franco, B., Hanon, K., & Malgrange, B. (2015).
Generation of Isogenic Human iPS Cell Line Precisely Corrected by Genome Editing Using the CRISPR/Cas9 System. Stem cell reviews and reports, 11(5), 774-87. [PubMed:26059412] [WorldCat] [DOI] - ↑
Gilbert, L.A., Larson, M.H., Morsut, L., Liu, Z., Brar, G.A., Torres, S.E., ..., & Qi, L.S. (2013).
CRISPR-mediated modular RNA-guided regulation of transcription in eukaryotes. Cell, 154(2), 442-51. [PubMed:23849981] [PMC] [WorldCat] [DOI] - ↑
Liu, Y., Han, J., Chen, Z., Wu, H., Dong, H., & Nie, G. (2017).
Engineering cell signaling using tunable CRISPR-Cpf1-based transcription factors. Nature communications, 8(1), 2095. [PubMed:29235474] [PMC] [WorldCat] [DOI] - ↑
Vora, S., Tuttle, M., Cheng, J., & Church, G. (2016).
Next stop for the CRISPR revolution: RNA-guided epigenetic regulators. The FEBS journal, 283(17), 3181-93. [PubMed:27248712] [WorldCat] [DOI] - ↑
Pulecio, J., Verma, N., Mejía-Ramírez, E., Huangfu, D., & Raya, A. (2017).
CRISPR/Cas9-Based Engineering of the Epigenome. Cell stem cell, 21(4), 431-447. [PubMed:28985525] [PMC] [WorldCat] [DOI] - ↑
Morita, S., Noguchi, H., Horii, T., Nakabayashi, K., Kimura, M., Okamura, K., ..., & Hatada, I. (2016).
Targeted DNA demethylation in vivo using dCas9-peptide repeat and scFv-TET1 catalytic domain fusions. Nature biotechnology, 34(10), 1060-1065. [PubMed:27571369] [WorldCat] [DOI] - ↑
Liu, X.S., Wu, H., Krzisch, M., Wu, X., Graef, J., Muffat, J., ..., & Jaenisch, R. (2018).
Rescue of Fragile X Syndrome Neurons by DNA Methylation Editing of the FMR1 Gene. Cell, 172(5), 979-992.e6. [PubMed:29456084] [PMC] [WorldCat] [DOI] - ↑
Ohtsuka, M., Sato, M., Miura, H., Takabayashi, S., Matsuyama, M., Koyano, T., ..., & Gurumurthy, C.B. (2018).
i-GONAD: a robust method for in situ germline genome engineering using CRISPR nucleases. Genome biology, 19(1), 25. [PubMed:29482575] [PMC] [WorldCat] [DOI] - ↑ 28.0 28.1
Wang, H., Yang, H., Shivalila, C.S., Dawlaty, M.M., Cheng, A.W., Zhang, F., & Jaenisch, R. (2013).
One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell, 153(4), 910-8. [PubMed:23643243] [PMC] [WorldCat] [DOI] - ↑
Yang, H., Wang, H., Shivalila, C.S., Cheng, A.W., Shi, L., & Jaenisch, R. (2013).
One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell, 154(6), 1370-9. [PubMed:23992847] [PMC] [WorldCat] [DOI] - ↑ 30.0 30.1
Aida, T., Chiyo, K., Usami, T., Ishikubo, H., Imahashi, R., Wada, Y., ..., & Tanaka, K. (2015).
Cloning-free CRISPR/Cas system facilitates functional cassette knock-in in mice. Genome biology, 16, 87. [PubMed:25924609] [PMC] [WorldCat] [DOI] - ↑
Quadros, R.M., Miura, H., Harms, D.W., Akatsuka, H., Sato, T., Aida, T., ..., & Gurumurthy, C.B. (2017).
Easi-CRISPR: a robust method for one-step generation of mice carrying conditional and insertion alleles using long ssDNA donors and CRISPR ribonucleoproteins. Genome biology, 18(1), 92. [PubMed:28511701] [PMC] [WorldCat] [DOI] - ↑
Nakade, S., Tsubota, T., Sakane, Y., Kume, S., Sakamoto, N., Obara, M., ..., & Suzuki, K.T. (2014).
Microhomology-mediated end-joining-dependent integration of donor DNA in cells and animals using TALENs and CRISPR/Cas9. Nature communications, 5, 5560. [PubMed:25410609] [PMC] [WorldCat] [DOI] - ↑
Aida, T., Nakade, S., Sakuma, T., Izu, Y., Oishi, A., Mochida, K., ..., & Tanaka, K. (2016).
Gene cassette knock-in in mammalian cells and zygotes by enhanced MMEJ. BMC genomics, 17(1), 979. [PubMed:27894274] [PMC] [WorldCat] [DOI] - ↑
Niu, Y., Shen, B., Cui, Y., Chen, Y., Wang, J., Wang, L., ..., & Sha, J. (2014).
Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell, 156(4), 836-43. [PubMed:24486104] [WorldCat] [DOI] - ↑
Liu, H., Chen, Y., Niu, Y., Zhang, K., Kang, Y., Ge, W., ..., & Ji, W. (2014).
TALEN-mediated gene mutagenesis in rhesus and cynomolgus monkeys. Cell stem cell, 14(3), 323-328. [PubMed:24529597] [PMC] [WorldCat] [DOI] - ↑
Liu, Z., Zhou, X., Zhu, Y., Chen, Z.F., Yu, B., Wang, Y., ..., & Qiu, Z. (2014).
Generation of a monkey with MECP2 mutations by TALEN-based gene targeting. Neuroscience bulletin, 30(3), 381-6. [PubMed:24838303] [PMC] [WorldCat] [DOI] - ↑
Wan, H., Feng, C., Teng, F., Yang, S., Hu, B., Niu, Y., ..., & Zhou, Q. (2015).
One-step generation of p53 gene biallelic mutant Cynomolgus monkey via the CRISPR/Cas system. Cell research, 25(2), 258-61. [PubMed:25430965] [PMC] [WorldCat] [DOI] - ↑
Yao, X., Liu, Z., Wang, X., Wang, Y., Nie, Y.H., Lai, L., ..., & Yang, H. (2018).
Generation of knock-in cynomolgus monkey via CRISPR/Cas9 editing. Cell research, 28(3), 379-382. [PubMed:29327726] [PMC] [WorldCat] [DOI] - ↑
Cui, Y., Niu, Y., Zhou, J., Chen, Y., Cheng, Y., Li, S., ..., & Ji, W. (2018).
Generation of a precise Oct4-hrGFP knockin cynomolgus monkey model via CRISPR/Cas9-assisted homologous recombination. Cell research, 28(3), 383-386. [PubMed:29327727] [PMC] [WorldCat] [DOI] - ↑
Zhao, H., Tu, Z., Xu, H., Yan, S., Yan, H., Zheng, Y., ..., & Zhang, Y.Q. (2017).
Altered neurogenesis and disrupted expression of synaptic proteins in prefrontal cortex of SHANK3-deficient non-human primate. Cell research, 27(10), 1293-1297. [PubMed:28741620] [PMC] [WorldCat] [DOI] - ↑
Chen, Y., Yu, J., Niu, Y., Qin, D., Liu, H., Li, G., ..., & Sun, Y.E. (2017).
Modeling Rett Syndrome Using TALEN-Edited MECP2 Mutant Cynomolgus Monkeys. Cell, 169(5), 945-955.e10. [PubMed:28525759] [PMC] [WorldCat] [DOI] - ↑
Liu, Z., Cai, Y., Wang, Y., Nie, Y., Zhang, C., Xu, Y., ..., & Sun, Q. (2018).
Cloning of Macaque Monkeys by Somatic Cell Nuclear Transfer. Cell, 172(4), 881-887.e7. [PubMed:29395327] [WorldCat] [DOI] - ↑ Ibraheim E, Song CQ, Mir A, Amrani N, Xue W, Sontheimer EJ.
All-in-One adeno-associated virus delivery and genome editing.
bioRxiv Posted April 4, 2018 - ↑
Long, C., Amoasii, L., Mireault, A.A., McAnally, J.R., Li, H., Sanchez-Ortiz, E., ..., & Olson, E.N. (2016).
Postnatal genome editing partially restores dystrophin expression in a mouse model of muscular dystrophy. Science (New York, N.Y.), 351(6271), 400-3. [PubMed:26721683] [PMC] [WorldCat] [DOI] - ↑
Nelson, C.E., Hakim, C.H., Ousterout, D.G., Thakore, P.I., Moreb, E.A., Castellanos Rivera, R.M., ..., & Gersbach, C.A. (2016).
In vivo genome editing improves muscle function in a mouse model of Duchenne muscular dystrophy. Science (New York, N.Y.), 351(6271), 403-7. [PubMed:26721684] [PMC] [WorldCat] [DOI] - ↑
Tabebordbar, M., Zhu, K., Cheng, J.K.W., Chew, W.L., Widrick, J.J., Yan, W.X., ..., & Wagers, A.J. (2016).
In vivo gene editing in dystrophic mouse muscle and muscle stem cells. Science (New York, N.Y.), 351(6271), 407-411. [PubMed:26721686] [PMC] [WorldCat] [DOI] - ↑
Suzuki, K., Tsunekawa, Y., Hernandez-Benitez, R., Wu, J., Zhu, J., Kim, E.J., ..., & Belmonte, J.C. (2016).
In vivo genome editing via CRISPR/Cas9 mediated homology-independent targeted integration. Nature, 540(7631), 144-149. [PubMed:27851729] [PMC] [WorldCat] [DOI] - ↑
Nishiyama, J., Mikuni, T., & Yasuda, R. (2017).
Virus-Mediated Genome Editing via Homology-Directed Repair in Mitotic and Postmitotic Cells in Mammalian Brain. Neuron, 96(4), 755-768.e5. [PubMed:29056297] [PMC] [WorldCat] [DOI] - ↑
Liang, P., Xu, Y., Zhang, X., Ding, C., Huang, R., Zhang, Z., ..., & Huang, J. (2015).
CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein & cell, 6(5), 363-372. [PubMed:25894090] [PMC] [WorldCat] [DOI] - ↑
Tang, L., Zeng, Y., Du, H., Gong, M., Peng, J., Zhang, B., ..., & Liu, J. (2017).
CRISPR/Cas9-mediated gene editing in human zygotes using Cas9 protein. Molecular genetics and genomics : MGG, 292(3), 525-533. [PubMed:28251317] [WorldCat] [DOI] - ↑
Kang, X., He, W., Huang, Y., Yu, Q., Chen, Y., Gao, X., ..., & Fan, Y. (2016).
Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. Journal of assisted reproduction and genetics, 33(5), 581-588. [PubMed:27052831] [PMC] [WorldCat] [DOI] - ↑
Ma, H., Marti-Gutierrez, N., Park, S.W., Wu, J., Lee, Y., Suzuki, K., ..., & Mitalipov, S. (2017).
Correction of a pathogenic gene mutation in human embryos. Nature, 548(7668), 413-419. [PubMed:28783728] [WorldCat] [DOI] - ↑
Zhou, C., Zhang, M., Wei, Y., Sun, Y., Sun, Y., Pan, H., ..., & Chen, Z.J. (2017).
Highly efficient base editing in human tripronuclear zygotes. Protein & cell, 8(10), 772-775. [PubMed:28875305] [PMC] [WorldCat] [DOI] - ↑
Liang, P., Ding, C., Sun, H., Xie, X., Xu, Y., Zhang, X., ..., & Huang, J. (2017).
Correction of β-thalassemia mutant by base editor in human embryos. Protein & cell, 8(11), 811-822. [PubMed:28942539] [PMC] [WorldCat] [DOI]