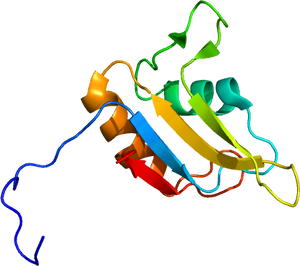
Fused in sarcoma
石垣診祐
名古屋大学大学院医学系研究科総合医学専攻 脳神経病態制御学 神経内科学分野
DOI:10.14931/bsd.9576 原稿受付日:2020年12月17日 原稿完成日:2021年1月18日
担当編集委員:山中 宏二(名古屋大学 環境医学研究所 病態神経科学)
英略称:FUS
Fused in sarcoma (FUS)はTLS(translocated in liposarcoma)とも称されるRNA結合タンパク質で、主として核内に局在し転写、選択的スプライシング、RNA輸送などRNA代謝全般に機能する分子である。Chopなどとの融合タンパク質が癌の原因遺伝子となることで発見され命名されたが、近年は筋萎縮性側索硬化症(ALS)の原因遺伝子および病態関連分子として注目されている。
Fused in sarcomaとは
Fused in sarcoma (FUS)は脂肪肉腫などの癌において、転座に伴う融合タンパク質(FUS-CHOP)として同定された[1][2][3]。N末端のFUSと、C末端のCHOPなどの様々な転写因子が融合することで非常に強力な転写活性を獲得し、癌化に寄与するものと考えられる[4][5]。
これとは別に、FUSはpre-mRNAの成熟に関わる複合体のサブユニットhnRNP P2としても同定された[6]。そして2009年に2つのグループから家族性ALSの原因遺伝子であることが報告され[7][8]、神経研究の分野で大きな注目を集めることになった。それ以降、TDP-43を含む多くのRNA結合タンパク質と同様にALSおよびその類縁疾患である前頭側頭葉変性症 (FTLD) の病態に強く関係することが明らかにされてきた。
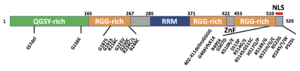
家族性ALSで認められた変異を示す。
2LA6による
構造
N末端にQGSY-rich領域、高度に保存されているRNA認識モチーフ(RRM)、アルギニン残基のジメチル化が特徴であるRGGリピート、およびC末端のzinc finger motifの構造を持つ(図1)。タンパク質立体構造については、RNA認識モチーフ(RRM)がRNAのステムループ部分に、C末端のzinc finger (ZnF) motifがRNAのGGU配列を認識して結合することが報告されている(図2)[9]。
サブファミリー
FUSはFET protein familyに含まれる。FET protein family にはEWS、TAF15も含まれるが、どれも家族性ALSの原因遺伝子であることがわかっている[4][10][11]。これら3つのFET protein familyはドメイン構造が共通である[2][5][10][12]。
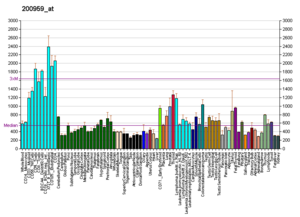
Wikipediaより。元データーは[13]による。
組織分布、細胞内分布
FUSはユビキタスな組織分布をとり、細胞内では主に核に局在するが、一部は細胞質すなわち神経細胞の軸索や樹状突起にも局在する(The Human Protein Atlas)。
機能
In vitro解析で,FUSはRNAおよび一本鎖DNAに結合性を示すものの二本鎖DNAにはほとんど結合しない[2][14][15][16]。実際の生体内におけるFUSのRNA結合部位は複数のグループからFUSのCLIP-seq解析結果が報告されており、主にFUSはスプライシングを受ける前のpre-mRNAに結合し,特に選択的スプライシングを受けるexon周囲や,選択的転写開始・終結点を持つ領域に結合する。FUS認識RNAモチーフは明瞭ではないが,GUリッチな配列に指向性が認められ、FUS結合領域ではRNAは二次構造をとりやすい[17][18][19][20][21]。
FUSは転写制御に関与していることが知られており[22]、合成途中のRNAへの結合を介してRNAポリメラーゼIIの転写速度を減弱させる。FUSのPrion like domainは,線維状の構造物を作ってRNAポリメラーゼIIのC末端領域と結合し、RNP顆粒の形成に関わっている[17][21][23][24]。FUSはRNA結合を介し,積極的にポリアデニル化制御を行っており、mRNA長制御を中心にRNAプロセシングに深く関与し、神経分化やシナプス形成に関係する遺伝子の制御に関わると推測される[25][26][27][28]。
一方、FUSはspliceosomeに含まれ、SFPQ、NONO、hnRNPA1、TDP-43、SMN、他のFET proteinなど多くのRNPと結合し選択的スプライシングに関与することが知られている[22][25][29][30][31][32]。FUSはMAPT、Camk2a、およびFmr1といった神経変性に関連する分子などの選択的スプライシングを制御する[18][19][20][33][34]。とくに、タウの遺伝子であるMAPTはexon10の制御はタウの代表的アイソフォームである3-repeat tauと4-repeat tauの生成に直結し、FUSの機能低下が結果としてタウのisoformである4-repeat tauが増加させることが複数のグループから報告されている[18][33][34][35]。
また、FUSはDNA修復機能に関与することが数多く報告されている。FUSはHDAC1への結合を介して神経細胞におけるDNA損傷に関与することや、PARP依存的なDNA修復への機能がALS関連変異で障害されることなどが知られている[36][37][38][39]。
FUSは核だけではなく、細胞質すなわち樹状突起や軸索内にも存在してRNA輸送や局所翻訳、軸索ガイダンスなどの制御を介して神経細胞の機能維持に働いている[40][41][42]。
FUSの全身ノックアウトマウスは背景により異なったフェノタイプを示す。C57BL/6の近交系では免疫系の異常により出生してすぐに死に至る一方で、非近交系では精子形成異常以外はほぼ正常に生育することが報告されている[43][44]。
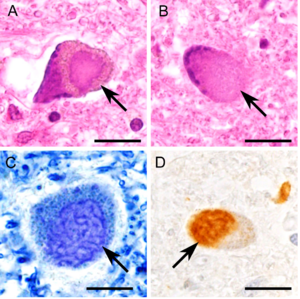
68歳男性、臨床診断ALS、全経過6年。
A. ヘマトキシリン・エオジン(HE)染色では境界明瞭な好塩基性の細胞質内封入体がみられる。
B. 境界不明瞭で染色の淡い封入体も多いので、注意する。
C. 好塩基性封入体は、Klüver-Barrera染色で観察しやすい。内部に線条を呈する封入体として観察される。
D. 好塩基性封入体は抗FUS免疫組織化学で標識される。
愛知医科大学加齢医科学研究所の陸雄一先生による。スケール=10 μm。
疾患との関わり
FUSは家族性ALS(ALS6)の原因遺伝子であり、家族性ALS の原因遺伝子として、わが国ではSOD1に次いで頻度が高い遺伝子と考えられており家族性ALSのうち4-9%を占める。ほとんどの症例は優性遺伝(顕性遺伝)形式である。変異はC末側に多く認められほとんどがpoint mutationである(図1)[45]。病理学的にはFUSが核内から細胞質に局在が変化してユビキチン陽性の細胞質封入体として神経細胞内に認められることが特徴である。FUSに変異が存在しない孤発性のALSにおいても、このようなFUSの病理所見が陽性の症例が存在し、塩基性封入体が特徴であることからbasophilic inclusion body disease (BIBD)と呼ばれる。ALSは認知症の1つであるFTLDと臨床的、病理学的、遺伝学的に同一の疾患スペクトラムを形成すると考えられ、FTLD症例にもFUS陽性の病理像を示すグループが存在し、FTLD-FUSと称される。
FUS変異による家族性ALSの中には、発達障害や精神疾患を併発するものあること、FUSの変異によって非特異的なタウオパチーを呈する症例があること、核内でのFUSの局在変化がALS, FTLDのみならず、4Rタウオパチーである進行性核上性麻痺(PSP)や大脳皮質基底核変性症(CBD)症例でも認められること、などからFUSはより広範な神経精神疾患のメカニズムに関わる可能性が示唆されている[46][47][48][49]。
FUSが神経変性や神経機能障害を引き起こすメカニズムについては毒性獲得、機能喪失など様々な仮説が提唱されているが、未だ多くが不明のままである。TDP-43のモデルと同様に、FUSの過剰発現モデルでALS様のフェノタイプを示すことがこれまで知られており、これは毒性獲得仮説の根拠となっている[50][51]。単純な過剰発現ではフェノタイプを呈さないこともあるが、FUSの発現は転写、翻訳のレベルで自己制御されているため、自己制御が破綻すると毒性獲得に至るものと推測されている[52]。
一方で、運動神経特異的にFUSをKOしてもALSにはならないことから、機能喪失のALSへの関与は限定的と考えられているが[50][53]、大脳の神経細胞での機能喪失はFTLD様の高次脳機能障害を引き起こすことが、マウス、ゼブラフィッシュやショウジョウバエなどのFUS喪失動物モデルから明らかにされている[27][30][54][55][56]。ALS/FTLDを含むFTLDスペクトラムで核内でのFUSとSFPQの結合性低下が存在することなどを考え合わせると[46]、前述のようにFUSの機能は大脳の神経細胞にも重要であり広範な神経疾患メカニズムに関わるものと推定される。
FUSの病態との関わりについて特筆すべきは、液-液相分離(liquid-liquid phase separation: LLPS)との関連である。液-液相分離は狭義にはin vitroで観察される現象を示し、特定の高分子が膜の存在しない集合体を形成する現象を称する。FUSは精製後にタグから切断するとすぐに液滴 (droplet)を形成し、その液-液相分離は変異やリン酸化、RNA濃度など様々な条件で変遷する。またそのことが核膜を通じたFUSの細胞局在変化に関与することなどが明らかにされ、病態におけるストレス顆粒や細胞質封入体、FUS複合体との関連性にも注目を集めており、FUS以外の多くのRNA結合タンパク質についても同様に液-液相分離が病態に関わる可能性が示唆されている[57][58][59][60][61][62][63]。しかしながら、神経変性疾患の封入体を含めて疾患の病理所見にどのように関連するのかについては、直接的な証拠が乏しいためにまだ明らかではない。
近年、FUSが関連するALS/FTLDがこれまでに報告されてきたFUS陽性のALS/FTLDという範疇にとどまらず、より広い範囲のALS/FTLDに関与することを示唆する報告がなされており[46][64][65][66]、詳細な液-液相分離と疾患の病態との直接的な関連が明らかにされることで、FUSの構造、機能に着目した新たな疾患バイオマーカーや治療法の標的が見いだされることが期待される。
関連語
参考文献
- ↑
Rabbitts, T.H., Forster, A., Larson, R., & Nathan, P. (1993).
Fusion of the dominant negative transcription regulator CHOP with a novel gene FUS by translocation t(12;16) in malignant liposarcoma. Nature genetics, 4(2), 175-80. [PubMed:7503811] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2
Crozat, A., Aman, P., Mandahl, N., & Ron, D. (1993).
Fusion of CHOP to a novel RNA-binding protein in human myxoid liposarcoma. Nature, 363(6430), 640-4. [PubMed:8510758] [WorldCat] [DOI] - ↑
Eneroth, M., Mandahl, N., Heim, S., Willén, H., Rydholm, A., Alberts, K.A., & Mitelman, F. (1990).
Localization of the chromosomal breakpoints of the t(12;16) in liposarcoma to subbands 12q13.3 and 16p11.2. Cancer genetics and cytogenetics, 48(1), 101-7. [PubMed:2372777] [WorldCat] [DOI] - ↑ 4.0 4.1
Bertolotti, A., Bell, B., & Tora, L. (1999).
The N-terminal domain of human TAFII68 displays transactivation and oncogenic properties. Oncogene, 18(56), 8000-10. [PubMed:10637511] [WorldCat] [DOI] - ↑ 5.0 5.1
Prasad, D.D., Ouchida, M., Lee, L., Rao, V.N., & Reddy, E.S. (1994).
TLS/FUS fusion domain of TLS/FUS-erg chimeric protein resulting from the t(16;21) chromosomal translocation in human myeloid leukemia functions as a transcriptional activation domain. Oncogene, 9(12), 3717-29. [PubMed:7970732] [WorldCat] - ↑
Calvio, C., Neubauer, G., Mann, M., & Lamond, A.I. (1995).
Identification of hnRNP P2 as TLS/FUS using electrospray mass spectrometry. RNA (New York, N.Y.), 1(7), 724-33. [PubMed:7585257] [PMC] [WorldCat] - ↑
Kwiatkowski, T.J., Bosco, D.A., Leclerc, A.L., Tamrazian, E., Vanderburg, C.R., Russ, C., ..., & Brown, R.H. (2009).
Mutations in the FUS/TLS gene on chromosome 16 cause familial amyotrophic lateral sclerosis. Science (New York, N.Y.), 323(5918), 1205-8. [PubMed:19251627] [WorldCat] [DOI] - ↑
Vance, C., Rogelj, B., Hortobágyi, T., De Vos, K.J., Nishimura, A.L., Sreedharan, J., ..., & Shaw, C.E. (2009).
Mutations in FUS, an RNA processing protein, cause familial amyotrophic lateral sclerosis type 6. Science (New York, N.Y.), 323(5918), 1208-1211. [PubMed:19251628] [PMC] [WorldCat] [DOI] - ↑
Loughlin, F.E., Lukavsky, P.J., Kazeeva, T., Reber, S., Hock, E.M., Colombo, M., ..., & Allain, F.H. (2019).
The Solution Structure of FUS Bound to RNA Reveals a Bipartite Mode of RNA Recognition with Both Sequence and Shape Specificity. Molecular cell, 73(3), 490-504.e6. [PubMed:30581145] [WorldCat] [DOI] - ↑ 10.0 10.1
Morohoshi, F., Ootsuka, Y., Arai, K., Ichikawa, H., Mitani, S., Munakata, N., & Ohki, M. (1998).
Genomic structure of the human RBP56/hTAFII68 and FUS/TLS genes. Gene, 221(2), 191-8. [PubMed:9795213] [WorldCat] [DOI] - ↑
Svetoni, F., Frisone, P., & Paronetto, M.P. (2016).
Role of FET proteins in neurodegenerative disorders. RNA biology, 13(11), 1089-1102. [PubMed:27415968] [PMC] [WorldCat] [DOI] - ↑
Iko, Y., Kodama, T.S., Kasai, N., Oyama, T., Morita, E.H., Muto, T., ..., & Morikawa, K. (2004).
Domain architectures and characterization of an RNA-binding protein, TLS. The Journal of biological chemistry, 279(43), 44834-40. [PubMed:15299008] [WorldCat] [DOI] - ↑
Su, A.I., Wiltshire, T., Batalov, S., Lapp, H., Ching, K.A., Block, D., ..., & Hogenesch, J.B. (2004).
A gene atlas of the mouse and human protein-encoding transcriptomes. Proceedings of the National Academy of Sciences of the United States of America, 101(16), 6062-7. [PubMed:15075390] [PMC] [WorldCat] [DOI] - ↑
Wang, X., Arai, S., Song, X., Reichart, D., Du, K., Pascual, G., ..., & Kurokawa, R. (2008).
Induced ncRNAs allosterically modify RNA-binding proteins in cis to inhibit transcription. Nature, 454(7200), 126-30. [PubMed:18509338] [PMC] [WorldCat] [DOI] - ↑
Baechtold, H., Kuroda, M., Sok, J., Ron, D., Lopez, B.S., & Akhmedov, A.T. (1999).
Human 75-kDa DNA-pairing protein is identical to the pro-oncoprotein TLS/FUS and is able to promote D-loop formation. The Journal of biological chemistry, 274(48), 34337-42. [PubMed:10567410] [WorldCat] [DOI] - ↑
Zinszner, H., Sok, J., Immanuel, D., Yin, Y., & Ron, D. (1997).
TLS (FUS) binds RNA in vivo and engages in nucleo-cytoplasmic shuttling. Journal of cell science, 110 ( Pt 15), 1741-50. [PubMed:9264461] [WorldCat] - ↑ 17.0 17.1
Masuda, A., & Ohno, K. (2016).
[Regulation of mRNA length by FUS]. Seikagaku. The Journal of Japanese Biochemical Society, 88(2), 244-7. [PubMed:27192881] [WorldCat] - ↑ 18.0 18.1 18.2
Ishigaki, S., Masuda, A., Fujioka, Y., Iguchi, Y., Katsuno, M., Shibata, A., ..., & Ohno, K. (2012).
Position-dependent FUS-RNA interactions regulate alternative splicing events and transcriptions. Scientific reports, 2, 529. [PubMed:22829983] [PMC] [WorldCat] [DOI] - ↑ 19.0 19.1
Lagier-Tourenne, C., Polymenidou, M., Hutt, K.R., Vu, A.Q., Baughn, M., Huelga, S.C., ..., & Yeo, G.W. (2012).
Divergent roles of ALS-linked proteins FUS/TLS and TDP-43 intersect in processing long pre-mRNAs. Nature neuroscience, 15(11), 1488-97. [PubMed:23023293] [PMC] [WorldCat] [DOI] - ↑ 20.0 20.1
Rogelj, B., Easton, L.E., Bogu, G.K., Stanton, L.W., Rot, G., Curk, T., ..., & Ule, J. (2012).
Widespread binding of FUS along nascent RNA regulates alternative splicing in the brain. Scientific reports, 2, 603. [PubMed:22934129] [PMC] [WorldCat] [DOI] - ↑ 21.0 21.1
Lerga, A., Hallier, M., Delva, L., Orvain, C., Gallais, I., Marie, J., & Moreau-Gachelin, F. (2001).
Identification of an RNA binding specificity for the potential splicing factor TLS. The Journal of biological chemistry, 276(9), 6807-16. [PubMed:11098054] [WorldCat] [DOI] - ↑ 22.0 22.1
Coady, T.H., & Manley, J.L. (2015).
ALS mutations in TLS/FUS disrupt target gene expression. Genes & development, 29(16), 1696-706. [PubMed:26251528] [PMC] [WorldCat] [DOI] - ↑
Masuda, A., Takeda, J., Okuno, T., Okamoto, T., Ohkawara, B., Ito, M., ..., & Ohno, K. (2015).
Position-specific binding of FUS to nascent RNA regulates mRNA length. Genes & development, 29(10), 1045-57. [PubMed:25995189] [PMC] [WorldCat] [DOI] - ↑
Schwartz, J.C., Wang, X., Podell, E.R., & Cech, T.R. (2013).
RNA seeds higher-order assembly of FUS protein. Cell reports, 5(4), 918-25. [PubMed:24268778] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1
Sun, S., Ling, S.C., Qiu, J., Albuquerque, C.P., Zhou, Y., Tokunaga, S., ..., & Cleveland, D.W. (2015).
ALS-causative mutations in FUS/TLS confer gain and loss of function by altered association with SMN and U1-snRNP. Nature communications, 6, 6171. [PubMed:25625564] [PMC] [WorldCat] [DOI] - ↑
Yamazaki, T., Chen, S., Yu, Y., Yan, B., Haertlein, T.C., Carrasco, M.A., ..., & Reed, R. (2012).
FUS-SMN protein interactions link the motor neuron diseases ALS and SMA. Cell reports, 2(4), 799-806. [PubMed:23022481] [PMC] [WorldCat] [DOI] - ↑ 27.0 27.1
Udagawa, T., Fujioka, Y., Tanaka, M., Honda, D., Yokoi, S., Riku, Y., ..., & Sobue, G. (2015).
FUS regulates AMPA receptor function and FTLD/ALS-associated behaviour via GluA1 mRNA stabilization. Nature communications, 6, 7098. [PubMed:25968143] [PMC] [WorldCat] [DOI] - ↑
Yokoi, S., Udagawa, T., Fujioka, Y., Honda, D., Okado, H., Watanabe, H., ..., & Sobue, G. (2017).
3'UTR Length-Dependent Control of SynGAP Isoform α2 mRNA by FUS and ELAV-like Proteins Promotes Dendritic Spine Maturation and Cognitive Function. Cell reports, 20(13), 3071-3084. [PubMed:28954225] [WorldCat] [DOI] - ↑
An, H., Skelt, L., Notaro, A., Highley, J.R., Fox, A.H., La Bella, V., ..., & Shelkovnikova, T.A. (2019).
ALS-linked FUS mutations confer loss and gain of function in the nucleus by promoting excessive formation of dysfunctional paraspeckles. Acta neuropathologica communications, 7(1), 7. [PubMed:30642400] [PMC] [WorldCat] [DOI] - ↑ 30.0 30.1
Ishigaki, S., Fujioka, Y., Okada, Y., Riku, Y., Udagawa, T., Honda, D., ..., & Sobue, G. (2017).
Altered Tau Isoform Ratio Caused by Loss of FUS and SFPQ Function Leads to FTLD-like Phenotypes. Cell reports, 18(5), 1118-1131. [PubMed:28147269] [WorldCat] [DOI] - ↑
Kahl, A., Blanco, I., Jackman, K., Baskar, J., Milaganur Mohan, H., Rodney-Sandy, R., ..., & Hochrainer, K. (2018).
Cerebral ischemia induces the aggregation of proteins linked to neurodegenerative diseases. Scientific reports, 8(1), 2701. [PubMed:29426953] [PMC] [WorldCat] [DOI] - ↑
Tsuiji, H., Iguchi, Y., Furuya, A., Kataoka, A., Hatsuta, H., Atsuta, N., ..., & Yamanaka, K. (2013).
Spliceosome integrity is defective in the motor neuron diseases ALS and SMA. EMBO molecular medicine, 5(2), 221-34. [PubMed:23255347] [PMC] [WorldCat] [DOI] - ↑ 33.0 33.1
Orozco, D., & Edbauer, D. (2013).
FUS-mediated alternative splicing in the nervous system: consequences for ALS and FTLD. Journal of molecular medicine (Berlin, Germany), 91(12), 1343-54. [PubMed:23974990] [WorldCat] [DOI] - ↑ 34.0 34.1
Fujioka, Y., Ishigaki, S., Masuda, A., Iguchi, Y., Udagawa, T., Watanabe, H., ..., & Sobue, G. (2013).
FUS-regulated region- and cell-type-specific transcriptome is associated with cell selectivity in ALS/FTLD. Scientific reports, 3, 2388. [PubMed:23925123] [PMC] [WorldCat] [DOI] - ↑
Orozco, D., Tahirovic, S., Rentzsch, K., Schwenk, B.M., Haass, C., & Edbauer, D. (2012).
Loss of fused in sarcoma (FUS) promotes pathological Tau splicing. EMBO reports, 13(8), 759-64. [PubMed:22710833] [PMC] [WorldCat] [DOI] - ↑
Naumann, M., Pal, A., Goswami, A., Lojewski, X., Japtok, J., Vehlow, A., ..., & Hermann, A. (2018).
Impaired DNA damage response signaling by FUS-NLS mutations leads to neurodegeneration and FUS aggregate formation. Nature communications, 9(1), 335. [PubMed:29362359] [PMC] [WorldCat] [DOI] - ↑
Singatulina, A.S., Hamon, L., Sukhanova, M.V., Desforges, B., Joshi, V., Bouhss, A., ..., & Pastré, D. (2019).
PARP-1 Activation Directs FUS to DNA Damage Sites to Form PARG-Reversible Compartments Enriched in Damaged DNA. Cell reports, 27(6), 1809-1821.e5. [PubMed:31067465] [WorldCat] [DOI] - ↑
Wang, H., Guo, W., Mitra, J., Hegde, P.M., Vandoorne, T., Eckelmann, B.J., ..., & Hegde, M.L. (2018).
Mutant FUS causes DNA ligation defects to inhibit oxidative damage repair in Amyotrophic Lateral Sclerosis. Nature communications, 9(1), 3683. [PubMed:30206235] [PMC] [WorldCat] [DOI] - ↑
Qiu, H., Lee, S., Shang, Y., Wang, W.Y., Au, K.F., Kamiya, S., ..., & Huang, E.J. (2014).
ALS-associated mutation FUS-R521C causes DNA damage and RNA splicing defects. The Journal of clinical investigation, 124(3), 981-99. [PubMed:24509083] [PMC] [WorldCat] [DOI] - ↑
Fujii, R., & Takumi, T. (2005).
TLS facilitates transport of mRNA encoding an actin-stabilizing protein to dendritic spines. Journal of cell science, 118(Pt 24), 5755-65. [PubMed:16317045] [WorldCat] [DOI] - ↑
Sephton, C.F., Tang, A.A., Kulkarni, A., West, J., Brooks, M., Stubblefield, J.J., ..., & Yu, G. (2014).
Activity-dependent FUS dysregulation disrupts synaptic homeostasis. Proceedings of the National Academy of Sciences of the United States of America, 111(44), E4769-78. [PubMed:25324524] [PMC] [WorldCat] [DOI] - ↑
Errichelli, L., Dini Modigliani, S., Laneve, P., Colantoni, A., Legnini, I., Capauto, D., ..., & Bozzoni, I. (2017).
FUS affects circular RNA expression in murine embryonic stem cell-derived motor neurons. Nature communications, 8, 14741. [PubMed:28358055] [PMC] [WorldCat] [DOI] - ↑
Hicks, G.G., Singh, N., Nashabi, A., Mai, S., Bozek, G., Klewes, L., ..., & Ruley, H.E. (2000).
Fus deficiency in mice results in defective B-lymphocyte development and activation, high levels of chromosomal instability and perinatal death. Nature genetics, 24(2), 175-9. [PubMed:10655065] [WorldCat] [DOI] - ↑
Kuroda, M., Sok, J., Webb, L., Baechtold, H., Urano, F., Yin, Y., ..., & Ron, D. (2000).
Male sterility and enhanced radiation sensitivity in TLS(-/-) mice. The EMBO journal, 19(3), 453-62. [PubMed:10654943] [PMC] [WorldCat] [DOI] - ↑ 青木正志 (2013).
FUS変異によるALS臨床病理と病態, 臨床神経 53:1080-1083 [PDF] - ↑ 46.0 46.1 46.2
Ishigaki, S., Riku, Y., Fujioka, Y., Endo, K., Iwade, N., Kawai, K., ..., & Sobue, G. (2020).
Aberrant interaction between FUS and SFPQ in neurons in a wide range of FTLD spectrum diseases. Brain : a journal of neurology, 143(8), 2398-2405. [PubMed:32770214] [WorldCat] [DOI] - ↑
Ferrer, I., Legati, A., García-Monco, J.C., Gomez-Beldarrain, M., Carmona, M., Blanco, R., ..., & Coppola, G. (2015).
Familial behavioral variant frontotemporal dementia associated with astrocyte-predominant tauopathy. Journal of neuropathology and experimental neurology, 74(4), 370-9. [PubMed:25756587] [PMC] [WorldCat] [DOI] - ↑
Bäumer, D., Hilton, D., Paine, S.M., Turner, M.R., Lowe, J., Talbot, K., & Ansorge, O. (2010).
Juvenile ALS with basophilic inclusions is a FUS proteinopathy with FUS mutations. Neurology, 75(7), 611-8. [PubMed:20668261] [PMC] [WorldCat] [DOI] - ↑
Yamashita, S., Mori, A., Sakaguchi, H., Suga, T., Ishihara, D., Ueda, A., ..., & Hirano, T. (2012).
Sporadic juvenile amyotrophic lateral sclerosis caused by mutant FUS/TLS: possible association of mental retardation with this mutation. Journal of neurology, 259(6), 1039-44. [PubMed:22057404] [WorldCat] [DOI] - ↑ 50.0 50.1
Scekic-Zahirovic, J., Sendscheid, O., El Oussini, H., Jambeau, M., Sun, Y., Mersmann, S., ..., & Dupuis, L. (2016).
Toxic gain of function from mutant FUS protein is crucial to trigger cell autonomous motor neuron loss. The EMBO journal, 35(10), 1077-97. [PubMed:26951610] [PMC] [WorldCat] [DOI] - ↑
Gao, F.B., Almeida, S., & Lopez-Gonzalez, R. (2017).
Dysregulated molecular pathways in amyotrophic lateral sclerosis-frontotemporal dementia spectrum disorder. The EMBO journal, 36(20), 2931-2950. [PubMed:28916614] [PMC] [WorldCat] [DOI] - ↑
Ling, S.C., Dastidar, S.G., Tokunaga, S., Ho, W.Y., Lim, K., Ilieva, H., ..., & Cleveland, D.W. (2019).
Overriding FUS autoregulation in mice triggers gain-of-toxic dysfunctions in RNA metabolism and autophagy-lysosome axis. eLife, 8. [PubMed:30747709] [PMC] [WorldCat] [DOI] - ↑
Sharma, A., Lyashchenko, A.K., Lu, L., Nasrabady, S.E., Elmaleh, M., Mendelsohn, M., ..., & Shneider, N.A. (2016).
ALS-associated mutant FUS induces selective motor neuron degeneration through toxic gain of function. Nature communications, 7, 10465. [PubMed:26842965] [PMC] [WorldCat] [DOI] - ↑
Kino, Y., Washizu, C., Kurosawa, M., Yamada, M., Miyazaki, H., Akagi, T., ..., & Nukina, N. (2015).
FUS/TLS deficiency causes behavioral and pathological abnormalities distinct from amyotrophic lateral sclerosis. Acta neuropathologica communications, 3, 24. [PubMed:25907258] [PMC] [WorldCat] [DOI] - ↑
Kabashi, E., Bercier, V., Lissouba, A., Liao, M., Brustein, E., Rouleau, G.A., & Drapeau, P. (2011).
FUS and TARDBP but not SOD1 interact in genetic models of amyotrophic lateral sclerosis. PLoS genetics, 7(8), e1002214. [PubMed:21829392] [PMC] [WorldCat] [DOI] - ↑
Sasayama, H., Shimamura, M., Tokuda, T., Azuma, Y., Yoshida, T., Mizuno, T., ..., & Yamaguchi, M. (2012).
Knockdown of the Drosophila fused in sarcoma (FUS) homologue causes deficient locomotive behavior and shortening of motoneuron terminal branches. PloS one, 7(6), e39483. [PubMed:22724023] [PMC] [WorldCat] [DOI] - ↑
Chong, P.A., & Forman-Kay, J.D. (2016).
Liquid-liquid phase separation in cellular signaling systems. Current opinion in structural biology, 41, 180-186. [PubMed:27552079] [WorldCat] [DOI] - ↑
Monahan, Z., Ryan, V.H., Janke, A.M., Burke, K.A., Rhoads, S.N., Zerze, G.H., ..., & Fawzi, N.L. (2017).
Phosphorylation of the FUS low-complexity domain disrupts phase separation, aggregation, and toxicity. The EMBO journal, 36(20), 2951-2967. [PubMed:28790177] [PMC] [WorldCat] [DOI] - ↑
Murakami, T., Qamar, S., Lin, J.Q., Schierle, G.S., Rees, E., Miyashita, A., ..., & St George-Hyslop, P. (2015).
ALS/FTD Mutation-Induced Phase Transition of FUS Liquid Droplets and Reversible Hydrogels into Irreversible Hydrogels Impairs RNP Granule Function. Neuron, 88(4), 678-90. [PubMed:26526393] [PMC] [WorldCat] [DOI] - ↑
Murray, D.T., Kato, M., Lin, Y., Thurber, K.R., Hung, I., McKnight, S.L., & Tycko, R. (2017).
Structure of FUS Protein Fibrils and Its Relevance to Self-Assembly and Phase Separation of Low-Complexity Domains. Cell, 171(3), 615-627.e16. [PubMed:28942918] [PMC] [WorldCat] [DOI] - ↑
Patel, A., Lee, H.O., Jawerth, L., Maharana, S., Jahnel, M., Hein, M.Y., ..., & Alberti, S. (2015).
A Liquid-to-Solid Phase Transition of the ALS Protein FUS Accelerated by Disease Mutation. Cell, 162(5), 1066-77. [PubMed:26317470] [WorldCat] [DOI] - ↑
Shiina, N. (2019).
Liquid- and solid-like RNA granules form through specific scaffold proteins and combine into biphasic granules. The Journal of biological chemistry, 294(10), 3532-3548. [PubMed:30606735] [PMC] [WorldCat] [DOI] - ↑
Yoshizawa, T., Ali, R., Jiou, J., Fung, H.Y.J., Burke, K.A., Kim, S.J., ..., & Chook, Y.M. (2018).
Nuclear Import Receptor Inhibits Phase Separation of FUS through Binding to Multiple Sites. Cell, 173(3), 693-705.e22. [PubMed:29677513] [PMC] [WorldCat] [DOI] - ↑
Ikenaka, K., Ishigaki, S., Iguchi, Y., Kawai, K., Fujioka, Y., Yokoi, S., ..., & Sobue, G. (2020).
Characteristic Features of FUS Inclusions in Spinal Motor Neurons of Sporadic Amyotrophic Lateral Sclerosis. Journal of neuropathology and experimental neurology, 79(4), 370-377. [PubMed:32142134] [WorldCat] [DOI] - ↑
Tyzack, G.E., Luisier, R., Taha, D.M., Neeves, J., Modic, M., Mitchell, J.S., ..., & Patani, R. (2019).
Widespread FUS mislocalization is a molecular hallmark of amyotrophic lateral sclerosis. Brain : a journal of neurology, 142(9), 2572-2580. [PubMed:31368485] [PMC] [WorldCat] [DOI] - ↑
Fujimori, K., Ishikawa, M., Otomo, A., Atsuta, N., Nakamura, R., Akiyama, T., ..., & Okano, H. (2018).
Modeling sporadic ALS in iPSC-derived motor neurons identifies a potential therapeutic agent. Nature medicine, 24(10), 1579-1589. [PubMed:30127392] [WorldCat] [DOI]