「チロシンリン酸化」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| (3人の利用者による、間の124版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | |||
<font size="+1">[http://researchmap.jp/cnsfaust 林 崇]</font><br> | |||
''国立精神・神経医療研究センター 神経研究所 病態生化学研究部 細胞生化学研究室''<br> | |||
DOI:<selfdoi /> 原稿受付日:2013年4月10日 原稿完成日:2013年4月13日<br> | |||
担当編集委員:[http://researchmap.jp/2rikenbsi 林 康紀](独立行政法人理化学研究所 脳科学総合研究センター)<br> | |||
</div> | |||
英:tyrosine phosphorylation 英略語:PY、P-Tyr 独:Tyrosin Phosphorylierung 羅:phosphorylation tyrosine | |||
{{box|text= | |||
[[wj:真核生物|真核生物]]に存在する[[wj:タンパク質|タンパク質]]の細胞内領域[[wj:チロシン|チロシン]]残基に起こる可逆的[[wj:リン酸基|リン酸基]]付加反応。チロシンリン酸化の状態は、チロシンリン酸化酵素(チロシンキナーゼ、protein tyrosine kinase、PTK)およびチロシン脱リン酸化酵素(チロシンホスファターゼ、protein tyrosine phosphatase、PTP)の活性のバランスにより制御される。高等生物の神経系において、チロシンリン酸化は、様々な[[神経発生]]や[[神経可塑性]]の過程で、タンパク質の活性や局在、タンパク質間の結合、[[イオンチャンネル]]の性質、[[細胞内情報伝達]]系等を制御することが知られている。 | |||
}} | |||
[[Image:Hayashi tyrosine phosphorylation.png|thumb|right|400px| '''図 チロシンリン酸化、脱リン酸化反応''']] | |||
== 反応 == | |||
タンパク質リン酸化は、最もよく見られるタンパク質[[wj:翻訳後修飾|翻訳後修飾]]機構である。チロシンリン酸化酵素は、[[wj:アデノシン三リン酸|アデノシン三リン酸]](ATP)のγ位の[[wj:高エネルギーリン酸結合|高エネルギーリン酸基]]を、基質チロシン残基側鎖にある[[wj:水酸基|水酸基]]に移動させ、リン酸[[wj:エステル|エステル]]化により[[wj:共有結合|共有結合]]させる(図)。一般に、リン酸化に伴って、基質チロシン残基部位に[[wj:負電荷|負電荷]]が導入される。チロシン残基前後のアミノ酸配列により、チロシンキナーゼの[[基質特異性]]が決まる。チロシンホスファターゼは、チロシンキナーゼと比較してより基質特異性が広く、リン酸化[[wj:セリン|セリン]]・[[wj:スレオニン|スレオニン]]をも基質とするものも存在する。タンパク質中のリン酸化残基の99%以上はセリンとスレオニンであるが、0.1%に満たないチロシンのリン酸化は生物学的に重要な役割を果たす。 | |||
== | == チロシンキナーゼ == | ||
{{Pfam_box | |||
| Symbol = SH2 | |||
| Name = SH2 domain | |||
| image = 1lkkA_SH2_domain.png | |||
| width = 220 | |||
| caption = Crystallographic structure of the SH2 domain. The structure consists of a large [[w:beta sheet|beta sheet]] (green) flanked by two [[w:alpha helix|alpha-helices]] (orange and blue)<ref name="pmid8604142"><pubmed>8604142</pubmed></ref>. | |||
| Pfam= PF00017 | |||
| InterPro= IPR000980 | |||
| SMART= SH2 | |||
| PROSITE=PDOC50001 | |||
| SCOP = 1sha | |||
| TCDB = | |||
| OPM family= | |||
| OPM protein= 1xa6 | |||
| CDD = cd00173 | |||
| PDB= | |||
{{PDB3|1k9a}}B:82-156 {{PDB3|1jwo}}A:122-196 {{PDB3|1a81}}G:168-244 | |||
{{PDB3|1csz}}A:168-244 {{PDB3|1csy}}A:168-244 {{PDB3|1m61}}A:163-239 | |||
{{PDB3|1nrv}}B:493-574 {{PDB3|1mw4}}A:431-512 {{PDB3|2aug}}A:439-520 | |||
{{PDB3|2shp}}A:112-197 {{PDB3|1x6c}}A:110-194 {{PDB3|2b3o}}A:110-194 | |||
{{PDB3|1ayc}}A:6-81 {{PDB3|1aya}}B:6-81 {{PDB3|1ayb}}A:6-81 | |||
{{PDB3|1ayd}} :6-81 {{PDB3|1ju5}}A:13-104 {{PDB3|1sps}}A:148-230 | |||
{{PDB3|1spr}}C:148-230 {{PDB3|1bkl}} :148-230 {{PDB3|1bkm}} :148-230 | |||
{{PDB3|1kc2}}A:148-230 {{PDB3|1is0}}A:148-230 {{PDB3|1nzv}}A:148-230 | |||
{{PDB3|1nzl}}A:148-230 {{PDB3|1skj}} :148-230 {{PDB3|1shb}}A:148-230 | |||
{{PDB3|1sha}}A:148-230 {{PDB3|1p13}}B:148-230 {{PDB3|2ptk}}A:148-230 | |||
{{PDB3|1f1w}}A:148-230 {{PDB3|1f2f}}A:148-230 {{PDB3|1o4k}}A:151-233 | |||
{{PDB3|1o4i}}A:151-233 {{PDB3|1o4h}}A:151-233 {{PDB3|1a09}}B:151-233 | |||
{{PDB3|1o46}}A:151-233 {{PDB3|1o43}}A:151-233 {{PDB3|1o4d}}A:151-233 | |||
{{PDB3|1o4b}}A:151-233 {{PDB3|1ksw}}A:151-233 {{PDB3|1a1b}}B:151-233 | |||
{{PDB3|1a1e}}B:151-233 {{PDB3|1o4j}}A:151-233 {{PDB3|1o4m}}A:151-233 | |||
{{PDB3|1o4c}}A:151-233 {{PDB3|1a08}}A:151-233 {{PDB3|1a07}}B:151-233 | |||
{{PDB3|1o49}}A:151-233 {{PDB3|1fmk}} :151-233 {{PDB3|1o4n}}A:151-233 | |||
{{PDB3|1hcs}}B:151-233 {{PDB3|2src}} :151-233 {{PDB3|1a1a}}B:151-233 | |||
{{PDB3|1a1c}}A:151-233 {{PDB3|1o4f}}A:151-233 {{PDB3|1o4e}}A:151-233 | |||
{{PDB3|1o4q}}A:151-233 {{PDB3|1o4r}}A:151-233 {{PDB3|1o48}}A:151-233 | |||
{{PDB3|1shd}}A:151-233 {{PDB3|1y57}}A:151-233 {{PDB3|1hct}}B:151-233 | |||
{{PDB3|1o45}}A:151-233 {{PDB3|1o4g}}A:151-233 {{PDB3|1o4o}}A:151-233 | |||
{{PDB3|1o47}}A:151-233 {{PDB3|1o42}}A:151-233 {{PDB3|1o44}}A:151-233 | |||
{{PDB3|1o4a}}A:151-233 {{PDB3|1o41}}A:151-233 {{PDB3|1o4p}}A:151-233 | |||
{{PDB3|1o4l}}A:151-233 {{PDB3|1g83}}A:149-231 {{PDB3|1aot}}F:149-231 | |||
{{PDB3|1aou}}F:149-231 {{PDB3|1lkk}}A:127-209 {{PDB3|1ijr}}A:127-209 | |||
{{PDB3|1fbz}}B:127-209 {{PDB3|1cwe}}A:127-209 {{PDB3|1lcj}}A:127-209 | |||
{{PDB3|1cwd}}L:127-209 {{PDB3|1lck}}A:127-209 {{PDB3|1bhf}}A:127-209 | |||
{{PDB3|1lkl}}A:127-209 {{PDB3|1qcf}}A:144-226 {{PDB3|1ad5}}A:144-226 | |||
{{PDB3|2hck}}B:144-226 {{PDB3|3hck}} :144-226 {{PDB3|1blj}} :118-199 | |||
{{PDB3|1blk}} :118-199 {{PDB3|1pic}}A:624-698 {{PDB3|1h9o}}A:624-698 | |||
{{PDB3|1bfj}} :624-698 {{PDB3|1bfi}} :624-698 {{PDB3|1qad}}A:624-698 | |||
{{PDB3|1z3k}}A:285-359 {{PDB3|1fhs}} :60-135 {{PDB3|1cj1}}A:60-135 | |||
{{PDB3|1fyr}}A:60-135 {{PDB3|2aob}}C:60-135 {{PDB3|1bm2}}A:60-135 | |||
{{PDB3|1bmb}}A:60-135 {{PDB3|1gri}}A:60-135 {{PDB3|1x0n}}A:60-135 | |||
{{PDB3|1jyu}}A:60-135 {{PDB3|1qg1}}E:60-135 {{PDB3|2aoa}}B:60-135 | |||
{{PDB3|1jyq}}B:60-135 {{PDB3|1zfp}}E:60-135 {{PDB3|1jyr}}A:60-135 | |||
{{PDB3|1ghu}} :60-135 {{PDB3|1r1q}}B:58-132 {{PDB3|1r1s}}A:58-132 | |||
{{PDB3|1r1p}}D:58-132 {{PDB3|1wcp}}A:488-559 {{PDB3|1mil}} :488-559 | |||
{{PDB3|1opk}}A:127-202 {{PDB3|2abl}} :127-202 {{PDB3|1opl}}A:127-202 | |||
{{PDB3|1ab2}} :127-202 {{PDB3|1ka6}}A:7-87 {{PDB3|1d4w}}A:7-87 | |||
{{PDB3|1d1z}}A:7-87 {{PDB3|1m27}}A:7-87 {{PDB3|1d4t}}A:7-87 | |||
{{PDB3|1ka7}}A:7-87 {{PDB3|1i3z}}A:5-86 {{PDB3|2cs0}}A:34-109 | |||
{{PDB3|2fci}}A:668-741 {{PDB3|2ple}}A:668-741 {{PDB3|2pld}}A:668-741 | |||
{{PDB3|1wqu}}A:460-530 {{PDB3|1rqq}}C:409-486 {{PDB3|1rpy}}A:409-486 | |||
{{PDB3|2crh}}A:671-745 {{PDB3|1xa6}}A:59-130 {{PDB3|2pna}} :333-408 | |||
{{PDB3|2pnb}} :333-408 {{PDB3|1oo4}}A:333-408 {{PDB3|1oo3}}A:333-408 | |||
{{PDB3|1fu5}}A:333-408 {{PDB3|1fu6}}A:333-408 {{PDB3|1lui}}A:245-328 | |||
{{PDB3|1luk}}A:245-328 {{PDB3|1lum}}A:245-328 {{PDB3|1lun}}A:245-328 | |||
{{PDB3|1yvl}}A:573-668 {{PDB3|1bf5}}A:573-668 {{PDB3|1bg1}}A:584-674 | |||
{{PDB3|1y1u}}A:589-670 {{PDB3|1uur}}A:587-658 {{PDB3|1uus}}A:587-658 | |||
}} {{Pfam box | |||
| Symbol = SH3_1 | |||
| Name = SH3 domain | |||
| image =1shg SH3 domain.png | |||
| width =200 | |||
| caption =Ribbon diagram of the SH3 domain, alpha spectrin, from [[w:chicken|chicken]] (PDB accession code 1SHG), colored from blue (N-terminus) to red (C-terminus). | |||
| Pfam= PF00018 | |||
| Pfam_clan= CL0010 | |||
| InterPro= IPR001452 | |||
| SMART= SM00326 | |||
| PROSITE = PS50002 | |||
| SuperFamily = SSF50044 | |||
| SCOP = 1shf | |||
| TCDB = | |||
| CDD = cd00174 | |||
| OPM family= | |||
| OPM protein= | |||
| PDB= | |||
}} 1979年Tony Hunterらにより、[[wj:癌遺伝子|癌遺伝子]]産物[[V-Src]]および[[wj:癌遺伝子|癌原遺伝子]]産物[[C-Src]]がチロシンリン酸化活性を持つことが発見された<ref><pubmed>19269802</pubmed></ref>。これが最初のチロシンキナーゼの報告例であり、以後、多くのチロシンキナーゼが同定された。 | |||
現在では真核生物[[wj:ゲノム|ゲノム]]の全遺伝子の約2%は[[セリン・スレオニンキナーゼ]]およびチロシンキナーゼをコードする事が知られている。[[wj:細菌|細菌]]や[[wj:酵母|酵母]]にはチロシンキナーゼは存在せず、[[線虫]] ''C. elegans''(19,100遺伝子)には全キナーゼ数454(2.4%)の内チロシンキナーゼは90種、[[ショウジョウバエ]] ''D. melanogaster''(13,600遺伝子)には全キナーゼ数239(1.8%)の内チロシンキナーゼは32種、[[wj:ヒト|ヒト]] ''H. sapiens''(23,000遺伝子)には全キナーゼ数518(2.2%)の内チロシンキナーゼは90種が存在する。ただしヒトの場合キナーゼ518種の内、約50種には活性がなく、また106種は[[wj:偽遺伝子|偽遺伝子]]であると考えられる。 | |||
構造的に、[[w:Transmembrane domain|膜貫通領域]]を持つ[[受容体]]型と膜貫通領域を持たない非受容体型とに大別される(表1)。ヒトには58種の[[受容体型チロシンキナーゼ]]と32種の[[非受容体型チロシンキナーゼ]]が存在する。受容体型は[[細胞膜]]上に、非受容体型は[[wj:細胞質|細胞質]]に存在する。 | |||
== | === 受容体型チロシンキナーゼ === | ||
受容体型チロシンキナーゼは、細胞外のリガンド結合ドメイン、細胞内のチロンシンキナーゼドメイン、そして両者をつなぐ膜貫通ドメインを持つ。神経系で重要な役割を果たすものとして、[[TrkA]]、[[TrkB]]、[[TrkC]]、[[FGF受容体]]、[[インスリン受容体]]、[[Eph受容体]]などがある。リガンド結合ドメインへのリガンド結合により、二量体化したチロシンキナーゼが活性化され、さらに下流の[[分裂促進因子活性化タンパク質キナーゼ#ERK|細胞外シグナル調節キナーゼ]] ([[分裂促進因子活性化タンパク質キナーゼ#ERK|extracellular signal-regulated kinase]] (ERK)/[[分裂促進因子活性化タンパク質キナーゼ|Mitogen-activated Protein Kinase]] (MAPK)系、[[ホスホイノシチド-3キナーゼ]] ([[PI3K]])/[[Akt]]系、[[ホスホリパーゼC]]γ ([[PLC]]γ)/[[カルシウム|Ca<sup>2+</sup>]]系等の[[シグナル伝達]]を制御している。キナーゼドメイン中には自己リン酸化部位およびATP結合部位を含み、自己リン酸化によりキナーゼ活性を調節している。 | |||
''詳細は[[受容体型チロシンキナーゼ]]の項目参照。'' | |||
=== 非受容体型チロシンキナーゼ === | |||
非受容体型チロシンキナーゼは、分子構造として細胞外領域をもたず、細胞内領域にチロシンキナーゼドメインをもつ。受容体型チロシンキナーゼと異なり、非受容体型チロシンキナーゼには、直接的に結合するリガンドはない。上位の制御因子は細胞膜上に存在する種々の受容体タンパク質であり、非受容体型チロシンキナーゼは、様々な膜受容体と会合して、膜受容体から細胞内への情報伝達を担う。受容体型チロシンキナーゼと同様に、非受容体型チロシンキナーゼもキナーゼドメイン中には自己リン酸化部位およびATP結合部位を含み、自己リン酸化によりキナーゼ活性を調節している。 | |||
代表的な非受容体型チロシンキナーゼとして[[Src]]ファミリーチロシンキナーゼがある。現在までにSrc、[[Yes]]、[[Fyn]]、[[Fgr]]、[[Lyn]]、[[Lck]]、[[Hck]]、[[Blk]]、[[Frk]]の9種が同定されており、脳では、Src、Yes、Fyn、Lyn、Lckが高発現を示す。発現部位ごとに[[wj:スプライシング|スプライシング]]多様性がみられるものもある。Srcファミリーチロシンキナーゼの場合、N末端領域に[[ミリストイル化]]部位や[[パルミトイル化]]部位を有し、これらの[[wj:脂肪酸|脂肪酸]]結合により細胞膜に付着し、膜近辺に局在する様になる。Srcファミリーチロシンキナーゼが会合する受容体に関しては、[[Cadherin-related neuronal receptor]] (CNR)/[[プロトカドヘリン]]ファミリーに属する分子がFynと会合しシナプス機能を制御する、等の研究報告がある<ref><pubmed>11672833</pubmed></ref>。 | |||
脳神経系で機能する他の非受容体型チロシンキナーゼには、SrcファミリーチロシンキナーゼC末端のチロシン残基をリン酸化して不活性化する[[C-Src tyrosine kinase]] ([[Csk]])、[[細胞接着]]に関わる[[Focal Adhesion Kinase]] ([[FAK]])および[[Pyk2]]([[FAKファミリーチロシンキナーゼ]])、神経突起伸長に関わる[[Fes]]/[[Fps]]および[[Fer]](Fesファミリーチロシンキナーゼ)があり、それぞれ脳神経機能に重要な役割を担っている。 | |||
多くの非受容体型チロシンキナーゼには、[[wj:SH2ドメイン|Src Homology 2]] (SH2)ドメインおよび[[w:SH3 domain|SH3]]ドメインとよばれるドメイン構造が存在する。SH2ドメインはリン酸化チロシン残基(pTyr)を、SH3ドメインは[[wj:プロリン|プロリン]]リッチ領域(X-Pro-X-X-Pro)を、それぞれ認識して結合することで、細胞内情報伝達系におけるタンパク質-タンパク質結合を制御する。これらのドメインは構造的に保存されたアミノ酸配列を持ち、Srcファミリーチロシンキナーゼにおいて最初に見出された。更に、[[Abl]]、[[Fes]]、[[Syk]]/Zap70、[[Tec]]、[[Ack]]、[[Csk]]、[[Srm]]、[[Rak]]等の非受容体型チロシンキナーゼや、[[ホスファチジルイノシトール-3キナーゼ]] (PI3K)、[[ホスホリパーゼC]] (PLC)-γ等のセリン・スレオニンキナーゼ、また[[Grb2]]、[[Nck]]等のアダプタータンパク質もこれらのドメイン構造を持つことが明らかになった。SH2ドメインは、約100アミノ酸残基の領域であり、2つの[[wj:αヘリックス|αヘリックス]]と7つの[[wj:βシート|βシート]]から構成される。SH3ドメインは、約60アミノ酸残基の領域であり、5つないし6つのβシートからなる典型的な[[wj:βバレル|βバレル]]構造をもつ。 | |||
{| style="text-align: left;" | |||
|+ '''表1. チロシンキナーゼの一覧''' wより改変 | |||
|- | |||
! colspan="3" align="center" style="background-color:lightskyblue;" | [[受容体型チロシンキナーゼ]] | |||
|- | |||
! rowspan="7" style="background-color:lightskyblue;" | [[成長因子受容体]] | |||
| style="background-color:lightskyblue;" | [[上皮成長因子受容体]]ファミリー | |||
| [[EGFR]], [[HER2/neu|ERBB2]], [[ERBB3]], [[ERBB4]] | |||
|- | |||
| style="background-color:lightskyblue;" | [[インスリン受容体]]ファミリー | |||
| [[IGF1R]], [[INSR]], [[INSRR]] | |||
|- | |||
| style="background-color:lightskyblue;" | [[PDGF受容体]]ファミリー | |||
| [[CSF1R]], [[FLT3]], [[KIT]], [[PDGFR]] ([[PDGFRA]], [[PDGFRB]]) | |||
|- | |||
| style="background-color:lightskyblue;" | [[FGF受容体]]ファミリー | |||
| [[FGFR1]], [[FGFR2]], [[FGFR3]], [[FGFR4]] | |||
|- | |||
| style="background-color:lightskyblue;" | [[VEGF受容体s]]ファミリー | |||
| [[FLT1|VEGFR1]], [[VEGFR2]], [[VEGFR3]], [[VEGFR4]] | |||
|- | |||
| style="background-color:lightskyblue;" | [[C-Met|HGF受容体]]ファミリー | |||
| [[MET]], [[RON]] | |||
|- | |||
| style="background-color:lightskyblue;" | [[Trk受容体]]ファミリー | |||
| [[NTRK1]], [[NTRK2]], [[NTRK3]] | |||
|- | |||
! align="left" colspan="2" style="background-color:lightskyblue;" | [[Eph受容体]]ファミリー | |||
| [[EPHA1]], [[EPHA2]], [[EEPHA3]], [[EPHA4]], [[EPHA5]], [[EPHA6]], [[EPHA7]], [[EPHA8]], [[EPHB1]], [[EPHB2]], [[EPHB3]], [[EPHB4]], [[EPHB5]], [[EPHB6]], [[EPHX]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Leukocyte受容体 チロシンキナーゼ|LTK受容体]]ファミリー | |||
| [[Leukocyte受容体 チロシンキナーゼ|LTK]], [[Anaplastic lymphoma kinase|ALK]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Angiopoietin受容体|TIE受容体]]ファミリー | |||
| [[TIE]], [[TEK]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[受容体チロシンキナーゼ様オーファン受容体|ROR受容体]]ファミリー | |||
| [[ROR1]], [[ROR2]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[DDR1|DDR受容体]]ファミリー | |||
| [[DDR1]], [[DDR2]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[PTK7|PTK7受容体]]ファミリー | |||
| [[PTK7]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Related to受容体 チロシンキナーゼ|RYK受容体]]ファミリー | |||
| [[RYK]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[MuSK protein|MuSK受容体]]ファミリー | |||
| [[MUSK]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[ROS1 (gene)|ROS受容体]]ファミリー | |||
| [[ROS1]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[AATK|AATYK受容体]]ファミリー | |||
| [[AATYK]], [[AATYK2]], [[AATYK3]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[AXL受容体 チロシンキナーゼ|AXL受容体]]ファミリー | |||
| [[AXL]], [[MER]], [[TYRO3]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[RET proto-oncogene|RET受容体]]ファミリー | |||
| [[RET]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | 未分類 | |||
| [[STYK1]] | |||
|- | |||
! colspan="3" align="center" style="background-color:lightskyblue;" | [[非受容体型チロシンキナーゼ]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Abl gene|ABL]]ファミリー | |||
| [[ABL1]], [[ARG]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[TNK2|ACK]]ファミリー | |||
| [[ACK1]], [[TNK1]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[CSK]]ファミリー | |||
| [[CSK]], [[MATK]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[FAK]]ファミリー | |||
| [[FAK]], [[PYK2]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Feline sarcoma oncogene|FES]]ファミリー | |||
| [[FES]], [[FER]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Fyn-related kinase|FRK]]ファミリー | |||
| [[FRK]], [[BRK]], [[SRMS]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Janus kinase|JAK]]ファミリー | |||
| [[JAK1]], [[JAK2]], [[JAK3]], [[TYK2]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[SRC-A]]ファミリー | |||
| [[SRC]], [[FGR]], [[FYN]], [[YES1]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[SRC-B]]ファミリー | |||
| [[BLK]], [[HCK]], [[LCK]], [[LYN]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[TEC (gene)|TEC]]ファミリー | |||
| [[TEC]], [[BMX]], [[BTK]], [[ITK]], [[TXK]] | |||
|- | |||
! colspan="2" style="background-color:lightskyblue;" | [[Syk|SYK]]ファミリー | |||
| [[SYK]], [[ZAP70]] | |||
|} | |||
<br> | |||
== チロシンホスファターゼ == | |||
チロシンホスファターゼには、107種が存在する。チロシンキナーゼと同様に、チロシンホスファターゼは、膜貫通領域を持つ[[受容体型チロシンホスファターゼ]]および膜貫通領域を持たない[[非受容体型チロシンホスファターゼ]]に大別される<ref><pubmed>17057753</pubmed></ref>(表2)。チロシンキナーゼ同様に、チロシンホスファターゼも、受容体型は細胞膜上に、非受容体型は細胞質に存在する。チロシンホスファターゼはチロシンキナーゼと違いその基質特異性は低く、リン酸化チロシンを含む多くのタンパク質を認識してそれぞれ脱リン酸化する。神経発生の過程における[[細胞接着|神経接着]]や[[軸索]]の伸長と[[軸索ガイダンス|ガイダンス ]]、[[シナプス形成]]、[[シナプス可塑性]]の調節におけるチロシンホスファターゼの機能が明らかにされている。 | |||
=== 受容体型チロシンホスファターゼ === | |||
全ての受容体型チロシンホスファターゼは、細胞外ドメイン、膜貫通ドメイン、細胞内の1つあるいは2つのホスファターゼドメイン(D1およびD2ドメイン)を持つ。細胞外には、[[免疫グロブリンスーパーファミリー|免疫グロブリンドメイン]]や[[フィブロネクチン]]Ⅲ型ドメインの繰り返し配列を持つ例が知られる。 | |||
細胞内のD1およびD2ドメインはそれぞれ280個程のアミノ酸残基からなり、高度に保存されている。この内、細胞膜に近い側のD1ドメインのみがホスファターゼ活性を持ち、その活性中心配列は(I/V)HCXAGXXR(S/T)Gである。一方、C末端側のD2ドメインはホスファターゼ活性を持たず、様々な結合タンパク質と会合する<ref><pubmed>17085782</pubmed></ref>。 | |||
神経系で重要な役割を果たすものとして、受容体型では[[PTPRF|LAR]]、[[PTPRS|PTPσ]]、[[PTPRD|PTPδ]]、[[PTPζ]]等がある。受容体型チロシンホスファターゼのリガンド分子に付いては未だ不明な点が多いが、これまでに、 | |||
#[[ヘパラン硫酸プロテオグリカン]]である[[シンデカン]]がLARと結合してそのホスファターゼ活性を抑制し、軸索伸長を制御する<ref><pubmed>16213816</pubmed></ref> | |||
#[[プレイオトロフィン]]がリガンドとしてPTPζに結合し、その活性を低下させて基質である[[βカテニン]]のチロシンリン酸化を亢進し、[[神経細胞移動]]を制御する<ref><pubmed>10706604</pubmed></ref><ref><pubmed>16105548</pubmed></ref> | |||
#[[シナプス前膜]]側のPTPδが[[シナプス後膜]]側の精神疾患原因遺伝子[[IL1RAPL1]]と''trans''-synapticに結合し、シナプス形成を促進する<ref><pubmed>21940441</pubmed></ref> | |||
等の研究報告が知られている。 | |||
=== 非受容体型チロシンホスファターゼ === | |||
非受容体型チロシンホスファターゼは受容体型チロシンホスファターゼと異なり、膜貫通ドメインを持たず、1つのホスファターゼドメインとそのN末かC末側に様々なドメインを有する。神経系で重要な役割を果たすものとして、[[PTP1B]]、[[PTEN]]、[[STEP]]、[[PTP-SL]]、[[SHP1]]、[[SHP-2]]、[[PTPMEG]]等などがある<ref><pubmed>14625689</pubmed></ref>。 | |||
小脳において、PTPMEGが[[グルタミン酸受容体]][[GluD2]]に結合し、[[AMPA型グルタミン酸受容体]][[GluA2]]サブユニットのチロシンリン酸化状態を制御して、シナプス上のAMPA型グルタミン酸受容体発現数を調節することにより、[[長期抑圧]]に関与する、等の研究報告が知られている<ref><pubmed>23431139</pubmed></ref>。<br> | |||
{| style="text-align: left;" | |||
|+ '''表2. チロシンホスファターゼ一覧''' wより改変。 | |||
|- | |||
! rowspan="9" align="left" style="background-color:lightskyblue;" | [[クラスI]] | |||
! rowspan="2" align="left" style="background-color:lightskyblue;" | Classical PTPs | |||
! align="left" style="background-color:lightskyblue;" | '''[[受容体型チロシンフォスファターゼ|受容体型チロシン]]'''ホスファターゼ | |||
| [[PTPRA]], [[PTPRB]], [[PTPRC]], [[PTPRD]], [[PTPRE]], [[PTPRF]], [[PTPRG]], [[PTPRH]], [[PTPRJ]], [[PTPRK]], [[PTPRM]], [[PTPRN]], [[PTPRN2]], [[PTPRO]], [[PTPRQ]], [[PTPRR]], [[PTPRS]], [[PTPRT]], [[PTPRU]], [[PTPRZ1]], [[PTPRZ2]] | |||
|- | |||
! align="left" style="background-color:lightskyblue;" | '''[[非受容体型チロシンフォスファターゼ|非受容体型チロシン]]'''ホスファターゼ | |||
| [[PTPN1]], [[PTPN2]], [[PTPN3]], [[PTPN4]], [[PTPN5]], [[PTPN6]], [[PTPN7]], [[PTPN9]], [[PTPN11]], [[PTPN12]], [[PTPN13]], [[PTPN14]], [[PTPN18]], [[PTPN20]], [[PTPN21]], [[PTPN22]], [[PTPN23]] | |||
|- | |||
! rowspan="7" align="left" style="background-color:lightskyblue;" | [[VH1様ホスファターゼ]]または[[二重特異性ホスファターゼ]] | |||
! style="background-color:lightskyblue;" | '''[[MAPK phosphatase]]s (MKPs)''' | |||
| [[DUSP1]], [[DUSP2]], [[DUSP4]], [[DUSP5]], [[DUSP6]], [[DUSP7]], [[DUSP8]], [[DUSP9]], [[DUSP10]], [[DUSP16]], [[MK-STYX]] | |||
|- | |||
! style="background-color:lightskyblue;" | '''Slingshots''' | |||
| [[SSH1]], [[SSH2]], [[SSH3]] | |||
|- | |||
! style="background-color:lightskyblue;" | '''PRLs''' | |||
| [[PTP4A1]], [[PTP4A2]], [[PTP4A3]] | |||
|- | |||
! style="background-color:lightskyblue;" | '''CDC14s''' | |||
| [[CDC14A]], [[CDC14B]], [[CDKN3]], [[PTP9Q22]] | |||
|- | |||
! style="background-color:lightskyblue;" | '''Atypical DSPs''' | |||
| [[DUSP3]], [[DUSP11]], [[DUSP12]], [[DUSP13A]], [[DUSP13B]], [[DUSP14]], [[DUSP15]], [[DUSP18]], [[DUSP19]], [[DUSP21]], [[DUSP22]], [[DUSP23]], [[DUSP24]], [[DUSP25]], [[DUSP26]], [[DUSP27]], [[EMP2A]], [[RNGTT]], [[STYX]] | |||
|- | |||
! style="background-color:lightskyblue;" | '''Phosphatase and tensin homologs (PTENs)''' | |||
| [[PTEN (gene)|PTEN]], [[TPIP]], [[TPTE]], [[TNS (gene)|TNS]], [[TENC1]] | |||
|- | |||
! style="background-color:lightskyblue;" | '''[[Myotubularin]]s''' | |||
| [[Myotubularin 1|MTM1]], [[MTMR2]], [[MTMR3]], [[MTMR4]], [[MTMR5]], [[MTMR6]], [[MTMR7]], [[MTMR8]], [[MTMR9]], [[MTMR10]], [[MTMR11]], [[MTMR12]], [[MTMR13]], [[MTMR14]], [[MTMR15]] | |||
|- | |||
! colspan="3" align="left" style="background-color:lightskyblue;" | [[クラスII]] | |||
| [[ACP1]] | |||
|- | |||
! colspan="3" align="left" style="background-color:lightskyblue;" | [[クラスIII]] | |||
| [[Cdc25]], [[CDC25A]], [[CDC25B]], [[CDC25C]] | |||
|- | |||
! colspan="3" style="background-color:lightskyblue;" | [[クラスIV]] | |||
| [[Eyes absent homolog]], [[EYA1]], [[EYA2]], [[EYA3]], [[EYA4]] | |||
|} | |||
== 生理機能 == | |||
チロシンリン酸化の神経機能における役割としては、[[シナプス前膜]]側からの[[神経伝達物質]]放出の調節、様々な[[電位依存性イオンチャネル]]および[[リガンド依存性イオンチャネル]]の[[コンダクタンス]]と[[開口確率]]の制御<ref><pubmed>11668044</pubmed></ref>、[[グルタミン酸受容体]]をはじめとした多くのタンパク質分子の[[シナプス]]での局在と輸送過程の制御が報告されている。更に、それらに伴い、神経可塑性と個体レベルの行動に変化がおこることが知られている。また、他の役割として、神経回路、[[神経筋接合部]]や[[ミエリン]]構造の形成、[[樹状突起]]の形態形成や[[軸索]]伸長等の過程において、チロシンリン酸化依存的な制御が挙げられる<ref><pubmed>21508038</pubmed></ref>。 | |||
== 関連項目 == | |||
*[[リン酸化]] | |||
*[[タンパク質リン酸化酵素]] | |||
*[[受容体型チロシンキナーゼ]] | |||
*[[非受容体型チロシンキナーゼ]] | |||
*[[受容体型チロシンホスファターゼ]] | |||
*[[非受容体型チロシンホファターゼ]] | |||
== 参考文献 == | |||
<references /> | |||
2020年7月9日 (木) 10:39時点における最新版
林 崇
国立精神・神経医療研究センター 神経研究所 病態生化学研究部 細胞生化学研究室
DOI:10.14931/bsd.353 原稿受付日:2013年4月10日 原稿完成日:2013年4月13日
担当編集委員:林 康紀(独立行政法人理化学研究所 脳科学総合研究センター)
英:tyrosine phosphorylation 英略語:PY、P-Tyr 独:Tyrosin Phosphorylierung 羅:phosphorylation tyrosine
真核生物に存在するタンパク質の細胞内領域チロシン残基に起こる可逆的リン酸基付加反応。チロシンリン酸化の状態は、チロシンリン酸化酵素(チロシンキナーゼ、protein tyrosine kinase、PTK)およびチロシン脱リン酸化酵素(チロシンホスファターゼ、protein tyrosine phosphatase、PTP)の活性のバランスにより制御される。高等生物の神経系において、チロシンリン酸化は、様々な神経発生や神経可塑性の過程で、タンパク質の活性や局在、タンパク質間の結合、イオンチャンネルの性質、細胞内情報伝達系等を制御することが知られている。
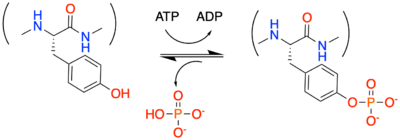
反応
タンパク質リン酸化は、最もよく見られるタンパク質翻訳後修飾機構である。チロシンリン酸化酵素は、アデノシン三リン酸(ATP)のγ位の高エネルギーリン酸基を、基質チロシン残基側鎖にある水酸基に移動させ、リン酸エステル化により共有結合させる(図)。一般に、リン酸化に伴って、基質チロシン残基部位に負電荷が導入される。チロシン残基前後のアミノ酸配列により、チロシンキナーゼの基質特異性が決まる。チロシンホスファターゼは、チロシンキナーゼと比較してより基質特異性が広く、リン酸化セリン・スレオニンをも基質とするものも存在する。タンパク質中のリン酸化残基の99%以上はセリンとスレオニンであるが、0.1%に満たないチロシンのリン酸化は生物学的に重要な役割を果たす。
チロシンキナーゼ
| SH2 domain | |||||||||
|---|---|---|---|---|---|---|---|---|---|
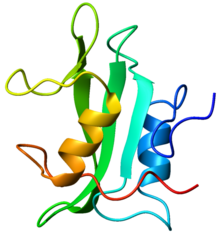 Crystallographic structure of the SH2 domain. The structure consists of a large beta sheet (green) flanked by two alpha-helices (orange and blue)[1]. | |||||||||
| Identifiers | |||||||||
| Symbol | SH2 | ||||||||
| Pfam | PF00017 | ||||||||
| InterPro | IPR000980 | ||||||||
| SMART | SH2 | ||||||||
| PROSITE | PDOC50001 | ||||||||
| SCOP | 1sha | ||||||||
| SUPERFAMILY | 1sha | ||||||||
| OPM protein | 1xa6 | ||||||||
| CDD | cd00173 | ||||||||
| |||||||||
| SH3 domain | |||||||||
|---|---|---|---|---|---|---|---|---|---|
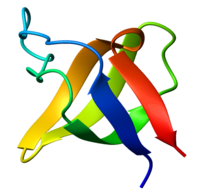 Ribbon diagram of the SH3 domain, alpha spectrin, from chicken (PDB accession code 1SHG), colored from blue (N-terminus) to red (C-terminus). | |||||||||
| Identifiers | |||||||||
| Symbol | SH3_1 | ||||||||
| Pfam | PF00018 | ||||||||
| Pfam clan | CL0010 | ||||||||
| InterPro | IPR001452 | ||||||||
| SMART | SM00326 | ||||||||
| PROSITE | PS50002 | ||||||||
| SCOP | 1shf | ||||||||
| SUPERFAMILY | 1shf | ||||||||
| CDD | cd00174 | ||||||||
| |||||||||
1979年Tony Hunterらにより、癌遺伝子産物V-Srcおよび癌原遺伝子産物C-Srcがチロシンリン酸化活性を持つことが発見された[2]。これが最初のチロシンキナーゼの報告例であり、以後、多くのチロシンキナーゼが同定された。
現在では真核生物ゲノムの全遺伝子の約2%はセリン・スレオニンキナーゼおよびチロシンキナーゼをコードする事が知られている。細菌や酵母にはチロシンキナーゼは存在せず、線虫 C. elegans(19,100遺伝子)には全キナーゼ数454(2.4%)の内チロシンキナーゼは90種、ショウジョウバエ D. melanogaster(13,600遺伝子)には全キナーゼ数239(1.8%)の内チロシンキナーゼは32種、ヒト H. sapiens(23,000遺伝子)には全キナーゼ数518(2.2%)の内チロシンキナーゼは90種が存在する。ただしヒトの場合キナーゼ518種の内、約50種には活性がなく、また106種は偽遺伝子であると考えられる。
構造的に、膜貫通領域を持つ受容体型と膜貫通領域を持たない非受容体型とに大別される(表1)。ヒトには58種の受容体型チロシンキナーゼと32種の非受容体型チロシンキナーゼが存在する。受容体型は細胞膜上に、非受容体型は細胞質に存在する。
受容体型チロシンキナーゼ
受容体型チロシンキナーゼは、細胞外のリガンド結合ドメイン、細胞内のチロンシンキナーゼドメイン、そして両者をつなぐ膜貫通ドメインを持つ。神経系で重要な役割を果たすものとして、TrkA、TrkB、TrkC、FGF受容体、インスリン受容体、Eph受容体などがある。リガンド結合ドメインへのリガンド結合により、二量体化したチロシンキナーゼが活性化され、さらに下流の細胞外シグナル調節キナーゼ (extracellular signal-regulated kinase (ERK)/Mitogen-activated Protein Kinase (MAPK)系、ホスホイノシチド-3キナーゼ (PI3K)/Akt系、ホスホリパーゼCγ (PLCγ)/Ca2+系等のシグナル伝達を制御している。キナーゼドメイン中には自己リン酸化部位およびATP結合部位を含み、自己リン酸化によりキナーゼ活性を調節している。
詳細は受容体型チロシンキナーゼの項目参照。
非受容体型チロシンキナーゼ
非受容体型チロシンキナーゼは、分子構造として細胞外領域をもたず、細胞内領域にチロシンキナーゼドメインをもつ。受容体型チロシンキナーゼと異なり、非受容体型チロシンキナーゼには、直接的に結合するリガンドはない。上位の制御因子は細胞膜上に存在する種々の受容体タンパク質であり、非受容体型チロシンキナーゼは、様々な膜受容体と会合して、膜受容体から細胞内への情報伝達を担う。受容体型チロシンキナーゼと同様に、非受容体型チロシンキナーゼもキナーゼドメイン中には自己リン酸化部位およびATP結合部位を含み、自己リン酸化によりキナーゼ活性を調節している。
代表的な非受容体型チロシンキナーゼとしてSrcファミリーチロシンキナーゼがある。現在までにSrc、Yes、Fyn、Fgr、Lyn、Lck、Hck、Blk、Frkの9種が同定されており、脳では、Src、Yes、Fyn、Lyn、Lckが高発現を示す。発現部位ごとにスプライシング多様性がみられるものもある。Srcファミリーチロシンキナーゼの場合、N末端領域にミリストイル化部位やパルミトイル化部位を有し、これらの脂肪酸結合により細胞膜に付着し、膜近辺に局在する様になる。Srcファミリーチロシンキナーゼが会合する受容体に関しては、Cadherin-related neuronal receptor (CNR)/プロトカドヘリンファミリーに属する分子がFynと会合しシナプス機能を制御する、等の研究報告がある[3]。
脳神経系で機能する他の非受容体型チロシンキナーゼには、SrcファミリーチロシンキナーゼC末端のチロシン残基をリン酸化して不活性化するC-Src tyrosine kinase (Csk)、細胞接着に関わるFocal Adhesion Kinase (FAK)およびPyk2(FAKファミリーチロシンキナーゼ)、神経突起伸長に関わるFes/FpsおよびFer(Fesファミリーチロシンキナーゼ)があり、それぞれ脳神経機能に重要な役割を担っている。
多くの非受容体型チロシンキナーゼには、Src Homology 2 (SH2)ドメインおよびSH3ドメインとよばれるドメイン構造が存在する。SH2ドメインはリン酸化チロシン残基(pTyr)を、SH3ドメインはプロリンリッチ領域(X-Pro-X-X-Pro)を、それぞれ認識して結合することで、細胞内情報伝達系におけるタンパク質-タンパク質結合を制御する。これらのドメインは構造的に保存されたアミノ酸配列を持ち、Srcファミリーチロシンキナーゼにおいて最初に見出された。更に、Abl、Fes、Syk/Zap70、Tec、Ack、Csk、Srm、Rak等の非受容体型チロシンキナーゼや、ホスファチジルイノシトール-3キナーゼ (PI3K)、ホスホリパーゼC (PLC)-γ等のセリン・スレオニンキナーゼ、またGrb2、Nck等のアダプタータンパク質もこれらのドメイン構造を持つことが明らかになった。SH2ドメインは、約100アミノ酸残基の領域であり、2つのαヘリックスと7つのβシートから構成される。SH3ドメインは、約60アミノ酸残基の領域であり、5つないし6つのβシートからなる典型的なβバレル構造をもつ。
| 受容体型チロシンキナーゼ | ||
|---|---|---|
| 成長因子受容体 | 上皮成長因子受容体ファミリー | EGFR, ERBB2, ERBB3, ERBB4 |
| インスリン受容体ファミリー | IGF1R, INSR, INSRR | |
| PDGF受容体ファミリー | CSF1R, FLT3, KIT, PDGFR (PDGFRA, PDGFRB) | |
| FGF受容体ファミリー | FGFR1, FGFR2, FGFR3, FGFR4 | |
| VEGF受容体sファミリー | VEGFR1, VEGFR2, VEGFR3, VEGFR4 | |
| HGF受容体ファミリー | MET, RON | |
| Trk受容体ファミリー | NTRK1, NTRK2, NTRK3 | |
| Eph受容体ファミリー | EPHA1, EPHA2, EEPHA3, EPHA4, EPHA5, EPHA6, EPHA7, EPHA8, EPHB1, EPHB2, EPHB3, EPHB4, EPHB5, EPHB6, EPHX | |
| LTK受容体ファミリー | LTK, ALK | |
| TIE受容体ファミリー | TIE, TEK | |
| ROR受容体ファミリー | ROR1, ROR2 | |
| DDR受容体ファミリー | DDR1, DDR2 | |
| PTK7受容体ファミリー | PTK7 | |
| RYK受容体ファミリー | RYK | |
| MuSK受容体ファミリー | MUSK | |
| ROS受容体ファミリー | ROS1 | |
| AATYK受容体ファミリー | AATYK, AATYK2, AATYK3 | |
| AXL受容体ファミリー | AXL, MER, TYRO3 | |
| RET受容体ファミリー | RET | |
| 未分類 | STYK1 | |
| 非受容体型チロシンキナーゼ | ||
| ABLファミリー | ABL1, ARG | |
| ACKファミリー | ACK1, TNK1 | |
| CSKファミリー | CSK, MATK | |
| FAKファミリー | FAK, PYK2 | |
| FESファミリー | FES, FER | |
| FRKファミリー | FRK, BRK, SRMS | |
| JAKファミリー | JAK1, JAK2, JAK3, TYK2 | |
| SRC-Aファミリー | SRC, FGR, FYN, YES1 | |
| SRC-Bファミリー | BLK, HCK, LCK, LYN | |
| TECファミリー | TEC, BMX, BTK, ITK, TXK | |
| SYKファミリー | SYK, ZAP70 | |
チロシンホスファターゼ
チロシンホスファターゼには、107種が存在する。チロシンキナーゼと同様に、チロシンホスファターゼは、膜貫通領域を持つ受容体型チロシンホスファターゼおよび膜貫通領域を持たない非受容体型チロシンホスファターゼに大別される[4](表2)。チロシンキナーゼ同様に、チロシンホスファターゼも、受容体型は細胞膜上に、非受容体型は細胞質に存在する。チロシンホスファターゼはチロシンキナーゼと違いその基質特異性は低く、リン酸化チロシンを含む多くのタンパク質を認識してそれぞれ脱リン酸化する。神経発生の過程における神経接着や軸索の伸長とガイダンス 、シナプス形成、シナプス可塑性の調節におけるチロシンホスファターゼの機能が明らかにされている。
受容体型チロシンホスファターゼ
全ての受容体型チロシンホスファターゼは、細胞外ドメイン、膜貫通ドメイン、細胞内の1つあるいは2つのホスファターゼドメイン(D1およびD2ドメイン)を持つ。細胞外には、免疫グロブリンドメインやフィブロネクチンⅢ型ドメインの繰り返し配列を持つ例が知られる。
細胞内のD1およびD2ドメインはそれぞれ280個程のアミノ酸残基からなり、高度に保存されている。この内、細胞膜に近い側のD1ドメインのみがホスファターゼ活性を持ち、その活性中心配列は(I/V)HCXAGXXR(S/T)Gである。一方、C末端側のD2ドメインはホスファターゼ活性を持たず、様々な結合タンパク質と会合する[5]。
神経系で重要な役割を果たすものとして、受容体型ではLAR、PTPσ、PTPδ、PTPζ等がある。受容体型チロシンホスファターゼのリガンド分子に付いては未だ不明な点が多いが、これまでに、
- ヘパラン硫酸プロテオグリカンであるシンデカンがLARと結合してそのホスファターゼ活性を抑制し、軸索伸長を制御する[6]
- プレイオトロフィンがリガンドとしてPTPζに結合し、その活性を低下させて基質であるβカテニンのチロシンリン酸化を亢進し、神経細胞移動を制御する[7][8]
- シナプス前膜側のPTPδがシナプス後膜側の精神疾患原因遺伝子IL1RAPL1とtrans-synapticに結合し、シナプス形成を促進する[9]
等の研究報告が知られている。
非受容体型チロシンホスファターゼ
非受容体型チロシンホスファターゼは受容体型チロシンホスファターゼと異なり、膜貫通ドメインを持たず、1つのホスファターゼドメインとそのN末かC末側に様々なドメインを有する。神経系で重要な役割を果たすものとして、PTP1B、PTEN、STEP、PTP-SL、SHP1、SHP-2、PTPMEG等などがある[10]。
小脳において、PTPMEGがグルタミン酸受容体GluD2に結合し、AMPA型グルタミン酸受容体GluA2サブユニットのチロシンリン酸化状態を制御して、シナプス上のAMPA型グルタミン酸受容体発現数を調節することにより、長期抑圧に関与する、等の研究報告が知られている[11]。
生理機能
チロシンリン酸化の神経機能における役割としては、シナプス前膜側からの神経伝達物質放出の調節、様々な電位依存性イオンチャネルおよびリガンド依存性イオンチャネルのコンダクタンスと開口確率の制御[12]、グルタミン酸受容体をはじめとした多くのタンパク質分子のシナプスでの局在と輸送過程の制御が報告されている。更に、それらに伴い、神経可塑性と個体レベルの行動に変化がおこることが知られている。また、他の役割として、神経回路、神経筋接合部やミエリン構造の形成、樹状突起の形態形成や軸索伸長等の過程において、チロシンリン酸化依存的な制御が挙げられる[13]。
関連項目
参考文献
- ↑
Tong, L., Warren, T.C., King, J., Betageri, R., Rose, J., & Jakes, S. (1996).
Crystal structures of the human p56lck SH2 domain in complex with two short phosphotyrosyl peptides at 1.0 A and 1.8 A resolution. Journal of molecular biology, 256(3), 601-10. [PubMed:8604142] [WorldCat] [DOI] - ↑
Hunter, T. (2009).
Tyrosine phosphorylation: thirty years and counting. Current opinion in cell biology, 21(2), 140-6. [PubMed:19269802] [PMC] [WorldCat] [DOI] - ↑
Hamada, S., & Yagi, T. (2001).
The cadherin-related neuronal receptor family: a novel diversified cadherin family at the synapse. Neuroscience research, 41(3), 207-15. [PubMed:11672833] [WorldCat] [DOI] - ↑
Tonks, N.K. (2006).
Protein tyrosine phosphatases: from genes, to function, to disease. Nature reviews. Molecular cell biology, 7(11), 833-46. [PubMed:17057753] [WorldCat] [DOI] - ↑
Lamprianou, S., & Harroch, S. (2006).
Receptor protein tyrosine phosphatase from stem cells to mature glial cells of the central nervous system. Journal of molecular neuroscience : MN, 29(3), 241-55. [PubMed:17085782] [WorldCat] [DOI] - ↑
Fox, A.N., & Zinn, K. (2005).
The heparan sulfate proteoglycan syndecan is an in vivo ligand for the Drosophila LAR receptor tyrosine phosphatase. Current biology : CB, 15(19), 1701-11. [PubMed:16213816] [WorldCat] [DOI] - ↑
Meng, K., Rodriguez-Peña, A., Dimitrov, T., Chen, W., Yamin, M., Noda, M., & Deuel, T.F. (2000).
Pleiotrophin signals increased tyrosine phosphorylation of beta beta-catenin through inactivation of the intrinsic catalytic activity of the receptor-type protein tyrosine phosphatase beta/zeta. Proceedings of the National Academy of Sciences of the United States of America, 97(6), 2603-8. [PubMed:10706604] [PMC] [WorldCat] [DOI] - ↑
Pariser, H., Perez-Pinera, P., Ezquerra, L., Herradon, G., & Deuel, T.F. (2005).
Pleiotrophin stimulates tyrosine phosphorylation of beta-adducin through inactivation of the transmembrane receptor protein tyrosine phosphatase beta/zeta. Biochemical and biophysical research communications, 335(1), 232-9. [PubMed:16105548] [WorldCat] [DOI] - ↑
Yoshida, T., Yasumura, M., Uemura, T., Lee, S.J., Ra, M., Taguchi, R., ..., & Mishina, M. (2011).
IL-1 receptor accessory protein-like 1 associated with mental retardation and autism mediates synapse formation by trans-synaptic interaction with protein tyrosine phosphatase δ. The Journal of neuroscience : the official journal of the Society for Neuroscience, 31(38), 13485-99. [PubMed:21940441] [PMC] [WorldCat] [DOI] - ↑
Paul, S., & Lombroso, P.J. (2003).
Receptor and nonreceptor protein tyrosine phosphatases in the nervous system. Cellular and molecular life sciences : CMLS, 60(11), 2465-82. [PubMed:14625689] [WorldCat] [DOI] - ↑
Kohda, K., Kakegawa, W., Matsuda, S., Yamamoto, T., Hirano, H., & Yuzaki, M. (2013).
The δ2 glutamate receptor gates long-term depression by coordinating interactions between two AMPA receptor phosphorylation sites. Proceedings of the National Academy of Sciences of the United States of America, 110(10), E948-57. [PubMed:23431139] [PMC] [WorldCat] [DOI] - ↑
Davis, M.J., Wu, X., Nurkiewicz, T.R., Kawasaki, J., Gui, P., Hill, M.A., & Wilson, E. (2001).
Regulation of ion channels by protein tyrosine phosphorylation. American journal of physiology. Heart and circulatory physiology, 281(5), H1835-62. [PubMed:11668044] [WorldCat] [DOI] - ↑
Dabrowski, A., & Umemori, H. (2011).
Orchestrating the synaptic network by tyrosine phosphorylation signalling. Journal of biochemistry, 149(6), 641-53. [PubMed:21508038] [PMC] [WorldCat] [DOI]