コネクトーム
英:connectome 独:Konnektom 仏:connectome 西:Conectoma 露:Коннектом 中:连接组
コネクトームは、神経系のすべての神経細胞が接続することでできた神経回路の全体を意味する。本来は、ある動物個体、あるいはある動物種の神経系において、全神経細胞の接続の総体を意味する概念である。しかしながら、神経系の一部で神経細胞が化学シナプスや電気シナプスで接続された局所的な接続ダイアグラム(Wiring diagram)に言及する場合にも、しばしば使われる。更に、神経細胞レベルでの接続だけでなく、脳の大まかな領域同士が接続する様相の説明にも使われている。コネクトミクス(connectomics)は、コネクトームを理解するための研究法、つまり実験、検出などの方法、その方法を適用した結果の解釈、そして複数の方法論を統合した研究の戦略についての概念である。
用語「コネクトーム」の起源
コネクトームは、生命科学分野で広く使われているゲノム(genome)、プロテオーム(proteome)などにならって、英語のconnect(ラテン語由来)とギリシア語の「完全、すべて、総体」を意味する接尾辞(-ome、ギリシア語-ωμα)を組み合わせた造語であり、英語の複数形は、connectomesである。コネクトームが、文献上、初めて使われたのは、2005年、Indiana大学のOlaf Spornsが、神経接続のマップを説明するために、ヒト・コネクトーム(Human Connectome)という概念を記述した論文である[1][2]。それとは独立して、Lausanne大学病院のPatric Hagmannが、その博士論文の中で同時期に同様に言及しているとされる[3]。特に、このコネクトームという概念が普及され始めたのは、2008年、Harvard大学のJeffrey W. LichtmanとJoshua R. Sanesによる共著総説論文の発表後である[4]。また、用語の定着には、Sebastian Seung(当時MIT、現Princeton大学)らのアウトリーチによる努力もあった[5]。しかし、これらの研究者の貢献は、あくまでコネクトームという用語を提案、普及、定着させたということで認知されるべきもので、コネクトームとは呼んでいないものの、それに相当する概念はこれらの研究者が提案する以前から存在していたと考えるべきであろう[6]。その典型例は、1986年に発表されたJohn Whiteらによるセンチュウ(Caenohabditis elegans)という一つの生物の神経細胞の結合性を網羅的に記述した研究である(後述)[7][8]。
コネクトームの研究史と階層
歴史的には、簡素な解剖用具などを用いて神経線維を観察することから、目と脳など神経組織同士を接続している構造が存在することは想像されていた(例:デカルト、1677年)。19世紀末になると、Santiago Rámon y Cajalが、個々の神経細胞の形態を明確に染め出すことを可能にしたGolgi染色と光学顕微鏡を用いることで、脳が多数の神経細胞とそれらの結合によって成り立っていることを提唱した。以後、神経細胞の間の結合を記述する研究は盛んに行われてきた[9]。Golgi染色やNissl染色などを施した連続切片を観察する時代を経て、20世紀中頃になると脳損傷後の変性神経線維をNauta法などで染色することで、神経回路の存在を確認する時代になった。1970年ごろになると、放射性アミノ酸や、酵素(HRP)などの軸索輸送を利用することで、神経回路の観察が簡便に行われるようになった。更に、1980年代には、脂溶性carbocyanine蛍光色素などの生体結合特性を持った蛍光色素(DiIなど)、植物レクチン(WGA、PHA-Lなど)、ビオチン誘導体(Biocytinなど)、軸索を効率的に移動できるコレラ毒素等の高感度トレーサーが開発され、多くの研究者に汎用されるようになった。そして、1990年代になると、蛍光顕微鏡に加えて、共焦点レーザー顕微鏡が普及し始め、デジタル画像として大規模なデータの保存と解析が扱えるようになってきた。コネクトーム研究の観点から、このような組織学的解剖と染色によって得られた知見をまとめた重要な研究が、1991年、FellemanとDavid van Essenらによるマカクサルの視覚系の結合性マッピングの概念の提出であった[10][11]。
このような研究手法は、Allen Brain InstituteのAllen Mouse Brain Connectivity Atlas[12]、Mouse Connectome Project (南カリフォルニア大学)[13]、マカクサルのCoCoMac(ドイツ)[14]などで、まとめられているようなコネクトーム収集プロジェクトにつながっている。これらは、神経系の解剖学的知見と組織学的研究を組み合わせたものであり、解像度的にはマイクロメーター からサブマイクロメーターレベルの「メソスケール Mesoscale」のコネクトームの情報となっている。このレベルのコネクトーム構築のもう一つのアプローチとしては、このような形態学的なアプローチとともに、電気生理学的アプローチ、更に光遺伝学、神経活動を間接的あるいは直接的に観察する細胞、組織レベルのアプローチ(カルシウム、活動電位、血流変化など)もある。しかし、現時点では、これらの方法論の多くは、大規模アプローチとしては適さず、局所的な回路に焦点を当てているか、あくまで予備的な解釈に用いられているのが現状である。
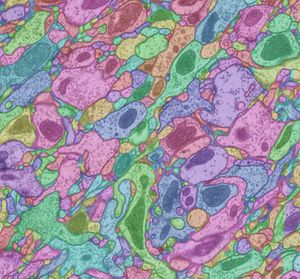
メソスケールのコネクトーム情報は、神経線維の走行や終末部位についての情報を大雑把に収集したものであり、シナプスレベルでの「ミクロスケール Microscale」の神経細胞間の結合性を記述したものではない。化学シナプスおよび電気シナプスを形態的に観察するためには、ナノメーターレベルの解像度がある電子顕微鏡が必要である。電子顕微鏡レベルで、ほぼ完全なコネクトーム解読に成功したのは、Sydney Brennerの大学院生であったJohn WhiteらによるセンチュウCaenohabditis elegansの神経系である(1986年発表)[15]。センチュウの場合、体のサイズが小型で、神経細胞の数が少なく(雌雄同体で302個)、その結合性も個体差が少なくステレオタイプである(75%再現性があると言われる)ので、コネクトームの構築が可能であった。一方、神経系のサイズが大きく、非常に多くの細胞と、その結合性に多様性がある脊椎動物の神経系のコネクトームの構築は極めて困難である。ミクロスケールなコネクトーム構築には、電子顕微鏡で観察するための多数の連続切片を失うことなく作製し、撮影し、その画像を保存し、結合性を解析していくための技術開発が行われてきている(下記参考)。その結果、マウス網膜、ショウジョウバエ視覚系、マウス大脳視覚野の部分的なコネクトームなどが構築された。これらの情報を総合的に収集しているのは、Open Connectome Project[16]である。
一方、Olaf Spornsによるヒト・コネクトームの提唱以来、脳の機能と病態を理解するためにヒトの脳で研究されているのは、メソレベルのコネクトームより更にスケールの大きな「マクロスケール Macroscale」のコネクトームである。これは小型の動物ではなく、ヒト、サル(マーモセットを含む)など比較的大型の動物での。この情報を収集しているのは、Human Connectome Project[17]である。これには、非侵襲なテンソルMRIなどを中心に用い神経線維の走行など解剖学的な側面に注目しているThe Harvard/MGH-UCLA Project[18]、および脳におけるfMRIによる活動領域の検出やゲノム情報など機能的な側面に重点を置く国際プロジェクトThe WU-Minn Project [19]がある。いずれも、解像度が上がれば、メソスケールのコネクトームにも近づくが、非侵襲で得られる解像度は、最大でもミリメートル程度であり、侵襲的な方法で得られる解像度とは違いがある。
以上、肉眼、光学顕微鏡のレベルである「メソスケール」、電子顕微鏡レベルである「ミクロスケール」、そして非侵襲で観察される脳の構造や活動を観察する「マクロスケール」の3つの階層での断絶が、コネクトームの研究では認識されているのが現状である。しかし、例えば、深度のある組織の観察を可能にする多光子励起顕微鏡、広い範囲を高速で観察できる光シート顕微鏡、光学顕微鏡の解像度を著しく向上させるナノスコピー(PALM, STORMなど)[20]、が改良されれば、これらのスケールの間の断絶を埋めることができる。
細胞レベルのコネクトームとコネクトミクス
解像度の観点からは、メソスケール、ミクロスケール、マクロスケールに分類されるコネクトームであるが、特にメソスケール、ミクロスケールで注目されるのは、神経細胞レベル、神経細胞のSubcellularレベルでの神経細胞同士の結合性である。つまり、どの神経細胞同士がシナプスで結合しているか、更にシナプスが細胞体や樹状突起のどの部分に存在しているか、という中核的な情報である。神経細胞同士の結合性を決定するには、上述した歴史的に利用されてきた方法論に加えて、最近の動向としては、以下のような6つの現代的なアプローチがあるが、それぞれのアプローチに長所、短所があり、互いに相補的なアプローチとなっていくものと予想される。
1)生理学的方法
生理学的な方法を利用し、神経細胞間の結合性を調べる。これには、複数神経細胞の全細胞記録法、ケージド神経伝達物質のレーザー光刺激法、光遺伝学、カルシウムイオンのセンサー(カルシウム感受性蛍光色素、GCaMPなどの遺伝学的なリポーター)、電位感受性センサーなどが利用される[21][22][23][24]。将来的に、哺乳類の神経系全体のコネクトームの解明には大規模生理学に適した方法論の開発が必要である。
2)シリアル電子顕微鏡
電子顕微鏡写真に基づき、形態的にコネクトームを構築することは、センチュウのコネクトーム構築でも利用された効果的な方法である[25][26][27][28][29]。しかしながら、哺乳類の脳のようにサイズの大きな構造におけるコネクトームの構築では、薄い切片を失うことなく、巨大な数の電子顕微鏡写真撮影を行い、それぞれの写真上の神経細胞とその突起、結合性を、多数の写真上で逐一トレースしていく必要がある。その情報量は、ビッグデータの典型であり、方法論の開発が進められてきている[30]。特に重要なのは、電子顕微鏡写真のトレースを一箇所間違えると、全く違う細胞をトレースすることになるという危険性があることである。そのため、Sebastian Seungらは、網膜のコネクトームを理解するために、ゲーム感覚で、神経細胞のコネクトーム構築に、一般市民を参加させようとするEyeWire[31]と名付けたクラウドサイトを構築している。これは、現状では、ヒトという作業者の目で電子顕微鏡写真を見て、それをトレースしていくことが、最も確実であるという見地から実施されているものであり、将来は、人工知能などにより、コネクトーム構築の作業が自動化される可能性も高い。このアプローチにおいては、神経細胞の広がりが小さく局所的なケースでは、電子顕微鏡写真上での追跡も比較的容易であろうが、例えば長い神経線維でつながった細胞同士のコネクトームを構築することは困難になる。
3)遺伝学的標識法
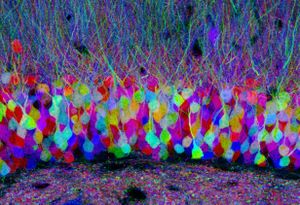
神経細胞を遺伝学的なレポーター(例、蛍光タンパク質)で標識し、神経細胞の形態と結合性を理解する方法論である。この方法論の特徴は、光学顕微鏡レベルでの観察が可能であるので、長い神経線維でつながった細胞同士のコネクトームの構築にも利用できることである。また、遺伝学的に標識できるため様々な神経細胞で特異的に発現するような遺伝子をドライバー(例、Cre、GAL4システム)を利用して、特定の神経回路のコネクトームについての知見を深めることができる。当初は、個々の神経細胞を蛍光タンパク質などで標識する方法が用いられていたが、コネクトーム構築には、多数の神経細胞を同時に観察する必要がある。そのために開発された方法論の1つが、Brainbowと呼ばれる技術である[32]。この技術は、確率論的、いくつかの蛍光団(XFPを)の組み合わせの発現を利用したもので、 各ニューロンは、Creリコンビナーゼとその基質となるlox配列を巧妙に利用することで、多数の異なる色によって区別できる。
これらの遺伝学的なツールの利用には、トランスジェニック動物、ノックイン動物、そして各種ウイルスベクターを用いることができる。中でも、神経細胞に効率的に遺伝子導入が可能であるアデノ随伴ウイルス(AAV)は、広く用いられている。一方、CRISPR・CAS9によるゲノム編集技術の発達とともに、このような遺伝学的ツールは広汎に用いられるようになると予想される。
特に、遺伝的なリポーターとして、電顕でその発現を観察できる方法は、2)の全体を再構築する方法と併用することで、様々なコンテキストで利用可能になるので注目される。とりわけ、最近開発されたARTEMIS法は、ペルオキシダーゼ活性を持つレポーター遺伝子を発現した神経細胞を、高品質な電顕画像の中で識別することができる[33]。
また、シナプス結合しているパートナーを調べるために、GRASPという方法が開発され、センチュウ、ショウジョウバエなどで利用されている[34]。また、GRASP法の他にも、その感度の低さを補うことが可能なsplit HRP法が開発され、哺乳類の神経系でも利用できることが示された。
4)Trans-synapticな方法
前項、遺伝学的標識法と類似しているが、より積極的にシナプス結合している神経細胞を探査していくコネクトームの構築法である。その1つは、小麦胚レクチン(WGA)などが、前シナプス部の細胞に導入された物質が、細胞質を介して直接は繋がっていない後シナプス部にシナプスを介して移行(Trans-synaptic)するということを利用するものである。これは、歴史的には、物質そのものを注入することで行われてきたが、ウィルスベクター、トランスジェニックマウスのような形で、遺伝学的に利用することが可能になっている。
もう1つの重要なアプローチは、同様な性質を持ったウィルスベクターを利用することである[35]。例えば、リポーター遺伝子を有するRabiesウィルスベクターは、Trans-synapticな移動をし、逆行性に輸送されることが知られており、前シナプス細胞のパートナーとなる神経細胞の標識が可能である。特に、ウイルスベクターのエンベロップタンパク質を変更することで、感染細胞を変更することができる[36][37]。
5)生体試料観察の工夫
細胞レベルでのコネクトームを理解するためには、それぞれの神経細胞とその突起の形態を高解像度で観察することが重要である。その一つの方法は顕微鏡の改良であるが、もう一つの方法論は神経組織の生体試料を観察しやすいように処理することである。特に、神経組織をホールマウントでその深部まで明確に観察できるようにするための方法論の開発が盛んである[38]。古くから、グリセロールなどを用いて組織を透明化する方法は用いられてきたが、Atsushi MiyawakiらによるScaleの発表以来、CLARITY, iDisco, SeeDBやその改良法により、より優れたプロトコールの開発が行われてきている[39][40]。また、Edward Boydenのグループは、組織そのものを拡大することができるExpansion Microscopyと名付けた方法を開発して注目されている[41]。
6)構成論的手法
システムを製作し、動作させることにより理解しようという方法論であり、例えば、シミュレーションやロボティクスを用いる。小規模な研究は、既に多くの神経科学者によって実施されてきている。とりわけ、この手法の代表的なものが、Henry Markumらによって開始されたBlue Brain Projectであり、欧州を中心とする神経科学、情報科学プロジェクトであるHuman Brain Projectの中核的プロジェクトと位置づけられている[42][43]。人工知能への関心の高まりとともに、今後の展開が注目される。
巨視的なコネクトーム
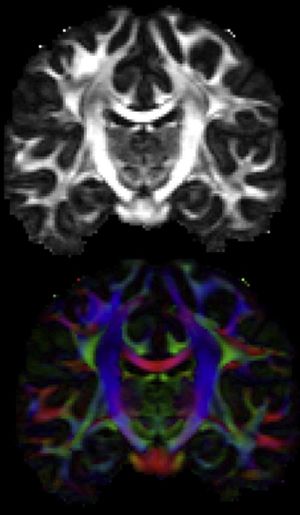
特にこのプロジェクトで最もよく利⽤されているのが、⾮侵襲脳計測法として、現在、ヒトの脳活動解析の主役となっている「fMRI」(functional MRI)だ。これは、1992年、Bell研究所にいた物理学者である⼩川誠⼆博⼠が磁気共鳴画像法(MRI)を応⽤して開発したものである。ニューロンが活動するとき、その近辺の⽑細⾎管を流れる⾚⾎球のヘモグロビンが運搬している酸素が消費され、脱酸素化ヘモグロビンが⽣じる。fMRIでは、MRIにより、⾎流の流れと、脱酸素化ヘモグロビンの濃度変化をみている(BOLD効果: Blood Oxygenation Level Dependent)。つまり、ニューロンの活動を直接観察しているわけではないので、実際のニューロンの活動とは、秒単位の時間的なズレがある。fMRIができるのはリアルタイムより少し遅れた時点での活動場所を推定するということである。 ⼀⽅、休⽌状態の⼤脳のある領域と別の領域が同調して⾃発的に変動するということが、結合状態にあるということを意味していると仮定すれば、fMRIを使って、領域間のつながりも推定することもできる(休⽌状態fMRI、図3)。この⽅法は、領域間の結合関係、つまりコネクトーム推定の有⼒な⼿段になっている。MRIは、⼀般に、⾼磁場を⽤いる装置ほど、⾼い空間分解能を持っているとされる。磁場は、テスラ(Tesla)という単位(1テスラは1万ガウスで,地球の磁場が約0.5ガウス)で表わされる。研究機関では、より⾼解像度を得るために、より⾼磁場のMRI装置を導⼊しようという努⼒している。 以下に紹介するような脳科学の先端研究では、現在は⼀般に3テスラから7テスラのものが使われており、1mm程度の空間的解像度があると⾔われている。しかし、原理的には、ニューロンの活動を観察しているわけではないので、解像度が上がっても、ある同じ領域でのシグナルが、全く同じニューロンの活動であるのか、というのは不明である。これは、脳研究や臨床診断でよく使われる脳電計(EEG)や脳磁計(MEG)についても当てはまる。EEGとMEGは、原理的には、ニューロンの神経活動の結果として⽣じる脳での電流や、その電流が⾮常に弱い磁場を引き起こすことを利⽤しているので、この場合は神経活動を直接計測している。しかし、多数のニューロンから⽣じる極めて弱い電流や磁気を検出するものなので、空間的解像度が低く、現状では脳マップやコネクトームを解析するというような⽬的には適さない。 実際に⼤規模なデータを集めてきているのは、世界で初めてサル脳の神経回路をコネクトーム様に描き出したDavid Van Essen博⼠(コネクトームへの挑戦(2)参照)をリーダーとする⽶Washington University(ミズーリ州セントルイス)と⽶University ofMinnesota、そして英Oxford Universityを中⼼とするコンソーシアムである。このプロジェクトでは、双⽣児ペアと300家族の家系を含む1200⼈の健康な成⼈を⽬標に、さまざまな⼿法のMRI、EEG、MEGを使うことで、それぞれの脳についてのデータ収集を続けている。また、同時にさまざまな⾏動関係のテストを実施している。被験者は、ボランティアで400ドルの⾦銭が⽀払われて、1⼈あたり2⽇間以上にわたるデータ取得が⾏われ、遺伝⼦検査のために⾎液も採集された。これは、遺伝⼦データと得られたコネクトーム情報を⽐較することで、脳回路形成に関わる遺伝⼦と環境の寄与を解明し、さらには、関連した遺伝⼦を特定するためという。 ⼀⽅、Harvard Universityの関連病院であるMassachusetts General HospitalのMartinos Centerと⽶University of California, Los Angels(現在はUniversity ofSouthern California)では、MRIの新しい⼿法、特に拡散MRIを⽤いたヒトコネクトームの解析に有⽤な⼿法を開発している。拡散MRIは、脳内にある軸索の束となった⻑距離の接続の様⼦をマッピングするものだ。⽔分⼦は脳の⼤部分ではランダムに運動している。ところが、軸索の束となった神経線維の中では、その線維の形に沿って⽔分⼦が流れるように動いている。拡散MRIでは、神経線維上で⽔分⼦が⽅向性を持って動いている部域とランダムに動いている部域の違いを⾒ることでシグナルを得ることになる。この⽅法を使うと、⽣きた脳の中で、そのまま神経の⾛⾏を観察することができるので、極めて便利である。もちろん、神経線維の⾛⾏をみているだけで、実際の結合性を⾒ているものではない。 このプロジェクトは、分解能、質、スピードを向上させることを⽬的とした技術開発である。 特に、Martinos CenterのVan Wedeen医師のグループは、拡散スペクトラムイメージング(diffusion spectrum imaging ;DSI)という⼿法を開発し、2012年のScience誌に掲載された美しい画像は、ヒトコネクトームプロジェクトの代表的成果として、先端脳科学の現状に関する紹介記事で頻繁に⾒かけるものになっている。
機能的コネクトーム
2012年6⽉の神経科学専⾨誌Neuronに、⽶Columbia University Kavli研究所のRichardYuste博⼠、⽶Harvard UniversityのGeorge Church博⼠らから「The Brain Activity MapProject and the Challenge of Functional Connectomics」と題する提案が掲載された。先回も紹介した⺠間財団Kavli財団が熱⼼なBrain Activity Map(BAM)プロジェクトについての提案である。 このBAM提案のキーパーソンとも⾔えるのが、Harvard Medical School遺伝学科の著名な分⼦⽣物学者George Church博⼠である。Church博⼠は、Walter Gilbert博⼠とゲノムシーケンシングの概念を提出し、これは現在、ゲノム解析の分野で重要な技術となっている次世代シーケンシングの⽯杖となった。2013年には、ゲノムエディティング技術として注⽬を集めているCRISPR-CAS9技術に関する論⽂を世界に先駆けて発表した。ゲノム解析において、このような先駆的な提案や技術開発を次々と⾏ってきたこともあり、バイオ研究界イノベーションの旗⼿としての同⽒の動向からは⽬が離せない。⼀⽅で、このBAMプロジェクトは、ヒトゲノムプロジェクトと⽐較した場合、その到達⽬標が実体としてあいまいであること、そして⽅法論や戦略が不⼗分であることが問題であると多くの研究者から指摘されている(これについては、別稿で改めて議論したい)。BRAIN Initiativeの中核をなす先⾏プロジェクトとして、Kavli 使用して機能的MRI(fMRIの)で休止状態とタスクの間に、コネクトーム回路の機能が検討されている。 [28]はちょうど地球の表面のように詳細なロードマップは、これらの道路を走行や車両の種類について多くを私たちに教えていません彼らはそのような意識のような特定の機能の動作を引き起こす、それは解剖学的接続に機能を関連付けるの理論を構築することが必要であるか神経構造を理解するために、どのような貨物運搬されている。[29]
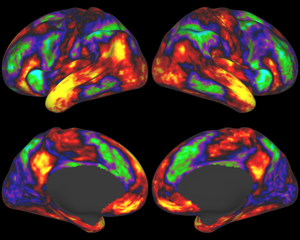
最後に、統計グラフ理論は、これらの脳のグラフ(ゴールデンら 、2009)を解析するための高度なパターン認識や推論ツールを開発している新興分野です。 「メソスケール」コネクトームは、数百マイクロメートルの空間分解能に相当します。 各個々のニューロンをマッピングしようとするのではなく、メソスケールでコネクトームは、ローカル回路個々のニューロンの数百または数千のリンク(例えば皮質カラム)によって形成された解剖学的および/または機能的に異なる神経細胞集団を、捕獲しようとしていました。 このスケールは、まだこの時点では非常に野心的な技術的課題を提示し、ローカルスケールでの侵襲性の技術または超高磁場MRIと小規模でプローブすることができます。
人間の脳では、コネクトームの意義は、人間の脳の構造と機能が複雑に複数のレベルと脳の接続のモードを介して、リンクされているという認識に由来します。 ニューロンまたは神経集団が相互に作用する、またはどのように強いまたはそれらの相互作用がある向けることが可能な強力な自然な制約があります。 実際、人間の認知の基礎はコネクトームによって形作ら動的相互作用のパターンです。 しかし、脳内の構造 - 機能関係は、単純な1対1マッピングに低減しにくいです。 実際には、コネクトームは明らかに現在の感覚入力、グローバルな脳の状態、学習と発展に応じて、変数の動的状態の大きな数をサポートすることができます。 機能状態でいくつかの変更樹状突起棘(ボンヘッファーとユステ、2002)の急速な出現と消失を示す二光子イメージング実験によって明らかにされているように、シナプスレベルでの構造的な接続性の急激な変化を伴うことがある。[11] このような複雑な変数の構造 - 機能マッピングにもかかわらず、コネクトームは、単一細胞記録から機能的神経画像に、動的な脳データの機構的な解釈に不可欠な基礎です。 :connectomicsの究極の目標は、マッピングすることである人間の脳を 。 この取り組みは、によって追求されたヒトコネクトームプロジェクトが主催する、 国立衛生研究所 、その焦点健康、生きている成人の人間の脳のネットワークマップを構築することです。脳ネットワーク脳画像の空間分解能(コッター、2007、Sporns、2010)のレベルに対応する、規模の異なるレベルで定義することができます。 [13] [14]これらのスケールは、大きくマイクロ、メソスケールとマクロスケールとして分類することができます。 最終的には、単一ニューロンからの皮質領域のような大規模システムへの神経細胞の集団の範囲で所定の種の神経組織の単一階層マップに異なるスケールで得られconnectomicマップに参加することが可能です。 主な実験データからの接続を推論に関わる方法論的不確実性を考慮すると、異なる個体のconnectomesに大きな差がある可能性が高いことを考えると、任意の統一されたマップは、おそらくSporns ら (接続のデータの確率的表現に依存しています。2005年)。[7]
マクロスケール(ミリメートル分解能)でコネクトームは、それぞれの接続の明確なパターンを有する、解剖学的に別個のモジュール(エリア、小包またはノード)にparcellatedすることができ、大きな脳のシステムをキャプチャしようとします。 メソスケールとマクロスケールでConnectomicデータベースは、携帯の解像度でよりも有意によりコンパクトかもしれないが、彼 らは、ネットワーク・ノードへの神経ボリュームの正確な解剖学的または機能的parcellationための効果的な戦略を必要とする(複雑さについては、例えば、ウォレスら、2004) 。[16]
コネクトームの利用
コネクトームを用いてネットワークとして研究することができるネットワークの科学とグラフ理論 。 マイクロスケールコネクトームの場合には、このネットワーク(またはノードのグラフが )ニューロンであり、エッジは、これらのニューロン間のシナプスに相当します。 グラフのエッジがそれらの領域を相互接続する軸索から誘導されながら、マクロスケールのコネクトームの場合、ノードは、のROI(関心領域)に対応しています。 彼らは実際に脳(または、広義には、全体の神経系)内の接続を説明する数学的な意味でのグラフであるとしてこのようconnectomesは時々 、 脳のグラフと呼ばれています。
研究者の一群(Iturria-メディナら 、2008)[30]使用してコネクトームデータセットを構築した拡散テンソル画像 (DTI)[31] [32] 70-90皮質と基礎の間で平均接続確率の導出に続いての脳の灰白質領域。 すべてのネットワークは、小さな世界の属性と「広いスケール」度分布を有することが見出されました。 これらのネットワークでの間隔度の中心性の分析は、高中心性を実証した楔前部 、 島 、 頭頂および前頭皮質。 別のグループ(功ら2008)[33]は 78皮質領域間の解剖学的接続のネットワークをマッピングするためにDTIを適用しております。 この研究はまた楔前部と上前頭回を含む、ヒトの脳のいくつかのハブ領域を同定しました。
ハグマンら (2007)[34]は 500と4000の間に約1000個のROIと約のために取得した接続行列の定量分析をナンバリング利息(ROIを)の均一に分布し、同じサイズの領域の間に測定された繊維の密度からの接続行列を構築2人の被験者から50,000繊維経路は指数関数(1スケール)次数分布だけでなく、堅牢なスモールワールドネットワークの属性を明らかにしました。 データセットは、拡散スペクトルイメージング(DSI)(Wedeen、2005)から誘導された[35]拡散強調イメージングの変異体[36] [37]線維トラクトを交配することによって引き起こされる拡散方向のイントラボクセル不均一に敏感ですしたがって、他の拡散イメージング手法(Wedeen、2008)よりも軸索の軌跡をより正確にマッピングすることができます。[38]全頭DSIデータセットの組み合わせは、ハグマンらによって開発されたアプローチに従って取得し、 処理することができる。(2007)[34] ;グラフ分析ツールを用いて動物の追跡研究(Sporns、2007 Sporns、2006)のために最初に考案[39] [40]人間の皮質接続のネットワーク構造の詳細な研究を許可(ハグマンら、2008)。[41]人間の脳のネットワークは、コア分解、モジュール性分析、ハブの分類と中心性を含むネットワーク解析法の広範な配列を使用して特徴付けられました。 ハグマンら 。 後部内側および頭頂葉皮質に主に位置し、高度かつ相互に相互接続された脳領域の構造コアの存在の証拠を提示しました。 コアはすべて、両方の大脳半球に位置する後部帯状皮質、楔前部、楔状部、傍小葉、帯状の地峡、上側頭溝の銀行、劣ると優れた頭頂皮質の部分を含みます。
connectomicsのサブフィールドは、複数の人の脳のグラフの比較を扱っています。 それは、次のようなコンセンサスグラフ構築することが可能であるブダペストリファレンスコネクトームを特定の方法でいくつかのconnectomesを「平均化」することによって。 一方、一部の研究者は、個々のconnectomesの違いの原因を調査しました。 それは、女性のマクロスケールconnectomesは男性よりも有意に多くのエッジが含まれていることが見出されており、女性のconnectomesにおけるエッジの大部分は、2つの半球の間で実行する。[42] [43]また、概してconnectomes展示スモールワールド年齢とともに減少し、全体的な皮質の接続を持つ文字を、。 [44]として2015年の継続的な目的のHCP寿命パイロットプロジェクトが 6の年齢層(4-6、8-9、14-間のコネクトームの違いを識別することです15、25-35、45-55、65-75)。 Connectogramsはローブが主催する円の周りの皮質領域を配置することによって、完全な脳データを視覚化するために使用されている。 [45] [46]内側の円は、カラースケールで皮質のメトリックを示しています。 DTIデータにおける白質のファイバ接続は、これらの皮質領域の間に引かれたとによって重み付けされる異方性比率との接続強度。 このようなグラフは、さらに有名な外傷性脳損傷患者にダメージを分析するために使用されているフィニアスゲージ 。[47]統計グラフ理論は、これらの脳のグラフを解析するための高度なパターン認識や推論ツールを開発している新興分野です(ゴールデンら、2009)。
参考文献
- ↑
Sporns, O., Tononi, G., & Kötter, R. (2005).
The human connectome: A structural description of the human brain. PLoS computational biology, 1(4), e42. [PubMed:16201007] [PMC] [WorldCat] [DOI] - ↑ Olaf Sporns (2010) Scholarpedia, 5(2):5584. [DOI]
- ↑ Patric Hagmann (2005) From diffusion MRI to brain connectomics [PhD Thesis]. Lausanne: Ecole Polytechnique Fédérale de Lausanne (EPFL). [DOI].
- ↑
Lichtman, J.W., & Sanes, J.R. (2008).
Ome sweet ome: what can the genome tell us about the connectome? Current opinion in neurobiology, 18(3), 346-53. [PubMed:18801435] [PMC] [WorldCat] [DOI] - ↑ Sebastian Seung (2013) Connectome: How the Brain's Wiring Makes Us Who We Are (Mariner Books) ISBN-10: 9780547678597 (邦訳「コネクトーム:脳の配線はどのように「わたし」をつくり出すのか」草思社 セバスチャン・スン (著), 青木 薫 (翻訳))
- ↑
Bargmann, C.I., & Marder, E. (2013).
From the connectome to brain function. Nature methods, 10(6), 483-90. [PubMed:23866325] [WorldCat] - ↑
White, J.G., Southgate, E., Thomson, J.N., & Brenner, S. (1986).
The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 314(1165), 1-340. [PubMed:22462104] [WorldCat] [DOI] - ↑
Emmons, S.W. (2015).
The beginning of connectomics: a commentary on White et al. (1986) 'The structure of the nervous system of the nematode Caenorhabditis elegans'. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 370(1666). [PubMed:25750233] [PMC] [WorldCat] [DOI] - ↑
Lanciego, J.L., & Wouterlood, F.G. (2011).
A half century of experimental neuroanatomical tracing. Journal of chemical neuroanatomy, 42(3), 157-83. [PubMed:21782932] [WorldCat] [DOI] - ↑ • Felleman DJ, Van Essen DC (1991) Distributed hierarchical processing in the primate cerebral cortex. Cereb Cortex 1, 1-47.
- ↑ Science. 1992 Jan 24;255(5043):419-23. Information processing in the primate visual system: an integrated systems perspective. Van Essen DC1, Anderson CH, Felleman DJ.
- ↑ http://connectivity.brain-map.org/
- ↑ http://www.mouseconnectome.org/
- ↑ http://cocomac.g-node.org/
- ↑ White JG, Southgate E, Thomson JN, Brenner S (1986) The structure of the nervous system of the nematode Caenorhabditis elegans. Phil. Trans. Royal Soc. London. B 314, 1-340.
- ↑ http://www.openconnectomeproject.org/
- ↑ http://www.neuroscienceblueprint.nih.gov/connectome/
- ↑ http://www.humanconnectomeproject.org/
- ↑ http://humanconnectome.org/
- ↑ Tønnesen, J. & Nägerl, U. V. Superresolution imaging for neuroscience. Experimental neurology 242, 33–40 (2013).
- ↑ Large-scale recording technology: Scaling up neuroscience Francesco P Battaglia Mark J Schnitzer Current Opinion in Neurobiology 2015, 32:iv–vi doi:10.1016/j.conb.2015.03.002
- ↑ Cell. 2016 Mar 10;164(6):1136-50. doi: 10.1016/j.cell.2016.02.027. Communication in Neural Circuits: Tools, Opportunities, and Challenges. Lerner TN1, Ye L1, Deisseroth K2.
- ↑ Cell. 2016 Apr 21;165(3):524-34. doi: 10.1016/j.cell.2016.03.047. Targeting Neural Circuits. Rajasethupathy P1, Ferenczi E1, Deisseroth K2.
- ↑ All-Optical Interrogation of Neural Circuits Valentina Emiliani1, Adam E. Cohen2,3, Karl Deisseroth4,5, and Michael Häusser6,7 +Show Affiliations The Journal of Neuroscience, 14 October 2015, 35(41): 13917-13926; doi: 10.1523/JNEUROSCI.2916-15.2015
- ↑ Briggman, K. L., Helmstaedter, M. & Denk, W. Wiring specificity in the direction- selectivity circuit of the retina. Nature 471, 183–8 (2011).
- ↑ Bock, D. D. et al. Network anatomy and in vivo physiology of visual cortical neurons. Nature 471, 177–82 (2011).
- ↑ Takemura, S. Y. et al. A visual motion detection circuit suggested by Drosophila connectomics. Nature 500, 175–81 (2013).
- ↑ Kasthuri, N. et al. Saturated Reconstruction of a Volume of Neocortex. Cell 162, 648–61 (2015).
- ↑ Morgan, J.L, Berger, D.R., Wetzel, A.W., & Lichtman, J.W. The fuzzy logic of neuronal connectivity in the mouse visual thalamus. Cell 165, 192–206 (2016).
- ↑ Plaza, S. M., Scheffer, L. K. & Chklovskii, D. B. Toward large-scale connectome reconstructions. Current opinion in neurobiology 25, 201–10 (2014).
- ↑ http://eyewire.org/
- ↑ A technicolour approach to the connectome Jeff W. Lichtman, Joshua R. Sanes, Jean Livet Nat Rev Neurosci. Author manuscript; available in PMC 2008 December 1.
- ↑ Reconstruction of genetically identified neurons imaged by serial-section electron microscopy
- ↑ Wickersham, I. R. & Feinberg, E. H. New technologies for imaging synaptic partners. Current opinion in neurobiology 22, 121–7 (2012).
- ↑ Wickersham, I. R. & Feinberg, E. H. New technologies for imaging synaptic partners. Current opinion in neurobiology 22, 121–7 (2012).
- ↑
Callaway, E.M., & Luo, L. (2015).
Monosynaptic Circuit Tracing with Glycoprotein-Deleted Rabies Viruses. The Journal of neuroscience : the official journal of the Society for Neuroscience, 35(24), 8979-85. [PubMed:26085623] [PMC] [WorldCat] [DOI] - ↑
Schwarz, L.A., Miyamichi, K., Gao, X.J., Beier, K.T., Weissbourd, B., DeLoach, K.E., ..., & Luo, L. (2015).
Viral-genetic tracing of the input-output organization of a central noradrenaline circuit. Nature, 524(7563), 88-92. [PubMed:26131933] [PMC] [WorldCat] [DOI] - ↑
Marx, V. (2016).
Optimizing probes to image cleared tissue. Nature methods, 13(3), 205-9. [PubMed:26914203] [WorldCat] [DOI] - ↑
Miyawaki, A. (2015).
Brain clearing for connectomics. Microscopy (Oxford, England), 64(1), 5-8. [PubMed:25652426] [WorldCat] [DOI] - ↑ http://clarityresourcecenter.org/
- ↑
Chen, F., Tillberg, P.W., & Boyden, E.S. (2015).
Optical imaging. Expansion microscopy. Science (New York, N.Y.), 347(6221), 543-8. [PubMed:25592419] [PMC] [WorldCat] [DOI] - ↑ http://bluebrain.epfl.ch/
- ↑
Markram, H., Muller, E., Ramaswamy, S., Reimann, M.W., Abdellah, M., Sanchez, C.A., ..., & Schürmann, F. (2015).
Reconstruction and Simulation of Neocortical Microcircuitry. Cell, 163(2), 456-92. [PubMed:26451489] [WorldCat] [DOI]