「プロテアソーム」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| 1行目: | 1行目: | ||
<div align="right"> | <div align="right"> | ||
<font size="+1">[http://researchmap.jp/read0160429/?lang=japanese 田中 啓二]、[ | <font size="+1">[http://researchmap.jp/read0160429/?lang=japanese 田中 啓二]、[http://kaken.nii.ac.jp/d/r/80462779.en.html 佐伯 泰]</font><br> | ||
''東京都医学総合研究所 | ''東京都医学総合研究所 蛋[[白質]]代謝研究室''<br> | ||
DOI:<selfdoi /> 原稿受付日:2013年3月14日 原稿完成日:2014年3月6日<br> | DOI:<selfdoi /> 原稿受付日:2013年3月14日 原稿完成日:2014年3月6日<br> | ||
担当編集委員:[http://researchmap.jp/2rikenbsi/?lang=japanese 林 康紀](独立行政法人理化学研究所)<br> | 担当編集委員:[http://researchmap.jp/2rikenbsi/?lang=japanese 林 康紀](独立行政法人理化学研究所)<br> | ||
2015年9月2日 (水) 11:50時点における版
田中 啓二、佐伯 泰
東京都医学総合研究所 蛋白質代謝研究室
DOI:10.14931/bsd.3479 原稿受付日:2013年3月14日 原稿完成日:2014年3月6日
担当編集委員:林 康紀(独立行政法人理化学研究所)
英語名:proteasome 独:Proteasom 仏:protéasome
真核生物の細胞内には進化的に保存された二つのタンパク質分解系、ユビキチン・プロテアソームシステム(UPS)とオートファジー・リソソームシステムが存在している。UPSは選択的タンパク質分解を担う中心的な酵素系であり、一方、オートファジー・リソソームシステムは一般に非選択的なタンパク質分解を担っているが、最近障害したタンパク質凝集体、異常・過剰オルガネラ、細胞内に侵入した細菌等を得特異的に排除する選択的オートファジーが注目されている。プロテアソームは、ユビキチン化されたタンパク質を選択的に分解する巨大で複雑なタンパク質分解酵素複合体である。ユビキチン化されたタンパク質の除去により、細胞周期・アポトーシス・シグナル伝達・転写制御・品質管理・シナプス可塑性などに関与する。これらの破綻により異常タンパク質の細胞内蓄積を来たし、神経変成疾患の発症につながる。
プロテアソームとは
タンパク質は、細胞内で絶えず合成と分解を繰り返しており、動的なリサイクル(新陳代謝)システムを構成している。特にタンパク質分解は多様な生体反応を不可逆的に制御する方法として発生や分化など様々な生命現象に不可欠な役割を果たしている。また細胞内に生じた不良品の積極的な除去に深く関与しているほか、良品であっても不要な(細胞活動に支障をきたす)場合、あるいは緊急時の栄養素の確保のために、積極的に動員される。このようにタンパク質は細胞内でリサイクルし, 動的平衡を保つことによって身体の中を浄化(不要品のクリアランス)して新鮮さを保ち、健康を維持している。
真核生物の細胞内には進化的に保存された二つのタンパク質分解系、ユビキチン・プロテアソームシステム(UPS)とオートファジー・リソソームシステムが存在しているが、このうちUPSは選択的タンパク質を担う中心的な酵素系で、プロテアソームは、ユビキチン化されたタンパク質を選択的に分解する細胞内装置である。
ユビキチンシステム
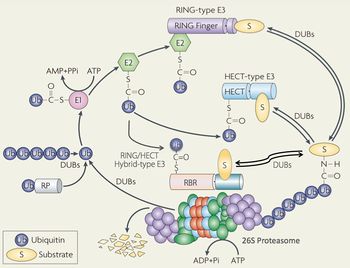
Ub:ユビキチン、E1: Ub活性化酵素、E2: Ub結合酵素、E3: Ubリガーゼ(連結酵素)、DUB: 脱ユビキチン酵素。基質を選別するE3酵素は、HECT型とRING型に大別される(最近では、HECT型とRING型のハイブリッド型E3としてRBR型E3ファミリーが提唱されている)。26Sプロテアソームは不要なユビキチン化タンパク質をエネルギー依存的に分解する巨大なタンパク質分解酵素複合体である[1]。UPSの作動機構においてATPの加水分解は、基質のユビキチン化(E1の作用)と26Sプロテアソームによる分解作用(基質のアンフォールディング)の二つのプロセスに必要である。
1977年、米国ハーバード大学のGoldbergのグループは網状赤血球の抽出液がエネルギー依存性のタンパク質分解活性を示すことを見いだした[2]。その後間もなく、イスラエルのHershkoとCiechanoverは、米国のRoseと共に、熱安定性の小さなタンパク質であるユビキチンがその主役であることを見出した。
ユビキチンは76個のアミノ酸からなる小さなタンパク質であり、進化的保存性が高くそのアミノ酸配列は全ての真核生物でほとんど同じである。1980年頃までに彼らは、ユビキチンが活性化酵素(E1:2種のヒト遺伝子にコードされる)・結合酵素(E2:約20種)・リガーゼ(E3:約600種)から構成された複合酵素系(ユビキチンシステム)によって標的タンパク質に共有結合(ユビキチンのC末端のカルボキシル基とタンパク質中のリジン残基のε-アミノ基が縮合したイソペプチド結合)する翻訳後修飾分子であることを明らかにした(図1)[3] [4]。このE1の作用にはATPの加水分解が必要である。そしてタンパク質に結合したユビキチン内の(主として48番目の)リジン残基と新しいユビキチン分子内のC末端のグリシンの間でイソペプチド結合ができ、さらにユビキチン分子間での縮合反応を繰り返すことによって、多数のユビキチン分子が鎖状に伸長したポリユビキチン鎖が形成される。
Hershkoら及びVarshavskyらは生じたポリユビキチン鎖が基質タンパク質を分解装置に輸送するためのシグナル(目印)として機能するという“ユビキチンシグナル”仮説を提唱した[3] [4] [5]。この仮説は、ポリユビキチン鎖の形成が(全体の反応としては)分解シグナルの提示反応であるが、実際に起きている化学反応は(イソ)ペプチド結合の形成(タンパク質合成と類似の反応)であり、エネルギー要求性を合理的に説明できた。興味深いことに細胞内には、ユビキチン化の逆反応を触媒する脱ユビキチン酵素(deubiquitylating enzyme:DUB)あるいはubiquitin specific protease(USP)が存在し、それらは生物種を問わず大きな遺伝子ファミリー(約80種)を形成している。多数のDUBが存在することは、ユビキチン化による翻訳後修飾が可逆的かつ多面的であることを示唆している。2004年、ユビキチンシステムの発見者たち3名は、ノーベル化学賞を受賞した。
ユビキチンは二つの異なったタイプの遺伝子にコードされている。一つは、ユビキチンとリボソームタンパク質の融合遺伝子であり、もう一つは数個〜10数個のユビキチンがタンデムに連なったポリユビキチン遺伝子である。これらの融合タンパク質からユビキチンを切り出す際にも、上述のDUBが使用される。ポリユビキチン遺伝子は、1回の転写・翻訳で多数のユビキチンを合成することができる点で秀逸であり、かつ熱ショック応答遺伝子でもあることから、細胞は環境ストレスに曝されたとき、必要とするユビキチンを迅速に大量生成することができるよう合理的に設計されている。このことは、細胞内のユビキチンレベルが外環境の変化に応答して厳格に制御されていることを示唆している。実際、ユビキチンの量は、多くても少なくても細胞は異常になり、常に適切に保たれる必要がある。
プロテアソームの発見
1983年、われわれはエネルギーを必要とするユビキチン化反応のステップに加えて、ユビキチン化タンパク質の分解にATPのエネルギーが必要であることを見出し、“エネルギー依存性タンパク質分解機構の2段階説”を発表した[6]。後に、このATP要求性のタンパク質分解反応を触媒する酵素が、真核生物のATP依存性プロテアーゼであることが判明し、1988年、プロテアソーム(プロテアーゼ活性を有した巨大粒子〜some)と命名した。
プロテアソームの分子構造
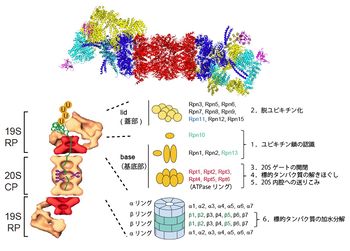
上図:26Sプロテアソームの単粒子解析とX線結晶構造解析に基づいた高次構造。
左図:26Sプロテアソーム(CPとRPの複合体)の電子顕微鏡による分子形状(単粒子解析: 独マックスプランク研究所W. Baumeister、S. Nickellから供与)。U:ユビキチン。
右図:サブユニットの構成モデル.触媒粒子(CP、20Sプロテアソーム)はα/βリングがαββαの順に会合した円柱状粒子(図3参照)。調節粒子(RP、PA700)はlid(蓋部)とbase(基底部)から構成された複合体。RPはRpn(RP non-ATPase)とRpt(RP triple-ATPase)サブユニット群から構成されている。Rpn10、Rpn13:ポリユビキチンリセプター、Rpn11:DUB(脱ユビキチン酵素)、 β1 (カスパーゼ様活性), β2(トリプシン様活性), β5(キモトリプシン様活性):触媒サブユニット。図には示していないが、DUBであるUSP14はRpn1に、そして Uch37はRpn13を介してRpn2に会合している(図4参照)。
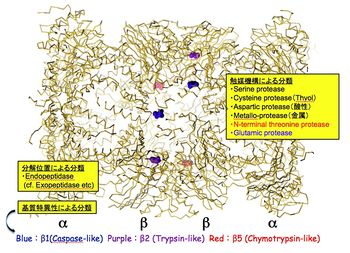
詳細は本文参照。プロテアソームは、N-末端スレオニンプロテアーゼ(エンドペプチダーゼ)というユニークな酵素である。
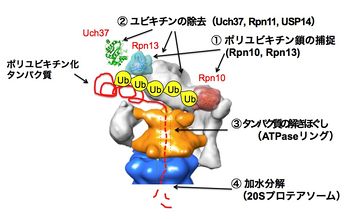
Ub:ユビキチン、Rpn10、Rpn13:ユビキチンリセプター、Rpn11、USP14/(酵母のUbp6)、Uch37:脱ユビキチン酵素
触媒粒子(core particle, CP、20Sプロテアソーム)の両端に調節粒子(regulatory particle, 19S RP)が会合した分子量250万、総サブユニット数66個から構成されたATP依存性プロテアーゼ多成分複合体を26Sプロテアソームという(図2下パネル)[9] [10] [11]。原子レベルでの構造は不明であり、現在、極低温電子顕微鏡(cryo-electron microscopy; Cryo-EM)による単粒子解析と個々の構成サブユニットの結晶構造を組み合わせた解析が進行中である[8] [12](図2上パネル)。
また19S RP(別称:PA700)以外の活性化因子(PA28, PA200等)の存在や、20Sプロテアソームが活性化因子の介在なしに天然変成タンパク質や酸化修飾タンパク質を直接分解することも報告されている[11] [13] [14]。
触媒粒子
触媒粒子はαリングとβリング(各々7種のサブユニットから構成)がαββαの順で会合した分子量75万の円筒型粒子(α1-7β1-7β1-7α1-7)である。本酵素はカスパーゼ型(β1)、トリプシン型(β2)、キモトリプシン型(β5)の触媒活性を有しており、これらの活性中心はβリングの内表面に露出している。CPは、通常、αリングが閉じているため細胞内では不活性型として存在している(図3)。
調節粒子
PA700
PA700は体表的な調節粒子である。Lid(蓋部)とbase(基底部)から構成されており,lid複合体とbase複合体は、各々10個と9個のサブユニットから構成されている。二つのユビキチンリセプターRpn10とRpn13は分子表面の離れた位置に存在してユビキチン化タンパク質を捕捉している[7]。調節粒子にはポリユビキチン鎖を根本から切断して解離するRpn11と、それ以外に末端からユビキチンを1個ずつ解離させる酵素USP14(酵母のUbp6)とUch37(酵母には存在しない)の3つの脱ユビキチン酵素が存在する。
低温電子顕微鏡よる解析からlidサブユニット群の位置情報が明らかにされている[8]。またbaseは6種のAAA型ATPaseサブユニット(Rpt1〜Rpt6)を含んでおり、この冠(Crown)型構造のATPaseリングは,触媒粒子のαリングと結合してその中央部のゲートを開き,基質タンパク質の通過を可能にさせる機能を有している他、ATPの加水分解エネルギーを利用してタンパク質の3次元構造を破壊(アンフォールディング)し,変性した基質がαリングを通ってβリングの内部に到達できるようにするアンチシャペロン作用を持っている[15] [16] [17](図4)。
PA28
RP/PA700以外の活性化因子としてPA28α、β、γが知られている[18][19]。
PA28α/βからなるヘテロ7量体は細胞質局在に局在し、インターフェロンγによって強く誘導され、内在性抗原のプロセッシングに関与している[11]。
ホモ7量体を形成しているPA28γは核に局在し、その欠損マウスは成長が遅延する[11]。
また20Sプロテアソームの両端にPA700とPA28の両調節ユニットを併せ持った“ハイブリッドプロテアソーム”も存在する[20]。
PA200
PA200(酵母のBlm10)も活性化因子として知られ、酵母からヒトまで普遍的に存在するが、その役割は諸説あって確定していない[21]。
プロテアソーム複合体形成に関与するシャペロン分子
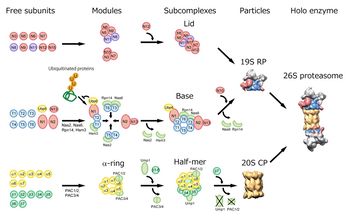
詳細は本文参照。
プロテアソームの分子集合には専門的な多数のシャペロン分子が関与している(図5)[1] [22] [23]。
Proteasome Assembling Chaperone
20Sプロテアソームの形成に特化した分子シャペロンProteasome Assembling Chaperone (PAC)1-4は、7種のαサブユニットと階層性をもって結合し、αリングの形成を促進する。PAC1/PAC2ヘテロ二量体はαリング同士の凝集体の形成を阻止する働きを示し、PAC3/PAC4ヘテロ二量体はαリング上へのαサブユニットの段階的な会合を促進して、迅速に正確なαリングを形成させる。βサブユニット群は、逐次的にαリング上に会合してβリングを形成する。この段階的な会合にはβ2やβ5のプロペプチド及びβ2のC末端伸長領域などが“分子内シャペロン”として作用する[1]。
Ump1/POMP/Proteassemblin
βサブユニットの会合やハーフ・プロテアソームの重合プロセスに関与している[1] [24]。
RP assembling chaperones
19S RPのbaseを構成するATPase リングの分子集合に関与するシャペロン分子である。これらは、それぞれ当初プロテアソームと一時的に結合するタンパク質(proteasome-interacting proteins、 PIPs:数十個存在)として同定されていた分子群、即ちNas2/p27、Nas6/gankyrin (p28)、Rpn14/PAAF1、Hsm3/S5b(出芽酵母/ヒト)であった[22]が最近、RP assembling chaperones (RAC) 1-4と呼ばれている[23]。
UBP6/USP14
ユビキチン鎖のbase中間体への偶発的な結合を阻止し、base複合体の形成を促進する[25]。
生理機能
詳細は本文参照。
ユビキチン-プロテアソーム系をコードする遺伝子の数は、ゲノム総遺伝子数の3〜5 %を占めると推定されており、細胞周期・DNA修復・アポトーシス・シグナル伝達・シナプス可塑性・転写制御・代謝調節・免疫応答・タンパク質の品質管理・ストレス応答・感染応答など、迅速に、順序よく、一過的にかつ一方向に決定する手段としての役割を担っている[26]。これらの生理作用は、細胞内における標的タンパク質の量の厳密な制御を反映しており、とくにユビキチンシステムの多様性に負うところが大きい。一方、プロテアソームは分解系としての役割以外に、前駆体タンパク質のプロセシングによる活性型への転換(例えば、NF-κBの成熟プロセス)やその生成ペプチドを抗原エピトープとして利用するなどポジティブな生命応答に貢献していることも知られている。
免疫系
免疫プロテアソーム
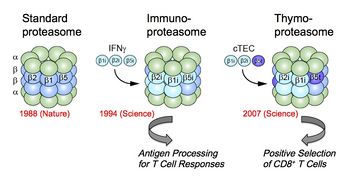
適応(獲得)免疫の中心的なテーマである自己と非自己の識別(細胞性免疫応答)において必須な役割を果たすために、プロテアソームのアイソフォーム(免疫型プロテアソーム)(図5)が存在する[11] [27][28] [29] [30]。免疫型プロテアソームの遺伝子は、進化的には適応免疫と同時期に誕生した[28] [31]。 主要組織適合性遺伝子複合体 (major histocompatibility antigen comple; MHC)を獲得した有顎脊椎動物では、プロテアソームはMHCクラスI結合ペプチド産生の必須酵素でもあり、CD8+T細胞を介した細胞性免疫応答に不可欠な役割を果たしている。ウイルスやガン抗原等の内在性抗原のプロセシング酵素として専門的に作用する酵素が存在する。これは標準/構成型プロテアソーム(standard/constitutive proteasome)と区別して、“免疫プロテアソーム(immunoproteasome)”と呼ばれる[32] [33] [34]。この亜型酵素はインターフェロンγ(IFNγ)などのサイトカインにより強く誘導される3種の新しいβ型触媒サブユニット(β1i, β2i, β5i)が優先的分子集合機構によって分子内置換した酵素である(図6中モデル)。
免疫プロテアソームは高いキモトリプシン様活性を有し、MHCクラスIのペプチド収容溝に高い親和性をもつペプチドを効率的に産生することができる(分子レベルでの自己と非自己の識別)。当初免疫プロテアソームは抗原プロセシングに特化した酵素と見られていたが、最近、免疫プロテアソームが有害タンパク質の凝集阻止を通してインターフェロン依存的な酸化ストレスによる細胞死を防御していること[35]やβ5iの特異的な阻害剤PR-957がサイトカインの産生や自己抗体レベルを低下させることから自己免疫疾患に関与していること[36] [37]が報告されている。
胸腺プロテアソーム
脊椎動物の胸腺皮質上皮細胞cTECにはβ5tという新規な触媒サブユニットが特異的に発現している。β5tの組み込まれた(β1iとβ2iをパートナーとする)亜型酵素は、胸腺プロテアソーム(thymoproteasome)と呼ばれる(図6右モデル)[38] [39]。
胸腺プロテアソームは、MHCクラスIに結合するリガンド(抗原エピトープ)の種類を変化させている。β5t欠損マウスではCD8+T細胞が減少し、リンパ球分化(様々なT細胞受容体を持った有用なCD8+T細胞のレパトア形成)に異常をきたしたことから、胸腺プロテアソームが胸腺における“正の選択”を駆動する抗原ペプチドを生成している(細胞レベルでの自己と非自己の識別)と考えられている[27] [39]。
シナプス可塑性
外界の刺激などに適応したシナプス(あるいは神経)の可塑性(信号伝達能力や形態の変化)は、脳の発生過程、老化や障害からの回復、あるいは記憶や学習などの高次の神経機能の基盤となっている。このシナプス可塑性においてタンパク質合成が重要であることは、以前から明確になっていたが、ニューロンにおけるタンパク質品質管理の研究が進展し、最近ではUPSによる選択的なタンパク質分解の重要性を示唆する報告が相次いでいる[40] [41]。UPSはシナプス可塑性の関与する神経伝達物質受容体、タンパク質キナーゼ、転写因子など様々なシナプスに存在するタンパク質を厳格に調節しているほか、海馬にける長期増強(long-term potentiation:LTP)などニューロンの局部での役割も注目されている[42]。またこれらの破綻による疾病も急増している[43]。さらにごく最近、長期記憶(long-term memory:LTM)におけるUPSの役割が注目されている[44] [45]。
老化
老化と共にプロテアソームの機能が低下するとの報告は、数多くある[46]が、実際にはプロテアソームの機能評価は必ずしも容易でなく、それらの信憑性には疑義がもたれていた。多くの場合、蛍光合成基質を用いたペプチダーゼ活性を指標とした報告であるが、これらの実験値が真にこの酵素の細胞内での機能レベルを正確に反映していることの保証はないからである。ところが最近、ショウジョウバエを用いた遺伝学的スクリーンから老化に依存したニューロンのproteotoxity(異常タンパク質の蓄積による細胞障害)を抑圧する遺伝子としてプロテアソームのRPサブユニット(Rpn11)が分離され、26Sプロテアソームの障害を起因としたプロテアソーム活性の低下が明らかとなった[47]。この結果は、プロテアソームの機能破綻が寿命の短縮に寄与していることを直接的に示しており、エイジングにおけるプロテアソームの役割の重要性が具体的に示唆された。
神経変性疾患

20Sプロテアソーム(αリング)の形成に必須な分子集合因子PAC1(図4参照)の条件的ノックアウトマウスをNestin-Creトランスジェニックマウス を交配させたマウスの生後3週間後の表現系と行動 [14]。
詳細は本文参照。
プロテアソームと神経凝集体
通常、活発に分裂している細胞の細胞質や核に蓄積した異常タンパク質(アンフォールド/ミスフォールドした変異タンパク質)は、細胞増殖によってクリアランス(浄化)できるが、非分裂細胞であるニューロンにおいては、それらを処理できないために、タンパク質の品質管理(不要タンパク質の処理)が細胞の生存に不可欠である[48] [49]。Kopitoらは、細胞内に異常タンパク質を強制発現させると、プロテアソームがそれらを処理できずに活性の低下を引き起こし[50]、蓄積した異常タンパク質が凝集しアグレゾーム(様々な神経変性疾患・患者脳の残存ニューロンに同定されている封入体と類似の凝集構造体)を形成することを示した[51]。この結果は、プロテアソームの機能減弱と神経変性の関連性を示唆している。
多くの神経変性疾患に観察される封入体が抗ユビキチン抗体で染色されること[52]から、UPSの破綻が神経変性疾患の発症原因であると考えられた[53]。McNaughtらはプロテアソーム阻害剤を直接マウス小脳に注入してパーキンソン病と類似の症状を引き起こすことを報告し、プロテアソームの抑制とニューロン死の直接的な関係を示唆した[54]が、再現性が乏しく、決定的な結論が得られていなかった[55]。
しかし最近、生後間もないマウスへのプロテアソーム阻害剤の長期間・連続投与によって神経変性が誘導されることが報告された[56]。また、プロテアソームRPを構成するATPaseサブユニットRpt2を脳特異的にノックアウトすると、ユビキチン陽性のLewy体様の封入体が蓄積すると共に神経変性が誘導される[57]。他方、Rpt3を運動ニューロン特異的にノックアウトすると、TAR DNA-binding protein 43 kDa (TDP-43)、fused in sarcoma (FUS)、ユビキチン2、optineurinなどの筋萎縮性側索硬化症(ALS)の神経変性原因遺伝子産物が細胞内分布異常あるいは過剰蓄積して ALS様の症状を呈した[58]。われわれも20Sプロテアソームの分子集合因子PAC1をマウス中枢神経系で欠損させてニューロンのプロテアソームレベルを持続的に低下させると、小脳変性を誘発して神経変性疾患様の症状に陥ることを見出した(図7)。これらの結果により、プロテアソームが神経細胞の恒常性維持に必須であることが遺伝学的に証明された[14]。
家族性パーキンソン病とPINK1・パーキン系
ユビキチン系E3リガーゼをコードしているパーキンが常染色体劣性若年性パーキンソン病の原因遺伝子であることが同定[59][60]され、UPSの破綻と神経変性疾患の関係が注目された。即ち、パーキンの標的分子がドーパミンニューロンに蓄積し、細胞死を誘導すると考えられた。
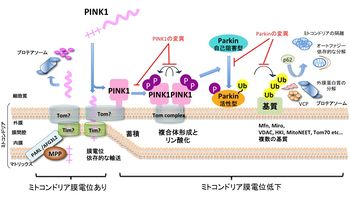
この機構としてYouleらやわれわれは若年性に発症する常染色体劣性遺伝性パーキンソン病の原因遺伝子産物であるセリン/スレオニン型タンパク質リン酸化酵素PINK1と パーキンとの関連に着目した。[61] [62]。
通常ミトコンドリア外膜局在型のPINK1は健常なミトコンドリアにおいては、PARL酵素とプロテアソーム系による恒常的な分解を受けているが、膜電位が低下すると、これらの分解系から免れてミトコンドリア外膜上に蓄積する。蓄積したPINK1は細胞質の不活性型パーキンを損傷ミトコンドリアに移行・活性型に変換させる。その結果、複数のミトコンドリア外膜タンパク質がユビキチン化されプロテアソームにより分解される。これが引き金となって損傷ミトコンドリアはオートファジーにより分解除去される(マイトファジー)[63]。
言い換えると、PINK1・パーキンは膜電位が低下した時のみに発動するように巧妙に制御されているので、損傷ミトコンドリアだけが細胞から除去されることになる。この品質管理が適切に行われずにニューロン内に異常ミトコンドリアが蓄積すると、ドーパミンニューロンの変性を引き起こしパーキンソン病が発症すると想定される(図8)。
このスキームにおける核心は、不良ミトコンドリアのモニタリングであり、その機序としてYouleらは、膜電位依存的なPINK1の(PARLが局在する)ミトコンドリア内膜への輸送仮説を提案しており、その骨子は「膜電位が低下するとPINK1の内膜への輸送が障害されてPINK1が外膜に蓄積する:量的制御」ことである[64]。一方われわれは「膜電位依存的な不活性型PINK1の自己リン酸化による活性化が不良Mtを感知する:質的制御」もう一つのメカニズムであることを突き止めた[65] [66]。そして活性化されたPINK1がパーキンをリン酸化して活性化・不良ミトコンドリアにリクルートすることも判明した[67]。
不良なミトコンドリアの累積は、活性酸素(ROS)を増産させ、DNA・タンパク質・脂質などを修飾して細胞障害を引き起こす。従ってミトコンドリアの品質管理(不良品の処理)は、細胞分裂によって損傷ミトコンドリアを浄化(クリアランス)できないニューロンなどの非分裂細胞にとっては、健康を維持するために必須である。実際、孤発型パーキンソン病におけるミトコンドリアの機能異常(呼吸鎖の低下やミトコンドリアDNAの欠失など)が報告されている[68]。ミトコンドリアの良・不良をモニター(監視)することは、ニューロンが健全に活動するために不可欠である。そしてミコンドリアは分裂と融合を繰り返しながら自律的に増殖できるので、不良ミトコンドリアを適切に処分できれば、正常な量を維持することができる。
癌
細胞周期制御におけるUPSの重要性は、その基質であるサイクリンをHuntらが発見したことを端緒とする(2001年、ノーベル生理学医学賞)。その後、細胞周期に関わる様々な制御因子がリン酸化とUPSに率いられたタンパク質分解で調節されていること、そしてその破綻が[wj:癌|癌]]化の要因となることが明らかになった。癌遺伝子や癌抑制遺伝子の多くが短寿命タンパク質であり、これらがUPSで厳格に調節されている。その代表例は癌抑制遺伝子p53である。これらの多くの例は、ユビキチンシステムによる基質の識別が注目されてきており、プロテアソームは調節というよりは寧ろ分解装置としての役割で貢献してきた。
免疫疾患
免疫プロテアソームのキモトリプシン型活性をコードするβ5i遺伝子(PSMB8)のミスセンス変異が、中条-西村症候群(凍瘡様皮疹と限局性脂肪萎縮を伴う遺伝性周期熱症候群)を引き起こすことが判明した[69] [70]。中条-西村症候群は、1939に発見された遺伝性疾病で、プロテアソームの分子集合異常に起因した機能不全のためにタンパク質の品質管理が破綻し、IL-6が過剰に産生されることによって引き起こされる炎症性疾患である。これはプロテアソームのヒト遺伝性疾患の最初の例であり、国内外で注目されている。
オートファジーとの関連
神経変性疾患に関係して、協同的に作用するオートファジーの役割が注目されている。オートファジーの最も重要な機能は[wj:飢餓適応|飢餓適応]]([wj:低栄養応答|低栄養応答]])である。即ちオートファジーによる自己成分の分解はアミノ酸供給を通じてエネルギーの恒常的な確保に関与しており、飢餓に対するバックアップシステムと考えられる(誘導的オートファジー)。この意味でオートファジーは自律的な栄養素確保を可能にする生存戦略となっている。しかしオートファジーは日常的にも低いレベルでおこっている。この基底レベルのオートファジー(恒常的オートファジー)は、細胞内タンパク質の選択的な浄化機構として重要であると考えられている[71]。
異常タンパク質あるいは変性タンパク質の細胞内蓄積はさまざまな変性疾患や老化過程でも観察されており、プロテアソームは大きな凝集体は処理できないために、これらの病態発症におけるオートファジーの関与が注目されている。実際、多く神経変性疾患においてオートファジーの亢進が報告されている。そして中枢神経系特異的にオートファジーを欠損させたマウスは神経変性疾患の症状を引き起こす[72] [73]。さらにプロテアソームを阻害すると、オートファジーが亢進することから、この二つのUPSとオートファジーには、相互補完的な役割があると考えられる[74]。
阻害剤・活性化剤
天然の阻害剤としてラクタシスチンやエポキソミシンが知られている。
プロテアソーム阻害剤であるPS-341(別名ボルテゾミブ、商品名ベルケイド)[75] [76]が多発性骨髄腫細胞のアポトーシス誘導を示すことが報告され、2003年再発・難治性骨髄腫を対象疾患として臨床応用されている。さらに副作用の少ない多くのプロテアソーム阻害剤の開発が進められており、既存の抗ガン剤との併用を視野に固形ガンを含め臨床治験が進行中である[77]。
またプロテアソームの脱ユビキチン酵素の一つUSP14の阻害剤IU1が、プロテアソームを活性化することが見出された[78]。またプロテアソームに会合している二種の脱ユビキチン酵素(USP14とUch37/UCHL5)を同時に抑制する阻害薬として開発されたb-AP15が、ガン細胞のアポトーシシスを誘導することが見出され、がん治療への貢献が期待されている[79]。さらにユビキチンリガーゼや脱ユビキチン化酵素の異常で様々な病気が発症することが知られており、これらに対する阻害剤の開発が欧米を中心に活発に行われている。従ってこれらをターゲットとした新薬が開発され、健康科学の発展に大きく寄与することが期待されている[80]。
現在、プロテアソームの活性が様々な疾病で大きく変動することが知られているほか[81]、プロテアソームの細胞内動態について再先端技術を用いた解析が精力的に勧められており[82]、今後UPSが創薬の大きなターゲットになることが、期待されている。
関連項目
参考文献
- ↑ 1.0 1.1 1.2 1.3 1.4
Murata, S., Yashiroda, H., & Tanaka, K. (2009).
Molecular mechanisms of proteasome assembly. Nature reviews. Molecular cell biology, 10(2), 104-15. [PubMed:19165213] [WorldCat] [DOI] - ↑
Etlinger, J.D., & Goldberg, A.L. (1977).
A soluble ATP-dependent proteolytic system responsible for the degradation of abnormal proteins in reticulocytes. Proceedings of the National Academy of Sciences of the United States of America, 74(1), 54-8. [PubMed:264694] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1
Hershko, A., & Ciechanover, A. (1992).
The ubiquitin system for protein degradation. Annual review of biochemistry, 61, 761-807. [PubMed:1323239] [WorldCat] [DOI] - ↑ 4.0 4.1
Hershko, A., & Ciechanover, A. (1998).
The ubiquitin system. Annual review of biochemistry, 67, 425-79. [PubMed:9759494] [WorldCat] [DOI] - ↑
Hershko, A., Ciechanover, A., & Varshavsky, A. (2000).
Basic Medical Research Award. The ubiquitin system. Nature medicine, 6(10), 1073-81. [PubMed:11017125] [WorldCat] [DOI] - ↑
Tanaka, K., Waxman, L., & Goldberg, A.L. (1983).
ATP serves two distinct roles in protein degradation in reticulocytes, one requiring and one independent of ubiquitin. The Journal of cell biology, 96(6), 1580-5. [PubMed:6304111] [PMC] [WorldCat] [DOI] - ↑ 7.0 7.1
Sakata, E., Bohn, S., Mihalache, O., Kiss, P., Beck, F., Nagy, I., ..., & Baumeister, W. (2012).
Localization of the proteasomal ubiquitin receptors Rpn10 and Rpn13 by electron cryomicroscopy. Proceedings of the National Academy of Sciences of the United States of America, 109(5), 1479-84. [PubMed:22215586] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1 8.2
Lander, G.C., Estrin, E., Matyskiela, M.E., Bashore, C., Nogales, E., & Martin, A. (2012).
Complete subunit architecture of the proteasome regulatory particle. Nature, 482(7384), 186-91. [PubMed:22237024] [PMC] [WorldCat] [DOI] - ↑
Coux, O., Tanaka, K., & Goldberg, A.L. (1996).
Structure and functions of the 20S and 26S proteasomes. Annual review of biochemistry, 65, 801-47. [PubMed:8811196] [WorldCat] [DOI] - ↑
Baumeister, W., Walz, J., Zühl, F., & Seemüller, E. (1998).
The proteasome: paradigm of a self-compartmentalizing protease. Cell, 92(3), 367-80. [PubMed:9476896] [WorldCat] [DOI] - ↑ 11.0 11.1 11.2 11.3 11.4
Tanaka, K. (2009).
The proteasome: overview of structure and functions. Proceedings of the Japan Academy. Series B, Physical and biological sciences, 85(1), 12-36. [PubMed:19145068] [PMC] [WorldCat] [DOI] - ↑
Worden, E.J., Padovani, C., & Martin, A. (2014).
Structure of the Rpn11-Rpn8 dimer reveals mechanisms of substrate deubiquitination during proteasomal degradation. Nature structural & molecular biology, 21(3), 220-7. [PubMed:24463465] [WorldCat] [DOI] - ↑
Jung, T., & Grune, T. (2008).
The proteasome and its role in the degradation of oxidized proteins. IUBMB life, 60(11), 743-52. [PubMed:18636510] [WorldCat] [DOI] - ↑ 14.0 14.1 14.2
Sasaki, K., Hamazaki, J., Koike, M., Hirano, Y., Komatsu, M., Uchiyama, Y., ..., & Murata, S. (2010).
PAC1 gene knockout reveals an essential role of chaperone-mediated 20S proteasome biogenesis and latent 20S proteasomes in cellular homeostasis. Molecular and cellular biology, 30(15), 3864-74. [PubMed:20498273] [PMC] [WorldCat] [DOI] - ↑
Finley, D. (2009).
Recognition and processing of ubiquitin-protein conjugates by the proteasome. Annual review of biochemistry, 78, 477-513. [PubMed:19489727] [PMC] [WorldCat] [DOI] - ↑
Saeki, Y., & Tanaka, K. (2007).
Unlocking the proteasome door. Molecular cell, 27(6), 865-7. [PubMed:17889660] [WorldCat] [DOI] - ↑
Smith, D.M., Fraga, H., Reis, C., Kafri, G., & Goldberg, A.L. (2011).
ATP binds to proteasomal ATPases in pairs with distinct functional effects, implying an ordered reaction cycle. Cell, 144(4), 526-38. [PubMed:21335235] [PMC] [WorldCat] [DOI] - ↑
Rechsteiner, M., Realini, C., & Ustrell, V. (2000).
The proteasome activator 11 S REG (PA28) and class I antigen presentation. The Biochemical journal, 345 Pt 1, 1-15. [PubMed:10600633] [PMC] [WorldCat] - ↑
Stadtmueller, B.M., & Hill, C.P. (2011).
Proteasome activators. Molecular cell, 41(1), 8-19. [PubMed:21211719] [PMC] [WorldCat] [DOI] - ↑
Tanahashi, N., Murakami, Y., Minami, Y., Shimbara, N., Hendil, K.B., & Tanaka, K. (2000).
Hybrid proteasomes. Induction by interferon-gamma and contribution to ATP-dependent proteolysis. The Journal of biological chemistry, 275(19), 14336-45. [PubMed:10799514] [WorldCat] [DOI] - ↑
Savulescu, A.F., & Glickman, M.H. (2011).
Proteasome activator 200: the heat is on... Molecular & cellular proteomics : MCP, 10(5), R110.006890. [PubMed:21389348] [PMC] [WorldCat] [DOI] - ↑ 22.0 22.1 22.2
Saeki, Y., & Tanaka, K. (2012).
Assembly and function of the proteasome. Methods in molecular biology (Clifton, N.J.), 832, 315-37. [PubMed:22350895] [WorldCat] [DOI] - ↑ 23.0 23.1
Tomko, R.J., & Hochstrasser, M. (2013).
Molecular architecture and assembly of the eukaryotic proteasome. Annual review of biochemistry, 82, 415-45. [PubMed:23495936] [PMC] [WorldCat] [DOI] - ↑
Ramos, P.C., & Dohmen, R.J. (2008).
PACemakers of proteasome core particle assembly. Structure (London, England : 1993), 16(9), 1296-304. [PubMed:18786393] [WorldCat] [DOI] - ↑
Sakata, E., Stengel, F., Fukunaga, K., Zhou, M., Saeki, Y., Förster, F., ..., & Robinson, C.V. (2011).
The catalytic activity of Ubp6 enhances maturation of the proteasomal regulatory particle. Molecular cell, 42(5), 637-49. [PubMed:21658604] [WorldCat] [DOI] - ↑
Weissman, A.M., Shabek, N., & Ciechanover, A. (2011).
The predator becomes the prey: regulating the ubiquitin system by ubiquitylation and degradation. Nature reviews. Molecular cell biology, 12(9), 605-20. [PubMed:21860393] [PMC] [WorldCat] [DOI] - ↑ 27.0 27.1
Klein, L., Hinterberger, M., Wirnsberger, G., & Kyewski, B. (2009).
Antigen presentation in the thymus for positive selection and central tolerance induction. Nature reviews. Immunology, 9(12), 833-44. [PubMed:19935803] [WorldCat] [DOI] - ↑ 28.0 28.1
Tanaka, K., & Kasahara, M. (1998).
The MHC class I ligand-generating system: roles of immunoproteasomes and the interferon-gamma-inducible proteasome activator PA28. Immunological reviews, 163, 161-76. [PubMed:9700509] [WorldCat] [DOI] - ↑
Rock, K.L., York, I.A., Saric, T., & Goldberg, A.L. (2002).
Protein degradation and the generation of MHC class I-presented peptides. Advances in immunology, 80, 1-70. [PubMed:12078479] [WorldCat] - ↑
Sijts, E.J., & Kloetzel, P.M. (2011).
The role of the proteasome in the generation of MHC class I ligands and immune responses. Cellular and molecular life sciences : CMLS, 68(9), 1491-502. [PubMed:21387144] [PMC] [WorldCat] [DOI] - ↑
Sutoh, Y., Kondo, M., Ohta, Y., Ota, T., Tomaru, U., Flajnik, M.F., & Kasahara, M. (2012).
Comparative genomic analysis of the proteasome β5t subunit gene: implications for the origin and evolution of thymoproteasomes. Immunogenetics, 64(1), 49-58. [PubMed:21748441] [PMC] [WorldCat] [DOI] - ↑
Akiyama, K., Yokota, K., Kagawa, S., Shimbara, N., Tamura, T., Akioka, H., ..., & Ichihara, A. (1994).
cDNA cloning and interferon gamma down-regulation of proteasomal subunits X and Y. Science (New York, N.Y.), 265(5176), 1231-4. [PubMed:8066462] [WorldCat] [DOI] - ↑
Hisamatsu, H., Shimbara, N., Saito, Y., Kristensen, P., Hendil, K.B., Fujiwara, T., ..., & Tanaka, K. (1996).
Newly identified pair of proteasomal subunits regulated reciprocally by interferon gamma. The Journal of experimental medicine, 183(4), 1807-16. [PubMed:8666937] [PMC] [WorldCat] [DOI] - ↑
Tanaka, K. (1994).
Role of proteasomes modified by interferon-gamma in antigen processing. Journal of leukocyte biology, 56(5), 571-5. [PubMed:7964165] [WorldCat] [DOI] - ↑
Seifert, U., Bialy, L.P., Ebstein, F., Bech-Otschir, D., Voigt, A., Schröter, F., ..., & Krüger, E. (2010).
Immunoproteasomes preserve protein homeostasis upon interferon-induced oxidative stress. Cell, 142(4), 613-24. [PubMed:20723761] [WorldCat] [DOI] - ↑
Muchamuel, T., Basler, M., Aujay, M.A., Suzuki, E., Kalim, K.W., Lauer, C., ..., & Groettrup, M. (2009).
A selective inhibitor of the immunoproteasome subunit LMP7 blocks cytokine production and attenuates progression of experimental arthritis. Nature medicine, 15(7), 781-7. [PubMed:19525961] [WorldCat] [DOI] - ↑
Groettrup, M., Kirk, C.J., & Basler, M. (2010).
Proteasomes in immune cells: more than peptide producers? Nature reviews. Immunology, 10(1), 73-8. [PubMed:20010787] [WorldCat] [DOI] - ↑
Murata, S., Sasaki, K., Kishimoto, T., Niwa, S., Hayashi, H., Takahama, Y., & Tanaka, K. (2007).
Regulation of CD8+ T cell development by thymus-specific proteasomes. Science (New York, N.Y.), 316(5829), 1349-53. [PubMed:17540904] [WorldCat] [DOI] - ↑ 39.0 39.1
Nitta, T., Murata, S., Sasaki, K., Fujii, H., Ripen, A.M., Ishimaru, N., ..., & Takahama, Y. (2010).
Thymoproteasome shapes immunocompetent repertoire of CD8+ T cells. Immunity, 32(1), 29-40. [PubMed:20045355] [WorldCat] [DOI] - ↑
Cajigas, I.J., Will, T., & Schuman, E.M. (2010).
Protein homeostasis and synaptic plasticity. The EMBO journal, 29(16), 2746-52. [PubMed:20717144] [PMC] [WorldCat] [DOI] - ↑
Hegde, A.N. (2010).
The ubiquitin-proteasome pathway and synaptic plasticity. Learning & memory (Cold Spring Harbor, N.Y.), 17(7), 314-27. [PubMed:20566674] [PMC] [WorldCat] [DOI] - ↑
Fonseca, R., Vabulas, R.M., Hartl, F.U., Bonhoeffer, T., & Nägerl, U.V. (2006).
A balance of protein synthesis and proteasome-dependent degradation determines the maintenance of LTP. Neuron, 52(2), 239-45. [PubMed:17046687] [WorldCat] [DOI] - ↑
Bingol, B., & Sheng, M. (2011).
Deconstruction for reconstruction: the role of proteolysis in neural plasticity and disease. Neuron, 69(1), 22-32. [PubMed:21220096] [WorldCat] [DOI] - ↑
Jarome, T.J., & Helmstetter, F.J. (2013).
The ubiquitin-proteasome system as a critical regulator of synaptic plasticity and long-term memory formation. Neurobiology of learning and memory, 105, 107-16. [PubMed:23623827] [PMC] [WorldCat] [DOI] - ↑
Kaang, B.K., & Choi, J.H. (2012).
Synaptic protein degradation in memory reorganization. Advances in experimental medicine and biology, 970, 221-40. [PubMed:22351058] [WorldCat] [DOI] - ↑
Kourtis, N., & Tavernarakis, N. (2011).
Cellular stress response pathways and ageing: intricate molecular relationships. The EMBO journal, 30(13), 2520-31. [PubMed:21587205] [PMC] [WorldCat] [DOI] - ↑
Tonoki, A., Kuranaga, E., Tomioka, T., Hamazaki, J., Murata, S., Tanaka, K., & Miura, M. (2009).
Genetic evidence linking age-dependent attenuation of the 26S proteasome with the aging process. Molecular and cellular biology, 29(4), 1095-106. [PubMed:19075009] [PMC] [WorldCat] [DOI] - ↑
Rubinsztein, D.C. (2006).
The roles of intracellular protein-degradation pathways in neurodegeneration. Nature, 443(7113), 780-6. [PubMed:17051204] [WorldCat] [DOI] - ↑
Goldberg, A.L. (2003).
Protein degradation and protection against misfolded or damaged proteins. Nature, 426(6968), 895-9. [PubMed:14685250] [WorldCat] [DOI] - ↑
Bence, N.F., Sampat, R.M., & Kopito, R.R. (2001).
Impairment of the ubiquitin-proteasome system by protein aggregation. Science (New York, N.Y.), 292(5521), 1552-5. [PubMed:11375494] [WorldCat] [DOI] - ↑
Kopito, R.R. (2000).
Aggresomes, inclusion bodies and protein aggregation. Trends in cell biology, 10(12), 524-30. [PubMed:11121744] [WorldCat] - ↑
Mori, H., Kondo, J., & Ihara, Y. (1987).
Ubiquitin is a component of paired helical filaments in Alzheimer's disease. Science (New York, N.Y.), 235(4796), 1641-4. [PubMed:3029875] [WorldCat] [DOI] - ↑
Alves-Rodrigues, A., Gregori, L., & Figueiredo-Pereira, M.E. (1998).
Ubiquitin, cellular inclusions and their role in neurodegeneration. Trends in neurosciences, 21(12), 516-20. [PubMed:9881849] [WorldCat] [DOI] - ↑
Mytilineou, C., McNaught, K.S., Shashidharan, P., Yabut, J., Baptiste, R.J., Parnandi, A., & Olanow, C.W. (2004).
Inhibition of proteasome activity sensitizes dopamine neurons to protein alterations and oxidative stress. Journal of neural transmission (Vienna, Austria : 1996), 111(10-11), 1237-51. [PubMed:15480836] [WorldCat] [DOI] - ↑
Matsuda, N., & Tanaka, K. (2010).
Does impairment of the ubiquitin-proteasome system or the autophagy-lysosome pathway predispose individuals to neurodegenerative disorders such as Parkinson's disease? Journal of Alzheimer's disease : JAD, 19(1), 1-9. [PubMed:20061621] [WorldCat] [DOI] - ↑
Romero-Granados, R., Fontán-Lozano, Á., Aguilar-Montilla, F.J., & Carrión, Á.M. (2011).
Postnatal proteasome inhibition induces neurodegeneration and cognitive deficiencies in adult mice: a new model of neurodevelopment syndrome. PloS one, 6(12), e28927. [PubMed:22174927] [PMC] [WorldCat] [DOI] - ↑
Bedford, L., Hay, D., Devoy, A., Paine, S., Powe, D.G., Seth, R., ..., & Mayer, R.J. (2008).
Depletion of 26S proteasomes in mouse brain neurons causes neurodegeneration and Lewy-like inclusions resembling human pale bodies. The Journal of neuroscience : the official journal of the Society for Neuroscience, 28(33), 8189-98. [PubMed:18701681] [PMC] [WorldCat] [DOI] - ↑
Tashiro, Y., Urushitani, M., Inoue, H., Koike, M., Uchiyama, Y., Komatsu, M., ..., & Takahashi, R. (2012).
Motor neuron-specific disruption of proteasomes, but not autophagy, replicates amyotrophic lateral sclerosis. The Journal of biological chemistry, 287(51), 42984-94. [PubMed:23095749] [PMC] [WorldCat] [DOI] - ↑
Kitada, T., Asakawa, S., Hattori, N., Matsumine, H., Yamamura, Y., Minoshima, S., ..., & Shimizu, N. (1998).
Mutations in the parkin gene cause autosomal recessive juvenile parkinsonism. Nature, 392(6676), 605-8. [PubMed:9560156] [WorldCat] [DOI] - ↑
Shimura, H., Hattori, N., Kubo, S.i., Mizuno, Y., Asakawa, S., Minoshima, S., ..., & Suzuki, T. (2000).
Familial Parkinson disease gene product, parkin, is a ubiquitin-protein ligase. Nature genetics, 25(3), 302-5. [PubMed:10888878] [WorldCat] [DOI] - ↑
Narendra, D., Tanaka, A., Suen, D.F., & Youle, R.J. (2008).
Parkin is recruited selectively to impaired mitochondria and promotes their autophagy. The Journal of cell biology, 183(5), 795-803. [PubMed:19029340] [PMC] [WorldCat] [DOI] - ↑
Matsuda, N., Sato, S., Shiba, K., Okatsu, K., Saisho, K., Gautier, C.A., ..., & Tanaka, K. (2010).
PINK1 stabilized by mitochondrial depolarization recruits Parkin to damaged mitochondria and activates latent Parkin for mitophagy. The Journal of cell biology, 189(2), 211-21. [PubMed:20404107] [PMC] [WorldCat] [DOI] - ↑
Narendra, D.P., Jin, S.M., Tanaka, A., Suen, D.F., Gautier, C.A., Shen, J., ..., & Youle, R.J. (2010).
PINK1 is selectively stabilized on impaired mitochondria to activate Parkin. PLoS biology, 8(1), e1000298. [PubMed:20126261] [PMC] [WorldCat] [DOI] - ↑
Youle, R.J., & Narendra, D.P. (2011).
Mechanisms of mitophagy. Nature reviews. Molecular cell biology, 12(1), 9-14. [PubMed:21179058] [PMC] [WorldCat] [DOI] - ↑
Okatsu, K., Oka, T., Iguchi, M., Imamura, K., Kosako, H., Tani, N., ..., & Matsuda, N. (2012).
PINK1 autophosphorylation upon membrane potential dissipation is essential for Parkin recruitment to damaged mitochondria. Nature communications, 3, 1016. [PubMed:22910362] [PMC] [WorldCat] [DOI] - ↑
Okatsu, K., Uno, M., Koyano, F., Go, E., Kimura, M., Oka, T., ..., & Matsuda, N. (2013).
A dimeric PINK1-containing complex on depolarized mitochondria stimulates Parkin recruitment. The Journal of biological chemistry, 288(51), 36372-84. [PubMed:24189060] [PMC] [WorldCat] [DOI] - ↑
Iguchi, M., Kujuro, Y., Okatsu, K., Koyano, F., Kosako, H., Kimura, M., ..., & Matsuda, N. (2013).
Parkin-catalyzed ubiquitin-ester transfer is triggered by PINK1-dependent phosphorylation. The Journal of biological chemistry, 288(30), 22019-32. [PubMed:23754282] [PMC] [WorldCat] [DOI] - ↑
Abou-Sleiman, P.M., Muqit, M.M., & Wood, N.W. (2006).
Expanding insights of mitochondrial dysfunction in Parkinson's disease. Nature reviews. Neuroscience, 7(3), 207-19. [PubMed:16495942] [WorldCat] [DOI] - ↑
Arima, K., Kinoshita, A., Mishima, H., Kanazawa, N., Kaneko, T., Mizushima, T., ..., & Yoshiura, K. (2011).
Proteasome assembly defect due to a proteasome subunit beta type 8 (PSMB8) mutation causes the autoinflammatory disorder, Nakajo-Nishimura syndrome. Proceedings of the National Academy of Sciences of the United States of America, 108(36), 14914-9. [PubMed:21852578] [PMC] [WorldCat] [DOI] - ↑
Kitamura, A., Maekawa, Y., Uehara, H., Izumi, K., Kawachi, I., Nishizawa, M., ..., & Yasutomo, K. (2011).
A mutation in the immunoproteasome subunit PSMB8 causes autoinflammation and lipodystrophy in humans. The Journal of clinical investigation, 121(10), 4150-60. [PubMed:21881205] [PMC] [WorldCat] [DOI] - ↑
Mizushima, N., & Komatsu, M. (2011).
Autophagy: renovation of cells and tissues. Cell, 147(4), 728-41. [PubMed:22078875] [WorldCat] [DOI] - ↑
Komatsu, M., Waguri, S., Chiba, T., Murata, S., Iwata, J., Tanida, I., ..., & Tanaka, K. (2006).
Loss of autophagy in the central nervous system causes neurodegeneration in mice. Nature, 441(7095), 880-4. [PubMed:16625205] [WorldCat] [DOI] - ↑
Hara, T., Nakamura, K., Matsui, M., Yamamoto, A., Nakahara, Y., Suzuki-Migishima, R., ..., & Mizushima, N. (2006).
Suppression of basal autophagy in neural cells causes neurodegenerative disease in mice. Nature, 441(7095), 885-9. [PubMed:16625204] [WorldCat] [DOI] - ↑
Harris, H., & Rubinsztein, D.C. (2011).
Control of autophagy as a therapy for neurodegenerative disease. Nature reviews. Neurology, 8(2), 108-17. [PubMed:22187000] [WorldCat] [DOI] - ↑
Adams, J. (2004).
The proteasome: a suitable antineoplastic target. Nature reviews. Cancer, 4(5), 349-60. [PubMed:15122206] [WorldCat] [DOI] - ↑
Goldberg, A.L. (2012).
Development of proteasome inhibitors as research tools and cancer drugs. The Journal of cell biology, 199(4), 583-8. [PubMed:23148232] [PMC] [WorldCat] [DOI] - ↑
Kortuem, K.M., & Stewart, A.K. (2013).
Carfilzomib. Blood, 121(6), 893-7. [PubMed:23393020] [WorldCat] [DOI] - ↑
Lee, B.H., Lee, M.J., Park, S., Oh, D.C., Elsasser, S., Chen, P.C., ..., & Finley, D. (2010).
Enhancement of proteasome activity by a small-molecule inhibitor of USP14. Nature, 467(7312), 179-84. [PubMed:20829789] [PMC] [WorldCat] [DOI] - ↑
D'Arcy, P., Brnjic, S., Olofsson, M.H., Fryknäs, M., Lindsten, K., De Cesare, M., ..., & Linder, S. (2011).
Inhibition of proteasome deubiquitinating activity as a new cancer therapy. Nature medicine, 17(12), 1636-40. [PubMed:22057347] [WorldCat] [DOI] - ↑
Bedford, L., Lowe, J., Dick, L.R., Mayer, R.J., & Brownell, J.E. (2011).
Ubiquitin-like protein conjugation and the ubiquitin-proteasome system as drug targets. Nature reviews. Drug discovery, 10(1), 29-46. [PubMed:21151032] [WorldCat] [DOI] - ↑
Schmidt, M., & Finley, D. (2014).
Regulation of proteasome activity in health and disease. Biochimica et biophysica acta, 1843(1), 13-25. [PubMed:23994620] [PMC] [WorldCat] [DOI] - ↑ Chan-Gi Pack, Haruka Yukii, Akio Toh-e, Tai Kudo, Hikaru Tsuchiya, Ai Kaiho, Eri Sakata, Shigeo Murata, Hideyoshi Yokosawa, Yasushi Sako, Wolfgang Baumeister, Keiji Tanaka, Yasushi Saeki
Quantitative Live-Cell Imaging Reveals Spatio-Temporal Dynamics and Cytoplasmic Assembly of the 26S Proteasome.
Nat. Commmun. 2014 in press.