「コーニション」の版間の差分
細 →分布 |
Tokiwayamasaki (トーク | 投稿記録) 細 →機能 |
||
| (3人の利用者による、間の6版が非表示) | |||
| 2行目: | 2行目: | ||
<font size="+1">山崎 世和</font><br> | <font size="+1">山崎 世和</font><br> | ||
''イェール大学''<br> | ''イェール大学''<br> | ||
DOI:<selfdoi /> | DOI:<selfdoi /> 原稿受付日:2016年1月22日 原稿完成日:2016年月日<br> | ||
担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | 担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | ||
</div> | </div> | ||
| 192行目: | 192行目: | ||
[[image:コーニション1.png|thumb|300px|'''図1.CNIH2の構造''']] | [[image:コーニション1.png|thumb|300px|'''図1.CNIH2の構造''']] | ||
コーニションとそのホモログは3回膜貫通型タンパク質であり、N末端が細胞質側に突出した形をとっていると考えられている<ref name=ref1 />(TMHMM, | コーニションとそのホモログは3回膜貫通型タンパク質であり、N末端が細胞質側に突出した形をとっていると考えられている<ref name=ref1 />(TMHMM, TMpred)<u>編集部コメント:TMHMM, TMpredとは何か説明するか、コンピューター予測ではなどとしても良いかと思います。</u>。図1にCNIH2の模式図を示す。 CNIH2/3については、細胞外ループがAMPA受容体の機能制御に重要であることが報告されている<ref name=ref6><pubmed>21172611</pubmed></ref> <ref name=ref7><pubmed>25186755</pubmed></ref>。 | ||
==ファミリー== | ==ファミリー== | ||
[[image:コーニション2.png|thumb|300px|'''図2.コーニションホモログの系統樹''']] | [[image:コーニション2.png|thumb|300px|'''図2.コーニションホモログの系統樹''']] | ||
哺乳類においてはCNIH1-4の4つの分子が報告されている。ほかに、線虫ではコーニション-1(CNI- | 哺乳類においてはCNIH1-4の4つの分子が報告されている。ほかに、線虫ではコーニション-1(CNI-1)が1つ、ショウジョウバエではコーニション(CNI)とコーニション関連(CNIR)の2つが同定されており<ref name=ref1 />、[[酵母]]においてこれらと相同性の高い分子として[[Erv14p]]が知られている<ref name=ref8><pubmed>9732282</pubmed></ref>(図2)<ref name=ref1 />。 | ||
==分布== | ==分布== | ||
酵母、ショウジョウバエ、線虫、哺乳類において、蛍光タグなどとの融合タンパク質の発現によって、[[小胞体]]と[[ゴルジ体]] への局在が示されている<ref name=ref1 /> <ref name=ref4 /> <ref name=ref8 />。また線虫や哺乳類においてCNI-1、CNIH2/3は、細胞表面・[[シナプス]]へも局在することが示唆されており<ref name=ref4 /> <ref name=ref6><pubmed>21172611</pubmed></ref>、実際にCNIH2/3遺伝子欠損マウスがAMPA型グルタミン酸受容体のチャネル特性を変化させることから<ref name=ref3 />、CNIH2/3とAMPA型グルタミン酸受容体は細胞表面やシナプスにおいて共局在すると考えられている。ただし、内在性CNIH2/3が、脳内において小胞体・ゴルジ体・細胞表面・シナプスのどの部位に局在するのかは示されていない。 | 酵母、ショウジョウバエ、線虫、哺乳類において、蛍光タグなどとの融合タンパク質の発現によって、[[小胞体]]と[[ゴルジ体]] への局在が示されている<ref name=ref1 /> <ref name=ref4 /> <ref name=ref8 />。また線虫や哺乳類においてCNI-1、CNIH2/3は、細胞表面・[[シナプス]]へも局在することが示唆されており<ref name=ref4 /> <ref name=ref6><pubmed>21172611</pubmed></ref>、実際にCNIH2/3遺伝子欠損マウスがAMPA型グルタミン酸受容体のチャネル特性を変化させることから<ref name=ref3 />、CNIH2/3とAMPA型グルタミン酸受容体は細胞表面やシナプスにおいて共局在すると考えられている。ただし、内在性CNIH2/3が、脳内において小胞体・ゴルジ体・細胞表面・シナプスのどの部位に局在するのかは示されていない。 | ||
組織レベルでは、 哺乳類ではCNIH2/3 | 組織レベルでは、 哺乳類ではCNIH2/3 mRNAが脳において、CNIH1 mRNAが末梢の様々な組織において<ref name=ref9><pubmed>10209299</pubmed></ref>発現することが、それぞれ[[ノザンブロット]]、[[in situハイブリダイザーション]]によって示されている(Allen Brain Atlas)。CNIH2については脳、特に[[海馬]]において強く発現していることがタンパク質レベルで示されており<ref name=ref6><pubmed>21172611</pubmed></ref>、これはin situハイブリダイザーションの結果と一致する 。また、内在性プロモーターを用いた[[トランスジェニック動物]]によって、[[wj:生殖細胞|生殖細胞]]系・[[wj:胚濾胞上皮細胞|胚濾胞上皮細胞]](<u>編集部コメント:これはなんでしょうか?</u>)・成体体細胞(ショウジョウバエ)<ref name=ref1 />、[[GLR-1]]陽性細胞を含む神経系の細胞(線虫)<ref name=ref4 />で発現が確認されている。 | ||
==機能== | ==機能== | ||
| 218行目: | 218行目: | ||
===AMPA受容体とCNIH2/3の結合についての議論=== | ===AMPA受容体とCNIH2/3の結合についての議論=== | ||
図3に示すように、海馬においてAMPA受容体機能複合体はAMPA受容体であるGluAサブユニット4量体、TARP、CNIHによって形成されている。AMPA受容体は、海馬においてGluA1/2のヘテロマー、GluA2/3のヘテロマーが主に存在する。Herringらのグループは野生型マウスの海馬でGluA2と免疫共沈降されるCNIH2/3がGluA1欠損マウスでは共沈降されなくなることから、CNIH2/3はGluA1と選択的に結合していると議論している<ref name=ref3 />。一方で、CNIH2/3の発現にはAMPA受容体との結合が必須であるにもかかわらずGluA1欠損マウスの海馬においてもCNIH2/3のタンパク発現が消失しないことから、GluA1以外のサブユニットもCNIH2/3に結合していると考えられている。実際、GluA2欠損マウスにおいてCNIH2/ | 図3に示すように、海馬においてAMPA受容体機能複合体はAMPA受容体であるGluAサブユニット4量体、TARP、CNIHによって形成されている。AMPA受容体は、海馬においてGluA1/2のヘテロマー、GluA2/3のヘテロマーが主に存在する。Herringらのグループは野生型マウスの海馬でGluA2と免疫共沈降されるCNIH2/3がGluA1欠損マウスでは共沈降されなくなることから、CNIH2/3はGluA1と選択的に結合していると議論している<ref name=ref3 />。一方で、CNIH2/3の発現にはAMPA受容体との結合が必須であるにもかかわらずGluA1欠損マウスの海馬においてもCNIH2/3のタンパク発現が消失しないことから、GluA1以外のサブユニットもCNIH2/3に結合していると考えられている。実際、GluA2欠損マウスにおいてCNIH2/3のタンパク発現が減少するなど、GluA1/2と結合していないCNIH2/3の存在を支持する報告がなされている<ref name=ref11><pubmed>24853943</pubmed></ref>。 | ||
==関連項目== | ==関連項目== | ||
2016年2月7日 (日) 15:52時点における版
山崎 世和
イェール大学
DOI:10.14931/bsd.6742 原稿受付日:2016年1月22日 原稿完成日:2016年月日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
同義語:コーニションホモログ
英語名: Cornichon, Cornichon homolog
英略称: CNI, CNIH
コーニションは酵母から哺乳類まで保存されているタンパク質ファミリーである[1]。その一部は、イオンチャネル型のグルタミン酸受容体と特異的に結合し、補助サブユニットとしてチャネル活性や細胞膜発現を制御すると考えられている。哺乳類CNIH2/3はAMPA型グルタミン酸受容体と結合しチャネル活性を制御することが[2] [3]、線虫ホモログのCNI-1は グルタミン酸受容体GLR-1の局在を調整することが報告されている[4]。
| Cornichon family AMPA receptor auxiliary protein 1 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||
| Symbols | CNIH1; CNIH; CNIH-1; CNIL; TGAM77 | ||||||||||
| External IDs | OMIM: 611287 MGI: 1277202 HomoloGene: 4219 GeneCards: CNIH1 Gene | ||||||||||
| |||||||||||
| RNA expression pattern | |||||||||||
 | |||||||||||
| More reference expression data | |||||||||||
| Orthologs | |||||||||||
| Species | Human | Mouse | |||||||||
| Entrez | 10175 | 12793 | |||||||||
| Ensembl | ENSG00000100528 | ENSMUSG00000015759 | |||||||||
| UniProt | O95406 | O35372 | |||||||||
| RefSeq (mRNA) | NM_001009551 | NM_009919 | |||||||||
| RefSeq (protein) | NP_005767 | NP_034049 | |||||||||
| Location (UCSC) |
Chr 14: 54.42 – 54.44 Mb |
Chr 14: 46.78 – 46.79 Mb | |||||||||
| PubMed search | [1] | [2] | |||||||||
| Cornichon family AMPA receptor auxiliary protein 3 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | CNIH3; CNIH-3 | ||||||||||||
| External IDs | OMIM: None GeneCards: CNIH3 Gene | ||||||||||||
| |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 149111 | n/a | |||||||||||
| Ensembl | ENSG00000143786 | n/a | |||||||||||
| UniProt | Q8TBE1 | n/a | |||||||||||
| RefSeq (mRNA) | NM_152495 | n/a | |||||||||||
| RefSeq (protein) | NP_689708 | n/a | |||||||||||
| Location (UCSC) |
Chr 1: 224.43 – 224.74 Mb | n/a | |||||||||||
| PubMed search | [4] | n/a | |||||||||||
| Cornichon family AMPA receptor auxiliary protein 4 | |||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Identifiers | |||||||||||||
| Symbols | CNIH4; CNIH-4; HSPC163 | ||||||||||||
| External IDs | MGI: 1925828 HomoloGene: 5932 GeneCards: CNIH4 Gene | ||||||||||||
| |||||||||||||
| RNA expression pattern | |||||||||||||
 | |||||||||||||
| More reference expression data | |||||||||||||
| Orthologs | |||||||||||||
| Species | Human | Mouse | |||||||||||
| Entrez | 29097 | 98417 | |||||||||||
| Ensembl | ENSG00000143771 | ENSMUSG00000062169 | |||||||||||
| UniProt | Q9P003 | Q9CX13 | |||||||||||
| RefSeq (mRNA) | NM_001277197 | NM_030131 | |||||||||||
| RefSeq (protein) | NP_001264126 | NP_084407 | |||||||||||
| Location (UCSC) |
Chr 1: 224.36 – 224.38 Mb |
Chr 1: 181.14 – 181.17 Mb | |||||||||||
| PubMed search | [5] | [6] | |||||||||||
イントロダクション
コーニションのショウジョウバエ変異体は、gurken (grk)(GRK、TGF様成長因子)変異体と類似の卵軸形成異常を示す変異体として見出された[5]。これらの変異体の卵は、背側付属器(dorsal appendage)を欠損し長く伸びた形態を示す(gurkenはキュウリ(独)、cornichonはキュウリの漬物(仏)の意)。CNIはショウジョウバエにおいて小胞体からのGRKの搬出・分泌を制御する[1]。
一方で近年、哺乳類のCornichon homolog isoform 2/3(CNIH2/3)がイオンチャネル共役型のAMPA型グルタミン酸受容体(AMPA受容体)の結合因子として同定された[2]。
構造
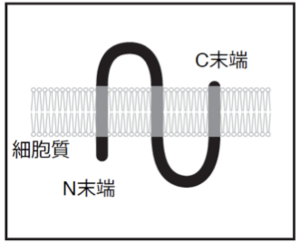
コーニションとそのホモログは3回膜貫通型タンパク質であり、N末端が細胞質側に突出した形をとっていると考えられている[1](TMHMM, TMpred)編集部コメント:TMHMM, TMpredとは何か説明するか、コンピューター予測ではなどとしても良いかと思います。。図1にCNIH2の模式図を示す。 CNIH2/3については、細胞外ループがAMPA受容体の機能制御に重要であることが報告されている[6] [7]。
ファミリー
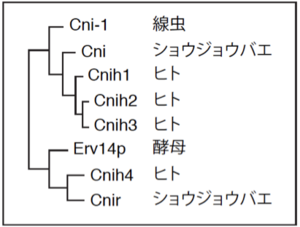
哺乳類においてはCNIH1-4の4つの分子が報告されている。ほかに、線虫ではコーニション-1(CNI-1)が1つ、ショウジョウバエではコーニション(CNI)とコーニション関連(CNIR)の2つが同定されており[1]、酵母においてこれらと相同性の高い分子としてErv14pが知られている[8](図2)[1]。
分布
酵母、ショウジョウバエ、線虫、哺乳類において、蛍光タグなどとの融合タンパク質の発現によって、小胞体とゴルジ体 への局在が示されている[1] [4] [8]。また線虫や哺乳類においてCNI-1、CNIH2/3は、細胞表面・シナプスへも局在することが示唆されており[4] [6]、実際にCNIH2/3遺伝子欠損マウスがAMPA型グルタミン酸受容体のチャネル特性を変化させることから[3]、CNIH2/3とAMPA型グルタミン酸受容体は細胞表面やシナプスにおいて共局在すると考えられている。ただし、内在性CNIH2/3が、脳内において小胞体・ゴルジ体・細胞表面・シナプスのどの部位に局在するのかは示されていない。
組織レベルでは、 哺乳類ではCNIH2/3 mRNAが脳において、CNIH1 mRNAが末梢の様々な組織において[9]発現することが、それぞれノザンブロット、in situハイブリダイザーションによって示されている(Allen Brain Atlas)。CNIH2については脳、特に海馬において強く発現していることがタンパク質レベルで示されており[6]、これはin situハイブリダイザーションの結果と一致する 。また、内在性プロモーターを用いたトランスジェニック動物によって、生殖細胞系・胚濾胞上皮細胞(編集部コメント:これはなんでしょうか?)・成体体細胞(ショウジョウバエ)[1]、GLR-1陽性細胞を含む神経系の細胞(線虫)[4]で発現が確認されている。
機能
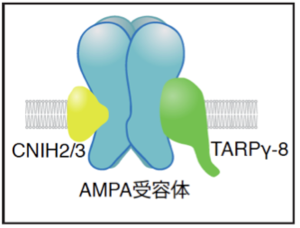
哺乳類においてCNIH2/3がAMPA型グルタミン酸受容体と直接結合し、補助サブユニットとして機能することが報告されている[3] [4]。CNIH2/3は海馬に強く発現しており、別の補助サブユニット(transmembrane AMPA receptor regulatory protein : TARP)の海馬アイソフォームであるTARPγ-8とともに図3のような3者AMPA型グルタミン酸受容体機能複合体を形成することで、チャネル特性や細胞表面発現などの生理機能を制御していると考えられている[3] [4]。
一方、ショウジョウバエのcni変異体の卵は軸形成異常を示し、背足付属器が欠損し長く伸びた形態となる[5]。この表現型はTGF様成長因子grkとその受容体torpedoの変異体においても観察される[10]。cni変異体ではGRKの局在・分泌に異常が見られることからCNIはGRKの小胞体からの搬出を制御していると考えられている[1]。
CNIH2/3欠損マウス
CNIH2/3の条件付き遺伝子欠損マウスが作出、解析されている[3]。Cre組換え酵素発現AAVの導入、またはNEX-CreによってCNIH2を欠損した神経細胞は、AMPA型グルタミン酸受容体依存的シナプス後電流の振幅減少およびシナプス減衰速度短縮の表現型を示すことから、CNIH2はAMPA型グルタミン酸受容体のシナプスにおけるチャネル活性を制御することが明らかとなった。またCNIH2とCNIH3の同時欠損によってこれらの表現型が増強することから、CNIH2とCNIH3は相補的に機能していると考えられる。
線虫cni-1変異体
線虫cni-1変異体は、個体の後方移動の頻度が増加する表現型を示し、これはグルタミン酸受容体を過剰刺激した時の表現型と一致する[4]。実際、cni-1変異体ではシナプスに局在するGLR-1の量が増加し、グルタミン酸に対する神経細胞の応答が増強される[4]。また線虫神経細胞への過剰発現やアフリカツメガエル卵母細胞を用いた実験で、CNI-1がGLR-1のシナプス発現量、細胞表面発現量を減少させることが明らかとなった[4]。これらの結果から線虫においてはCNI-1がグルタミン酸受容体の細胞表面への発現を制御することで、神経細胞のグルタミン酸受容体依存的な興奮活性を調節していると考えられている。
AMPA受容体とCNIH2/3の結合についての議論
図3に示すように、海馬においてAMPA受容体機能複合体はAMPA受容体であるGluAサブユニット4量体、TARP、CNIHによって形成されている。AMPA受容体は、海馬においてGluA1/2のヘテロマー、GluA2/3のヘテロマーが主に存在する。Herringらのグループは野生型マウスの海馬でGluA2と免疫共沈降されるCNIH2/3がGluA1欠損マウスでは共沈降されなくなることから、CNIH2/3はGluA1と選択的に結合していると議論している[3]。一方で、CNIH2/3の発現にはAMPA受容体との結合が必須であるにもかかわらずGluA1欠損マウスの海馬においてもCNIH2/3のタンパク発現が消失しないことから、GluA1以外のサブユニットもCNIH2/3に結合していると考えられている。実際、GluA2欠損マウスにおいてCNIH2/3のタンパク発現が減少するなど、GluA1/2と結合していないCNIH2/3の存在を支持する報告がなされている[11]。
関連項目
参考文献
- ↑ 1.0 1.1 1.2 1.3 1.4 1.5 1.6 1.7
Bökel, C., Dass, S., Wilsch-Bräuninger, M., & Roth, S. (2006).
Drosophila Cornichon acts as cargo receptor for ER export of the TGFalpha-like growth factor Gurken. Development (Cambridge, England), 133(3), 459-70. [PubMed:16396907] [WorldCat] [DOI] - ↑ 2.0 2.1
Schwenk, J., Harmel, N., Zolles, G., Bildl, W., Kulik, A., Heimrich, B., ..., & Klöcker, N. (2009).
Functional proteomics identify cornichon proteins as auxiliary subunits of AMPA receptors. Science (New York, N.Y.), 323(5919), 1313-9. [PubMed:19265014] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2 3.3 3.4 3.5
Herring, B.E., Shi, Y., Suh, Y.H., Zheng, C.Y., Blankenship, S.M., Roche, K.W., & Nicoll, R.A. (2013).
Cornichon proteins determine the subunit composition of synaptic AMPA receptors. Neuron, 77(6), 1083-96. [PubMed:23522044] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1 4.2 4.3 4.4 4.5 4.6 4.7 4.8
Brockie, P.J., Jensen, M., Mellem, J.E., Jensen, E., Yamasaki, T., Wang, R., ..., & Maricq, A.V. (2013).
Cornichons control ER export of AMPA receptors to regulate synaptic excitability. Neuron, 80(1), 129-42. [PubMed:24094107] [PMC] [WorldCat] [DOI] - ↑ 5.0 5.1
Ashburner, M., Thompson, P., Roote, J., Lasko, P.F., Grau, Y., el Messal, M., ..., & Simpson, P. (1990).
The genetics of a small autosomal region of Drosophila melanogaster containing the structural gene for alcohol dehydrogenase. VII. Characterization of the region around the snail and cactus loci. Genetics, 126(3), 679-94. [PubMed:2123463] [PMC] [WorldCat] - ↑ 6.0 6.1 6.2
Kato, A.S., Gill, M.B., Ho, M.T., Yu, H., Tu, Y., Siuda, E.R., ..., & Bredt, D.S. (2010).
Hippocampal AMPA receptor gating controlled by both TARP and cornichon proteins. Neuron, 68(6), 1082-96. [PubMed:21172611] [PMC] [WorldCat] [DOI] - ↑
Shanks, N.F., Cais, O., Maruo, T., Savas, J.N., Zaika, E.I., Azumaya, C.M., ..., & Nakagawa, T. (2014).
Molecular dissection of the interaction between the AMPA receptor and cornichon homolog-3. The Journal of neuroscience : the official journal of the Society for Neuroscience, 34(36), 12104-20. [PubMed:25186755] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1
Powers, J., & Barlowe, C. (1998).
Transport of axl2p depends on erv14p, an ER-vesicle protein related to the Drosophila cornichon gene product. The Journal of cell biology, 142(5), 1209-22. [PubMed:9732282] [PMC] [WorldCat] [DOI] - ↑
Utku, N., Bulwin, G.C., Beinke, S., Heinemann, T., Beato, F., Randall, J., ..., & Gullans, S.R. (1999).
The human homolog of Drosophila cornichon protein is differentially expressed in alloactivated T-cells. Biochimica et biophysica acta, 1449(3), 203-10. [PubMed:10209299] [WorldCat] [DOI] - ↑
Schüpbach, T. (1987).
Germ line and soma cooperate during oogenesis to establish the dorsoventral pattern of egg shell and embryo in Drosophila melanogaster. Cell, 49(5), 699-707. [PubMed:3107840] [WorldCat] [DOI] - ↑
Boudkkazi, S., Brechet, A., Schwenk, J., & Fakler, B. (2014).
Cornichon2 dictates the time course of excitatory transmission at individual hippocampal synapses. Neuron, 82(4), 848-58. [PubMed:24853943] [WorldCat] [DOI]