「時計遺伝子」の版間の差分
細編集の要約なし |
|||
| (同じ利用者による、間の4版が非表示) | |||
| 4行目: | 4行目: | ||
''1. 名古屋大学 トランスフォーマティブ生命分子研究所''<br> | ''1. 名古屋大学 トランスフォーマティブ生命分子研究所''<br> | ||
''2. 名古屋大学 環境医学研究所''<br> | ''2. 名古屋大学 環境医学研究所''<br> | ||
DOI:<selfdoi /> | DOI:<selfdoi /> 原稿受付日:2023年4月25日 原稿完成日:2023年5月1日<br> | ||
担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | 担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | ||
</div></div> | </div></div> | ||
| 13行目: | 13行目: | ||
==時計遺伝子とは== | ==時計遺伝子とは== | ||
時計遺伝子は[[概日リズム]]生成の分子メカニズム関わる一群の遺伝子を指す。概日リズムは約24時間周期の生物リズムであり、以下の3つの特徴的な性質を示す<ref name=田澤2009>'''田澤 仁 (2009).'''<br>マメから生まれた生物時計―エルヴィン・ビュニングの物語、学会出版センター</ref> | 時計遺伝子は[[概日リズム]]生成の分子メカニズム関わる一群の遺伝子を指す。概日リズムは約24時間周期の生物リズムであり、以下の3つの特徴的な性質を示す<ref name=田澤2009>'''田澤 仁 (2009).'''<br>マメから生まれた生物時計―エルヴィン・ビュニングの物語、学会出版センター</ref>。 | ||
# 明暗などの環境変化の無い恒常条件下でも約24時間周期のリズムを示す (自律振動性) | # 明暗などの環境変化の無い恒常条件下でも約24時間周期のリズムを示す (自律振動性) | ||
| 19行目: | 19行目: | ||
# リズムの周期が温度の影響をほとんど受けない (温度補償性) | # リズムの周期が温度の影響をほとんど受けない (温度補償性) | ||
概日リズムの周期長などの性質は遺伝することから、概日リズムを生み出す仕組みは遺伝子によって規定されていると考えられた<ref name=田澤2009 /> (1)。1971年に[[ショウジョウバエ]]の概日リズム変異体[[period]] ([[per]])が報告され、その後、[[アカパンカビ]]の[[frequency]] ([[frq]]) 変異体、[[ハムスター]]の[[tau]]変異体、[[マウス]]の[[Clock]]変異体、[[シアノバクテリア]]の[[kai]]変異体群が報告された<ref name=岡村2004>'''岡村 均, 深田 吉孝 (2004).'''<br>時計遺伝子の分子生物学、シュプリンガーフェアラーク東京 </ref> | 概日リズムの周期長などの性質は遺伝することから、概日リズムを生み出す仕組みは遺伝子によって規定されていると考えられた<ref name=田澤2009 /> (1)。1971年に[[ショウジョウバエ]]の概日リズム変異体[[period]] ([[per]])が報告され、その後、[[アカパンカビ]]の[[frequency]] ([[frq]]) 変異体、[[ハムスター]]の[[tau]]変異体、[[マウス]]の[[Clock]]変異体、[[シアノバクテリア]]の[[kai]]変異体群が報告された<ref name=岡村2004>'''岡村 均, 深田 吉孝 (2004).'''<br>時計遺伝子の分子生物学、シュプリンガーフェアラーク東京 </ref>。そして、1984年にショウジョウバエper遺伝子がクローニングされ、続いて概日リズム変異体の原因遺伝子が続々と明らかになった<ref name=岡村2004/><ref name=海老原2012>'''海老原 史樹文, 吉村 崇 (2012).'''<br>時間生物学 化学同人.</ref>。そのため、時計遺伝子という用語は、[[順遺伝学]]的な手法から概日リズム変異体の原因遺伝子が同定された際に、その原因遺伝子を指すために元々は使用された。 | ||
その後、分子生物学的に概日リズムの生成に関与する遺伝子が数多く同定されたため、これら遺伝子を広義の時計遺伝子として扱うことがある<ref name=日本時間生物学会> [https://chronobiology.jp/TechnicalTerms.html 日本時間生物学会 時間生物学用語集]</ref> | その後、分子生物学的に概日リズムの生成に関与する遺伝子が数多く同定されたため、これら遺伝子を広義の時計遺伝子として扱うことがある<ref name=日本時間生物学会> [https://chronobiology.jp/TechnicalTerms.html 日本時間生物学会 時間生物学用語集]</ref>。また、概日性の周期で発現リズムを示す遺伝子をccg (clock-controlled gene)と呼ぶこともある。 | ||
==概日振動体の構成要素== | ==概日振動体の構成要素== | ||
時計遺伝子は、狭義では概日リズムを生成する仕組みのうち、[[振動体]]を構成する要素を指すという考え方がある<ref name=日本時間生物学会 /><ref name=Aronson1994><pubmed>8128244</pubmed></ref> | 時計遺伝子は、狭義では概日リズムを生成する仕組みのうち、[[振動体]]を構成する要素を指すという考え方がある<ref name=日本時間生物学会 /><ref name=Aronson1994><pubmed>8128244</pubmed></ref>。概日リズムは概日振動体によって生み出され、出力系を介して生理的な表出リズム(overt rhythm)として観察される<ref name= Dunlap2003> Jay C. Dunlap, Jennifer J. Loros, Patricia J. Decoursey. (2003)<br>Chronobiology: Biological Timekeeping, Sinauer Associates Inc.</ref><ref name=Eskin1979><pubmed>499572</pubmed></ref>。そのため、表出リズムを消失させる要因としては、振動体の構成要素が障害された場合と、出力系が障害された場合が考えられる。ある因子が振動体を構成する要素であるか否かは、以下のような検証によって確認することができる<ref name=Aronson1994><pubmed>8128244</pubmed></ref>。 | ||
# その因子の欠損によって振動体は大きく影響を受け、表出リズムは消失する。 | # その因子の欠損によって振動体は大きく影響を受け、表出リズムは消失する。 | ||
# その因子は約24時間の周期で自律振動する。 | # その因子は約24時間の周期で自律振動する。 | ||
| 31行目: | 31行目: | ||
# その因子の増減レベルを一定にすると、表出リズムは消失する。 | # その因子の増減レベルを一定にすると、表出リズムは消失する。 | ||
[[ファイル:Kon clock genes Fig.png|サムネイル|'''図. 転写翻訳を介したフィードバックループ'''<br>[[bHLH-PAS型転写因子]]CLOCK(あるいは[[Neuronal PAS Domain Protein 2]] ([[NPAS2]]))は[[Basic Helix-Loop-Helix ARNT Like 1]] ([[BMAL1]])とヘテロ二量体を形成し、[[E-box]]と呼ばれる[[DNAシスエレメント]]を介してPerや[[Cry]]遺伝子の転写を活性化する。翻訳されたPERやCRYタンパク質は複合体を形成し、細胞質から核に移行してCLOCK- | [[ファイル:Kon clock genes Fig.png|サムネイル|'''図. 転写翻訳を介したフィードバックループ'''<br>[[bHLH-PAS型転写因子]]CLOCK(あるいは[[Neuronal PAS Domain Protein 2]] ([[NPAS2]]))は[[Basic Helix-Loop-Helix ARNT Like 1]] ([[BMAL1]])とヘテロ二量体を形成し、[[E-box]]と呼ばれる[[DNAシスエレメント]]を介してPerや[[Cry]]遺伝子の転写を活性化する。翻訳されたPERやCRYタンパク質は複合体を形成し、細胞質から核に移行してCLOCK-BMAL1の転写活性化を抑制する。その結果、PerやCry遺伝子の発現レベルは概日リズムを示す。文献<ref>'''金 尚宏, 深田 吉孝 (2019).'''<br>生物時計と体のリズム. 学術の動向 24, 8-19</ref>を元に改変)。]] | ||
==転写翻訳フィードバックループ== | ==転写翻訳フィードバックループ== | ||
===コアループ=== | ===コアループ=== | ||
[[哺乳類]]において、大部分の遺伝子発現リズムおよび生理リズムは、主に4種類の因子によって形成される転写翻訳フィードバックループ (transcription-translation feedback loop, TTFL)によって生み出されている<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 /> | [[哺乳類]]において、大部分の遺伝子発現リズムおよび生理リズムは、主に4種類の因子によって形成される転写翻訳フィードバックループ (transcription-translation feedback loop, TTFL)によって生み出されている<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 />。4種類の因子とは、 | ||
# PERIOD (PER)タンパク質、 | # PERIOD (PER)タンパク質、 | ||
# CRYPTOCHROME (CRY)タンパク質、 | # CRYPTOCHROME (CRY)タンパク質、 | ||
| 41行目: | 41行目: | ||
である。 | である。 | ||
これらの遺伝子産物は遺伝的冗長性があるものが多く、PERはPer1, Per2およびPer3遺伝子、CRYはCry1およびCry2遺伝子、class I bHLH-PAS型転写因子はClockおよびNpas2遺伝子、class II bHLH-PAS型転写因子はBmal1 およびBmal2遺伝子にコードされている<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 /> | これらの遺伝子産物は遺伝的冗長性があるものが多く、PERはPer1, Per2およびPer3遺伝子、CRYはCry1およびCry2遺伝子、class I bHLH-PAS型転写因子はClockおよびNpas2遺伝子、class II bHLH-PAS型転写因子はBmal1 およびBmal2遺伝子にコードされている<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 />。CLOCK(あるいはNPAS2)はBMAL1とヘテロ二量体を形成し、E-boxと呼ばれるDNAシスエレメントを介して[[Per1]]/[[Per2|2]]や[[Cry1]]/[[Cry2|2]]遺伝子の転写を活性化する。翻訳されたPER1/2やCRY1/2タンパク質は複合体を形成し、[[細胞質]]から[[核]]に移行してCLOCK-BMAL1の転写活性化を抑制する。その結果、Per1/2やCry1/2遺伝子の発現レベルは概日リズムを示す ('''図''')。これらの4因子による転写翻訳フィードバック機構をコアループと呼ぶ。 | ||
Per1/2二重欠損マウス、Cry1/2二重欠損マウス、Clock/Npas2二重欠損マウス、あるいはBmal1欠損マウスは、行動リズムの消失や遺伝子発現リズムの消失が引き起こされる<ref name=Bunger2000><pubmed>11163178</pubmed></ref><ref name=Zheng2001><pubmed>11389837</pubmed></ref><ref name=DeBruyne2007><pubmed>17417633</pubmed></ref><ref name=vanderHorst1999><pubmed>10217146</pubmed></ref> | Per1/2二重欠損マウス、Cry1/2二重欠損マウス、Clock/Npas2二重欠損マウス、あるいはBmal1欠損マウスは、行動リズムの消失や遺伝子発現リズムの消失が引き起こされる<ref name=Bunger2000><pubmed>11163178</pubmed></ref><ref name=Zheng2001><pubmed>11389837</pubmed></ref><ref name=DeBruyne2007><pubmed>17417633</pubmed></ref><ref name=vanderHorst1999><pubmed>10217146</pubmed></ref>。そのため、これら因子は概日性の生理リズムを生成する必須因子である。転写を介した[[フィードバックループ]]は[[動物]]、[[菌類]]、[[植物]]において遺伝子発現リズムを生成するメカニズムとして共通している。一方、これらの生物系統の間で、概日リズムの生成に関わる転写関連因子の配列相同性は限定的であるため、転写を介したフィードバックループは各生物界で独立に進化したと考えられている<ref name=岡村2004/>。 | ||
===サブループ=== | ===サブループ=== | ||
コアループに共役する形でいくつかのサブループが報告されている。 | コアループに共役する形でいくつかのサブループが報告されている。 | ||
# ROR-response element (RORE)配列を介したBmal1遺伝子の制御<br>[[視交叉上核]]において主観的夜 (circadian time 16; CT16)に発現ピークを示すBmal1などの遺伝子の上流にはRORE配列が存在する<ref name=Ueda2002><pubmed>12152080</pubmed></ref><ref name=Ueda2005><pubmed>15665827</pubmed></ref> | # ROR-response element (RORE)配列を介したBmal1遺伝子の制御<br>[[視交叉上核]]において主観的夜 (circadian time 16; CT16)に発現ピークを示すBmal1などの遺伝子の上流にはRORE配列が存在する<ref name=Ueda2002><pubmed>12152080</pubmed></ref><ref name=Ueda2005><pubmed>15665827</pubmed></ref>。RORE配列には転写抑制因子としてREV-ERBαおよび[[REV-ERBβ]]、転写活性化因子として[[RAR Related Orphan Receptorα|RAR Related Orphan Receptor (ROR)α]]、[[RORβ]]、[[RORγ]]が作用することが報告されている。 | ||
# D-boxを介したPer1/2遺伝子の制御<br>Per1/2遺伝子の上流には[[D-box]]が存在する。D-boxには[[転写抑制因子]]として[[E4 promoter Binding Protein 4]] ([[E4BP4]])、[[転写活性化因子]]として[[D-Box Binding PAR BZIP Transcription Factor]] ([[DBP]]), [[Hepatic leukemia factor]] ([[HLF]]), [[Thyrotroph embryonic factor]] ([[TEF]])が結合することが報告されている<ref name=Ueda2005 /><ref name=Mitsui2001><pubmed>11316793</pubmed></ref> | # D-boxを介したPer1/2遺伝子の制御<br>Per1/2遺伝子の上流には[[D-box]]が存在する。D-boxには[[転写抑制因子]]として[[E4 promoter Binding Protein 4]] ([[E4BP4]])、[[転写活性化因子]]として[[D-Box Binding PAR BZIP Transcription Factor]] ([[DBP]]), [[Hepatic leukemia factor]] ([[HLF]]), [[Thyrotroph embryonic factor]] ([[TEF]])が結合することが報告されている<ref name=Ueda2005 /><ref name=Mitsui2001><pubmed>11316793</pubmed></ref>。 | ||
# 転写因子[[Deleted in esophageal cancer]] (DEC)1/2によるE-box配列の制御<br>上記のCLOCK-BMAL1による制御に加え、E-boxには転写抑制因子として[[DEC1]]/[[DEC2|2]]が結合することが報告されている<ref name=Ueda2005 /><ref name=Honma2002><pubmed>12397359</pubmed></ref> | # 転写因子[[Deleted in esophageal cancer]] (DEC)1/2によるE-box配列の制御<br>上記のCLOCK-BMAL1による制御に加え、E-boxには転写抑制因子として[[DEC1]]/[[DEC2|2]]が結合することが報告されている<ref name=Ueda2005 /><ref name=Honma2002><pubmed>12397359</pubmed></ref>。 | ||
これらのサブループは多くの遺伝子に対してさまざまな位相のリズムを生み出していると考えられている。 | これらのサブループは多くの遺伝子に対してさまざまな位相のリズムを生み出していると考えられている。 | ||
==振動体に関する議論== | ==振動体に関する議論== | ||
転写を介したフィードバックループが概日性の遺伝子発現リズムを生み出す基本骨格であることは広く受け入れられているが<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 /> | 転写を介したフィードバックループが概日性の遺伝子発現リズムを生み出す基本骨格であることは広く受け入れられているが<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 />、動物においてその仕組みが概日性の振動体を構成するか否かに関しては、上記の『[[時計遺伝子#概日振動体の構成要素|概日振動体の構成要素]]』'''1-5.'''の基準において、実験的な検証と議論が続いている<ref name=Lakin-Thomas2006><pubmed>16603673</pubmed></ref>。基準'''5.'''に関して、概日リズムに関わる転写関連因子を一定に発現した場合に、表出リズムが消失するか否かの検証実験が報告されている。ショウジョウバエにおいて、per遺伝子を欠損した個体において、per遺伝子を一定に発現させて遺伝子補完を行った場合でも、行動リズムは観察される<ref name=Yang2001><pubmed>11239435</pubmed></ref>。また、哺乳類の細胞においてCRY1/2タンパク質を一定に発現させたり、あるいは[[ラット]]においてPer1 [[mRNA]]を一定に発現させても、概日リズムは観察される<ref name=Fan2007><pubmed>17583506</pubmed></ref><ref name=Numano2006><pubmed>16537451</pubmed></ref>。 | ||
2011年、[[ヒト]][[赤血球]]において[[ペルオキシレドキシン]]というタンパク質の酸化還元状態が概日リズムを示すことが報告された<ref name=ONeill2011><pubmed>21270888</pubmed></ref> | 2011年、[[ヒト]][[赤血球]]において[[ペルオキシレドキシン]]というタンパク質の酸化還元状態が概日リズムを示すことが報告された<ref name=ONeill2011><pubmed>21270888</pubmed></ref>。赤血球には核がないため、本報告は転写を介したフィードバックループに依存しない振動機構が存在することを示唆するものであった。その後、Cry1/2欠損やPer1/2欠損、Bmal1欠損マウスにおいても、細胞レベルにおいては遺伝子やタンパク質の発現リズムが観察されることが報告されている<ref name=Ono2013><pubmed>23575670</pubmed></ref><ref name=Maywood2013><pubmed>23690615</pubmed></ref><ref name=Ko2010><pubmed>20967239</pubmed></ref><ref name=Ray2020><pubmed>32054765</pubmed></ref>。 | ||
==翻訳後修飾酵素の役割と進化的保存性== | ==翻訳後修飾酵素の役割と進化的保存性== | ||
ハムスターの行動リズムの短周期化を引き起こすtau変異の原因遺伝子として、[[カゼインキナーゼ]]1ε([[カゼインキナーゼ|CK1ε]])が報告されている<ref name=Lowrey2000><pubmed>10775102</pubmed></ref> | ハムスターの行動リズムの短周期化を引き起こすtau変異の原因遺伝子として、[[カゼインキナーゼ]]1ε([[カゼインキナーゼ|CK1ε]])が報告されている<ref name=Lowrey2000><pubmed>10775102</pubmed></ref>。CK1εは[[カゼインキナーゼ|CK1δ]]とともに、概日リズムの周期制御や温度補償性に関わり、CK1ε/δの低分子阻害剤は培養細胞における転写リズムの周期を延長する。CK1ε/δのin vitroの基質としては、PER2がよく解析されている<ref name=Lowrey2000 /><ref name=Isojima2009><pubmed>19805222</pubmed></ref>。 また、CK1εによるPER2のリン酸化は、[[ユビキチンE3リガーゼ]] [[β-transducin repeat containing]] ([[β-TrCP]]) に認識され、PER2は[[ユビキチン化]]された後に[[プロテアソーム]]によって分解される<ref name=田澤2009 /><ref name=岡村2004/><ref name=海老原2012 />。 | ||
CRYタンパク質レベルは[[ユビキチンリガーゼ]][[F-box/LRR-repeat protein 3]] ([[FBXL3]])や[[F-box and leucine rich repeat protein 21]] ([[FBXL21]])により制御されている<ref name=Hirano2013><pubmed>23452856</pubmed></ref><ref name=Yoo2013><pubmed>23452855</pubmed></ref> | CRYタンパク質レベルは[[ユビキチンリガーゼ]][[F-box/LRR-repeat protein 3]] ([[FBXL3]])や[[F-box and leucine rich repeat protein 21]] ([[FBXL21]])により制御されている<ref name=Hirano2013><pubmed>23452856</pubmed></ref><ref name=Yoo2013><pubmed>23452855</pubmed></ref>。FBXL3の欠損マウスは約28時間の行動リズムを示し、FBXL3とFBXL21の二重欠損マウスは行動リズムが消失する。 | ||
[[Casein Kinase 2]] (CK2) はショウジョウバエにおいて、長周期性を示す変異体として同定され<ref name=Lin2002><pubmed>12447397</pubmed></ref> | [[Casein Kinase 2]] (CK2) はショウジョウバエにおいて、長周期性を示す変異体として同定され<ref name=Lin2002><pubmed>12447397</pubmed></ref>、哺乳類の培養細胞においてもリズム周期や振幅の制御に関わることが報告されている<ref name=Tamaru2009><pubmed>19330005</pubmed></ref><ref name=Tsuchiya2009><pubmed>19491384</pubmed></ref>。CK2の基質としては、BMAL1やPER2が報告されている。 | ||
[[Glycogen synthase kinase 3]] ([[GSK3]]) の活性抑制によりショウジョウバエの行動リズムは長周期化し<ref name=Martinek2001><pubmed>11440719</pubmed></ref> | [[Glycogen synthase kinase 3]] ([[GSK3]]) の活性抑制によりショウジョウバエの行動リズムは長周期化し<ref name=Martinek2001><pubmed>11440719</pubmed></ref>、Gsk3βヘテロ欠損マウスも行動リズムが長周期となる<ref name=Lavoie2013 />。GSK3βの基質としては、PER2や[[REV-ERBα]]などが報告されている<ref name=Martinek2001><pubmed>11440719</pubmed></ref><ref name=Lavoie2013><pubmed>23919927</pubmed></ref><ref name=Yin2006><pubmed>16484495</pubmed></ref>。 | ||
[[Ca2+/カルモジュリン依存性タンパク質キナーゼII|Ca<sup>2+</sup>/カルモジュリン依存性タンパク質キナーゼII]] ([[CaMKII]])は哺乳類の細胞において転写リズムの振幅制御および温度補償性に関わり、[[CaMKIIα]]のキナーゼ活性欠失マウスにおいては行動リズムの長周期化やリズム消失が観察される<ref name=Kon2014><pubmed>24831701</pubmed></ref> | [[Ca2+/カルモジュリン依存性タンパク質キナーゼII|Ca<sup>2+</sup>/カルモジュリン依存性タンパク質キナーゼII]] ([[CaMKII]])は哺乳類の細胞において転写リズムの振幅制御および温度補償性に関わり、[[CaMKIIα]]のキナーゼ活性欠失マウスにおいては行動リズムの長周期化やリズム消失が観察される<ref name=Kon2014><pubmed>24831701</pubmed></ref>。CaMKIIの基質としては[[サイクリックAMP応答配列結合タンパク質]] ([[cAMP response element binding protein]], [[CREB]])やCLOCKが報告されている<ref name=Kon2014><pubmed>24831701</pubmed></ref><ref name=Yokota2001><pubmed>11299324</pubmed></ref>。 | ||
転写を介したフィードバックループを構成する転写関連因子は動物界、菌界、植物界で異なるのに対し、CK1やCK2, GSK-3β、CaMKIIなどの[[リン酸化酵素]]は[[真核生物]]で生物界を超えて役割が保存されている<ref name=O'Neill2011><pubmed>21270895</pubmed></ref><ref name=Uehara2019><pubmed>31097584</pubmed></ref><ref name=Mehra2009><pubmed>19450520</pubmed></ref><ref name=Wang2021><pubmed>34182778</pubmed></ref><ref name=Kon2021><pubmed>33931447</pubmed></ref> | 転写を介したフィードバックループを構成する転写関連因子は動物界、菌界、植物界で異なるのに対し、CK1やCK2, GSK-3β、CaMKIIなどの[[リン酸化酵素]]は[[真核生物]]で生物界を超えて役割が保存されている<ref name=O'Neill2011><pubmed>21270895</pubmed></ref><ref name=Uehara2019><pubmed>31097584</pubmed></ref><ref name=Mehra2009><pubmed>19450520</pubmed></ref><ref name=Wang2021><pubmed>34182778</pubmed></ref><ref name=Kon2021><pubmed>33931447</pubmed></ref>。 | ||
==シアノバクテリアにおけるタンパク質翻訳後振動== | ==シアノバクテリアにおけるタンパク質翻訳後振動== | ||
原核生物である[[シアノバクテリア]]では、概日振動体は[[Kaiタンパク質]]群によって形成される<ref name=岡村2004/><ref name=海老原2012 /> | 原核生物である[[シアノバクテリア]]では、概日振動体は[[Kaiタンパク質]]群によって形成される<ref name=岡村2004/><ref name=海老原2012 />。シアノバクテリアは恒暗条件では[[光合成]]が抑制されるため[[転写]]・[[翻訳]]を含めた細胞内の様々な活性が著しく低下するが、このように転写がほとんど起こらない恒暗条件においても[[KaiC]]タンパク質のリン酸化リズムは安定に継続する<ref name=Tomita2005><pubmed>15550625</pubmed></ref>。さらに、精製した[[KaiA]], [[KaiB]] およびKaiCタンパク質を[[ATP]]と共に混合することにより、KaiCタンパク質のリン酸化リズムを試験管内で再構成できることが示されている<ref name=Nakajima2005><pubmed>15831759</pubmed></ref>。 | ||
==関連項目== | ==関連項目== | ||
* [[細胞時計]] | * [[細胞時計]] | ||
2023年5月7日 (日) 14:03時点における最新版
金 尚宏1
小野 大輔2
1. 名古屋大学 トランスフォーマティブ生命分子研究所
2. 名古屋大学 環境医学研究所
DOI:10.14931/bsd.10189 原稿受付日:2023年4月25日 原稿完成日:2023年5月1日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
英:clock gene
概日リズムは約24時間周期の生物リズムであり、自律振動性、同調性、温度補償性という3つの特徴的な性質を有する。時計遺伝子は概日リズム生成の分子メカニズムに関わり、時計遺伝子の変異体では概日リズムの周期変化や概日リズム消失が観察される。哺乳類において、ほとんどの概日リズムは転写リズムによって生み出されており、時計遺伝子の多くは転写関連因子をコードしている。class I bHLH-PAS型転写因子CLOCK(あるいはNPAS2)はclass II bHLH-PAS型転写因子BMAL1とヘテロ二量体を形成し、E-boxと呼ばれるDNAシスエレメントを介してPer1/2やCry1/2遺伝子の転写を活性化する。翻訳されたPER1/2やCRY1/2タンパク質は複合体を形成し、細胞質から核に移行してCLOCK-BMAL1ヘテロ二量体の転写活性化を抑制する。この転写翻訳フィードバックループ (transcription-translation feedback loop, TTFL)が転写リズム生成の基本骨格である。時計遺伝子産物は翻訳後修飾によって制御されており、カゼインキナーゼ1ε/δ (CK1ε/δ、カゼインキナーゼ2 (CK2)、グリコーゲン合成酵素キナーゼ3β (GSK-3β)、Ca2+/カルモジュリン依存性タンパク質キナーゼII (CaMKII)などのリン酸化酵素が概日リズム生成に関与している。
時計遺伝子とは
時計遺伝子は概日リズム生成の分子メカニズム関わる一群の遺伝子を指す。概日リズムは約24時間周期の生物リズムであり、以下の3つの特徴的な性質を示す[1]。
- 明暗などの環境変化の無い恒常条件下でも約24時間周期のリズムを示す (自律振動性)
- 明暗や温度などの環境サイクルに同調する (同調性)
- リズムの周期が温度の影響をほとんど受けない (温度補償性)
概日リズムの周期長などの性質は遺伝することから、概日リズムを生み出す仕組みは遺伝子によって規定されていると考えられた[1] (1)。1971年にショウジョウバエの概日リズム変異体period (per)が報告され、その後、アカパンカビのfrequency (frq) 変異体、ハムスターのtau変異体、マウスのClock変異体、シアノバクテリアのkai変異体群が報告された[2]。そして、1984年にショウジョウバエper遺伝子がクローニングされ、続いて概日リズム変異体の原因遺伝子が続々と明らかになった[2][3]。そのため、時計遺伝子という用語は、順遺伝学的な手法から概日リズム変異体の原因遺伝子が同定された際に、その原因遺伝子を指すために元々は使用された。
その後、分子生物学的に概日リズムの生成に関与する遺伝子が数多く同定されたため、これら遺伝子を広義の時計遺伝子として扱うことがある[4]。また、概日性の周期で発現リズムを示す遺伝子をccg (clock-controlled gene)と呼ぶこともある。
概日振動体の構成要素
時計遺伝子は、狭義では概日リズムを生成する仕組みのうち、振動体を構成する要素を指すという考え方がある[4][5]。概日リズムは概日振動体によって生み出され、出力系を介して生理的な表出リズム(overt rhythm)として観察される[6][7]。そのため、表出リズムを消失させる要因としては、振動体の構成要素が障害された場合と、出力系が障害された場合が考えられる。ある因子が振動体を構成する要素であるか否かは、以下のような検証によって確認することができる[5]。
- その因子の欠損によって振動体は大きく影響を受け、表出リズムは消失する。
- その因子は約24時間の周期で自律振動する。
- その因子を増加(あるいは減少)させると、フィードバックにより、その因子の減少(あるいは増加)が引き起こされる。
- 明暗サイクルの位相シフトは、その因子の増減リズムおよび表出リズムの位相シフトを引き起こす。
- その因子の増減レベルを一定にすると、表出リズムは消失する。
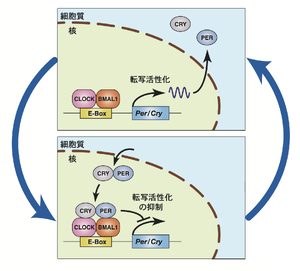
bHLH-PAS型転写因子CLOCK(あるいはNeuronal PAS Domain Protein 2 (NPAS2))はBasic Helix-Loop-Helix ARNT Like 1 (BMAL1)とヘテロ二量体を形成し、E-boxと呼ばれるDNAシスエレメントを介してPerやCry遺伝子の転写を活性化する。翻訳されたPERやCRYタンパク質は複合体を形成し、細胞質から核に移行してCLOCK-BMAL1の転写活性化を抑制する。その結果、PerやCry遺伝子の発現レベルは概日リズムを示す。文献[8]を元に改変)。
転写翻訳フィードバックループ
コアループ
哺乳類において、大部分の遺伝子発現リズムおよび生理リズムは、主に4種類の因子によって形成される転写翻訳フィードバックループ (transcription-translation feedback loop, TTFL)によって生み出されている[1][2][3]。4種類の因子とは、
- PERIOD (PER)タンパク質、
- CRYPTOCHROME (CRY)タンパク質、
- class I basic helix–loop–helix/Per-ARNT-SIM (bHLH-PAS)型転写因子、
- class II bHLH-PAS型転写因子
である。
これらの遺伝子産物は遺伝的冗長性があるものが多く、PERはPer1, Per2およびPer3遺伝子、CRYはCry1およびCry2遺伝子、class I bHLH-PAS型転写因子はClockおよびNpas2遺伝子、class II bHLH-PAS型転写因子はBmal1 およびBmal2遺伝子にコードされている[1][2][3]。CLOCK(あるいはNPAS2)はBMAL1とヘテロ二量体を形成し、E-boxと呼ばれるDNAシスエレメントを介してPer1/2やCry1/2遺伝子の転写を活性化する。翻訳されたPER1/2やCRY1/2タンパク質は複合体を形成し、細胞質から核に移行してCLOCK-BMAL1の転写活性化を抑制する。その結果、Per1/2やCry1/2遺伝子の発現レベルは概日リズムを示す (図)。これらの4因子による転写翻訳フィードバック機構をコアループと呼ぶ。
Per1/2二重欠損マウス、Cry1/2二重欠損マウス、Clock/Npas2二重欠損マウス、あるいはBmal1欠損マウスは、行動リズムの消失や遺伝子発現リズムの消失が引き起こされる[9][10][11][12]。そのため、これら因子は概日性の生理リズムを生成する必須因子である。転写を介したフィードバックループは動物、菌類、植物において遺伝子発現リズムを生成するメカニズムとして共通している。一方、これらの生物系統の間で、概日リズムの生成に関わる転写関連因子の配列相同性は限定的であるため、転写を介したフィードバックループは各生物界で独立に進化したと考えられている[2]。
サブループ
コアループに共役する形でいくつかのサブループが報告されている。
- ROR-response element (RORE)配列を介したBmal1遺伝子の制御
視交叉上核において主観的夜 (circadian time 16; CT16)に発現ピークを示すBmal1などの遺伝子の上流にはRORE配列が存在する[13][14]。RORE配列には転写抑制因子としてREV-ERBαおよびREV-ERBβ、転写活性化因子としてRAR Related Orphan Receptor (ROR)α、RORβ、RORγが作用することが報告されている。 - D-boxを介したPer1/2遺伝子の制御
Per1/2遺伝子の上流にはD-boxが存在する。D-boxには転写抑制因子としてE4 promoter Binding Protein 4 (E4BP4)、転写活性化因子としてD-Box Binding PAR BZIP Transcription Factor (DBP), Hepatic leukemia factor (HLF), Thyrotroph embryonic factor (TEF)が結合することが報告されている[14][15]。 - 転写因子Deleted in esophageal cancer (DEC)1/2によるE-box配列の制御
上記のCLOCK-BMAL1による制御に加え、E-boxには転写抑制因子としてDEC1/2が結合することが報告されている[14][16]。
これらのサブループは多くの遺伝子に対してさまざまな位相のリズムを生み出していると考えられている。
振動体に関する議論
転写を介したフィードバックループが概日性の遺伝子発現リズムを生み出す基本骨格であることは広く受け入れられているが[1][2][3]、動物においてその仕組みが概日性の振動体を構成するか否かに関しては、上記の『概日振動体の構成要素』1-5.の基準において、実験的な検証と議論が続いている[17]。基準5.に関して、概日リズムに関わる転写関連因子を一定に発現した場合に、表出リズムが消失するか否かの検証実験が報告されている。ショウジョウバエにおいて、per遺伝子を欠損した個体において、per遺伝子を一定に発現させて遺伝子補完を行った場合でも、行動リズムは観察される[18]。また、哺乳類の細胞においてCRY1/2タンパク質を一定に発現させたり、あるいはラットにおいてPer1 mRNAを一定に発現させても、概日リズムは観察される[19][20]。
2011年、ヒト赤血球においてペルオキシレドキシンというタンパク質の酸化還元状態が概日リズムを示すことが報告された[21]。赤血球には核がないため、本報告は転写を介したフィードバックループに依存しない振動機構が存在することを示唆するものであった。その後、Cry1/2欠損やPer1/2欠損、Bmal1欠損マウスにおいても、細胞レベルにおいては遺伝子やタンパク質の発現リズムが観察されることが報告されている[22][23][24][25]。
翻訳後修飾酵素の役割と進化的保存性
ハムスターの行動リズムの短周期化を引き起こすtau変異の原因遺伝子として、カゼインキナーゼ1ε(CK1ε)が報告されている[26]。CK1εはCK1δとともに、概日リズムの周期制御や温度補償性に関わり、CK1ε/δの低分子阻害剤は培養細胞における転写リズムの周期を延長する。CK1ε/δのin vitroの基質としては、PER2がよく解析されている[26][27]。 また、CK1εによるPER2のリン酸化は、ユビキチンE3リガーゼ β-transducin repeat containing (β-TrCP) に認識され、PER2はユビキチン化された後にプロテアソームによって分解される[1][2][3]。
CRYタンパク質レベルはユビキチンリガーゼF-box/LRR-repeat protein 3 (FBXL3)やF-box and leucine rich repeat protein 21 (FBXL21)により制御されている[28][29]。FBXL3の欠損マウスは約28時間の行動リズムを示し、FBXL3とFBXL21の二重欠損マウスは行動リズムが消失する。
Casein Kinase 2 (CK2) はショウジョウバエにおいて、長周期性を示す変異体として同定され[30]、哺乳類の培養細胞においてもリズム周期や振幅の制御に関わることが報告されている[31][32]。CK2の基質としては、BMAL1やPER2が報告されている。
Glycogen synthase kinase 3 (GSK3) の活性抑制によりショウジョウバエの行動リズムは長周期化し[33]、Gsk3βヘテロ欠損マウスも行動リズムが長周期となる[34]。GSK3βの基質としては、PER2やREV-ERBαなどが報告されている[33][34][35]。
Ca2+/カルモジュリン依存性タンパク質キナーゼII (CaMKII)は哺乳類の細胞において転写リズムの振幅制御および温度補償性に関わり、CaMKIIαのキナーゼ活性欠失マウスにおいては行動リズムの長周期化やリズム消失が観察される[36]。CaMKIIの基質としてはサイクリックAMP応答配列結合タンパク質 (cAMP response element binding protein, CREB)やCLOCKが報告されている[36][37]。
転写を介したフィードバックループを構成する転写関連因子は動物界、菌界、植物界で異なるのに対し、CK1やCK2, GSK-3β、CaMKIIなどのリン酸化酵素は真核生物で生物界を超えて役割が保存されている[38][39][40][41][42]。
シアノバクテリアにおけるタンパク質翻訳後振動
原核生物であるシアノバクテリアでは、概日振動体はKaiタンパク質群によって形成される[2][3]。シアノバクテリアは恒暗条件では光合成が抑制されるため転写・翻訳を含めた細胞内の様々な活性が著しく低下するが、このように転写がほとんど起こらない恒暗条件においてもKaiCタンパク質のリン酸化リズムは安定に継続する[43]。さらに、精製したKaiA, KaiB およびKaiCタンパク質をATPと共に混合することにより、KaiCタンパク質のリン酸化リズムを試験管内で再構成できることが示されている[44]。
関連項目
参考文献
- ↑ 1.0 1.1 1.2 1.3 1.4 1.5 田澤 仁 (2009).
マメから生まれた生物時計―エルヴィン・ビュニングの物語、学会出版センター - ↑ 2.0 2.1 2.2 2.3 2.4 2.5 2.6 2.7 岡村 均, 深田 吉孝 (2004).
時計遺伝子の分子生物学、シュプリンガーフェアラーク東京 - ↑ 3.0 3.1 3.2 3.3 3.4 3.5 海老原 史樹文, 吉村 崇 (2012).
時間生物学 化学同人. - ↑ 4.0 4.1 日本時間生物学会 時間生物学用語集
- ↑ 5.0 5.1
Aronson, B.D., Johnson, K.A., Loros, J.J., & Dunlap, J.C. (1994).
Negative feedback defining a circadian clock: autoregulation of the clock gene frequency. Science (New York, N.Y.), 263(5153), 1578-84. [PubMed:8128244] [WorldCat] [DOI] - ↑ Jay C. Dunlap, Jennifer J. Loros, Patricia J. Decoursey. (2003)
Chronobiology: Biological Timekeeping, Sinauer Associates Inc. - ↑
Eskin, A. (1979).
Identification and physiology of circadian pacemakers. Introduction. Federation proceedings, 38(12), 2570-2. [PubMed:499572] [WorldCat] - ↑ 金 尚宏, 深田 吉孝 (2019).
生物時計と体のリズム. 学術の動向 24, 8-19 - ↑
Bunger, M.K., Wilsbacher, L.D., Moran, S.M., Clendenin, C., Radcliffe, L.A., Hogenesch, J.B., ..., & Bradfield, C.A. (2000).
Mop3 is an essential component of the master circadian pacemaker in mammals. Cell, 103(7), 1009-17. [PubMed:11163178] [PMC] [WorldCat] [DOI] - ↑
Zheng, B., Albrecht, U., Kaasik, K., Sage, M., Lu, W., Vaishnav, S., ..., & Lee, C.C. (2001).
Nonredundant roles of the mPer1 and mPer2 genes in the mammalian circadian clock. Cell, 105(5), 683-94. [PubMed:11389837] [WorldCat] [DOI] - ↑
DeBruyne, J.P., Weaver, D.R., & Reppert, S.M. (2007).
CLOCK and NPAS2 have overlapping roles in the suprachiasmatic circadian clock. Nature neuroscience, 10(5), 543-5. [PubMed:17417633] [PMC] [WorldCat] [DOI] - ↑
van der Horst, G.T., Muijtjens, M., Kobayashi, K., Takano, R., Kanno, S., Takao, M., ..., & Yasui, A. (1999).
Mammalian Cry1 and Cry2 are essential for maintenance of circadian rhythms. Nature, 398(6728), 627-30. [PubMed:10217146] [WorldCat] [DOI] - ↑
Ueda, H.R., Chen, W., Adachi, A., Wakamatsu, H., Hayashi, S., Takasugi, T., ..., & Hashimoto, S. (2002).
A transcription factor response element for gene expression during circadian night. Nature, 418(6897), 534-9. [PubMed:12152080] [WorldCat] [DOI] - ↑ 14.0 14.1 14.2
Ueda, H.R., Hayashi, S., Chen, W., Sano, M., Machida, M., Shigeyoshi, Y., ..., & Hashimoto, S. (2005).
System-level identification of transcriptional circuits underlying mammalian circadian clocks. Nature genetics, 37(2), 187-92. [PubMed:15665827] [WorldCat] [DOI] - ↑
Mitsui, S., Yamaguchi, S., Matsuo, T., Ishida, Y., & Okamura, H. (2001).
Antagonistic role of E4BP4 and PAR proteins in the circadian oscillatory mechanism. Genes & development, 15(8), 995-1006. [PubMed:11316793] [PMC] [WorldCat] [DOI] - ↑
Honma, S., Kawamoto, T., Takagi, Y., Fujimoto, K., Sato, F., Noshiro, M., ..., & Honma, K. (2002).
Dec1 and Dec2 are regulators of the mammalian molecular clock. Nature, 419(6909), 841-4. [PubMed:12397359] [WorldCat] [DOI] - ↑
Lakin-Thomas, P.L. (2006).
Transcriptional feedback oscillators: maybe, maybe not.. Journal of biological rhythms, 21(2), 83-92. [PubMed:16603673] [WorldCat] [DOI] - ↑
Yang, Z., & Sehgal, A. (2001).
Role of molecular oscillations in generating behavioral rhythms in Drosophila. Neuron, 29(2), 453-67. [PubMed:11239435] [WorldCat] [DOI] - ↑
Fan, Y., Hida, A., Anderson, D.A., Izumo, M., & Johnson, C.H. (2007).
Cycling of CRYPTOCHROME proteins is not necessary for circadian-clock function in mammalian fibroblasts. Current biology : CB, 17(13), 1091-100. [PubMed:17583506] [PMC] [WorldCat] [DOI] - ↑
Numano, R., Yamazaki, S., Umeda, N., Samura, T., Sujino, M., Takahashi, R., ..., & Tei, H. (2006).
Constitutive expression of the Period1 gene impairs behavioral and molecular circadian rhythms. Proceedings of the National Academy of Sciences of the United States of America, 103(10), 3716-21. [PubMed:16537451] [PMC] [WorldCat] [DOI] - ↑
O'Neill, J.S., & Reddy, A.B. (2011).
Circadian clocks in human red blood cells. Nature, 469(7331), 498-503. [PubMed:21270888] [PMC] [WorldCat] [DOI] - ↑
Ono, D., Honma, S., & Honma, K. (2013).
Cryptochromes are critical for the development of coherent circadian rhythms in the mouse suprachiasmatic nucleus. Nature communications, 4, 1666. [PubMed:23575670] [WorldCat] [DOI] - ↑
Maywood, E.S., Drynan, L., Chesham, J.E., Edwards, M.D., Dardente, H., Fustin, J.M., ..., & Hastings, M.H. (2013).
Analysis of core circadian feedback loop in suprachiasmatic nucleus of mCry1-luc transgenic reporter mouse. Proceedings of the National Academy of Sciences of the United States of America, 110(23), 9547-52. [PubMed:23690615] [PMC] [WorldCat] [DOI] - ↑
Ko, C.H., Yamada, Y.R., Welsh, D.K., Buhr, E.D., Liu, A.C., Zhang, E.E., ..., & Takahashi, J.S. (2010).
Emergence of noise-induced oscillations in the central circadian pacemaker. PLoS biology, 8(10), e1000513. [PubMed:20967239] [PMC] [WorldCat] [DOI] - ↑
Ray, S., Valekunja, U.K., Stangherlin, A., Howell, S.A., Snijders, A.P., Damodaran, G., & Reddy, A.B. (2020).
Circadian rhythms in the absence of the clock gene Bmal1. Science (New York, N.Y.), 367(6479), 800-806. [PubMed:32054765] [WorldCat] [DOI] - ↑ 26.0 26.1
Lowrey, P.L., Shimomura, K., Antoch, M.P., Yamazaki, S., Zemenides, P.D., Ralph, M.R., ..., & Takahashi, J.S. (2000).
Positional syntenic cloning and functional characterization of the mammalian circadian mutation tau. Science (New York, N.Y.), 288(5465), 483-92. [PubMed:10775102] [PMC] [WorldCat] [DOI] - ↑
Isojima, Y., Nakajima, M., Ukai, H., Fujishima, H., Yamada, R.G., Masumoto, K.H., ..., & Ueda, H.R. (2009).
CKIepsilon/delta-dependent phosphorylation is a temperature-insensitive, period-determining process in the mammalian circadian clock. Proceedings of the National Academy of Sciences of the United States of America, 106(37), 15744-9. [PubMed:19805222] [PMC] [WorldCat] [DOI] - ↑
Hirano, A., Yumimoto, K., Tsunematsu, R., Matsumoto, M., Oyama, M., Kozuka-Hata, H., ..., & Fukada, Y. (2013).
FBXL21 regulates oscillation of the circadian clock through ubiquitination and stabilization of cryptochromes. Cell, 152(5), 1106-18. [PubMed:23452856] [WorldCat] [DOI] - ↑
Yoo, S.H., Mohawk, J.A., Siepka, S.M., Shan, Y., Huh, S.K., Hong, H.K., ..., & Takahashi, J.S. (2013).
Competing E3 ubiquitin ligases govern circadian periodicity by degradation of CRY in nucleus and cytoplasm. Cell, 152(5), 1091-105. [PubMed:23452855] [PMC] [WorldCat] [DOI] - ↑
Lin, J.M., Kilman, V.L., Keegan, K., Paddock, B., Emery-Le, M., Rosbash, M., & Allada, R. (2002).
A role for casein kinase 2alpha in the Drosophila circadian clock. Nature, 420(6917), 816-20. [PubMed:12447397] [WorldCat] [DOI] - ↑
Tamaru, T., Hirayama, J., Isojima, Y., Nagai, K., Norioka, S., Takamatsu, K., & Sassone-Corsi, P. (2009).
CK2alpha phosphorylates BMAL1 to regulate the mammalian clock. Nature structural & molecular biology, 16(4), 446-8. [PubMed:19330005] [PMC] [WorldCat] [DOI] - ↑
Tsuchiya, Y., Akashi, M., Matsuda, M., Goto, K., Miyata, Y., Node, K., & Nishida, E. (2009).
Involvement of the protein kinase CK2 in the regulation of mammalian circadian rhythms. Science signaling, 2(73), ra26. [PubMed:19491384] [WorldCat] [DOI] - ↑ 33.0 33.1
Martinek, S., Inonog, S., Manoukian, A.S., & Young, M.W. (2001).
A role for the segment polarity gene shaggy/GSK-3 in the Drosophila circadian clock. Cell, 105(6), 769-79. [PubMed:11440719] [WorldCat] [DOI] - ↑ 34.0 34.1
Lavoie, J., Hébert, M., & Beaulieu, J.M. (2013).
Glycogen synthase kinase-3β haploinsufficiency lengthens the circadian locomotor activity period in mice. Behavioural brain research, 253, 262-5. [PubMed:23919927] [WorldCat] [DOI] - ↑
Yin, L., Wang, J., Klein, P.S., & Lazar, M.A. (2006).
Nuclear receptor Rev-erbalpha is a critical lithium-sensitive component of the circadian clock. Science (New York, N.Y.), 311(5763), 1002-5. [PubMed:16484495] [WorldCat] [DOI] - ↑ 36.0 36.1
Kon, N., Yoshikawa, T., Honma, S., Yamagata, Y., Yoshitane, H., Shimizu, K., ..., & Fukada, Y. (2014).
CaMKII is essential for the cellular clock and coupling between morning and evening behavioral rhythms. Genes & development, 28(10), 1101-10. [PubMed:24831701] [PMC] [WorldCat] [DOI] - ↑
Yokota, S., Yamamoto, M., Moriya, T., Akiyama, M., Fukunaga, K., Miyamoto, E., & Shibata, S. (2001).
Involvement of calcium-calmodulin protein kinase but not mitogen-activated protein kinase in light-induced phase delays and Per gene expression in the suprachiasmatic nucleus of the hamster. Journal of neurochemistry, 77(2), 618-27. [PubMed:11299324] [WorldCat] [DOI] - ↑
O'Neill, J.S., van Ooijen, G., Dixon, L.E., Troein, C., Corellou, F., Bouget, F.Y., ..., & Millar, A.J. (2011).
Circadian rhythms persist without transcription in a eukaryote. Nature, 469(7331), 554-8. [PubMed:21270895] [PMC] [WorldCat] [DOI] - ↑
Uehara, T.N., Mizutani, Y., Kuwata, K., Hirota, T., Sato, A., Mizoi, J., ..., & Nakamichi, N. (2019).
Casein kinase 1 family regulates PRR5 and TOC1 in the Arabidopsis circadian clock. Proceedings of the National Academy of Sciences of the United States of America, 116(23), 11528-11536. [PubMed:31097584] [PMC] [WorldCat] [DOI] - ↑
Mehra, A., Shi, M., Baker, C.L., Colot, H.V., Loros, J.J., & Dunlap, J.C. (2009).
A role for casein kinase 2 in the mechanism underlying circadian temperature compensation. Cell, 137(4), 749-60. [PubMed:19450520] [PMC] [WorldCat] [DOI] - ↑
Wang, B., Zhou, X., Gerber, S.A., Loros, J.J., & Dunlap, J.C. (2021).
Cellular Calcium Levels Influenced by NCA-2 Impact Circadian Period Determination in Neurospora. mBio, 12(3), e0149321. [PubMed:34182778] [PMC] [WorldCat] [DOI] - ↑
Kon, N., Wang, H.T., Kato, Y.S., Uemoto, K., Kawamoto, N., Kawasaki, K., ..., & Fukada, Y. (2021).
Na+/Ca2+ exchanger mediates cold Ca2+ signaling conserved for temperature-compensated circadian rhythms. Science advances, 7(18). [PubMed:33931447] [PMC] [WorldCat] [DOI] - ↑
Tomita, J., Nakajima, M., Kondo, T., & Iwasaki, H. (2005).
No transcription-translation feedback in circadian rhythm of KaiC phosphorylation. Science (New York, N.Y.), 307(5707), 251-4. [PubMed:15550625] [WorldCat] [DOI] - ↑
Nakajima, M., Imai, K., Ito, H., Nishiwaki, T., Murayama, Y., Iwasaki, H., ..., & Kondo, T. (2005).
Reconstitution of circadian oscillation of cyanobacterial KaiC phosphorylation in vitro. Science (New York, N.Y.), 308(5720), 414-5. [PubMed:15831759] [WorldCat] [DOI]