「ヒストンアセチル基転移酵素」の版間の差分
細 →タイプB |
細 →分類 |
||
| 33行目: | 33行目: | ||
{| class="wikitable" | {| class="wikitable" | ||
|+表1. | |+表1. ヒストンアセチル基転移酵素の分類 | ||
! ファミリー !! メンバー !! 他の名称 !! 基質 | ! ファミリー !! メンバー !! 他の名称 !! 基質 | ||
|- | |- | ||
2023年5月31日 (水) 23:13時点における版
内田 周作
京都大学大学院医学研究科
DOI:10.14931/bsd.7707 原稿受付日:2023年5月22日 原稿完成日:2023年5月31日
担当編集委員:和田 圭司(国立研究開発法人国立精神・神経医療研究センター)
英: histone acetyltransferase
略称: HAT
ヒストンアセチル基転移酵素は、アセチルCoAからアセチル基を転移して、ヒストンタンパク質のリジン残基にアセチル基を結合させる酵素。一般的に、ヒストンのアセチル化は遺伝子発現の活性化と関連する。
ヒストンアセチル基転移酵素とは
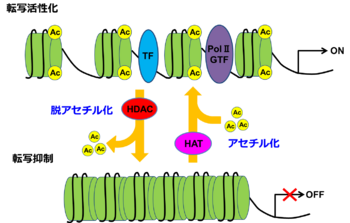
ヒストンがヒストンアセチル基転移酵素 (HAT)によりアセチル化された状態ではヒストン-DNA間の結合が緩むことで、TFやPolⅡの結合が可能となり、転写は活性化される。逆にヒストン脱アセチル化酵素 (HDAC)により、ヒストンが脱アセチル化されるとTF、PolⅡが結合出来ないため転写は抑制される。
GTF:general transcription factor:基本転写因子群、Ac:acetylation:アセチル化。
村尾 直哉作成。アセチル化の項目より。
ヒストンタンパク質はリジンやアルギニン残基を豊富に含む塩基性の高いタンパク質で、ゲノムDNAが巻きつくことでヌクレオソームと呼ばれる構造単位を作り出している。ヒストンには、H1/H5(リンカーヒストン)、H2、H3、およびH4(コアヒストン)の5つのファミリーがある。ヌクレオソームは、2つのH2A-H2B二量体と1つのH3-H4四量体から形成されている。ヒストンタンパク質に対する化学修飾の1つにリジン残基のアセチル化がある。ヒストンタンパク質のリジン残基のアセチル化は、リジンの正電荷を中性化し、ヒストンとDNAの間の静電引力を弱めることでDNAを部分的に解くことができ、これにより転写活性化因子がリクルートされるため、遺伝子発現の活性化と関連している(図1)。ヒストンタンパク質のリジン残基のアセチル化を担う酵素を総称してヒストンアセチル基転移酵素と呼んでいる。ヒストンアセチル基転移酵素の主な標的はヒストンH3とH4であるが、ヒストンH2AとH2Bもアセチル化されることが知られている[1] 。
分類
ヒストンアセチル基転移酵素はその細胞内局在に基づいて核局在型のタイプAと細胞質局在型のタイプBの2つのクラスに大別されている(表1)[2]。しかし、核・細胞質の両方で機能するなど、特定のクラスに分類できないヒストンアセチル基転移酵素もある。
タイプA
主に5つのファミリーに分類されている。1) Gcn5関連N-アセチルトランスフェラーゼ(GNAT)ファミリー、2) CBP/p300、3) MOZ、酵母YBF2、SAS2、TIP60(MYST)ファミリー、4) 転写因子関連ヒストンアセチル基転移酵素、5) 核受容体関連ヒストンアセチル基転移酵素がある。これらタイプAのヒストンアセチル基転移酵素はブロモドメインを含んでおり、ヒストンのリジン残基をアセチル化することによって遺伝子発現の調節に関与している。
Gcn5関連N-アセチルトランスフェラーゼファミリー
Gcn5関連N-アセチルトランスフェラーゼ(GNAT)ファミリーの主要メンバーには、最初に同定されたgeneral control of amino acid synthesis 5 (Gcn5)に加えてelongation protein 3 (ELP3)、p300/CBP-associated factor (PCAF)、chromodomain on chromosome Y protein、establishment of cohesion 1、establishment of cohesion 1 homolog 1、establishment of cohesion 1 homolog 2(establishment of cohesion 1 homolog 1のパラログ)、ADA-two-A containing 2、mechanosensory abnormal 17などが含まれる。これらのヒストンアセチル基転移酵素にはブロモドメインが存在し、ヒストンH2B、H3、H4のリジン残基をアセチル化することが示されている[3] 。
MYSTファミリー
MOZ、酵母YBF2、SAS2、TIP60(MYST)ファミリーには、Tip60、MOZ、MOF、MORF、およびHBO1が含まれる。これらのヒストンアセチル基転移酵素には、カノニカルなアセチルCoA結合部位とC2HC型ジンクフィンガーモチーフを持つ240アミノ酸長のMYST領域がある。MYSTファミリーのほとんどのヒストンアセチル基転移酵素は、タンパク質-タンパク質相互作用に関与するクロモドメインを有している。構造的なアライメントは、これらのクロモドメインがカノニカルなポリコンブ関連クロモドメインとわずかに異なることを示唆している[4] 。MYSTファミリーのヒストンアセチル基転移酵素は、ヒストンH2A、H3、H4のリジン残基をアセチル化することが知られている。
CBP/p300
CBPとp300はCBP/p300ファミリーのパラログである。CBPとp300の主要なタンパク質相互作用ドメインは、核受容体相互作用ドメイン、リン酸化型CREB結合ドメイン、システイン/ヒスチジン領域(CH1およびCH3)、インターフェロン応答結合ドメインである[5] 。CBP/p300は、アセチルトランスフェラーゼドメイン、ブロモドメイン、プラントホメオドメインタイプのジンクフィンガーモチーフを持っている。CBP/p300はヒストン以外にも多くの基質を有している。
タイプB
細胞質に存在し、新しく合成されたヒストンをヌクレオソームに組み込む前にアセチル化する。タイプBのヒストンアセチル基転移酵素によってヒストンに付加されたアセチル基は、核に移行してヒストン脱アセチル化酵素(HDAC)によって脱アセチル化される。Hat1は、タイプBのヒストンアセチル基転移酵素の数少ない例の1つであるが、構造的・機能的にはGNATファミリーに属するとも考えられている。
| ファミリー | メンバー | 他の名称 | 基質 |
|---|---|---|---|
| Gcn5関連N-アセチルトランスフェラーゼ(GNAT)ファミリー | |||
| KAT1 | HAT1 | H4, (H2A) | |
| KAT2A | GCN5 | H3, (H4, H2B) | |
| KAT2B | PCAF | H3, H4 | |
| MYSTファミリー | |||
| KAT5 | Tip60 | H2A, H4, (H3) | |
| KAT6A | MOZ, MYST3 | H3, H4 | |
| KAT6B | MORF, MYST4 | H3, H4 | |
| KAT7 | HBO1, MYST2 | H3, H4 | |
| KAT8 | MOF, MYST1 | H4 | |
| CBP/p300ファミリー | |||
| KAT3A | CBP | H2A, H2B, H3, H4 | |
| KAT3B | p300 | H2A, H2B, H3, H4 | |
| 転写因子関連ヒストンアセチル基転移酵素 | |||
| KAT4 | TAF1, TBP | H3, H4, (H2A) | |
| KAT12 | TIFIIIC90 | H3 | |
| 核受容体関連ヒストンアセチル基転移酵素 | |||
| KAT13A | SRC1 | H3, H4 | |
| KAT13C | SRC2 | H3, H4 | |
| KAT13B | SRC3 | H3, H4 | |
| KAT13D | CLOCK | H3, H4 | |
| 細胞質型ヒストンアセチル基転移酵素 | |||
| KAT1 | HAT1 | H4, (H2A) | |
| HAT4 | NAA60 | H4 |
機能
触媒活性
ヒストンH3の9番、14番、18番、23番目のリジン残基、ヒストンH4の5番、8番、12番、16番目のリジン残基は全てアセチル化の標的となる [6][1] 。ヒストンH2Aでは5番と9番目リジン残基、ヒストンH2Bでは5番、12番、15番、20番目のリジン残基がアセチル化される[6][1] 。
活性調節
ヒストンアセチル基転移酵素の活性は、自己アセチル化、細胞内シグナル、細胞内代謝系など様々なメカニズムによって調節されると考えられている。自己アセチル化は、ヒストンタンパク質リジン残基のアセチル化を介して酵素活性が調節されることを意味する。
Tip60は最初に自己アセチル化が報告されたヒストンアセチル基転移酵素で、MYSTドメインの活性部位である327番目のリジン残基のアセチル化は酵素活性の重要な調節マーカーとして機能する。
p300/CBP-associated factor(PCAF)の自己アセチル化は、核局在化シグナルの5つのリジン残基を特異的にターゲットとする。自己アセチル化されたPCAFは核に局在し、遺伝子発現を活性化する。一方、脱アセチル化されたPCAFは、アポトーシス細胞の細胞質に主に存在していることから、PCAFの自己アセチル化による転写誘導が細胞の生存に関わることが示唆されている[7] 。同様に、p300およびCBPの活性も自己アセチル化を介して調節される[8] 。
CBPによるアセチル化は、カルシウムイオンやcAMPなどのセカンドメッセンジャー経路の下流に存在するリン酸化カスケードなどのシグナル伝達経路によって調節されることで酵素活性に影響を与える[9][10] 。
神経発達における役割
CBP/p300が神経発生において重要であることを示す研究がいくつかある。これらヒストンアセチル基転移酵素の遺伝子欠損マウスにおいて、神経発生の初期段階である胎生(E)9.0からE11.5で胚性致死となることが報告されており、造血、血管新生、心臓、肺、および腸臓器形成が障害されている[11][12][13][14] 。
マウス発生過程でのp300メッセンジャーRNA(mRNA)は、神経組織での発現が高いことが示されており、p300が神経発生に関与していることを示唆している。事実、CBPまたはp300をノックアウトすると神経管の異常が生じ、twist、paired box 3(PAX3)、およびactivating enhancer binding protein 2(AP2)などの他の転写因子に影響を与えることで、神経発生に障害を及ぼすことが示されている[15][14] 。
シナプス可塑性における役割
マウス海馬CA1領域におけるヒストンH3のアセチル化は文脈学習後に亢進し、このヒストンアセチル化の上昇は、N-メチル-D-アスパラギン酸型グルタミン酸受容体(NMDA受容体)とERKシグナル伝達経路の両方の活性化と関連している[16] 。2010年、Pelegらは、ゲノムワイド解析により、ヒストンH4の12番目のリジン残基(H4K12)のアセチル化が長期記憶形成に必須の転写活性化と関連していることを見出した[17] 。多くの可塑性関連遺伝子(c-FosやBdnf遺伝子)の発現上昇も、CBPなどのヒストンアセチル基転移酵素活性の増加に関連して同定されている。マウス前脳特異的CBP欠損マウスは、長期記憶形成の障害、シナプス可塑性の障害、シナプス機能や可塑性に関与する遺伝子の発現低下を示した[18] 。別の研究においても、CBPノックアウトマウス(ヘテロ型)において、海馬依存性の学習と記憶課題の障害や、学習・記憶に関わるとされる長期増強(LTP)の低下が報告されている[19] 。これらの知見から、CBPは、特に学習・記憶やシナプス可塑性において重要な役割を担っていることが示唆されている。
MYSTファミリーのTip60は、転写調節、DNA修復、クロマチンリモデリングを含む様々な細胞機能において重要な役割を果たすヒストンアセチル基転移酵素である。Tip60は、記憶形成やシナプス可塑性に関与していることが示唆されている。海馬においてTip60をノックダウンしたマウスは、H4K12アセチル化レベルの低下、長期記憶形成の阻害、長期増強(LTP)の維持に障害を認めた[20] 。ショウジョウバエにおいて、Tip60とGNATファミリーのGcn5は長期記憶の維持に必要であることが示されている[21] 。このように、ヒストンアセチル基転移酵素によるヒストンアセチル化が記憶形成やシナプス可塑性に重要な役割を果たすことが示唆されている。
疾患との関わり
神経変性疾患においてヒストンアセチル基転移酵素活性およびヒストンタンパク質のアセチル化の障害が報告されており、認知機能の低下や運動障害との関連が示唆されている。CBPがpolyQ凝集体に存在することが示され、神経変性疾患との関連が想定されている[22] 。事実、CAGリピートの異常な伸長はCBPやPCAFを含む転写制御因子の機能低下を誘発し、遺伝子発現の低下が示されている[23] 。ハンチントン病モデルマウス及び患者脳において、CBPを介した転写活性化異常の存在が示唆されている[24] 。
CBP/p300は発生において重要な酵素であり、これらヒストンアセチル基転移酵素の突然変異や欠失は、知的障害などの症状を呈するRubinstein-Taybi症候群との関連が示唆されている[25] 。
筋萎縮性側索硬化症マウスモデル(SOD1 G86R)では、腰部脊髄コリン作動性運動ニューロンにおいてCBPの減少とヒストンH3アセチル化の減少が見出されている[26] 。
野生型Presenilin 1(PS1)はCBPとp300の転写活性能を促進するが、家族性アルツハイマー病関連遺伝子のPS1変異はこの効果を示さなかったことから、CBP/p300とアルツハイマー病との関連が示唆されている[27] 。アルツハイマー病モデルマウスにおいて、CBPの発現誘導はBdnfの発現亢進を介して記憶学習能力を回復させることが示されている[28] 。また、CBP/p300の内因性阻害分子であるEID1は、アルツハイマー病患者の海馬において細胞質から核へ移行すること、EID1の過剰発現は、ヒストンH3のアセチル化の低下、海馬におけるLTPの低下、空間記憶障害を引き起こすことが示された。この結果はCBP/p300がアルツハイマー病態に関わることを示唆している[29] 。
CBP/p300以外のヒストンアセチル基転移酵素と脳疾患との関連研究はまだ少なく、今後の解析が待たれる。
関連語
参考文献
- ↑ 1.0 1.1 1.2
Sun, H., Kennedy, P.J., & Nestler, E.J. (2013).
Epigenetics of the depressed brain: role of histone acetylation and methylation. Neuropsychopharmacology : official publication of the American College of Neuropsychopharmacology, 38(1), 124-37. [PubMed:22692567] [PMC] [WorldCat] [DOI] - ↑
Sheikh, B.N., & Akhtar, A. (2019).
The many lives of KATs - detectors, integrators and modulators of the cellular environment. Nature reviews. Genetics, 20(1), 7-23. [PubMed:30390049] [WorldCat] [DOI] - ↑
Lee, K.K., & Workman, J.L. (2007).
Histone acetyltransferase complexes: one size doesn't fit all. Nature reviews. Molecular cell biology, 8(4), 284-95. [PubMed:17380162] [WorldCat] [DOI] - ↑
Sanjuán, R., & Marín, I. (2001).
Tracing the origin of the compensasome: evolutionary history of DEAH helicase and MYST acetyltransferase gene families. Molecular biology and evolution, 18(3), 330-43. [PubMed:11230534] [WorldCat] [DOI] - ↑
Livengood, J.A., Scoggin, K.E., Van Orden, K., McBryant, S.J., Edayathumangalam, R.S., Laybourn, P.J., & Nyborg, J.K. (2002).
p53 Transcriptional activity is mediated through the SRC1-interacting domain of CBP/p300. The Journal of biological chemistry, 277(11), 9054-61. [PubMed:11782467] [WorldCat] [DOI] - ↑ 6.0 6.1
Roth, S.Y., Denu, J.M., & Allis, C.D. (2001).
Histone acetyltransferases. Annual review of biochemistry, 70, 81-120. [PubMed:11395403] [WorldCat] [DOI] - ↑
Blanco, J.C., Minucci, S., Lu, J., Yang, X.J., Walker, K.K., Chen, H., ..., & Ozato, K. (1998).
The histone acetylase PCAF is a nuclear receptor coactivator. Genes & development, 12(11), 1638-51. [PubMed:9620851] [PMC] [WorldCat] [DOI] - ↑
Thompson, P.R., Wang, D., Wang, L., Fulco, M., Pediconi, N., Zhang, D., ..., & Cole, P.A. (2004).
Regulation of the p300 HAT domain via a novel activation loop. Nature structural & molecular biology, 11(4), 308-15. [PubMed:15004546] [WorldCat] [DOI] - ↑
Bengtson, C.P., & Bading, H. (2012).
Nuclear calcium signaling. Advances in experimental medicine and biology, 970, 377-405. [PubMed:22351065] [WorldCat] [DOI] - ↑
Vo, N., & Goodman, R.H. (2001).
CREB-binding protein and p300 in transcriptional regulation. The Journal of biological chemistry, 276(17), 13505-8. [PubMed:11279224] [WorldCat] [DOI] - ↑
Kung, A.L., Rebel, V.I., Bronson, R.T., Ch'ng, L.E., Sieff, C.A., Livingston, D.M., & Yao, T.P. (2000).
Gene dose-dependent control of hematopoiesis and hematologic tumor suppression by CBP. Genes & development, 14(3), 272-7. [PubMed:10673499] [PMC] [WorldCat] - ↑
Oike, Y., Hata, A., Mamiya, T., Kaname, T., Noda, Y., Suzuki, M., ..., & Yamamura, K. (1999).
Truncated CBP protein leads to classical Rubinstein-Taybi syndrome phenotypes in mice: implications for a dominant-negative mechanism. Human molecular genetics, 8(3), 387-96. [PubMed:9949198] [WorldCat] [DOI] - ↑
Shikama, N., Lutz, W., Kretzschmar, R., Sauter, N., Roth, J.F., Marino, S., ..., & Eckner, R. (2003).
Essential function of p300 acetyltransferase activity in heart, lung and small intestine formation. The EMBO journal, 22(19), 5175-85. [PubMed:14517255] [PMC] [WorldCat] [DOI] - ↑ 14.0 14.1
Yao, T.P., Oh, S.P., Fuchs, M., Zhou, N.D., Ch'ng, L.E., Newsome, D., ..., & Eckner, R. (1998).
Gene dosage-dependent embryonic development and proliferation defects in mice lacking the transcriptional integrator p300. Cell, 93(3), 361-72. [PubMed:9590171] [WorldCat] [DOI] - ↑
Tanaka, Y., Naruse, I., Hongo, T., Xu, M., Nakahata, T., Maekawa, T., & Ishii, S. (2000).
Extensive brain hemorrhage and embryonic lethality in a mouse null mutant of CREB-binding protein. Mechanisms of development, 95(1-2), 133-45. [PubMed:10906457] [WorldCat] [DOI] - ↑
Levenson, J.M., O'Riordan, K.J., Brown, K.D., Trinh, M.A., Molfese, D.L., & Sweatt, J.D. (2004).
Regulation of histone acetylation during memory formation in the hippocampus. The Journal of biological chemistry, 279(39), 40545-59. [PubMed:15273246] [WorldCat] [DOI] - ↑
Peleg, S., Sananbenesi, F., Zovoilis, A., Burkhardt, S., Bahari-Javan, S., Agis-Balboa, R.C., ..., & Fischer, A. (2010).
Altered histone acetylation is associated with age-dependent memory impairment in mice. Science (New York, N.Y.), 328(5979), 753-6. [PubMed:20448184] [WorldCat] [DOI] - ↑
Korzus, E., Rosenfeld, M.G., & Mayford, M. (2004).
CBP histone acetyltransferase activity is a critical component of memory consolidation. Neuron, 42(6), 961-72. [PubMed:15207240] [PMC] [WorldCat] [DOI] - ↑
Alarcón, J.M., Malleret, G., Touzani, K., Vronskaya, S., Ishii, S., Kandel, E.R., & Barco, A. (2004).
Chromatin acetylation, memory, and LTP are impaired in CBP+/- mice: a model for the cognitive deficit in Rubinstein-Taybi syndrome and its amelioration. Neuron, 42(6), 947-59. [PubMed:15207239] [WorldCat] [DOI] - ↑
Uchida, S., Teubner, B.J.W., Hevi, C., Hara, K., Kobayashi, A., Dave, R.M., ..., & Shumyatsky, G.P. (2017).
CRTC1 Nuclear Translocation Following Learning Modulates Memory Strength via Exchange of Chromatin Remodeling Complexes on the Fgf1 Gene. Cell reports, 18(2), 352-366. [PubMed:28076781] [PMC] [WorldCat] [DOI] - ↑
Hirano, Y., Ihara, K., Masuda, T., Yamamoto, T., Iwata, I., Takahashi, A., ..., & Saitoe, M. (2016).
Shifting transcriptional machinery is required for long-term memory maintenance and modification in Drosophila mushroom bodies. Nature communications, 7, 13471. [PubMed:27841260] [PMC] [WorldCat] [DOI] - ↑
Kazantsev, A., Preisinger, E., Dranovsky, A., Goldgaber, D., & Housman, D. (1999).
Insoluble detergent-resistant aggregates form between pathological and nonpathological lengths of polyglutamine in mammalian cells. Proceedings of the National Academy of Sciences of the United States of America, 96(20), 11404-9. [PubMed:10500189] [PMC] [WorldCat] [DOI] - ↑
Steffan, J.S., Bodai, L., Pallos, J., Poelman, M., McCampbell, A., Apostol, B.L., ..., & Thompson, L.M. (2001).
Histone deacetylase inhibitors arrest polyglutamine-dependent neurodegeneration in Drosophila. Nature, 413(6857), 739-43. [PubMed:11607033] [WorldCat] [DOI] - ↑
Wilburn, B., Rudnicki, D.D., Zhao, J., Weitz, T.M., Cheng, Y., Gu, X., ..., & Yang, X.W. (2011).
An antisense CAG repeat transcript at JPH3 locus mediates expanded polyglutamine protein toxicity in Huntington's disease-like 2 mice. Neuron, 70(3), 427-40. [PubMed:21555070] [PMC] [WorldCat] [DOI] - ↑
Kalkhoven, E., Roelfsema, J.H., Teunissen, H., den Boer, A., Ariyurek, Y., Zantema, A., ..., & Peters, D.J. (2003).
Loss of CBP acetyltransferase activity by PHD finger mutations in Rubinstein-Taybi syndrome. Human molecular genetics, 12(4), 441-50. [PubMed:12566391] [WorldCat] [DOI] - ↑
Rouaux, C., Jokic, N., Mbebi, C., Boutillier, S., Loeffler, J.P., & Boutillier, A.L. (2003).
Critical loss of CBP/p300 histone acetylase activity by caspase-6 during neurodegeneration. The EMBO journal, 22(24), 6537-49. [PubMed:14657026] [PMC] [WorldCat] [DOI] - ↑
Francis, Y.I., Diss, J.K., Kariti, M., Stephanou, A., & Latchman, D.S. (2007).
p300 activation by Presenilin 1 but not by its M146L mutant. Neuroscience letters, 413(2), 137-40. [PubMed:17197080] [WorldCat] [DOI] - ↑
Caccamo, A., Maldonado, M.A., Bokov, A.F., Majumder, S., & Oddo, S. (2010).
CBP gene transfer increases BDNF levels and ameliorates learning and memory deficits in a mouse model of Alzheimer's disease. Proceedings of the National Academy of Sciences of the United States of America, 107(52), 22687-92. [PubMed:21149712] [PMC] [WorldCat] [DOI] - ↑
Liu, R., Lei, J.X., Luo, C., Lan, X., Chi, L., Deng, P., ..., & Liu, Q.Y. (2012).
Increased EID1 nuclear translocation impairs synaptic plasticity and memory function associated with pathogenesis of Alzheimer's disease. Neurobiology of disease, 45(3), 902-12. [PubMed:22186421] [PMC] [WorldCat] [DOI]