「細胞質ポリアデニル化要素結合タンパク質」の版間の差分
ページの作成:「{{}}」 |
細編集の要約なし |
||
| (同じ利用者による、間の18版が非表示) | |||
| 1行目: | 1行目: | ||
{{}} | <div align="right"> | ||
<font size="+1">[https://researchmap.jp/TsuyoshiUdagawa 宇田川 剛]</font><br> | |||
名古屋市立大学大学院薬学研究科''<br> | |||
DOI:<selfdoi /> 原稿受付日:2023年5月8日 原稿完成日:2023年5月24日<br> | |||
担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | |||
</div></div> | |||
英:cytoplasmic polyadenylation element binding protein<br> | |||
英略語:CPEB | |||
{{box|text= 細胞質ポリアデニル化要素結合タンパク質(cytoplasmic polyadenylation element binding protein, CPEB)はmRNA の3’UTRに存在するCPE配列(典型的には5’-UUUUAAU-3’)に結合し、ポリA鎖の長さを調節することで、翻訳(タンパク質合成)を制御するRNA結合タンパク質である。CPEBはもともとアフリカツメガエルの卵母細胞において母性mRNAの翻訳制御を介して減数分裂進行を制御する因子として知られていた。その後、CPEBは神経細胞においてシナプスへのmRNA輸送とシナプス刺激に応答した局所翻訳を制御することが明らかにされた。CPEBノックアウトマウスはシナプス可塑性や学習と記憶に異常を示すことからCPEBによるシナプス刺激に応答したmRNA輸送と局所翻訳は学習と記憶に重要な役割を担うと考えられる。}} | |||
== 細胞質ポリアデニル化要素結合タンパク質とは == | |||
[[アフリカツメガエル]]の[[卵母細胞]]では[[cyclinB1]]、[[サイクリン依存性キナーゼ2]] ([[cdk2]])、[[c-mos]]等の母性[[mRNA]]が、[[ポリA鎖]]が短い状態で保存されている。卵母細胞が[[プロジェステロン]]の刺激を受けると、ポリA鎖が伸長し、それらのmRNAの翻訳が促進されることで[[減数分裂]]が進行することが知られていた。これらのmRNAの[[3’UTR]]にはしばしばcytoplasmic polyadenylation element (CPE)配列(典型的には5’-UUUUAAU-3’)と呼ばれるシス配列が存在し、細胞質ポリアデニル化要素結合タンパク質([[cytoplasmic polyadenylation element binding protein]], [[CPEB]])はこのCPE配列に結合する配列特異的RNA結合タンパク質として同定された<ref name=Hake1994><pubmed>7954828</pubmed></ref><ref name=Stebbins-Boaz1996><pubmed>8665866</pubmed></ref>。 | |||
CPEBは卵母細胞の成熟過程だけでなく、[[精子]]形成にも必要とされることが明らかにされ<ref name=Luitjens2000><pubmed>11040214</pubmed></ref>、[[生殖細胞]]における重要な[[翻訳制御因子]]として、その分子制御機構の解明が進められてきた。 | |||
その後、CPEBは神経系においても高発現し、シナプス局所へのmRNA輸送とシナプス刺激に応じた特異的mRNAのポリA鎖伸長と翻訳促進を担うことが明らかにされ<ref name=Huang2002><pubmed>11980711</pubmed></ref><ref name=Huang2003><pubmed>12629046</pubmed></ref><ref name=Udagawa2012><pubmed>22727665</pubmed></ref>、さらに、[[シナプス可塑性]]や[[学習]]と[[記憶]]にもCPEBが重要な役割を担うことが示された<ref name=Alarcon2004><pubmed>15169862</pubmed></ref><ref name=Berger-Sweeney2006><pubmed>16452649</pubmed></ref>。CPEB(後に、[[CPEB1]]と遺伝子名が変更)とCPEBファミリータンパク質は、生殖系と神経系以外にも、[[細胞老化]]、[[がん]]、[[ストレス応答]]、[[器官形成]]などの様々な局面で働くことが現在までに報告されており、[[脊椎動物]]のmRNAの20-30%がCPEsを介した翻訳制御を受けると推定されている<ref name=Groisman2006><pubmed>17015432</pubmed></ref><ref name=Burns2011><pubmed>21478871</pubmed></ref><ref name=Nagaoka2012><pubmed>22334078</pubmed></ref><ref name=Davidson2016><pubmed>26947065</pubmed></ref><ref name=Calderone2016><pubmed>26627607</pubmed></ref><ref name=Maillo2017><pubmed>28092655</pubmed></ref><ref name=Pascual2020><pubmed>32440535</pubmed></ref><ref name=Pique2008><pubmed>18267074</pubmed></ref>。 | |||
[[ファイル:Udagawa CPEB Fig1.png|サムネイル|'''図1. CPEBファミリータンパク質の系統樹'''<br>MEGA11により作成したマウス、アフリカツメガエル、ショウジョウバエ、アメフラシのCPEBタンパク質の最尤法系統樹。CPEB 1サブファミリーにはマウスCPEB1(MmCPEB)、アフリカツメガエルCPEB1(XtCPEB)、ショウジョウバエOrb(DmOrb)、アメフラシCPEB(ApCPEB)が含まれ、それ以外のCPEBタンパク質はすべてCPEB2サブファミリーに含まれる。]] | |||
[[ファイル:Udagawa CPEB Fig2.png|サムネイル|'''図2. CPEBファミリータンパク質の一次構造'''<br>ヒトのCPEBファミリータンパク質CPEB1-4の一次構造。CPEB1のN末端側には翻訳抑制型から翻訳促進型への複合体の変換に必要なリン酸化セリン残基がある。CPEB2-4のN末端側には明確なドメインがなく、液-液相分離やプリオン様性質に必要な天然変性領域が含まれる。C末端側には全てのCPEBファミリータンパク質に保存された2つのRNA結合ドメイン(RRM1とRRM2)と2つのZnイオンが配位する配列(ZZ domain)が存在する。]] | |||
== サブファミリー == | |||
CPEBは後生動物に保存されているRNA結合タンパク質であり、大きく2つのサブファミリーに分かれる('''図1''')。 | |||
CPEB1サブファミリーには哺乳類およびアフリカツメガエルのCPEB1、[[ショウジョウバエ]]の[[Oo18 RNA-binding protein]] ([[Orb]])、[[アメフラシ]]の[[ApCPEB]]が含まれる。その他のCPEBファミリータンパク質(哺乳類およびアフリカツメガエルの[[CPEB2]]-[[CPEB4|4]], ショウジョウバエの[[Orb2]])はCPEB2サブファミリーに含まれる。CPEB1-4は20%程度の共通したmRNAを標的にする一方で、それぞれに特徴的な標的を持つ。CPEB1は典型的なCPE配列(5’-UUUUAAU-3’)に近い配列を持つmRNAを標的とする傾向があるのに対し、CPEB2-4はG置換を含む非典型的なCPE配列(5’-UUUUGUA-3’)を標的とする傾向がある。CPEB2-4の標的は大部分が共通している<ref name=Duran-Arque2022><pubmed>36096799</pubmed></ref>。 | |||
== 構造 == | |||
N末端側の配列はCPEB1-4の間で多様性がある('''図2''')。哺乳類、及びアフリカツメガエルのCPEB1のN末端領域の[[セリン]]残基の[[リン酸化]]はCPEB複合体の翻訳抑制型から翻訳促進型への変換に必要とされる<ref name=Mendez2000><pubmed>11106762</pubmed></ref><ref name=Kim2006><pubmed>17052452</pubmed></ref>。CPEB2-4、Orb2、及びアメフラシのApCPEBのN末端側には[[天然変性領域]]が含まれ、これらのタンパク質は[[液-液相分離]]、または[[プリオン]]様の性質を示す<ref name=Si2003><pubmed>14697205</pubmed></ref><ref name=Majumdar2012><pubmed>22284910</pubmed></ref><ref name=Guillen-Boixet2016><pubmed>27802129</pubmed></ref><ref name=Hervas2020><pubmed>32165583</pubmed></ref>。 | |||
すべてのCPEBファミリータンパク質のC末端側には2つのRNA結合ドメイン([[RRM1]]と[[RRM2]])が存在し、これは標的mRNAとの結合に必要とされる。また、C末端付近には[[ZZドメイン]]と呼ばれる2つの[[亜鉛]]イオンが配位する領域を持ち、CPEB複合体のタンパク質間、及びタンパク質-RNA間相互作用に必要とされる<ref name=Afroz2014><pubmed>24990967</pubmed></ref>。 | |||
== 発現 == | |||
アフリカツメガエルのCPEB1とショウジョウバエのOrbはもともと卵母細胞特異的なタンパク質として同定されたが、実際にはほぼ全身に発現していると思われる。哺乳類ではすべてのCPEBファミリータンパク質はほぼ全身に発現しており、特に、生殖系と神経系で高い発現を示す。 | |||
== 機能 == | |||
===卵母細胞成熟の制御=== | |||
CPEB1は元々、卵母細胞の成熟や精子形成といった発生の初期過程で重要な役割を果たすと考えられていた。特に卵母細胞の成熟過程では新たな転写が起こらず、もともと蓄えられていた母性mRNAのポリA鎖の長さの調節を介した翻訳制御が減数分裂の進行に中心的な役割を果たすことが知られており、これらのmRNAに結合するCPEB1によるポリA鎖制御の分子機構の解明が進められてきた。 | |||
アフリカツメガエルの卵母細胞におけるCPEB複合体因子として、[[デアデニレース]]([[ポリA短鎖化酵素]] ([[poly(A)-specific ribonuclease]], [[PARN]]))、[[細胞質型ポリAポリメラーゼ]] [[Germ Line Development 2]] ([[Gld2]])、mRNAの3’末端プロセッシング因子[[cleavage and polyadenylation specificity factor]] ([[CPSF]])タンパク質群、[[足場タンパク質]][[Symplekin]]、[[翻訳開始因子4E]] ([[elongation initiation factor 4E]], [[eIF4E]])結合翻訳抑制因子[[Maskin]]等が同定されている<ref name=Kim2006><pubmed>17052452</pubmed></ref><ref name=Stebbins-Boaz1999><pubmed>10635326</pubmed></ref><ref name=Barnard2004><pubmed>15550246</pubmed></ref>。CPEB1を含むいくつかのCPEB複合体因子はmRNAの3’末端近傍の[[ポリA付加シグナル]](5’-AAUAAA-3’)、及びその近く位置するCPE配列に[[核]]内で結合した後に、[[細胞質]]に輸送されると考えられる。 | |||
細胞質に輸送されたmRNAはCPEB複合体因子PARNの働きで、速やかにそのポリA鎖が短鎖化される<ref name=Kim2006><pubmed>17052452</pubmed></ref>。同時に、CPEB複合体はMaskinを介してeIF4Eに結合し、[[eIF4G]]のeIF4Eとの結合を妨げ、翻訳を抑制状態に維持する<ref name=Stebbins-Boaz1999><pubmed>10635326</pubmed></ref>。プロジェステロンの刺激により、[[Aurora Kinase]]がCPEB1をリン酸化すると、PARNは複合体から排出され、Gld2によるポリA鎖伸長が優勢となる<ref name=Kim2006><pubmed>17052452</pubmed></ref>。これにより、複数のポリA結合タンパク質 (poly(A)-binding proteins, PABP)が伸長したポリA鎖に結合し、PABP・eIF4G・eIF4Eの結合が促進され、翻訳開始複合体がmRNAの5’末端に形成される。 | |||
このようにして、CPEB1を含む細胞質ポリA鎖制御複合体は、cyclinB1、cdk2、c-mos等の減数分裂の進行を担う因子の発現のスイッチを時期特異的に切り替えると考えられる。 | |||
===シナプス可塑性の制御=== | |||
哺乳類神経細胞では、CPEBはCPE配列を含むmRNAの翻訳を抑制した状態で[[細胞体]]から[[樹状突起]]の遠位に輸送する働きを持つことが報告されている<ref name=Huang2003><pubmed>12629046</pubmed></ref>。哺乳類神経細胞におけるCPEB複合体は概ねアフリカツメガエルのそれと同様であり、Symplekinを足場タンパク質として、[[PAP associated domain containing 4]] ([[PAPD4]], Gld2の哺乳類ホモログ)、PARN、[[cleavage and polyadenylation specificity factor]] ([[CPSF]])が結合した複合体が[[マウス]][[脳]]、及び、[[初代培養神経細胞]]の樹状突起で顆粒状に検出され、輸送顆粒を形成していると考えられる<ref name=Udagawa2012 />。アフリカツメガエルで同定されたeIF4E結合タンパク質Maskinは哺乳類細胞には保存されていないが、同じくeIF4E結合タンパク質である[[Neuroguidin]]([[Ngd]])が哺乳類細胞におけるCPEB複合体因子として同定されており、Maskinと同様にCPEB複合体による翻訳抑制の機能を担うと考えられる<ref name=Jung2006><pubmed>16705177</pubmed></ref>。 | |||
CPEB1は[[NMDA型グルタミン酸受容体]]刺激に応じてAurora kinaseによりリン酸化され、CaMKIIαやNMDA型グルタミン酸受容体[[GluN2A]]サブユニット等のシナプス機能に重要な因子をコードするmRNAのポリA鎖を伸長し、翻訳を活性化することが示されている<ref name=Huang2002><pubmed>11980711</pubmed></ref><ref name=Udagawa2012><pubmed>22727665</pubmed></ref>。CPEB1[[ノックアウトマウス]]では学習と記憶の分子基盤となるタンパク質合成依存的[[長期増強]]([[LTP]])が阻害されており、幾つかの[[海馬]]依存的な[[学習]]と[[記憶]]の試験において異常が認められることから、CPEB複合体によるmRNA輸送と[[局所翻訳]]がシナプス可塑性や学習と記憶に重要な役割を果たすと考えられる<ref name=Alarcon2004 /><ref name=Berger-Sweeney2006 />。 | |||
一方、CPEB複合体において翻訳を促進する機能を果たす[[PAPD4]]の発現抑制はLTPを阻害するものの<ref name=Udagawa2012><pubmed>22727665</pubmed></ref>、PAPD4ノックアウトマウスは海馬依存的な学習と記憶の試験で異常を示さないことも報告されており<ref name=Mansur2016><pubmed>27495319</pubmed></ref>、CPEB複合体による学習と記憶の制御機構についてはまだ未解明な点も多い。 | |||
===プリオン様性質と液-液相分離=== | |||
アメフラシにおいてもCPEB(ApCPEB)による局所翻訳が感覚ニューロンのシナプス可塑性の一種である[[長期促通]](LTF)に必要とされることが報告されている<ref name=Si2003b><pubmed>14697206</pubmed></ref>。興味深いことに、ApCPEBはN末端側にタンパク質凝集体を形成するプリオン様の性質を持ち、凝集体型のApCPEBが翻訳を活性化する機能を持つことが示されている<ref name=Si2010><pubmed>20144764</pubmed></ref>。同様に、ショウジョウバエのOrb2もまたプリオン様の性質を持ち、Orb2のオリゴマーがポリAの長鎖化と翻訳の促進を担う機能を持ち、Orb2のモノマーはポリAを短鎖化させ、翻訳を抑制することが報告されている<ref name=Majumdar2012><pubmed>22284910</pubmed></ref><ref name=Khan2015><pubmed>26638074</pubmed></ref>。いずれの場合にもオリゴマー形成が長期記憶の維持に重要であるとされている。 | |||
しかしながら、プリオンドメインが別のタンパク質との相互作用を介して機能している可能性もあり、プリオン仮説についてはさらなる検証が必要と考えられる。Orb2によるオリゴマー化を介したポリA鎖制御機構についても慎重な検証が必要である。なお、哺乳類のCPEB1ではプリオン様の性質は示されておらず、哺乳類CPEB1による翻訳制御と学習と記憶の制御機構がアメフラシのApCPEBによるポリA鎖制御機構と同様であるかは明らかでない。 | |||
一方、哺乳類のCPEB2-4はN末端ドメインを介して液-液相分離による液滴を形成し、その機能に重要な役割を果たす可能性が指摘されている<ref name=Guillen-Boixet2016><pubmed>27802129</pubmed></ref>。CPEB4はN末端の天然変性領域を介して液-液相分離による液滴を形成するが、細胞周期の[[M期]]において[[Cdk1]]および[[Erk2]]によりN末端ドメインが過リン酸化されるとモノマー型に変換され活性化されることが報告されている<ref name=Guillen-Boixet2016><pubmed>27802129</pubmed></ref>。CPEB2-4による翻訳制御機構の詳細はまだ明らかでないが、[[CPEB3]]ノックアウトマウスは海馬依存的な学習と記憶を亢進させることが報告されている<ref name=Chao2013><pubmed>24155305</pubmed></ref>。 | |||
===その他=== | |||
哺乳類のCPEB1を含むすべてのCPEBファミリータンパク質は体細胞においてPARNではなく、[[デアデニース複合体]][[CCR4-NOT]]をリクルートしmRNAの不安定化に関わることも報告されている<ref name=Ogami2014><pubmed>23178487</pubmed></ref>。一方、その制御機構はCPEB1とCPEB2-4で異なっているらしい。CPEB1は上述のようにN末端ドメインの単一アミノ酸残基のリン酸化で複合体の構成と翻訳抑制型・翻訳活性化型の変換が制御されているが、CPEB2-4ではN末端ドメインの複数の[[プロリン]]残基のリン酸化がタンパク質の液-液相分離を制御し、それが標的mRNAの制御に重要であるとされている<ref name=Guillen-Boixet2016><pubmed>27802129</pubmed></ref>。 | |||
==疾患との関わり== | |||
===脆弱X精神遅滞症候群=== | |||
CPEBタンパク質が特定のヒト疾患の原因因子として報告された例はこれまでのところないが、いくつかの疾患との関連が報告されている。CPEB1は[[脆弱X精神遅滞症候群]](FXS)の原因因子であるRNA結合タンパク質[[fragile X mental retardation protein]] ([[FMRP]])と相互作用する<ref name=Udagawa2013><pubmed>24141422</pubmed></ref>。FXSはFMRPの機能欠失により発症する。FMRPは翻訳抑制因子として機能し、FMRPノックアウトマウスの脳や、FXS患者由来の細胞ではタンパク質合成が亢進していることが報告されており、これが病態の一つと考えられている<ref name=Darnell2013><pubmed>23584741</pubmed></ref>。FMRPはシナプス刺激に応答した翻訳の過剰な活性化を抑える働きを持つと考えられている。一方CPEBはFMRPとは逆にシナプス刺激に応答して翻訳を促進する機能を持つ。FMRPとCPEB1は多くの共通した標的mRNAを持つことから両者が共通した標的mRNA発現のバランスを調整していると考えられる。実際、FMRPと反対の働きをするCPEB1の発現をFMRPノックアウトマウスにおいて抑制することにより、亢進したタンパク質合成レベルが正常レベルに低下し、FXS病態が改善されることが示されている<ref name=Udagawa2013 />。 | |||
===ハンチントン病=== | |||
[[ハンチントン病]]患者やマウスモデルの[[線条体]]ではCPEB1の発現が上昇し、CPEB4の発現が低下していることが報告されている<ref name=Pico2021><pubmed>34586830</pubmed></ref>。この発現変化により全転写産物の17.3%のポリA鎖長が変化し、多くの[[神経変性疾患]]関連因子の発現が変動していることが明らかにされた。興味深いことに、このCPEB1 ,4の発現変化により別の神経疾患である[[ビオチン]]-[[チアミン]]応答性[[大脳基底核]]疾患の原因となる[[SLC19C]]の発現が低下することが見出された。さらに、ハンチントン病マウスモデルにチアミンピロリン酸を処理することにより一部のハンチントン病様表現系が改善されることが明らかにされた<ref name=Pico2021 />。 | |||
===その他=== | |||
CPEB1は生殖細胞と神経細胞以外の体細胞にも発現し、[[がん原遺伝子]][[myc]]や[[がん抑制遺伝子]][[p53]]の制御を介した[[細胞老化]]の制御<ref name=Groisman2006><pubmed>17015432</pubmed></ref><ref name=Burns2011><pubmed>21478871</pubmed></ref>、mRNAの局在化を介した[[細胞極性]]の形成<ref name=Nagaoka2012><pubmed>22334078</pubmed></ref>、[[器官形成]]等にも関わることが報告されている<ref name=Calderone2016><pubmed>26627607</pubmed></ref><ref name=Zeng2022><pubmed>35177647</pubmed></ref>。また、その他のCPEBファミリータンパク質と[[がん]]、[[肝]]機能障害、神経機能障害との関わりについても報告されている<ref name=Calderone2016><pubmed>26627607</pubmed></ref><ref name=Pascual2020><pubmed>32440535</pubmed></ref><ref name=Chao2013><pubmed>24155305</pubmed></ref><ref name=Villanueva2017><pubmed>28300077</pubmed></ref><ref name=Huang2014><pubmed>25404896</pubmed></ref>。 | |||
== 関連語 == | |||
* [[RNA結合タンパク質]] | |||
* [[プリオン]] | |||
* [[fragile X mental retardation protein]] | |||
== 参考文献 == | |||
<references /> | |||
2023年5月26日 (金) 08:09時点における最新版
宇田川 剛
名古屋市立大学大学院薬学研究科
DOI:10.14931/bsd.10326 原稿受付日:2023年5月8日 原稿完成日:2023年5月24日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
英:cytoplasmic polyadenylation element binding protein
英略語:CPEB
細胞質ポリアデニル化要素結合タンパク質(cytoplasmic polyadenylation element binding protein, CPEB)はmRNA の3’UTRに存在するCPE配列(典型的には5’-UUUUAAU-3’)に結合し、ポリA鎖の長さを調節することで、翻訳(タンパク質合成)を制御するRNA結合タンパク質である。CPEBはもともとアフリカツメガエルの卵母細胞において母性mRNAの翻訳制御を介して減数分裂進行を制御する因子として知られていた。その後、CPEBは神経細胞においてシナプスへのmRNA輸送とシナプス刺激に応答した局所翻訳を制御することが明らかにされた。CPEBノックアウトマウスはシナプス可塑性や学習と記憶に異常を示すことからCPEBによるシナプス刺激に応答したmRNA輸送と局所翻訳は学習と記憶に重要な役割を担うと考えられる。
細胞質ポリアデニル化要素結合タンパク質とは
アフリカツメガエルの卵母細胞ではcyclinB1、サイクリン依存性キナーゼ2 (cdk2)、c-mos等の母性mRNAが、ポリA鎖が短い状態で保存されている。卵母細胞がプロジェステロンの刺激を受けると、ポリA鎖が伸長し、それらのmRNAの翻訳が促進されることで減数分裂が進行することが知られていた。これらのmRNAの3’UTRにはしばしばcytoplasmic polyadenylation element (CPE)配列(典型的には5’-UUUUAAU-3’)と呼ばれるシス配列が存在し、細胞質ポリアデニル化要素結合タンパク質(cytoplasmic polyadenylation element binding protein, CPEB)はこのCPE配列に結合する配列特異的RNA結合タンパク質として同定された[1][2]。
CPEBは卵母細胞の成熟過程だけでなく、精子形成にも必要とされることが明らかにされ[3]、生殖細胞における重要な翻訳制御因子として、その分子制御機構の解明が進められてきた。
その後、CPEBは神経系においても高発現し、シナプス局所へのmRNA輸送とシナプス刺激に応じた特異的mRNAのポリA鎖伸長と翻訳促進を担うことが明らかにされ[4][5][6]、さらに、シナプス可塑性や学習と記憶にもCPEBが重要な役割を担うことが示された[7][8]。CPEB(後に、CPEB1と遺伝子名が変更)とCPEBファミリータンパク質は、生殖系と神経系以外にも、細胞老化、がん、ストレス応答、器官形成などの様々な局面で働くことが現在までに報告されており、脊椎動物のmRNAの20-30%がCPEsを介した翻訳制御を受けると推定されている[9][10][11][12][13][14][15][16]。

MEGA11により作成したマウス、アフリカツメガエル、ショウジョウバエ、アメフラシのCPEBタンパク質の最尤法系統樹。CPEB 1サブファミリーにはマウスCPEB1(MmCPEB)、アフリカツメガエルCPEB1(XtCPEB)、ショウジョウバエOrb(DmOrb)、アメフラシCPEB(ApCPEB)が含まれ、それ以外のCPEBタンパク質はすべてCPEB2サブファミリーに含まれる。
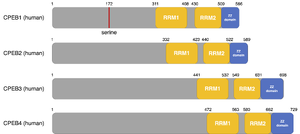
ヒトのCPEBファミリータンパク質CPEB1-4の一次構造。CPEB1のN末端側には翻訳抑制型から翻訳促進型への複合体の変換に必要なリン酸化セリン残基がある。CPEB2-4のN末端側には明確なドメインがなく、液-液相分離やプリオン様性質に必要な天然変性領域が含まれる。C末端側には全てのCPEBファミリータンパク質に保存された2つのRNA結合ドメイン(RRM1とRRM2)と2つのZnイオンが配位する配列(ZZ domain)が存在する。
サブファミリー
CPEBは後生動物に保存されているRNA結合タンパク質であり、大きく2つのサブファミリーに分かれる(図1)。
CPEB1サブファミリーには哺乳類およびアフリカツメガエルのCPEB1、ショウジョウバエのOo18 RNA-binding protein (Orb)、アメフラシのApCPEBが含まれる。その他のCPEBファミリータンパク質(哺乳類およびアフリカツメガエルのCPEB2-4, ショウジョウバエのOrb2)はCPEB2サブファミリーに含まれる。CPEB1-4は20%程度の共通したmRNAを標的にする一方で、それぞれに特徴的な標的を持つ。CPEB1は典型的なCPE配列(5’-UUUUAAU-3’)に近い配列を持つmRNAを標的とする傾向があるのに対し、CPEB2-4はG置換を含む非典型的なCPE配列(5’-UUUUGUA-3’)を標的とする傾向がある。CPEB2-4の標的は大部分が共通している[17]。
構造
N末端側の配列はCPEB1-4の間で多様性がある(図2)。哺乳類、及びアフリカツメガエルのCPEB1のN末端領域のセリン残基のリン酸化はCPEB複合体の翻訳抑制型から翻訳促進型への変換に必要とされる[18][19]。CPEB2-4、Orb2、及びアメフラシのApCPEBのN末端側には天然変性領域が含まれ、これらのタンパク質は液-液相分離、またはプリオン様の性質を示す[20][21][22][23]。
すべてのCPEBファミリータンパク質のC末端側には2つのRNA結合ドメイン(RRM1とRRM2)が存在し、これは標的mRNAとの結合に必要とされる。また、C末端付近にはZZドメインと呼ばれる2つの亜鉛イオンが配位する領域を持ち、CPEB複合体のタンパク質間、及びタンパク質-RNA間相互作用に必要とされる[24]。
発現
アフリカツメガエルのCPEB1とショウジョウバエのOrbはもともと卵母細胞特異的なタンパク質として同定されたが、実際にはほぼ全身に発現していると思われる。哺乳類ではすべてのCPEBファミリータンパク質はほぼ全身に発現しており、特に、生殖系と神経系で高い発現を示す。
機能
卵母細胞成熟の制御
CPEB1は元々、卵母細胞の成熟や精子形成といった発生の初期過程で重要な役割を果たすと考えられていた。特に卵母細胞の成熟過程では新たな転写が起こらず、もともと蓄えられていた母性mRNAのポリA鎖の長さの調節を介した翻訳制御が減数分裂の進行に中心的な役割を果たすことが知られており、これらのmRNAに結合するCPEB1によるポリA鎖制御の分子機構の解明が進められてきた。
アフリカツメガエルの卵母細胞におけるCPEB複合体因子として、デアデニレース(ポリA短鎖化酵素 (poly(A)-specific ribonuclease, PARN))、細胞質型ポリAポリメラーゼ Germ Line Development 2 (Gld2)、mRNAの3’末端プロセッシング因子cleavage and polyadenylation specificity factor (CPSF)タンパク質群、足場タンパク質Symplekin、翻訳開始因子4E (elongation initiation factor 4E, eIF4E)結合翻訳抑制因子Maskin等が同定されている[19][25][26]。CPEB1を含むいくつかのCPEB複合体因子はmRNAの3’末端近傍のポリA付加シグナル(5’-AAUAAA-3’)、及びその近く位置するCPE配列に核内で結合した後に、細胞質に輸送されると考えられる。
細胞質に輸送されたmRNAはCPEB複合体因子PARNの働きで、速やかにそのポリA鎖が短鎖化される[19]。同時に、CPEB複合体はMaskinを介してeIF4Eに結合し、eIF4GのeIF4Eとの結合を妨げ、翻訳を抑制状態に維持する[25]。プロジェステロンの刺激により、Aurora KinaseがCPEB1をリン酸化すると、PARNは複合体から排出され、Gld2によるポリA鎖伸長が優勢となる[19]。これにより、複数のポリA結合タンパク質 (poly(A)-binding proteins, PABP)が伸長したポリA鎖に結合し、PABP・eIF4G・eIF4Eの結合が促進され、翻訳開始複合体がmRNAの5’末端に形成される。
このようにして、CPEB1を含む細胞質ポリA鎖制御複合体は、cyclinB1、cdk2、c-mos等の減数分裂の進行を担う因子の発現のスイッチを時期特異的に切り替えると考えられる。
シナプス可塑性の制御
哺乳類神経細胞では、CPEBはCPE配列を含むmRNAの翻訳を抑制した状態で細胞体から樹状突起の遠位に輸送する働きを持つことが報告されている[5]。哺乳類神経細胞におけるCPEB複合体は概ねアフリカツメガエルのそれと同様であり、Symplekinを足場タンパク質として、PAP associated domain containing 4 (PAPD4, Gld2の哺乳類ホモログ)、PARN、cleavage and polyadenylation specificity factor (CPSF)が結合した複合体がマウス脳、及び、初代培養神経細胞の樹状突起で顆粒状に検出され、輸送顆粒を形成していると考えられる[6]。アフリカツメガエルで同定されたeIF4E結合タンパク質Maskinは哺乳類細胞には保存されていないが、同じくeIF4E結合タンパク質であるNeuroguidin(Ngd)が哺乳類細胞におけるCPEB複合体因子として同定されており、Maskinと同様にCPEB複合体による翻訳抑制の機能を担うと考えられる[27]。
CPEB1はNMDA型グルタミン酸受容体刺激に応じてAurora kinaseによりリン酸化され、CaMKIIαやNMDA型グルタミン酸受容体GluN2Aサブユニット等のシナプス機能に重要な因子をコードするmRNAのポリA鎖を伸長し、翻訳を活性化することが示されている[4][6]。CPEB1ノックアウトマウスでは学習と記憶の分子基盤となるタンパク質合成依存的長期増強(LTP)が阻害されており、幾つかの海馬依存的な学習と記憶の試験において異常が認められることから、CPEB複合体によるmRNA輸送と局所翻訳がシナプス可塑性や学習と記憶に重要な役割を果たすと考えられる[7][8]。
一方、CPEB複合体において翻訳を促進する機能を果たすPAPD4の発現抑制はLTPを阻害するものの[6]、PAPD4ノックアウトマウスは海馬依存的な学習と記憶の試験で異常を示さないことも報告されており[28]、CPEB複合体による学習と記憶の制御機構についてはまだ未解明な点も多い。
プリオン様性質と液-液相分離
アメフラシにおいてもCPEB(ApCPEB)による局所翻訳が感覚ニューロンのシナプス可塑性の一種である長期促通(LTF)に必要とされることが報告されている[29]。興味深いことに、ApCPEBはN末端側にタンパク質凝集体を形成するプリオン様の性質を持ち、凝集体型のApCPEBが翻訳を活性化する機能を持つことが示されている[30]。同様に、ショウジョウバエのOrb2もまたプリオン様の性質を持ち、Orb2のオリゴマーがポリAの長鎖化と翻訳の促進を担う機能を持ち、Orb2のモノマーはポリAを短鎖化させ、翻訳を抑制することが報告されている[21][31]。いずれの場合にもオリゴマー形成が長期記憶の維持に重要であるとされている。
しかしながら、プリオンドメインが別のタンパク質との相互作用を介して機能している可能性もあり、プリオン仮説についてはさらなる検証が必要と考えられる。Orb2によるオリゴマー化を介したポリA鎖制御機構についても慎重な検証が必要である。なお、哺乳類のCPEB1ではプリオン様の性質は示されておらず、哺乳類CPEB1による翻訳制御と学習と記憶の制御機構がアメフラシのApCPEBによるポリA鎖制御機構と同様であるかは明らかでない。
一方、哺乳類のCPEB2-4はN末端ドメインを介して液-液相分離による液滴を形成し、その機能に重要な役割を果たす可能性が指摘されている[22]。CPEB4はN末端の天然変性領域を介して液-液相分離による液滴を形成するが、細胞周期のM期においてCdk1およびErk2によりN末端ドメインが過リン酸化されるとモノマー型に変換され活性化されることが報告されている[22]。CPEB2-4による翻訳制御機構の詳細はまだ明らかでないが、CPEB3ノックアウトマウスは海馬依存的な学習と記憶を亢進させることが報告されている[32]。
その他
哺乳類のCPEB1を含むすべてのCPEBファミリータンパク質は体細胞においてPARNではなく、デアデニース複合体CCR4-NOTをリクルートしmRNAの不安定化に関わることも報告されている[33]。一方、その制御機構はCPEB1とCPEB2-4で異なっているらしい。CPEB1は上述のようにN末端ドメインの単一アミノ酸残基のリン酸化で複合体の構成と翻訳抑制型・翻訳活性化型の変換が制御されているが、CPEB2-4ではN末端ドメインの複数のプロリン残基のリン酸化がタンパク質の液-液相分離を制御し、それが標的mRNAの制御に重要であるとされている[22]。
疾患との関わり
脆弱X精神遅滞症候群
CPEBタンパク質が特定のヒト疾患の原因因子として報告された例はこれまでのところないが、いくつかの疾患との関連が報告されている。CPEB1は脆弱X精神遅滞症候群(FXS)の原因因子であるRNA結合タンパク質fragile X mental retardation protein (FMRP)と相互作用する[34]。FXSはFMRPの機能欠失により発症する。FMRPは翻訳抑制因子として機能し、FMRPノックアウトマウスの脳や、FXS患者由来の細胞ではタンパク質合成が亢進していることが報告されており、これが病態の一つと考えられている[35]。FMRPはシナプス刺激に応答した翻訳の過剰な活性化を抑える働きを持つと考えられている。一方CPEBはFMRPとは逆にシナプス刺激に応答して翻訳を促進する機能を持つ。FMRPとCPEB1は多くの共通した標的mRNAを持つことから両者が共通した標的mRNA発現のバランスを調整していると考えられる。実際、FMRPと反対の働きをするCPEB1の発現をFMRPノックアウトマウスにおいて抑制することにより、亢進したタンパク質合成レベルが正常レベルに低下し、FXS病態が改善されることが示されている[34]。
ハンチントン病
ハンチントン病患者やマウスモデルの線条体ではCPEB1の発現が上昇し、CPEB4の発現が低下していることが報告されている[36]。この発現変化により全転写産物の17.3%のポリA鎖長が変化し、多くの神経変性疾患関連因子の発現が変動していることが明らかにされた。興味深いことに、このCPEB1 ,4の発現変化により別の神経疾患であるビオチン-チアミン応答性大脳基底核疾患の原因となるSLC19Cの発現が低下することが見出された。さらに、ハンチントン病マウスモデルにチアミンピロリン酸を処理することにより一部のハンチントン病様表現系が改善されることが明らかにされた[36]。
その他
CPEB1は生殖細胞と神経細胞以外の体細胞にも発現し、がん原遺伝子mycやがん抑制遺伝子p53の制御を介した細胞老化の制御[9][10]、mRNAの局在化を介した細胞極性の形成[11]、器官形成等にも関わることが報告されている[13][37]。また、その他のCPEBファミリータンパク質とがん、肝機能障害、神経機能障害との関わりについても報告されている[13][15][32][38][39]。
関連語
参考文献
- ↑
Hake, L.E., & Richter, J.D. (1994).
CPEB is a specificity factor that mediates cytoplasmic polyadenylation during Xenopus oocyte maturation. Cell, 79(4), 617-27. [PubMed:7954828] [WorldCat] [DOI] - ↑
Stebbins-Boaz, B., Hake, L.E., & Richter, J.D. (1996).
CPEB controls the cytoplasmic polyadenylation of cyclin, Cdk2 and c-mos mRNAs and is necessary for oocyte maturation in Xenopus. The EMBO journal, 15(10), 2582-92. [PubMed:8665866] [PMC] [WorldCat] - ↑
Luitjens, C., Gallegos, M., Kraemer, B., Kimble, J., & Wickens, M. (2000).
CPEB proteins control two key steps in spermatogenesis in C. elegans. Genes & development, 14(20), 2596-609. [PubMed:11040214] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1
Huang, Y.S., Jung, M.Y., Sarkissian, M., & Richter, J.D. (2002).
N-methyl-D-aspartate receptor signaling results in Aurora kinase-catalyzed CPEB phosphorylation and alpha CaMKII mRNA polyadenylation at synapses. The EMBO journal, 21(9), 2139-48. [PubMed:11980711] [PMC] [WorldCat] [DOI] - ↑ 5.0 5.1
Huang, Y.S., Carson, J.H., Barbarese, E., & Richter, J.D. (2003).
Facilitation of dendritic mRNA transport by CPEB. Genes & development, 17(5), 638-53. [PubMed:12629046] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2 6.3
Udagawa, T., Swanger, S.A., Takeuchi, K., Kim, J.H., Nalavadi, V., Shin, J., ..., & Richter, J.D. (2012).
Bidirectional control of mRNA translation and synaptic plasticity by the cytoplasmic polyadenylation complex. Molecular cell, 47(2), 253-66. [PubMed:22727665] [PMC] [WorldCat] [DOI] - ↑ 7.0 7.1
Alarcon, J.M., Hodgman, R., Theis, M., Huang, Y.S., Kandel, E.R., & Richter, J.D. (2004).
Selective modulation of some forms of schaffer collateral-CA1 synaptic plasticity in mice with a disruption of the CPEB-1 gene. Learning & memory (Cold Spring Harbor, N.Y.), 11(3), 318-27. [PubMed:15169862] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1
Berger-Sweeney, J., Zearfoss, N.R., & Richter, J.D. (2006).
Reduced extinction of hippocampal-dependent memories in CPEB knockout mice. Learning & memory (Cold Spring Harbor, N.Y.), 13(1), 4-7. [PubMed:16452649] [WorldCat] [DOI] - ↑ 9.0 9.1
Groisman, I., Ivshina, M., Marin, V., Kennedy, N.J., Davis, R.J., & Richter, J.D. (2006).
Control of cellular senescence by CPEB. Genes & development, 20(19), 2701-12. [PubMed:17015432] [PMC] [WorldCat] [DOI] - ↑ 10.0 10.1
Burns, D.M., D'Ambrogio, A., Nottrott, S., & Richter, J.D. (2011).
CPEB and two poly(A) polymerases control miR-122 stability and p53 mRNA translation. Nature, 473(7345), 105-8. [PubMed:21478871] [PMC] [WorldCat] [DOI] - ↑ 11.0 11.1
Nagaoka, K., Udagawa, T., & Richter, J.D. (2012).
CPEB-mediated ZO-1 mRNA localization is required for epithelial tight-junction assembly and cell polarity. Nature communications, 3, 675. [PubMed:22334078] [PMC] [WorldCat] [DOI] - ↑
Davidson, A., Parton, R.M., Rabouille, C., Weil, T.T., & Davis, I. (2016).
Localized Translation of gurken/TGF-α mRNA during Axis Specification Is Controlled by Access to Orb/CPEB on Processing Bodies. Cell reports, 14(10), 2451-62. [PubMed:26947065] [PMC] [WorldCat] [DOI] - ↑ 13.0 13.1 13.2
Calderone, V., Gallego, J., Fernandez-Miranda, G., Garcia-Pras, E., Maillo, C., Berzigotti, A., ..., & Mendez, R. (2016).
Sequential Functions of CPEB1 and CPEB4 Regulate Pathologic Expression of Vascular Endothelial Growth Factor and Angiogenesis in Chronic Liver Disease. Gastroenterology, 150(4), 982-97.e30. [PubMed:26627607] [WorldCat] [DOI] - ↑
Maillo, C., Martín, J., Sebastián, D., Hernández-Alvarez, M., García-Rocha, M., Reina, O., ..., & Méndez, R. (2017).
Circadian- and UPR-dependent control of CPEB4 mediates a translational response to counteract hepatic steatosis under ER stress. Nature cell biology, 19(2), 94-105. [PubMed:28092655] [WorldCat] [DOI] - ↑ 15.0 15.1
Pascual, R., Martín, J., Salvador, F., Reina, O., Chanes, V., Millanes-Romero, A., ..., & Méndez, R. (2020).
The RNA binding protein CPEB2 regulates hormone sensing in mammary gland development and luminal breast cancer. Science advances, 6(20), eaax3868. [PubMed:32440535] [PMC] [WorldCat] [DOI] - ↑
Piqué, M., López, J.M., Foissac, S., Guigó, R., & Méndez, R. (2008).
A combinatorial code for CPE-mediated translational control. Cell, 132(3), 434-48. [PubMed:18267074] [WorldCat] [DOI] - ↑
Duran-Arqué, B., Cañete, M., Castellazzi, C.L., Bartomeu, A., Ferrer-Caelles, A., Reina, O., ..., & Mendez, R. (2022).
Comparative analyses of vertebrate CPEB proteins define two subfamilies with coordinated yet distinct functions in post-transcriptional gene regulation. Genome biology, 23(1), 192. [PubMed:36096799] [PMC] [WorldCat] [DOI] - ↑
Mendez, R., Murthy, K.G., Ryan, K., Manley, J.L., & Richter, J.D. (2000).
Phosphorylation of CPEB by Eg2 mediates the recruitment of CPSF into an active cytoplasmic polyadenylation complex. Molecular cell, 6(5), 1253-9. [PubMed:11106762] [WorldCat] [DOI] - ↑ 19.0 19.1 19.2 19.3
Kim, J.H., & Richter, J.D. (2006).
Opposing polymerase-deadenylase activities regulate cytoplasmic polyadenylation. Molecular cell, 24(2), 173-83. [PubMed:17052452] [WorldCat] [DOI] - ↑
Si, K., Lindquist, S., & Kandel, E.R. (2003).
A neuronal isoform of the aplysia CPEB has prion-like properties. Cell, 115(7), 879-91. [PubMed:14697205] [WorldCat] [DOI] - ↑ 21.0 21.1
Majumdar, A., Cesario, W.C., White-Grindley, E., Jiang, H., Ren, F., Khan, M.R., ..., & Si, K. (2012).
Critical role of amyloid-like oligomers of Drosophila Orb2 in the persistence of memory. Cell, 148(3), 515-29. [PubMed:22284910] [WorldCat] [DOI] - ↑ 22.0 22.1 22.2 22.3
Guillén-Boixet, J., Buzon, V., Salvatella, X., & Méndez, R. (2016).
CPEB4 is regulated during cell cycle by ERK2/Cdk1-mediated phosphorylation and its assembly into liquid-like droplets. eLife, 5. [PubMed:27802129] [PMC] [WorldCat] [DOI] - ↑
Hervas, R., Rau, M.J., Park, Y., Zhang, W., Murzin, A.G., Fitzpatrick, J.A.J., ..., & Si, K. (2020).
Cryo-EM structure of a neuronal functional amyloid implicated in memory persistence in Drosophila. Science (New York, N.Y.), 367(6483), 1230-1234. [PubMed:32165583] [PMC] [WorldCat] [DOI] - ↑
Afroz, T., Skrisovska, L., Belloc, E., Guillén-Boixet, J., Méndez, R., & Allain, F.H. (2014).
A fly trap mechanism provides sequence-specific RNA recognition by CPEB proteins. Genes & development, 28(13), 1498-514. [PubMed:24990967] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1
Stebbins-Boaz, B., Cao, Q., de Moor, C.H., Mendez, R., & Richter, J.D. (1999).
Maskin is a CPEB-associated factor that transiently interacts with elF-4E. Molecular cell, 4(6), 1017-27. [PubMed:10635326] [WorldCat] [DOI] - ↑
Barnard, D.C., Ryan, K., Manley, J.L., & Richter, J.D. (2004).
Symplekin and xGLD-2 are required for CPEB-mediated cytoplasmic polyadenylation. Cell, 119(5), 641-51. [PubMed:15550246] [WorldCat] [DOI] - ↑
Jung, M.Y., Lorenz, L., & Richter, J.D. (2006).
Translational control by neuroguidin, a eukaryotic initiation factor 4E and CPEB binding protein. Molecular and cellular biology, 26(11), 4277-87. [PubMed:16705177] [PMC] [WorldCat] [DOI] - ↑
Mansur, F., Ivshina, M., Gu, W., Schaevitz, L., Stackpole, E., Gujja, S., ..., & Richter, J.D. (2016).
Gld2-catalyzed 3' monoadenylation of miRNAs in the hippocampus has no detectable effect on their stability or on animal behavior. RNA (New York, N.Y.), 22(10), 1492-9. [PubMed:27495319] [PMC] [WorldCat] [DOI] - ↑
Si, K., Giustetto, M., Etkin, A., Hsu, R., Janisiewicz, A.M., Miniaci, M.C., ..., & Kandel, E.R. (2003).
A neuronal isoform of CPEB regulates local protein synthesis and stabilizes synapse-specific long-term facilitation in aplysia. Cell, 115(7), 893-904. [PubMed:14697206] [WorldCat] [DOI] - ↑
Si, K., Choi, Y.B., White-Grindley, E., Majumdar, A., & Kandel, E.R. (2010).
Aplysia CPEB can form prion-like multimers in sensory neurons that contribute to long-term facilitation. Cell, 140(3), 421-35. [PubMed:20144764] [WorldCat] [DOI] - ↑
Khan, M.R., Li, L., Pérez-Sánchez, C., Saraf, A., Florens, L., Slaughter, B.D., ..., & Si, K. (2015).
Amyloidogenic Oligomerization Transforms Drosophila Orb2 from a Translation Repressor to an Activator. Cell, 163(6), 1468-83. [PubMed:26638074] [PMC] [WorldCat] [DOI] - ↑ 32.0 32.1
Chao, H.W., Tsai, L.Y., Lu, Y.L., Lin, P.Y., Huang, W.H., Chou, H.J., ..., & Huang, Y.S. (2013).
Deletion of CPEB3 enhances hippocampus-dependent memory via increasing expressions of PSD95 and NMDA receptors. The Journal of neuroscience : the official journal of the Society for Neuroscience, 33(43), 17008-22. [PubMed:24155305] [PMC] [WorldCat] [DOI] - ↑
Ogami, K., Hosoda, N., Funakoshi, Y., & Hoshino, S. (2014).
Antiproliferative protein Tob directly regulates c-myc proto-oncogene expression through cytoplasmic polyadenylation element-binding protein CPEB. Oncogene, 33(1), 55-64. [PubMed:23178487] [WorldCat] [DOI] - ↑ 34.0 34.1
Udagawa, T., Farny, N.G., Jakovcevski, M., Kaphzan, H., Alarcon, J.M., Anilkumar, S., ..., & Richter, J.D. (2013).
Genetic and acute CPEB1 depletion ameliorate fragile X pathophysiology. Nature medicine, 19(11), 1473-7. [PubMed:24141422] [PMC] [WorldCat] [DOI] - ↑
Darnell, J.C., & Klann, E. (2013).
The translation of translational control by FMRP: therapeutic targets for FXS. Nature neuroscience, 16(11), 1530-6. [PubMed:23584741] [PMC] [WorldCat] [DOI] - ↑ 36.0 36.1
Picó, S., Parras, A., Santos-Galindo, M., Pose-Utrilla, J., Castro, M., Fraga, E., ..., & Lucas, J.J. (2021).
CPEB alteration and aberrant transcriptome-polyadenylation lead to a treatable SLC19A3 deficiency in Huntington's disease. Science translational medicine, 13(613), eabe7104. [PubMed:34586830] [WorldCat] [DOI] - ↑
Zeng, W., Yue, L., Lam, K.S.W., Zhang, W., So, W.K., Tse, E.H.Y., & Cheung, T.H. (2022).
CPEB1 directs muscle stem cell activation by reprogramming the translational landscape. Nature communications, 13(1), 947. [PubMed:35177647] [PMC] [WorldCat] [DOI] - ↑
Villanueva, E., Navarro, P., Rovira-Rigau, M., Sibilio, A., Méndez, R., & Fillat, C. (2017).
Translational reprogramming in tumour cells can generate oncoselectivity in viral therapies. Nature communications, 8, 14833. [PubMed:28300077] [PMC] [WorldCat] [DOI] - ↑
Huang, W.H., Chao, H.W., Tsai, L.Y., Chung, M.H., & Huang, Y.S. (2014).
Elevated activation of CaMKIIα in the CPEB3-knockout hippocampus impairs a specific form of NMDAR-dependent synaptic depotentiation. Frontiers in cellular neuroscience, 8, 367. [PubMed:25404896] [PMC] [WorldCat] [DOI]