「知的障害関連遺伝子」の版間の差分
細編集の要約なし |
|||
| (4人の利用者による、間の20版が非表示) | |||
| 1行目: | 1行目: | ||
英語名: Human genes in relation to Intellectual Disability | 英語名: Human genes in relation to Intellectual Disability | ||
英語略名: ID genes | 英語略名: ID genes | ||
[[知的障害]](Intellectual Disability、以下IDと称す)は全人口のうち約2-3%に認めるといわれ、特徴的な身体所見や検査所見を伴う症候性IDと、ID以外に付随症状を伴わない非症候性IDに分類される。IDのうち約50-60%で遺伝的原因が解明、それらは[[wikipedia:ja:染色体異常|染色体異常]]、[[wikipedia:ja:ゲノム構造異常|ゲノム構造異常]]、単一遺伝子異常など多岐にわたる。[[マイクロアレイ]]や[[wikipedia:DNA_sequencing#High-throughput_sequencing|次世代シーケンサー]]など[[wikipedia:ja:分子遺伝学|分子遺伝学]]検査の急速な進歩・普及により、現在ID関連遺伝子は約450遺伝子以上も報告され、そのうち症候性IDの原因として約400遺伝子、非症候性IDの原因として約50遺伝子が認知されている。また[[wikipedia:ja:X連鎖性遺伝子|X連鎖性遺伝子]]は約22%(約100遺伝子)で、男性にIDの頻度が高い要因となっている。非症候性IDに対する[[wikipedia:Exome sequencing|エクソーム解析]]によって、[[wikipedia:ja:常染色体|常染色体]]上の関連遺伝子の同定が相次いでいる。高速かつ網羅的な解析が可能となることで、疾患の重症度に複数の遺伝子異常の関与も示唆され始めている。ID関連遺伝子の多くは、[[シナプス形成]]・機能に関連する蛋白質をコードし、さらに多くの関連遺伝子の同定が進むことで、ID発症メカニズムの詳細が明らかになると期待される。 | |||
[[知的障害]](Intellectual Disability、以下IDと称す)は全人口のうち約2-3%に認めるといわれ、特徴的な身体所見や検査所見を伴う症候性IDと、ID以外に付随症状を伴わない非症候性IDに分類される。IDのうち約50-60% | |||
==概略== | ==概略== | ||
| 22行目: | 13行目: | ||
[[Image:図3. シナプス前終末におけるID関連分子群.png|thumb|300px|'''図3. [[シナプス前終末]]におけるID関連分子群'''<br />[[シナプス小胞]]形成から[[神経伝達物質]]の放出までの代表的な経路を示す。ID関連遺伝子がコードする分子は紫色で提示されている。(H. van Bokhoven, 2011より改変引用)<br />]] | [[Image:図3. シナプス前終末におけるID関連分子群.png|thumb|300px|'''図3. [[シナプス前終末]]におけるID関連分子群'''<br />[[シナプス小胞]]形成から[[神経伝達物質]]の放出までの代表的な経路を示す。ID関連遺伝子がコードする分子は紫色で提示されている。(H. van Bokhoven, 2011より改変引用)<br />]] | ||
[[Image:図4. シナプス後部におけるID関連分子群.png|thumb|300px|'''図4.[[シナプス後部]]におけるID関連分子群'''<br />[[シナプス後肥厚形成]]、[[細胞骨格]]再構成、[[シグナル]]伝達、[[ | [[Image:図4. シナプス後部におけるID関連分子群.png|thumb|300px|'''図4.[[シナプス後部]]におけるID関連分子群'''<br />[[シナプス後肥厚形成]]、[[細胞骨格]]再構成、[[シグナル]]伝達、[[エピジェネティックス]]な[[転写制御]]に関わる代表的な経路を示す。図3と同様ID関連遺伝子がコードする分子は紫色で提示されている。(H. van Bokhoven , 2011より改変引用)]] | ||
[[wikipedia:American Psychiatric Association|アメリカ精神医学会]]の定めている「精神疾患の診断・統計マニュアル[[DSM-IV-TR]]」の定義によると、IDは | [[wikipedia:American Psychiatric Association|アメリカ精神医学会]]の定めている「精神疾患の診断・統計マニュアル[[DSM-IV-TR]]」の定義によると、IDは | ||
| 31行目: | 22行目: | ||
としている。軽度(IQ 50-69)、中等度(IQ 35-49)、重度(IQ 20-34)、最重度(IQ 20以下)に分類される。 | としている。軽度(IQ 50-69)、中等度(IQ 35-49)、重度(IQ 20-34)、最重度(IQ 20以下)に分類される。 | ||
全人口に占めるIDの頻度は約2-3%、中等度~重度ID(IQ<50)は0.3-0.5%で、IDの約85%は軽症である。男女比は1.3:1で、男性が女性より多い。遺伝的な要因が同定されているのは約50-60%(中等度から重度で約65%、軽度で約20%) | 全人口に占めるIDの頻度は約2-3%、中等度~重度ID(IQ<50)は0.3-0.5%で、IDの約85%は軽症である。男女比は1.3:1で、男性が女性より多い。遺伝的な要因が同定されているのは約50-60%(中等度から重度で約65%、軽度で約20%)で、軽度IDは環境要因が大きいと推定されている。<ref name=ref1><pubmed> 21910631 </pubmed></ref> | ||
IDと、[[精神遅滞]](mental retardation: MR)はほぼ同義語であるが、日本では1998年9月公布の精神薄弱の用語の整理のための関係法律の一部を改正する法律において、米国では2010年5月公布のRosa’s Lawにおいて、知的障害(Intellectual Disability)へ呼称を変更することが、法律に明記された。 | IDと、[[精神遅滞]](mental retardation: MR)はほぼ同義語であるが、日本では1998年9月公布の精神薄弱の用語の整理のための関係法律の一部を改正する法律において、米国では2010年5月公布のRosa’s Lawにおいて、知的障害(Intellectual Disability)へ呼称を変更することが、法律に明記された。 | ||
| 39行目: | 30行目: | ||
==IDの遺伝学的原因== | ==IDの遺伝学的原因== | ||
IDの最も頻度の高い原因は[[ダウン症候群|21トリソミー]]であり、21トリソミーも含む染色体異常はIDの約10% | IDの最も頻度の高い原因は[[ダウン症候群|21トリソミー]]であり、21トリソミーも含む染色体異常はIDの約10%に認めるといわれている。<ref name=ref2><pubmed>20466091</pubmed></ref>染色体異常には数的異常と[[wikipedia:ja:染色体異常#.E3.81.9D.E3.81.AE.E4.BB.96.E3.81.AE.E6.9F.93.E8.89.B2.E4.BD.93.E7.95.B0.E5.B8.B8|転座]]や[[wikipedia:ja:逆位|逆位]]などの質的構造異常が含まれ、複数の遺伝子の量的異常や構造異常により切断された遺伝子が原因となりうる。通常の染色体検査(核型検査)で正常と判断されたIDの約2.5%に[[wikipedia:ja:サブテロメア|サブテロメア]](テロメアに隣接する約100~300 Kbの領域)構造異常を認めることも知られており、[[wikipedia:ja:蛍光in situハイブリダイゼーション|蛍光in situハイブリダイゼーション]](FISH)法や[[wikipedia:multiplex ligation-dependent probe amplification|multiplex ligation-dependent probe amplification]] (MLPA) 法を用いて、サブテロメア領域の欠失・重複を検出することが可能である。<ref name=ref1 />また[[wikipedia:ja:核型検査|核型検査]]正常例のマイクロアレイスクリーニングで、数Kbから数Mbにわたる正常[[wikipedia:ja:ヒトゲノム|ヒトゲノム]]に認めない[[wikipedia:ja:コピー数異常|コピー数異常]](copy number variation : CNV)をIDの約5-20%に認めるといわれており、CNV内の量的感受性遺伝子や近傍の遺伝子発現変化、切断点上の遺伝子破壊や再構成による[[wikipedia:ja:融合遺伝子|融合遺伝子]]などが原因となりうる。<ref name=ref2 />単一遺伝子異常には遺伝子全体もしくは部分欠失、重複、点変異などが含まれる。その多くは、症候性IDやX連鎖性ID(X-linked Intellectual Disability、以下XLIDと称す)に関連した染色体・ゲノム構造異常や家系による[[wikipedia:ja:連鎖解析|連鎖解析]]より単離されてきたが、遺伝子座位同定のみ判明し原因遺伝子が未同定のものも多い。 | ||
==ID関連遺伝子の分類== | ==ID関連遺伝子の分類== | ||
ID関連遺伝子は現在450遺伝子以上報告され、そのうち症候性ID関連が約400遺伝子、非症候性ID関連が約50遺伝子である。<ref name=ref1 />X連鎖性遺伝子は約100遺伝子 (約22%)であり、XLIDが高頻度(男性の約1/600-1/1000人)でIDが男性に多いことをよく説明する。<ref name=ref3><pubmed>22482801</pubmed></ref>XLIDも、非症候性 XLID (従来MRXと呼ばれた)と、症候性XLID (従来のMRXS)に分類される。症候性XLIDには現在150症候群以上が知られ、そのうち約80遺伝子が単離、約30症候群の遺伝子座位が同定されている(図1)。非症候性 XLIDでは45家系以上で遺伝子座位が同定され、現在約40遺伝子が単離されているが、そのうち約20遺伝子は同時に症候性XLIDの責任遺伝子でもある(図2)。<ref name=ref1 /><ref name=ref3 /><ref>’’’ Greenwood Genetic Center XLID Update of the Atlas on X-linked Mental Retardation. 2011. http://www.ggc.org/research/molecular-studies/xlid.html’’’</ref>近年、常染色体に責任遺伝子を持つIDの報告が相次いでおり、相対的にXLIDの頻度は低下している。<ref name=ref1 /><ref name=ref5><pubmed>20797689</pubmed></ref><ref name=ref6><pubmed>21076407</pubmed></ref><ref name=ref7><pubmed>21572417</pubmed></ref><ref name=ref8><pubmed>21937992</pubmed></ref> | |||
Najmabadiら(2011年)は、[[wikipedia:ja:常染色体劣性遺伝|常染色体劣性遺伝]]形式を示す非症候性IDの136[[wikipedia:ja:血族婚|血族婚]]家系に対し、次世代シーケンサーを用いたエクソーム解析を行い、新規原因遺伝子として50遺伝子を同定したと報告している。<ref name=ref8 />Awadallaら(2010年)やVissersら(2010年)は、家族歴のない非症候性ID例([[自閉症]]や[[統合失調症]]を含む)において、エクソーム解析により常染色体上の遺伝子に多数の[[新生突然変異]](de novo mutation)を報告しており、今後さらに常染色体上のID関連遺伝子の同定が進むと考えられる。<ref name=ref5 /><ref name=ref6 />ID関連遺伝子は、コードする蛋白質の機能別に、①シナプス形成に関与、②細胞骨格に関与、③シグナル伝達系群に関与、④遺伝子転写制御やエピジェネティックス機構に関与、⑤代謝系に関与するものなどに分けられる。中でもシナプス形成・機能に関連する遺伝子は多い。また一症例に疾患関連CNVや関連遺伝子変異を同時に認める報告も相次いでおり、複数の遺伝子異常がID重症度と関連している可能性が示唆される。<ref name=ref1 /><ref name=ref7 /><ref name=ref9><pubmed>21841781</pubmed></ref>Bokhoven(2011年)が提示したシナプス形成・機能に関与する分子群を示す(図3、4)。 | |||
==IDの分子遺伝学的診断フローチャート== | |||
[[wikipedia:American Academy of Neurology|アメリカ神経学会]](ANN)からの[[全般性発達障害]](global developmental delay)/知的障害(Intellectual Disability)の評価を行うガイドライン(2003年)では、少なくとも三世代にわたる家系図、出生前後の病歴、特徴的な身体所見、画像所見、核型検査などの遺伝学的検査、代謝系の検査、行動などについての評価を段階的に行うことが勧められている。<ref name=ref10><pubmed>21956720</pubmed></ref>そのなかで遺伝学的検査は3.5~10%に診断的意義を認め、形態異常が認められない症例でも行うべきであるとしている。<ref><pubmed>12578916</pubmed></ref>遺伝学的検査は段階的に行うことが推奨される(既知の原因探索を除く)。まず染色体異常の検出方法として、[[wikipedia:ja:Gバンド分染法|Gバンド分染法]]による核型検査、FISH法が挙げられ、これらの検査では約数Mb~数十Kbレベルの構造異常が検出可能である。より微細なゲノム構造異常、CNVの検出方法として、マイクロアレイ解析、MLPA法、[[wikipedia:ja:定量PCR法|定量PCR法]]などがあり、これらの検査では約数Kbレベルの構造異常を検出することができる。Michelsonら(2011年)の報告によると、核型検査での異常検出率は約4% (症候性は約19%)、サブテロメアFISHでの異常検出率は約3-6% (症候性が約5%)であった。<ref name=ref10 />マイクロアレイ解析での異常検出率は約7% (症候性は約11%)であり、欧米ではGバンド分染法に替わるFirst tier testとして提唱された。<ref name=ref2 />単一遺伝子異常のうち、[[FMR1]]遺伝子変異は軽症の患児の2%に認め、[[MECP2]]遺伝子変異は中等度から重度の女児の1.5%に認めており、家族歴のないID患者でもよく認めることから、ルーチンで行うことが推奨されている。<ref name=ref10 /> | |||
現時点でのIDの分子遺伝学的検査の流れを以下にまとめる(図5)。 | |||
[[Image:図5. IDの分子遺伝学的検査のフローチャート.png|thumb|300px|図'''5. IDの分子遺伝学的検査のフローチャート''']] | [[Image:図5. IDの分子遺伝学的検査のフローチャート.png|thumb|300px|図'''5. IDの分子遺伝学的検査のフローチャート''']] | ||
症候性IDが疑われる症例は、[[wikipedia:ja:表現型|表現型]]より推定される既知の原因探索を行う。既知の原因を認めなかった場合は、非症候性IDと同様の段階的な原因探索を行う。非症候性IDが疑われる症例は、まず核型検査を行い染色体異常の有無を確認し、異常を認めなければ、より微細なゲノム構造異常をみるために、マイクロアレイ解析を行う。ゲノム構造異常を認めた場合は、その領域に存在する候補遺伝子の探索、あるいは切断点近傍の遺伝子解析を行う。ゲノム構造異常を認めない場合は、高密度[[SNP]]マイクロアレイを用いて連鎖解析や[[wikipedia:homozygosity mapping|homozygosity mapping]]を行い、候補領域の絞り込みを行う。候補領域内の責任遺伝子探索を続いて行うが、直接シーケンス法で遺伝子変異の探索を行う他に、コントロール配列と変異のある配列の二本鎖が解離する温度の違いを検出することで遺伝子変異の検出を行う、[[wikipedia:ja:高解像度融解曲線分析|高解像度融解曲線分析]] high-resolution melting curve (HRM) 法を用いることも有用である。近年では次世代シーケンサーを用いた全エクソーム・全ゲノムシーケンス解析を行うことで、高速で網羅的な解析が可能となり遺伝子特定のスピードが上がっている。 | |||
症候性IDが疑われる症例は、[[wikipedia:ja:表現型|表現型]] | |||
==展望== | ==展望== | ||
マイクロアレイ解析や次世代シーケンス解析などの新しい分子遺伝学検査の進歩・普及により、多くの症例を詳細に解析することができるようになった。その一端としてID関連遺伝子の多くが、シナプス形成・機能に関わる蛋白質をコードするなど、今後も多くの関連遺伝子が同定されIDの総合的理解が進むとともに、ID予防や治療につながる戦略の開発も可能になることが期待される。 | |||
==参考文献== | ==参考文献== | ||
<references /> | <references /> | ||
(執筆者:近藤有希子、松本直通 担当編集委員:加藤忠史)<br> | |||
2012年5月10日 (木) 21:51時点における版
英語名: Human genes in relation to Intellectual Disability
英語略名: ID genes
知的障害(Intellectual Disability、以下IDと称す)は全人口のうち約2-3%に認めるといわれ、特徴的な身体所見や検査所見を伴う症候性IDと、ID以外に付随症状を伴わない非症候性IDに分類される。IDのうち約50-60%で遺伝的原因が解明、それらは染色体異常、ゲノム構造異常、単一遺伝子異常など多岐にわたる。マイクロアレイや次世代シーケンサーなど分子遺伝学検査の急速な進歩・普及により、現在ID関連遺伝子は約450遺伝子以上も報告され、そのうち症候性IDの原因として約400遺伝子、非症候性IDの原因として約50遺伝子が認知されている。またX連鎖性遺伝子は約22%(約100遺伝子)で、男性にIDの頻度が高い要因となっている。非症候性IDに対するエクソーム解析によって、常染色体上の関連遺伝子の同定が相次いでいる。高速かつ網羅的な解析が可能となることで、疾患の重症度に複数の遺伝子異常の関与も示唆され始めている。ID関連遺伝子の多くは、シナプス形成・機能に関連する蛋白質をコードし、さらに多くの関連遺伝子の同定が進むことで、ID発症メカニズムの詳細が明らかになると期待される。
概略

症候性XLIDの100症候群とその責任遺伝子75遺伝子を示す。(Greenwood Genetic Center, updated June 2011より改変引用)

左側に非症候性XLID (MRX)に関わる22遺伝子、右側に非症候性XLID (MRX)と症候性XLID (MRXS)の両方に関わる17遺伝子を示す。(Greenwood Genetic Center, updated July 2011より改変引用)
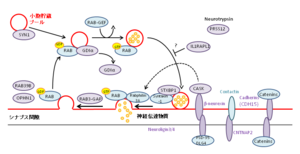
シナプス小胞形成から神経伝達物質の放出までの代表的な経路を示す。ID関連遺伝子がコードする分子は紫色で提示されている。(H. van Bokhoven, 2011より改変引用)

シナプス後肥厚形成、細胞骨格再構成、シグナル伝達、エピジェネティックスな転写制御に関わる代表的な経路を示す。図3と同様ID関連遺伝子がコードする分子は紫色で提示されている。(H. van Bokhoven , 2011より改変引用)
アメリカ精神医学会の定めている「精神疾患の診断・統計マニュアルDSM-IV-TR」の定義によると、IDは
- 明らかに平均以下の知的機能: 個別施行による知能検査で、およそ70またはそれ以下のIQ (幼児においては、明らかに平均以下の知的機能であるという臨床的判断による)
- 同時に、現在の適応機能 (すなわち、その文化圏でその年齢に対して期待される基準に適応する有能さ)の欠陥または不全が、以下のうち2つ以上の領域で存在: コミュニケーション、自己管理、家庭生活、社会的/対人的技能、地域社会資源の利用、自律性、発揮される学習能力、仕事、余暇、健康、安全、
- 発症は18歳以前である状態
としている。軽度(IQ 50-69)、中等度(IQ 35-49)、重度(IQ 20-34)、最重度(IQ 20以下)に分類される。
全人口に占めるIDの頻度は約2-3%、中等度~重度ID(IQ<50)は0.3-0.5%で、IDの約85%は軽症である。男女比は1.3:1で、男性が女性より多い。遺伝的な要因が同定されているのは約50-60%(中等度から重度で約65%、軽度で約20%)で、軽度IDは環境要因が大きいと推定されている。[1]
IDと、精神遅滞(mental retardation: MR)はほぼ同義語であるが、日本では1998年9月公布の精神薄弱の用語の整理のための関係法律の一部を改正する法律において、米国では2010年5月公布のRosa’s Lawにおいて、知的障害(Intellectual Disability)へ呼称を変更することが、法律に明記された。
IDは、症候性と非症候性に大別できる。症候性IDは、特徴的な身体所見、臨床経過および生化学的所見などから他の疾患と区別可能な場合をいう。染色体異常やゲノム構造異常による複数の遺伝子の欠失・重複、または単一遺伝子異常が原因であることが多い。一方非症候性IDは、他のIDをきたす疾患と区別されるような知能以外の特徴的な症状を伴わない場合をいい、単一遺伝子異常や環境要因が原因とされている。小児期にはID以外の身体的特徴が明らかでない場合もあり、両者の区別は時に困難である。
IDの遺伝学的原因
IDの最も頻度の高い原因は21トリソミーであり、21トリソミーも含む染色体異常はIDの約10%に認めるといわれている。[2]染色体異常には数的異常と転座や逆位などの質的構造異常が含まれ、複数の遺伝子の量的異常や構造異常により切断された遺伝子が原因となりうる。通常の染色体検査(核型検査)で正常と判断されたIDの約2.5%にサブテロメア(テロメアに隣接する約100~300 Kbの領域)構造異常を認めることも知られており、蛍光in situハイブリダイゼーション(FISH)法やmultiplex ligation-dependent probe amplification (MLPA) 法を用いて、サブテロメア領域の欠失・重複を検出することが可能である。[1]また核型検査正常例のマイクロアレイスクリーニングで、数Kbから数Mbにわたる正常ヒトゲノムに認めないコピー数異常(copy number variation : CNV)をIDの約5-20%に認めるといわれており、CNV内の量的感受性遺伝子や近傍の遺伝子発現変化、切断点上の遺伝子破壊や再構成による融合遺伝子などが原因となりうる。[2]単一遺伝子異常には遺伝子全体もしくは部分欠失、重複、点変異などが含まれる。その多くは、症候性IDやX連鎖性ID(X-linked Intellectual Disability、以下XLIDと称す)に関連した染色体・ゲノム構造異常や家系による連鎖解析より単離されてきたが、遺伝子座位同定のみ判明し原因遺伝子が未同定のものも多い。
ID関連遺伝子の分類
ID関連遺伝子は現在450遺伝子以上報告され、そのうち症候性ID関連が約400遺伝子、非症候性ID関連が約50遺伝子である。[1]X連鎖性遺伝子は約100遺伝子 (約22%)であり、XLIDが高頻度(男性の約1/600-1/1000人)でIDが男性に多いことをよく説明する。[3]XLIDも、非症候性 XLID (従来MRXと呼ばれた)と、症候性XLID (従来のMRXS)に分類される。症候性XLIDには現在150症候群以上が知られ、そのうち約80遺伝子が単離、約30症候群の遺伝子座位が同定されている(図1)。非症候性 XLIDでは45家系以上で遺伝子座位が同定され、現在約40遺伝子が単離されているが、そのうち約20遺伝子は同時に症候性XLIDの責任遺伝子でもある(図2)。[1][3][4]近年、常染色体に責任遺伝子を持つIDの報告が相次いでおり、相対的にXLIDの頻度は低下している。[1][5][6][7][8]
Najmabadiら(2011年)は、常染色体劣性遺伝形式を示す非症候性IDの136血族婚家系に対し、次世代シーケンサーを用いたエクソーム解析を行い、新規原因遺伝子として50遺伝子を同定したと報告している。[8]Awadallaら(2010年)やVissersら(2010年)は、家族歴のない非症候性ID例(自閉症や統合失調症を含む)において、エクソーム解析により常染色体上の遺伝子に多数の新生突然変異(de novo mutation)を報告しており、今後さらに常染色体上のID関連遺伝子の同定が進むと考えられる。[5][6]ID関連遺伝子は、コードする蛋白質の機能別に、①シナプス形成に関与、②細胞骨格に関与、③シグナル伝達系群に関与、④遺伝子転写制御やエピジェネティックス機構に関与、⑤代謝系に関与するものなどに分けられる。中でもシナプス形成・機能に関連する遺伝子は多い。また一症例に疾患関連CNVや関連遺伝子変異を同時に認める報告も相次いでおり、複数の遺伝子異常がID重症度と関連している可能性が示唆される。[1][7][9]Bokhoven(2011年)が提示したシナプス形成・機能に関与する分子群を示す(図3、4)。
IDの分子遺伝学的診断フローチャート
アメリカ神経学会(ANN)からの全般性発達障害(global developmental delay)/知的障害(Intellectual Disability)の評価を行うガイドライン(2003年)では、少なくとも三世代にわたる家系図、出生前後の病歴、特徴的な身体所見、画像所見、核型検査などの遺伝学的検査、代謝系の検査、行動などについての評価を段階的に行うことが勧められている。[10]そのなかで遺伝学的検査は3.5~10%に診断的意義を認め、形態異常が認められない症例でも行うべきであるとしている。[11]遺伝学的検査は段階的に行うことが推奨される(既知の原因探索を除く)。まず染色体異常の検出方法として、Gバンド分染法による核型検査、FISH法が挙げられ、これらの検査では約数Mb~数十Kbレベルの構造異常が検出可能である。より微細なゲノム構造異常、CNVの検出方法として、マイクロアレイ解析、MLPA法、定量PCR法などがあり、これらの検査では約数Kbレベルの構造異常を検出することができる。Michelsonら(2011年)の報告によると、核型検査での異常検出率は約4% (症候性は約19%)、サブテロメアFISHでの異常検出率は約3-6% (症候性が約5%)であった。[10]マイクロアレイ解析での異常検出率は約7% (症候性は約11%)であり、欧米ではGバンド分染法に替わるFirst tier testとして提唱された。[2]単一遺伝子異常のうち、FMR1遺伝子変異は軽症の患児の2%に認め、MECP2遺伝子変異は中等度から重度の女児の1.5%に認めており、家族歴のないID患者でもよく認めることから、ルーチンで行うことが推奨されている。[10]
現時点でのIDの分子遺伝学的検査の流れを以下にまとめる(図5)。
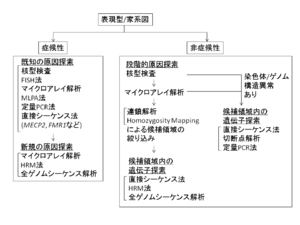
症候性IDが疑われる症例は、表現型より推定される既知の原因探索を行う。既知の原因を認めなかった場合は、非症候性IDと同様の段階的な原因探索を行う。非症候性IDが疑われる症例は、まず核型検査を行い染色体異常の有無を確認し、異常を認めなければ、より微細なゲノム構造異常をみるために、マイクロアレイ解析を行う。ゲノム構造異常を認めた場合は、その領域に存在する候補遺伝子の探索、あるいは切断点近傍の遺伝子解析を行う。ゲノム構造異常を認めない場合は、高密度SNPマイクロアレイを用いて連鎖解析やhomozygosity mappingを行い、候補領域の絞り込みを行う。候補領域内の責任遺伝子探索を続いて行うが、直接シーケンス法で遺伝子変異の探索を行う他に、コントロール配列と変異のある配列の二本鎖が解離する温度の違いを検出することで遺伝子変異の検出を行う、高解像度融解曲線分析 high-resolution melting curve (HRM) 法を用いることも有用である。近年では次世代シーケンサーを用いた全エクソーム・全ゲノムシーケンス解析を行うことで、高速で網羅的な解析が可能となり遺伝子特定のスピードが上がっている。
展望
マイクロアレイ解析や次世代シーケンス解析などの新しい分子遺伝学検査の進歩・普及により、多くの症例を詳細に解析することができるようになった。その一端としてID関連遺伝子の多くが、シナプス形成・機能に関わる蛋白質をコードするなど、今後も多くの関連遺伝子が同定されIDの総合的理解が進むとともに、ID予防や治療につながる戦略の開発も可能になることが期待される。
参考文献
- ↑ 1.0 1.1 1.2 1.3 1.4 1.5
van Bokhoven, H. (2011).
Genetic and epigenetic networks in intellectual disabilities. Annual review of genetics, 45, 81-104. [PubMed:21910631] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2
Miller, D.T., Adam, M.P., Aradhya, S., Biesecker, L.G., Brothman, A.R., Carter, N.P., ..., & Ledbetter, D.H. (2010).
Consensus statement: chromosomal microarray is a first-tier clinical diagnostic test for individuals with developmental disabilities or congenital anomalies. American journal of human genetics, 86(5), 749-64. [PubMed:20466091] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1
Lubs, H.A., Stevenson, R.E., & Schwartz, C.E. (2012).
Fragile X and X-linked intellectual disability: four decades of discovery. American journal of human genetics, 90(4), 579-90. [PubMed:22482801] [PMC] [WorldCat] [DOI] - ↑ ’’’ Greenwood Genetic Center XLID Update of the Atlas on X-linked Mental Retardation. 2011. http://www.ggc.org/research/molecular-studies/xlid.html’’’
- ↑ 5.0 5.1
Awadalla, P., Gauthier, J., Myers, R.A., Casals, F., Hamdan, F.F., Griffing, A.R., ..., & Rouleau, G.A. (2010).
Direct measure of the de novo mutation rate in autism and schizophrenia cohorts. American journal of human genetics, 87(3), 316-24. [PubMed:20797689] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Vissers, L.E., de Ligt, J., Gilissen, C., Janssen, I., Steehouwer, M., de Vries, P., ..., & Veltman, J.A. (2010).
A de novo paradigm for mental retardation. Nature genetics, 42(12), 1109-12. [PubMed:21076407] [WorldCat] [DOI] - ↑ 7.0 7.1
O'Roak, B.J., Deriziotis, P., Lee, C., Vives, L., Schwartz, J.J., Girirajan, S., ..., & Eichler, E.E. (2011).
Exome sequencing in sporadic autism spectrum disorders identifies severe de novo mutations. Nature genetics, 43(6), 585-9. [PubMed:21572417] [PMC] [WorldCat] [DOI] - ↑ 8.0 8.1
Najmabadi, H., Hu, H., Garshasbi, M., Zemojtel, T., Abedini, S.S., Chen, W., ..., & Ropers, H.H. (2011).
Deep sequencing reveals 50 novel genes for recessive cognitive disorders. Nature, 478(7367), 57-63. [PubMed:21937992] [WorldCat] [DOI] - ↑
Cooper, G.M., Coe, B.P., Girirajan, S., Rosenfeld, J.A., Vu, T.H., Baker, C., ..., & Eichler, E.E. (2011).
A copy number variation morbidity map of developmental delay. Nature genetics, 43(9), 838-46. [PubMed:21841781] [PMC] [WorldCat] [DOI] - ↑ 10.0 10.1 10.2
Michelson, D.J., Shevell, M.I., Sherr, E.H., Moeschler, J.B., Gropman, A.L., & Ashwal, S. (2011).
Evidence report: Genetic and metabolic testing on children with global developmental delay: report of the Quality Standards Subcommittee of the American Academy of Neurology and the Practice Committee of the Child Neurology Society. Neurology, 77(17), 1629-35. [PubMed:21956720] [WorldCat] [DOI] - ↑
Shevell, M., Ashwal, S., Donley, D., Flint, J., Gingold, M., Hirtz, D., ..., & Practice Committee of the Child Neurology Society (2003).
Practice parameter: evaluation of the child with global developmental delay: report of the Quality Standards Subcommittee of the American Academy of Neurology and The Practice Committee of the Child Neurology Society. Neurology, 60(3), 367-80. [PubMed:12578916] [WorldCat] [DOI]
(執筆者:近藤有希子、松本直通 担当編集委員:加藤忠史)