RNA干渉
塩見 美喜子
慶應義塾大学 医学部 分子生物学
DOI:10.14931/bsd.3744 原稿受付日:2013年4月30日 原稿完成日:2015年1月9日
担当編集委員:上口 裕之(独立行政法人理化学研究所 脳科学総合研究センター)
英語名:RNA interference 独:RNA-Interferenz 仏:Interférence par ARN
英略名:RNAi
RNA干渉とはmRNAに対して相補的な配列をもつ一本鎖RNA(アンチセンス鎖)、その逆鎖である一本鎖RNA(センス鎖)からなる二本鎖RNAによって、遺伝子発現抑制効果を示す現象である。当初、線虫の遺伝子Lin-4遺伝子産物が、タンパク質をコードせずに遺伝子発現を制御することから示されたが、その後、単細胞生物から哺乳動物に至る様々な生物で内在性の小分子RNAがRNA干渉のメカニズムにより遺伝子制御に関わることが見いだされ、発生や代謝、感染防御など生命維持に欠かせない多くの現象を制御し、生体の恒常性を維持する働きを有することが分かっている。RNA干渉関連分子の機能異常が発症原因となる疾患も見つかってきている。またさらに外来に二本鎖RNAを投与することによっても同様のメカニズムによって遺伝子発現を制御することができることから、RNA干渉は、遺伝子機能探索の技術として細胞や個体でも応用が可能で、創薬に繋がる大きな可能性を秘めている。
RNA干渉とは
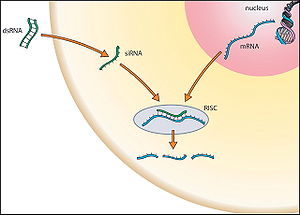
投与された二本鎖RNAはshort interfering RNA(siRNA)に変換され、mRNAとRNA induced silencing complex (RISC)を形成し、mRNAの不安定化を引き起こす。
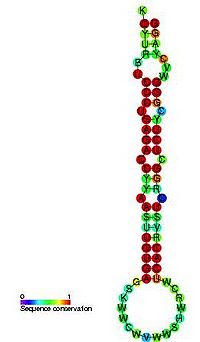
1993年、Victor Ambros博士らは、C. elegansのlin-4遺伝子産物がタンパク質をコードしないのにもかかわらず、lin-14遺伝子産物であるLIN-14タンパク質の発現を負に調節する因子であることを見出した[1]。この遺伝子産物は、ヘアピン型の小分子RNAであり、標的遺伝子と結合することにより機能すると考えられた(図1)。その後、microRNA(miRNA)と呼ばれるようになり、現在までに単細胞生物から哺乳動物に至る様々な生物で内在性の小分子RNAがRNA干渉のメカニズムにより遺伝子制御に関わることが見いだされ、発生や代謝、ウイルス感染防御など生命維持に欠かせない多くの現象を制御し、生体の恒常性を維持する働きを有することが分かっている[2] [3] [4]。RNAi関連分子の機能異常が発症原因となる疾患も見つかってきている[5]。
一方、1998年に、線虫の発生遺伝学者Craig MelloとAndrew Fire両博士らは、発生に関わる遺伝子の機能解析を目的として、線虫体内に、標的mRNAに対して相補的な配列をもつ一本鎖RNA(アンチセンス鎖)、その逆鎖である一本鎖RNA(センス鎖)、その両者からなる二本鎖RNAを別途投与することによって、二本鎖RNAが高い遺伝子発現抑制効果を示すことを見出した[6]。この発見は、小分子RNAを外来性に投与することにより、任意の遺伝子の発現を調節する可能性を示唆した(図2)。
これらの発見をきっかけにRNA干渉の研究は飛躍的にすすみ、現在では二本鎖RNA がshort interfering RNA(siRNA)に変換されて遺伝子発現を調節するプロセスが明らかとなっている[2] [3] [4]。siRNAもmiRNAもその生合成にDicerを必要とする、また遺伝子の発現を抑制するにはArgonauteタンパク質とRNA induced silencing complex (RISC)を形成する必要がある、などお互い類似したメカニズムで起こることなどが明らかにされている[2] [3] [4]。
また、RNA干渉は、遺伝子機能探索の技術として細胞や個体でも応用が可能で、創薬に繋がる大きな可能性を秘めているため、基礎・応用に限らず国内外の多くの研究者がRNAサイレンシングを研究対象としている。
内在性小分子RNAによるもの
miRNA
動物や植物に限らず多くの生物はmiRNAを発現する[7] [8]。miRNAは恒常的であるが、各miRNAの発現は個体において時空間的に調節されている。
ゲノム上に位置するmiRNA遺伝子から発現したmiRNA転写産物は一本鎖RNAであるが、ヘアピン構造をとることを特徴とする[9] [10] [11]。核でまずRNaseIIIドメインをもったDroshaによって第一次プロセシングを受け細胞質へ移行する。細胞質ではDicerによって第二次プロセシングを受け、二本鎖miRNAとして切り出される。その後1本鎖となったmiRNAは、siRNAと同様にArgonauteタンパク質と結合することによってmiRISCを形成する。哺乳動物のmiRNAの場合、標的mRNAへの対合にはシード配列とよばれる5’末端から2~7塩基が関わる。つまり、siRNAと異なり、標的RNAとの対合がmiRNAの5’末端から10塩基目と11塩基目まで及ばないため、Argonauteタンパク質は標的RNAを切断する事が出来ない。miRISC にはGW182タンパク質を介してRNA分解酵素が結合するが、これら因子の助けを借りて、miRISCは標的RNAの不安定性を導く。標的mRNAの翻訳を阻害することも知られる。つまり、siRNAとmiRNAでは標的RNAの発現抑制の分子メカニズムが異なることを特徴とする。
PIWI interacting RNA
PIWI interacting RNA(piRNA)は生殖組織特異的に発現する小分子RNAで、やはり生殖組織特異的に発現するArgonauteタンパク質群(これをPIWIタンパク質と称する)と特異的に結合することによってpiRISCを形成する。piRNAの多くはpiRNAクラスターとよばれる遺伝子間領域から発現する一本鎖非コードRNAを前駆体とし、Dicer非依存的に発現する[12] [13] [14] [15]。piRNAはトランスポゾンに対してアンチセンスである場合が多く、それらはトランスポゾンの発現抑制に関わる。トランスポゾンはゲノム損傷の原因となるため、次世代にゲノム情報を伝搬する生殖組織はこれを抑える独自の仕組みを獲得したと考えられる。細胞質局在型piRISCは、標的RNA(トランスポゾン転写産物)を切断することによってその発現を抑制するが、核局在型piRISCは転写レベルでトランスポゾンを抑制すると考えられている。piRNA生合成およびpiRISCによる転写レベルでのトランスポゾン発現抑制の分子メカニズムは未だ不明な点を多く残す。
外来小分子RNAによるもの
RNA干渉を用いて、任意の遺伝子の発現を抑制することが可能である。ヘアピン状になったshort hairpin RNA (shRNA)を用いることが多い。配列はSiDirect 2.0などでデザインするが、実際には複数個作成して効果を確認することが推奨される。shRNAはRNAとして合成して導入するほか、ウイルスベクターやプラスミドベクターとして導入することも可能である。
細胞内に導入された二本鎖RNAは、21塩基長程度の一本鎖RNAであるsiRNAへと変換され、標的RNAへの対合及び発現抑制が可能になる[2] [3] [4]。二本鎖RNAのプロセシングにはRNaseIIIドメインをもったDicerタンパク質が働く。Dicerは二本鎖RNAの端から21塩基長程度の二本鎖RNAを切り出す。Dicer切断による産物は、5'末端にリン酸基を有し、3'末端が反対鎖より2塩基突出していることを特徴とする。切り出された二本鎖RNA はArgonauteタンパク質に取り込まれ、Argonauteタンパク質がもつRNA切断活性(Slicer活性と呼ぶ)によって一方鎖の中央が切断され一本鎖RNAとなる。残った一本鎖siRNAとArgonauteタンパク質からなる複合体をRISCとよぶ。二本鎖siRNAのうち、どちらの鎖がArgonauteによって切られるかは、二本鎖siRNAの両端の対合性の強弱によって決定される。
RISCに結合した標的RNAは、Argonauteタンパク質のSlicer活性によって切断される。試験管内でRNA干渉反応を誘導すると、切断された標的RNAを検出することが出来るが、細胞内ではSlicer活性によって切断されたmRNAはその他のRNA分解酵素によって分解されるため、タンパク質合成の鋳型となりえない。RNA干渉を遺伝子発現抑制機構とする所以はここにある。
Argonauteタンパク質はPAZドメインとMIDドメインを介してsiRNAと結合する。標的RNAの切断は、PIWIドメインが担う。切断はsiRNAの5’末端から10塩基目と11塩基目をつなぐリン酸基に相対する部位で起こり、その切り口は、5'末端にリン酸基を、3'末端は水酸基をもつことを特徴とする。
疾患との関わり
miRNAは生体内で様々な遺伝子の発現を制御する。ヒトの場合、約6割の遺伝子がmiRNAの制御下にあると見積もられている[16]。miRNAの発現や機能に異常が生じると、その下流に位置する標的遺伝子の発現も異常となり、これが原因で癌などの疾患を導きだすことが知られている。肺がんの発症との関連が唱われているmiRNAにlet-7やmiR-17-92クラスターがある[5]。miRNAの発現量をバイオマーカーとして診断に用いる試みもある[17]。
関連項目
参考文献
- ↑
Lee, R.C., Feinbaum, R.L., & Ambros, V. (1993).
The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell, 75(5), 843-54. [PubMed:8252621] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2 2.3
Ghildiyal, M., & Zamore, P.D. (2009).
Small silencing RNAs: an expanding universe. Nature reviews. Genetics, 10(2), 94-108. [PubMed:19148191] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2 3.3
Kim, V.N., Han, J., & Siomi, M.C. (2009).
Biogenesis of small RNAs in animals. Nature reviews. Molecular cell biology, 10(2), 126-39. [PubMed:19165215] [WorldCat] [DOI] - ↑ 4.0 4.1 4.2 4.3
Siomi, H., & Siomi, M.C. (2009).
On the road to reading the RNA-interference code. Nature, 457(7228), 396-404. [PubMed:19158785] [WorldCat] [DOI] - ↑ 5.0 5.1
Osada, H., & Takahashi, T. (2011).
let-7 and miR-17-92: small-sized major players in lung cancer development. Cancer science, 102(1), 9-17. [PubMed:20735434] [WorldCat] [DOI] - ↑
Fire, A., Xu, S., Montgomery, M.K., Kostas, S.A., Driver, S.E., & Mello, C.C. (1998).
Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature, 391(6669), 806-11. [PubMed:9486653] [WorldCat] [DOI] - ↑
Pasquinelli, A.E., Reinhart, B.J., Slack, F., Martindale, M.Q., Kuroda, M.I., Maller, B., ..., & Ruvkun, G. (2000).
Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature, 408(6808), 86-9. [PubMed:11081512] [WorldCat] [DOI] - ↑
Bartel, D.P. (2004).
MicroRNAs: genomics, biogenesis, mechanism, and function. Cell, 116(2), 281-97. [PubMed:14744438] [WorldCat] [DOI] - ↑
Huntzinger, E., & Izaurralde, E. (2011).
Gene silencing by microRNAs: contributions of translational repression and mRNA decay. Nature reviews. Genetics, 12(2), 99-110. [PubMed:21245828] [WorldCat] [DOI] - ↑
Krol, J., Loedige, I., & Filipowicz, W. (2010).
The widespread regulation of microRNA biogenesis, function and decay. Nature reviews. Genetics, 11(9), 597-610. [PubMed:20661255] [WorldCat] [DOI] - ↑
Czech, B., & Hannon, G.J. (2011).
Small RNA sorting: matchmaking for Argonautes. Nature reviews. Genetics, 12(1), 19-31. [PubMed:21116305] [PMC] [WorldCat] [DOI] - ↑
Malone, C.D., & Hannon, G.J. (2009).
Small RNAs as guardians of the genome. Cell, 136(4), 656-68. [PubMed:19239887] [PMC] [WorldCat] [DOI] - ↑
Thomson, T., & Lin, H. (2009).
The biogenesis and function of PIWI proteins and piRNAs: progress and prospect. Annual review of cell and developmental biology, 25, 355-76. [PubMed:19575643] [PMC] [WorldCat] [DOI] - ↑
Siomi, M.C., Sato, K., Pezic, D., & Aravin, A.A. (2011).
PIWI-interacting small RNAs: the vanguard of genome defence. Nature reviews. Molecular cell biology, 12(4), 246-58. [PubMed:21427766] [WorldCat] [DOI] - ↑
Ishizu, H., Siomi, H., & Siomi, M.C. (2012).
Biology of PIWI-interacting RNAs: new insights into biogenesis and function inside and outside of germlines. Genes & development, 26(21), 2361-73. [PubMed:23124062] [PMC] [WorldCat] [DOI] - ↑
Friedman, R.C., Farh, K.K., Burge, C.B., & Bartel, D.P. (2009).
Most mammalian mRNAs are conserved targets of microRNAs. Genome research, 19(1), 92-105. [PubMed:18955434] [PMC] [WorldCat] [DOI] - ↑
Allegra, A., Alonci, A., Campo, S., Penna, G., Petrungaro, A., Gerace, D., & Musolino, C. (2012).
Circulating microRNAs: new biomarkers in diagnosis, prognosis and treatment of cancer (review). International journal of oncology, 41(6), 1897-912. [PubMed:23026890] [WorldCat] [DOI]