「コネクトーム」の版間の差分
Masahitoyamagata (トーク | 投稿記録) (ページの作成:「connectome コネクトーム 英語名:Connectome コネクトームは、神経系のすべての神経細胞が接続することでできた神経回路の総体...」) |
細 (→マクロスケールのコネクトーム) |
||
| (3人の利用者による、間の151版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | |||
<font size="+1">山形方人</font><br> | |||
''Harvard University''<br> | |||
DOI:<selfdoi /> 原稿受付日:2016年6月21日 原稿完成日:2016年6月29日<br> | |||
担当編集委員:[http://researchmap.jp/tsuyoshimiyakawa 宮川 剛](藤田保健衛生大学)<br> | |||
</div> | |||
英:connectome 独:Konnektom 仏:connectome 西:conectoma 露:Коннектом 中:连接组 | |||
{{box|text= | |||
コネクトームは、神経系のすべての神経細胞が接続することでできた神経回路の全体を意味する。本来は、ある動物個体、あるいはある動物種の神経系において、全神経細胞の接続の総体を意味する概念である。しかしながら、神経系の一部で神経細胞が[[化学シナプス]]や[[電気シナプス]]で接続された局所的な接続ダイアグラム(Wiring diagram)に言及する場合にも、しばしば使われる。更に、神経細胞レベルでの接続だけでなく、脳の大まかな領域同士が接続する様相の説明にも使われている。'''コネクトミクス(connectomics)'''は、コネクトームを理解するための研究法、つまり実験、検出などの方法、その方法を適用した結果の解釈、そして複数の方法論を統合した研究の戦略についての概念である。}} | |||
用語「コネクトーム」の起源 | ==用語「コネクトーム」の起源== | ||
コネクトームは、生命科学分野で広く使われている[[ゲノム]](genome)、[[プロテオーム]](proteome)などにならって、英語のconnect(ラテン語由来)とギリシア語の「完全、すべて、総体」を意味する接尾辞(-ome、ギリシア語-ωμα)を組み合わせた造語であり、英語の複数形は、connectomesである。 | |||
文献上、初めて使われたのは、2005年、[[wj:インディアナ大学|Indiana大学]]のOlaf Spornsが、神経接続のマップを説明するために、[[ヒト]]・コネクトーム(Human Connectome)という概念を記述した論文である<ref><pubmed>16201007</pubmed></ref><ref>'''Olaf Sporns'''<br>Connectome<br>Scholarpedia, 5(2):5584, 2010. [[doi:10.4249/scholarpedia.5584|[DOI]]]</ref>。それとは独立して、[[wj:ローザンヌ大学|Lausanne大学]]病院のPatric Hagmannが、その博士論文の中で同時期に同様に言及していた<ref>'''Patric Hagmann'''<br>From diffusion MRI to brain connectomics [PhD Thesis].<br>Lausanne: Ecole Polytechnique Fédérale de Lausanne (EPFL). 2005 [[doi:10.5075/epfl-thesis-3230|[DOI]]]. </ref>。 | |||
特に、このコネクトームという概念が普及され始めたのは、2008年、[[wj:ハーバード大学|Harvard大学]]の[[w:Jeffrey W. Lichtman|Jeffrey W. Lichtman]]と[[w:Joshua R. Sanes|Joshua R. Sanes]]による共著総説論文の発表後である<ref><pubmed>18801435</pubmed></ref>。また、用語の定着には、[[w:Sebastian Seung|Sebastian Seung]](当時[[wj:MIT|MIT]]、現[[wj:プリンストン大学|Princeton大学]])らのアウトリーチによる努力もあった<ref name=seung>'''Sebastian Seung'''<br>Connectome: How the Brain's Wiring Makes Us Who We Are<br>''Mariner Books'', 2013 ISBN 9780547678597<br>邦訳'''セバスチャン・スン''' (著)、 '''青木 薫''' (訳)<br>コネクトーム:脳の配線はどのように「わたし」をつくり出すのか<br>草思社</ref>。 | |||
しかし、これらの研究者の貢献は、あくまでコネクトームという用語を提案、普及、定着させたということで認知されるべきもので、コネクトームとは呼んでいないものの、それに相当する概念はこれらの研究者が提案する以前から存在していたと考えるべきである<ref><pubmed>23866325</pubmed></ref>。その典型例は、1986年に発表されたJohn Whiteらによる[[線虫]](''[[Caenorhabditis elegans]]'')という一つの生物の神経細胞の結合性を網羅的に記述した研究である(後述)<ref><pubmed>22462104</pubmed></ref><ref><pubmed>25750233</pubmed></ref>。 | |||
==コネクトームの研究史と階層== | |||
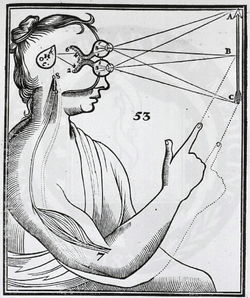
[[ファイル:Descartes3.jpg |サムネイル|250px|'''図1.Descartes 「視覚と外部刺激への応答メカニズム」(1677年)'''<br>目から入力した視覚情報が、つながった線で脳に伝えられているという仮説が表現されている。]] | |||
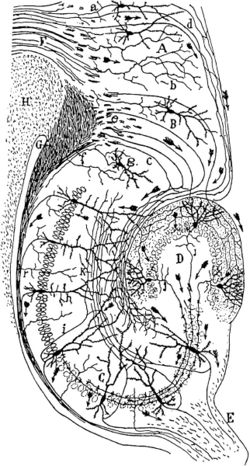
[[ファイル:Cajalhippocampus.jpg|サムネイル|250px|'''図2.Santiago Rámon y Cajalによって描写されたゴルジ染色された海馬(1911年)'''<br>脳の組織が神経細胞から成立し、神経回路を作っていることを認識された。]] | |||
歴史的には、簡素な解剖用具などを用いて[[神経線維]]を観察することから、[[目]]と[[脳]]など神経組織同士を接続している構造が存在することは想像されていた(例:[[wj:ルネ・デカルト|デカルト]]、1677年、'''図1''')。 | |||
19世紀末になると、[[wj:サンティアゴ・ラモン・イ・カハール|Santiago Rámon y Cajal]](1852-1934)が、個々の神経細胞の形態を明確に染め出すことを可能にした[[ゴルジ染色|Golgi染色]]と[[光学顕微鏡]]を用いることで、脳が多数の神経細胞とそれらの結合によって成り立っていることを提唱した('''図2''')。以後、神経細胞の間の結合を記述する研究は盛んに行われてきた<ref><pubmed>21782932</pubmed></ref><ref><pubmed>27442070</pubmed></ref>。Golgi染色や[[ニッスル染色|Nissl染色]]などを施した連続切片を観察する時代を経て、20世紀中頃になると脳損傷後の変性神経線維をNauta法などで染色することで、神経回路の存在を確認する時代になった。 | |||
1970年ごろになると、放射性[[wj:アミノ酸|アミノ酸]]や、[[wj:酵素|酵素]]([[西洋ワサビペルオキシダーゼ]] ([[horseradish peroxidase]]; [[HRP]]))などの[[軸索輸送]]を利用することで、神経回路の観察が簡便に行われるようになった。更に、1980年代には、脂溶性carbocyanine[[蛍光色素]]などの生体結合特性を持った蛍光色素([[DiI]]など)、[[wj:植物レクチン|植物レクチン]]([[wheat germ agglutinin]] ([[wheat germ agglutinin|WGA]])、[[phytohaemagglutinin]] ([[phytohaemagglutinin|PHA-L]])など)、[[ビオチン]]誘導体 ([[biocytin]], [[Neurobiotin]]など)、軸索を効率的に移動する[[コレラ毒素]]サブユニット等の高感度トレーサーが開発され、多くの研究者に汎用されるようになった。 | |||
そして、1990年代になると、[[蛍光顕微鏡]]に加えて、[[共焦点レーザー顕微鏡]]が普及し始め、デジタル画像として大規模なデータの保存と解析が行われるようになった。コネクトーム研究の観点から、このような組織学的解剖と染色によって得られた知見をまとめた重要な研究が、1991年、FellemanとDavid van Essenらによる[[マカクサル]]の[[視覚系]]の結合性マッピングの概念の提出であった<ref><pubmed>1822724</pubmed></ref><ref><pubmed>1734518</pubmed></ref>。 | |||
このような研究手法は、[[w:Allen_Institute_for_Brain_Science#Allen_Mouse_Brain_Connectivity_Atlas|Allen Mouse Brain Connectivity Atlas]]([[wj:アレン脳科学研究所|Allen Brain Institute]])、[http://www.mouseconnectome.org/ Mouse Connectome Project] ([[wj:南カリフォルニア大学|南カリフォルニア大学]])、マカクサルの[http://cocomac.g-node.org/ CoCoMac](ドイツ)などで、まとめられているようなコネクトーム収集プロジェクトにつながっている。これらは、神経系の解剖学的知見と組織学的研究を組み合わせたものであり、解像度的にはµmからサブµmレベルの'''「メソスケール Mesoscale」'''のコネクトームの情報となっている。このレベルのコネクトーム構築のもう一つのアプローチとしては、このような形態学的なアプローチとともに、生理学的アプローチもある(「細胞レベルのコネクトームとコネクトミクス 生理学的方法」の項参考)。しかし、現時点では、生理学的アプローチの多くは、大規模アプローチとしては適さず、局所的な回路に焦点を当てているか、あくまで予備的な解釈に用いられているのが現状である。 | |||
メソスケールのコネクトーム情報は、神経線維の走行や終末部位についての情報を大雑把に収集したものであり、[[シナプス]]レベルでの'''「ミクロスケール Microscale」'''の神経細胞間の結合性を記述したものではない。[[化学シナプス]]および[[電気シナプス]]を形態的に観察するためには、ナノメーターレベルの解像度がある[[電子顕微鏡]]が必要である。 | |||
電子顕微鏡レベルで、ほぼ完全なコネクトーム解読に成功したのは、[[wj:シドニー・ブレンナー|Sydney Brenner]]の大学院生であったJohn Whiteらによる線虫''Caenorhabditis elegans''の神経系である(1986年)<ref><pubmed>22462104</pubmed></ref>。線虫の場合、体のサイズが小型で、神経細胞の数が少なく(雌雄同体で302個)、その結合性も個体差が少なくステレオタイプである(75%再現性があると言われる<ref><pubmed>21304930</pubmed></ref>)ので、コネクトームの構築が可能であった。一方、神経系のサイズが大きく、非常に多くの細胞と、その結合性に多様性がある脊椎動物の神経系のコネクトームの構築は極めて困難である。 | |||
ミクロスケールなコネクトーム構築には、電子顕微鏡で観察するための多数の連続切片を失うことなく作製し、撮影し、その画像を保存し、結合性を解析していくための技術開発が行われてきている(後述)。その結果、[[マウス]][[網膜]]、[[ショウジョウバエ]]視覚系、マウス[[大脳]][[視覚野]]の部分的なコネクトームなどが構築された。これらの情報を総合的に収集しているのは、[http://www.openconnectomeproject.org/ Open Connectome project]である。 | |||
一方、Olaf Spornsによるヒト・コネクトームの提唱以来、脳の機能と病態を理解するためにヒトの脳で研究されているのは、メソレベルのコネクトームより更にスケールの大きな'''「マクロスケール Macroscale」'''のコネクトームである<ref name=sporns>'''Olaf Sporns'''<br>Discovering the Human Connectome<br>''MIT Press'' 2012 ISBN 0262017903</ref><ref>'''Henry Kennedy, David C. Van Essen, Yves Christen (eds.)'''<br>Micro-, Meso- and Macro-Connectomics of the Brain<br>''Springer'', 2016 [http://link.springer.com/book/10.1007%2F978-3-319-27777-6 [OpenAccess]]</ref>。これは小型の動物ではなく、ヒト、サルなど比較的大型の動物での脳の活動部位から推定されるコネクトームである。ヒトを中心にこの情報を収集しているのは、[http://www.neuroscienceblueprint.nih.gov/connectome/ Human Connectome Project]である。これには、非侵襲な[[テンソルMRI]]などを中心に用い神経線維の走行など解剖学的な側面に注目している[http://www.humanconnectomeproject.org/ The Harvard/MGH-USC Project](かつて、UCLA)、および脳における[[fMRI]]による活動領域の検出やゲノム情報など機能的な側面に重点を置く国際プロジェクト[http://humanconnectome.org/ The WU-Minn Project]がある。いずれも、解像度が上がれば、メソスケールのコネクトームにも近づくが、非侵襲で得られる解像度は、最大でもミリメートル程度であり、侵襲的な方法で得られる解像度とは違いがある。 | |||
以上、肉眼、光学顕微鏡のレベルである「メソスケール」、電子顕微鏡レベルである「ミクロスケール」、そして非侵襲で観察される脳の構造や活動を観察する「マクロスケール」の3つの階層での断絶が、コネクトームの研究では認識されているのが現状である。しかし、例えば、深度のある組織の観察を可能にする[[多光子励起顕微鏡]]、広い範囲を高速で観察できる[[光シート顕微鏡]]、光学顕微鏡の解像度を著しく向上させる[[超高解像度顕微鏡]]([[PALM]]、[[STORM]]など)<ref><pubmed>23063602</pubmed></ref>、が改良されれば、これらのスケールの間の断絶を埋めることができる。 | |||
==細胞レベルのコネクトームとコネクトミクス== | |||
解像度の観点からは、メソスケール、ミクロスケール、マクロスケールに分類されるコネクトームであるが、特にメソスケール、ミクロスケールで注目されるのは、神経細胞レベル、神経細胞のSubcellularレベルでの神経細胞同士の結合性である。つまり、どの神経細胞同士がシナプスで結合しているか、更にシナプスが[[細胞体]]や[[樹状突起]]のどの部分に存在しているか、という中核的な情報である。更に、ミエリンに代表されるように、[[グリア細胞]]の存在も様々な機作で神経細胞間のコミュニケーションに大きな影響を与えるので、神経回路の理解には無視はできないが、ここでは神経細胞同士の結合性の問題に焦点を絞る。 | |||
神経細胞同士の結合性を決定するには、上述した歴史的に利用されてきた方法論に加えて、最近の動向としては、以下のような6つの現代的なアプローチがあるが、それぞれのアプローチに長所、短所があり、互いに相補的なアプローチとなっていくものと予想される。 | |||
====生理学的方法==== | |||
生理学的な方法を利用し、神経細胞間の結合性を調べる。これには、複数神経細胞の[[全細胞記録]]法、[[ケージド試薬|ケージド神経伝達物質]]のレーザー光刺激法、[[光遺伝学]]、[[カルシウム]]イオンのセンサー([[カルシウム感受性蛍光色素]]、[[GCaMP]]などの遺伝学的なリポーター)、[[電位感受性センサー]]などが利用される<ref><pubmed>25959713</pubmed></ref><ref><pubmed>26967281</pubmed></ref><ref><pubmed>27104976</pubmed></ref><ref><pubmed>26468193</pubmed></ref>。また、神経活動によって誘起される[[最初期遺伝子]](immediate early genes)の発現(レトロスペクティブな方法)を利用する方法もある<ref><pubmed>25558063</pubmed></ref>。 | |||
将来的に、[[哺乳類]]の神経系全体のコネクトームの解明には大規模生理学に適した方法論の開発が必要である(下記、「機能的コネクトーム」の項参考)。 | |||
====電子顕微鏡==== | |||
[[ファイル:Fly.jpg|サムネイル|250px|'''図3.ショウジョウバエ視覚系の連続切片の電子顕微鏡写真に現れた細胞をトレースすることでコネクトームを理解'''<br>[http://openconnecto.me/takemura13] <ref name=takemura />]] | |||
電子顕微鏡写真に基づき、形態的にコネクトームを構築することは、線虫のコネクトーム構築でも利用された効果的な方法であり、マウス網膜、マウス大脳皮質視覚野、ショウジョウバエ視覚系('''図3''')などで部分的なコネクトーム的な報告がなされてきている<ref><pubmed>21390125</pubmed></ref><ref><pubmed>21390124</pubmed></ref><ref name=takemura><pubmed>23925240</pubmed></ref><ref><pubmed>26232230</pubmed></ref><ref><pubmed>27015312</pubmed></ref><ref>http://www.openconnectomeproject.org/</ref>。 | |||
哺乳類の脳のようにサイズの大きな構造におけるコネクトームの構築では、薄い連続切片を失うことなく、巨大な数の電子顕微鏡写真撮影を行い、それぞれの写真上の神経細胞とその突起、結合性を、多数の写真上で逐一トレース、全体を再構築していく必要がある。その情報量は、近年の大容量デジタル情報の保存媒体とコンピューティングの発達が可能にした[[ビッグデータ]]の典型であり、各種の方法論の開発が進められてきている<ref><pubmed>24598270</pubmed></ref>。 | |||
特に重要なのは、神経細胞の電子顕微鏡写真のトレースを一箇所間違えると、全く違う神経細胞をトレースすることになるという危険性があることである。そのため、Sebastian Seungらは、網膜のコネクトームを理解するために、ゲーム感覚で、神経細胞のコネクトーム構築に、一般市民を参加させようとする[http://eyewire.org/ EyeWire]と名付けたウェッブサイトを構築している。これは、現状では、ヒトという作業者の目で電子顕微鏡写真を見て、それをトレースしていくことが、最も確実であるということから実施されているものである。 | |||
将来は、[[ディープラーニング]]を行う[[人工知能]]により、コネクトーム構築の精密作業が自動化される可能性も高い。このアプローチにおいては、神経細胞の広がりが小さく局所的なケースでは電子顕微鏡写真上での追跡も比較的容易であろうが、例えば長い神経線維でつながった神経細胞同士のコネクトームを構築することは困難である。この問題の解決には、次項の遺伝学的標識法との組み合わせを利用するのが有用であろう。 | |||
====遺伝学的標識法==== | |||
[[ファイル:Brainbow.jpg|サムネイル|250px|'''図4.Brainbow'''<br> http://www.cellimagelibrary.org/images/42753 (Creative Commons Attribution)]] | |||
神経細胞を遺伝学的なレポーター(例、[[蛍光タンパク質]])で標識し、神経細胞の形態と結合性を理解する方法論である。この方法論の特徴は、光学顕微鏡レベルでの観察が可能であるので、長い神経線維でつながった細胞同士のコネクトームの構築にも利用できることである。また、遺伝学的に標識できるため様々な神経細胞で特異的に発現するような遺伝子をドライバー(例、[[Creリコンビナーゼ]]、[[GAL4システム]])を利用して、特定の神経回路のコネクトームについての知見を深めることができる。 | |||
当初は、個々の神経細胞を蛍光タンパク質などで標識する方法が用いられていたが、コネクトーム構築には、多数の神経細胞を同時に観察する必要がある。そのために開発された方法論の1つが、[[Brainbow]]と呼ばれる技術である<ref><pubmed>18446160</pubmed></ref>(図4)。この技術は、ランダムに、異なる色を持ついくつかの蛍光タンパク質の組み合わせの発現を利用したもので、 各ニューロンは、Creリコンビナーゼによる基質となる[[lox配列]]の組み換えを利用することで、異なる色の蛍光を発色することになり、細胞体とその突起が異なる色として区別することが可能になっている。 | |||
これらの遺伝学的なツール(ドライバー、レポーター)の利用には、トランスジェニック動物、[[ノックイン動物]]、そして各種ウイルスベクターを用いることができる。中でも、神経細胞に効率的に遺伝子導入が可能である[[アデノ随伴ウイルスベクター|アデノ随伴ウイルス(AAV)ベクター]]は、広く用いられている。また、[[CRISPR/Cas9]]法の開発によって簡便になった[[ゲノム編集]]の技術の発達とともに、このような遺伝学的ツールはますます広汎に用いられるようになると予想される。 | |||
特に、遺伝的なリポーターとして、電子顕微鏡でその発現を観察できる方法は、[[コネクトーム#電子顕微鏡|上記]]の全体を電子顕微鏡で再構築する方法と併用することで、様々なコンテキストで利用可能になるので注目される<ref><pubmed>25362474</pubmed></ref>。とりわけ、最近開発された[[ARTEMIS法]]は、[[ペルオキシダーゼ]]活性を持つレポーター遺伝子を発現した神経細胞を、高品質な電子顕微鏡画像の中で識別することができる<ref><pubmed>27383271</pubmed></ref>。 | |||
また、神経細胞同士の結合を記述するコネクトームの本質の一面は、シナプスを介した神経細胞間の[[細胞接着]]の記述でもある。シナプス結合しているパートナーを調べるために、シナプス結合したパートナー細胞同士のシナプス結合を[[シナプス接着分子]]に融合させた分割GFPで検出する[[GRASP法]]という方法が開発され、線虫、ショウジョウバエなどで利用されている<ref><pubmed>22221865</pubmed></ref><ref><pubmed>22355283</pubmed></ref>。また、GRASP法の他にも、その感度の低さを補うことが可能な[[split HRP法]]が開発され、哺乳類の神経系でも利用できることが示された<ref><pubmed>27240195</pubmed></ref>。 | |||
====Trans-synapticな方法==== | |||
前項目と類似しているが、より積極的にシナプス結合している神経細胞を探査していくコネクトームの構築法である。その1つは、小麦胚レクチン(WGA)などが、[[前シナプス部]]の細胞に導入された物質が、[[細胞質]]を介して直接は繋がっていない[[後シナプス部]]にシナプスを介して移行(Trans-synaptic)するということを利用するものである。これは、歴史的には、物質そのものを注入することで行われてきたが、[[ウイルスベクター]]、[[トランスジェニックマウス]]のような形で、遺伝学的な手法を利用することが可能になった<ref><pubmed>20297728</pubmed></ref>。 | |||
特に重要なアプローチは、同様な性質を持ったウイルスベクターを利用することである<ref><pubmed>22221865</pubmed></ref>。例えば、リポーター遺伝子を有する[[狂犬病ウイルスベクター]]は、Trans-synapticな移動をし、[[逆行性]]に輸送されることが知られており、前シナプス細胞のパートナーとなる神経細胞の標識が可能である。特に、ウイルスベクターの[[エンベロープ]]タンパク質を変更することで、感染細胞を変更することができ、このような方法とCreレコンビナーゼなどを用いることでシナプス結合しているパートナーを標識できる[[TRIO法]]が最近開発されたコネクトミクスの手法の一つである<ref><pubmed>26085623</pubmed></ref><ref><pubmed>26131933</pubmed></ref>。 | |||
====生体試料観察の工夫 ==== | |||
細胞レベルでのコネクトームを理解するためには、それぞれの神経細胞とその突起の形態やシナプス局在分子を高解像度で観察することが重要である。その一つの方法は顕微鏡の改良であるが、もう一つの方法論は神経組織の生体試料を観察しやすいように処理することである(例、Array Tomography<ref><pubmed>21041404</pubmed></ref>)。特に、近年、神経組織をホールマウントでその深部まで明確に観察できるようにするための方法論の開発が活発である<ref><pubmed>26914203</pubmed></ref>。 | |||
古くからグリセロールなどを用いて組織を透明化する技術は用いられてきたが、[[wj:宮脇敦|Atsushi Miyawaki]]らによる[[Scale]]の発表以来、[[CLARITY]]、[[iDisco]]、[[SeeDB]]やその改良法など、より優れたプロトコールの開発と改良が行われてきている<ref><pubmed>25652426</pubmed></ref><ref>http://clarityresourcecenter.org/</ref>。また、[[w:Edward Boyden|Edward Boyden]]のグループは、組織そのものを拡大することができる[[Expansion Microscopy]]と名付けた方法を開発して注目されている<ref><pubmed>25592419</pubmed></ref>。 | |||
====構成論的手法==== | |||
システムを製作し、動作させることにより理解しようという方法論であり、例えば、シミュレーションやロボティクスを用いる。小規模な研究は、既に多くの神経科学者によって実施されてきている。この手法の代表的で大規模な試みが、コネクトームも考慮して、[[w:Henry Markum| Henry Markum]]らによって着手された[[Blue Brain Project]]であり、欧州を中心とする神経科学、情報科学プロジェクトである[[Human Brain Project]]の中核的プロジェクトと位置づけられている<ref>http://bluebrain.epfl.ch/</ref><ref><pubmed>26451489</pubmed></ref>。人工知能への関心の高まりとともに、今後の展開が注目される。 | |||
==マクロスケールのコネクトーム== | |||
[[ファイル:Story-vs-math Task-fMRI.png|サムネイル|250px|'''図5.fMRI'''<br>Story vs Math. Image courtesy D. Barch, M. Harms, G. Burgess for the WU-Minn HCP consortium - http://humanconnectome.org]] | |||
[[ファイル:Diffusion FA.JPG|サムネイル|250px|'''図6.dMRI'''<br>Fractional anisotropy (top), and principal diffusion directions (bottom) images from the HCP dMRI. <br>Image courtesy of the [http://humanconnectome.org WU-Minn HCP consortium]]] | |||
Olaf Spornsによるヒト・コネクトームの提唱以来、脳の機能と病態を理解するために[[ヒト]]の脳で研究されているのは、メソレベルのコネクトームより更に大きく、脳全体を視野にいれた「マクロスケール Macroscale」の巨視的なコネクトームである。これは、しばしば、様々なタスクに伴う脳の活動領域を観察する[[脳マッピング]]と同時に関心を持たれている。米国の脳科学プロジェクトである[[BRAINイニシアティブ]]の一部として実施されている国際プロジェクトで[http://www.neuroscienceblueprint.nih.gov/connectome Human Connectome Project]では、[[functional MRI]] ([[fMRI]])('''図5''')による活動領域の検出など機能的な側面に重点を置く国際プロジェクト[https://www.humanconnectome.org/ The WU-Minn Project]と、非侵襲なテンソルMRIなどを中心に用い神経線維の走行を重視する[http://www.humanconnectomeproject.org/ The Harvard/MGH-USC Project]が実施されてきた('''図6''')。いずれも、解像度が上がれば、メソスケールのコネクトームにも近づくが、非侵襲で得られる解像度は、最大でもミリメートル程度であり、侵襲的な方法で得られる解像度とは違いがある。 | |||
⾮侵襲脳計測法として、現在、ヒトの脳活動解析技術の主役となっているのは、[[fMRI]]である。fMRIでは、MRIにより、⾎流の流れと、脱酸素化[[wj:ヘモグロビン|ヘモグロビン]]の濃度変化をみる([[BOLD効果]])ことで、神経活動に伴う変動を検出する。つまり、ニューロンの活動を直接観察しているわけではないので、実際のニューロンの活動とは、秒単位の時間的なズレがある。そして、休⽌状態の⼤脳のある領域と別の領域が同調して⾃発的に変動するということが、結合状態にあるということを意味していると仮定すれば、fMRIを使って、領域間のつながりも推定することもできる(休⽌状態fMRI)。この⽅法は、領域間の結合関係、つまりコネクトーム推定の有⼒な⼿段になっている。 | |||
実際に⼤規模なデータを集めてきているのは、⽶[[wj: Washington大学|Washington大学]](ミズーリ州)と⽶[[wj:ミネソタ大学|University of Minnesota]]、そして英[[wj:オックスフォード大学|Oxford大学]]を中⼼とするコンソーシアムである。このプロジェクトでは、健常な成⼈を⽬標に、fMRI、[[PET]]、[[脳波]] ([[EEG]])、[[脳磁計]] ([[MEG]])を使うことで、それぞれの脳や⾏動関係の情報を収集してきている。 | |||
fMRIは、高度な脳機能の理解に利用されている。例えば、2016年、[[wj:カリフォルニア大学バークレー校|California大学Berkeley校]]のグループは、自然[[言語]]のそれぞれの単語と大脳皮質活動領域を関連づける脳マップを作製した<ref><pubmed>27121839</pubmed></ref>。このような脳マップの中で、コネクトームを理解することが重要である。 | |||
更に、安静状態にある脳での活動の同調の観察から、「[[Default mode network(DMN)]]」と呼ばれる複数脳領域を結ぶネットワークが意識や精神・神経疾患などについての理解の観点から注目されている<ref><pubmed>25938726</pubmed></ref>。DMNは、機能的にはネットワーク内の領域同士の何らかの結合性と関係していると考えられるが、物理的な結合性であるコネクトームとどのような関係にあるのかは、今後の課題である。 | |||
⼀⽅、米[[wj: Harvard大学|Harvard大学]]/MGH(マサチューセッツ総合病院Martinos Center)と南カリフォルニア大学(以前はカリフォルニア大学ロスアンゼルス校)を中心に進められてきたのは、[[拡散MRI]]([[dMRI]])、テンソルMRIを用いる方法である。更に、新しい方法である[[拡散スペクトラムイメージング]] ([[diffusion spectrum imaging]], [[diffusion spectrum imaging|DSI]])と[[拡散強調イメージング]] ([[High angular resolution diffusion imaging, HARDI]])は、脳内にある[[軸索]]の束となった⻑距離の接続の様⼦をマッピングする。これらの⽅法を使うと、⽣きた脳の中で、そのまま神経の⾛⾏を観察することができる。しかし、神経線維の⾛⾏をみているだけで、実際の結合性を⾒ているものではないが、今後のコネクトーム理解の方法論として期待ができる<ref>http://www.the-scientist.com/?articles.view/articleNo/41266/title/White-s-the-Matter/</ref>。 | |||
==機能的コネクトーム== | |||
2012年、[[w:Rafael Yuste|Rafael Yuste]]([[wj:コロンビア大学|Columbia大学]])、George Church(Harvard大学)らが、「The Brain Activity Map Project and the Challenge of Functional Connectomics」と題する提案をした<ref><pubmed>22726828</pubmed></ref><ref><pubmed>23470729</pubmed></ref>。民間財団である[[w:Kavli foundation|Kavli財団]]を中心としたBrain Activity Map(BAM)プロジェクトについての提案であり、主に電子顕微鏡の利用などを想定した「構造的コネクトーム(Structural connectome)」に対して、すべての神経細胞の活動のパターンを動的にマッピングする「機能的コネクトーム(functional connectome)」の解明を提唱している。 | |||
その後の技術的進歩により、その方法論については、当初イメージしていたことから徐々に変更になっているが、基本的には、神経細胞の活動に着眼して、「細胞レベルのコネクトームとコネクトミクス 生理学的方法」で上述した大規模生理学的な方法とfMRIなどの方法を、新技術の開発により達成して機能面からのコネクトームを理解しようという提案である<ref><pubmed>25654253</pubmed></ref>。 | |||
機能的コネクトームの情報が、構造的コネクトームの情報とともに不可欠なのは、線虫とは対照的に、ヒトを含めた哺乳類の神経系では構造的な[[可塑性]]が見られるということである。例えば、神経細胞の[[スパイン]]は動的な構造であり、出現したり消失したりする<ref><pubmed>14708001</pubmed></ref>。 | |||
つまり、シナプスの結合性や構造は神経系が機能する際、大きく変化しているので、構造的コネクトームだけで、神経回路の働きを把握することは全く不十分であり、神経活動の情報を含めた機能的コネクトームの考慮が大切であるという議論が背景にある。環境による変化(広義の[[エピジェネティックス]])や個体ごとの差をどのようにコネクトーム研究の中で考慮していくのかは、今後の大きな課題であろう。 | |||
==コネクトームの利用== | |||
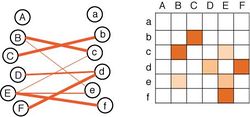
[[ファイル:Connectome.jpg|サムネイル|250px|'''図7.コネクトームを簡便に表現する模式的なイメージ図'''<br>A, aなどの各要素は、脳の各領域または各神経細胞で、オレンジ色の線や升は結合の強さの情報を示す。(左)結合ダイアグラム。(右)結合マトリックス。]] | |||
コネクトームのプロジェクトは、[[wj:ヒトゲノムプロジェクト|ヒトゲノムプロジェクト]]がそうであったように、多数の研究者や技術者の共同作業によって行なわれきており、米国のオバマ政権が提唱した脳科学研究プロジェクトであるBRAINイニシアティブなどによって積極的にサポートされてきた。また、欧州のHuman Brain Project、日本の[[Brain/MINDSプロジェクト]]などを含めた国際的な脳科学プロジェクトの主要なテーマの一つになっている。 | |||
このような電子顕微鏡やfMRIなどを用いて得られたデータは、神経科学の大規模データとして、誰でもそのデータが利用できるオープンデータとなる<ref><pubmed>24401992</pubmed></ref><ref><pubmed>25349916</pubmed></ref>。コネクトームのデータは生データとしてサーバーに保存されるが、コネクトームの簡便な表現法としては、'''図7'''に示したような結合のダイアグラムを示す方法や結合マトリックスを用いた方法がしばしば用いられてきている。 | |||
また、コネクトームに関係したプロジェクトは大きな研究費と多くの研究者の存在が必要となり、そのデータの取得と公開が神経科学者などのコミュニティに広く有用であるため、米国では[[National Brain Observatory]](国立脳天文台)設立の提案がなされ<ref><pubmed>26481036</pubmed></ref>、[[w:Argonne National Laboratory|Argonne National Laboratory]](イリノイ州)などにそのような組織を作ることが計画されている<ref>http://www.sciencemag.org/news/2015/10/neuroscientist-team-calls-national-brain-observatory</ref>。 | |||
このようなコネクトームのデータは、脳科学、神経科学全般の基礎研究情報として、ヒトと動物の脳の働き、神経回路の構造と機能の理解のために広く利用されることになる。 | |||
特に、医学領域では、[[認知症]]、[[うつ病]]、[[統合失調症]]を含めた[[精神疾患|精神]]・神経疾患、[[自閉症]]などの[[発達障害]]、神経組織の損傷などに伴う様々な病態や症状の理解の基礎データにもなる。将来はこれらの疾患の客観的な診断への活用が注目されている。そして遺伝子情報を活用した[[精密医療(Precision medicine)]]<ref>https://www.nih.gov/precision-medicine-initiative-cohort-program</ref>とともに、有効な治療法の開発と個々の患者への適用にも貢献するであろう。 | |||
更に、脳を神経回路の仕組みを模倣した人工知能の開発のための基礎情報としても役立つと思われる。コネクトームの知見を活用することで、[[心理学]]、[[言語学]]、経済学、商業、犯罪、保険、教育、政治、芸術、宗教、倫理学などのヒトの脳活動が関係した分野に、神経回路の観点から、これまでにない概念が提供されることも考えられ、その社会的な影響も大きいとの予想もある<ref name=seung>'''Sebastian Seung'''<br>Connectome: How the Brain's Wiring Makes Us Who We Are<br>''Mariner Books'', 2013 ISBN 9780547678597<br>邦訳'''セバスチャン・スン''' (著)、 '''青木 薫''' (訳)<br>コネクトーム:脳の配線はどのように「わたし」をつくり出すのか<br>草思社</ref><ref name=sporns>'''Olaf Sporns'''<br>Discovering the Human Connectome<br>''MIT Press'' 2012 ISBN 0262017903</ref><ref>'''Gary Marcus, Jeremy Freeman (Eds), May-Britt Moser, Edvard I. Moser (Contributors)'''<br>The Future of the Brain: Essays by the World's Leading Neuroscientists<br> ''Princeton University Press'', 2014, ISBN 069116276X</ref><ref>'''Michio Kaku'''<br> | |||
The Future of the Mind: The Scientific Quest to Understand, Enhance, and Empower the Mind <br>''Doubleday'', 2015, ISBN 038553082X<br>邦訳'''ミチオ・カク''' (著)、 '''斉藤 隆央''' (訳) <br>フューチャー・オブ・マインド―心の未来を科学する<br>NHK出版</ref>。 | |||
コネクトームの情報をどのように利用していくのか、その是非を含めて、社会の広い分野での議論の必要性が高まってきている。 | |||
==参考文献== | |||
<references/> | |||
2020年1月11日 (土) 23:08時点における最新版
山形方人
Harvard University
DOI:10.14931/bsd.7096 原稿受付日:2016年6月21日 原稿完成日:2016年6月29日
担当編集委員:宮川 剛(藤田保健衛生大学)
英:connectome 独:Konnektom 仏:connectome 西:conectoma 露:Коннектом 中:连接组
コネクトームは、神経系のすべての神経細胞が接続することでできた神経回路の全体を意味する。本来は、ある動物個体、あるいはある動物種の神経系において、全神経細胞の接続の総体を意味する概念である。しかしながら、神経系の一部で神経細胞が化学シナプスや電気シナプスで接続された局所的な接続ダイアグラム(Wiring diagram)に言及する場合にも、しばしば使われる。更に、神経細胞レベルでの接続だけでなく、脳の大まかな領域同士が接続する様相の説明にも使われている。コネクトミクス(connectomics)は、コネクトームを理解するための研究法、つまり実験、検出などの方法、その方法を適用した結果の解釈、そして複数の方法論を統合した研究の戦略についての概念である。
用語「コネクトーム」の起源
コネクトームは、生命科学分野で広く使われているゲノム(genome)、プロテオーム(proteome)などにならって、英語のconnect(ラテン語由来)とギリシア語の「完全、すべて、総体」を意味する接尾辞(-ome、ギリシア語-ωμα)を組み合わせた造語であり、英語の複数形は、connectomesである。
文献上、初めて使われたのは、2005年、Indiana大学のOlaf Spornsが、神経接続のマップを説明するために、ヒト・コネクトーム(Human Connectome)という概念を記述した論文である[1][2]。それとは独立して、Lausanne大学病院のPatric Hagmannが、その博士論文の中で同時期に同様に言及していた[3]。
特に、このコネクトームという概念が普及され始めたのは、2008年、Harvard大学のJeffrey W. LichtmanとJoshua R. Sanesによる共著総説論文の発表後である[4]。また、用語の定着には、Sebastian Seung(当時MIT、現Princeton大学)らのアウトリーチによる努力もあった[5]。
しかし、これらの研究者の貢献は、あくまでコネクトームという用語を提案、普及、定着させたということで認知されるべきもので、コネクトームとは呼んでいないものの、それに相当する概念はこれらの研究者が提案する以前から存在していたと考えるべきである[6]。その典型例は、1986年に発表されたJohn Whiteらによる線虫(Caenorhabditis elegans)という一つの生物の神経細胞の結合性を網羅的に記述した研究である(後述)[7][8]。
コネクトームの研究史と階層
歴史的には、簡素な解剖用具などを用いて神経線維を観察することから、目と脳など神経組織同士を接続している構造が存在することは想像されていた(例:デカルト、1677年、図1)。
19世紀末になると、Santiago Rámon y Cajal(1852-1934)が、個々の神経細胞の形態を明確に染め出すことを可能にしたGolgi染色と光学顕微鏡を用いることで、脳が多数の神経細胞とそれらの結合によって成り立っていることを提唱した(図2)。以後、神経細胞の間の結合を記述する研究は盛んに行われてきた[9][10]。Golgi染色やNissl染色などを施した連続切片を観察する時代を経て、20世紀中頃になると脳損傷後の変性神経線維をNauta法などで染色することで、神経回路の存在を確認する時代になった。
1970年ごろになると、放射性アミノ酸や、酵素(西洋ワサビペルオキシダーゼ (horseradish peroxidase; HRP))などの軸索輸送を利用することで、神経回路の観察が簡便に行われるようになった。更に、1980年代には、脂溶性carbocyanine蛍光色素などの生体結合特性を持った蛍光色素(DiIなど)、植物レクチン(wheat germ agglutinin (WGA)、phytohaemagglutinin (PHA-L)など)、ビオチン誘導体 (biocytin, Neurobiotinなど)、軸索を効率的に移動するコレラ毒素サブユニット等の高感度トレーサーが開発され、多くの研究者に汎用されるようになった。
そして、1990年代になると、蛍光顕微鏡に加えて、共焦点レーザー顕微鏡が普及し始め、デジタル画像として大規模なデータの保存と解析が行われるようになった。コネクトーム研究の観点から、このような組織学的解剖と染色によって得られた知見をまとめた重要な研究が、1991年、FellemanとDavid van Essenらによるマカクサルの視覚系の結合性マッピングの概念の提出であった[11][12]。
このような研究手法は、Allen Mouse Brain Connectivity Atlas(Allen Brain Institute)、Mouse Connectome Project (南カリフォルニア大学)、マカクサルのCoCoMac(ドイツ)などで、まとめられているようなコネクトーム収集プロジェクトにつながっている。これらは、神経系の解剖学的知見と組織学的研究を組み合わせたものであり、解像度的にはµmからサブµmレベルの「メソスケール Mesoscale」のコネクトームの情報となっている。このレベルのコネクトーム構築のもう一つのアプローチとしては、このような形態学的なアプローチとともに、生理学的アプローチもある(「細胞レベルのコネクトームとコネクトミクス 生理学的方法」の項参考)。しかし、現時点では、生理学的アプローチの多くは、大規模アプローチとしては適さず、局所的な回路に焦点を当てているか、あくまで予備的な解釈に用いられているのが現状である。
メソスケールのコネクトーム情報は、神経線維の走行や終末部位についての情報を大雑把に収集したものであり、シナプスレベルでの「ミクロスケール Microscale」の神経細胞間の結合性を記述したものではない。化学シナプスおよび電気シナプスを形態的に観察するためには、ナノメーターレベルの解像度がある電子顕微鏡が必要である。
電子顕微鏡レベルで、ほぼ完全なコネクトーム解読に成功したのは、Sydney Brennerの大学院生であったJohn Whiteらによる線虫Caenorhabditis elegansの神経系である(1986年)[13]。線虫の場合、体のサイズが小型で、神経細胞の数が少なく(雌雄同体で302個)、その結合性も個体差が少なくステレオタイプである(75%再現性があると言われる[14])ので、コネクトームの構築が可能であった。一方、神経系のサイズが大きく、非常に多くの細胞と、その結合性に多様性がある脊椎動物の神経系のコネクトームの構築は極めて困難である。
ミクロスケールなコネクトーム構築には、電子顕微鏡で観察するための多数の連続切片を失うことなく作製し、撮影し、その画像を保存し、結合性を解析していくための技術開発が行われてきている(後述)。その結果、マウス網膜、ショウジョウバエ視覚系、マウス大脳視覚野の部分的なコネクトームなどが構築された。これらの情報を総合的に収集しているのは、Open Connectome projectである。
一方、Olaf Spornsによるヒト・コネクトームの提唱以来、脳の機能と病態を理解するためにヒトの脳で研究されているのは、メソレベルのコネクトームより更にスケールの大きな「マクロスケール Macroscale」のコネクトームである[15][16]。これは小型の動物ではなく、ヒト、サルなど比較的大型の動物での脳の活動部位から推定されるコネクトームである。ヒトを中心にこの情報を収集しているのは、Human Connectome Projectである。これには、非侵襲なテンソルMRIなどを中心に用い神経線維の走行など解剖学的な側面に注目しているThe Harvard/MGH-USC Project(かつて、UCLA)、および脳におけるfMRIによる活動領域の検出やゲノム情報など機能的な側面に重点を置く国際プロジェクトThe WU-Minn Projectがある。いずれも、解像度が上がれば、メソスケールのコネクトームにも近づくが、非侵襲で得られる解像度は、最大でもミリメートル程度であり、侵襲的な方法で得られる解像度とは違いがある。
以上、肉眼、光学顕微鏡のレベルである「メソスケール」、電子顕微鏡レベルである「ミクロスケール」、そして非侵襲で観察される脳の構造や活動を観察する「マクロスケール」の3つの階層での断絶が、コネクトームの研究では認識されているのが現状である。しかし、例えば、深度のある組織の観察を可能にする多光子励起顕微鏡、広い範囲を高速で観察できる光シート顕微鏡、光学顕微鏡の解像度を著しく向上させる超高解像度顕微鏡(PALM、STORMなど)[17]、が改良されれば、これらのスケールの間の断絶を埋めることができる。
細胞レベルのコネクトームとコネクトミクス
解像度の観点からは、メソスケール、ミクロスケール、マクロスケールに分類されるコネクトームであるが、特にメソスケール、ミクロスケールで注目されるのは、神経細胞レベル、神経細胞のSubcellularレベルでの神経細胞同士の結合性である。つまり、どの神経細胞同士がシナプスで結合しているか、更にシナプスが細胞体や樹状突起のどの部分に存在しているか、という中核的な情報である。更に、ミエリンに代表されるように、グリア細胞の存在も様々な機作で神経細胞間のコミュニケーションに大きな影響を与えるので、神経回路の理解には無視はできないが、ここでは神経細胞同士の結合性の問題に焦点を絞る。
神経細胞同士の結合性を決定するには、上述した歴史的に利用されてきた方法論に加えて、最近の動向としては、以下のような6つの現代的なアプローチがあるが、それぞれのアプローチに長所、短所があり、互いに相補的なアプローチとなっていくものと予想される。
生理学的方法
生理学的な方法を利用し、神経細胞間の結合性を調べる。これには、複数神経細胞の全細胞記録法、ケージド神経伝達物質のレーザー光刺激法、光遺伝学、カルシウムイオンのセンサー(カルシウム感受性蛍光色素、GCaMPなどの遺伝学的なリポーター)、電位感受性センサーなどが利用される[18][19][20][21]。また、神経活動によって誘起される最初期遺伝子(immediate early genes)の発現(レトロスペクティブな方法)を利用する方法もある[22]。
将来的に、哺乳類の神経系全体のコネクトームの解明には大規模生理学に適した方法論の開発が必要である(下記、「機能的コネクトーム」の項参考)。
電子顕微鏡
電子顕微鏡写真に基づき、形態的にコネクトームを構築することは、線虫のコネクトーム構築でも利用された効果的な方法であり、マウス網膜、マウス大脳皮質視覚野、ショウジョウバエ視覚系(図3)などで部分的なコネクトーム的な報告がなされてきている[24][25][23][26][27][28]。
哺乳類の脳のようにサイズの大きな構造におけるコネクトームの構築では、薄い連続切片を失うことなく、巨大な数の電子顕微鏡写真撮影を行い、それぞれの写真上の神経細胞とその突起、結合性を、多数の写真上で逐一トレース、全体を再構築していく必要がある。その情報量は、近年の大容量デジタル情報の保存媒体とコンピューティングの発達が可能にしたビッグデータの典型であり、各種の方法論の開発が進められてきている[29]。
特に重要なのは、神経細胞の電子顕微鏡写真のトレースを一箇所間違えると、全く違う神経細胞をトレースすることになるという危険性があることである。そのため、Sebastian Seungらは、網膜のコネクトームを理解するために、ゲーム感覚で、神経細胞のコネクトーム構築に、一般市民を参加させようとするEyeWireと名付けたウェッブサイトを構築している。これは、現状では、ヒトという作業者の目で電子顕微鏡写真を見て、それをトレースしていくことが、最も確実であるということから実施されているものである。
将来は、ディープラーニングを行う人工知能により、コネクトーム構築の精密作業が自動化される可能性も高い。このアプローチにおいては、神経細胞の広がりが小さく局所的なケースでは電子顕微鏡写真上での追跡も比較的容易であろうが、例えば長い神経線維でつながった神経細胞同士のコネクトームを構築することは困難である。この問題の解決には、次項の遺伝学的標識法との組み合わせを利用するのが有用であろう。
遺伝学的標識法
神経細胞を遺伝学的なレポーター(例、蛍光タンパク質)で標識し、神経細胞の形態と結合性を理解する方法論である。この方法論の特徴は、光学顕微鏡レベルでの観察が可能であるので、長い神経線維でつながった細胞同士のコネクトームの構築にも利用できることである。また、遺伝学的に標識できるため様々な神経細胞で特異的に発現するような遺伝子をドライバー(例、Creリコンビナーゼ、GAL4システム)を利用して、特定の神経回路のコネクトームについての知見を深めることができる。
当初は、個々の神経細胞を蛍光タンパク質などで標識する方法が用いられていたが、コネクトーム構築には、多数の神経細胞を同時に観察する必要がある。そのために開発された方法論の1つが、Brainbowと呼ばれる技術である[30](図4)。この技術は、ランダムに、異なる色を持ついくつかの蛍光タンパク質の組み合わせの発現を利用したもので、 各ニューロンは、Creリコンビナーゼによる基質となるlox配列の組み換えを利用することで、異なる色の蛍光を発色することになり、細胞体とその突起が異なる色として区別することが可能になっている。
これらの遺伝学的なツール(ドライバー、レポーター)の利用には、トランスジェニック動物、ノックイン動物、そして各種ウイルスベクターを用いることができる。中でも、神経細胞に効率的に遺伝子導入が可能であるアデノ随伴ウイルス(AAV)ベクターは、広く用いられている。また、CRISPR/Cas9法の開発によって簡便になったゲノム編集の技術の発達とともに、このような遺伝学的ツールはますます広汎に用いられるようになると予想される。
特に、遺伝的なリポーターとして、電子顕微鏡でその発現を観察できる方法は、上記の全体を電子顕微鏡で再構築する方法と併用することで、様々なコンテキストで利用可能になるので注目される[31]。とりわけ、最近開発されたARTEMIS法は、ペルオキシダーゼ活性を持つレポーター遺伝子を発現した神経細胞を、高品質な電子顕微鏡画像の中で識別することができる[32]。
また、神経細胞同士の結合を記述するコネクトームの本質の一面は、シナプスを介した神経細胞間の細胞接着の記述でもある。シナプス結合しているパートナーを調べるために、シナプス結合したパートナー細胞同士のシナプス結合をシナプス接着分子に融合させた分割GFPで検出するGRASP法という方法が開発され、線虫、ショウジョウバエなどで利用されている[33][34]。また、GRASP法の他にも、その感度の低さを補うことが可能なsplit HRP法が開発され、哺乳類の神経系でも利用できることが示された[35]。
Trans-synapticな方法
前項目と類似しているが、より積極的にシナプス結合している神経細胞を探査していくコネクトームの構築法である。その1つは、小麦胚レクチン(WGA)などが、前シナプス部の細胞に導入された物質が、細胞質を介して直接は繋がっていない後シナプス部にシナプスを介して移行(Trans-synaptic)するということを利用するものである。これは、歴史的には、物質そのものを注入することで行われてきたが、ウイルスベクター、トランスジェニックマウスのような形で、遺伝学的な手法を利用することが可能になった[36]。
特に重要なアプローチは、同様な性質を持ったウイルスベクターを利用することである[37]。例えば、リポーター遺伝子を有する狂犬病ウイルスベクターは、Trans-synapticな移動をし、逆行性に輸送されることが知られており、前シナプス細胞のパートナーとなる神経細胞の標識が可能である。特に、ウイルスベクターのエンベロープタンパク質を変更することで、感染細胞を変更することができ、このような方法とCreレコンビナーゼなどを用いることでシナプス結合しているパートナーを標識できるTRIO法が最近開発されたコネクトミクスの手法の一つである[38][39]。
生体試料観察の工夫
細胞レベルでのコネクトームを理解するためには、それぞれの神経細胞とその突起の形態やシナプス局在分子を高解像度で観察することが重要である。その一つの方法は顕微鏡の改良であるが、もう一つの方法論は神経組織の生体試料を観察しやすいように処理することである(例、Array Tomography[40])。特に、近年、神経組織をホールマウントでその深部まで明確に観察できるようにするための方法論の開発が活発である[41]。
古くからグリセロールなどを用いて組織を透明化する技術は用いられてきたが、Atsushi MiyawakiらによるScaleの発表以来、CLARITY、iDisco、SeeDBやその改良法など、より優れたプロトコールの開発と改良が行われてきている[42][43]。また、Edward Boydenのグループは、組織そのものを拡大することができるExpansion Microscopyと名付けた方法を開発して注目されている[44]。
構成論的手法
システムを製作し、動作させることにより理解しようという方法論であり、例えば、シミュレーションやロボティクスを用いる。小規模な研究は、既に多くの神経科学者によって実施されてきている。この手法の代表的で大規模な試みが、コネクトームも考慮して、 Henry Markumらによって着手されたBlue Brain Projectであり、欧州を中心とする神経科学、情報科学プロジェクトであるHuman Brain Projectの中核的プロジェクトと位置づけられている[45][46]。人工知能への関心の高まりとともに、今後の展開が注目される。
マクロスケールのコネクトーム

Story vs Math. Image courtesy D. Barch, M. Harms, G. Burgess for the WU-Minn HCP consortium - http://humanconnectome.org
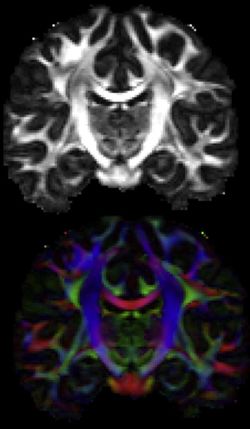
Fractional anisotropy (top), and principal diffusion directions (bottom) images from the HCP dMRI.
Image courtesy of the WU-Minn HCP consortium
Olaf Spornsによるヒト・コネクトームの提唱以来、脳の機能と病態を理解するためにヒトの脳で研究されているのは、メソレベルのコネクトームより更に大きく、脳全体を視野にいれた「マクロスケール Macroscale」の巨視的なコネクトームである。これは、しばしば、様々なタスクに伴う脳の活動領域を観察する脳マッピングと同時に関心を持たれている。米国の脳科学プロジェクトであるBRAINイニシアティブの一部として実施されている国際プロジェクトでHuman Connectome Projectでは、functional MRI (fMRI)(図5)による活動領域の検出など機能的な側面に重点を置く国際プロジェクトThe WU-Minn Projectと、非侵襲なテンソルMRIなどを中心に用い神経線維の走行を重視するThe Harvard/MGH-USC Projectが実施されてきた(図6)。いずれも、解像度が上がれば、メソスケールのコネクトームにも近づくが、非侵襲で得られる解像度は、最大でもミリメートル程度であり、侵襲的な方法で得られる解像度とは違いがある。
⾮侵襲脳計測法として、現在、ヒトの脳活動解析技術の主役となっているのは、fMRIである。fMRIでは、MRIにより、⾎流の流れと、脱酸素化ヘモグロビンの濃度変化をみる(BOLD効果)ことで、神経活動に伴う変動を検出する。つまり、ニューロンの活動を直接観察しているわけではないので、実際のニューロンの活動とは、秒単位の時間的なズレがある。そして、休⽌状態の⼤脳のある領域と別の領域が同調して⾃発的に変動するということが、結合状態にあるということを意味していると仮定すれば、fMRIを使って、領域間のつながりも推定することもできる(休⽌状態fMRI)。この⽅法は、領域間の結合関係、つまりコネクトーム推定の有⼒な⼿段になっている。
実際に⼤規模なデータを集めてきているのは、⽶Washington大学(ミズーリ州)と⽶University of Minnesota、そして英Oxford大学を中⼼とするコンソーシアムである。このプロジェクトでは、健常な成⼈を⽬標に、fMRI、PET、脳波 (EEG)、脳磁計 (MEG)を使うことで、それぞれの脳や⾏動関係の情報を収集してきている。
fMRIは、高度な脳機能の理解に利用されている。例えば、2016年、California大学Berkeley校のグループは、自然言語のそれぞれの単語と大脳皮質活動領域を関連づける脳マップを作製した[47]。このような脳マップの中で、コネクトームを理解することが重要である。
更に、安静状態にある脳での活動の同調の観察から、「Default mode network(DMN)」と呼ばれる複数脳領域を結ぶネットワークが意識や精神・神経疾患などについての理解の観点から注目されている[48]。DMNは、機能的にはネットワーク内の領域同士の何らかの結合性と関係していると考えられるが、物理的な結合性であるコネクトームとどのような関係にあるのかは、今後の課題である。
⼀⽅、米Harvard大学/MGH(マサチューセッツ総合病院Martinos Center)と南カリフォルニア大学(以前はカリフォルニア大学ロスアンゼルス校)を中心に進められてきたのは、拡散MRI(dMRI)、テンソルMRIを用いる方法である。更に、新しい方法である拡散スペクトラムイメージング (diffusion spectrum imaging, DSI)と拡散強調イメージング (High angular resolution diffusion imaging, HARDI)は、脳内にある軸索の束となった⻑距離の接続の様⼦をマッピングする。これらの⽅法を使うと、⽣きた脳の中で、そのまま神経の⾛⾏を観察することができる。しかし、神経線維の⾛⾏をみているだけで、実際の結合性を⾒ているものではないが、今後のコネクトーム理解の方法論として期待ができる[49]。
機能的コネクトーム
2012年、Rafael Yuste(Columbia大学)、George Church(Harvard大学)らが、「The Brain Activity Map Project and the Challenge of Functional Connectomics」と題する提案をした[50][51]。民間財団であるKavli財団を中心としたBrain Activity Map(BAM)プロジェクトについての提案であり、主に電子顕微鏡の利用などを想定した「構造的コネクトーム(Structural connectome)」に対して、すべての神経細胞の活動のパターンを動的にマッピングする「機能的コネクトーム(functional connectome)」の解明を提唱している。
その後の技術的進歩により、その方法論については、当初イメージしていたことから徐々に変更になっているが、基本的には、神経細胞の活動に着眼して、「細胞レベルのコネクトームとコネクトミクス 生理学的方法」で上述した大規模生理学的な方法とfMRIなどの方法を、新技術の開発により達成して機能面からのコネクトームを理解しようという提案である[52]。
機能的コネクトームの情報が、構造的コネクトームの情報とともに不可欠なのは、線虫とは対照的に、ヒトを含めた哺乳類の神経系では構造的な可塑性が見られるということである。例えば、神経細胞のスパインは動的な構造であり、出現したり消失したりする[53]。
つまり、シナプスの結合性や構造は神経系が機能する際、大きく変化しているので、構造的コネクトームだけで、神経回路の働きを把握することは全く不十分であり、神経活動の情報を含めた機能的コネクトームの考慮が大切であるという議論が背景にある。環境による変化(広義のエピジェネティックス)や個体ごとの差をどのようにコネクトーム研究の中で考慮していくのかは、今後の大きな課題であろう。
コネクトームの利用
コネクトームのプロジェクトは、ヒトゲノムプロジェクトがそうであったように、多数の研究者や技術者の共同作業によって行なわれきており、米国のオバマ政権が提唱した脳科学研究プロジェクトであるBRAINイニシアティブなどによって積極的にサポートされてきた。また、欧州のHuman Brain Project、日本のBrain/MINDSプロジェクトなどを含めた国際的な脳科学プロジェクトの主要なテーマの一つになっている。
このような電子顕微鏡やfMRIなどを用いて得られたデータは、神経科学の大規模データとして、誰でもそのデータが利用できるオープンデータとなる[54][55]。コネクトームのデータは生データとしてサーバーに保存されるが、コネクトームの簡便な表現法としては、図7に示したような結合のダイアグラムを示す方法や結合マトリックスを用いた方法がしばしば用いられてきている。
また、コネクトームに関係したプロジェクトは大きな研究費と多くの研究者の存在が必要となり、そのデータの取得と公開が神経科学者などのコミュニティに広く有用であるため、米国ではNational Brain Observatory(国立脳天文台)設立の提案がなされ[56]、Argonne National Laboratory(イリノイ州)などにそのような組織を作ることが計画されている[57]。
このようなコネクトームのデータは、脳科学、神経科学全般の基礎研究情報として、ヒトと動物の脳の働き、神経回路の構造と機能の理解のために広く利用されることになる。
特に、医学領域では、認知症、うつ病、統合失調症を含めた精神・神経疾患、自閉症などの発達障害、神経組織の損傷などに伴う様々な病態や症状の理解の基礎データにもなる。将来はこれらの疾患の客観的な診断への活用が注目されている。そして遺伝子情報を活用した精密医療(Precision medicine)[58]とともに、有効な治療法の開発と個々の患者への適用にも貢献するであろう。
更に、脳を神経回路の仕組みを模倣した人工知能の開発のための基礎情報としても役立つと思われる。コネクトームの知見を活用することで、心理学、言語学、経済学、商業、犯罪、保険、教育、政治、芸術、宗教、倫理学などのヒトの脳活動が関係した分野に、神経回路の観点から、これまでにない概念が提供されることも考えられ、その社会的な影響も大きいとの予想もある[5][15][59][60]。
コネクトームの情報をどのように利用していくのか、その是非を含めて、社会の広い分野での議論の必要性が高まってきている。
参考文献
- ↑
Sporns, O., Tononi, G., & Kötter, R. (2005).
The human connectome: A structural description of the human brain. PLoS computational biology, 1(4), e42. [PubMed:16201007] [PMC] [WorldCat] [DOI] - ↑ Olaf Sporns
Connectome
Scholarpedia, 5(2):5584, 2010. [DOI] - ↑ Patric Hagmann
From diffusion MRI to brain connectomics [PhD Thesis].
Lausanne: Ecole Polytechnique Fédérale de Lausanne (EPFL). 2005 [DOI]. - ↑
Lichtman, J.W., & Sanes, J.R. (2008).
Ome sweet ome: what can the genome tell us about the connectome? Current opinion in neurobiology, 18(3), 346-53. [PubMed:18801435] [PMC] [WorldCat] [DOI] - ↑ 5.0 5.1 Sebastian Seung
Connectome: How the Brain's Wiring Makes Us Who We Are
Mariner Books, 2013 ISBN 9780547678597
邦訳セバスチャン・スン (著)、 青木 薫 (訳)
コネクトーム:脳の配線はどのように「わたし」をつくり出すのか
草思社 - ↑
Bargmann, C.I., & Marder, E. (2013).
From the connectome to brain function. Nature methods, 10(6), 483-90. [PubMed:23866325] [WorldCat] - ↑
White, J.G., Southgate, E., Thomson, J.N., & Brenner, S. (1986).
The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 314(1165), 1-340. [PubMed:22462104] [WorldCat] [DOI] - ↑
Emmons, S.W. (2015).
The beginning of connectomics: a commentary on White et al. (1986) 'The structure of the nervous system of the nematode Caenorhabditis elegans'. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 370(1666). [PubMed:25750233] [PMC] [WorldCat] [DOI] - ↑
Lanciego, J.L., & Wouterlood, F.G. (2011).
A half century of experimental neuroanatomical tracing. Journal of chemical neuroanatomy, 42(3), 157-83. [PubMed:21782932] [WorldCat] [DOI] - ↑
Swanson, L.W., & Lichtman, J.W. (2016).
From Cajal to Connectome and Beyond. Annual review of neuroscience, 39, 197-216. [PubMed:27442070] [WorldCat] [DOI] - ↑
Felleman, D.J., & Van Essen, D.C. (1991).
Distributed hierarchical processing in the primate cerebral cortex. Cerebral cortex (New York, N.Y. : 1991), 1(1), 1-47. [PubMed:1822724] [WorldCat] [DOI] - ↑
Van Essen, D.C., Anderson, C.H., & Felleman, D.J. (1992).
Information processing in the primate visual system: an integrated systems perspective. Science (New York, N.Y.), 255(5043), 419-23. [PubMed:1734518] [WorldCat] [DOI] - ↑
White, J.G., Southgate, E., Thomson, J.N., & Brenner, S. (1986).
The structure of the nervous system of the nematode Caenorhabditis elegans. Philosophical transactions of the Royal Society of London. Series B, Biological sciences, 314(1165), 1-340. [PubMed:22462104] [WorldCat] [DOI] - ↑
Varshney, L.R., Chen, B.L., Paniagua, E., Hall, D.H., & Chklovskii, D.B. (2011).
Structural properties of the Caenorhabditis elegans neuronal network. PLoS computational biology, 7(2), e1001066. [PubMed:21304930] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1 Olaf Sporns
Discovering the Human Connectome
MIT Press 2012 ISBN 0262017903 - ↑ Henry Kennedy, David C. Van Essen, Yves Christen (eds.)
Micro-, Meso- and Macro-Connectomics of the Brain
Springer, 2016 [OpenAccess] - ↑
Tønnesen, J., & Nägerl, U.V. (2013).
Superresolution imaging for neuroscience. Experimental neurology, 242, 33-40. [PubMed:23063602] [WorldCat] [DOI] - ↑
Battaglia, F.P., & Schnitzer, M.J. (2015).
Editorial overview: Large-scale recording technology: Scaling up neuroscience. Current opinion in neurobiology, 32, iv-vi. [PubMed:25959713] [WorldCat] [DOI] - ↑
Lerner, T.N., Ye, L., & Deisseroth, K. (2016).
Communication in Neural Circuits: Tools, Opportunities, and Challenges. Cell, 164(6), 1136-1150. [PubMed:26967281] [PMC] [WorldCat] [DOI] - ↑
Rajasethupathy, P., Ferenczi, E., & Deisseroth, K. (2016).
Targeting Neural Circuits. Cell, 165(3), 524-34. [PubMed:27104976] [PMC] [WorldCat] [DOI] - ↑
Emiliani, V., Cohen, A.E., Deisseroth, K., & Häusser, M. (2015).
All-Optical Interrogation of Neural Circuits. The Journal of neuroscience : the official journal of the Society for Neuroscience, 35(41), 13917-26. [PubMed:26468193] [PMC] [WorldCat] [DOI] - ↑
Kim, Y., Venkataraju, K.U., Pradhan, K., Mende, C., Taranda, J., Turaga, S.C., ..., & Osten, P. (2015).
Mapping social behavior-induced brain activation at cellular resolution in the mouse. Cell reports, 10(2), 292-305. [PubMed:25558063] [PMC] [WorldCat] [DOI] - ↑ 23.0 23.1
Takemura, S.Y., Bharioke, A., Lu, Z., Nern, A., Vitaladevuni, S., Rivlin, P.K., ..., & Chklovskii, D.B. (2013).
A visual motion detection circuit suggested by Drosophila connectomics. Nature, 500(7461), 175-81. [PubMed:23925240] [PMC] [WorldCat] [DOI] - ↑
Briggman, K.L., Helmstaedter, M., & Denk, W. (2011).
Wiring specificity in the direction-selectivity circuit of the retina. Nature, 471(7337), 183-8. [PubMed:21390125] [WorldCat] [DOI] - ↑
Bock, D.D., Lee, W.C., Kerlin, A.M., Andermann, M.L., Hood, G., Wetzel, A.W., ..., & Reid, R.C. (2011).
Network anatomy and in vivo physiology of visual cortical neurons. Nature, 471(7337), 177-82. [PubMed:21390124] [PMC] [WorldCat] [DOI] - ↑
Kasthuri, N., Hayworth, K.J., Berger, D.R., Schalek, R.L., Conchello, J.A., Knowles-Barley, S., ..., & Lichtman, J.W. (2015).
Saturated Reconstruction of a Volume of Neocortex. Cell, 162(3), 648-61. [PubMed:26232230] [WorldCat] [DOI] - ↑
Morgan, J.L., Berger, D.R., Wetzel, A.W., & Lichtman, J.W. (2016).
The Fuzzy Logic of Network Connectivity in Mouse Visual Thalamus. Cell, 165(1), 192-206. [PubMed:27015312] [PMC] [WorldCat] [DOI] - ↑ http://www.openconnectomeproject.org/
- ↑
Plaza, S.M., Scheffer, L.K., & Chklovskii, D.B. (2014).
Toward large-scale connectome reconstructions. Current opinion in neurobiology, 25, 201-10. [PubMed:24598270] [WorldCat] [DOI] - ↑
Lichtman, J.W., Livet, J., & Sanes, J.R. (2008).
A technicolour approach to the connectome. Nature reviews. Neuroscience, 9(6), 417-22. [PubMed:18446160] [PMC] [WorldCat] [DOI] - ↑
Atasoy, D., Betley, J.N., Li, W.P., Su, H.H., Sertel, S.M., Scheffer, L.K., ..., & Sternson, S.M. (2014).
A genetically specified connectomics approach applied to long-range feeding regulatory circuits. Nature neuroscience, 17(12), 1830-9. [PubMed:25362474] [PMC] [WorldCat] [DOI] - ↑
Joesch, M., Mankus, D., Yamagata, M., Shahbazi, A., Schalek, R., Suissa-Peleg, A., ..., & Sanes, J.R. (2016).
Reconstruction of genetically identified neurons imaged by serial-section electron microscopy. eLife, 5. [PubMed:27383271] [PMC] [WorldCat] [DOI] - ↑
Wickersham, I.R., & Feinberg, E.H. (2012).
New technologies for imaging synaptic partners. Current opinion in neurobiology, 22(1), 121-7. [PubMed:22221865] [WorldCat] [DOI] - ↑
Yamagata, M., & Sanes, J.R. (2012).
Transgenic strategy for identifying synaptic connections in mice by fluorescence complementation (GRASP). Frontiers in molecular neuroscience, 5, 18. [PubMed:22355283] [PMC] [WorldCat] [DOI] - ↑
Martell, J.D., Yamagata, M., Deerinck, T.J., Phan, S., Kwa, C.G., Ellisman, M.H., ..., & Ting, A.Y. (2016).
A split horseradish peroxidase for the detection of intercellular protein-protein interactions and sensitive visualization of synapses. Nature biotechnology, 34(7), 774-80. [PubMed:27240195] [PMC] [WorldCat] [DOI] - ↑
Yoshihara, Y. (2010).
[Visualization of selective transsynaptic neural pathways using a genetic method]. Brain and nerve = Shinkei kenkyu no shinpo, 62(3), 233-42. [PubMed:20297728] [WorldCat] - ↑
Wickersham, I.R., & Feinberg, E.H. (2012).
New technologies for imaging synaptic partners. Current opinion in neurobiology, 22(1), 121-7. [PubMed:22221865] [WorldCat] [DOI] - ↑
Callaway, E.M., & Luo, L. (2015).
Monosynaptic Circuit Tracing with Glycoprotein-Deleted Rabies Viruses. The Journal of neuroscience : the official journal of the Society for Neuroscience, 35(24), 8979-85. [PubMed:26085623] [PMC] [WorldCat] [DOI] - ↑
Schwarz, L.A., Miyamichi, K., Gao, X.J., Beier, K.T., Weissbourd, B., DeLoach, K.E., ..., & Luo, L. (2015).
Viral-genetic tracing of the input-output organization of a central noradrenaline circuit. Nature, 524(7563), 88-92. [PubMed:26131933] [PMC] [WorldCat] [DOI] - ↑
Micheva, K.D., O'Rourke, N., Busse, B., & Smith, S.J. (2010).
Array tomography: high-resolution three-dimensional immunofluorescence. Cold Spring Harbor protocols, 2010(11), pdb.top89. [PubMed:21041404] [WorldCat] [DOI] - ↑
Marx, V. (2016).
Optimizing probes to image cleared tissue. Nature methods, 13(3), 205-9. [PubMed:26914203] [WorldCat] [DOI] - ↑
Miyawaki, A. (2015).
Brain clearing for connectomics. Microscopy (Oxford, England), 64(1), 5-8. [PubMed:25652426] [WorldCat] [DOI] - ↑ http://clarityresourcecenter.org/
- ↑
Chen, F., Tillberg, P.W., & Boyden, E.S. (2015).
Optical imaging. Expansion microscopy. Science (New York, N.Y.), 347(6221), 543-8. [PubMed:25592419] [PMC] [WorldCat] [DOI] - ↑ http://bluebrain.epfl.ch/
- ↑
Markram, H., Muller, E., Ramaswamy, S., Reimann, M.W., Abdellah, M., Sanchez, C.A., ..., & Schürmann, F. (2015).
Reconstruction and Simulation of Neocortical Microcircuitry. Cell, 163(2), 456-92. [PubMed:26451489] [WorldCat] [DOI] - ↑
Huth, A.G., de Heer, W.A., Griffiths, T.L., Theunissen, F.E., & Gallant, J.L. (2016).
Natural speech reveals the semantic maps that tile human cerebral cortex. Nature, 532(7600), 453-8. [PubMed:27121839] [PMC] [WorldCat] [DOI] - ↑
Raichle, M.E. (2015).
The brain's default mode network. Annual review of neuroscience, 38, 433-47. [PubMed:25938726] [WorldCat] [DOI] - ↑ http://www.the-scientist.com/?articles.view/articleNo/41266/title/White-s-the-Matter/
- ↑
Alivisatos, A.P., Chun, M., Church, G.M., Greenspan, R.J., Roukes, M.L., & Yuste, R. (2012).
The brain activity map project and the challenge of functional connectomics. Neuron, 74(6), 970-4. [PubMed:22726828] [PMC] [WorldCat] [DOI] - ↑
Alivisatos, A.P., Chun, M., Church, G.M., Deisseroth, K., Donoghue, J.P., Greenspan, R.J., ..., & Yuste, R. (2013).
Neuroscience. The brain activity map. Science (New York, N.Y.), 339(6125), 1284-5. [PubMed:23470729] [PMC] [WorldCat] [DOI] - ↑
Keller, P.J., & Ahrens, M.B. (2015).
Visualizing whole-brain activity and development at the single-cell level using light-sheet microscopy. Neuron, 85(3), 462-83. [PubMed:25654253] [WorldCat] [DOI] - ↑
Yuste, R., & Bonhoeffer, T. (2004).
Genesis of dendritic spines: insights from ultrastructural and imaging studies. Nature reviews. Neuroscience, 5(1), 24-34. [PubMed:14708001] [WorldCat] [DOI] - ↑
Burns, R., Roncal, W.G., Kleissas, D., Lillaney, K., Manavalan, P., Perlman, E., ..., & Vogelstein, R.J. (2013).
The Open Connectome Project Data Cluster: Scalable Analysis and Vision for High-Throughput Neuroscience. Scientific and statistical database management : International Conference, SSDBM ... : proceedings. International Conference on Scientific and Statistical Database Management. [PubMed:24401992] [PMC] [DOI] - ↑
Poldrack, R.A., & Gorgolewski, K.J. (2014).
Making big data open: data sharing in neuroimaging. Nature neuroscience, 17(11), 1510-7. [PubMed:25349916] [WorldCat] [DOI] - ↑
Alivisatos, A.P., Chun, M., Church, G.M., Greenspan, R.J., Roukes, M.L., & Yuste, R. (2015).
A National Network of Neurotechnology Centers for the BRAIN Initiative. Neuron, 88(3), 445-8. [PubMed:26481036] [PMC] [WorldCat] [DOI] - ↑ http://www.sciencemag.org/news/2015/10/neuroscientist-team-calls-national-brain-observatory
- ↑ https://www.nih.gov/precision-medicine-initiative-cohort-program
- ↑ Gary Marcus, Jeremy Freeman (Eds), May-Britt Moser, Edvard I. Moser (Contributors)
The Future of the Brain: Essays by the World's Leading Neuroscientists
Princeton University Press, 2014, ISBN 069116276X - ↑ Michio Kaku
The Future of the Mind: The Scientific Quest to Understand, Enhance, and Empower the Mind
Doubleday, 2015, ISBN 038553082X
邦訳ミチオ・カク (著)、 斉藤 隆央 (訳)
フューチャー・オブ・マインド―心の未来を科学する
NHK出版