ヒストンメチル基転移酵素
久能 修、中島 欽一
九州大学 大学院医学研究院 応用幹細胞医科学部門 基盤幹細胞学分野
DOI:10.14931/bsd.4479 原稿受付日:2023年5月13日 原稿完成日:2023年5月21日
担当編集委員:古屋敷 智之(神戸大学大学院医学研究科・医学部 薬理学分野)
英:histone methyl transferase
英略称:HMT
ヒストンメチル基転移酵素はヒストンは特定のアミノ酸残基をメチル化する。それにより、遺伝子発現、細胞周期、ゲノムの安定性、核構造の調節など、細胞の重要な機能が制御される。ヒストンメチル基転移酵素はヒストンリジンメチル基転移酵素(KMT)とタンパク質アルギニンメチル基転移酵素(PRMT)の2つに分類され、ヒストンリジンメチル基転移酵素はさらにSETドメイン型と非SETドメイン型に分類される。神経系では、ヒストンメチル基転移酵素はニューロンの発生、成熟、機能、維持に関与しており、その異常な活性化または不活性化は、神経疾患に繋がる。
歴史・背景
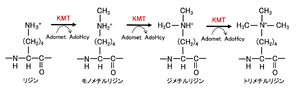
AdoMet: S-adenosyl-L-methionine、AdoHcy: S adenosyl-L-homocysteine。文献[1] より改変。
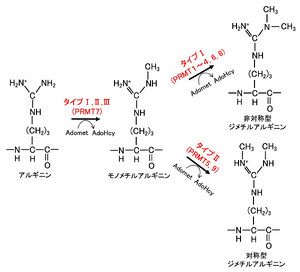
AdoMet: S-adenosyl-L-methionine、AdoHcy: S adenosyl-L-homocysteine。文献[1] より改変。
1964年にゲノムDNAのmRNAへの転写がヒストンタンパク質のリジンのε-アミノ基のメチル化によって調節されることが見出された[2](図1)。しかし、Thomas Jenuweinらにより、最初のヒストンリジンメチル基転移酵素であるヒトおよびマウス suppressor of variegation 3-9 homolog1 (SUV39H1、KMT1Aとしても知られる) が報告されたのは2000年になってからのことであった。その後、ショウジョウバエの3つのタンパク質、Su(var)3-9、Enhancer of Zeste、Trithoraxが共通して持つSETドメインとのホモロジー検索により、多くのヒストンリジンメチル基転移酵素が同定された[3][4]。
一方、タンパク質アルギニンメチル基転移酵素は、ヒストンやその他のタンパク質のアルギニン残基のメチル化を触媒することによって様々な生物学的プロセスに関与する(図2)。ヒストンタンパク質のアルギニン残基のメチル化が発見された1967年以降から現在までに哺乳類で9つが同定されている。
いずれもS-アデノシル-L-メチオニン (S-adenosyl-L-methionine, SAM/Adomet)を補因子として、リジンあるいはアルギニン残基にメチル基を転移させる。酵母からヒトまで広く真核生物で進化的に保存されている[5]。
分類
ドメイン構造、標的分子となるヒストン残基とメチル化の数で分類される(表1)。
ヒストンリジンメチル基転移酵素
SETドメイン型、非SETドメイン型の2つのクラスから構成される。
SETドメイン型
SETドメインを持ち、ヒストンリジンメチル基転移酵素の大部分を占める。
SETドメインは、一次配列のN末端とC末端によって形成される2つの領域からなり、それぞれSET-NとSET-Cという。それぞれの領域には、3〜4本の短いβ-ストランド、短いヘリックス、およびこれらの二次構造をつなぐいくつかのループが含まれている[6]。またSETに隣接する領域は、SETドメイン構造の安定化とともにメチル化酵素の活性に必要である[6]。
非SETドメイン型
SETドメインの代わりにseven-beta-strand (7βS)ドメインを持ち、このドメインがヌクレオソーム表面に露出しているヒストン H3 の79番目のリジン残基(H3K79)に対するメチル基供与活性を持つ[7][8][9]。Dot1 like protein (DOT1L。KMT4としても知られる)の1種類のみが属する[10][11]。
タンパク質アルギニンメチル基転移酵素
共通のメチルトランスフェラーゼ(MTase)ドメインを持つ。また一部には、リン酸化や酸化などの修飾を介してシグナル伝達に関与するドメインを持つものも存在する[12][13]。
メチル化機構の様式によりタイプI(PRMT1~4, 6, 8; PRMT4はCARM1とも呼ばれる)、タイプII(PRMT5, 9)、タイプIII(PRMT7)に分類される。タイプIとIIの タンパク質アルギニンメチル基転移酵素のみが、モノメチル化されたアルギニンをさらに二次メチル化する触媒作用を持ち、タイプIIIタンパク質アルギニンメチル基転移酵素はモノメチル化活性のみが知られている[14]。タイプIとIIの違いは、タイプIは非対称型ジメチルアルギニンを形成し、タイプIIは対称型ジメチルアルギニン(SDMA)を形成する点である(図2)。タイプI タンパク質アルギニンメチル基転移酵素のうち,PRMT1は哺乳類において85%の非対称型ジメチルアルギニンの生合成を担っている[15]。
| 標的 | 酵素名(供与できるメチル基) | |
|---|---|---|
| ヒストンリジンメチル基転移酵素(SETドメイン型) | ||
| H3K4 | SETD1A (me1, me2, me3) | |
| SETD1B (me1, me2, me3) | ||
| MLL1 (me1, me2, me3) | ||
| MLL2 (me1, me2, me3) | ||
| MLL3 (me1, me2, me3) | ||
| MLL4 (me1, me2, me3) | ||
| SETD7 (me1) | ||
| SMYD3 (me2, me3) | ||
| PRDM9 (me3) | ||
| H3K9 | SETDB1 (me1, me2, me3) | |
| G9a (me1, me2) | ||
| EHMT1 (me1, me2) | ||
| PRDM2 (me1, me2) | ||
| SUV39H1 (me2, me3) | ||
| SUV39H2 (me2, me3) | ||
| H3K27 | EZH1 (me2, me3) | |
| EZH2 (me2, me3) | ||
| H3K36 | SETD2 (me1, me2, me3) | |
| NSD1 (me1, me2) | ||
| NSD2 (me1, me2) | ||
| NSD3 (me1, me2) | ||
| ASH1L (me1, me2) | ||
| SMYD2 (me2) | ||
| ヒストンリジンメチル基転移酵素(非SETドメイン型) | H3K79 | DOT1L (me1, me2, me3) |
| ヒストンリジンメチル基転移酵素(SETドメイン型) | H4K20 | SETD8 (me1) |
| SUV420H1 (me2, me3) | ||
| SUV420H2 (me2, me3) | ||
| タンパク質アルギニンメチル基転移酵素 | H3R2 | CARM1 (PRMT4) (me1, me2(a)) |
| PRMT5 (me1, me2(s)) | ||
| PRMT6 (me1, me2(a)) | ||
| PRMT7 (me1, me2(s)) | ||
| H3R8 | PRMT5 (me1, me2(s)) | |
| H3R17 | CARM1 (me1, me2(a)) | |
| H3R26 | CARM1 (me1, me2(a)) | |
| H4R3 | PRMT1 (me1, me2(a)) | |
| PRMT5 (me1, me2(s)) | ||
| PRMT6 (me1, me2(a)) | ||
| 未同定 | PRMT2 | |
| PRMT3 |
凡例:me1, me2, me3はそれぞれモノメチル化、ジメチル化、トリメチル化を表す。
(s)は対称 (symmetric), (a)は非対称 (asymmetric) なジメチル化を表す。
それぞれのメチル化機構は図1にまとめた。
組織・細胞分布
多くのヒストンメチル基転移酵素は様々な組織で発現しているが、中でもいくつかのヒストンリジンメチル基転移酵素は組織特異的な発現、細胞特異的な発現を示す。組織特異的な発現のある主なヒストンメチル基転移酵素を表2、組織特異的な発現は示さないが、細胞特異的な発現を示すヒストンメチル基転移酵素を表3に示した。詳細はThe Human Protein Atlasを参考。
| 酵素名 | 特異的に発現する組織 | 特異的に発現する細胞 |
|---|---|---|
| PRDM9 | 脳、精巣上体、精巣 | 精母細胞 |
| SUV39H2 | 精巣 | 初期精子細胞 |
| EZH1 | 網膜 | |
| EZH2 | 骨髄、リンパ系組織、精巣 | 初期精子細胞、有糸分裂細胞(大腸) |
| SMYD2 | 心筋 | 心筋細胞、壁細胞(胃)、セルトリ細胞(精巣) |
| DOT1L | 精巣 | 初期精子細胞 |
| CARM1 | 骨格筋 | 脂肪前駆細胞(内臓脂肪) |
| PRMT8 | 脳、網膜、精巣 | 腸管内分泌細胞(大腸)、線毛上皮細胞(肺)、精子細胞 |
| 酵素名 | 特異的に発現する細胞 |
|---|---|
| SETD1A | 精原細胞 |
| MLL2 | 精母細胞、精原細胞 |
| SETD7 | セルトリ細胞 |
| SMYD3 | 脂肪前駆細胞 |
| SETDB1 | 精母細胞 |
| SUV39H1 | 脂肪細胞、セルトリ細胞 |
| SETD2 | 精母細胞 |
| NSD1 | 甲状腺細胞 |
| NSD2 | 精母細胞 |
| ASH1L | 精母細胞 |
| SETD8 | 角化細胞、甲状腺細胞 |
| PRMT5 | 精母細胞、精原細胞 |
| PRMT6 | 甲状腺細胞 |
| PRMT9 | 初期精子細胞、精母細胞 |
分子機能
各クラスは触媒ドメインは異なるものの、いずれもメチル基供与体として S-アデノシル-L-メチオニン (SAM/Adomet) を使用する[3][16]。リジン残基には1~3個のメチル基が、アルギニン残基に1あるいは2個のメチル基が付加される(図1, 2)。モノ・ジ・トリメチル化のどの状態まで触媒するかは表1に示したようにそれぞれの特異性によって分かれている。ヒストンH3のリジン残基ではK4(4番目のリジン残基)、K9、K27、K36、K79が、アルギニン残基ではR2(2番目のアルギニン残基)、R8、R17、R26がメチル化され、ヒストンH4ではK20、R3がメチル化される。
それぞれメチル化される残基の位置によって転写の活性化に関与するものと抑制に関与するものが存在しており、一般的にはH3K4、K36、K79、R17は転写活性化、H3K9、K27、H4K20は転写抑制に関与すると考えられている[17][9][18]。
神経細胞での機能
ヒストンリジンメチル基転移酵素
神経幹細胞は自己複製能とニューロン、アストロサイト及びオリゴデンドロサイトへの多分化能を持っており、脳の発達と恒常性にとって重要な役割を果たしているが、神経幹細胞の増殖・複製と分化には多くのヒストンリジンメチル基転移酵素が関与している。増殖にはSetd8の関与が示唆されており、Setd8に変異を加えることで機能が失われると、脳内のH4K20モノメチル化が失われるとともに休止状態にある神経幹細胞の再活性化が遅れることが報告されている[19]。また、Setd8はサイクリン依存性キナーゼ1(Cdk1)やWntシグナル経路の転写コアクチベーターであるearthbound1/jerky(Ebd1)のプロモーター領域に結合し、脳内でのCdk1およびEbd1の発現に必要であることも報告されている [20]。Setd8と同様に、Cdk1とEbd1は神経幹細胞の再活性化を促進することがわかっており、Cdk1とEbd1の過剰発現によってSetd8変異脳で観察された神経幹細胞再活性化の障害を改善できることが示されている。これらの結果からSetd8は、Wntシグナル伝達と細胞周期の進行を調節することにより、神経幹細胞の再活性化を促進すると考えられる[20]。
神経前駆細胞からニューロンへの分化においてはSetdb1が重要な役割を果たしている。Setdb1は脳の発生初期の神経前駆細胞において高発現しており、ニューロン分化に必要な遺伝子の発現を抑制しているが、発生進行に伴いSetdb1の発現は低下するため、ニューロンへの分化が可能になる。しかし、発生初期からSetdb1が欠失すると、メカニズムは不明であるが、ニューロンへの分化と成熟に必要な遺伝子の発現が阻害され、ニューロン生成は妨げられる[21]。一方で、分化後のニューロンにおいてSetdb1を過剰発現させると、空間認知障害やうつ様行動が誘発されるなど、Setdb1はニューロンのそれぞれの分化過程において異なる作用を持っている[22][23]。
G9aやG9a-like protein (GLP)も神経系において重要な役割を果たしている。前脳ニューロン特異的なG9aまたはGlpの欠損(ノックアウト)マウスにおいて、生後間もない状態では、明らかなニューロンの早期発達や構造上の欠陥は見られないが、生後6~8週齢のマウスでは、成熟ニューロン特異的な遺伝子発現パターンの変化や学習・記憶や意欲・報酬系の障害が見られる[24]。さらにコカインの反復曝露によって、G9aの発現レベルが側坐核ニューロンで特異的に低下し、H3K9me2のレベルが有意に低下する[25]。G9aの発現レベル低下を補完すると、コカインによるニューロンの形態変化、薬物依存行動としての常同行動の増加やストレスへの強い反応などに代表される行動変化は抑制される。また、側座核ニューロンにおいてG9aを特異的に不活性化すると、コカインに曝露せずともニューロン形態変化が変化し、コカインに対する嗜好性も増強される。以上よりG9aの発現抑制の解除はコカインへの渇望を抑制するための効果的治療法となりうると考えられる[26]。
Ezh2とSuv4-20hは、グリア線維性酸性タンパク質(glial fibrillary acidic protein, GFAP)陽性の放射状グリア細胞においてニューロンへの分化を共同で調節しており、両遺伝子の欠失は海馬の発達に劇的な欠陥を引き起こす(単一ノックアウトでは観察されない)[27]。また成体海馬においては、Ezh2とSuv4-20hが神経前駆細胞で異なる役割を果たすことが報告されており[28] 、Ezh2は早期分化を抑制することにより神経前駆細胞集団の維持に大きな役割を果たし、一方でSuv4-20hは神経前駆細胞の細胞周期のS期進行を仲介することで神経前駆細胞の増殖に影響を与えると考えられている[29][30]。
Suv39h1/2によるH3K9メチル化は、成体海馬の神経前駆細胞からニューロンへの分化を制御していることがわかっている。成体海馬の神経前駆細胞におけるSuv39h1/2の薬理学的阻害は、ニューロン分化を阻害する一方で増殖を亢進させた[31]。さらに、歯状回でSuv39h1/2をノックダウンするとニューロン新生が阻害されたことから、Suv39h1/2を介したH3K9me3が成体海馬のニューロン新生に重要な役割を果たしていると考えられる[31]。
成体での脳室下帯のニューロン新生においてはMll1が必須であり、Mll1欠損の神経幹細胞ではニューロンへの分化がほとんど起こらず、グリア系列に分化したという報告がある[32][33]。脳室下帯に存在する神経前駆細胞では、通常Dlx2遺伝子の転写開始点が高レベルのH3K4me3を有することで転写が活性化されている。一方Mll1欠損神経前駆細胞では、Dlx2遺伝子のクロマチンでこのH3K4me3が維持され、また転写抑制に関与するH3K27me3により二重にマークされることで、Dlx2の発現が抑制されている[33]。
タンパク質アルギニンメチル基転移酵素
近年の研究により、ヒストンリジンメチル基転移酵素に加え、タンパク質アルギニンメチル基転移酵素も中枢神経系の発生や維持に関与していることが報告されており、Prmt6は、発達中のマウス大脳皮質における神経前駆細胞の分化・増殖に必須であることが明らかになった[34]。
Prmt1はマウス胚の神経幹/前駆細胞で高発現しており、神経幹/前駆細胞でのPrmt1のノックダウンはアストロサイトへの分化を抑制するという報告がある[35]。神経幹/前駆細胞のアストロサイト分化には転写因子であるシグナル伝達兼転写活性化因子3 (signal transducer and activator of transcription 3, STAT3)が重要であることが知られているが[36] 、Prmt1はSTAT3のアルギニン残基をメチル化してSTAT3の転写活性化能を増強することにより、神経幹/前駆細胞のアストロサイト分化を促進する[35]。またPrmt1の中枢神経系特異的欠損マウスでは、野生型マウスと同様に出生するものの生後2週間ほどで死亡し、脳では成熟オリゴデンドロサイトやその前駆細胞の減少が認められた[37]。
Prmt5は大脳皮質の神経幹細胞発生時にSchwann cell factor 1(SC1)によって標的遺伝子にリクルートされることで、神経幹細胞の増殖能と多分化能を維持することが報告されている[38]。
疾患との関わり
脳神経系疾患
ヒストンリジンメチル基転移酵素
Setdb1は、脳腫瘍において一般的に過剰発現され、主にヒストンH3K9me3を誘導し、腫瘍抑制遺伝子の発現を抑制する[39]。また、統合失調症やうつ病などの気分障害、認知障害では、Setdb1の発現が上昇し、NMDA型グルタミン酸受容体サブユニットGrin2Bの発現を抑制する[22][40]。これらの疾患では、陰性症状優位の表現型や疾患予後の悪化と関連している。さらに、ハンチントン病ではSetdb1の発現が上昇し、ハンチントン病の病因に関与する重要な遺伝子の発現低下と関連するとの報告がある[41]。
一方、Setdb1遺伝子が欠損すると、シナプス形成に重要なIL1RAPL1(Interleukin-1 receptor accessory protein-like 1)遺伝子のエンハンサーが活性化され、その発現が亢進する[42][43]。この遺伝子の発現亢進が自閉スペクトラム症を含む精神・神経疾患の原因である可能性が示されている。
また、レット症候群の原因遺伝子Mecp2との関与も報告されており、レット症候群のモデル動物であるMecp2ノックアウトマウスの、ニューロンにおけるSetdb1を介したH3K9の過度なメチル化は、MeCP2の欠損のみによる表現型を、さらに悪化させることがわかっている[44]。
タンパク質アルギニンメチル基転移酵素ファミリー
Prmt1が神経系疾患に関与していることが報告されている。fused in sarcoma (FUS)遺伝子の変異は筋萎縮性側索硬化症 (amyotrophic lateral sclerosis, ALS)の原因の1つであり、前頭側頭型認知症 (frontotemporal dementia, FTD)にも関与するが、Prmt1をノックアウトするとこれらの疾患に関連するFUS変異体による細胞毒性が増強される[45][46][47]。この結果から、Prmt1がFUSのメチル化を介して、細胞内での凝集体形成や細胞毒性の抑制に寄与することが示唆されている。
さらにPrmt1 は、アポトーシス促進性のBCL-2 antagonist of cell death(BAD)タンパク質をメチル化することによりAktからのリン酸化を阻害し、その結果アポトーシスを亢進させることが示されている[48]。最近の報告によると、Btg1ノックアウト髄芽腫モデルマウスでは、Prmt1の発現が増加し、このPrmt1-BAD軸を介して腫瘍のアポトーシスを亢進することで、腫瘍の発生を抑制している。[49]。一方で神経膠腫細胞株においてPrmt1をノックダウンすると、細胞周期が停止し、アポトーシスに至った[50]。これらの結果は、Prmt1が様々な種類の腫瘍細胞において異なる機能を果たす可能性を示唆している。
腫瘍
ヒストンリジンメチル基転移酵素
ポリコーム抑制複合体(polycomb repressive complex, PRC)は、発生過程において体の前後軸や体節を決定するホメオボックス遺伝子の発現制御にかかわるものとして知られるが、その制御には特にPRC2複合体に含まれるEzh2の作用が重要である。Ezh2は、そのヒストンメチル基転移酵素活性を使用してH3K27me3を触媒し、E-カドへリン、p16INK4α、p57、PSP94などの腫瘍抑制遺伝子を抑制しており[51] 、Ezh2の過剰発現は子宮内膜がん、肺がん、黒色腫、乳がん、膀胱がんおよび結腸直腸がんにおけるがん細胞の増殖、移動、および浸潤を促進する[52][53][54]。
タンパク質アルギニンメチル基転移酵素
タンパク質アルギニンメチル基転移酵素の過剰発現によってがんの増殖と転移を促進するとの報告がある。PRMT1は、急性骨髄性白血病における白血病細胞の増殖を促進することが示されており[55][56][57] 、他の研究では、PRMT1 の過剰発現によってE-カドへリンと Twist1(Twist family BHLH transcription factor 1)のメチル化を介して肺がんの転移と浸潤を促進する[58]。またPRMT1がc-GASのアルギニン残基をメチル化して二量体化を阻害し、腫瘍細胞において、自然免疫経路であるc-GAS/STINGシグナルを抑制することで、腫瘍化を促進することも報告された[59]。PRMT5 については、上皮成長因子受容体 (epidermal growth factor receptor, EGFR)/ AKT / β-カテニンシグナル伝達経路を介して膵臓がん細胞の悪性度を高める[60]。
関連語
参考文献
- ↑ 1.0 1.1
Tsukada, Y. (2007).
[Histone methylation and demethylation--focusing on demethylation--]. Seikagaku. The Journal of Japanese Biochemical Society, 79(7), 691-7. [PubMed:17763704] [WorldCat] PDF - ↑
Allfrey, V.G., & Mirsky, A.E. (1964).
Structural Modifications of Histones and their Possible Role in the Regulation of RNA Synthesis. Science (New York, N.Y.), 144(3618), 559. [PubMed:17836360] [WorldCat] [DOI] - ↑ 3.0 3.1
Dillon, S.C., Zhang, X., Trievel, R.C., & Cheng, X. (2005).
The SET-domain protein superfamily: protein lysine methyltransferases. Genome biology, 6(8), 227. [PubMed:16086857] [PMC] [WorldCat] [DOI] - ↑
Jenuwein, T. (2006).
The epigenetic magic of histone lysine methylation. The FEBS journal, 273(14), 3121-35. [PubMed:16857008] [WorldCat] [DOI] - ↑
Rea, S., Eisenhaber, F., O'Carroll, D., Strahl, B.D., Sun, Z.W., Schmid, M., ..., & Jenuwein, T. (2000).
Regulation of chromatin structure by site-specific histone H3 methyltransferases. Nature, 406(6796), 593-9. [PubMed:10949293] [WorldCat] [DOI] - ↑ 6.0 6.1
Marmorstein, R. (2003).
Structure of SET domain proteins: a new twist on histone methylation. Trends in biochemical sciences, 28(2), 59-62. [PubMed:12575990] [WorldCat] [DOI] - ↑
Husmann, D., & Gozani, O. (2019).
Histone lysine methyltransferases in biology and disease. Nature structural & molecular biology, 26(10), 880-889. [PubMed:31582846] [PMC] [WorldCat] [DOI] - ↑
Kim, W., Choi, M., & Kim, J.E. (2014).
The histone methyltransferase Dot1/DOT1L as a critical regulator of the cell cycle. Cell cycle (Georgetown, Tex.), 13(5), 726-38. [PubMed:24526115] [PMC] [WorldCat] [DOI] - ↑ 9.0 9.1
Park, J., Lee, K., Kim, K., & Yi, S.J. (2022).
The role of histone modifications: from neurodevelopment to neurodiseases. Signal transduction and targeted therapy, 7(1), 217. [PubMed:35794091] [PMC] [WorldCat] [DOI] - ↑
Okada, Y., Feng, Q., Lin, Y., Jiang, Q., Li, Y., Coffield, V.M., ..., & Zhang, Y. (2005).
hDOT1L links histone methylation to leukemogenesis. Cell, 121(2), 167-78. [PubMed:15851025] [WorldCat] [DOI] - ↑
van Leeuwen, F., Gafken, P.R., & Gottschling, D.E. (2002).
Dot1p modulates silencing in yeast by methylation of the nucleosome core. Cell, 109(6), 745-56. [PubMed:12086673] [WorldCat] [DOI] - ↑
Lee, J., Sayegh, J., Daniel, J., Clarke, S., & Bedford, M.T. (2005).
PRMT8, a new membrane-bound tissue-specific member of the protein arginine methyltransferase family. The Journal of biological chemistry, 280(38), 32890-6. [PubMed:16051612] [WorldCat] [DOI] - ↑
Iwasaki, H., & Yada, T. (2007).
Protein arginine methylation regulates insulin signaling in L6 skeletal muscle cells. Biochemical and biophysical research communications, 364(4), 1015-21. [PubMed:17971302] [WorldCat] [DOI] - ↑
Hashimoto, M., Fukamizu, A., Nakagawa, T., & Kizuka, Y. (2021).
Roles of protein arginine methyltransferase 1 (PRMT1) in brain development and disease. Biochimica et biophysica acta. General subjects, 1865(1), 129776. [PubMed:33127433] [WorldCat] [DOI] - ↑
Tang, J., Frankel, A., Cook, R.J., Kim, S., Paik, W.K., Williams, K.R., ..., & Herschman, H.R. (2000).
PRMT1 is the predominant type I protein arginine methyltransferase in mammalian cells. The Journal of biological chemistry, 275(11), 7723-30. [PubMed:10713084] [WorldCat] [DOI] - ↑
Nguyen, A.T., & Zhang, Y. (2011).
The diverse functions of Dot1 and H3K79 methylation. Genes & development, 25(13), 1345-58. [PubMed:21724828] [PMC] [WorldCat] [DOI] - ↑
Di Nisio, E., Lupo, G., Licursi, V., & Negri, R. (2021).
The Role of Histone Lysine Methylation in the Response of Mammalian Cells to Ionizing Radiation. Frontiers in genetics, 12, 639602. [PubMed:33859667] [PMC] [WorldCat] [DOI] - ↑
Bauer, U.M., Daujat, S., Nielsen, S.J., Nightingale, K., & Kouzarides, T. (2002).
Methylation at arginine 17 of histone H3 is linked to gene activation. EMBO reports, 3(1), 39-44. [PubMed:11751582] [PMC] [WorldCat] [DOI] - ↑
Wickramasekara, R.N., & Stessman, H.A.F. (2019).
Histone 4 Lysine 20 Methylation: A Case for Neurodevelopmental Disease. Biology, 8(1). [PubMed:30832413] [PMC] [WorldCat] [DOI] - ↑ 20.0 20.1
Huang, J., Gujar, M.R., Deng, Q., Y Chia, S., Li, S., Tan, P., ..., & Wang, H. (2021).
Histone lysine methyltransferase Pr-set7/SETD8 promotes neural stem cell reactivation. EMBO reports, 22(4), e50994. [PubMed:33565211] [PMC] [WorldCat] [DOI] - ↑
Tan, S.L., Nishi, M., Ohtsuka, T., Matsui, T., Takemoto, K., Kamio-Miura, A., ..., & Kageyama, R. (2012).
Essential roles of the histone methyltransferase ESET in the epigenetic control of neural progenitor cells during development. Development (Cambridge, England), 139(20), 3806-16. [PubMed:22991445] [WorldCat] [DOI] - ↑ 22.0 22.1
Bharadwaj, R., Peter, C.J., Jiang, Y., Roussos, P., Vogel-Ciernia, A., Shen, E.Y., ..., & Akbarian, S. (2014).
Conserved higher-order chromatin regulates NMDA receptor gene expression and cognition. Neuron, 84(5), 997-1008. [PubMed:25467983] [PMC] [WorldCat] [DOI] - ↑
Jiang, Y., Jakovcevski, M., Bharadwaj, R., Connor, C., Schroeder, F.A., Lin, C.L., ..., & Akbarian, S. (2010).
Setdb1 histone methyltransferase regulates mood-related behaviors and expression of the NMDA receptor subunit NR2B. The Journal of neuroscience : the official journal of the Society for Neuroscience, 30(21), 7152-67. [PubMed:20505083] [PMC] [WorldCat] [DOI] - ↑
Schaefer, A., Sampath, S.C., Intrator, A., Min, A., Gertler, T.S., Surmeier, D.J., ..., & Greengard, P. (2009).
Control of cognition and adaptive behavior by the GLP/G9a epigenetic suppressor complex. Neuron, 64(5), 678-91. [PubMed:20005824] [PMC] [WorldCat] [DOI] - ↑
Maze, I., Covington, H.E., Dietz, D.M., LaPlant, Q., Renthal, W., Russo, S.J., ..., & Nestler, E.J. (2010).
Essential role of the histone methyltransferase G9a in cocaine-induced plasticity. Science (New York, N.Y.), 327(5962), 213-6. [PubMed:20056891] [PMC] [WorldCat] [DOI] - ↑
Shinkai, Y., & Tachibana, M. (2011).
H3K9 methyltransferase G9a and the related molecule GLP. Genes & development, 25(8), 781-8. [PubMed:21498567] [PMC] [WorldCat] [DOI] - ↑
Chang, K.C., Rhodes, C.T., Zhang, J.Q., Moseley, M.C., Cardona, S.M., Huang, S.A., ..., & Lin, C.A. (2022).
The chromatin repressors EZH2 and Suv4-20h coregulate cell fate specification during hippocampal development. FEBS letters, 596(3), 294-308. [PubMed:34890048] [WorldCat] [DOI] - ↑
Rhodes, C.T., & Lin, C.A. (2023).
Role of the histone methyltransferases Ezh2 and Suv4-20h1/Suv4-20h2 in neurogenesis. Neural regeneration research, 18(3), 469-473. [PubMed:36018148] [PMC] [WorldCat] [DOI] - ↑
Rhodes, C.T., Zunino, G., Huang, S.A., Cardona, S.M., Cardona, A.E., Berger, M.S., ..., & Lin, C.A. (2018).
Region specific knock-out reveals distinct roles of chromatin modifiers in adult neurogenic niches. Cell cycle (Georgetown, Tex.), 17(3), 377-389. [PubMed:29433384] [PMC] [WorldCat] [DOI] - ↑ Rhodes, C. (2017).
Epigenetic Repression in the Context of Adult Neurogenesis. 2017, The University of Texas at San Antonio. - ↑ 31.0 31.1
Guerra, M.V., Cáceres, M.I., Herrera-Soto, A., Arredondo, S.B., Varas-Godoy, M., van Zundert, B., & Varela-Nallar, L. (2021).
H3K9 Methyltransferases Suv39h1 and Suv39h2 Control the Differentiation of Neural Progenitor Cells in the Adult Hippocampus. Frontiers in cell and developmental biology, 9, 778345. [PubMed:35096813] [PMC] [WorldCat] [DOI] - ↑
Potts, M.B., Siu, J.J., Price, J.D., Salinas, R.D., Cho, M.J., Ramos, A.D., ..., & Lim, D.A. (2014).
Analysis of Mll1 deficiency identifies neurogenic transcriptional modules and Brn4 as a factor for direct astrocyte-to-neuron reprogramming. Neurosurgery, 75(4), 472-82; discussion 482. [PubMed:24887289] [PMC] [WorldCat] [DOI] - ↑ 33.0 33.1
Lim, D.A., Huang, Y.C., Swigut, T., Mirick, A.L., Garcia-Verdugo, J.M., Wysocka, J., ..., & Alvarez-Buylla, A. (2009).
Chromatin remodelling factor Mll1 is essential for neurogenesis from postnatal neural stem cells. Nature, 458(7237), 529-33. [PubMed:19212323] [PMC] [WorldCat] [DOI] - ↑
Bouchard, C., Sahu, P., Meixner, M., Nötzold, R.R., Rust, M.B., Kremmer, E., ..., & Bauer, U.M. (2018).
Genomic Location of PRMT6-Dependent H3R2 Methylation Is Linked to the Transcriptional Outcome of Associated Genes. Cell reports, 24(12), 3339-3352. [PubMed:30232013] [WorldCat] [DOI] - ↑ 35.0 35.1
Honda, M., Nakashima, K., & Katada, S. (2017).
PRMT1 regulates astrocytic differentiation of embryonic neural stem/precursor cells. Journal of neurochemistry, 142(6), 901-907. [PubMed:28695568] [WorldCat] [DOI] - ↑
Nakashima, K., Yanagisawa, M., Arakawa, H., Kimura, N., Hisatsune, T., Kawabata, M., ..., & Taga, T. (1999).
Synergistic signaling in fetal brain by STAT3-Smad1 complex bridged by p300. Science (New York, N.Y.), 284(5413), 479-82. [PubMed:10205054] [WorldCat] [DOI] - ↑
Hashimoto, M., Murata, K., Ishida, J., Kanou, A., Kasuya, Y., & Fukamizu, A. (2016).
Severe Hypomyelination and Developmental Defects Are Caused in Mice Lacking Protein Arginine Methyltransferase 1 (PRMT1) in the Central Nervous System. The Journal of biological chemistry, 291(5), 2237-45. [PubMed:26637354] [PMC] [WorldCat] [DOI] - ↑
Chittka, A., Nitarska, J., Grazini, U., & Richardson, W.D. (2012).
Transcription factor positive regulatory domain 4 (PRDM4) recruits protein arginine methyltransferase 5 (PRMT5) to mediate histone arginine methylation and control neural stem cell proliferation and differentiation. The Journal of biological chemistry, 287(51), 42995-3006. [PubMed:23048031] [PMC] [WorldCat] [DOI] - ↑
Sepsa, A., Levidou, G., Gargalionis, A., Adamopoulos, C., Spyropoulou, A., Dalagiorgou, G., ..., & Korkolopoulou, P. (2015).
Emerging role of linker histone variant H1x as a biomarker with prognostic value in astrocytic gliomas. A multivariate analysis including trimethylation of H3K9 and H4K20. PloS one, 10(1), e0115101. [PubMed:25602259] [PMC] [WorldCat] [DOI] - ↑
Avramopoulos, D., Lasseter, V.K., Fallin, M.D., Wolyniec, P.S., McGrath, J.A., Nestadt, G., ..., & Pulver, A.E. (2007).
Stage II follow-up on a linkage scan for bipolar disorder in the Ashkenazim provides suggestive evidence for chromosome 12p and the GRIN2B gene. Genetics in medicine : official journal of the American College of Medical Genetics, 9(11), 745-51. [PubMed:18007143] [WorldCat] [DOI] - ↑
Ryu, H., Lee, J., Hagerty, S.W., Soh, B.Y., McAlpin, S.E., Cormier, K.A., ..., & Ferrante, R.J. (2006).
ESET/SETDB1 gene expression and histone H3 (K9) trimethylation in Huntington's disease. Proceedings of the National Academy of Sciences of the United States of America, 103(50), 19176-81. [PubMed:17142323] [PMC] [WorldCat] [DOI] - ↑
Sun, Z., & Chadwick, B.P. (2018).
Loss of SETDB1 decompacts the inactive X chromosome in part through reactivation of an enhancer in the IL1RAPL1 gene. Epigenetics & chromatin, 11(1), 45. [PubMed:30103804] [PMC] [WorldCat] [DOI] - ↑
Markouli, M., Strepkos, D., Chlamydas, S., & Piperi, C. (2021).
Histone lysine methyltransferase SETDB1 as a novel target for central nervous system diseases. Progress in neurobiology, 200, 101968. [PubMed:33279625] [WorldCat] [DOI] - ↑
Jiang, Y., Matevossian, A., Guo, Y., & Akbarian, S. (2011).
Setdb1-mediated histone H3K9 hypermethylation in neurons worsens the neurological phenotype of Mecp2-deficient mice. Neuropharmacology, 60(7-8), 1088-97. [PubMed:20869373] [PMC] [WorldCat] [DOI] - ↑
Vance, C., Rogelj, B., Hortobágyi, T., De Vos, K.J., Nishimura, A.L., Sreedharan, J., ..., & Shaw, C.E. (2009).
Mutations in FUS, an RNA processing protein, cause familial amyotrophic lateral sclerosis type 6. Science (New York, N.Y.), 323(5918), 1208-1211. [PubMed:19251628] [PMC] [WorldCat] [DOI] - ↑
Tradewell, M.L., Yu, Z., Tibshirani, M., Boulanger, M.C., Durham, H.D., & Richard, S. (2012).
Arginine methylation by PRMT1 regulates nuclear-cytoplasmic localization and toxicity of FUS/TLS harbouring ALS-linked mutations. Human molecular genetics, 21(1), 136-49. [PubMed:21965298] [WorldCat] [DOI] - ↑
Yamaguchi, A., & Kitajo, K. (2012).
The effect of PRMT1-mediated arginine methylation on the subcellular localization, stress granules, and detergent-insoluble aggregates of FUS/TLS. PloS one, 7(11), e49267. [PubMed:23152885] [PMC] [WorldCat] [DOI] - ↑
Sakamaki, J., Daitoku, H., Ueno, K., Hagiwara, A., Yamagata, K., & Fukamizu, A. (2011).
Arginine methylation of BCL-2 antagonist of cell death (BAD) counteracts its phosphorylation and inactivation by Akt. Proceedings of the National Academy of Sciences of the United States of America, 108(15), 6085-90. [PubMed:21444773] [PMC] [WorldCat] [DOI] - ↑
Ceccarelli, M., D'Andrea, G., Micheli, L., & Tirone, F. (2020).
Deletion of Btg1 Induces Prmt1-Dependent Apoptosis and Increased Stemness in Shh-Type Medulloblastoma Cells Without Affecting Tumor Frequency. Frontiers in oncology, 10, 226. [PubMed:32231994] [PMC] [WorldCat] [DOI] - ↑
Wang, S., Tan, X., Yang, B., Yin, B., Yuan, J., Qiang, B., & Peng, X. (2012).
The role of protein arginine-methyltransferase 1 in gliomagenesis. BMB reports, 45(8), 470-5. [PubMed:22917032] [WorldCat] [DOI] - ↑
Cao, R., Wang, L., Wang, H., Xia, L., Erdjument-Bromage, H., Tempst, P., ..., & Zhang, Y. (2002).
Role of histone H3 lysine 27 methylation in Polycomb-group silencing. Science (New York, N.Y.), 298(5595), 1039-43. [PubMed:12351676] [WorldCat] [DOI] - ↑
Eskander, R.N., Ji, T., Huynh, B., Wardeh, R., Randall, L.M., & Hoang, B. (2013).
Inhibition of enhancer of zeste homolog 2 (EZH2) expression is associated with decreased tumor cell proliferation, migration, and invasion in endometrial cancer cell lines. International journal of gynecological cancer : official journal of the International Gynecological Cancer Society, 23(6), 997-1005. [PubMed:23792601] [PMC] [WorldCat] [DOI] - ↑
Serresi, M., Siteur, B., Hulsman, D., Company, C., Schmitt, M.J., Lieftink, C., ..., & Gargiulo, G. (2018).
Ezh2 inhibition in Kras-driven lung cancer amplifies inflammation and associated vulnerabilities. The Journal of experimental medicine, 215(12), 3115-3135. [PubMed:30487290] [PMC] [WorldCat] [DOI] - ↑
Yomtoubian, S., Lee, S.B., Verma, A., Izzo, F., Markowitz, G., Choi, H., ..., & Mittal, V. (2020).
Inhibition of EZH2 Catalytic Activity Selectively Targets a Metastatic Subpopulation in Triple-Negative Breast Cancer. Cell reports, 30(3), 755-770.e6. [PubMed:31968251] [WorldCat] [DOI] - ↑
He, X., Zhu, Y., Lin, Y.C., Li, M., Du, J., Dong, H., ..., & Li, L. (2019).
PRMT1-mediated FLT3 arginine methylation promotes maintenance of FLT3-ITD+ acute myeloid leukemia. Blood, 134(6), 548-560. [PubMed:31217189] [PMC] [WorldCat] [DOI] - ↑
Zhu, Y., He, X., Lin, Y.C., Dong, H., Zhang, L., Chen, X., ..., & Li, L. (2019).
Targeting PRMT1-mediated FLT3 methylation disrupts maintenance of MLL-rearranged acute lymphoblastic leukemia. Blood, 134(15), 1257-1268. [PubMed:31395602] [PMC] [WorldCat] [DOI] - ↑
Liu, Z., Zhang, R., Chen, X., Yao, P., Yan, T., Liu, W., ..., & Zhao, S. (2019).
Identification of hub genes and small-molecule compounds related to intracerebral hemorrhage with bioinformatics analysis. PeerJ, 7, e7782. [PubMed:31667013] [PMC] [WorldCat] [DOI] - ↑
Avasarala, S., Van Scoyk, M., Karuppusamy Rathinam, M.K., Zerayesus, S., Zhao, X., Zhang, W., ..., & Bikkavilli, R.K. (2015).
PRMT1 Is a Novel Regulator of Epithelial-Mesenchymal-Transition in Non-small Cell Lung Cancer. The Journal of biological chemistry, 290(21), 13479-89. [PubMed:25847239] [PMC] [WorldCat] [DOI] - ↑
Liu, J., Bu, X., Chu, C., Dai, X., Asara, J.M., Sicinski, P., ..., & Wei, W. (2023).
PRMT1 mediated methylation of cGAS suppresses anti-tumor immunity. Nature communications, 14(1), 2806. [PubMed:37193698] [PMC] [WorldCat] [DOI] - ↑
Ge, L., Wang, H., Xu, X., Zhou, Z., He, J., Peng, W., ..., & Xu, M. (2020).
PRMT5 promotes epithelial-mesenchymal transition via EGFR-β-catenin axis in pancreatic cancer cells. Journal of cellular and molecular medicine, 24(2), 1969-1979. [PubMed:31851779] [PMC] [WorldCat] [DOI]