Hesファミリー
小林 妙子、影山 龍一郎
京都大学 ウイルス研究所
DOI:10.14931/bsd.1880 原稿受付日:2013年3月25日 原稿完成日:2013年5月7日
担当編集委員:大隅 典子(東北大学 大学院医学系研究科 附属創生応用医学研究センター 脳神経科学コアセンター 発生発達神経科学分野)
| basic helix-loop-helix DNA-binding domain | |||||||||
|---|---|---|---|---|---|---|---|---|---|
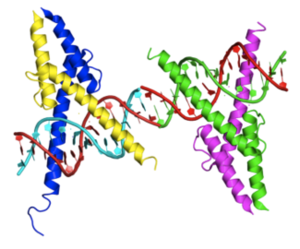 Crystal structure of MyoD bHLH domain bound to DNA based on 1MDY | |||||||||
| Identifiers | |||||||||
| Symbol | bHLH | ||||||||
| Pfam | PF00010 | ||||||||
| InterPro | IPR001092 | ||||||||
| SMART | SM00353 | ||||||||
| PROSITE | PDOC00038 | ||||||||
| SCOP | 1mdy | ||||||||
| SUPERFAMILY | 1mdy | ||||||||
| |||||||||
英語名:hairy and enhancer of split 英略語:HES
ショウジョウバエにおいて神経分化を抑制するhairyおよびenhancer of split (E(spl))の哺乳類相同遺伝子群。hairy and enhancer of splitの頭文字からHESと名付けられた[1]。転写を負に制御する抑制型のbasic helix-loop-helix (bHLH)型転写因子であり、多くはNotchシグナルのエフェクターである。ホモあるいはヘテロ二量体を形成して機能する。構造や機能で類似するHey/Hesrファミリー(Hes-related with YRPW motif)も存在する[2]。Hesは主に分化促進型のbHLH因子を抑制することによって、幹細胞や未分化細胞の維持に機能していると考えられている[3]。
サブファミリー
ヒトでHes1からHes7まで同定されている。マウスではHes4は存在しない。Notchシグナルにより、Hes1, Hes5, Hes7の発現誘導が報告されている[4][5]。Notchシグナル以外にも、Hes1についてはBMP、FGF、 NF-κB、 Shh、Wntシグナルにより[6] [7]、Hes7についてもFGFシグナルによる発現誘導が報告されており[8]、それぞれ細胞・組織特異的な発現パターンを示す。Hes1は最も解析が進んでおり、神経、感覚器、膵臓、内分泌細胞、血球細胞の発生に中心的な役割を持つことが報告されている[3]。Hes3、Hes5はHes1と相補的に働く[9]。Hes7は体節形成に必須である[10]。Hes6はDNA結合能力が無く、Hes1とヘテロダイマーを形成することによってHes1機能を阻害する[11]。
| |||||||
| 遺伝子名はAllen Brain Atlasのin situハイブリダイゼーションデーターへリンクしている。 |
ドメイン構造

bHLH、Orange、WRPWの三つのドメインが保存されている[3]。bHLHドメインはDNA結合と二量体形成に必須である。Basic領域の中央には他のbHLH因子にはないプロリン残基が保存されており、Hesファミリーに特異的なDNA結合配列であるClass C配列(CACG(C/A)G)、N box配列(CACNAG)に強く結合する。Orangeドメインには二つの両親媒性ヘリックスがあり、ヘテロ二量体形成時のパートナーの選択に関与する。WRPWドメインはTrp-Arg-Pro-Trpの4アミノ酸からなり、コリプレッサーであるTransducin-like E(spl) (TLE1-4)/ Groucho-related gene (grg)との結合に必須である。HeyファミリーではこのWRPWがYRPWに置換されており、TLEと強く結合できない[12]。
機能
リプレッサー機能

Hesは次の二つの異なるメカニズムで転写を抑制することができる。
Active repression
ホモ二量体、あるいは、他の抑制型bHLH転写因子とヘテロ二量体を形成し、ターゲットDNA配列に結合する。WRPWドメインでコリプレッサーであるTLEと結合し、TLEに結合しているヒストン脱アセチル化酵素をリクルートすることによってクロマチン構造変化を引き起こし、ターゲット遺伝子の転写を抑制する。
一方、TLE非依存型の転写抑制活性も報告されており、Hes1に関しては、bHLHドメインでSilent information regulator 2 (Sir2)ホモログであるSirt1をリクルートする[13] 。(ショウジョウバエホモログであるhairyやE(spl)では、WRPWモチーフ近傍でC-terminal binding protein (CtBP)と結合してヒストン脱アセチル化酵素をリクルートすると報告されている。[14])
Passive repression
活性化型のbHLH型転写因子と結合して認識DNA配列への結合を阻害することにより、間接的にターゲット遺伝子の転写を抑制する。例えば、構成的に発現しているbHLH転写因子であるEタンパク質と結合し、認識配列であるE-boxへの結合を阻害する[15]。ショウジョウバエにおいて神経分化を促進するachate-scute遺伝子のマウスホモログMash1は、Eタンパク質とヘテロダイマーを形成して機能する活性化型の転写因子だが、HesはEタンパク質との結合によるpassive repressionと、Mash1のプロモーターのclass C siteに直接結合するactive repressionの二つのメカニズムで、二重にMash1の活性を制御している[3]。
発現のオシレーション

Hes遺伝子の発現は、特徴的なネガティブフィードバックループを形成している。Hes1、Hes7は自身のプロモーター配列に複数のN-box配列を持っており、自身の転写発現を抑制することができる。しかし、Hes1、Hes7共にmRNA、タンパク質が不安定で速やかに分解されるため、転写抑制が解除される結果、再び発現が誘導される[3]。この繰り返しによって、Hes1、Hes7の発現は、特徴的な短周期の周期的振動(オシレーション)を示すことが報告されており、マウスでは約二時間の周期性を示す。
この周期的な発現パターンは、胚発生の過程において重要な役割を担っていることが分かってきた。Hes1は神経分化における未分化細胞の維持や分化のタイミングの制御に[16]、Hes7については周期的な体節形成のタイミングを測る時計として機能する[10]。Hes1については、線維芽細胞での発現振動や、ES細胞での発現振動が報告されており、前者では細胞の増殖に、後者では分化の不均一性に寄与している[17][18][19][20]。
神経発生における機能
Hes1、Hes3、Hes5は発生期の脳に発現し、神経分化を抑制して神経幹/前駆細胞の増殖、維持に働く。発生期の脳では、神経幹/前駆細胞として、初期では神経上皮細胞が、その後(マウスでは胎生約8.5日目から)放射状グリア細胞が現れるが、ノックアウトマウスによる解析から、Hes1、Hes5は放射状グリア細胞の維持に、Hes1、Hes3、Hes5は神経上皮細胞の維持に必須であることが示されている[3][4]。
Hes1, Hes3, Hes5は発生期の脳の境界構造の維持にも重要である[21][22]。間脳の前視床と視床を区切る境界構造であるzona limitans intrathalamica (ZLI)は、Hes1、Hes5が無いと構造自体が失われる。Hes1、Hes3、Hes5が無いと、中脳と後脳の境界構造であるisthmusで、また、神経管の左右を区切る境界構造(背側側の蓋板、腹側側の底板)で、遺伝子発現や構造の異常が生じる。この異常は、これらの領域でHesにより抑制されていた神経分化が昂進することによると考えられる。
関連項目
参考文献
- ↑
Sasai, Y., Kageyama, R., Tagawa, Y., Shigemoto, R., & Nakanishi, S. (1992).
Two mammalian helix-loop-helix factors structurally related to Drosophila hairy and Enhancer of split. Genes & development, 6(12B), 2620-34. [PubMed:1340473] [WorldCat] [DOI] - ↑
Iso, T., Sartorelli, V., Chung, G., Shichinohe, T., Kedes, L., & Hamamori, Y. (2001).
HERP, a new primary target of Notch regulated by ligand binding. Molecular and cellular biology, 21(17), 6071-9. [PubMed:11486044] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2 3.3 3.4 3.5
Kageyama, R., Ohtsuka, T., & Kobayashi, T. (2007).
The Hes gene family: repressors and oscillators that orchestrate embryogenesis. Development (Cambridge, England), 134(7), 1243-51. [PubMed:17329370] [WorldCat] [DOI] - ↑ 4.0 4.1
Ohtsuka, T., Ishibashi, M., Gradwohl, G., Nakanishi, S., Guillemot, F., & Kageyama, R. (1999).
Hes1 and Hes5 as notch effectors in mammalian neuronal differentiation. The EMBO journal, 18(8), 2196-207. [PubMed:10205173] [PMC] [WorldCat] [DOI] - ↑
Bessho, Y., Miyoshi, G., Sakata, R., & Kageyama, R. (2001).
Hes7: a bHLH-type repressor gene regulated by Notch and expressed in the presomitic mesoderm. Genes to cells : devoted to molecular & cellular mechanisms, 6(2), 175-85. [PubMed:11260262] [WorldCat] - ↑ Ryoichiro Kageyama, Jun Hatakeyama, Toshiyuki Ohtsuka, Transcription factors in the nervous system, Wiley-VDH
- ↑
Nakayama, K., Satoh, T., Igari, A., Kageyama, R., & Nishida, E. (2008).
FGF induces oscillations of Hes1 expression and Ras/ERK activation. Current biology : CB, 18(8), R332-4. [PubMed:18430630] [WorldCat] [DOI] - ↑
Niwa, Y., Masamizu, Y., Liu, T., Nakayama, R., Deng, C.X., & Kageyama, R. (2007).
The initiation and propagation of Hes7 oscillation are cooperatively regulated by Fgf and notch signaling in the somite segmentation clock. Developmental cell, 13(2), 298-304. [PubMed:17681139] [WorldCat] [DOI] - ↑
Hatakeyama, J., Bessho, Y., Katoh, K., Ookawara, S., Fujioka, M., Guillemot, F., & Kageyama, R. (2004).
Hes genes regulate size, shape and histogenesis of the nervous system by control of the timing of neural stem cell differentiation. Development (Cambridge, England), 131(22), 5539-50. [PubMed:15496443] [WorldCat] [DOI] - ↑ 10.0 10.1
Bessho, Y., Sakata, R., Komatsu, S., Shiota, K., Yamada, S., & Kageyama, R. (2001).
Dynamic expression and essential functions of Hes7 in somite segmentation. Genes & development, 15(20), 2642-7. [PubMed:11641270] [PMC] [WorldCat] [DOI] - ↑
Bae, S., Bessho, Y., Hojo, M., & Kageyama, R. (2000).
The bHLH gene Hes6, an inhibitor of Hes1, promotes neuronal differentiation. Development (Cambridge, England), 127(13), 2933-43. [PubMed:10851137] [WorldCat] - ↑
Fischer, A., & Gessler, M. (2007).
Delta-Notch--and then? Protein interactions and proposed modes of repression by Hes and Hey bHLH factors. Nucleic acids research, 35(14), 4583-96. [PubMed:17586813] [PMC] [WorldCat] [DOI] - ↑
Takata, T., & Ishikawa, F. (2003).
Human Sir2-related protein SIRT1 associates with the bHLH repressors HES1 and HEY2 and is involved in HES1- and HEY2-mediated transcriptional repression. Biochemical and biophysical research communications, 301(1), 250-7. [PubMed:12535671] [WorldCat] [DOI] - ↑
Poortinga, G., Watanabe, M., & Parkhurst, S.M. (1998).
Drosophila CtBP: a Hairy-interacting protein required for embryonic segmentation and hairy-mediated transcriptional repression. The EMBO journal, 17(7), 2067-78. [PubMed:9524128] [PMC] [WorldCat] [DOI] - ↑
Akazawa, C., Sasai, Y., Nakanishi, S., & Kageyama, R. (1992).
Molecular characterization of a rat negative regulator with a basic helix-loop-helix structure predominantly expressed in the developing nervous system. The Journal of biological chemistry, 267(30), 21879-85. [PubMed:1400497] [WorldCat] - ↑
Shimojo, H., Ohtsuka, T., & Kageyama, R. (2008).
Oscillations in notch signaling regulate maintenance of neural progenitors. Neuron, 58(1), 52-64. [PubMed:18400163] [WorldCat] [DOI] - ↑
Hirata, H., Yoshiura, S., Ohtsuka, T., Bessho, Y., Harada, T., Yoshikawa, K., & Kageyama, R. (2002).
Oscillatory expression of the bHLH factor Hes1 regulated by a negative feedback loop. Science (New York, N.Y.), 298(5594), 840-3. [PubMed:12399594] [WorldCat] [DOI] - ↑
Masamizu, Y., Ohtsuka, T., Takashima, Y., Nagahara, H., Takenaka, Y., Yoshikawa, K., ..., & Kageyama, R. (2006).
Real-time imaging of the somite segmentation clock: revelation of unstable oscillators in the individual presomitic mesoderm cells. Proceedings of the National Academy of Sciences of the United States of America, 103(5), 1313-8. [PubMed:16432209] [PMC] [WorldCat] [DOI] - ↑
Yoshiura, S., Ohtsuka, T., Takenaka, Y., Nagahara, H., Yoshikawa, K., & Kageyama, R. (2007).
Ultradian oscillations of Stat, Smad, and Hes1 expression in response to serum. Proceedings of the National Academy of Sciences of the United States of America, 104(27), 11292-7. [PubMed:17592117] [PMC] [WorldCat] [DOI] - ↑
Kobayashi, T., Mizuno, H., Imayoshi, I., Furusawa, C., Shirahige, K., & Kageyama, R. (2009).
The cyclic gene Hes1 contributes to diverse differentiation responses of embryonic stem cells. Genes & development, 23(16), 1870-5. [PubMed:19684110] [PMC] [WorldCat] [DOI] - ↑
Hirata, H., Tomita, K., Bessho, Y., & Kageyama, R. (2001).
Hes1 and Hes3 regulate maintenance of the isthmic organizer and development of the mid/hindbrain. The EMBO journal, 20(16), 4454-66. [PubMed:11500373] [PMC] [WorldCat] [DOI] - ↑
Baek, J.H., Hatakeyama, J., Sakamoto, S., Ohtsuka, T., & Kageyama, R. (2006).
Persistent and high levels of Hes1 expression regulate boundary formation in the developing central nervous system. Development (Cambridge, England), 133(13), 2467-76. [PubMed:16728479] [WorldCat] [DOI]