「14-3-3タンパク質」の版間の差分
細 →共通認識配列 |
細編集の要約なし |
||
| (同じ利用者による、間の16版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | |||
<font size="+1">[https://researchmap.jp/read0009425 市村 徹]</font><br> | |||
''元防衛大学校 応用科学群応用化学科''<br> | |||
<font size="+1">[https://researchmap.jp/taokamasato 田岡万悟]</font><br> | |||
''東京都立大学 理学部化学科''<br> | |||
{{box|text= 14-3-3タンパク質は分子量約30kDaのサブユニットから構成される2量体タンパク質のファミリーである。ヒトでは9つの分子種(α~σアイソフォーム)の存在が確認されており、各アイソフォームは分子のN末端構造を介してホモ或いはヘテロに結合することで、その内部にU字型の溝構造を形成している。14-3-3は、この溝構造を利用することで多種多様なリン酸化タンパク質と、リン酸化に依存して結合する。脳神経系において、14-3-3は神経突起の伸長、神経分化、細胞移動と生存、神経伝達物質の合成や放出など、さまざまな細胞プロセスに関わることが報告されている。さらに、14-3- | DOI:<selfdoi /> 原稿受付日:2025年2月27日 原稿完成日:2025年3月10日<br> | ||
担当編集委員:[http://researchmap.jp/wadancnp 和田 圭司](国立研究開発法人国立精神・神経医療研究センター) | |||
</div> | |||
英:14-3-3 protein | |||
{{box|text= 14-3-3タンパク質は分子量約30kDaのサブユニットから構成される2量体タンパク質のファミリーである。ヒトでは9つの分子種(α~σアイソフォーム)の存在が確認されており、各アイソフォームは分子のN末端構造を介してホモ或いはヘテロに結合することで、その内部にU字型の溝構造を形成している。14-3-3は、この溝構造を利用することで多種多様なリン酸化タンパク質と、リン酸化に依存して結合する。脳神経系において、14-3-3は神経突起の伸長、神経分化、細胞移動と生存、神経伝達物質の合成や放出など、さまざまな細胞プロセスに関わることが報告されている。さらに、14-3-3は、クロイツフェルト-ヤコブ病やパーキンソン病をはじめとする神経変性疾患、神経発達疾患、神経精神疾患など、さまざまな神経疾患と遺伝的に関連することも報告されている。}} | |||
== 研究の歴史 == | == 研究の歴史 == | ||
14-3-3 タンパク質(以下14-3-3)は、1965年B.W. | 14-3-3 タンパク質(以下14-3-3)は、1965年B. W. Mooreによって、[[ウシ]][[脳]]の可溶性画分において発見され、[[DEAEセルロースクロマトグラフィー]]での分画番号とその後の[[ゲル電気移動]]での移動位置の組み合わせによって命名された一群の酸性タンパク質である<ref name=Moore1965><pubmed>4953930</pubmed></ref>[1]。[[神経細胞]]に特異的に発現し(可溶性タンパク質の約1%)、神経[[軸索流]]によって[[神経終末]]([[シナプス]]部)に輸送されることが報告されていたが<ref name=Erickson1980><pubmed>6161216</pubmed></ref><ref name=Boston1982><pubmed>7038050</pubmed></ref>[2][3]、その構造と機能は発見から約20年間にわたり不明であった。 | ||
1987年、このタンパク質の最初の機能として、[[カテコールアミン]]と[[セロトニン]]生合成経路の[[律速段階酵素]]([[チロシン水酸化酵素]][TH]と[[トリプトファン水酸化酵素]][TPH])を、[[カルシウム/カルモデュリン依存性タンパク質キナーゼII]] ([[CaMKII]]) <ref name=Yamauchi1981><pubmed>6113235</pubmed></ref>[4]によるリン酸化反応に依存して活性化する能力が報告された<ref name=Ichimura1987><pubmed>2885229</pubmed></ref>[5]。次いで、このタンパク質の分子構造<ref name=Ichimura1988><pubmed>2902623</pubmed></ref>[6]と[[リン酸化]]依存性相互作用<ref name=Furukawa1993><pubmed>8101440</pubmed></ref>[7]が明らかにされ、さらにこれが互いに構造が類似した複数の成分からなるmulti-geneファミリーを形成していることが証明された<ref name=Shinkai1996><pubmed>8721374</pubmed></ref>[8]。 | |||
現在ではこのファミリーが[[ヒト]]から[[植物]]、[[酵母]]に至る[[真核生物]]に広汎に分布すること、また[[細胞膜]]から[[核]]内に至る細胞反応の調節に深く関与していることが分かっている。 | |||
== 構造 == | == 構造 == | ||
=== 一次構造 === | === 一次構造 === | ||
14-3- | 14-3-3は[[等電点]] 4.6~5.1、分子量約30kDaのサブユニットから構成される二量体分子である<ref name=Ichimura1988 /> [6]。複数の分子種([[アイソフォーム]])の存在が確認されており、それらの一次構造は生物種間において高度に(60%以上の一致性)保存されている(後述)。各アイソフォームのアミノ酸配列には、リーダー配列や[[膜貫通領域]]などの極端な[[疎水性]]領域は含まれていない。また結合[[糖鎖]]の存在も確認されていない。14-3-3はこれらのアイソフォームがホモ或いはヘテロに結合することで形成される可溶性タンパク質の集団である<ref name=Ichimura1988 /><ref name=Shinkai1996 /> [6][8]。 | ||
=== 高次構造 === | === 高次構造 === | ||
14-3- | 14-3-3を構成する各サブユニットは、9個の[[α-ヘリックス]]とそれらをつなぐ短いリンカーからなり、U字型の溝構造を作るためにダイマーを形成する<ref name=Xiao1995><pubmed>7603573</pubmed></ref><ref name=Liu1995><pubmed>7603574</pubmed></ref>[9][10]('''図1A''')。片方のサブユニットの第1および第2ヘリックスと、他方のサブユニットの第3および第4ヘリックスは[[静電相互作用]]と[[疎水結合]]によって結合し、14-3-3のダイマー形成に関与している。一方、それぞれのサブユニットの第3、5、7、9ヘリックスは全体として屈曲し、U字型構造の内部に標的(リガンド)に対する結合部位を形成している('''図1B''')。14-3-3の各サブユニットはこのリガンド結合部位を介して、1サブユニットあたり1つのリガンドと、ダイマー分子あたり2個のリガンドを同時に結合できる。 | ||
上記したように14-3-3ファミリーのアミノ酸配列は生物種を超えて高度に保存されているが、U字構造内部の配列はとりわけ高く保存されている<ref name=Liu1995 /> [10]('''図1''')。このU字構造と[[nonsense mediated mRNA decay factor]] ([[SMG7]])タンパク質([[ナンセンスmRNA]]の崩壊因子)のN末端領域が形成する構造は、立体的に相同であることが報告されている<ref name=Fukuhara2005><pubmed>15721257</pubmed></ref>[11]。 | |||
[[ファイル:Ichimura 14-3-3 Fig2.png|サムネイル|'''図2. 14-3-3オルソログの系統樹'''<br>14-3-3オルソログ配列をClustalWを用いてアラインメントし、FastTreeアルゴリズムを用いて作成した。AT, ''Arabidopsis thaliana''; CE, ''C. elegans''; DM, ''Drosophila melanogaster''; DR, ''Danio rerio''; GG, ''Gallus gallus''; HS, ''Homo sapiens''; SC, ''Saccharomyces cerevisiae''; SP, ''Schizosaccharomyces pombe''; and XL, ''Xenopus laevis''.<br>Aristizábal-Corralesら<ref name=Aristizabal-Corrales2012><pubmed>22328524</pubmed></ref>から改変して転載。]] | |||
== ファミリー == | == ファミリー == | ||
複数の14-3- | 複数の14-3-3アイソフォームが真核生物([[動物界]]、[[植物界]]、[[菌界]])を構成するさまざまな細胞から見つかっている。 | ||
ヒトでは9つのアイソフォーム(α~σアイソフォーム)が報告されており<ref name=Shinkai1996 /> [8]、これらのうち、αとδアイソフォームは、それぞれβとζアイソフォームのリン酸化型である<ref name=Aitken1995><pubmed>7890696</pubmed></ref>[12]。同様のアイソフォームは、[[ラット]]、[[マウス]]、[[ウシ]]、[[ヒツジ]]、[[ニワトリ]]、[[ツメガエル]]、[[ゼブラフィシュ]]でも確認されている。各アイソフォームは種内でもまた種間でも高い配列保存が見られる。 | |||
[[線虫]]([[C. elegans|''C. elegans'']])と[[ショウジョウバエ]]([[D. melanogaster|''D. melanogaster'']])では、それぞれ2つのアイソフォーム([[PAR-5]]と[[FTT-2]]:[[Dm14-3-3ε]]と[[Dm14-3-3ζ]])の発現が検出されている<ref name=Berdichevsky2006><pubmed>16777605</pubmed></ref><ref name=Skoulakis1996><pubmed>8938125</pubmed></ref><ref name=Benton2002><pubmed>12431373</pubmed></ref>[13][14][15]。 | |||
<ref name=Skoulakis1996><pubmed>8938125</pubmed></ref><ref name=Benton2002><pubmed>12431373</pubmed></ref>[13][14][15]。 | |||
植物では[[シロイヌナズナ]]で13のアイソフォーム(μ~χアイソフォーム)の存在が確認されている<ref name=DeLille2001><pubmed>11351068</pubmed></ref>[16]。また、[[ワタ]]には6つ、[[イネ]]には8つ、[[オオムギ]]には5つ、[[タバコ]]には17の潜在的アイソフォームの存在が報告されている<ref name=Denison2011><pubmed>21907297</pubmed></ref>[17]。菌類の[[出芽酵母]]([[S. cerevisiae|''S. cerevisiae'']])と[[分裂酵母]]([[S. pombe|''S. pombe'']])では、それぞれ2つのアイソフォーム([[BMH1]]と[[BMH2]];[[Rad24]]と[[Rad25]])が見つかっている<ref name=vanHeusden1995><pubmed>7744048</pubmed></ref><ref name=Peng1997><pubmed>9278512</pubmed></ref>[18][19]。 | |||
[[系統樹]]('''図2''')から考えると、共通の祖先は、まず、[[14-3-3ε|ε]]と[[14-3-3β|β]][[14-3-3γ|γ]][[14-3-3ζ|ζ]][[14-3-3η|η]][[14-3-3θ|θ]][[14-3-3σ|σ]]のアイソフォームグループの2つに分かれ、その後εは酵母や植物各種アイソフォームと分岐し、βζθσはγηアイソフォームと分岐している。εは原始14-3-3に最も近い配列を持っており、その一方でσは最も遠い配列を持っている('''図2''')。なお、[[原核生物]]([[新生細菌]]や[[古細菌]])の細胞からは、14-3-3ファミリーに属するタンパク質は見つかっていない。 | |||
== 脳神経系における分布 == | == 脳神経系における分布 == | ||
各アイソフォームに特異的な[[cDNA]]を用いた[[in situ hybridization|''in situ'' hybridization]]解析では、[[ラット]]脳の14-3-3の[[mRNA]]はモノアミン合成ニューロンだけでなく、ほとんどすべての領域で発現していることが確認されている。いずれの領域でも、発現は神経細胞で顕著であり、特に[[嗅球]][[僧帽細胞]]、[[海馬]][[錐体細胞]]、[[小脳]][[プルキンエ細胞]]、[[脳幹]]や[[脊髄]]の[[運動ニューロン]]など、比較的大きな[[細胞体]]と高度に発達した[[樹状突起]]野を持つ[[投射型ニューロン]]で多量に検出されている<ref name=Watanabe1991><pubmed>1649368</pubmed></ref>[20]。一方、こうしたmRNAの発現には、ラットの発生初期から一貫して高レベルの発現を示すニューロン、発生初期に高発現を示し、その後成熟期と成体期に発現が減少するニューロン、発生を通じて低レベルの発現を示すニューロンの3つのタイプがあることが確認されている。さらに、ある種のニューロンでは、mRNAの発現にアイソフォーム特異性があることが知られている<ref name=Watanabe1994><pubmed>7984035</pubmed></ref>[21]。 | |||
各アイソフォームに特異的な[[抗体]]を用いた[[免疫組織化学]]では、神経細胞の[[軸索]]や樹状突起を含む細胞体で発現が確認されている。また、ほとんどの原発性ヒト神経系腫瘍において14-3-3の発現亢進が認められている<ref name=Cao2006><pubmed>16292484</pubmed></ref>[22]。一方、細胞分画法を組み合わせた生化学的な方法では、14-3-3は主に細胞質画分に検出されるが、かなりの割合が[[シナプス膜]]などの不溶性画分にアイソフォーム特異的に検出されている<ref name=Martin1994><pubmed>7964746</pubmed></ref>[23]。 | |||
== 機能 == | == 機能 == | ||
=== 共通認識配列 === | === 共通認識配列 === | ||
14-3-3は、分子全体で形成するU字型構造のリガンド結合部位を介して、タンパク質キナーゼによってリン酸化された様々な標的分子と相互作用する<ref name=Tzivion2002><pubmed>11709560</pubmed></ref>[24]。14-3-3が認識するリン酸化の共通配列として、モード1(RxxpS/TxP)<ref name=Muslin1996><pubmed>8601312</pubmed></ref>[25]、モード2(RxxxpS/TxP)<ref name=Yaffe1997><pubmed>9428519</pubmed></ref>[26],モード3(pS/TxxCOOH)<ref name=Ganguly2005><pubmed>15644438</pubmed></ref>[27]が報告されている(pS/ | 14-3-3は、分子全体で形成するU字型構造のリガンド結合部位を介して、タンパク質キナーゼによってリン酸化された様々な標的分子と相互作用する<ref name=Tzivion2002><pubmed>11709560</pubmed></ref>[24]。14-3-3が認識するリン酸化の共通配列として、モード1(RxxpS/TxP)<ref name=Muslin1996><pubmed>8601312</pubmed></ref>[25]、モード2(RxxxpS/TxP)<ref name=Yaffe1997><pubmed>9428519</pubmed></ref>[26],モード3(pS/TxxCOOH)<ref name=Ganguly2005><pubmed>15644438</pubmed></ref>[27]が報告されている(pS/Tはリン酸化セリンまたはリン酸化[[トレオニン]]、xは任意の[[アミノ酸]])。共通配列のうち、モード1の配列を含む標的分子が最も多く、その配列中にProが含まれないことも多い。プレ配列と呼ばれるシグナル配列をN末端にもつ[[ミトコンドリア]]タンパク質の前駆体や[[緑膿菌]][[ADP-ribosyltransferase Exoenzyme S]] ([[ExoS]])などは、14-3-3との結合にリン酸化を必要としないが、リン酸化タンパク質の場合と同様にU字型構造のリガンド結合部位を介して相互作用すると考えられている<ref name=Masters1999><pubmed>10213629</pubmed></ref>[28]。 | ||
=== 阻害物質と安定化物質 === | === 阻害物質と安定化物質 === | ||
| 47行目: | 50行目: | ||
==== 阻害物質 ==== | ==== 阻害物質 ==== | ||
14-3-3タンパク質のリガンド結合部位に結合して14-3-3のタンパク質- | 14-3-3タンパク質のリガンド結合部位に結合して14-3-3のタンパク質-タンパク質間相互作用をマスクする阻害剤として、[[ペプチド]]性あるいは非ペプチド性の物質が報告されている。ペプチド性物質としては、[[ファージディスプレイ]]によって発見された[[R18ペプチド]](PHCVPRDLSWLDLEANMCLP)や、2分子のR18を短いペプチド鎖で連結した[[difopein]]などがある<ref name=Wang1999><pubmed>10493820</pubmed></ref><ref name=Masters2001><pubmed>11577088</pubmed></ref>[77][78]。非ペプチド性の阻害物質としては、[[フタルイミド]]誘導体である[[BV02]]がある<ref name=Mancini2011><pubmed>21041536</pubmed></ref>[79]。 | ||
==== 安定化物質 ==== | ==== 安定化物質 ==== | ||
14-3-3タンパク質- | 14-3-3タンパク質-タンパク質間相互作用の安定化剤としては[[真菌]]が産生する[[ジテルペン配糖体]]である[[フシコクシン-A]]([[FC-A]])とそれの誘導体である[[コチレニン-A]]([[CN-A]])などがある。これらは14-3-3と[[H+-exporting P2-type ATPase|H<sup>+</sup>-exporting P2-type ATPase]] (PMA2) <ref name=Wurtele2003><pubmed>12606564</pubmed></ref>[80]、[[嚢胞性線維症膜コンダクタンス制御因子]] ([[cystic fibrosis transmembrane conductance regulator]], [[CFTR]])<ref name=Stevers2016><pubmed>26888287</pubmed></ref>[81]、[[Raf-1]]<ref name=Molzan2013><pubmed>23808890</pubmed></ref>[82]とのタンパク質-タンパク質間を安定化することが報告されている。 | ||
[[ファイル:Ichimura 14-3-3 Fig3.png|サムネイル|'''図3. タンパク質レベルにおける14-3-3の働き'''<br>14-3-3はタンパク質キナーゼによってリン酸化された標的と相互作用することで、 (1)標的酵素の活性を調節する、(2)標的タンパク質の細胞内輸送を制御する、(3)標的分子を安定化させる、など多彩な役割をもつことが知られている。]] | |||
=== タンパク質レベルにおける役割 === | === タンパク質レベルにおける役割 === | ||
14-3- | 14-3-3が標的とするタンパク質は現在300種を超えており、その中には細胞内[[シグナル伝達]]経路を構成する一連の酵素・タンパク質が共通して含まれている<ref name=Kakiuchi2007><pubmed>17559233</pubmed></ref>[29]。こうした14-3-3の標的には、[[タンパク質リン酸化酵素]]、[[ユビキチンリガーゼ]]、代謝酵素、[[転写因子]]、[[細胞骨格]]成分、[[イオンチャネル]]などがあげられる。14-3-3の結合は、標的リン酸化タンパク質のコンフォメーションや分子間相互作用に影響を与えることで<ref name=Tzivion2002><pubmed>11709560</pubmed></ref><ref name=Yaffe1997><pubmed>9428519</pubmed></ref><ref name=Obsil2001><pubmed>11336675</pubmed></ref><ref name=Yaffe2002><pubmed>11911880</pubmed></ref><ref name=Park2019><pubmed>31581174</pubmed></ref>[24][26][30][31][32]、結果として(1)標的酵素の活性を調節する、(2)標的タンパク質の細胞内輸送(局在)を制御する、(3)標的分子を安定化させる、など多彩な役割をもつことが知られている('''図3''')。14-3-3の相互作用にはアイソフォーム特異性があることが報告されている<ref name=Shinkai1996><pubmed>8721374</pubmed></ref>[8]。 | ||
=== 細胞と個体レベルにおける役割 === | === 細胞と個体レベルにおける役割 === | ||
14-3-3はリン酸化シグナルに応じて多種多様な細胞機能を調節している<ref name=Morrison2009><pubmed>19027299</pubmed></ref>[33]。例えば、14-3- | 14-3-3はリン酸化シグナルに応じて多種多様な細胞機能を調節している<ref name=Morrison2009><pubmed>19027299</pubmed></ref>[33]。例えば、14-3-3は[[Raf-1]]<ref name=Freed1994><pubmed>8085158</pubmed></ref><ref name=Irie1994><pubmed>8085159</pubmed></ref><ref name=Fantl1994><pubmed>7935795</pubmed></ref><ref name=Fu1994><pubmed>7939632</pubmed></ref>[34][35][36][37]や[[BRAF]]<ref name=Park2019><pubmed>31581174</pubmed></ref><ref name=Kondo2019><pubmed>31604311</pubmed></ref>[32][38]、[[MEKK]]<ref name=Fanger1998><pubmed>9452471</pubmed></ref>[39]などに結合して、それらのキナーゼ活性を制御することで細胞の増殖や分化を調節している。また、[[PI3K]]<ref name=Bonnefoy-Berard1995><pubmed>7479742</pubmed></ref>[40]や[[Akt]]<ref name=Zhang1999><pubmed>10411906</pubmed></ref>[41]などと相互作用し細胞の生存維持や運動を、あるいは[[Bad]]<ref name=Zha1996><pubmed>8929531</pubmed></ref>[42]や[[FKHRL1]]<ref name=Brunet1999><pubmed>10102273</pubmed></ref>[43]などと複合体を形成し[[細胞死]]([[アポトーシス]])抑制に関与していることが報告されている。分裂酵母では、[[Cdc25C]][[ホスファターゼ]]に結合することで、[[DNA修復]]にかかわる[[細胞周期]]チェックポイントシグナルの調節に重要な役割を果たしている<ref name=Peng1997 /><ref name=Ford1994><pubmed>8036497</pubmed></ref>[19][44]。出芽酵母では2つの14-3-3遺伝子の欠損は細胞死を引き起こすことが知られている<ref name=vanHeusden1995 />[18]。 | ||
=== 脳神経系での機能 === | === 脳神経系での機能 === | ||
脳神経系において、14-3- | 脳神経系において、14-3-3は上記したチロシン水酸化酵素やトリプトファン水酸化酵素、[[セロトニン-N-アセチル転移酵素|セロトニン-''N''-アセチル転移酵素]](NAT)<ref name=Obsil2001 /> [30]と相互作用し酵素活性を制御することで細胞内モノアミンレベルの調節に関わっている。また[[カルシウム|Ca<sup>2+</sup>]]シグナルに応じて[[神経伝達物質]]の放出([[エキソサイトーシス]])を促進する役割が確認されている<ref name=Morgan1992><pubmed>1538762</pubmed></ref>[45]。シナプスにおける働きとしてよりよく理解されているのは、さまざまなイオンチャネルのモジュレーターとしての役割である。代表的な標的チャネルとして、[[ニコチン性アセチルコリン受容体]](α4β2nAChR <ref name=Jeanclos2001><pubmed>11352901</pubmed></ref>[46])、[[電位依存性カルシウムチャネル]]([[電位依存性カルシウムチャネル#Cav2_(N,_P/Q,_R型)|Ca<sub>V</sub>2.2]] <ref name=Li2006><pubmed>16982421</pubmed></ref>[47])、[[カリウムチャネル]]([[KCNK3]] | ||
<ref name=O'Kelly2002><pubmed>12437930</pubmed></ref>[48] | <ref name=O'Kelly2002><pubmed>12437930</pubmed></ref>[48])、[[塩素イオンチャネル|塩素チャネル]](CFTR | ||
<ref name=Liang2012><pubmed>22278744</pubmed></ref>[49] | <ref name=Liang2012><pubmed>22278744</pubmed></ref>[49])、[[NMDA受容体]] ([[NR2C]]サブユニット<ref name=Chen2009><pubmed>19477150</pubmed></ref>[50])などが上げられる。14-3-3のリン酸化依存性相互作用は、チャネル活性を調節し、あるいは細胞内輸送や構造安定性に影響を与えることによって、効率的なシナプス伝達の維持・制御に寄与していることが報告されている。 | ||
[[マウス]][[海馬]]において阻害ペプチドであるdifopein(dimeric fourteen-three-three peptide inhibitor)を用い14-3-3を阻害すると、[[CA3]]-[[CA1]]シナプスにおける[[連想学習]]と[[記憶行動]]を障害され、[[長期増強]]([[LTP]])を抑制されることが明らかになっている<ref name=Qiao2014><pubmed>24695700</pubmed></ref>[51]。またdifopeinは14-3-3と[[LRRK2]]キナーゼの相互作用を妨害し、マウス海馬の[[初代培養]]ニューロンの[[神経突起]]伸長を短縮化させることも知られている<ref name=Lavalley2016><pubmed>26546614</pubmed></ref>[52]。14-3-3は[[δカテニン]]、[[NUDEL]]、[[LIMドメイン含有キナーゼ]] ([[LIMK]])や[[コフィリン]]などに相互作用することで、神経細胞の移動、神経分化、形態形成、[[構造可塑性]]を制御することが報告されている<ref name=Cornell2017><pubmed>29075177</pubmed></ref>[53]。これらの標的に加えて、14-3-3は、次項に列挙したものを含む多種多様なタンパク質と結合することによって、脳神経系で機能していると考えられる。 | |||
== 疾患との関わり == | == 疾患との関わり == | ||
| 69行目: | 72行目: | ||
=== クロイツフェルト-ヤコブ病 === | === クロイツフェルト-ヤコブ病 === | ||
[[プリオン]]タンパク質(PrP)のミスフォールディングによって引き起こされる致死性の[[海綿状脳症]]である。治療法は現在開発されておらず、対症療法が主体である。生前の確定診断法はないが、[[脳脊髄液]]由来の14-3-3タンパク質が[[クロイツフェルト-ヤコブ病]]の信頼できるマーカーになりうることが報告されている<ref name=Hsich1996><pubmed>8782499</pubmed></ref>[54]。14-3-3タンパク質の脳脊髄液への漏出は、クロイツフェルト-ヤコブ病による脳神経細胞の広範な破壊に依存している可能性が指摘されている<ref name=Muayqil2012><pubmed>22993290</pubmed></ref>。[55] | |||
[54]。14-3- | |||
=== アルツハイマー病 === | === アルツハイマー病 === | ||
[[アルツハイマー病]]とは数百万人が罹患しており、徐々に悪化する神経変性疾患である。[[アミロイド斑]]と[[神経原線維変化]]という2つの病理学的特徴があげられる。神経原線維変化は、異常にリン酸化されたタウタンパク質の凝集によって主に構成されており、14-3-3は、リン酸化されたタウに結合し<ref name=Hashiguchi2000><pubmed>10840038</pubmed></ref>[56]その機能や安定性を制御することで、神経原線維変化へのタウの凝集を調節していると推定されている<ref name=Shimada2013><pubmed>24364034</pubmed></ref><ref name=Abdi2024><pubmed>38375509</pubmed></ref>[57][58]。また、14-3-3は、アルツハイマー病の[[アミロイド斑]]の主成分である[[アミロイドβ]]のクリアランスに関係することも報告されている<ref name=Abdi2024 />[58]。さらに、14-3-3はアルツハイマー病患者の脳や脳脊髄液中で発現レベルが異常化していることも判明しており、その異常がアルツハイマー病の病態マーカーになる可能性も示唆されている。 | |||
=== パーキンソン病 === | === パーキンソン病 === | ||
[[パーキンソン病]]とは[[黒質]]の[[ドーパミン]]神経細胞の障害によって発症する[[神経変性疾患]]である。患者の脳では、[[レビー小体]]と呼ばれる線維状タンパク質を含む不溶性の凝集体が観察され、14-3-3はレビー小体に存在することが証明されている<ref name=Kawamoto2002><pubmed>11895039</pubmed></ref>[59]。さらに14-3-3は、パーキンソン病の発症と進行に関係する3大主要因子であるLRRK2、[[α-シヌクレイン]]、[[パーキン]]らのすべてと相互作用し、その活性を制御したり細胞内局在を変化させたり、あるいは安定化するなどの役割が報告されている<ref name=Giusto2021><pubmed>34548498</pubmed></ref>[60]。14-3-3のアミノ酸配列にはα-シヌクレインと相同的な領域がある<ref name=Ostrerova1999><pubmed>10407019</pubmed></ref>[61]。 | |||
=== 筋萎縮性側索硬化症 === | === 筋萎縮性側索硬化症 === | ||
[[筋萎縮性側索硬化症]]とは筋力低下と筋萎縮を特徴とする致死的な運動ニューロン疾患である。神経病理学的特徴として、[[ニューロフィラメント]](NF)や[[TDP-43]]、[[SOD]]などを含む[[レビー小体様ヒアリン封入体]](LBHI)の存在があげられる。患者でも動物モデルでも、14-3-3はLBHIに存在することが証明されている<ref name=Kawamoto2004><pubmed>15378322</pubmed></ref>[62]。14-3-3は、NF軽鎖にリン酸化依存的に結合して安定化させることで、ニューロフィラメント軽鎖を介する[[凝集体]]形成を抑制すると推定されている<ref name=Miao2013><pubmed>23230147</pubmed></ref>[63]。また、14-3-3θのmRNA発現レベルが患者の脊髄で上昇していることも明らかにされている<ref name=Malaspina2000><pubmed>11080204</pubmed></ref> [64]。 | |||
=== ポリグルタミン病 === | === ポリグルタミン病 === | ||
==== 脊髄小脳失調症 ==== | |||
[[脊髄小脳失調症]]1型([[SCA1]])は、[[アタキシン-1]]における[[ポリグルタミン]](ポリQ)鎖の異状伸長によって引き起こされる致死性の神経変性疾患である。SCA1における病態の顕著な部位は小脳プルキンエ細胞であり、そこでは異状アタキシン-1が核に入り込み、封入体を形成する。14-3-3は、リン酸化されたアタキシン-1に結合し<ref name=Chen2003><pubmed>12757707</pubmed></ref>[65]、[[脱リン酸化]]を防ぐとともに核への移行を阻害する役割が報告されている<ref name=Lai2011><pubmed>21835928</pubmed></ref>[66]。14-3-3εの部分欠損がSCA1の小脳表現型を改善するという所見は、14-3-3がSCA1の病因に寄与していることを示唆している<ref name=Jafar-Nejad2011><pubmed>21245341</pubmed></ref>[67]。 | |||
=== ハンチントン病 === | ==== ハンチントン病 ==== | ||
[[ハンチントン病]]とは[[常染色体顕性遺伝]]の進行性神経変性疾患であり、[[ハンチンチン]]タンパク質におけるポリQ鎖の伸長によって引き起こされる。14-3-3は、この異常ハンチンチンの封入体形成を促進することで、ミスフォールディングしたハンチンチンを除去する役割が示唆されている<ref name=Kaneko2006><pubmed>16516399</pubmed></ref>[68]。[[siRNA]]による14-3-3ζの減少は異状ハンチンチンの封入体形成を阻害したことから<ref name=Omi2008><pubmed>18078716</pubmed></ref>[69]、14-3-3が封入体形成に関与することも示唆されている。 | |||
=== ミラー・ディーカー症候群 === | === ミラー・ディーカー症候群 === | ||
[[ミラー・ディーカー症候群]]とは[[lissencephaly]]([[滑脳症]])のより重篤な型であり、ヒトとマウスに脳の異常を伴うまれな[[神経細胞移動]]障害を引き起こす。14-3-3ε遺伝子YWHAEが存在する[[染色体]]領域17p13-3はミラー・ディーカー症候群患者で常に欠損していることが明らかになっている<ref name=Toyo-oka2003><pubmed>12796778</pubmed></ref>[70]。14-3-3εは、[[CDK5]]でリン酸化されたNUDELに結合し、NUDELのリン酸化状態を保護することで、神経細胞の移動を制御していることが報告されている<ref name=Toyo-oka2003 /> [70]。NUDELは、[[LIS1]]結合タンパク質として知られており、この複合体は細胞質[[ダイニン]]重鎖機能を制御する、神経細胞の移動に必須な因子である<ref name=Taya2007><pubmed>17202468</pubmed></ref>[71]。このことは、14-3-3εが神経細胞移動に必須の役割を担っていることを示唆している。実際、14-3-3ε欠損マウスは、海馬の欠損、皮質の菲薄化、移動距離の減少、神経細胞死の増加などの脳構造の異状を示す。しかし14-3-3εの単独欠失では、発症しないことが報告されている<ref name=Denomme-Pichon2023><pubmed>36999555</pubmed></ref>[72]。 | |||
=== 統合失調症 === | === 統合失調症 === | ||
[[統合失調症]]とは[[陽性症状]]、[[陰性症状]]、[[認知症状]]の組み合わせによって特徴付けられる[[精神神経疾患]]であり、高い遺伝性があることが示されている。遺伝子解析により、14-3-3η遺伝子YWHAHが存在する染色体領域22q12-13との関連が示唆されている。実際、14-3-3η遺伝子の[[一塩基多型]](SNP)との有意な関連は、様々なヒトサンプルを用いた多くの研究で証明されている<ref name=Toyooka1999><pubmed>10206237</pubmed></ref><ref name=Bell2000><pubmed>11121172</pubmed></ref>[73][74]。患者の脳サンプルにおいて、14-3-3ηを含む多くの14-3-3アイソフォームのmRNA発現レベルが変化していることが明らかになっている。また、14-3-3εヘテロ接合体ノックアウトマウスは、海馬や皮質の構造変化や[[ワーキングメモリー]]障害などの表現型を表すことから、初めての動物モデルとして提唱されている<ref name=Ikeda2008><pubmed>18658164</pubmed></ref>[75]。 | |||
==参考文献== | |||
2025年3月13日 (木) 20:26時点における版
市村 徹
元防衛大学校 応用科学群応用化学科
田岡万悟
東京都立大学 理学部化学科
DOI:10.14931/bsd.10664 原稿受付日:2025年2月27日 原稿完成日:2025年3月10日
担当編集委員:和田 圭司(国立研究開発法人国立精神・神経医療研究センター)
英:14-3-3 protein
14-3-3タンパク質は分子量約30kDaのサブユニットから構成される2量体タンパク質のファミリーである。ヒトでは9つの分子種(α~σアイソフォーム)の存在が確認されており、各アイソフォームは分子のN末端構造を介してホモ或いはヘテロに結合することで、その内部にU字型の溝構造を形成している。14-3-3は、この溝構造を利用することで多種多様なリン酸化タンパク質と、リン酸化に依存して結合する。脳神経系において、14-3-3は神経突起の伸長、神経分化、細胞移動と生存、神経伝達物質の合成や放出など、さまざまな細胞プロセスに関わることが報告されている。さらに、14-3-3は、クロイツフェルト-ヤコブ病やパーキンソン病をはじめとする神経変性疾患、神経発達疾患、神経精神疾患など、さまざまな神経疾患と遺伝的に関連することも報告されている。
研究の歴史
14-3-3 タンパク質(以下14-3-3)は、1965年B. W. Mooreによって、ウシ脳の可溶性画分において発見され、DEAEセルロースクロマトグラフィーでの分画番号とその後のゲル電気移動での移動位置の組み合わせによって命名された一群の酸性タンパク質である[1][1]。神経細胞に特異的に発現し(可溶性タンパク質の約1%)、神経軸索流によって神経終末(シナプス部)に輸送されることが報告されていたが[2][3][2][3]、その構造と機能は発見から約20年間にわたり不明であった。
1987年、このタンパク質の最初の機能として、カテコールアミンとセロトニン生合成経路の律速段階酵素(チロシン水酸化酵素[TH]とトリプトファン水酸化酵素[TPH])を、カルシウム/カルモデュリン依存性タンパク質キナーゼII (CaMKII) [4][4]によるリン酸化反応に依存して活性化する能力が報告された[5][5]。次いで、このタンパク質の分子構造[6][6]とリン酸化依存性相互作用[7][7]が明らかにされ、さらにこれが互いに構造が類似した複数の成分からなるmulti-geneファミリーを形成していることが証明された[8][8]。
現在ではこのファミリーがヒトから植物、酵母に至る真核生物に広汎に分布すること、また細胞膜から核内に至る細胞反応の調節に深く関与していることが分かっている。
構造
一次構造
14-3-3は等電点 4.6~5.1、分子量約30kDaのサブユニットから構成される二量体分子である[6] [6]。複数の分子種(アイソフォーム)の存在が確認されており、それらの一次構造は生物種間において高度に(60%以上の一致性)保存されている(後述)。各アイソフォームのアミノ酸配列には、リーダー配列や膜貫通領域などの極端な疎水性領域は含まれていない。また結合糖鎖の存在も確認されていない。14-3-3はこれらのアイソフォームがホモ或いはヘテロに結合することで形成される可溶性タンパク質の集団である[6][8] [6][8]。
高次構造
14-3-3を構成する各サブユニットは、9個のα-ヘリックスとそれらをつなぐ短いリンカーからなり、U字型の溝構造を作るためにダイマーを形成する[9][10][9][10](図1A)。片方のサブユニットの第1および第2ヘリックスと、他方のサブユニットの第3および第4ヘリックスは静電相互作用と疎水結合によって結合し、14-3-3のダイマー形成に関与している。一方、それぞれのサブユニットの第3、5、7、9ヘリックスは全体として屈曲し、U字型構造の内部に標的(リガンド)に対する結合部位を形成している(図1B)。14-3-3の各サブユニットはこのリガンド結合部位を介して、1サブユニットあたり1つのリガンドと、ダイマー分子あたり2個のリガンドを同時に結合できる。
上記したように14-3-3ファミリーのアミノ酸配列は生物種を超えて高度に保存されているが、U字構造内部の配列はとりわけ高く保存されている[10] [10](図1)。このU字構造とnonsense mediated mRNA decay factor (SMG7)タンパク質(ナンセンスmRNAの崩壊因子)のN末端領域が形成する構造は、立体的に相同であることが報告されている[11][11]。
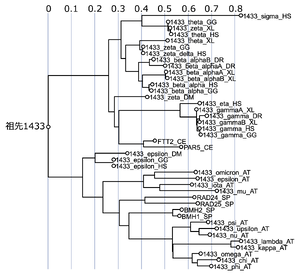
14-3-3オルソログ配列をClustalWを用いてアラインメントし、FastTreeアルゴリズムを用いて作成した。AT, Arabidopsis thaliana; CE, C. elegans; DM, Drosophila melanogaster; DR, Danio rerio; GG, Gallus gallus; HS, Homo sapiens; SC, Saccharomyces cerevisiae; SP, Schizosaccharomyces pombe; and XL, Xenopus laevis.
Aristizábal-Corralesら[12]から改変して転載。
ファミリー
複数の14-3-3アイソフォームが真核生物(動物界、植物界、菌界)を構成するさまざまな細胞から見つかっている。
ヒトでは9つのアイソフォーム(α~σアイソフォーム)が報告されており[8] [8]、これらのうち、αとδアイソフォームは、それぞれβとζアイソフォームのリン酸化型である[13][12]。同様のアイソフォームは、ラット、マウス、ウシ、ヒツジ、ニワトリ、ツメガエル、ゼブラフィシュでも確認されている。各アイソフォームは種内でもまた種間でも高い配列保存が見られる。
線虫(C. elegans)とショウジョウバエ(D. melanogaster)では、それぞれ2つのアイソフォーム(PAR-5とFTT-2:Dm14-3-3εとDm14-3-3ζ)の発現が検出されている[14][15][16][13][14][15]。
植物ではシロイヌナズナで13のアイソフォーム(μ~χアイソフォーム)の存在が確認されている[17][16]。また、ワタには6つ、イネには8つ、オオムギには5つ、タバコには17の潜在的アイソフォームの存在が報告されている[18][17]。菌類の出芽酵母(S. cerevisiae)と分裂酵母(S. pombe)では、それぞれ2つのアイソフォーム(BMH1とBMH2;Rad24とRad25)が見つかっている[19][20][18][19]。
系統樹(図2)から考えると、共通の祖先は、まず、εとβγζηθσのアイソフォームグループの2つに分かれ、その後εは酵母や植物各種アイソフォームと分岐し、βζθσはγηアイソフォームと分岐している。εは原始14-3-3に最も近い配列を持っており、その一方でσは最も遠い配列を持っている(図2)。なお、原核生物(新生細菌や古細菌)の細胞からは、14-3-3ファミリーに属するタンパク質は見つかっていない。
脳神経系における分布
各アイソフォームに特異的なcDNAを用いたin situ hybridization解析では、ラット脳の14-3-3のmRNAはモノアミン合成ニューロンだけでなく、ほとんどすべての領域で発現していることが確認されている。いずれの領域でも、発現は神経細胞で顕著であり、特に嗅球僧帽細胞、海馬錐体細胞、小脳プルキンエ細胞、脳幹や脊髄の運動ニューロンなど、比較的大きな細胞体と高度に発達した樹状突起野を持つ投射型ニューロンで多量に検出されている[21][20]。一方、こうしたmRNAの発現には、ラットの発生初期から一貫して高レベルの発現を示すニューロン、発生初期に高発現を示し、その後成熟期と成体期に発現が減少するニューロン、発生を通じて低レベルの発現を示すニューロンの3つのタイプがあることが確認されている。さらに、ある種のニューロンでは、mRNAの発現にアイソフォーム特異性があることが知られている[22][21]。
各アイソフォームに特異的な抗体を用いた免疫組織化学では、神経細胞の軸索や樹状突起を含む細胞体で発現が確認されている。また、ほとんどの原発性ヒト神経系腫瘍において14-3-3の発現亢進が認められている[23][22]。一方、細胞分画法を組み合わせた生化学的な方法では、14-3-3は主に細胞質画分に検出されるが、かなりの割合がシナプス膜などの不溶性画分にアイソフォーム特異的に検出されている[24][23]。
機能
共通認識配列
14-3-3は、分子全体で形成するU字型構造のリガンド結合部位を介して、タンパク質キナーゼによってリン酸化された様々な標的分子と相互作用する[25][24]。14-3-3が認識するリン酸化の共通配列として、モード1(RxxpS/TxP)[26][25]、モード2(RxxxpS/TxP)[27][26],モード3(pS/TxxCOOH)[28][27]が報告されている(pS/Tはリン酸化セリンまたはリン酸化トレオニン、xは任意のアミノ酸)。共通配列のうち、モード1の配列を含む標的分子が最も多く、その配列中にProが含まれないことも多い。プレ配列と呼ばれるシグナル配列をN末端にもつミトコンドリアタンパク質の前駆体や緑膿菌ADP-ribosyltransferase Exoenzyme S (ExoS)などは、14-3-3との結合にリン酸化を必要としないが、リン酸化タンパク質の場合と同様にU字型構造のリガンド結合部位を介して相互作用すると考えられている[29][28]。
阻害物質と安定化物質
14-3-3のタンパク質-タンパク質間相互作用を標的とすることで、神経疾患をはじめとするさまざまな疾患に治療的に介入しようとする試みが新たに始まっている[30][76]。低分子量化合物による14-3-3タンパク質-タンパク質間の制御には,阻害的と増強的(安定化)という2つの方向性がある。
阻害物質
14-3-3タンパク質のリガンド結合部位に結合して14-3-3のタンパク質-タンパク質間相互作用をマスクする阻害剤として、ペプチド性あるいは非ペプチド性の物質が報告されている。ペプチド性物質としては、ファージディスプレイによって発見されたR18ペプチド(PHCVPRDLSWLDLEANMCLP)や、2分子のR18を短いペプチド鎖で連結したdifopeinなどがある[31][32][77][78]。非ペプチド性の阻害物質としては、フタルイミド誘導体であるBV02がある[33][79]。
安定化物質
14-3-3タンパク質-タンパク質間相互作用の安定化剤としては真菌が産生するジテルペン配糖体であるフシコクシン-A(FC-A)とそれの誘導体であるコチレニン-A(CN-A)などがある。これらは14-3-3とH+-exporting P2-type ATPase (PMA2) [34][80]、嚢胞性線維症膜コンダクタンス制御因子 (cystic fibrosis transmembrane conductance regulator, CFTR)[35][81]、Raf-1[36][82]とのタンパク質-タンパク質間を安定化することが報告されている。
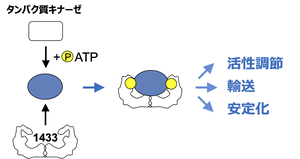
14-3-3はタンパク質キナーゼによってリン酸化された標的と相互作用することで、 (1)標的酵素の活性を調節する、(2)標的タンパク質の細胞内輸送を制御する、(3)標的分子を安定化させる、など多彩な役割をもつことが知られている。
タンパク質レベルにおける役割
14-3-3が標的とするタンパク質は現在300種を超えており、その中には細胞内シグナル伝達経路を構成する一連の酵素・タンパク質が共通して含まれている[37][29]。こうした14-3-3の標的には、タンパク質リン酸化酵素、ユビキチンリガーゼ、代謝酵素、転写因子、細胞骨格成分、イオンチャネルなどがあげられる。14-3-3の結合は、標的リン酸化タンパク質のコンフォメーションや分子間相互作用に影響を与えることで[25][27][38][39][40][24][26][30][31][32]、結果として(1)標的酵素の活性を調節する、(2)標的タンパク質の細胞内輸送(局在)を制御する、(3)標的分子を安定化させる、など多彩な役割をもつことが知られている(図3)。14-3-3の相互作用にはアイソフォーム特異性があることが報告されている[8][8]。
細胞と個体レベルにおける役割
14-3-3はリン酸化シグナルに応じて多種多様な細胞機能を調節している[41][33]。例えば、14-3-3はRaf-1[42][43][44][45][34][35][36][37]やBRAF[40][46][32][38]、MEKK[47][39]などに結合して、それらのキナーゼ活性を制御することで細胞の増殖や分化を調節している。また、PI3K[48][40]やAkt[49][41]などと相互作用し細胞の生存維持や運動を、あるいはBad[50][42]やFKHRL1[51][43]などと複合体を形成し細胞死(アポトーシス)抑制に関与していることが報告されている。分裂酵母では、Cdc25Cホスファターゼに結合することで、DNA修復にかかわる細胞周期チェックポイントシグナルの調節に重要な役割を果たしている[20][52][19][44]。出芽酵母では2つの14-3-3遺伝子の欠損は細胞死を引き起こすことが知られている[19][18]。
脳神経系での機能
脳神経系において、14-3-3は上記したチロシン水酸化酵素やトリプトファン水酸化酵素、セロトニン-N-アセチル転移酵素(NAT)[38] [30]と相互作用し酵素活性を制御することで細胞内モノアミンレベルの調節に関わっている。またCa2+シグナルに応じて神経伝達物質の放出(エキソサイトーシス)を促進する役割が確認されている[53][45]。シナプスにおける働きとしてよりよく理解されているのは、さまざまなイオンチャネルのモジュレーターとしての役割である。代表的な標的チャネルとして、ニコチン性アセチルコリン受容体(α4β2nAChR [54][46])、電位依存性カルシウムチャネル(CaV2.2 [55][47])、カリウムチャネル(KCNK3 [56][48])、塩素チャネル(CFTR [57][49])、NMDA受容体 (NR2Cサブユニット[58][50])などが上げられる。14-3-3のリン酸化依存性相互作用は、チャネル活性を調節し、あるいは細胞内輸送や構造安定性に影響を与えることによって、効率的なシナプス伝達の維持・制御に寄与していることが報告されている。
マウス海馬において阻害ペプチドであるdifopein(dimeric fourteen-three-three peptide inhibitor)を用い14-3-3を阻害すると、CA3-CA1シナプスにおける連想学習と記憶行動を障害され、長期増強(LTP)を抑制されることが明らかになっている[59][51]。またdifopeinは14-3-3とLRRK2キナーゼの相互作用を妨害し、マウス海馬の初代培養ニューロンの神経突起伸長を短縮化させることも知られている[60][52]。14-3-3はδカテニン、NUDEL、LIMドメイン含有キナーゼ (LIMK)やコフィリンなどに相互作用することで、神経細胞の移動、神経分化、形態形成、構造可塑性を制御することが報告されている[61][53]。これらの標的に加えて、14-3-3は、次項に列挙したものを含む多種多様なタンパク質と結合することによって、脳神経系で機能していると考えられる。
疾患との関わり
14-3-3は脳神経系に多量に存在すること、その発現にアイソフォーム特異性があること、さらに14-3-3が標的とするタンパク質の多くが神経機能に重要な役割を担っていることなどから、14-3-3と神経疾患との関わりについては多くの報告がある。
クロイツフェルト-ヤコブ病
プリオンタンパク質(PrP)のミスフォールディングによって引き起こされる致死性の海綿状脳症である。治療法は現在開発されておらず、対症療法が主体である。生前の確定診断法はないが、脳脊髄液由来の14-3-3タンパク質がクロイツフェルト-ヤコブ病の信頼できるマーカーになりうることが報告されている[62][54]。14-3-3タンパク質の脳脊髄液への漏出は、クロイツフェルト-ヤコブ病による脳神経細胞の広範な破壊に依存している可能性が指摘されている[63]。[55]
アルツハイマー病
アルツハイマー病とは数百万人が罹患しており、徐々に悪化する神経変性疾患である。アミロイド斑と神経原線維変化という2つの病理学的特徴があげられる。神経原線維変化は、異常にリン酸化されたタウタンパク質の凝集によって主に構成されており、14-3-3は、リン酸化されたタウに結合し[64][56]その機能や安定性を制御することで、神経原線維変化へのタウの凝集を調節していると推定されている[65][66][57][58]。また、14-3-3は、アルツハイマー病のアミロイド斑の主成分であるアミロイドβのクリアランスに関係することも報告されている[66][58]。さらに、14-3-3はアルツハイマー病患者の脳や脳脊髄液中で発現レベルが異常化していることも判明しており、その異常がアルツハイマー病の病態マーカーになる可能性も示唆されている。
パーキンソン病
パーキンソン病とは黒質のドーパミン神経細胞の障害によって発症する神経変性疾患である。患者の脳では、レビー小体と呼ばれる線維状タンパク質を含む不溶性の凝集体が観察され、14-3-3はレビー小体に存在することが証明されている[67][59]。さらに14-3-3は、パーキンソン病の発症と進行に関係する3大主要因子であるLRRK2、α-シヌクレイン、パーキンらのすべてと相互作用し、その活性を制御したり細胞内局在を変化させたり、あるいは安定化するなどの役割が報告されている[68][60]。14-3-3のアミノ酸配列にはα-シヌクレインと相同的な領域がある[69][61]。
筋萎縮性側索硬化症
筋萎縮性側索硬化症とは筋力低下と筋萎縮を特徴とする致死的な運動ニューロン疾患である。神経病理学的特徴として、ニューロフィラメント(NF)やTDP-43、SODなどを含むレビー小体様ヒアリン封入体(LBHI)の存在があげられる。患者でも動物モデルでも、14-3-3はLBHIに存在することが証明されている[70][62]。14-3-3は、NF軽鎖にリン酸化依存的に結合して安定化させることで、ニューロフィラメント軽鎖を介する凝集体形成を抑制すると推定されている[71][63]。また、14-3-3θのmRNA発現レベルが患者の脊髄で上昇していることも明らかにされている[72] [64]。
ポリグルタミン病
脊髄小脳失調症
脊髄小脳失調症1型(SCA1)は、アタキシン-1におけるポリグルタミン(ポリQ)鎖の異状伸長によって引き起こされる致死性の神経変性疾患である。SCA1における病態の顕著な部位は小脳プルキンエ細胞であり、そこでは異状アタキシン-1が核に入り込み、封入体を形成する。14-3-3は、リン酸化されたアタキシン-1に結合し[73][65]、脱リン酸化を防ぐとともに核への移行を阻害する役割が報告されている[74][66]。14-3-3εの部分欠損がSCA1の小脳表現型を改善するという所見は、14-3-3がSCA1の病因に寄与していることを示唆している[75][67]。
ハンチントン病
ハンチントン病とは常染色体顕性遺伝の進行性神経変性疾患であり、ハンチンチンタンパク質におけるポリQ鎖の伸長によって引き起こされる。14-3-3は、この異常ハンチンチンの封入体形成を促進することで、ミスフォールディングしたハンチンチンを除去する役割が示唆されている[76][68]。siRNAによる14-3-3ζの減少は異状ハンチンチンの封入体形成を阻害したことから[77][69]、14-3-3が封入体形成に関与することも示唆されている。
ミラー・ディーカー症候群
ミラー・ディーカー症候群とはlissencephaly(滑脳症)のより重篤な型であり、ヒトとマウスに脳の異常を伴うまれな神経細胞移動障害を引き起こす。14-3-3ε遺伝子YWHAEが存在する染色体領域17p13-3はミラー・ディーカー症候群患者で常に欠損していることが明らかになっている[78][70]。14-3-3εは、CDK5でリン酸化されたNUDELに結合し、NUDELのリン酸化状態を保護することで、神経細胞の移動を制御していることが報告されている[78] [70]。NUDELは、LIS1結合タンパク質として知られており、この複合体は細胞質ダイニン重鎖機能を制御する、神経細胞の移動に必須な因子である[79][71]。このことは、14-3-3εが神経細胞移動に必須の役割を担っていることを示唆している。実際、14-3-3ε欠損マウスは、海馬の欠損、皮質の菲薄化、移動距離の減少、神経細胞死の増加などの脳構造の異状を示す。しかし14-3-3εの単独欠失では、発症しないことが報告されている[80][72]。
統合失調症
統合失調症とは陽性症状、陰性症状、認知症状の組み合わせによって特徴付けられる精神神経疾患であり、高い遺伝性があることが示されている。遺伝子解析により、14-3-3η遺伝子YWHAHが存在する染色体領域22q12-13との関連が示唆されている。実際、14-3-3η遺伝子の一塩基多型(SNP)との有意な関連は、様々なヒトサンプルを用いた多くの研究で証明されている[81][82][73][74]。患者の脳サンプルにおいて、14-3-3ηを含む多くの14-3-3アイソフォームのmRNA発現レベルが変化していることが明らかになっている。また、14-3-3εヘテロ接合体ノックアウトマウスは、海馬や皮質の構造変化やワーキングメモリー障害などの表現型を表すことから、初めての動物モデルとして提唱されている[83][75]。
参考文献
- ↑
Moore, B.W. (1965).
A soluble protein characteristic of the nervous system. Biochemical and biophysical research communications, 19(6), 739-44. [PubMed:4953930] [WorldCat] [DOI] - ↑
Erickson, P.F., & Moore, B.W. (1980).
Investigation of the axonal transport of three acidic, soluble proteins (14-3-2, 14-3-3, and S-100) in the rabbit visual system. Journal of neurochemistry, 35(1), 232-41. [PubMed:6161216] [WorldCat] [DOI] - ↑
Boston, P.F., Jackson, P., Kynoch, P.A., & Thompson, R.J. (1982).
Purification, properties, and immunohistochemical localisation of human brain 14-3-3 protein. Journal of neurochemistry, 38(5), 1466-74. [PubMed:7038050] [WorldCat] [DOI] - ↑
Yamauchi, T., Nakata, H., & Fujisawa, H. (1981).
A new activator protein that activates tryptophan 5-monooxygenase and tyrosine 3-monooxygenase in the presence of Ca2+-, calmodulin-dependent protein kinase. Purification and characterization. The Journal of biological chemistry, 256(11), 5404-9. [PubMed:6113235] [WorldCat] - ↑
Ichimura, T., Isobe, T., Okuyama, T., Yamauchi, T., & Fujisawa, H. (1987).
Brain 14-3-3 protein is an activator protein that activates tryptophan 5-monooxygenase and tyrosine 3-monooxygenase in the presence of Ca2+,calmodulin-dependent protein kinase II. FEBS letters, 219(1), 79-82. [PubMed:2885229] [WorldCat] [DOI] - ↑ 6.0 6.1 6.2
Ichimura, T., Isobe, T., Okuyama, T., Takahashi, N., Araki, K., Kuwano, R., & Takahashi, Y. (1988).
Molecular cloning of cDNA coding for brain-specific 14-3-3 protein, a protein kinase-dependent activator of tyrosine and tryptophan hydroxylases. Proceedings of the National Academy of Sciences of the United States of America, 85(19), 7084-8. [PubMed:2902623] [PMC] [WorldCat] [DOI] - ↑
Furukawa, Y., Ikuta, N., Omata, S., Yamauchi, T., Isobe, T., & Ichimura, T. (1993).
Demonstration of the phosphorylation-dependent interaction of tryptophan hydroxylase with the 14-3-3 protein. Biochemical and biophysical research communications, 194(1), 144-9. [PubMed:8101440] [WorldCat] [DOI] - ↑ 8.0 8.1 8.2 8.3
Shinkai, F., & Isobe, T. (1996).
[14-3-3 protein family: the structure and function in cell signaling pathway]. Tanpakushitsu kakusan koso. Protein, nucleic acid, enzyme, 41(4), 313-26. [PubMed:8721374] [WorldCat] - ↑
Xiao, B., Smerdon, S.J., Jones, D.H., Dodson, G.G., Soneji, Y., Aitken, A., & Gamblin, S.J. (1995).
Structure of a 14-3-3 protein and implications for coordination of multiple signalling pathways. Nature, 376(6536), 188-91. [PubMed:7603573] [WorldCat] [DOI] - ↑ 10.0 10.1
Liu, D., Bienkowska, J., Petosa, C., Collier, R.J., Fu, H., & Liddington, R. (1995).
Crystal structure of the zeta isoform of the 14-3-3 protein. Nature, 376(6536), 191-4. [PubMed:7603574] [WorldCat] [DOI] - ↑
Fukuhara, N., Ebert, J., Unterholzner, L., Lindner, D., Izaurralde, E., & Conti, E. (2005).
SMG7 is a 14-3-3-like adaptor in the nonsense-mediated mRNA decay pathway. Molecular cell, 17(4), 537-47. [PubMed:15721257] [WorldCat] [DOI] - ↑
Aristizábal-Corrales, D., Fontrodona, L., Porta-de-la-Riva, M., Guerra-Moreno, A., Cerón, J., & Schwartz, S. (2012).
The 14-3-3 gene par-5 is required for germline development and DNA damage response in Caenorhabditis elegans. Journal of cell science, 125(Pt 7), 1716-26. [PubMed:22328524] [WorldCat] [DOI] - ↑
Aitken, A., Howell, S., Jones, D., Madrazo, J., & Patel, Y. (1995).
14-3-3 alpha and delta are the phosphorylated forms of raf-activating 14-3-3 beta and zeta. In vivo stoichiometric phosphorylation in brain at a Ser-Pro-Glu-Lys MOTIF. The Journal of biological chemistry, 270(11), 5706-9. [PubMed:7890696] [WorldCat] [DOI] - ↑
Berdichevsky, A., Viswanathan, M., Horvitz, H.R., & Guarente, L. (2006).
C. elegans SIR-2.1 interacts with 14-3-3 proteins to activate DAF-16 and extend life span. Cell, 125(6), 1165-77. [PubMed:16777605] [WorldCat] [DOI] - ↑
Skoulakis, E.M., & Davis, R.L. (1996).
Olfactory learning deficits in mutants for leonardo, a Drosophila gene encoding a 14-3-3 protein. Neuron, 17(5), 931-44. [PubMed:8938125] [WorldCat] [DOI] - ↑
Benton, R., Palacios, I.M., & St Johnston, D. (2002).
Drosophila 14-3-3/PAR-5 is an essential mediator of PAR-1 function in axis formation. Developmental cell, 3(5), 659-71. [PubMed:12431373] [WorldCat] [DOI] - ↑
DeLille, J.M., Sehnke, P.C., & Ferl, R.J. (2001).
The arabidopsis 14-3-3 family of signaling regulators. Plant physiology, 126(1), 35-8. [PubMed:11351068] [PMC] [WorldCat] [DOI] - ↑
Denison, F.C., Paul, A.L., Zupanska, A.K., & Ferl, R.J. (2011).
14-3-3 proteins in plant physiology. Seminars in cell & developmental biology, 22(7), 720-7. [PubMed:21907297] [WorldCat] [DOI] - ↑ 19.0 19.1
van Heusden, G.P., Griffiths, D.J., Ford, J.C., Chin-A-Woeng, T.F., Schrader, P.A., Carr, A.M., & Steensma, H.Y. (1995).
The 14-3-3 proteins encoded by the BMH1 and BMH2 genes are essential in the yeast Saccharomyces cerevisiae and can be replaced by a plant homologue. European journal of biochemistry, 229(1), 45-53. [PubMed:7744048] [WorldCat] - ↑ 20.0 20.1
Peng, C.Y., Graves, P.R., Thoma, R.S., Wu, Z., Shaw, A.S., & Piwnica-Worms, H. (1997).
Mitotic and G2 checkpoint control: regulation of 14-3-3 protein binding by phosphorylation of Cdc25C on serine-216. Science (New York, N.Y.), 277(5331), 1501-5. [PubMed:9278512] [WorldCat] [DOI] - ↑
Watanabe, M., Isobe, T., Okuyama, T., Ichimura, T., Kuwano, R., Takahashi, Y., & Kondo, H. (1991).
Molecular cloning of cDNA to rat 14-3-3 eta chain polypeptide and the neuronal expression of the mRNA in the central nervous system. Brain research. Molecular brain research, 10(2), 151-8. [PubMed:1649368] [WorldCat] [DOI] - ↑
Watanabe, M., Isobe, T., Ichimura, T., Kuwano, R., Takahashi, Y., Kondo, H., & Inoue, Y. (1994).
Molecular cloning of rat cDNAs for the zeta and theta subtypes of 14-3-3 protein and differential distributions of their mRNAs in the brain. Brain research. Molecular brain research, 25(1-2), 113-21. [PubMed:7984035] [WorldCat] [DOI] - ↑
Cao, W.D., Zhang, X., Zhang, J.N., Yang, Z.J., Zhen, H.N., Cheng, G., ..., & Gao, D. (2006).
Immunocytochemical detection of 14-3-3 in primary nervous system tumors. Journal of neuro-oncology, 77(2), 125-30. [PubMed:16292484] [WorldCat] [DOI] - ↑
Martin, H., Rostas, J., Patel, Y., & Aitken, A. (1994).
Subcellular localisation of 14-3-3 isoforms in rat brain using specific antibodies. Journal of neurochemistry, 63(6), 2259-65. [PubMed:7964746] [WorldCat] [DOI] - ↑ 25.0 25.1
Tzivion, G., & Avruch, J. (2002).
14-3-3 proteins: active cofactors in cellular regulation by serine/threonine phosphorylation. The Journal of biological chemistry, 277(5), 3061-4. [PubMed:11709560] [WorldCat] [DOI] - ↑
Muslin, A.J., Tanner, J.W., Allen, P.M., & Shaw, A.S. (1996).
Interaction of 14-3-3 with signaling proteins is mediated by the recognition of phosphoserine. Cell, 84(6), 889-97. [PubMed:8601312] [WorldCat] [DOI] - ↑ 27.0 27.1
Yaffe, M.B., Rittinger, K., Volinia, S., Caron, P.R., Aitken, A., Leffers, H., ..., & Cantley, L.C. (1997).
The structural basis for 14-3-3:phosphopeptide binding specificity. Cell, 91(7), 961-71. [PubMed:9428519] [WorldCat] [DOI] - ↑
Ganguly, S., Weller, J.L., Ho, A., Chemineau, P., Malpaux, B., & Klein, D.C. (2005).
Melatonin synthesis: 14-3-3-dependent activation and inhibition of arylalkylamine N-acetyltransferase mediated by phosphoserine-205. Proceedings of the National Academy of Sciences of the United States of America, 102(4), 1222-7. [PubMed:15644438] [PMC] [WorldCat] [DOI] - ↑
Masters, S.C., Pederson, K.J., Zhang, L., Barbieri, J.T., & Fu, H. (1999).
Interaction of 14-3-3 with a nonphosphorylated protein ligand, exoenzyme S of Pseudomonas aeruginosa. Biochemistry, 38(16), 5216-21. [PubMed:10213629] [WorldCat] [DOI] - ↑
Stevers, L.M., Sijbesma, E., Botta, M., MacKintosh, C., Obsil, T., Landrieu, I., ..., & Ottmann, C. (2018).
Modulators of 14-3-3 Protein-Protein Interactions. Journal of medicinal chemistry, 61(9), 3755-3778. [PubMed:28968506] [PMC] [WorldCat] [DOI] - ↑
Wang, B., Yang, H., Liu, Y.C., Jelinek, T., Zhang, L., Ruoslahti, E., & Fu, H. (1999).
Isolation of high-affinity peptide antagonists of 14-3-3 proteins by phage display. Biochemistry, 38(38), 12499-504. [PubMed:10493820] [WorldCat] [DOI] - ↑
Masters, S.C., & Fu, H. (2001).
14-3-3 proteins mediate an essential anti-apoptotic signal. The Journal of biological chemistry, 276(48), 45193-200. [PubMed:11577088] [WorldCat] [DOI] - ↑
Mancini, M., Corradi, V., Petta, S., Barbieri, E., Manetti, F., Botta, M., & Santucci, M.A. (2011).
A new nonpeptidic inhibitor of 14-3-3 induces apoptotic cell death in chronic myeloid leukemia sensitive or resistant to imatinib. The Journal of pharmacology and experimental therapeutics, 336(3), 596-604. [PubMed:21041536] [WorldCat] [DOI] - ↑
Würtele, M., Jelich-Ottmann, C., Wittinghofer, A., & Oecking, C. (2003).
Structural view of a fungal toxin acting on a 14-3-3 regulatory complex. The EMBO journal, 22(5), 987-94. [PubMed:12606564] [PMC] [WorldCat] [DOI] - ↑
Stevers, L.M., Lam, C.V., Leysen, S.F., Meijer, F.A., van Scheppingen, D.S., de Vries, R.M., ..., & Ottmann, C. (2016).
Characterization and small-molecule stabilization of the multisite tandem binding between 14-3-3 and the R domain of CFTR. Proceedings of the National Academy of Sciences of the United States of America, 113(9), E1152-61. [PubMed:26888287] [PMC] [WorldCat] [DOI] - ↑
Molzan, M., Kasper, S., Röglin, L., Skwarczynska, M., Sassa, T., Inoue, T., ..., & Ottmann, C. (2013).
Stabilization of physical RAF/14-3-3 interaction by cotylenin A as treatment strategy for RAS mutant cancers. ACS chemical biology, 8(9), 1869-75. [PubMed:23808890] [WorldCat] [DOI] - ↑
Kakiuchi, K., Yamauchi, Y., Taoka, M., Iwago, M., Fujita, T., Ito, T., ..., & Ichimura, T. (2007).
Proteomic analysis of in vivo 14-3-3 interactions in the yeast Saccharomyces cerevisiae. Biochemistry, 46(26), 7781-92. [PubMed:17559233] [WorldCat] [DOI] - ↑ 38.0 38.1
Obsil, T., Ghirlando, R., Klein, D.C., Ganguly, S., & Dyda, F. (2001).
Crystal structure of the 14-3-3zeta:serotonin N-acetyltransferase complex. a role for scaffolding in enzyme regulation. Cell, 105(2), 257-67. [PubMed:11336675] [WorldCat] [DOI] - ↑
Yaffe, M.B. (2002).
How do 14-3-3 proteins work?-- Gatekeeper phosphorylation and the molecular anvil hypothesis. FEBS letters, 513(1), 53-7. [PubMed:11911880] [WorldCat] [DOI] - ↑ 40.0 40.1
Park, E., Rawson, S., Li, K., Kim, B.W., Ficarro, S.B., Pino, G.G., ..., & Eck, M.J. (2019).
Architecture of autoinhibited and active BRAF-MEK1-14-3-3 complexes. Nature, 575(7783), 545-550. [PubMed:31581174] [PMC] [WorldCat] [DOI] - ↑
Morrison, D.K. (2009).
The 14-3-3 proteins: integrators of diverse signaling cues that impact cell fate and cancer development. Trends in cell biology, 19(1), 16-23. [PubMed:19027299] [PMC] [WorldCat] [DOI] - ↑
Freed, E., Symons, M., Macdonald, S.G., McCormick, F., & Ruggieri, R. (1994).
Binding of 14-3-3 proteins to the protein kinase Raf and effects on its activation. Science (New York, N.Y.), 265(5179), 1713-6. [PubMed:8085158] [WorldCat] [DOI] - ↑
Irie, K., Gotoh, Y., Yashar, B.M., Errede, B., Nishida, E., & Matsumoto, K. (1994).
Stimulatory effects of yeast and mammalian 14-3-3 proteins on the Raf protein kinase. Science (New York, N.Y.), 265(5179), 1716-9. [PubMed:8085159] [WorldCat] [DOI] - ↑
Fantl, W.J., Muslin, A.J., Kikuchi, A., Martin, J.A., MacNicol, A.M., Gross, R.W., & Williams, L.T. (1994).
Activation of Raf-1 by 14-3-3 proteins. Nature, 371(6498), 612-4. [PubMed:7935795] [WorldCat] [DOI] - ↑
Fu, H., Xia, K., Pallas, D.C., Cui, C., Conroy, K., Narsimhan, R.P., ..., & Roberts, T.M. (1994).
Interaction of the protein kinase Raf-1 with 14-3-3 proteins. Science (New York, N.Y.), 266(5182), 126-9. [PubMed:7939632] [WorldCat] [DOI] - ↑
Kondo, Y., Ognjenović, J., Banerjee, S., Karandur, D., Merk, A., Kulhanek, K., ..., & Kuriyan, J. (2019).
Cryo-EM structure of a dimeric B-Raf:14-3-3 complex reveals asymmetry in the active sites of B-Raf kinases. Science (New York, N.Y.), 366(6461), 109-115. [PubMed:31604311] [PMC] [WorldCat] [DOI] - ↑
Fanger, G.R., Widmann, C., Porter, A.C., Sather, S., Johnson, G.L., & Vaillancourt, R.R. (1998).
14-3-3 proteins interact with specific MEK kinases. The Journal of biological chemistry, 273(6), 3476-83. [PubMed:9452471] [WorldCat] [DOI] - ↑
Bonnefoy-Bérard, N., Liu, Y.C., von Willebrand, M., Sung, A., Elly, C., Mustelin, T., ..., & Altman, A. (1995).
Inhibition of phosphatidylinositol 3-kinase activity by association with 14-3-3 proteins in T cells. Proceedings of the National Academy of Sciences of the United States of America, 92(22), 10142-6. [PubMed:7479742] [PMC] [WorldCat] [DOI] - ↑
Zhang, L., Chen, J., & Fu, H. (1999).
Suppression of apoptosis signal-regulating kinase 1-induced cell death by 14-3-3 proteins. Proceedings of the National Academy of Sciences of the United States of America, 96(15), 8511-5. [PubMed:10411906] [PMC] [WorldCat] [DOI] - ↑
Zha, J., Harada, H., Yang, E., Jockel, J., & Korsmeyer, S.J. (1996).
Serine phosphorylation of death agonist BAD in response to survival factor results in binding to 14-3-3 not BCL-X(L). Cell, 87(4), 619-28. [PubMed:8929531] [WorldCat] [DOI] - ↑
Brunet, A., Bonni, A., Zigmond, M.J., Lin, M.Z., Juo, P., Hu, L.S., ..., & Greenberg, M.E. (1999).
Akt promotes cell survival by phosphorylating and inhibiting a Forkhead transcription factor. Cell, 96(6), 857-68. [PubMed:10102273] [WorldCat] [DOI] - ↑
Ford, J.C., al-Khodairy, F., Fotou, E., Sheldrick, K.S., Griffiths, D.J., & Carr, A.M. (1994).
14-3-3 protein homologs required for the DNA damage checkpoint in fission yeast. Science (New York, N.Y.), 265(5171), 533-5. [PubMed:8036497] [WorldCat] [DOI] - ↑
Morgan, A., & Burgoyne, R.D. (1992).
Exo1 and Exo2 proteins stimulate calcium-dependent exocytosis in permeabilized adrenal chromaffin cells. Nature, 355(6363), 833-6. [PubMed:1538762] [WorldCat] [DOI] - ↑
Jeanclos, E.M., Lin, L., Treuil, M.W., Rao, J., DeCoster, M.A., & Anand, R. (2001).
The chaperone protein 14-3-3eta interacts with the nicotinic acetylcholine receptor alpha 4 subunit. Evidence for a dynamic role in subunit stabilization. The Journal of biological chemistry, 276(30), 28281-90. [PubMed:11352901] [WorldCat] [DOI] - ↑
Li, Y., Wu, Y., & Zhou, Y. (2006).
Modulation of inactivation properties of CaV2.2 channels by 14-3-3 proteins. Neuron, 51(6), 755-71. [PubMed:16982421] [WorldCat] [DOI] - ↑
O'Kelly, I., Butler, M.H., Zilberberg, N., & Goldstein, S.A. (2002).
Forward transport. 14-3-3 binding overcomes retention in endoplasmic reticulum by dibasic signals. Cell, 111(4), 577-88. [PubMed:12437930] [WorldCat] [DOI] - ↑
Liang, X., Da Paula, A.C., Bozóky, Z., Zhang, H., Bertrand, C.A., Peters, K.W., ..., & Frizzell, R.A. (2012).
Phosphorylation-dependent 14-3-3 protein interactions regulate CFTR biogenesis. Molecular biology of the cell, 23(6), 996-1009. [PubMed:22278744] [PMC] [WorldCat] [DOI] - ↑
Chen, B.S., & Roche, K.W. (2009).
Growth factor-dependent trafficking of cerebellar NMDA receptors via protein kinase B/Akt phosphorylation of NR2C. Neuron, 62(4), 471-8. [PubMed:19477150] [PMC] [WorldCat] [DOI] - ↑
Qiao, H., Foote, M., Graham, K., Wu, Y., & Zhou, Y. (2014).
14-3-3 proteins are required for hippocampal long-term potentiation and associative learning and memory. The Journal of neuroscience : the official journal of the Society for Neuroscience, 34(14), 4801-8. [PubMed:24695700] [PMC] [WorldCat] [DOI] - ↑
Lavalley, N.J., Slone, S.R., Ding, H., West, A.B., & Yacoubian, T.A. (2016).
14-3-3 Proteins regulate mutant LRRK2 kinase activity and neurite shortening. Human molecular genetics, 25(1), 109-22. [PubMed:26546614] [PMC] [WorldCat] [DOI] - ↑
Cornell, B., & Toyo-Oka, K. (2017).
14-3-3 Proteins in Brain Development: Neurogenesis, Neuronal Migration and Neuromorphogenesis. Frontiers in molecular neuroscience, 10, 318. [PubMed:29075177] [PMC] [WorldCat] [DOI] - ↑
Hsich, G., Kenney, K., Gibbs, C.J., Lee, K.H., & Harrington, M.G. (1996).
The 14-3-3 brain protein in cerebrospinal fluid as a marker for transmissible spongiform encephalopathies. The New England journal of medicine, 335(13), 924-30. [PubMed:8782499] [WorldCat] [DOI] - ↑
Muayqil, T., Gronseth, G., & Camicioli, R. (2012).
Evidence-based guideline: diagnostic accuracy of CSF 14-3-3 protein in sporadic Creutzfeldt-Jakob disease: report of the guideline development subcommittee of the American Academy of Neurology. Neurology, 79(14), 1499-506. [PubMed:22993290] [PMC] [WorldCat] [DOI] - ↑
Hashiguchi, M., Sobue, K., & Paudel, H.K. (2000).
14-3-3zeta is an effector of tau protein phosphorylation. The Journal of biological chemistry, 275(33), 25247-54. [PubMed:10840038] [WorldCat] [DOI] - ↑
Shimada, T., Fournier, A.E., & Yamagata, K. (2013).
Neuroprotective function of 14-3-3 proteins in neurodegeneration. BioMed research international, 2013, 564534. [PubMed:24364034] [PMC] [WorldCat] [DOI] - ↑ 66.0 66.1
Abdi, G., Jain, M., Patil, N., Upadhyay, B., Vyas, N., Dwivedi, M., & Kaushal, R.S. (2024).
14-3-3 proteins-a moonlight protein complex with therapeutic potential in neurological disorder: in-depth review with Alzheimer's disease. Frontiers in molecular biosciences, 11, 1286536. [PubMed:38375509] [PMC] [WorldCat] [DOI] - ↑
Kawamoto, Y., Akiguchi, I., Nakamura, S., Honjyo, Y., Shibasaki, H., & Budka, H. (2002).
14-3-3 proteins in Lewy bodies in Parkinson disease and diffuse Lewy body disease brains. Journal of neuropathology and experimental neurology, 61(3), 245-53. [PubMed:11895039] [WorldCat] [DOI] - ↑
Giusto, E., Yacoubian, T.A., Greggio, E., & Civiero, L. (2021).
Pathways to Parkinson's disease: a spotlight on 14-3-3 proteins. NPJ Parkinson's disease, 7(1), 85. [PubMed:34548498] [PMC] [WorldCat] [DOI] - ↑
Ostrerova, N., Petrucelli, L., Farrer, M., Mehta, N., Choi, P., Hardy, J., & Wolozin, B. (1999).
alpha-Synuclein shares physical and functional homology with 14-3-3 proteins. The Journal of neuroscience : the official journal of the Society for Neuroscience, 19(14), 5782-91. [PubMed:10407019] [PMC] [WorldCat] [DOI] - ↑
Kawamoto, Y., Akiguchi, I., Nakamura, S., & Budka, H. (2004).
14-3-3 proteins in Lewy body-like hyaline inclusions in patients with sporadic amyotrophic lateral sclerosis. Acta neuropathologica, 108(6), 531-7. [PubMed:15378322] [WorldCat] [DOI] - ↑
Miao, L., Teng, J., Lin, J., Liao, X., & Chen, J. (2013).
14-3-3 proteins interact with neurofilament protein-L and regulate dynamic assembly of neurofilaments. Journal of cell science, 126(Pt 2), 427-36. [PubMed:23230147] [WorldCat] [DOI] - ↑
Malaspina, A., Kaushik, N., & de Belleroche, J. (2000).
A 14-3-3 mRNA is up-regulated in amyotrophic lateral sclerosis spinal cord. Journal of neurochemistry, 75(6), 2511-20. [PubMed:11080204] [WorldCat] [DOI] - ↑
Chen, H.K., Fernandez-Funez, P., Acevedo, S.F., Lam, Y.C., Kaytor, M.D., Fernandez, M.H., ..., & Zoghbi, H.Y. (2003).
Interaction of Akt-phosphorylated ataxin-1 with 14-3-3 mediates neurodegeneration in spinocerebellar ataxia type 1. Cell, 113(4), 457-68. [PubMed:12757707] [WorldCat] [DOI] - ↑
Lai, S., O'Callaghan, B., Zoghbi, H.Y., & Orr, H.T. (2011).
14-3-3 Binding to ataxin-1(ATXN1) regulates its dephosphorylation at Ser-776 and transport to the nucleus. The Journal of biological chemistry, 286(40), 34606-16. [PubMed:21835928] [PMC] [WorldCat] [DOI] - ↑
Jafar-Nejad, P., Ward, C.S., Richman, R., Orr, H.T., & Zoghbi, H.Y. (2011).
Regional rescue of spinocerebellar ataxia type 1 phenotypes by 14-3-3epsilon haploinsufficiency in mice underscores complex pathogenicity in neurodegeneration. Proceedings of the National Academy of Sciences of the United States of America, 108(5), 2142-7. [PubMed:21245341] [PMC] [WorldCat] [DOI] - ↑
Kaneko, K., & Hachiya, N.S. (2006).
The alternative role of 14-3-3 zeta as a sweeper of misfolded proteins in disease conditions. Medical hypotheses, 67(1), 169-71. [PubMed:16516399] [WorldCat] [DOI] - ↑
Omi, K., Hachiya, N.S., Tanaka, M., Tokunaga, K., & Kaneko, K. (2008).
14-3-3zeta is indispensable for aggregate formation of polyglutamine-expanded huntingtin protein. Neuroscience letters, 431(1), 45-50. [PubMed:18078716] [WorldCat] [DOI] - ↑ 78.0 78.1
Toyo-oka, K., Shionoya, A., Gambello, M.J., Cardoso, C., Leventer, R., Ward, H.L., ..., & Wynshaw-Boris, A. (2003).
14-3-3epsilon is important for neuronal migration by binding to NUDEL: a molecular explanation for Miller-Dieker syndrome. Nature genetics, 34(3), 274-85. [PubMed:12796778] [WorldCat] [DOI] - ↑
Taya, S., Shinoda, T., Tsuboi, D., Asaki, J., Nagai, K., Hikita, T., ..., & Kaibuchi, K. (2007).
DISC1 regulates the transport of the NUDEL/LIS1/14-3-3epsilon complex through kinesin-1. The Journal of neuroscience : the official journal of the Society for Neuroscience, 27(1), 15-26. [PubMed:17202468] [PMC] [WorldCat] [DOI] - ↑
Denommé-Pichon, A.S., Collins, S.C., Bruel, A.L., Mikhaleva, A., Wagner, C., Vancollie, V.E., ..., & Yalcin, B. (2023).
YWHAE loss of function causes a rare neurodevelopmental disease with brain abnormalities in human and mouse. Genetics in medicine : official journal of the American College of Medical Genetics, 25(7), 100835. [PubMed:36999555] [WorldCat] [DOI] - ↑
Toyooka, K., Muratake, T., Tanaka, T., Igarashi, S., Watanabe, H., Takeuchi, H., ..., & Takahashi, Y. (1999).
14-3-3 protein eta chain gene (YWHAH) polymorphism and its genetic association with schizophrenia. American journal of medical genetics, 88(2), 164-7. [PubMed:10206237] [WorldCat] - ↑
Bell, R., Munro, J., Russ, C., Powell, J.F., Bruinvels, A., Kerwin, R.W., & Collier, D.A. (2000).
Systematic screening of the 14-3-3 eta (eta) chain gene for polymorphic variants and case-control analysis in schizophrenia. American journal of medical genetics, 96(6), 736-43. [PubMed:11121172] [WorldCat] - ↑
Ikeda, M., Hikita, T., Taya, S., Uraguchi-Asaki, J., Toyo-oka, K., Wynshaw-Boris, A., ..., & Iwata, N. (2008).
Identification of YWHAE, a gene encoding 14-3-3epsilon, as a possible susceptibility gene for schizophrenia. Human molecular genetics, 17(20), 3212-22. [PubMed:18658164] [WorldCat] [DOI]