ミクログリア
津田 誠*、齊藤 秀俊、山下 智大、松田 烈士
九州大学大学院薬学研究院ライフイノベーション分野、薬理学分野
DOI:10.14931/bsd.6941 原稿受付日:2016年2月29日 原稿完成日:2016年3月15日
担当編集委員:村上 富士夫(大阪大学 大学院生命機能研究科)
*corresponding author
英語:microglia 独:Microglial-Zelle 仏:microglie
同義語:マイクログリア、小膠細胞、Hortega細胞
ミクログリアは中枢神経系グリア細胞の一つで、中枢の免疫担当細胞として知られ、中枢神経系に存在する常在性マクロファージとも呼ばれる。アストロサイトやオリゴデンドロサイトなどとは異なり、胎生期卵黄嚢で発生する前駆細胞を起源とする。正常状態では脳や脊髄に点在し、細胞同士がお互いに重ならず分布している。ミクログリアは細長い突起を有し、それをダイナミックに動かし、シナプスや軸索等に接触させその機能を監視・調節していることが徐々に明らかになっている。病態時には、細胞体の肥大化や細胞増殖を伴い活性化状態となる。細胞膜受容体を含む様々な分子の発現を変化させ、病巣部への移動、ダメージを受けた細胞やアミロイドβタンパク質(Aβ)などの細胞外タンパク質の貪食、液性因子(炎症性因子、細胞障害性因子、栄養因子など)の産生放出を引き起こす。中枢神経系疾患のメカニズムに大きな役割を有しており、治療薬開発における有望なターゲットとして注目されている。
歴史
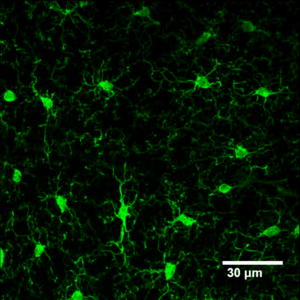
IIba1抗体による免疫組織染色(著者原図)
ミクログリアは1920年代にPio del Rio-Hortegaによって中枢神経系における「第3のエレメント」として位置付けられ、「ミクログリア」と命名された。彼の一連の研究から、ミクログリアの発達初期に脳への浸潤し、その細胞はアメボイド形態で中胚葉由来であろうということ、成体脳では枝分かれした形態で一定間隔を保って分布し、病態ではアメボイド形態になり、移動、増殖、貪食能を有するという仮説が立てられた[1]。これらは、現在までの多くの研究から明らかになったミクログリアの特徴や機能にマッチしており、非常に先駆的な研究といえる(彼はFather of Microgliaとも呼ばれている)。
その後、中枢神経系においてミクログリアを他の細胞と識別できるF4/80、Fc受容体、補体受容体[2]、Iba1[3] [4]に対する抗体や細胞培養法の開発により、ミクログリア研究が大きく発展した。さらに、1960年代後半にKreutzbergが、血液脳関門が正常なままである顔面神経切断モデルを開発し、神経損傷によるミクログリアの応答性の研究に飛躍的な発展をもたらした[1]。
その後、ミクログリアに蛍光タンパク質を発現するトランスジェニックマウスが開発され、免疫組織染色をすることなくミクログリアの可視化が実現し、生体イメージング技術との組み合わせで、ミクログリアのin vivoイメージングが可能になった(後述)。これにより、これまで正常時のミクログリアは「静止型(resting)」とされてきたが、ミクログリアは細胞突起をダイナミックに動かし、シナプスとの物理的コンタクトや細胞障害に鋭敏に応答することが明らかになった。
さらに、遺伝子改変による細胞ラベリング技術等により、長らく議論になっていたミクログリアの起源が骨髄由来細胞ではなく胎生期に卵黄嚢で発生する前駆細胞であることが発見され、更なるブレイクスルーとなった(後述)。また、生体からミクログリアのみをFACS等で分取し、マイクロアレイや次世代シーケンス技術等による網羅的遺伝子解析からミクログリアに高発現する遺伝子群(P2ry12、P2ry13、Tmem119、Gpr34、Siglech、Trem2、Cx3cr1など)も明らかになり、骨髄由来の単球やマクロファージとは異なる遺伝子発現パターンを有していることが報告された[5] [6] [7]。
最近では脳部位によってもミクログリアの遺伝子発現が異なることも報告されている[8]。加えて、ミクログリア選択的な遺伝子改変技術も開発され、ミクログリアの生理および病態における多くの興味深い役割が次々と明らかになってきており[1] [9] [10] [11] [12] [13] [14]、中枢神経系機能の維持や異常にこの「第3のエレメント」が世界的な注目を集めている。
発生
ミクログリアの発生や起源については古くから議論されてきた。以前は神経外胚葉由来という説もあったが、現在では中胚葉由来とされている。マクロファージマーカーでミクログリアを標識した組織学的研究から、脳でのミクログリアは胎生期の骨髄造血前に観察される[15]。ミクログリアの起源となる前駆細胞とその発生組織は最近まで未解明であったが、2010年にGinhouxらがfate-mapping解析によって、胎生7.5日の卵黄嚢に存在する前駆細胞が循環器系(胎生8.5~10日に形成)を介して脳へ移動しミクログリアに分化することを発見し[16]、それがミクログリアの起源であろうとされている。脳へ移動した卵黄嚢由来前駆細胞は増殖能を有する。この説は、骨髄系細胞の発生に必要な転写因子Mybの欠損マウスでミクログリアが正常に発生することからも裏付けられた[17]。卵黄嚢前駆細胞(おそらく赤血球系骨髄前駆細胞)からミクログリアへの分化には転写因子RUNX1、PU.1、IRF8が重要な役割を担っている[16] [18]。
成体におけるミクログリアについては、蛍光タンパク質GFPを発現したマウスの骨髄細胞を移植した骨髄キメラマウスの脳でGFP陽性のミクログリアが観察されたことから、骨髄由来の単球やマクロファージがミクログリアの供給源である可能性が考えられた。しかし、骨髄キメラマウスを作製するために用いる致死量放射線照射や骨髄細胞投与という実験的操作の影響が懸念されていた[19]。実際に、脳の遮蔽保護や放射線照射を利用しないキメラマウスでの実験では、GFP陽性のミクログリアがほとんど認められなかった[20] [21]。
コロニー刺激因子1受容体(CSF1R)はミクログリアに発現しており、その欠損マウスではミクログリアの細胞数が消失する[22]。また、CSF1R阻害薬を成体マウスに慢性的に処置することでもミクログリア数が著減する[23]。一方で、CSF1Rの内因性リガンドであるCSF1の欠損ではそのような劇的な減少は認められない。最近、CSF1Rの他の内因性リガンドとして同定されたインターロイキン-34(IL-34)は、生後マウス脳のニューロンに発現し、その欠損により卵黄嚢での前駆細胞は正常であるが、生後のミクログリア細胞数が減少することから、成体でのミクログリアの維持に重要であると考えられている[24] [25]。さらに興味深いことは、IL-34欠損マウスでのミクログリア数の減少は脳部位によって差異があり、各脳部位によってミクログリアの維持メカニズムが異なる可能性も示唆されている。また、TGF-βも成体でのミクログリアの機能維持に関与している[6]。
形態・分布
通常は小さな細胞体に複数の細かく枝分かれした突起をもつ細胞形態で存在しているが、細胞外からの刺激が加わると突起の短縮や細胞体の肥大化などの顕著な形態学的変化を伴い、活性化状態へと移行する。正常状態のミクログリアはラミファイド型、活性化しアメーバ様の形態をしたミクログリアはアメボイド型と呼ばれる。神経細胞の損傷や脳組織内への感染性細菌の侵入に応答して活性化型へと移行したミクログリアは貪食作用を示して病原菌や細胞の残骸を取り除く。活性化しアメーバ運動をしているミクログリアの姿は末梢の免疫細胞であるマクロファージと非常に良く似た形態をとる。
ミクログリアは細胞個々のテリトリーがあり、同一の脳部位においてはほぼ均一に分布している。しかし、その分布密度は脳部位によって異なっており、例えば成体マウスでは、皮質や脳梁におけるミクログリアの占める割合は細胞の5%ほどであるが、黒質におけるミクログリアの占める割合は細胞の12%にのぼる。
また、部位によって細胞体や突起構造の形にも違いがあり、灰白質と白質のミクログリアを比べると、灰白質のミクログリアは放射状に突起構造を伸ばしているが、白質のミクログリアは長細く突起を伸ばし、細胞体も細長い形態をとることが報告されている[26]。また、興味深いことに、ミクログリア突起長は昼間よりも夜間で長く、さらに突起の分岐数も夜間のほうが多く、より複雑な構造をとっている。この日内変化はミクログリア分子時計で制御されており、プリン受容体の一つであるP2Y12受容体の発現が日内変動しているためと考えられている。さらに、シナプスの密度と活動性も日内変化の原因の一つとしてミクログリアの突起構造日内変化が関与していることも示されている[27]。
突起伸長・細胞遊走

(黄色:レーザー照射による損傷部位, 緑色:ミクログリア)
CX3CR1-EGFPマウスと2光子顕微鏡を用いて, レーザー照射による障害を与えた際の成体マウス大脳皮質のin vivoイメージングを行った。脳が損傷を受けるとミクログリアは直ちにそれを感知し, 損傷部位へと突起を伸ばす。(著者原図)
2005年にNimmerjahnらとGanらの研究グループは2光子顕微鏡を用いたin vivoイメージングにより、生きたままのマウスの脳内ミクログリアを非侵襲的に観察することに成功し、従来静止状態とされてきたラミファイドミクログリアが常に突起を動かし伸縮を繰り返して活発に活動していることを発見した[28] [29]。固定組織標本からは認識されなかったこの発見はミクログリア研究のブレイクスルーとなり、ミクログリアの挙動とその生理的な役割に注目を集めることとなった。
ミクログリアの突起伸長や細胞遊走は化学誘引物質の濃度勾配に従う走化性によって起こる。ミクログリアの代表的走化性誘導因子としてはATPおよびADPが知られており、初代培養ミクログリア細胞を用いた研究からP2Y12受容体を介したシグナルが重要な役割を担っていることが明らかにされている[30] [31]。加えて、P2X4受容体やアデノシン受容体A1やA3受容体も細胞遊走に関与する[32] [33]。その一方で、ミクログリアの突起の退縮にはA2A受容体が関与することも報告されている[34]。ATPやADP以外にもAβやブラジキニン、グルタミン酸、補体C5a、CCL21、NGF、EGFといった多岐にわたる因子がミクログリア走化性誘導因子として報告されている[1] [35] [36] [37]。
機能
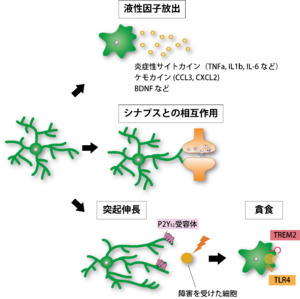
(A) 神経障害時やストレス、細菌感染などによって活性化したミクログリアからは炎症性サイトカインやケモカインが放出され、炎症応答や神経変性を引き起こす。
(B) ミクログリアは突起をシナプス構造と接触させ、相互作用を行っている。
(C) 感染や局所的な損傷時に、ミクログリアは異常部位へと突起を伸ばし、活性化を伴って異常部位へ遊走する. その後、異物や死細胞を貪食し、除去する。
液性因子産生放出
ミクログリアは中枢神経系の機能に様々な影響を及ぼすが、この生理機能への調節機構の手段の一つとして液性因子の産生や放出が挙げられる。神経障害時やストレス、細胞内感染などによって活性化したミクログリアからは腫瘍壊死因子(TNF-α)、IL-1β、IL-6などの炎症性サイトカインが放出され、神経変性や中枢神経系の炎症応答を引き起こす[38]。それ故、ミクログリア由来の炎症性サイトカインにより中枢神経系の機能に何らかの支障が生じることで、多発性硬化症やアルツハイマー病などの中枢神経系疾患の悪化につながることが示唆されている。
ミクログリアから放出されるケモカインもまた炎症応答や神経変性を引き起こすなど、生理学的および病的状態に大きく寄与する[39]。培養ミクログリア細胞からはCCL3(MIP-1α)やCXCL2(MIP-2)がプリン受容体であるP2X7受容体の刺激を介して産生および放出される[40] [41]。そして神経が障害される状況においては脊髄ミクログリアでCCL3の発現が増加し、持続した疼痛が起こる[42]。炎症性サイトカインやケモカイン以外にも、一酸化窒素(NO)、活性酸素(ROS)、グルタミン酸、ATPなどがミクログリアから放出され、神経細胞死を誘導することが示唆されている[43] [44] [45]。
一方、ミクログリアから産生放出される液性因子は、神経系の調節にも密接に関わる。例えば、神経系の異常時において活性化したミクログリアから放出される脳由来神経栄養因子(BDNF)は神経の興奮を引き起こす[46]。一方、ミクログリア特異的にBDNFを欠損させることで脳のシナプス可塑性に異常が認められることから、ミクログリア由来のBDNFは正常時には記憶や学習に重要な役割を担うことが分かる[47]。発達期や出生後早期においては、ミクログリアから分泌されるインスリン様成長因子1(IGF-1)がニューロンの生存維持に必要なこと[48]、また面白いことに神経障害作用を有する炎症性サイトカインIL-1βおよびIFN-γがニューロンの発生をむしろ促進することも報告されている[49]。他にも、中枢神経系ではミクログリアのみに発現するリソソーム性プロテアーゼのカテプシンSは、神経細胞に発現する膜結合型ケモカインのフラクタルカイン(CX3CL1)を切断し[50]、それがミクログリアのCX3CR1に作用することで様々な生理応答を示す。さらに、ミクログリアから放出されるカテプシンSは大脳皮質体性感覚野においてスパインの密度や活動の日内リズム形成に関与することが報告されている[27]。
シナプスとの相互作用
正常時のミクログリアは細かく枝分かれした突起を脳実質内に張り巡らせて脳内環境の異常を待ち構えていると考えられていたが、2光子励起顕微鏡を用いたマウス大脳皮質のイメージング法の利用により、その突起の動態は非常にダイナミックなもので常に一定の領域の中で突起の退縮を繰り返していることが証明された[28] [29]。この時の突起の動きは1 μm毎分で、数時間で脳全体の容積を検索できるような速度と推測される。
さらに、脳内に傷害が起きた場合はP2Y12受容体を介して、さらに動的に突起を動かして障害部位に集積する[51]。ミクログリアの突起がシナプス構造に接触するという直接の証拠は電子顕微鏡像で得られており[52]、体性感覚野又は視覚野皮質Ⅱ/Ⅲ層のシナプスにおいては、二光子励起観察像からミクログリアの突起がシナプスに短期的な接触を繰り返していることが生きたままのマウスで確認されている[53]。ミクログリアのシナプスへの接触は眼からの入力を取り除いた場合、テトロドトキシン処置や低温条件によって神経活動が抑制された条件で減少することから、神経活動依存的なものであると考えられている。
ミクログリアのシナプスへの接触はシナプス剪定(synaptic pruning)といった発達段階において不必要なシナプスを取り除く機能に深く関係していると考えられており[54]、シナプスリモデリングが活発な脳部位(皮質、海馬、視覚処理回路)におけるミクログリアの存在が注目されていた[2] [55] [56]。現在では、眼-視床経路におけるシナプスの左右眼選択的な神経回路構築時に補体シグナルを介したミクログリアによるシナプスの除去が重要なプロセスを担っていることが証明されている[57]。また、障害を受けた神経細胞のシナプス間にミクログリアが入り込むことでシナプス接続を断つ(synaptic stripping)現象も古くから報告されている[58]。
貪食
ミクログリアはその挙動からマクロファージに類似した細胞と認識されている。1900年代にRobertsonによって、神経細胞由来の崩壊物がミクログリアの細胞内に多数存在していることが見出されており、ミクログリアの貪食についての初めての観察とされている。ミクログリアは活性化型の形態の一つとして、通常は細く枝分かれした突起の退縮を引き起こし、アメボイド形態に変化する。このようなミクログリアは強い貪食作用を示し、死細胞やデブリ(障害を受けた細胞の破片など)を取り除く作用を持っている。ミクログリアが障害を受けた死細胞を取り除くことは、有害な細胞内因子の漏出を防いで脳内環境を保つ意味で重要なプロセスである。
現在では、神経細胞の自己死の一つの形態に、ミクログリアが生きた神経細胞を貪食して組織中から取り除くといった現象も報告されている(ファゴプトーシス phagoptosis)[59] [60]。これらミクログリアの貪食活性は死細胞に対してだけではなく、病原体や細胞からの分泌物や老廃物の除去という役割も持っており、ミクログリアの最も重要な機能の一つである。
また、不要物の除去はその後の脳組織の回復にも寄与すると考えられ、障害によって変性した軸索の再生の促進にも関与するとされる。
ミクログリアの貪食に関わる受容体としてはToll様受容体(TLR)など外因性病原体を認識する受容体と、TREM2などのアポトーシスを認識する受容体が主なものであるが、Fc受容体や補体受容体、スカベンジャー受容体、MAC-2、マンノース受容体、LRP受容体、P2Y6受容体などもミクログリアの貪食機能との関わりが示唆されている[61] [62] [63]。
中枢神経疾患における役割

Iba1抗体で標識したミクログリア(著者原図)
疼痛
神経系のダメージや機能不全により神経障害性疼痛と総称される慢性的な痛みが発症する。その発症と維持メカニズムはわかっていないが、近年脊髄におけるミクログリアの役割が注目されている。同疼痛のモデル動物である人為的な末梢神経損傷モデルや神経障害を伴う病態モデル(糖尿病、がん、脊髄損傷、帯状疱疹など)において、脊髄のミクログリアは肥大化し、突起の退縮が起こる。さらに、細胞マーカーCD11bやIba1の発現が増加し、損傷ニューロンで発現するCSF1によって細胞増殖が誘発され、細胞数が2~3倍に増加する[64] [65]。
神経障害性疼痛における脊髄ミクログリアの重要性は、プリン受容体のP2X4受容体の役割から見出された[66]。神経障害性疼痛動物モデルの脊髄後角では、転写因子IRF8とIRF5によってP2X4受容体がミクログリアで特異的に高発現し、その受容体を遮断すること、あるいは遺伝子をノックダウンや欠損させることで、アロディニアが著明に抑制された[66] [67] [68] [69]。
ミクログリアのP2X4受容体がATPで刺激されることでBDNFなどの液性因子が産生放出され[70]、それらが脊髄後角ニューロンの機能を変調し、神経障害性疼痛を発症することが報告されている[46]。したがって、ミクログリアとニューロン間の病的連関が神経障害性疼痛の原因であろうと考えられている[71]。ミクログリアにはP2X4受容体以外にも他の機能分子が発現し、神経障害性疼痛に関与している[72] [73] [74] [75]。
複合性局所疼痛症候群(CRPS)の患者の脊髄において、CD68陽性ミクログリアの活性化が報告されている[76]。
アルツハイマー病
アルツハイマー病(AD)モデルマウスやアルツハイマー病患者の脳では老人斑周囲にミクログリアや単球、マクロファージの集積が認められる。ミクログリアには可溶性Aβオリゴマーや線維性Aβが結合するscavenger receptor A、CD36、RAGEなど多くの受容体が発現しているが、これらの発現レベルやAβの分解酵素、オートファジーがアルツハイマー病モデルマウスで低下し[77] [78] [79]、それがAβ蓄積の原因の一つであろうと想定されている[80]。Aβクリアランスにはミクログリアの細胞貪食が重要であるとされている一方で、ミクログリアの除去マウスでAβに劇的な変化が認められないという報告もあり[81]、その関与は単純ではない。また、三次元電顕解析[82]やミクログリアのin vivoイメージング解析[83] [84]などでも、ミクログリアのAβプラークの貪食やクリアランスを支持する結果が得られていない。貪食関連分子の発現低下やミクログリアの老化などが同細胞の機能低下に関連している可能性も指摘されている[85]。
末梢からの単球の集積にはケモカイン受容体CCR2の関与が報告され、この病態初期の単球の集積を阻害することでアルツハイマー病モデルマウスの死亡率やAβの蓄積の増加がみられることから、脳へ浸潤した単球がアルツハイマー病病態に保護的に働いている可能性が示唆されている[86]。また、Aβの分解は末梢性マクロファージの方がより効率的であるという報告もある[85]。しかし、末梢単球やマクロファージの脳内浸潤については放射線照射による骨髄キメラマウス等が使われているため今後の検討が必要である。Aβの脳血管周囲への沈着はアルツハイマー病患者で高頻度に観察されるが、アルツハイマー病モデルマウスの血管周囲マクロファージの除去により脳血管周囲のAβ沈着が悪化することも報告されている[87]。
ミクログリアやその他のミエロイド系細胞によるAβクリアランスが不十分な場合、AβはミクログリアのCD36やTLR4などに作用することで、NLRP3インフラマソームなどを活性化して炎症因子の産生や放出を引き起こす[88] [89] [90]。炎症性サイトカインはミクログリアのAβクリアランス能などの細胞機能を低下させ、その影響が周辺の他の細胞に波及しタウオパチー、さらには神経細胞死を起こすという説が想定されている[90]。
さらに、アルツハイマー病リスクファクターとしてミクログリアによるAβプラークの貪食に関連するTREM2変異[91] [92] [93]、TREM2と相互作用するTYROBPの変異[94]が報告された。さらにミクログリアのAβ貪食を抑制するCD33の機能獲得変異も孤発性アルツハイマー病リスクファクターとして報告され[95]、アルツハイマー病におけるミクログリアの機能変化の関与が支持されている。しかし、アルツハイマー病モデルマウスでのTREM2の役割については一致した結果は得られていない[96] [97] [98]。その原因の一つとして解析するアルツハイマー病モデルマウスが考えられる。最近、西道らによって、新しいアルツハイマー病モデルマウスが開発され、同マウスでもミクログリアの活性化が認められている[99]。
筋萎縮性側索硬化症
筋萎縮性側索硬化症(ALS)においてミクログリアは、その症状の進行に寄与することが示唆されている。SOD1遺伝子の変異を原因とする家族性ALS患者では、脱落した下位または上位の運動神経細胞周囲にミクログリアやマクロファージの集積が見られている[100]。同様の事象はヒト変異SOD1を強制発現することでALS病態をしめすモデル動物の腰髄においても観察されており、麻痺症状の現れる前においてすでに貪食形態のミクログリアが運動神経の細胞体に接触している像が得られている[101]。
また、SOD1の変異の有無にかかわらずフォールディング異常を示すSOD1の凝集物がミクログリアに集積していることも示されている[102]。ALSモデルマウスのミクログリアで、SOD1を正常型に置き換えると病態発症の程度が軽くなり、ミクログリアで変異型SOD1を発現する動物を作成しても症状は現れないことから[103] [104]、病態の進行時にはミクログリアのSOD1が寄与していると予想される。
また、培養系を用いた研究では、外因的に与えられたSOD1によって活性化されたミクログリアの培養上清が培養運動神経の生存率を低下させることも明らかになっており、ミクログリアの活性化が病態の進行に寄与することを示唆している[105]。変異型SOD1によるALSモデルマウスでは、病巣部でのミクログリアの集積に末梢骨髄由来細胞の浸潤は寄与していないことがパラバイオーシスを用いた実験によって証明されている[20]。
また、CX3CR1欠損マウスでは変異型SOD1発現による生存率の低下が増悪しており、CX3CR1の病態への関与、さらにミクログリアの関与を示唆している[106]。しかしながら、運動神経の細胞死にミクログリアがどのように関与しているかに対しては未だ明確な答えが得られていない。
多発性硬化症
多発性硬化症(multiple sclerosis: MS)の実験モデルとして用いられている実験的自己免疫性脳脊髄炎(experimental autoimmune encephalomyelisis: EAE)は、中枢における自己免疫疾患研究に用いられている。末梢T細胞とミクログリアの相互作用がこの病態に大きく寄与することがわかっており、活性化ミクログリアと浸潤マクロファージによって、脱髄や神経傷害などの組織傷害が惹起される。CD11b-HSVTKマウスはガンシクロビルの投与によってミクログリアを欠失できる実験動物として用いられているが、この実験系を用いてミクログリアが欠失している動物では、EAEの発症の遅れや病態の程度の軽減が観察されている[107]。マクロファージ同様、ミクログリアにもM1、M2といった異なる活性化様式の存在が考えられており、脱髄・寛解の過程においては、M1様ミクログリアがTh1、Th17ヘルパーT細胞からの病態悪化シグナルによって炎症応答・脱髄を引き起こし、M2様ミクログリアは、組織修復・病態の寛解を誘導していると考えられている[108] [109]。ミクログリアの活性化に関わる因子としてマイクロRNAのmiR-124、JAK/STATシグナルの抑制経路に関わるSOCS3についての報告があり、miR-124は病態初期にその発現が抑制されることでC/EBPαやPU.1の発現を介したミクログリアの活性化に関与しており、miR-124の投与によってM2様ミクログリアの出現と病態の改善が見られることが証明されている[110]。SOCS3については骨髄性細胞でのSOCS3欠損の結果、STAT3/STAT4シグナルの過剰な活性化を介してM1様ミクログリアの出現へ繋がるとされている[111]。
参考文献
- ↑ 1.0 1.1 1.2 1.3
Kettenmann, H., Hanisch, U.K., Noda, M., & Verkhratsky, A. (2011).
Physiology of microglia. Physiological reviews, 91(2), 461-553. [PubMed:21527731] [WorldCat] [DOI] - ↑ 2.0 2.1
Perry, V.H., Hume, D.A., & Gordon, S. (1985).
Immunohistochemical localization of macrophages and microglia in the adult and developing mouse brain. Neuroscience, 15(2), 313-26. [PubMed:3895031] [WorldCat] [DOI] - ↑
Imai, Y., Ibata, I., Ito, D., Ohsawa, K., & Kohsaka, S. (1996).
A novel gene iba1 in the major histocompatibility complex class III region encoding an EF hand protein expressed in a monocytic lineage. Biochemical and biophysical research communications, 224(3), 855-62. [PubMed:8713135] [WorldCat] [DOI] - ↑
Ito, D., Imai, Y., Ohsawa, K., Nakajima, K., Fukuuchi, Y., & Kohsaka, S. (1998).
Microglia-specific localisation of a novel calcium binding protein, Iba1. Brain research. Molecular brain research, 57(1), 1-9. [PubMed:9630473] [WorldCat] [DOI] - ↑
Gautier, E.L., Shay, T., Miller, J., Greter, M., Jakubzick, C., Ivanov, S., ..., & Immunological Genome Consortium (2012).
Gene-expression profiles and transcriptional regulatory pathways that underlie the identity and diversity of mouse tissue macrophages. Nature immunology, 13(11), 1118-28. [PubMed:23023392] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Butovsky, O., Jedrychowski, M.P., Moore, C.S., Cialic, R., Lanser, A.J., Gabriely, G., ..., & Weiner, H.L. (2014).
Identification of a unique TGF-β-dependent molecular and functional signature in microglia. Nature neuroscience, 17(1), 131-43. [PubMed:24316888] [PMC] [WorldCat] [DOI] - ↑
Gosselin, D., Link, V.M., Romanoski, C.E., Fonseca, G.J., Eichenfield, D.Z., Spann, N.J., ..., & Glass, C.K. (2014).
Environment drives selection and function of enhancers controlling tissue-specific macrophage identities. Cell, 159(6), 1327-40. [PubMed:25480297] [PMC] [WorldCat] [DOI] - ↑
Grabert, K., Michoel, T., Karavolos, M.H., Clohisey, S., Baillie, J.K., Stevens, M.P., ..., & McColl, B.W. (2016).
Microglial brain region-dependent diversity and selective regional sensitivities to aging. Nature neuroscience, 19(3), 504-16. [PubMed:26780511] [PMC] [WorldCat] [DOI] - ↑
Ransohoff, R.M., & Cardona, A.E. (2010).
The myeloid cells of the central nervous system parenchyma. Nature, 468(7321), 253-62. [PubMed:21068834] [WorldCat] [DOI] - ↑
Hughes, V. (2012).
Microglia: The constant gardeners. Nature, 485(7400), 570-2. [PubMed:22660301] [WorldCat] [DOI] - ↑
Aguzzi, A., Barres, B.A., & Bennett, M.L. (2013).
Microglia: scapegoat, saboteur, or something else? Science (New York, N.Y.), 339(6116), 156-61. [PubMed:23307732] [PMC] [WorldCat] [DOI] - ↑
Prinz, M., & Priller, J. (2014).
Microglia and brain macrophages in the molecular age: from origin to neuropsychiatric disease. Nature reviews. Neuroscience, 15(5), 300-12. [PubMed:24713688] [WorldCat] [DOI] - ↑
Biber, K., Möller, T., Boddeke, E., & Prinz, M. (2016).
Central nervous system myeloid cells as drug targets: current status and translational challenges. Nature reviews. Drug discovery, 15(2), 110-24. [PubMed:26634996] [WorldCat] [DOI] - ↑
Salter, M.W., & Beggs, S. (2014).
Sublime microglia: expanding roles for the guardians of the CNS. Cell, 158(1), 15-24. [PubMed:24995975] [WorldCat] [DOI] - ↑
Ginhoux, F., Lim, S., Hoeffel, G., Low, D., & Huber, T. (2013).
Origin and differentiation of microglia. Frontiers in cellular neuroscience, 7, 45. [PubMed:23616747] [PMC] [WorldCat] [DOI] - ↑ 16.0 16.1
Ginhoux, F., Greter, M., Leboeuf, M., Nandi, S., See, P., Gokhan, S., ..., & Merad, M. (2010).
Fate mapping analysis reveals that adult microglia derive from primitive macrophages. Science (New York, N.Y.), 330(6005), 841-5. [PubMed:20966214] [PMC] [WorldCat] [DOI] - ↑
Schulz, C., Gomez Perdiguero, E., Chorro, L., Szabo-Rogers, H., Cagnard, N., Kierdorf, K., ..., & Geissmann, F. (2012).
A lineage of myeloid cells independent of Myb and hematopoietic stem cells. Science (New York, N.Y.), 336(6077), 86-90. [PubMed:22442384] [WorldCat] [DOI] - ↑
Kierdorf, K., Erny, D., Goldmann, T., Sander, V., Schulz, C., Perdiguero, E.G., ..., & Prinz, M. (2013).
Microglia emerge from erythromyeloid precursors via Pu.1- and Irf8-dependent pathways. Nature neuroscience, 16(3), 273-80. [PubMed:23334579] [WorldCat] [DOI] - ↑
Larochelle, A., Bellavance, M.A., Michaud, J.P., & Rivest, S. (2016).
Bone marrow-derived macrophages and the CNS: An update on the use of experimental chimeric mouse models and bone marrow transplantation in neurological disorders. Biochimica et biophysica acta, 1862(3), 310-22. [PubMed:26432480] [WorldCat] [DOI] - ↑ 20.0 20.1
Ajami, B., Bennett, J.L., Krieger, C., Tetzlaff, W., & Rossi, F.M. (2007).
Local self-renewal can sustain CNS microglia maintenance and function throughout adult life. Nature neuroscience, 10(12), 1538-43. [PubMed:18026097] [WorldCat] [DOI] - ↑
Kierdorf, K., Katzmarski, N., Haas, C.A., & Prinz, M. (2013).
Bone marrow cell recruitment to the brain in the absence of irradiation or parabiosis bias. PloS one, 8(3), e58544. [PubMed:23526995] [PMC] [WorldCat] [DOI] - ↑
Erblich, B., Zhu, L., Etgen, A.M., Dobrenis, K., & Pollard, J.W. (2011).
Absence of colony stimulation factor-1 receptor results in loss of microglia, disrupted brain development and olfactory deficits. PloS one, 6(10), e26317. [PubMed:22046273] [PMC] [WorldCat] [DOI] - ↑
Elmore, M.R., Najafi, A.R., Koike, M.A., Dagher, N.N., Spangenberg, E.E., Rice, R.A., ..., & Green, K.N. (2014).
Colony-stimulating factor 1 receptor signaling is necessary for microglia viability, unmasking a microglia progenitor cell in the adult brain. Neuron, 82(2), 380-97. [PubMed:24742461] [PMC] [WorldCat] [DOI] - ↑
Greter, M., Lelios, I., Pelczar, P., Hoeffel, G., Price, J., Leboeuf, M., ..., & Becher, B. (2012).
Stroma-derived interleukin-34 controls the development and maintenance of langerhans cells and the maintenance of microglia. Immunity, 37(6), 1050-1060. [PubMed:23177320] [PMC] [WorldCat] [DOI] - ↑
Wang, Y., Szretter, K.J., Vermi, W., Gilfillan, S., Rossini, C., Cella, M., ..., & Colonna, M. (2012).
IL-34 is a tissue-restricted ligand of CSF1R required for the development of Langerhans cells and microglia. Nature immunology, 13(8), 753-60. [PubMed:22729249] [PMC] [WorldCat] [DOI] - ↑
Lawson, L.J., Perry, V.H., Dri, P., & Gordon, S. (1990).
Heterogeneity in the distribution and morphology of microglia in the normal adult mouse brain. Neuroscience, 39(1), 151-70. [PubMed:2089275] [WorldCat] [DOI] - ↑ 27.0 27.1
Hayashi, Y., Koyanagi, S., Kusunose, N., Okada, R., Wu, Z., Tozaki-Saitoh, H., ..., & Nakanishi, H. (2013).
The intrinsic microglial molecular clock controls synaptic strength via the circadian expression of cathepsin S. Scientific reports, 3, 2744. [PubMed:24067868] [PMC] [WorldCat] [DOI] - ↑ 28.0 28.1
Nimmerjahn, A., Kirchhoff, F., & Helmchen, F. (2005).
Resting microglial cells are highly dynamic surveillants of brain parenchyma in vivo. Science (New York, N.Y.), 308(5726), 1314-8. [PubMed:15831717] [WorldCat] [DOI] - ↑ 29.0 29.1
Davalos, D., Grutzendler, J., Yang, G., Kim, J.V., Zuo, Y., Jung, S., ..., & Gan, W.B. (2005).
ATP mediates rapid microglial response to local brain injury in vivo. Nature neuroscience, 8(6), 752-8. [PubMed:15895084] [WorldCat] [DOI] - ↑
Honda, S., Sasaki, Y., Ohsawa, K., Imai, Y., Nakamura, Y., Inoue, K., & Kohsaka, S. (2001).
Extracellular ATP or ADP induce chemotaxis of cultured microglia through Gi/o-coupled P2Y receptors. The Journal of neuroscience : the official journal of the Society for Neuroscience, 21(6), 1975-82. [PubMed:11245682] [PMC] [WorldCat] - ↑
Sasaki, Y., Hoshi, M., Akazawa, C., Nakamura, Y., Tsuzuki, H., Inoue, K., & Kohsaka, S. (2003).
Selective expression of Gi/o-coupled ATP receptor P2Y12 in microglia in rat brain. Glia, 44(3), 242-50. [PubMed:14603465] [WorldCat] [DOI] - ↑
Ohsawa, K., Irino, Y., Nakamura, Y., Akazawa, C., Inoue, K., & Kohsaka, S. (2007).
Involvement of P2X4 and P2Y12 receptors in ATP-induced microglial chemotaxis. Glia, 55(6), 604-16. [PubMed:17299767] [WorldCat] [DOI] - ↑
Ohsawa, K., Sanagi, T., Nakamura, Y., Suzuki, E., Inoue, K., & Kohsaka, S. (2012).
Adenosine A3 receptor is involved in ADP-induced microglial process extension and migration. Journal of neurochemistry, 121(2), 217-27. [PubMed:22335470] [WorldCat] [DOI] - ↑
Orr, A.G., Orr, A.L., Li, X.J., Gross, R.E., & Traynelis, S.F. (2009).
Adenosine A(2A) receptor mediates microglial process retraction. Nature neuroscience, 12(7), 872-8. [PubMed:19525944] [PMC] [WorldCat] [DOI] - ↑
Tiffany, H.L., Lavigne, M.C., Cui, Y.H., Wang, J.M., Leto, T.L., Gao, J.L., & Murphy, P.M. (2001).
Amyloid-beta induces chemotaxis and oxidant stress by acting at formylpeptide receptor 2, a G protein-coupled receptor expressed in phagocytes and brain. The Journal of biological chemistry, 276(26), 23645-52. [PubMed:11316806] [WorldCat] [DOI] - ↑
Gilad, G.M., & Gilad, V.H. (1995).
Chemotaxis and accumulation of nerve growth factor by microglia and macrophages. Journal of neuroscience research, 41(5), 594-602. [PubMed:7563239] [WorldCat] [DOI] - ↑
Nolte, C., Kirchhoff, F., & Kettenmann, H. (1997).
Epidermal growth factor is a motility factor for microglial cells in vitro: evidence for EGF receptor expression. The European journal of neuroscience, 9(8), 1690-8. [PubMed:9283823] [WorldCat] [DOI] - ↑
Inoue, K. (2006).
The function of microglia through purinergic receptors: neuropathic pain and cytokine release. Pharmacology & therapeutics, 109(1-2), 210-26. [PubMed:16169595] [WorldCat] [DOI] - ↑
Ambrosini, E., & Aloisi, F. (2004).
Chemokines and glial cells: a complex network in the central nervous system. Neurochemical research, 29(5), 1017-38. [PubMed:15139300] [WorldCat] [DOI] - ↑
Kataoka, A., Tozaki-Saitoh, H., Koga, Y., Tsuda, M., & Inoue, K. (2009).
Activation of P2X7 receptors induces CCL3 production in microglial cells through transcription factor NFAT. Journal of neurochemistry, 108(1), 115-25. [PubMed:19014371] [WorldCat] [DOI] - ↑
Shiratori, M., Tozaki-Saitoh, H., Yoshitake, M., Tsuda, M., & Inoue, K. (2010).
P2X7 receptor activation induces CXCL2 production in microglia through NFAT and PKC/MAPK pathways. Journal of neurochemistry, 114(3), 810-9. [PubMed:20477948] [WorldCat] [DOI] - ↑
Matsushita, K., Tozaki-Saitoh, H., Kojima, C., Masuda, T., Tsuda, M., Inoue, K., & Hoka, S. (2014).
Chemokine (C-C motif) receptor 5 is an important pathological regulator in the development and maintenance of neuropathic pain. Anesthesiology, 120(6), 1491-503. [PubMed:24589480] [WorldCat] [DOI] - ↑
Spanos, J.P., Hsu, N.J., & Jacobs, M. (2015).
Microglia are crucial regulators of neuro-immunity during central nervous system tuberculosis. Frontiers in cellular neuroscience, 9, 182. [PubMed:26041993] [PMC] [WorldCat] [DOI] - ↑
Kim, S.Y., Moon, J.H., Lee, H.G., Kim, S.U., & Lee, Y.B. (2007).
ATP released from beta-amyloid-stimulated microglia induces reactive oxygen species production in an autocrine fashion. Experimental & molecular medicine, 39(6), 820-7. [PubMed:18160853] [WorldCat] [DOI] - ↑
Takaki, J., Fujimori, K., Miura, M., Suzuki, T., Sekino, Y., & Sato, K. (2012).
L-glutamate released from activated microglia downregulates astrocytic L-glutamate transporter expression in neuroinflammation: the 'collusion' hypothesis for increased extracellular L-glutamate concentration in neuroinflammation. Journal of neuroinflammation, 9, 275. [PubMed:23259598] [PMC] [WorldCat] [DOI] - ↑ 46.0 46.1
Coull, J.A., Beggs, S., Boudreau, D., Boivin, D., Tsuda, M., Inoue, K., ..., & De Koninck, Y. (2005).
BDNF from microglia causes the shift in neuronal anion gradient underlying neuropathic pain. Nature, 438(7070), 1017-21. [PubMed:16355225] [WorldCat] [DOI] - ↑
Parkhurst, C.N., Yang, G., Ninan, I., Savas, J.N., Yates, J.R., Lafaille, J.J., ..., & Gan, W.B. (2013).
Microglia promote learning-dependent synapse formation through brain-derived neurotrophic factor. Cell, 155(7), 1596-609. [PubMed:24360280] [PMC] [WorldCat] [DOI] - ↑
Ueno, M., Fujita, Y., Tanaka, T., Nakamura, Y., Kikuta, J., Ishii, M., & Yamashita, T. (2013).
Layer V cortical neurons require microglial support for survival during postnatal development. Nature neuroscience, 16(5), 543-51. [PubMed:23525041] [WorldCat] [DOI] - ↑
Shigemoto-Mogami, Y., Hoshikawa, K., Goldman, J.E., Sekino, Y., & Sato, K. (2014).
Microglia enhance neurogenesis and oligodendrogenesis in the early postnatal subventricular zone. The Journal of neuroscience : the official journal of the Society for Neuroscience, 34(6), 2231-43. [PubMed:24501362] [PMC] [WorldCat] [DOI] - ↑
Clark, A.K., Yip, P.K., & Malcangio, M. (2009).
The liberation of fractalkine in the dorsal horn requires microglial cathepsin S. The Journal of neuroscience : the official journal of the Society for Neuroscience, 29(21), 6945-54. [PubMed:19474321] [PMC] [WorldCat] [DOI] - ↑
Haynes, S.E., Hollopeter, G., Yang, G., Kurpius, D., Dailey, M.E., Gan, W.B., & Julius, D. (2006).
The P2Y12 receptor regulates microglial activation by extracellular nucleotides. Nature neuroscience, 9(12), 1512-9. [PubMed:17115040] [WorldCat] [DOI] - ↑
Tremblay, M.È., Lowery, R.L., & Majewska, A.K. (2010).
Microglial interactions with synapses are modulated by visual experience. PLoS biology, 8(11), e1000527. [PubMed:21072242] [PMC] [WorldCat] [DOI] - ↑
Wake, H., Moorhouse, A.J., Jinno, S., Kohsaka, S., & Nabekura, J. (2009).
Resting microglia directly monitor the functional state of synapses in vivo and determine the fate of ischemic terminals. The Journal of neuroscience : the official journal of the Society for Neuroscience, 29(13), 3974-80. [PubMed:19339593] [PMC] [WorldCat] [DOI] - ↑
Paolicelli, R.C., Bolasco, G., Pagani, F., Maggi, L., Scianni, M., Panzanelli, P., ..., & Gross, C.T. (2011).
Synaptic pruning by microglia is necessary for normal brain development. Science (New York, N.Y.), 333(6048), 1456-8. [PubMed:21778362] [WorldCat] [DOI] - ↑
Milligan, C.E., Cunningham, T.J., & Levitt, P. (1991).
Differential immunochemical markers reveal the normal distribution of brain macrophages and microglia in the developing rat brain. The Journal of comparative neurology, 314(1), 125-35. [PubMed:1797868] [WorldCat] [DOI] - ↑
Dalmau, I., Finsen, B., Zimmer, J., González, B., & Castellano, B. (1998).
Development of microglia in the postnatal rat hippocampus. Hippocampus, 8(5), 458-74. [PubMed:9825958] [WorldCat] [DOI] - ↑
Stevens, B., Allen, N.J., Vazquez, L.E., Howell, G.R., Christopherson, K.S., Nouri, N., ..., & Barres, B.A. (2007).
The classical complement cascade mediates CNS synapse elimination. Cell, 131(6), 1164-78. [PubMed:18083105] [WorldCat] [DOI] - ↑
Blinzinger, K., & Kreutzberg, G. (1968).
Displacement of synaptic terminals from regenerating motoneurons by microglial cells. Zeitschrift fur Zellforschung und mikroskopische Anatomie (Vienna, Austria : 1948), 85(2), 145-57. [PubMed:5706753] [WorldCat] [DOI] - ↑
Neher, J.J., Neniskyte, U., Zhao, J.W., Bal-Price, A., Tolkovsky, A.M., & Brown, G.C. (2011).
Inhibition of microglial phagocytosis is sufficient to prevent inflammatory neuronal death. Journal of immunology (Baltimore, Md. : 1950), 186(8), 4973-83. [PubMed:21402900] [WorldCat] [DOI] - ↑
Brown, G.C., & Neher, J.J. (2014).
Microglial phagocytosis of live neurons. Nature reviews. Neuroscience, 15(4), 209-16. [PubMed:24646669] [WorldCat] [DOI] - ↑
Veerhuis, R., Nielsen, H.M., & Tenner, A.J. (2011).
Complement in the brain. Molecular immunology, 48(14), 1592-603. [PubMed:21546088] [PMC] [WorldCat] [DOI] - ↑
Smith, M.E. (1999).
Phagocytosis of myelin in demyelinative disease: a review. Neurochemical research, 24(2), 261-8. [PubMed:9972873] [WorldCat] [DOI] - ↑
Inoue, K., & Tsuda, M. (2009).
Microglia and neuropathic pain. Glia, 57(14), 1469-79. [PubMed:19306358] [WorldCat] [DOI] - ↑
Tsuda, M., Inoue, K., & Salter, M.W. (2005).
Neuropathic pain and spinal microglia: a big problem from molecules in "small" glia. Trends in neurosciences, 28(2), 101-7. [PubMed:15667933] [WorldCat] [DOI] - ↑
Guan, Z., Kuhn, J.A., Wang, X., Colquitt, B., Solorzano, C., Vaman, S., ..., & Basbaum, A.I. (2016).
Injured sensory neuron-derived CSF1 induces microglial proliferation and DAP12-dependent pain. Nature neuroscience, 19(1), 94-101. [PubMed:26642091] [PMC] [WorldCat] [DOI] - ↑ 66.0 66.1
Tsuda, M., Shigemoto-Mogami, Y., Koizumi, S., Mizokoshi, A., Kohsaka, S., Salter, M.W., & Inoue, K. (2003).
P2X4 receptors induced in spinal microglia gate tactile allodynia after nerve injury. Nature, 424(6950), 778-83. [PubMed:12917686] [WorldCat] [DOI] - ↑
Tsuda, M., Kuboyama, K., Inoue, T., Nagata, K., Tozaki-Saitoh, H., & Inoue, K. (2009).
Behavioral phenotypes of mice lacking purinergic P2X4 receptors in acute and chronic pain assays. Molecular pain, 5, 28. [PubMed:19515262] [PMC] [WorldCat] [DOI] - ↑
Masuda, T., Tsuda, M., Yoshinaga, R., Tozaki-Saitoh, H., Ozato, K., Tamura, T., & Inoue, K. (2012).
IRF8 is a critical transcription factor for transforming microglia into a reactive phenotype. Cell reports, 1(4), 334-340. [PubMed:22832225] [PMC] [WorldCat] [DOI] - ↑
Masuda, T., Iwamoto, S., Yoshinaga, R., Tozaki-Saitoh, H., Nishiyama, A., Mak, T.W., ..., & Inoue, K. (2014).
Transcription factor IRF5 drives P2X4R+-reactive microglia gating neuropathic pain. Nature communications, 5, 3771. [PubMed:24818655] [PMC] [WorldCat] [DOI] - ↑
Masuda, T., Iwamoto, S., Yoshinaga, R., Tozaki-Saitoh, H., Nishiyama, A., Mak, T.W., ..., & Inoue, K. (2014).
Transcription factor IRF5 drives P2X4R+-reactive microglia gating neuropathic pain. Nature communications, 5, 3771. [PubMed:24818655] [PMC] [WorldCat] [DOI] - ↑
Beggs, S., Trang, T., & Salter, M.W. (2012).
P2X4R+ microglia drive neuropathic pain. Nature neuroscience, 15(8), 1068-73. [PubMed:22837036] [PMC] [WorldCat] [DOI] - ↑
Tsuda, M., Beggs, S., Salter, M.W., & Inoue, K. (2013).
Microglia and intractable chronic pain. Glia, 61(1), 55-61. [PubMed:22740331] [WorldCat] [DOI] - ↑
Scholz, J., & Woolf, C.J. (2007).
The neuropathic pain triad: neurons, immune cells and glia. Nature neuroscience, 10(11), 1361-8. [PubMed:17965656] [WorldCat] [DOI] - ↑
McMahon, S.B., & Malcangio, M. (2009).
Current challenges in glia-pain biology. Neuron, 64(1), 46-54. [PubMed:19840548] [WorldCat] [DOI] - ↑
Ji, R.R., Xu, Z.Z., & Gao, Y.J. (2014).
Emerging targets in neuroinflammation-driven chronic pain. Nature reviews. Drug discovery, 13(7), 533-48. [PubMed:24948120] [PMC] [WorldCat] [DOI] - ↑
Del Valle, L., Schwartzman, R.J., & Alexander, G. (2009).
Spinal cord histopathological alterations in a patient with longstanding complex regional pain syndrome. Brain, behavior, and immunity, 23(1), 85-91. [PubMed:18786633] [WorldCat] [DOI] - ↑
Hickman, S.E., Allison, E.K., & El Khoury, J. (2008).
Microglial dysfunction and defective beta-amyloid clearance pathways in aging Alzheimer's disease mice. The Journal of neuroscience : the official journal of the Society for Neuroscience, 28(33), 8354-60. [PubMed:18701698] [PMC] [WorldCat] [DOI] - ↑
Krabbe, G., Halle, A., Matyash, V., Rinnenthal, J.L., Eom, G.D., Bernhardt, U., ..., & Heppner, F.L. (2013).
Functional impairment of microglia coincides with Beta-amyloid deposition in mice with Alzheimer-like pathology. PloS one, 8(4), e60921. [PubMed:23577177] [PMC] [WorldCat] [DOI] - ↑
Lucin, K.M., O'Brien, C.E., Bieri, G., Czirr, E., Mosher, K.I., Abbey, R.J., ..., & Wyss-Coray, T. (2013).
Microglial beclin 1 regulates retromer trafficking and phagocytosis and is impaired in Alzheimer's disease. Neuron, 79(5), 873-86. [PubMed:24012002] [PMC] [WorldCat] [DOI] - ↑
Mawuenyega, K.G., Sigurdson, W., Ovod, V., Munsell, L., Kasten, T., Morris, J.C., ..., & Bateman, R.J. (2010).
Decreased clearance of CNS beta-amyloid in Alzheimer's disease. Science (New York, N.Y.), 330(6012), 1774. [PubMed:21148344] [PMC] [WorldCat] [DOI] - ↑
Grathwohl, S.A., Kälin, R.E., Bolmont, T., Prokop, S., Winkelmann, G., Kaeser, S.A., ..., & Jucker, M. (2009).
Formation and maintenance of Alzheimer's disease beta-amyloid plaques in the absence of microglia. Nature neuroscience, 12(11), 1361-3. [PubMed:19838177] [PMC] [WorldCat] [DOI] - ↑
Stalder, M., Deller, T., Staufenbiel, M., & Jucker, M. (2001).
3D-Reconstruction of microglia and amyloid in APP23 transgenic mice: no evidence of intracellular amyloid. Neurobiology of aging, 22(3), 427-34. [PubMed:11378249] [WorldCat] [DOI] - ↑
Meyer-Luehmann, M., Spires-Jones, T.L., Prada, C., Garcia-Alloza, M., de Calignon, A., Rozkalne, A., ..., & Hyman, B.T. (2008).
Rapid appearance and local toxicity of amyloid-beta plaques in a mouse model of Alzheimer's disease. Nature, 451(7179), 720-4. [PubMed:18256671] [PMC] [WorldCat] [DOI] - ↑
Bolmont, T., Haiss, F., Eicke, D., Radde, R., Mathis, C.A., Klunk, W.E., ..., & Calhoun, M.E. (2008).
Dynamics of the microglial/amyloid interaction indicate a role in plaque maintenance. The Journal of neuroscience : the official journal of the Society for Neuroscience, 28(16), 4283-92. [PubMed:18417708] [PMC] [WorldCat] [DOI] - ↑ 85.0 85.1
Meyer-Luehmann, M., & Prinz, M. (2015).
Myeloid cells in Alzheimer's disease: culprits, victims or innocent bystanders? Trends in neurosciences, 38(10), 659-668. [PubMed:26442698] [WorldCat] [DOI] - ↑
El Khoury, J., Toft, M., Hickman, S.E., Means, T.K., Terada, K., Geula, C., & Luster, A.D. (2007).
Ccr2 deficiency impairs microglial accumulation and accelerates progression of Alzheimer-like disease. Nature medicine, 13(4), 432-8. [PubMed:17351623] [WorldCat] [DOI] - ↑
Hawkes, C.A., & McLaurin, J. (2009).
Selective targeting of perivascular macrophages for clearance of beta-amyloid in cerebral amyloid angiopathy. Proceedings of the National Academy of Sciences of the United States of America, 106(4), 1261-6. [PubMed:19164591] [PMC] [WorldCat] [DOI] - ↑
Heneka, M.T., Kummer, M.P., Stutz, A., Delekate, A., Schwartz, S., Vieira-Saecker, A., ..., & Golenbock, D.T. (2013).
NLRP3 is activated in Alzheimer's disease and contributes to pathology in APP/PS1 mice. Nature, 493(7434), 674-8. [PubMed:23254930] [PMC] [WorldCat] [DOI] - ↑
Halle, A., Hornung, V., Petzold, G.C., Stewart, C.R., Monks, B.G., Reinheckel, T., ..., & Golenbock, D.T. (2008).
The NALP3 inflammasome is involved in the innate immune response to amyloid-beta. Nature immunology, 9(8), 857-65. [PubMed:18604209] [PMC] [WorldCat] [DOI] - ↑ 90.0 90.1
Heppner, F.L., Ransohoff, R.M., & Becher, B. (2015).
Immune attack: the role of inflammation in Alzheimer disease. Nature reviews. Neuroscience, 16(6), 358-72. [PubMed:25991443] [WorldCat] [DOI] - ↑
Kleinberger, G., Yamanishi, Y., Suárez-Calvet, M., Czirr, E., Lohmann, E., Cuyvers, E., ..., & Haass, C. (2014).
TREM2 mutations implicated in neurodegeneration impair cell surface transport and phagocytosis. Science translational medicine, 6(243), 243ra86. [PubMed:24990881] [WorldCat] [DOI] - ↑
Jonsson, T., Stefansson, H., Steinberg, S., Jonsdottir, I., Jonsson, P.V., Snaedal, J., ..., & Stefansson, K. (2013).
Variant of TREM2 associated with the risk of Alzheimer's disease. The New England journal of medicine, 368(2), 107-16. [PubMed:23150908] [PMC] [WorldCat] [DOI] - ↑
Guerreiro, R., Wojtas, A., Bras, J., Carrasquillo, M., Rogaeva, E., Majounie, E., ..., & Alzheimer Genetic Analysis Group (2013).
TREM2 variants in Alzheimer's disease. The New England journal of medicine, 368(2), 117-27. [PubMed:23150934] [PMC] [WorldCat] [DOI] - ↑
Zhang, B., Gaiteri, C., Bodea, L.G., Wang, Z., McElwee, J., Podtelezhnikov, A.A., ..., & Emilsson, V. (2013).
Integrated systems approach identifies genetic nodes and networks in late-onset Alzheimer's disease. Cell, 153(3), 707-20. [PubMed:23622250] [PMC] [WorldCat] [DOI] - ↑
Griciuc, A., Serrano-Pozo, A., Parrado, A.R., Lesinski, A.N., Asselin, C.N., Mullin, K., ..., & Tanzi, R.E. (2013).
Alzheimer's disease risk gene CD33 inhibits microglial uptake of amyloid beta. Neuron, 78(4), 631-43. [PubMed:23623698] [PMC] [WorldCat] [DOI] - ↑
Jay, T.R., Miller, C.M., Cheng, P.J., Graham, L.C., Bemiller, S., Broihier, M.L., ..., & Lamb, B.T. (2015).
TREM2 deficiency eliminates TREM2+ inflammatory macrophages and ameliorates pathology in Alzheimer's disease mouse models. The Journal of experimental medicine, 212(3), 287-95. [PubMed:25732305] [PMC] [WorldCat] [DOI] - ↑
Wang, Y., Cella, M., Mallinson, K., Ulrich, J.D., Young, K.L., Robinette, M.L., ..., & Colonna, M. (2015).
TREM2 lipid sensing sustains the microglial response in an Alzheimer's disease model. Cell, 160(6), 1061-71. [PubMed:25728668] [PMC] [WorldCat] [DOI] - ↑
Ulrich, J.D., Finn, M.B., Wang, Y., Shen, A., Mahan, T.E., Jiang, H., ..., & Holtzman, D.M. (2014).
Altered microglial response to Aβ plaques in APPPS1-21 mice heterozygous for TREM2. Molecular neurodegeneration, 9, 20. [PubMed:24893973] [PMC] [WorldCat] [DOI] - ↑
Saito, T., Matsuba, Y., Mihira, N., Takano, J., Nilsson, P., Itohara, S., ..., & Saido, T.C. (2014).
Single App knock-in mouse models of Alzheimer's disease. Nature neuroscience, 17(5), 661-3. [PubMed:24728269] [WorldCat] [DOI] - ↑
Ince, P.G., Shaw, P.J., Slade, J.Y., Jones, C., & Hudgson, P. (1996).
Familial amyotrophic lateral sclerosis with a mutation in exon 4 of the Cu/Zn superoxide dismutase gene: pathological and immunocytochemical changes. Acta neuropathologica, 92(4), 395-403. [PubMed:8891072] [WorldCat] [DOI] - ↑
Sanagi, T., Yuasa, S., Nakamura, Y., Suzuki, E., Aoki, M., Warita, H., ..., & Ohsawa, K. (2010).
Appearance of phagocytic microglia adjacent to motoneurons in spinal cord tissue from a presymptomatic transgenic rat model of amyotrophic lateral sclerosis. Journal of neuroscience research, 88(12), 2736-46. [PubMed:20648658] [WorldCat] [DOI] - ↑
Forsberg, K., Andersen, P.M., Marklund, S.L., & Brännström, T. (2011).
Glial nuclear aggregates of superoxide dismutase-1 are regularly present in patients with amyotrophic lateral sclerosis. Acta neuropathologica, 121(5), 623-34. [PubMed:21287393] [PMC] [WorldCat] [DOI] - ↑
Beers, D.R., Henkel, J.S., Zhao, W., Wang, J., Huang, A., Wen, S., ..., & Appel, S.H. (2011).
Endogenous regulatory T lymphocytes ameliorate amyotrophic lateral sclerosis in mice and correlate with disease progression in patients with amyotrophic lateral sclerosis. Brain : a journal of neurology, 134(Pt 5), 1293-314. [PubMed:21596768] [PMC] [WorldCat] [DOI] - ↑
Boillée, S., Yamanaka, K., Lobsiger, C.S., Copeland, N.G., Jenkins, N.A., Kassiotis, G., ..., & Cleveland, D.W. (2006).
Onset and progression in inherited ALS determined by motor neurons and microglia. Science (New York, N.Y.), 312(5778), 1389-92. [PubMed:16741123] [WorldCat] [DOI] - ↑
Roberts, K., Zeineddine, R., Corcoran, L., Li, W., Campbell, I.L., & Yerbury, J.J. (2013).
Extracellular aggregated Cu/Zn superoxide dismutase activates microglia to give a cytotoxic phenotype. Glia, 61(3), 409-19. [PubMed:23281114] [WorldCat] [DOI] - ↑
Cardona, A.E., Pioro, E.P., Sasse, M.E., Kostenko, V., Cardona, S.M., Dijkstra, I.M., ..., & Ransohoff, R.M. (2006).
Control of microglial neurotoxicity by the fractalkine receptor. Nature neuroscience, 9(7), 917-24. [PubMed:16732273] [WorldCat] [DOI] - ↑
Heppner, F.L., Greter, M., Marino, D., Falsig, J., Raivich, G., Hövelmeyer, N., ..., & Aguzzi, A. (2005).
Experimental autoimmune encephalomyelitis repressed by microglial paralysis. Nature medicine, 11(2), 146-52. [PubMed:15665833] [WorldCat] [DOI] - ↑
Ponomarev, E.D., Shriver, L.P., Maresz, K., Pedras-Vasconcelos, J., Verthelyi, D., & Dittel, B.N. (2007).
GM-CSF production by autoreactive T cells is required for the activation of microglial cells and the onset of experimental autoimmune encephalomyelitis. Journal of immunology (Baltimore, Md. : 1950), 178(1), 39-48. [PubMed:17182538] [WorldCat] [DOI] - ↑
Krausgruber, T., Blazek, K., Smallie, T., Alzabin, S., Lockstone, H., Sahgal, N., ..., & Udalova, I.A. (2011).
IRF5 promotes inflammatory macrophage polarization and TH1-TH17 responses. Nature immunology, 12(3), 231-8. [PubMed:21240265] [WorldCat] [DOI] - ↑
Ponomarev, E.D., Veremeyko, T., Barteneva, N., Krichevsky, A.M., & Weiner, H.L. (2011).
MicroRNA-124 promotes microglia quiescence and suppresses EAE by deactivating macrophages via the C/EBP-α-PU.1 pathway. Nature medicine, 17(1), 64-70. [PubMed:21131957] [PMC] [WorldCat] [DOI] - ↑
Qin, H., Yeh, W.I., De Sarno, P., Holdbrooks, A.T., Liu, Y., Muldowney, M.T., ..., & Benveniste, E.N. (2012).
Signal transducer and activator of transcription-3/suppressor of cytokine signaling-3 (STAT3/SOCS3) axis in myeloid cells regulates neuroinflammation. Proceedings of the National Academy of Sciences of the United States of America, 109(13), 5004-9. [PubMed:22411837] [PMC] [WorldCat] [DOI]