「Förster共鳴エネルギー移動」の版間の差分
タグ: 巻き戻し |
|||
| (3人の利用者による、間の23版が非表示) | |||
| 4行目: | 4行目: | ||
<font size="+1">[http://researchmap.jp/2rikenbsi 林 康紀]</font><br> | <font size="+1">[http://researchmap.jp/2rikenbsi 林 康紀]</font><br> | ||
''独立行政法人理化学研究所 脳科学総合研究センター''<br> | ''独立行政法人理化学研究所 脳科学総合研究センター''<br> | ||
DOI [[XXXX]]/XXXX 原稿受付日:2013年9月18日 原稿完成日:2013年月日<br> | |||
担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | 担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | ||
</div> | </div> | ||
英:[[Förster]] resonance energy transfer 英略称:FRET 独:Förster-Resonanzenergietransfer 仏:transfert d'énergie entre molécules fluorescentes | 英:[[Förster]] resonance energy transfer 英略称:FRET 独:Förster-Resonanzenergietransfer 仏:transfert d'énergie entre molécules fluorescentes | ||
{{box|text= 二つの[[wj:蛍光|蛍光]]分子がごく近接して存在する場合、一つの蛍光分子からもう一つの蛍光分子へエネルギーが移行する。これをFörster共鳴エネルギー移動(FRET)という<ref | {{box|text= 二つの[[wj:蛍光|蛍光]]分子がごく近接して存在する場合、一つの蛍光分子からもう一つの蛍光分子へエネルギーが移行する。これをFörster共鳴エネルギー移動(FRET)という<ref><pubmed>22352636</pubmed></ref>。FRETの効率は2つの蛍光分子の[[wj:スペクトル|スペクトル]]の重なりの大きさ、距離と角度により左右されるため、FRETを測定する事により蛍光分子間の空間配置を間接的に測定する事が可能である。特に近年の[[GFP]]ならびにその類縁タンパク質を用いた遺伝子にコードされるFRETプローブが作成され、タンパク質相互作用、生化学反応や細胞内シグナル伝達を可視化する事が出来るようになった。}} | ||
==Förster共鳴エネルギー移動とは== | ==Förster共鳴エネルギー移動とは== | ||
[[Image:FRET-図3.jpg|thumb|right|300px|<b>図1. ドナーとアクセプターの励起、蛍光スペクトル</b><br>ドナーの蛍光スペクトルとアクセプターの励起スペクトルに重なりがあるときに、FRETが起きる。黒破線で囲まれたスペクトルは、励起スペクトルを示す。緑色のタンパク質がドナー、赤色タンパク質がアクセプターを示す。]] | [[Image:FRET-図3.jpg|thumb|right|300px|<b>図1. ドナーとアクセプターの励起、蛍光スペクトル</b><br>ドナーの蛍光スペクトルとアクセプターの励起スペクトルに重なりがあるときに、FRETが起きる。黒破線で囲まれたスペクトルは、励起スペクトルを示す。緑色のタンパク質がドナー、赤色タンパク質がアクセプターを示す。]] | ||
二つの蛍光分子がごく近接して存在する場合、一つの蛍光分子からもう一つの蛍光分子へ、エネルギーが移行する事が知られている(図1)。この現象は、1946年[[w:Theodor Förster|Theodor Förster]]によって報告されたことから、Förster共鳴エネルギー移動(FRET)という<ref>'''Förster, T.'''<br>Energiewanderung und Fluorescenz<br>''Naturwissenscaft''. 1946, 33:166–175</ref> <ref | 二つの蛍光分子がごく近接して存在する場合、一つの蛍光分子からもう一つの蛍光分子へ、エネルギーが移行する事が知られている(図1)。この現象は、1946年[[w:Theodor Förster|Theodor Förster]]によって報告されたことから、Förster共鳴エネルギー移動(FRET)という<ref>'''Förster, T.'''<br>Energiewanderung und Fluorescenz<br>''Naturwissenscaft''. 1946, 33:166–175</ref><ref><pubmed>22352636</pubmed></ref>。かつてFRETは、fluorescence resonance energy transferの略称として用いたが、実際には蛍光を伴わないエネルギー移動であることから、現在ではFörster resonance energy transferと呼ぶ事がIUPACにより推奨されている。 | ||
かつては生細胞にてFRETを検出するのは、非常に煩雑であった。プローブとなるタンパク質を精製、化学的に[[wj:蛍光色素|蛍光色素]]でラベルし、細胞に導入するという操作が必要であり、生物学分野での応用はきわめて限定されたものであった。しかし、GFPとその変異体、類縁タンパク質の発見により今日においては様々な分野において、多くの蛍光タンパク質を基にした、完全に遺伝子によってコードされるFRETプローブが使用されている。 | かつては生細胞にてFRETを検出するのは、非常に煩雑であった。プローブとなるタンパク質を精製、化学的に[[wj:蛍光色素|蛍光色素]]でラベルし、細胞に導入するという操作が必要であり、生物学分野での応用はきわめて限定されたものであった。しかし、GFPとその変異体、類縁タンパク質の発見により今日においては様々な分野において、多くの蛍光タンパク質を基にした、完全に遺伝子によってコードされるFRETプローブが使用されている。 | ||
| 71行目: | 72行目: | ||
まず、ドナーの蛍光のアクセプターチャネルへの漏れ込みであり、信号/雑音比の減少の原因となる。漏れ込みを極力抑えるには、適切な[[wj:バンドパスフィルター|バンドパスフィルター]]を用いる。光量を犠牲にしても、ドナー蛍光が漏れ込まない波長を選ぶ方がFRETは特異的に検出できる。 | まず、ドナーの蛍光のアクセプターチャネルへの漏れ込みであり、信号/雑音比の減少の原因となる。漏れ込みを極力抑えるには、適切な[[wj:バンドパスフィルター|バンドパスフィルター]]を用いる。光量を犠牲にしても、ドナー蛍光が漏れ込まない波長を選ぶ方がFRETは特異的に検出できる。 | ||
また、蛍光画像に背景雑音がある事があるが、それがFRET変化に影響を与える。背景雑音を引き算することで、よりFRETが計算できるが、蛍光シグナルが暗いと、少しの背景雑音のぶれがシグナルを左右する。例えば細胞の周辺は暗いので背景雑音の引き算により[[wj:偽陽性|偽陽性]]が出やすいので注意を要する。 | |||
2分子間FRETのイメージングでは、ドナーとアクセプターの局在の違いは偽陽性を生じる。リンカーで連結し1分子にするか、局在しているアクセプターの蛍光強度を補正することで避けることが可能である。 | 2分子間FRETのイメージングでは、ドナーとアクセプターの局在の違いは偽陽性を生じる。リンカーで連結し1分子にするか、局在しているアクセプターの蛍光強度を補正することで避けることが可能である。 | ||
=== | === アクセプターブリーチング法 === | ||
適切な波長の光によって、アクセプターを褪色させることでFRETを解消することができる。つまり、F'<sub>D</sub> | 適切な波長の光によって、アクセプターを褪色させることでFRETを解消することができる。つまり、F'<sub>D</sub>がアクセプターの退色によりF<sub>D</sub>と等しくなる事により、E=0となる。その為、褪色前後の画像を比較する事によりEが検出可能である。しかしながら、この手法は不可逆的であるために経時的変化を追うことは困難である。 | ||
[[Image:FRET-図2new.jpg|thumb|right|300px|<b>図2:励起光によって励起された電子が基底状態に戻る際の減衰曲線</b><br>緑の太い線がFRETが起きていない時のドナー蛍光の減衰曲線。FRETを起こしたドナー蛍光の速度が加わることにより速度定数が大きくなり細い緑の線のように減衰曲線の傾斜が大きくなる。 ''N<sub>0</sub>''は励起光によって励起された電子の数、''k''は励起状態にある電子が基底状態に戻る速度定数。]] | [[Image:FRET-図2new.jpg|thumb|right|300px|<b>図2:励起光によって励起された電子が基底状態に戻る際の減衰曲線</b><br>緑の太い線がFRETが起きていない時のドナー蛍光の減衰曲線。FRETを起こしたドナー蛍光の速度が加わることにより速度定数が大きくなり細い緑の線のように減衰曲線の傾斜が大きくなる。 ''N<sub>0</sub>''は励起光によって励起された電子の数、''k''は励起状態にある電子が基底状態に戻る速度定数。]] | ||
=== 蛍光寿命イメージング=== | === 蛍光寿命イメージング=== | ||
蛍光体が励起されると、図2に示すような減衰曲線に従って蛍光を発する。蛍光寿命は、''k''は励起状態にある電子が基底状態に戻る速度定数''k''の[[wj:逆数|逆数]]である。蛍光として基底状態に戻る際の速度定数、熱を発して基底状態に戻るなどの無放射遷移の速度定数の和として表される。 | 蛍光体が励起されると、図2に示すような減衰曲線に従って蛍光を発する。蛍光寿命は、''k''は励起状態にある電子が基底状態に戻る速度定数''k''の[[wj:逆数|逆数]]である。蛍光として基底状態に戻る際の速度定数、熱を発して基底状態に戻るなどの無放射遷移の速度定数の和として表される。 | ||
| 103行目: | 103行目: | ||
[[Image:FRET-図4.jpg|thumb|right|300px|<b>図3. [[海馬]][[スライス]][[CA1]][[錐体細胞]]に発現させたGFPの蛍光寿命イメージおよび減衰曲線</b><br>(横軸は時間、縦軸は光子数)。実際には、20秒で数千個オーダーの光子を取得する。これらの光子の発生確率分布が減衰曲線を形成し、近似曲線をフィッティングさせることで蛍光寿命を取得する。]] | [[Image:FRET-図4.jpg|thumb|right|300px|<b>図3. [[海馬]][[スライス]][[CA1]][[錐体細胞]]に発現させたGFPの蛍光寿命イメージおよび減衰曲線</b><br>(横軸は時間、縦軸は光子数)。実際には、20秒で数千個オーダーの光子を取得する。これらの光子の発生確率分布が減衰曲線を形成し、近似曲線をフィッティングさせることで蛍光寿命を取得する。]] | ||
====時間ドメイン==== | ====時間ドメイン==== | ||
励起光によって発生した一つ一つの光子が検出器まで届くまでの時間(数nsec)を計測することで時定数τ | 励起光によって発生した一つ一つの光子が検出器まで届くまでの時間(数nsec)を計測することで時定数τを計算する。時間を横軸としてヒストグラムを作製することができる。通常蛍光寿命は指数関数に従い減衰していく。FRETを起こしている分子と起こしていない分子が共存する時には[[wj:二重指数関数|二重指数関数]]になるため、二重指数関数にフィッティングすることによって、FRETの起きている分子の割合が算出できる。得られる光子の数が少ない時には二重指数関数フィッティングは不正確になりやすい為、単に平均蛍光寿命を計算するだけで済ませる場合も有る。単一指数関数の場合は、平均蛍光寿命はτに等しくなる。 | ||
理論上は取得した蛍光を全てデータに反映させることができるが、実際には光子取得後、再び光子を取得する状態に戻るハードウェアのリセット時間(dead time)などがあり全ての光子を取得する事は出来ない。また、秒単位の経時変化を追うためには、低解像度で画像取得されているのが現状で有り、多数のピクセルから蛍光寿命を取得するためには、処理速度の速いハードウェアが必要となる。 | 理論上は取得した蛍光を全てデータに反映させることができるが、実際には光子取得後、再び光子を取得する状態に戻るハードウェアのリセット時間(dead time)などがあり全ての光子を取得する事は出来ない。また、秒単位の経時変化を追うためには、低解像度で画像取得されているのが現状で有り、多数のピクセルから蛍光寿命を取得するためには、処理速度の速いハードウェアが必要となる。 | ||
| 110行目: | 110行目: | ||
[[File:FRET-Heterodyning.png|thumb|right|300px|<b>図4. 周波数ドメインによる測定</b><br>光源の強度を高周波で変調させるのと同時に、検出器も高周波で変調させる。その時に光源の周波数(f<sub>ex</sub>)と検出器の周波数(f<sub>s</sub>)をずらし、そこから蛍光寿命を計算により求める。]] | [[File:FRET-Heterodyning.png|thumb|right|300px|<b>図4. 周波数ドメインによる測定</b><br>光源の強度を高周波で変調させるのと同時に、検出器も高周波で変調させる。その時に光源の周波数(f<sub>ex</sub>)と検出器の周波数(f<sub>s</sub>)をずらし、そこから蛍光寿命を計算により求める。]] | ||
====周波数ドメイン==== | ====周波数ドメイン==== | ||
光源の強度を高周波で変調させるのと同時に、検出器も高周波で変調させる。その時に光源と検出器の周波数をずらしておく(heterodyning) | 光源の強度を高周波で変調させるのと同時に、検出器も高周波で変調させる。その時に光源と検出器の周波数をずらしておく(heterodyning)。多数のサイクルを繰り返す事により、間接的に蛍光寿命を計算していく。画像の取得にかかる時間がtime domainと比較して短いのが特徴である。 | ||
時間ドメインと同様に光源にはパルスレーザーや光量が高頻度にて変調できるレーザーなどが用いられる。 | 時間ドメインと同様に光源にはパルスレーザーや光量が高頻度にて変調できるレーザーなどが用いられる。 | ||
| 124行目: | 124行目: | ||
===プローブの分解に伴うFRETの変化を検出するプローブ=== | ===プローブの分解に伴うFRETの変化を検出するプローブ=== | ||
この原理は、FRETプローブの最も初期に導入されたデザインである(図5A)。プロテアーゼによって分解される配列の両端にドナーとアクセプターを連結する。プロテアーゼによって、この配列が分解されるとドナーとアクセプターの間に起きていたFRETが解消されることによって、プロテアーゼの活性を評価する。例として、[[wj:第X因子|第X因子]]、[[カスパーゼ]]などの[[プロテアーゼ]]活性のプローブが挙げられる<ref | この原理は、FRETプローブの最も初期に導入されたデザインである(図5A)。プロテアーゼによって分解される配列の両端にドナーとアクセプターを連結する。プロテアーゼによって、この配列が分解されるとドナーとアクセプターの間に起きていたFRETが解消されることによって、プロテアーゼの活性を評価する。例として、[[wj:第X因子|第X因子]]、[[wikipedia:ja:カスパーゼ|カスパーゼ]]などの[[wikipedia:ja:プロテアーゼ|プロテアーゼ]]活性のプローブが挙げられる<ref><pubmed>8707050</pubmed></ref><ref><pubmed>9518501</pubmed></ref><ref><pubmed>12409609</pubmed></ref><ref><pubmed>21637712</pubmed></ref><ref><pubmed>17946841</pubmed></ref>。このプローブのデザインの短所としては、反応が不可逆的であるために、一つの実験系で何度も測定することが困難であることである。 | ||
===二分子間相互作用を利用したFRETプローブ=== | ===二分子間相互作用を利用したFRETプローブ=== | ||
| 133行目: | 133行目: | ||
タンパク質相互作用を測定する事により間接的にシグナル伝達系の活性化も測定する事が可能である。例えば、[[低分子量Gタンパク質]]が活性化に伴い、エフェクタータンパク質と相互作用が起こる事を利用し、低分子量Gタンパク質活性を測定する事が可能である。 | タンパク質相互作用を測定する事により間接的にシグナル伝達系の活性化も測定する事が可能である。例えば、[[低分子量Gタンパク質]]が活性化に伴い、エフェクタータンパク質と相互作用が起こる事を利用し、低分子量Gタンパク質活性を測定する事が可能である。 | ||
また、タンパク質相互作用はポリマーでも良い。これを利用して[[アクチン]]の重合状態の測定にも用いられた<ref name= | また、タンパク質相互作用はポリマーでも良い。これを利用して[[アクチン]]の重合状態の測定にも用いられた<ref name=ref15361876><pubmed>15361876</pubmed></ref>(図5C)。 | ||
===一分子内FRETプローブ=== | ===一分子内FRETプローブ=== | ||
| 139行目: | 139行目: | ||
====タンパク質の構造変化==== | ====タンパク質の構造変化==== | ||
興味のあるタンパク質が、活性化の際に構造変化を誘起することが知られている場合には構造変化を利用することができる(図5D)。タンパク質のC末およびN末にドナーおよびアクセプターを連結する。あるいは構造変化が起こすドメインの方が、FRETは起こりやすい可能性もある。この手法は、[[CaMKII]]<ref | 興味のあるタンパク質が、活性化の際に構造変化を誘起することが知られている場合には構造変化を利用することができる(図5D)。タンパク質のC末およびN末にドナーおよびアクセプターを連結する。あるいは構造変化が起こすドメインの方が、FRETは起こりやすい可能性もある。この手法は、[[CaMKII]]<ref><pubmed>15788767</pubmed></ref>、[[カルシニューリン]]<ref><pubmed>18493642</pubmed></ref>、[[raf]]<ref><pubmed>15711535</pubmed></ref><ref><pubmed>16858395</pubmed></ref>、[[膜電位]]測定<ref><pubmed>18622396</pubmed></ref>、などに用いられている。 | ||
==== | ====タンパク質結合に伴う構造変化==== | ||
ある種のタンパク質は活性化、不活性化に伴い、下流のタンパク質と結合する。このような相互作用を利用してタンパク質の活性化、不活性化を測定することができる。 | |||
例えば[[カルシウム]]FRETプローブ、カメレオンはこの原理を利用している<ref | 例えば[[カルシウム]]FRETプローブ、カメレオンはこの原理を利用している<ref><pubmed>9278050</pubmed></ref>。 この場合、ドナー、[[カルモジュリン]]、カルモジュリン結合配列であるM13ペプチド、アクセプターを1つの分子に融合する。カルモジュリンがCa<sup>2+</sup>と結合すると、M13ペプチドと結合し、蛍光強度が変化する。 | ||
また、[[低分子量Gタンパク質]]の活性化プローブは、低分子量Gタンパク質、シグナル伝達下流の結合タンパク質の結合ドメインをドナーとアクセプターで挟んだ形状をしている(図5E)。低分子Gタンパク質が[[GDP]]から[[GTP]]結合型になり活性化すると、結合ドメインと相互作用によりFRETが生じる<ref><pubmed>16429133</pubmed></ref>。 | |||
====共有結合修飾によって生じる構造変化==== | ====共有結合修飾によって生じる構造変化==== | ||
このプローブは、ドナー、アクセプター、[[wj:共有結合|共有結合]]修飾を受けるドメイン、これを認識するドメインからなる。プローブが共有結合修飾を受けると、認識するドメインが結合し、ドナーとアクセプターの距離が縮まりFRETが起きる(図5F)。 | このプローブは、ドナー、アクセプター、[[wj:共有結合|共有結合]]修飾を受けるドメイン、これを認識するドメインからなる。プローブが共有結合修飾を受けると、認識するドメインが結合し、ドナーとアクセプターの距離が縮まりFRETが起きる(図5F)。 | ||
このプローブは、[[キナーゼ]]の活性化を測定するために使用される<ref | このプローブは、[[キナーゼ]]の活性化を測定するために使用される<ref><pubmed>11875431</pubmed></ref><ref><pubmed>11752449</pubmed></ref>。この場合、キナーゼの基質となるタンパク質に[[14-3-3タンパク質]]のような[[リン酸化]]タンパク質を認識するドメインを融合し、その結合に伴うタンパク質構造変化を、両端に結合させたドナーとアクセプター間のFRETで測定する。 | ||
====生体膜上の小分子==== | ====生体膜上の小分子==== | ||
このプローブは、主に、[[wj:脂質|脂質]]分子に応用されてきた(図5G)。ドナー、脂質結合ドメイン、アクセプターがヘリックス構造で連結され、[[グリシン]]-グリシン配列をその途中に導入することで、そこを中心に一方の蛍光タンパク質が回転することができる。膜結合ドメインを用いて、プローブを結合させる。脂質分子が増えた際に、脂質結合ドメインが脂質分子を認識し、構造変化が起き、ドナーとアクセプターの距離が縮まりFRETが生じる。[[ジアシルグリセロール]]<ref | このプローブは、主に、[[wj:脂質|脂質]]分子に応用されてきた(図5G)。ドナー、脂質結合ドメイン、アクセプターがヘリックス構造で連結され、[[グリシン]]-グリシン配列をその途中に導入することで、そこを中心に一方の蛍光タンパク質が回転することができる。膜結合ドメインを用いて、プローブを結合させる。脂質分子が増えた際に、脂質結合ドメインが脂質分子を認識し、構造変化が起き、ドナーとアクセプターの距離が縮まりFRETが生じる。[[ジアシルグリセロール]]<ref><pubmed>16990811</pubmed></ref>、[[ホスファチジルイノシトール|イノシトールリン脂質]]群<ref><pubmed>14528311</pubmed></ref><ref><pubmed>18685081</pubmed></ref><ref><pubmed>18685081</pubmed></ref><ref><pubmed>18685081</pubmed></ref>を測定するために用いられている。 | ||
{| class="wikitable" | {| class="wikitable" | ||
| 161行目: | 161行目: | ||
| 分類||対象||プローブ名||年||プローブデザイン||参考文献 | | 分類||対象||プローブ名||年||プローブデザイン||参考文献 | ||
|- | |- | ||
|rowspan="21" | 生体内小分子|| [[カルシウム]] ||Cameleon|| 1997 || E || <ref | |rowspan="21" | 生体内小分子|| [[カルシウム]] ||Cameleon|| 1997 || E || <ref><pubmed>9278050</pubmed></ref> | ||
|- | |- | ||
|[[サイクリックGMP]] ([[cGMP]]) ||CGY, Cygnet, pGES-DE2, cGi|| 2000, 2001, 2006 || | |[[サイクリックGMP]] ([[cGMP]]) ||CGY, Cygnet, pGES-DE2, cGi|| 2000, 2001, 2006 || 3-1 || <ref><pubmed>11140757</pubmed></ref><ref><pubmed>11226257</pubmed></ref><ref><pubmed>16369548</pubmed></ref><ref><pubmed>23801067</pubmed></ref> | ||
|- | |- | ||
|[[サイクリックAMP]] ([[cAMP]]) ||Epac|| 2000,2004 || | |[[サイクリックAMP]] ([[cAMP]]) ||Epac|| 2000,2004 || 2, 3-1 || <ref><pubmed>11872839</pubmed></ref><ref><pubmed> 15231839</pubmed></ref> | ||
|- | |- | ||
|[[イノシトール3リン酸]] ([[IP3|IP<sub>3</sub>]]) ||LIBRA, Fretino, FIRE|| 2004, 2005, 2006 || | |[[イノシトール3リン酸]] ([[IP3|IP<sub>3</sub>]]) ||LIBRA, Fretino, FIRE|| 2004, 2005, 2006 || 3-1 || <ref><pubmed>15272011</pubmed></ref><ref><pubmed> 16053285</pubmed></ref><ref><pubmed> 16754959</pubmed></ref> | ||
|- | |- | ||
|[[一酸化窒素]] ([[NO]]) ||NOA-1, Piccell|| 2005, 2006 || | |[[一酸化窒素]] ([[NO]]) ||NOA-1, Piccell|| 2005, 2006 || 3-1 || <ref><pubmed>16176986</pubmed></ref><ref><pubmed> 17165805</pubmed></ref> | ||
|- | |- | ||
|[[アデノシン3リン酸]] ([[ATP]]) ||A Team 1.03-nD/nA|| 2012 || | |[[アデノシン3リン酸]] ([[ATP]]) ||A Team 1.03-nD/nA|| 2012 || 3-1 || <ref><pubmed>19720993</pubmed></ref> | ||
|- | |- | ||
|[[エストロゲン]] ||SCCoR|| 2004 || | |[[エストロゲン]] ||SCCoR|| 2004 || 3-3 || <ref><pubmed>15080726</pubmed></ref> | ||
|- | |- | ||
|[[アンドロゲン]] ||Ficaro|| 2006 || | |[[アンドロゲン]] ||Ficaro|| 2006 || 3-3 || <ref><pubmed>16555356</pubmed></ref> | ||
|- | |- | ||
|[[グルココルチコイド受容体]]リガンド ||GLUCOCOR|| 2007 || | |[[グルココルチコイド受容体]]リガンド ||GLUCOCOR|| 2007 || 3-3 || <ref><pubmed>15163683</pubmed></ref><ref><pubmed>17897691</pubmed></ref> | ||
|- | |- | ||
|[[神経成長因子]] ||ECaus|| 2008 || | |[[神経成長因子]] ||ECaus|| 2008 || 3-3 || <ref><pubmed>18510313</pubmed></ref> | ||
|- | |- | ||
|[[核内受容体]] ||conpro|| 2007 || | |[[核内受容体]] ||conpro|| 2007 || 3-2 || <ref><pubmed>17387660</pubmed></ref> | ||
|- | |- | ||
|[[O-N-アセチルグルコサミン]] ([[O-GlcNAc]]) || || 2006 || | |[[O-N-アセチルグルコサミン]] ([[O-GlcNAc]]) || || 2006 || 3-3 || <ref><pubmed>17105262</pubmed></ref> | ||
|- | |- | ||
|[[ビタミンA]] ([[レチノイン酸]]) ||GEPRAS|| 2013 || | |[[ビタミンA]] ([[レチノイン酸]]) ||GEPRAS|| 2013 || 3-1 || <ref><pubmed>23563268</pubmed></ref> | ||
|- | |- | ||
|[[wj:モリブデン酸|モリブデン酸]] ||MolyProbe|| 2013 || | |[[wj:モリブデン酸|モリブデン酸]] ||MolyProbe|| 2013 || 3-1 || <ref><pubmed>23472155</pubmed></ref> | ||
|- | |- | ||
|[[グルタミン酸]] ||FLIPE|| 2005 || | |[[グルタミン酸]] ||FLIPE|| 2005 || 3-1 || <ref><pubmed>15939876</pubmed></ref> | ||
|- | |- | ||
|[[wj:亜鉛|Zn<sup>2+</sup>]] ||eCALWY-1|| 2009 || | |[[wj:亜鉛|Zn<sup>2+</sup>]] ||eCALWY-1|| 2009 || 2 || <ref><pubmed>19718032</pubmed></ref> | ||
|- | |- | ||
|[[wj:塩素|Cl<sup>-</sup>]] ||Clomeleon|| 2000 || その他 || <ref><pubmed>11055428</pubmed></ref> | |[[wj:塩素|Cl<sup>-</sup>]] ||Clomeleon|| 2000 || その他 || <ref><pubmed>11055428</pubmed></ref> | ||
| 197行目: | 197行目: | ||
|[[wj:水素イオン濃度|水素イオン濃度]] (pH) ||GFpH, YFpH|| 2001 || その他 || <ref><pubmed>11716495</pubmed></ref> | |[[wj:水素イオン濃度|水素イオン濃度]] (pH) ||GFpH, YFpH|| 2001 || その他 || <ref><pubmed>11716495</pubmed></ref> | ||
|- | |- | ||
|[[wj:グルコース|グルコース]] ||FLIPglu|| 2003 || | |[[wj:グルコース|グルコース]] ||FLIPglu|| 2003 || 3-1 || <ref><pubmed>12649277</pubmed></ref> | ||
|- | |- | ||
|[[wj:マルトース|マルトース]]||FLIPmal|| 2002 || | |[[wj:マルトース|マルトース]]||FLIPmal|| 2002 || 3-1 || <ref><pubmed>12097642</pubmed></ref> | ||
|- | |- | ||
|[[wj:リボース|リボース]]||FLIPrib|| 2003 || | |[[wj:リボース|リボース]]||FLIPrib|| 2003 || 3-1 || <ref><pubmed>14550551</pubmed></ref> | ||
|- | |- | ||
|rowspan="25" | タンパク質リン酸化酵素 || [[カルシウム/カルモジュリン依存性タンパク質リン酸化酵素II]] ([[CaMKII]]) ||Camui α, green-Camui α, Camk2a reporter|| 2005, 2009, 2011, 2013 || D || <ref | |rowspan="25" | タンパク質リン酸化酵素 || [[カルシウム/カルモジュリン依存性タンパク質リン酸化酵素II]] ([[CaMKII]]) ||Camui α, green-Camui α, Camk2a reporter|| 2005, 2009, 2011, 2013 || D || <ref><pubmed>15788767</pubmed></ref><ref><pubmed> 19295602</pubmed></ref><ref name=ref23602566><pubmed> 23602566</pubmed></ref><ref name=ref21506563><pubmed> 21506563</pubmed></ref> | ||
|- | |- | ||
| [[Src]] ||Srcus|| 2005, 2005, 2007 || | | [[Src]] ||Srcus|| 2005, 2005, 2007 || 3-3 || <ref name=ref11752449><pubmed>11752449</pubmed></ref><ref><pubmed> 15846350</pubmed></ref><ref><pubmed> 17284441</pubmed></ref> | ||
|- | |- | ||
| [[Ca2+/リン脂質依存性タンパク質リン酸化酵素|Ca<sup>2+</sup>/リン脂質依存性タンパク質リン酸化酵素]] ([[Cキナーゼ]], [[プロテインキナーゼC]], [[PKC]]) ||CKAR, CY-PKCdelta|| 2003, 2005 || | | [[Ca2+/リン脂質依存性タンパク質リン酸化酵素|Ca<sup>2+</sup>/リン脂質依存性タンパク質リン酸化酵素]] ([[Cキナーゼ]], [[プロテインキナーゼC]], [[PKC]]) ||CKAR, CY-PKCdelta|| 2003, 2005 || 3-3, 3-1 || <ref><pubmed>12782683</pubmed></ref><ref><pubmed> 15611119</pubmed></ref> | ||
|- | |- | ||
| [[タンパク質リン酸化酵素D]] ([[Dキナーゼ]], [[プロテインキナーゼD]], [[PKD]]) ||DKAR|| 2007 || | | [[タンパク質リン酸化酵素D]] ([[Dキナーゼ]], [[プロテインキナーゼD]], [[PKD]]) ||DKAR|| 2007 || 3-3 || <ref><pubmed>17189263</pubmed></ref> | ||
|- | |- | ||
| [[cAMP依存性タンパク質リン酸化酵素]] ([[Aキナーゼ]], [[プロテインキナーゼA]], [[PKA]]) ||ART, AKAR|| 2000, 2001 || | | [[cAMP依存性タンパク質リン酸化酵素]] ([[Aキナーゼ]], [[プロテインキナーゼA]], [[PKA]]) ||ART, AKAR|| 2000, 2001 || 3-3 || <ref><pubmed>10700148</pubmed></ref><ref><pubmed>11752448</pubmed></ref> | ||
|- | |- | ||
| [[Abl]] ||Picchu|| 2001 || | | [[Abl]] ||Picchu|| 2001 || E || <ref name=ref11752449 /> | ||
|- | |- | ||
| [[Bcr]]-Abl ||Bcr-Abl activity sensor||2010 || | | [[Bcr]]-Abl ||Bcr-Abl activity sensor||2010 || E || <ref><pubmed>20817824</pubmed></ref> | ||
|- | |- | ||
| [[c-Raf]] ||Prin-cRaf|| 2005 || | | [[c-Raf]] ||Prin-cRaf|| 2005 || 3-1 || <ref><pubmed>15711535</pubmed></ref> | ||
|- | |- | ||
| [[P21 protein-activated kinase 1]] ([[PAK1]]) ||Pakabi|| 2009 || | | [[P21 protein-activated kinase 1]] ([[PAK1]]) ||Pakabi|| 2009 || 3-1 || <ref><pubmed>19574218</pubmed></ref> | ||
|- | |- | ||
| [[B-raf]] ||Prin-Braf|| 2006 || | | [[B-raf]] ||Prin-Braf|| 2006 || 3-1 || <ref><pubmed>16858395</pubmed></ref> | ||
|- | |- | ||
| [[Zeta-chain-associated protein kinase 70]] ([[ZAP-70]]) ||ROZA|| 2008 || | | [[Zeta-chain-associated protein kinase 70]] ([[ZAP-70]]) ||ROZA|| 2008 || 3-3 || <ref><pubmed>18231606</pubmed></ref> | ||
|- | |- | ||
| [[Akt]] ||Aktus, BKAR, Akind|| 2003, 2005, 2007 || | | [[Akt]] ||Aktus, BKAR, Akind|| 2003, 2005, 2007 || 3-3 || <ref><pubmed>12773546</pubmed></ref><ref><pubmed> 15583002</pubmed></ref><ref name=ref17407381><pubmed> 17407381</pubmed></ref> | ||
|- | |- | ||
| [[細胞外シグナル調節キナーゼ]] ([[Extracellular signal-regulated kinases]], [[ERK]]) ||Miu2, Erkus, EKAR|| 2006, 2007, 2008 || | | [[細胞外シグナル調節キナーゼ]] ([[Extracellular signal-regulated kinases]], [[ERK]]) ||Miu2, Erkus, EKAR|| 2006, 2007, 2008 || 3-1,3-3,3-3 || <ref><pubmed>16418172</pubmed></ref><ref><pubmed> 17261026</pubmed></ref><ref><pubmed> 19033456</pubmed></ref> | ||
|- | |- | ||
| [[インシュリン受容体]] ||Phocus|| 2002 || | | [[インシュリン受容体]] ||Phocus|| 2002 || 3-3 || <ref><pubmed>11875431</pubmed></ref> | ||
|- | |- | ||
| [[上皮成長因子受容体]] ([[EGFR]]) || ||2001 || | | [[上皮成長因子受容体]] ([[EGFR]]) || ||2001 || 3-3 || <ref name=ref11752449 /> | ||
|- | |- | ||
| [[Ataxia telangiectasia mutated]] ([[ATM]]) || || 2007 || | | [[Ataxia telangiectasia mutated]] ([[ATM]]) || || 2007 || 3-3 || <ref><pubmed>17428747</pubmed></ref> | ||
|- | |- | ||
|[[オーロラBキナーゼ]] || || 2008 || | |[[オーロラBキナーゼ]] || || 2008 || 3-3 || <ref><pubmed>18463638</pubmed></ref> | ||
|- | |- | ||
| [[サイクリンB1]]-[[サイクリン依存性キナーゼ1]] || || 2010 || | | [[サイクリンB1]]-[[サイクリン依存性キナーゼ1]] || || 2010 || 3-3 || <ref><pubmed>20412769</pubmed></ref> | ||
|- | |- | ||
| [[ミオシン軽鎖キナーゼ]] ||MLCK-FIP || 2002 || | | [[ミオシン軽鎖キナーゼ]] ||MLCK-FIP || 2002 || 3-1 || <ref><pubmed>11815633</pubmed></ref> | ||
|- | |- | ||
| [[c-Jun N端末キナーゼ]] ([[JNK]]) ||JNKAR1, JUNKAR1EV || 2010, 2011 || | | [[c-Jun N端末キナーゼ]] ([[JNK]]) ||JNKAR1, JUNKAR1EV || 2010, 2011 || 3-3 || <ref><pubmed>20212108</pubmed></ref><ref name=ref21976697><pubmed> 21976697</pubmed></ref> | ||
|- | |- | ||
| [[リボソームS6キナーゼ]] ([[RSK]], [[S6K]]) ||Eevee-RSK, Eevee-S6K|| 2011 || | | [[リボソームS6キナーゼ]] ([[RSK]], [[S6K]]) ||Eevee-RSK, Eevee-S6K|| 2011 || 3-3 || <ref name=ref21976697></ref> | ||
|- | |- | ||
| [[接着斑キナーゼ]] ([[Focal Adhesion Kinase]], [[FAK]]) ||CYFAK413, FERM-sensor|| 2008, 2009 || | | [[接着斑キナーゼ]] ([[Focal Adhesion Kinase]], [[FAK]]) ||CYFAK413, FERM-sensor|| 2008, 2009 || 2, 3-1 || <ref><pubmed>17967873</pubmed></ref><ref><pubmed> 19208768</pubmed></ref> | ||
|- | |- | ||
| [[Polo様キナーゼ1]] ([[polo-like kinase1]], [[PLK1]]) || || 2008 || | | [[Polo様キナーゼ1]] ([[polo-like kinase1]], [[PLK1]]) || || 2008 || 3-3 || <ref><pubmed>18615013</pubmed></ref> | ||
|- | |- | ||
| [[ストレス活性化プロテインキナーゼ3]] ([[Stress-Activated Protein Kinase Kinase Kinase]], [[stress-activated protein kinase 3]], [[SAP3K]]) || || 2009 || | | [[ストレス活性化プロテインキナーゼ3]] ([[Stress-Activated Protein Kinase Kinase Kinase]], [[stress-activated protein kinase 3]], [[SAP3K]]) || || 2009 || 3-3 || <ref><pubmed>19737916</pubmed></ref> | ||
|- | |- | ||
| [[細胞死関連タンパク質キナーゼ]] ([[death associated protein kinase 1]], [[DAPK1]]) ||DAPK1(334)-F40|| 2011 || | | [[細胞死関連タンパク質キナーゼ]] ([[death associated protein kinase 1]], [[DAPK1]]) ||DAPK1(334)-F40|| 2011 || 3-1 || <ref name=ref21506563 /> | ||
|- | |- | ||
| [[ホスファターゼ]] || [[カルシニューリン]] ||CaNAR1|| 2008, 2013 || | | [[ホスファターゼ]] || [[カルシニューリン]] ||CaNAR1|| 2008, 2013 || 3-1 || <ref name=ref23602566 /><ref><pubmed> 18493642</pubmed></ref> | ||
|- | |- | ||
|rowspan="8" | [[低分子量Gタンパク質]] || [[Ras]] ||Raichu-Ras, Fras|| 2001, 2006 || B, E || <ref | |rowspan="8" | [[低分子量Gタンパク質]] || [[Ras]] ||Raichu-Ras, Fras|| 2001, 2006 || B, E || <ref><pubmed>16429133</pubmed></ref><ref name=ref11429608><pubmed> 11429608</pubmed></ref> | ||
|- | |- | ||
| [[Rap]] ||Raichu-Rap|| 2001 || E || <ref name=ref11429608></ref> | | [[Rap]] ||Raichu-Rap|| 2001 || E || <ref name=ref11429608></ref> | ||
| 263行目: | 263行目: | ||
| [[Rab5]] ||Raichu-Rab5|| 2008 || E || <ref><pubmed>18385674</pubmed></ref> | | [[Rab5]] ||Raichu-Rab5|| 2008 || E || <ref><pubmed>18385674</pubmed></ref> | ||
|- | |- | ||
| [[Rho]] ||Raichu-RhoA|| 2003, 2011 || B, E || <ref name=ref21423166><pubmed>21423166</pubmed></ref> <ref><pubmed> 12860967</pubmed></ref> | | [[Rho]] ||Raichu-RhoA|| 2003, 2011 || B, E || <ref name=ref21423166><pubmed>21423166</pubmed></ref><ref><pubmed> 12860967</pubmed></ref> | ||
|- | |- | ||
| [[Cdc42]] ||Raichu-cdc42|| 2004, 2011 || B, E || <ref name=ref14570905 /><ref name=ref21423166 /> | | [[Cdc42]] ||Raichu-cdc42|| 2004, 2011 || B, E || <ref name=ref14570905 /><ref name=ref21423166 /> | ||
|- | |- | ||
| [[Ral]] ||Raichu-Ral|| 2004 || | | [[Ral]] ||Raichu-Ral|| 2004 || 3-3 || <ref><pubmed>15034142</pubmed></ref> | ||
|- | |- | ||
| [[TC10]] ||Raichu-TC10|| 2006 || | | [[TC10]] ||Raichu-TC10|| 2006 || 3-2 || <ref><pubmed>16950130</pubmed></ref> | ||
|- | |- | ||
|rowspan="6" | [[シグナル伝達]] || [[RCC1]]||CFP-RCC1-YFP|| 2008 || | |rowspan="6" | [[シグナル伝達]] || [[RCC1]] ([[Ran]]に対する[[GDP, GTP交換因子]]) ||CFP-RCC1-YFP|| 2008 || 3-1 || <ref><pubmed>18762580</pubmed></ref> | ||
|- | |- | ||
| [[CrkII]][[リン酸化]] ||Picchu|| 2001 || | | [[CrkII]][[リン酸化]] ||Picchu|| 2001 || 3-1 || <ref><pubmed>11406630</pubmed></ref> | ||
|- | |- | ||
| [[neuronal Wiskott–Aldrich syndrome protein]] | | [[Wiskott-Aldrich syndrome-like]] ([[WASL]], [[neuronal Wiskott–Aldrich syndrome protein]], [[N-WASP]]) ||Stinger|| 2004 || 3-1 || <ref><pubmed>15084285</pubmed></ref><ref><pubmed> 14732696</pubmed></ref> | ||
|- | |- | ||
| [[アドレナリン受容体]] ||α<sub>2A</sub>[[AR]]-cam|| 2003 || | | [[アドレナリン受容体]] ||α<sub>2A</sub>[[AR]]-cam|| 2003 || 3-1 || <ref name=ref12808462><pubmed>12808462</pubmed></ref> | ||
|- | |- | ||
| [[副甲状腺ホルモン受容体]] ||PTHR-cam|| 2003 || | | [[副甲状腺ホルモン受容体]] ||PTHR-cam|| 2003 || 3-1 || <ref name=ref12808462 /> | ||
|- | |- | ||
| [[細胞膜]][[カルシウムポンプ]] ||BFP-PMCA-GFP|| 2007 || | | [[細胞膜]][[カルシウムポンプ]] ||BFP-PMCA-GFP|| 2007 || 3-1 || <ref><pubmed>17901055</pubmed></ref> | ||
|- | |- | ||
| [[アセチル化]] || [[ヒストン]]アセチル化 ||Histac|| 2004, 2009 || | | [[アセチル化]] || [[ヒストン]]アセチル化 ||Histac|| 2004, 2009 || 3-3 || <ref><pubmed>15137760</pubmed></ref><ref><pubmed> 19805290</pubmed></ref> | ||
|- | |- | ||
|rowspan="6" | [[脂質]] || [[ホスファチジルイノシトール-3,4,5-三リン酸]] ([[PIP3|PIP<sub>3</sub>]]) ||Fllip, FLIMPA|| 2003 || G || <ref | |rowspan="6" | [[脂質]] || [[ホスファチジルイノシトール-3,4,5-三リン酸]] ([[PIP3|PIP<sub>3</sub>]]) ||Fllip, FLIMPA|| 2003 || G || <ref><pubmed>14528311</pubmed></ref> | ||
|- | |- | ||
| [[ホスファチジルイノシトール-4,5-二リン酸]] ([[PIP2|PI(4,5)P<sub>2</sub>]]) ||Pippi-PI(4,5)P<sub>2</sub>|| 2008 || G || <ref name=ref18685081 /> | | [[ホスファチジルイノシトール-4,5-二リン酸]] ([[PIP2|PI(4,5)P<sub>2</sub>]]) ||Pippi-PI(4,5)P<sub>2</sub>|| 2008 || G || <ref name=ref18685081><pubmed>18685081</pubmed></ref> | ||
|- | |- | ||
| [[ホスファチジルイノシトール-3,4-二リン酸]] ([[PI(3,4)P2|PI(3,4)P<sub>2</sub>]]) ||Pippi-PI(3,4)P<sub>2</sub>|| 2008 || G || <ref name=ref18685081 /> | | [[ホスファチジルイノシトール-3,4-二リン酸]] ([[PI(3,4)P2|PI(3,4)P<sub>2</sub>]]) ||Pippi-PI(3,4)P<sub>2</sub>|| 2008 || G || <ref name=ref18685081 /> | ||
| 295行目: | 295行目: | ||
| [[ホスファチジン酸]] ||Pii|| 2010 || G || <ref><pubmed>20826779</pubmed></ref> | | [[ホスファチジン酸]] ||Pii|| 2010 || G || <ref><pubmed>20826779</pubmed></ref> | ||
|- | |- | ||
| [[ジアシルグリセロール]] (DAG) ||Daglas, DIGDA|| 2006, 2008 || G || <ref name=ref18685081 /> <ref | | [[ジアシルグリセロール]] (DAG) ||Daglas, DIGDA|| 2006, 2008 || G || <ref name=ref18685081 /><ref><pubmed> 16990811</pubmed></ref> | ||
|- | |- | ||
|rowspan="6" | タンパク質相互作用 || [[アクチン]] || ||2004, 2008 || C || <ref | |rowspan="6" | タンパク質相互作用 || [[アクチン]] || ||2004, 2008 || C || <ref><pubmed>15361876</pubmed></ref><ref><pubmed> 18512154</pubmed></ref> | ||
|- | |- | ||
| [[3-ホスホイノシチド依存性プロテインキナーゼ1]]([[phosphoinositide-dependent protein kinase 1]], [[PDK1]])-[[タンパク質キナーゼB]] ([[protein kinase B]], [[PKB]], [[Akt]])相互作用 || ||2007 || | | [[3-ホスホイノシチド依存性プロテインキナーゼ1]]([[phosphoinositide-dependent protein kinase 1]], [[PDK1]])-[[タンパク質キナーゼB]] ([[protein kinase B]], [[PKB]], [[Akt]])相互作用 || ||2007 || 2 || <ref name=ref17407381 /> | ||
|- | |- | ||
| [[タンパク質チロシン脱リン酸化酵素1B]] ([[protein tyrosine phosphatase 1B]], [[PTP 1B]])-[[受容体型チロシンキナーゼ]] ([[receptor tyrosine kinase]]s, [[RTK]]s)相互作用 || || 2002 || | | [[タンパク質チロシン脱リン酸化酵素1B]] ([[protein tyrosine phosphatase 1B]], [[PTP 1B]])-[[受容体型チロシンキナーゼ]] ([[receptor tyrosine kinase]]s, [[RTK]]s)相互作用 || || 2002 || 2 || <ref><pubmed>11872838</pubmed></ref> | ||
|- | |- | ||
| [[乳癌耐性タンパク質]] ([[breast cancer resistance protein]], [[BCRP]])/[[ATP結合カセット輸送体]] ([[ATP-binding cassette sub-family G member]], [[ABCG]])相互作用 || ||2010 || | | [[乳癌耐性タンパク質]] ([[breast cancer resistance protein]], [[BCRP]])/[[ATP結合カセット輸送体]] ([[ATP-binding cassette sub-family G member]], [[ABCG]])相互作用 || ||2010 || 2 || <ref><pubmed>20622991</pubmed></ref> | ||
|- | |- | ||
| [[コフィリン]]-[[アクチン]]相互作用 || ||2008 || B || <ref><pubmed>17993279</pubmed></ref> | | [[コフィリン]]-[[アクチン]]相互作用 || ||2008 || B || <ref><pubmed>17993279</pubmed></ref> | ||
|- | |- | ||
| [[第10染色体ホスファターゼ・テンシン・ホモログ]] ([[Phosphatase and tensin homolog deleted from chromosome 10]], [[PTEN]])-[[ミオシンV]]相互作用 || ||2009 || | | [[第10染色体ホスファターゼ・テンシン・ホモログ]] ([[Phosphatase and tensin homolog deleted from chromosome 10]], [[PTEN]])-[[ミオシンV]]相互作用 || ||2009 || 2 || <ref><pubmed>19767745</pubmed></ref> | ||
|- | |- | ||
|rowspan="8" | [[プロテアーゼ]] || [[カスパーゼ-3]] ||EGFP-DEVD-EBFP|| 1998 || A || <ref | |rowspan="8" | [[プロテアーゼ]] || [[カスパーゼ-3]] ||EGFP-DEVD-EBFP|| 1998 || A || <ref><pubmed>9518501</pubmed></ref> | ||
|- | |- | ||
| [[カスパーゼ-8]] ||CFP-c3-YFP-c6-mRFP|| 2002 || A || <ref | | [[カスパーゼ-8]] ||CFP-c3-YFP-c6-mRFP|| 2002 || A || <ref><pubmed>12409609</pubmed></ref> | ||
|- | |- | ||
| [[カスパーゼ-9]] ||SCAT9|| 2011 || A || <ref | | [[カスパーゼ-9]] ||SCAT9|| 2011 || A || <ref><pubmed>21637712</pubmed></ref> | ||
|- | |- | ||
| [[カスパーゼ-7]] ||VDEVDc|| 2006 || A || <ref | | [[カスパーゼ-7]] ||VDEVDc|| 2006 || A || <ref><pubmed>17946841</pubmed></ref> | ||
|- | |- | ||
| [[マトリックスメタロプロテアーゼ]] ([[MMP]]) ||YFP-MSS-CFP<sup>display</sup>, MTI-MMP-FRET biosensor|| 2007, 2008 || A || <ref><pubmed>17187878</pubmed></ref><ref><pubmed> 18441011</pubmed></ref> | | [[マトリックスメタロプロテアーゼ]] ([[MMP]]) ||YFP-MSS-CFP<sup>display</sup>, MTI-MMP-FRET biosensor|| 2007, 2008 || A || <ref><pubmed>17187878</pubmed></ref><ref><pubmed> 18441011</pubmed></ref> | ||
|- | |- | ||
| [[wj:第Xa因子|第Xa因子]] || ||1996 || A || <ref | | [[wj:第Xa因子|第Xa因子]] || ||1996 || A || <ref><pubmed>8707050</pubmed></ref> | ||
|- | |- | ||
|[[カルパイン]]活性||pYSCS|| 2000 || A || <ref><pubmed>10688895</pubmed></ref> | |[[カルパイン]]活性||pYSCS|| 2000 || A || <ref><pubmed>10688895</pubmed></ref> | ||
|- | |- | ||
| [[プレセニリン]] ||GFP-PSI-RFP|| 2009 || | | [[プレセニリン]] ||GFP-PSI-RFP|| 2009 || 3-1 || <ref><pubmed>19924286</pubmed></ref> | ||
|- | |- | ||
|rowspan="5" | その他 ||細胞膜張力センサー ||stFRET|| 2008 || | |rowspan="5" | その他 ||細胞膜張力センサー ||stFRET|| 2008 || 3-1 || <ref><pubmed>18479457</pubmed></ref> | ||
|- | |- | ||
| [[膜電位]] ||VSFP, Mermaid, ArcLight, VSFP-Butterfly|| 2001, 2008, 2012 || D || <ref><pubmed>11454036</pubmed></ref> <ref | | [[膜電位]] ||VSFP, Mermaid, ArcLight, VSFP-Butterfly|| 2001, 2008, 2012 || D || <ref><pubmed>11454036</pubmed></ref><ref><pubmed> 18622396</pubmed></ref><ref><pubmed> 22958819</pubmed></ref><ref><pubmed> 23868559</pubmed></ref> | ||
|- | |- | ||
| [[ミオシンII]] ||GSldCB|| 1998, 2006 || | | [[ミオシンII]] ||GSldCB|| 1998, 2006 || 3-1 || <ref><pubmed>9845076</pubmed></ref><ref><pubmed> 16939200</pubmed></ref> | ||
|- | |- | ||
| [[wj:ヒト免疫不全ウイルス|ヒト免疫不全ウイルス]]Revタンパク質 ||YRGnC-11ad|| 2005 || | | [[wj:ヒト免疫不全ウイルス|ヒト免疫不全ウイルス]]Revタンパク質 ||YRGnC-11ad|| 2005 || 3-1 || <ref><pubmed>16013840</pubmed></ref> | ||
|- | |- | ||
|| [[wj:酸化還元|酸化還元]] ||Redoxfluor, Gaskins|| 2010, 2011 || | || [[wj:酸化還元|酸化還元]] ||Redoxfluor, Gaskins|| 2010, 2011 || 3-1 || <ref><pubmed>20498274</pubmed></ref><ref><pubmed> 21606117</pubmed></ref> | ||
|} | |} | ||
プローブデザインA- | プローブデザインA-Gは図4に対応している。京都大学医学部松田道行による[http://www.lif.kyoto-u.ac.jp/labs/fret/phogemon/index.htm Phogemon Project] などを参考に作成。 | ||
==蛍光色素の選択== | ==蛍光色素の選択== | ||
現在多くの場合GFPあるいはその関連タンパク質が用いられている。 | 現在多くの場合GFPあるいはその関連タンパク質が用いられている。 | ||
蛍光強度比イメージングの場合は、GFPの色彩変異体であるシアン色蛍光タンパク質CFPと黄色蛍光タンパク質YFPのFRETペアがよく用いられている。CFPの中でも、Ceruleanが明るい蛍光を示すためこれを用いるべきである。YFPの変異体の中では、Venus、Ypetがよい。いずれも若干の凝集傾向が有り、これはタンパク質表面にある三つのアラニン残基のメチル基によるものとされており、それを変異させたmonomeric GFP (A206K変異体)で凝集を避ける事が出来る<ref name=ref11988576><pubmed>11988576</pubmed></ref> <ref name=ref18512154 />。一方で、プロテアーゼプローブなどでは、FRETダイナミックレンジが改善するという報告もある<ref name=ref15696158><pubmed>15696158</pubmed></ref> | 蛍光強度比イメージングの場合は、GFPの色彩変異体であるシアン色蛍光タンパク質CFPと黄色蛍光タンパク質YFPのFRETペアがよく用いられている。CFPの中でも、Ceruleanが明るい蛍光を示すためこれを用いるべきである。YFPの変異体の中では、Venus、Ypetがよい。いずれも若干の凝集傾向が有り、これはタンパク質表面にある三つのアラニン残基のメチル基によるものとされており、それを変異させたmonomeric GFP (A206K変異体)で凝集を避ける事が出来る<ref name=ref11988576><pubmed>11988576</pubmed></ref><ref name=ref18512154><pubmed>18512154</pubmed></ref>。一方で、プロテアーゼプローブなどでは、FRETダイナミックレンジが改善するという報告もある<ref name=ref15696158><pubmed>15696158</pubmed></ref> | ||
<ref name=ref17586775><pubmed>17586775</pubmed></ref>。CFPとYFPはいずれもGFPの変異体で殆ど同一の配列である為か、トランスジェニック動物が作りにくい事が経験的に知られている{kamioka, 2012}。 | <ref name=ref17586775><pubmed>17586775</pubmed></ref>。CFPとYFPはいずれもGFPの変異体で殆ど同一の配列である為か、トランスジェニック動物が作りにくい事が経験的に知られている{kamioka, 2012}。 | ||
近年、Clover(緑色域)とmRuby2(赤色域)がより良いFRETペアであると報告されている<ref><pubmed>22961245</pubmed></ref>。 | 近年、Clover(緑色域)とmRuby2(赤色域)がより良いFRETペアであると報告されている<ref><pubmed>22961245</pubmed></ref>。 | ||
一方、蛍光寿命イメージングとしてはドナーとしてmGFP、アクセプターとしてmRFPもしくはmCherryが用いられる。この場合、アクセプターの蛍光強度は問題ではないので蛍光を発しないREACh, darkVenus, superREAChなども用いられる<ref name=ref16537489><pubmed>16537489</pubmed></ref><ref name=ref18512154 | 一方、蛍光寿命イメージングとしてはドナーとしてmGFP、アクセプターとしてmRFPもしくはmCherryが用いられる。この場合、アクセプターの蛍光強度は問題ではないので蛍光を発しないREACh, darkVenus, superREAChなども用いられる<ref name=ref16537489><pubmed>16537489</pubmed></ref><ref name=ref18512154><pubmed>18512154</pubmed></ref><ref name=ref18302935><pubmed>18302935</pubmed></ref>。 | ||
==神経科学分野への応用例 == | ==神経科学分野への応用例 == | ||
1997年、宮脇らによって、CFPおよびYFPを利用した、細胞内[[カルシウム]] プローブ、カメレオンが開発され<ref | 1997年、宮脇らによって、CFPおよびYFPを利用した、細胞内[[カルシウム]] プローブ、カメレオンが開発され<ref><pubmed>9278050</pubmed></ref>、さらに、cAMP<ref><pubmed>10620803</pubmed></ref>, cGMP<ref><pubmed>11140757</pubmed></ref>、 リン酸化<ref><pubmed>11752449</pubmed></ref>を初めとした主要な細胞内シグナル伝達分子のFRETプローブが次々と作製され、分子のリアルタイムな活性および局在の活性の解明に大きく貢献した。 | ||
2000年初期に記憶の形成に必須なシグナル分子、Ca<sup>2+</sup>/カルモデュリン依存性タンパク質キナーゼII (CaMKII)の活性化を評価するためのFRETプローブ、 Camuiが開発された<ref | 2000年初期に記憶の形成に必須なシグナル分子、Ca<sup>2+</sup>/カルモデュリン依存性タンパク質キナーゼII (CaMKII)の活性化を評価するためのFRETプローブ、 Camuiが開発された<ref><pubmed>15788767</pubmed></ref>。CaMKIIはそれまでは、一旦活性化されたらその活性が自己リン酸化により持続する事で、長期に亘る記憶に必要なシナプス反応の増強を維持すると考えられてきたが、実際にはCaMKIIの活性化は一過性である事が示された<ref name=ref19295602><pubmed>19295602</pubmed></ref>。 | ||
一方、[[樹状突起]][[スパイン]])の形態を制御する[[アクチン]]の重合を可視化するためのFRETプローブが開発され、アクチンの重合が長期増強現象に伴い引き起こされる事、またそれが長期間維持される事が示された<ref | 一方、[[樹状突起]][[スパイン]])の形態を制御する[[アクチン]]の重合を可視化するためのFRETプローブが開発され、アクチンの重合が長期増強現象に伴い引き起こされる事、またそれが長期間維持される事が示された<ref><pubmed>15361876</pubmed></ref>。その調節の上流にある[[Rho族低分子量Gタンパク質]][[Cdc42]]、[[RhoA]]の活性も同様に維持される事が判った <ref><pubmed>19295602</pubmed></ref><ref><pubmed>18556515</pubmed></ref><ref><pubmed>21423166</pubmed></ref>。 | ||
個体においてもFRET測定法が導入されている。神経回路ネットワークにおける[[シナプス]]の役割を解明する目的で、[[フェレット]]の[[大脳皮質]][[視覚野]]にCaMKIIプローブを発現し、[[片眼剥奪]]によって、神経回路ネットワークに変化を起こした時のCaMKIIの活性化の変化を観測している<ref><pubmed>22160721</pubmed></ref>。また、神経活動をモニターする膜電位プローブを開発し[[マウス]]の[[洞毛]] | 個体においてもFRET測定法が導入されている。神経回路ネットワークにおける[[シナプス]]の役割を解明する目的で、[[フェレット]]の[[大脳皮質]][[視覚野]]にCaMKIIプローブを発現し、[[片眼剥奪]]によって、神経回路ネットワークに変化を起こした時のCaMKIIの活性化の変化を観測している<ref><pubmed>22160721</pubmed></ref>。また、神経活動をモニターする膜電位プローブを開発し[[マウス]]の[[洞毛]]刺激の投射先である[[体性感覚野]][[バレル皮質]]での入力特異的な神経の活性化を観察している<ref><pubmed>20622860</pubmed></ref>。 | ||
病態との関係では、神経細胞内のカルシウム濃度を測定するために、[[オレゴングリーンBAPTA]]の蛍光寿命の変化から、カルシウム濃度を測定し、[[アストロサイト]]でのカルシウム濃度が、[[アルツハイマー病]]モデル[[マウス]]と正常マウスで異なることが報告されている<ref><pubmed>19251629</pubmed></ref>。 | 病態との関係では、神経細胞内のカルシウム濃度を測定するために、[[オレゴングリーンBAPTA]]の蛍光寿命の変化から、カルシウム濃度を測定し、[[アストロサイト]]でのカルシウム濃度が、[[アルツハイマー病]]モデル[[マウス]]と正常マウスで異なることが報告されている<ref><pubmed>19251629</pubmed></ref>。 | ||
Homo-FRETも応用されている。CaMKIIは12量体を形成しているが、異方性の変化を基に、その構造中に二量体の単位が存在し、活性化に伴う二量体同士の位置関係が変化することが明らかにされている<ref><pubmed>19339497</pubmed></ref>。 | Homo-FRETも応用されている。CaMKIIは12量体を形成しているが、異方性の変化を基に、その構造中に二量体の単位が存在し、活性化に伴う二量体同士の位置関係が変化することが明らかにされている<ref><pubmed>19339497</pubmed></ref>。 | ||
== 将来展望 == | == 将来展望 == | ||
| 379行目: | 368行目: | ||
*[http://www.lif.kyoto-u.ac.jp/labs/fret/phogemon/index.htm Phogemon Project] 京都大学医学部 松田道行よるFRETセンサー開発とイメージング方法の解説。文字化けする時にはブラウザーの設定をShift-JISにするよい。 | *[http://www.lif.kyoto-u.ac.jp/labs/fret/phogemon/index.htm Phogemon Project] 京都大学医学部 松田道行よるFRETセンサー開発とイメージング方法の解説。文字化けする時にはブラウザーの設定をShift-JISにするよい。 | ||
*[http://www.lif.kyoto-u.ac.jp/labs/fret/e-phogemon/index.htm | *[http://www.lif.kyoto-u.ac.jp/labs/fret/e-phogemon/index.htm] 同英語版。 | ||
== 参考文献 == | == 参考文献 == | ||
<references /> | <references /> | ||
2013年11月16日 (土) 05:29時点における版
上田 善文
金沢医科大学 血液免疫内科学
林 康紀
独立行政法人理化学研究所 脳科学総合研究センター
DOI XXXX/XXXX 原稿受付日:2013年9月18日 原稿完成日:2013年月日
担当編集委員:柚崎 通介(慶應義塾大学 医学部生理学)
英:Förster resonance energy transfer 英略称:FRET 独:Förster-Resonanzenergietransfer 仏:transfert d'énergie entre molécules fluorescentes
二つの蛍光分子がごく近接して存在する場合、一つの蛍光分子からもう一つの蛍光分子へエネルギーが移行する。これをFörster共鳴エネルギー移動(FRET)という[1]。FRETの効率は2つの蛍光分子のスペクトルの重なりの大きさ、距離と角度により左右されるため、FRETを測定する事により蛍光分子間の空間配置を間接的に測定する事が可能である。特に近年のGFPならびにその類縁タンパク質を用いた遺伝子にコードされるFRETプローブが作成され、タンパク質相互作用、生化学反応や細胞内シグナル伝達を可視化する事が出来るようになった。
Förster共鳴エネルギー移動とは
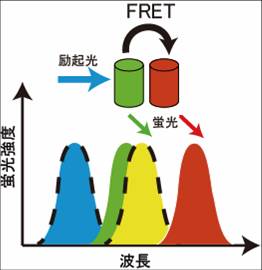
ドナーの蛍光スペクトルとアクセプターの励起スペクトルに重なりがあるときに、FRETが起きる。黒破線で囲まれたスペクトルは、励起スペクトルを示す。緑色のタンパク質がドナー、赤色タンパク質がアクセプターを示す。
二つの蛍光分子がごく近接して存在する場合、一つの蛍光分子からもう一つの蛍光分子へ、エネルギーが移行する事が知られている(図1)。この現象は、1946年Theodor Försterによって報告されたことから、Förster共鳴エネルギー移動(FRET)という[2][3]。かつてFRETは、fluorescence resonance energy transferの略称として用いたが、実際には蛍光を伴わないエネルギー移動であることから、現在ではFörster resonance energy transferと呼ぶ事がIUPACにより推奨されている。
かつては生細胞にてFRETを検出するのは、非常に煩雑であった。プローブとなるタンパク質を精製、化学的に蛍光色素でラベルし、細胞に導入するという操作が必要であり、生物学分野での応用はきわめて限定されたものであった。しかし、GFPとその変異体、類縁タンパク質の発見により今日においては様々な分野において、多くの蛍光タンパク質を基にした、完全に遺伝子によってコードされるFRETプローブが使用されている。
FRETの効率を決定する因子
蛍光分子のうち、エネルギーを受け渡す方をドナー、受け取る方をアクセプターと呼ぶ。FRETの効率はドナーとアクセプター間の次のような因子によって影響される。
- ドナーとアクセプター間の距離 r
- ドナーの発光スペクトルとアクセプターの吸収スペクトルの重なり J
- ドナーの発光双極子モーメントとアクセプターの吸収双極子モーメントとの相対分子配向κ
ドナーとアクセプター蛍光分子が決定されるとJは次のように計算される。
ここでfD(λ)はピーク値を1としたドナー発光スペクトル、 εA(λ)はアクセプターのモル吸光係数、λは波長である。
Jを用い特定のドナーとアクセプターの間にFörster距離R0が以下のように定義される。これはエネルギー移動効率が50%となるドナーとアクセプターの距離である。通常の蛍光分子の場合、5 nm程度である。
κはドナーとアクセプターの相互分子配向である。多くの場合、正確に求める事は困難であるため、しばしばκ2 =2/3 と仮定される。この値は、両方の色素が自由に回転しており、励起状態の間は等方的に配向していると考えられる場合に得られる。色素が固定されている場合や自由に回転することができないような場合、κ2 =2/3 とは仮定できない。Q0はアクセプターが無い場合のドナーの蛍光量子収率、nは媒体の屈折率(水、25 °Cの場合、1.3342)、NAはアボガドロ数である。これらの定数を当てはめると、κより前の部分は、8.786 x 1011 mol L-1 cm nm2となる[4]。
R0を用いると、FRET効率Eは次のように表す事が出来る。
Förster距離R0が大きいドナーとアクセプターの組み合わせの方が、FRET効率Eが良い。R0を大きくするためには蛍光量子収率Q0がよいドナー、モル吸光係数εA(λ)が良いアクセプター、またいずれも長波長域にあるドナーとアクセプターの組み合わせを選択する。
ドナーとアクセプターの蛍光スペクトルが変化しない状態では、ドナーとアクセプター間の距離rと配向κの変化をFRETの効率の変化として読み取る事が出来る。これを利用して、様々な細胞現象に対するプローブをデザインする事が可能である。
FRETの画像検出
FRETが起こるとドナーの蛍光強度の減少、アクセプターの蛍光の増加、ドナー蛍光寿命の減少が観察される。液体サンプルであれば蛍光分光光度計を用いる事で検出する事が可能であるが、画像として取得する場合には観察したい対象の特性を考慮しつつ、方法を選択していく。
蛍光強度比イメージング
ドナーおよびアクセプターの蛍光を取得し、ピクセルごとの蛍光強度比を計算する。FRETが起きると、ドナーの蛍光強度が減少し、アクセプターの蛍光強度が増加する。現在、最も広く使用されている手法である。タイムラプス解析も行える。
ドナー蛍光強度とFRET効率Eとは次のような関係に有る。
ただし、F'D、FDはそれぞれ、アクセプターがある場合と無い場合でのドナー蛍光強度である。つまり、FRET効率が良い程、F'D小さくなる、つまり画像が暗くなる。一方で、FRETによりアクセプター画像は明るくなるので、ドナー画像とアクセプター画像の比を計算する事で、FRETを検出する事が出来る。
データを取得、解釈する際に注意しなければいけないポイントがある。
まず、ドナーの蛍光のアクセプターチャネルへの漏れ込みであり、信号/雑音比の減少の原因となる。漏れ込みを極力抑えるには、適切なバンドパスフィルターを用いる。光量を犠牲にしても、ドナー蛍光が漏れ込まない波長を選ぶ方がFRETは特異的に検出できる。
また、蛍光画像に背景雑音がある事があるが、それがFRET変化に影響を与える。背景雑音を引き算することで、よりFRETが計算できるが、蛍光シグナルが暗いと、少しの背景雑音のぶれがシグナルを左右する。例えば細胞の周辺は暗いので背景雑音の引き算により偽陽性が出やすいので注意を要する。
2分子間FRETのイメージングでは、ドナーとアクセプターの局在の違いは偽陽性を生じる。リンカーで連結し1分子にするか、局在しているアクセプターの蛍光強度を補正することで避けることが可能である。
アクセプターブリーチング法
適切な波長の光によって、アクセプターを褪色させることでFRETを解消することができる。つまり、F'Dがアクセプターの退色によりFDと等しくなる事により、E=0となる。その為、褪色前後の画像を比較する事によりEが検出可能である。しかしながら、この手法は不可逆的であるために経時的変化を追うことは困難である。
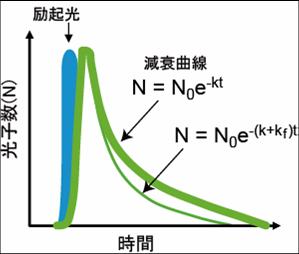
緑の太い線がFRETが起きていない時のドナー蛍光の減衰曲線。FRETを起こしたドナー蛍光の速度が加わることにより速度定数が大きくなり細い緑の線のように減衰曲線の傾斜が大きくなる。 N0は励起光によって励起された電子の数、kは励起状態にある電子が基底状態に戻る速度定数。
蛍光寿命イメージング
蛍光体が励起されると、図2に示すような減衰曲線に従って蛍光を発する。蛍光寿命は、kは励起状態にある電子が基底状態に戻る速度定数kの逆数である。蛍光として基底状態に戻る際の速度定数、熱を発して基底状態に戻るなどの無放射遷移の速度定数の和として表される。
FRETを起こしている時の速度定数kfは、以下の式で規定される。
ここで、kDはドナーの蛍光の速度定数である。
ドナー蛍光の速度定数とFRET効率Eとは次のような関係に有る。
ここでτ'DとτDはそれぞれ、アクセプターが存在する場合と存在しない場合でのドナー蛍光寿命である。つまり、FRETが起きると、蛍光寿命が短縮する(図3)。
蛍光寿命測定法は、アクセプターの蛍光は必要ないため、蛍光強度比測定法に比べて、蛍光の漏れ込み、ドナーとアクセプターの局在の違いなどによって生じる疑陽性を回避できる。
蛍光寿命の変化を測定する方法は2つある。
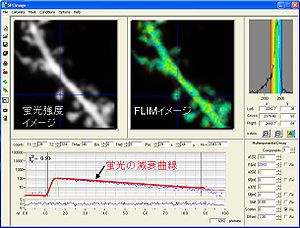
(横軸は時間、縦軸は光子数)。実際には、20秒で数千個オーダーの光子を取得する。これらの光子の発生確率分布が減衰曲線を形成し、近似曲線をフィッティングさせることで蛍光寿命を取得する。
時間ドメイン
励起光によって発生した一つ一つの光子が検出器まで届くまでの時間(数nsec)を計測することで時定数τを計算する。時間を横軸としてヒストグラムを作製することができる。通常蛍光寿命は指数関数に従い減衰していく。FRETを起こしている分子と起こしていない分子が共存する時には二重指数関数になるため、二重指数関数にフィッティングすることによって、FRETの起きている分子の割合が算出できる。得られる光子の数が少ない時には二重指数関数フィッティングは不正確になりやすい為、単に平均蛍光寿命を計算するだけで済ませる場合も有る。単一指数関数の場合は、平均蛍光寿命はτに等しくなる。
理論上は取得した蛍光を全てデータに反映させることができるが、実際には光子取得後、再び光子を取得する状態に戻るハードウェアのリセット時間(dead time)などがあり全ての光子を取得する事は出来ない。また、秒単位の経時変化を追うためには、低解像度で画像取得されているのが現状で有り、多数のピクセルから蛍光寿命を取得するためには、処理速度の速いハードウェアが必要となる。
光源にはパルスレーザーを用いる。神経系の研究によく用いられる二光子顕微鏡に後付けする事も可能である。
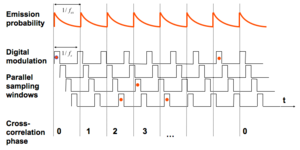
光源の強度を高周波で変調させるのと同時に、検出器も高周波で変調させる。その時に光源の周波数(fex)と検出器の周波数(fs)をずらし、そこから蛍光寿命を計算により求める。
周波数ドメイン
光源の強度を高周波で変調させるのと同時に、検出器も高周波で変調させる。その時に光源と検出器の周波数をずらしておく(heterodyning)。多数のサイクルを繰り返す事により、間接的に蛍光寿命を計算していく。画像の取得にかかる時間がtime domainと比較して短いのが特徴である。
時間ドメインと同様に光源にはパルスレーザーや光量が高頻度にて変調できるレーザーなどが用いられる。
異方性測定
一つの蛍光団のストークスシフトが小さい場合、励起スペクトルと蛍光スペクトルの重なりが大きいような蛍光団では、同一の蛍光団同士で、Homo-FRETが生じる。Homo-FRETは、蛍光強度および蛍光寿命は変化しないが、異方性が変わる。この原理を用いて分子同士のクラスターの度合いなどに応用されている。
プローブのデザイン

GFPとその変異体、類縁タンパク質の発見により今日においては様々な細胞生物学の分野において、多くの蛍光タンパク質を基にしたFRETプローブが使用されている。
これらのプローブを分類すると、以下のように分類される(表1、図5)。
プローブの分解に伴うFRETの変化を検出するプローブ
この原理は、FRETプローブの最も初期に導入されたデザインである(図5A)。プロテアーゼによって分解される配列の両端にドナーとアクセプターを連結する。プロテアーゼによって、この配列が分解されるとドナーとアクセプターの間に起きていたFRETが解消されることによって、プロテアーゼの活性を評価する。例として、第X因子、カスパーゼなどのプロテアーゼ活性のプローブが挙げられる[5][6][7][8][9]。このプローブのデザインの短所としては、反応が不可逆的であるために、一つの実験系で何度も測定することが困難であることである。
二分子間相互作用を利用したFRETプローブ
興味のあるタンパク質同士の相互作用を測定する際に、この原理が用いられる。一方にドナー、他方にアクセプターを連結する。タンパク質同士が結合していないときにはFRETは起きていないが、結合することによってFRETを生じる(図5B)。
距離のファクターを生かせるために、比較的大きなシグナルが得られる一方、内在性のタンパク質が反応に関与するために、その分FRET応答が減少する。ドナーとアクセプターの発現量の差によるFRETの応答の変化も問題になる。特に、アクセプターと結合しないドナーが多量に存在するとFRET応答が小さくなる。一般にアクセプターが多い系が、使用に適している。
タンパク質相互作用を測定する事により間接的にシグナル伝達系の活性化も測定する事が可能である。例えば、低分子量Gタンパク質が活性化に伴い、エフェクタータンパク質と相互作用が起こる事を利用し、低分子量Gタンパク質活性を測定する事が可能である。
また、タンパク質相互作用はポリマーでも良い。これを利用してアクチンの重合状態の測定にも用いられた[10](図5C)。
一分子内FRETプローブ
一分子内にドナーとアクセプターを連結し、これらの配向および距離の変化を利用する。2分子間FRETに生じるような発現量の違いやドナーとアクセプターの局在の変化によって生じる疑似陽性を考慮する必要がない。さらに活性に伴うタンパク質の構造変化などを利用するために、比較的容易に応答するプローブが作製できるが、ドナーとアクセプターを適切な位置に配置するなどの検討が必要である。
タンパク質の構造変化
興味のあるタンパク質が、活性化の際に構造変化を誘起することが知られている場合には構造変化を利用することができる(図5D)。タンパク質のC末およびN末にドナーおよびアクセプターを連結する。あるいは構造変化が起こすドメインの方が、FRETは起こりやすい可能性もある。この手法は、CaMKII[11]、カルシニューリン[12]、raf[13][14]、膜電位測定[15]、などに用いられている。
タンパク質結合に伴う構造変化
ある種のタンパク質は活性化、不活性化に伴い、下流のタンパク質と結合する。このような相互作用を利用してタンパク質の活性化、不活性化を測定することができる。
例えばカルシウムFRETプローブ、カメレオンはこの原理を利用している[16]。 この場合、ドナー、カルモジュリン、カルモジュリン結合配列であるM13ペプチド、アクセプターを1つの分子に融合する。カルモジュリンがCa2+と結合すると、M13ペプチドと結合し、蛍光強度が変化する。
また、低分子量Gタンパク質の活性化プローブは、低分子量Gタンパク質、シグナル伝達下流の結合タンパク質の結合ドメインをドナーとアクセプターで挟んだ形状をしている(図5E)。低分子Gタンパク質がGDPからGTP結合型になり活性化すると、結合ドメインと相互作用によりFRETが生じる[17]。
共有結合修飾によって生じる構造変化
このプローブは、ドナー、アクセプター、共有結合修飾を受けるドメイン、これを認識するドメインからなる。プローブが共有結合修飾を受けると、認識するドメインが結合し、ドナーとアクセプターの距離が縮まりFRETが起きる(図5F)。
このプローブは、キナーゼの活性化を測定するために使用される[18][19]。この場合、キナーゼの基質となるタンパク質に14-3-3タンパク質のようなリン酸化タンパク質を認識するドメインを融合し、その結合に伴うタンパク質構造変化を、両端に結合させたドナーとアクセプター間のFRETで測定する。
生体膜上の小分子
このプローブは、主に、脂質分子に応用されてきた(図5G)。ドナー、脂質結合ドメイン、アクセプターがヘリックス構造で連結され、グリシン-グリシン配列をその途中に導入することで、そこを中心に一方の蛍光タンパク質が回転することができる。膜結合ドメインを用いて、プローブを結合させる。脂質分子が増えた際に、脂質結合ドメインが脂質分子を認識し、構造変化が起き、ドナーとアクセプターの距離が縮まりFRETが生じる。ジアシルグリセロール[20]、イノシトールリン脂質群[21][22][23][24]を測定するために用いられている。
| 分類 | 対象 | プローブ名 | 年 | プローブデザイン | 参考文献 |
| 生体内小分子 | カルシウム | Cameleon | 1997 | E | [25] |
| サイクリックGMP (cGMP) | CGY, Cygnet, pGES-DE2, cGi | 2000, 2001, 2006 | 3-1 | [26][27][28][29] | |
| サイクリックAMP (cAMP) | Epac | 2000,2004 | 2, 3-1 | [30][31] | |
| イノシトール3リン酸 (IP3) | LIBRA, Fretino, FIRE | 2004, 2005, 2006 | 3-1 | [32][33][34] | |
| 一酸化窒素 (NO) | NOA-1, Piccell | 2005, 2006 | 3-1 | [35][36] | |
| アデノシン3リン酸 (ATP) | A Team 1.03-nD/nA | 2012 | 3-1 | [37] | |
| エストロゲン | SCCoR | 2004 | 3-3 | [38] | |
| アンドロゲン | Ficaro | 2006 | 3-3 | [39] | |
| グルココルチコイド受容体リガンド | GLUCOCOR | 2007 | 3-3 | [40][41] | |
| 神経成長因子 | ECaus | 2008 | 3-3 | [42] | |
| 核内受容体 | conpro | 2007 | 3-2 | [43] | |
| O-N-アセチルグルコサミン (O-GlcNAc) | 2006 | 3-3 | [44] | ||
| ビタミンA (レチノイン酸) | GEPRAS | 2013 | 3-1 | [45] | |
| モリブデン酸 | MolyProbe | 2013 | 3-1 | [46] | |
| グルタミン酸 | FLIPE | 2005 | 3-1 | [47] | |
| Zn2+ | eCALWY-1 | 2009 | 2 | [48] | |
| Cl- | Clomeleon | 2000 | その他 | [49] | |
| 水素イオン濃度 (pH) | GFpH, YFpH | 2001 | その他 | [50] | |
| グルコース | FLIPglu | 2003 | 3-1 | [51] | |
| マルトース | FLIPmal | 2002 | 3-1 | [52] | |
| リボース | FLIPrib | 2003 | 3-1 | [53] | |
| タンパク質リン酸化酵素 | カルシウム/カルモジュリン依存性タンパク質リン酸化酵素II (CaMKII) | Camui α, green-Camui α, Camk2a reporter | 2005, 2009, 2011, 2013 | D | [54][55][56][57] |
| Src | Srcus | 2005, 2005, 2007 | 3-3 | [58][59][60] | |
| Ca2+/リン脂質依存性タンパク質リン酸化酵素 (Cキナーゼ, プロテインキナーゼC, PKC) | CKAR, CY-PKCdelta | 2003, 2005 | 3-3, 3-1 | [61][62] | |
| タンパク質リン酸化酵素D (Dキナーゼ, プロテインキナーゼD, PKD) | DKAR | 2007 | 3-3 | [63] | |
| cAMP依存性タンパク質リン酸化酵素 (Aキナーゼ, プロテインキナーゼA, PKA) | ART, AKAR | 2000, 2001 | 3-3 | [64][65] | |
| Abl | Picchu | 2001 | E | [58] | |
| Bcr-Abl | Bcr-Abl activity sensor | 2010 | E | [66] | |
| c-Raf | Prin-cRaf | 2005 | 3-1 | [67] | |
| P21 protein-activated kinase 1 (PAK1) | Pakabi | 2009 | 3-1 | [68] | |
| B-raf | Prin-Braf | 2006 | 3-1 | [69] | |
| Zeta-chain-associated protein kinase 70 (ZAP-70) | ROZA | 2008 | 3-3 | [70] | |
| Akt | Aktus, BKAR, Akind | 2003, 2005, 2007 | 3-3 | [71][72][73] | |
| 細胞外シグナル調節キナーゼ (Extracellular signal-regulated kinases, ERK) | Miu2, Erkus, EKAR | 2006, 2007, 2008 | 3-1,3-3,3-3 | [74][75][76] | |
| インシュリン受容体 | Phocus | 2002 | 3-3 | [77] | |
| 上皮成長因子受容体 (EGFR) | 2001 | 3-3 | [58] | ||
| Ataxia telangiectasia mutated (ATM) | 2007 | 3-3 | [78] | ||
| オーロラBキナーゼ | 2008 | 3-3 | [79] | ||
| サイクリンB1-サイクリン依存性キナーゼ1 | 2010 | 3-3 | [80] | ||
| ミオシン軽鎖キナーゼ | MLCK-FIP | 2002 | 3-1 | [81] | |
| c-Jun N端末キナーゼ (JNK) | JNKAR1, JUNKAR1EV | 2010, 2011 | 3-3 | [82][83] | |
| リボソームS6キナーゼ (RSK, S6K) | Eevee-RSK, Eevee-S6K | 2011 | 3-3 | [83] | |
| 接着斑キナーゼ (Focal Adhesion Kinase, FAK) | CYFAK413, FERM-sensor | 2008, 2009 | 2, 3-1 | [84][85] | |
| Polo様キナーゼ1 (polo-like kinase1, PLK1) | 2008 | 3-3 | [86] | ||
| ストレス活性化プロテインキナーゼ3 (Stress-Activated Protein Kinase Kinase Kinase, stress-activated protein kinase 3, SAP3K) | 2009 | 3-3 | [87] | ||
| 細胞死関連タンパク質キナーゼ (death associated protein kinase 1, DAPK1) | DAPK1(334)-F40 | 2011 | 3-1 | [57] | |
| ホスファターゼ | カルシニューリン | CaNAR1 | 2008, 2013 | 3-1 | [56][88] |
| 低分子量Gタンパク質 | Ras | Raichu-Ras, Fras | 2001, 2006 | B, E | [89][90] |
| Rap | Raichu-Rap | 2001 | E | [90] | |
| Rac | Raichu-Rac1 | 2004 | E | [91] | |
| Rab5 | Raichu-Rab5 | 2008 | E | [92] | |
| Rho | Raichu-RhoA | 2003, 2011 | B, E | [93][94] | |
| Cdc42 | Raichu-cdc42 | 2004, 2011 | B, E | [91][93] | |
| Ral | Raichu-Ral | 2004 | 3-3 | [95] | |
| TC10 | Raichu-TC10 | 2006 | 3-2 | [96] | |
| シグナル伝達 | RCC1 (Ranに対するGDP, GTP交換因子) | CFP-RCC1-YFP | 2008 | 3-1 | [97] |
| CrkIIリン酸化 | Picchu | 2001 | 3-1 | [98] | |
| Wiskott-Aldrich syndrome-like (WASL, neuronal Wiskott–Aldrich syndrome protein, N-WASP) | Stinger | 2004 | 3-1 | [99][100] | |
| アドレナリン受容体 | α2AAR-cam | 2003 | 3-1 | [101] | |
| 副甲状腺ホルモン受容体 | PTHR-cam | 2003 | 3-1 | [101] | |
| 細胞膜カルシウムポンプ | BFP-PMCA-GFP | 2007 | 3-1 | [102] | |
| アセチル化 | ヒストンアセチル化 | Histac | 2004, 2009 | 3-3 | [103][104] |
| 脂質 | ホスファチジルイノシトール-3,4,5-三リン酸 (PIP3) | Fllip, FLIMPA | 2003 | G | [105] |
| ホスファチジルイノシトール-4,5-二リン酸 (PI(4,5)P2) | Pippi-PI(4,5)P2 | 2008 | G | [106] | |
| ホスファチジルイノシトール-3,4-二リン酸 (PI(3,4)P2) | Pippi-PI(3,4)P2 | 2008 | G | [106] | |
| ホスファチジルイノシトール-4-リン酸 (PI4P) | Pippi-PI(4)P | 2008 | G | [106] | |
| ホスファチジン酸 | Pii | 2010 | G | [107] | |
| ジアシルグリセロール (DAG) | Daglas, DIGDA | 2006, 2008 | G | [106][108] | |
| タンパク質相互作用 | アクチン | 2004, 2008 | C | [109][110] | |
| 3-ホスホイノシチド依存性プロテインキナーゼ1(phosphoinositide-dependent protein kinase 1, PDK1)-タンパク質キナーゼB (protein kinase B, PKB, Akt)相互作用 | 2007 | 2 | [73] | ||
| タンパク質チロシン脱リン酸化酵素1B (protein tyrosine phosphatase 1B, PTP 1B)-受容体型チロシンキナーゼ (receptor tyrosine kinases, RTKs)相互作用 | 2002 | 2 | [111] | ||
| 乳癌耐性タンパク質 (breast cancer resistance protein, BCRP)/ATP結合カセット輸送体 (ATP-binding cassette sub-family G member, ABCG)相互作用 | 2010 | 2 | [112] | ||
| コフィリン-アクチン相互作用 | 2008 | B | [113] | ||
| 第10染色体ホスファターゼ・テンシン・ホモログ (Phosphatase and tensin homolog deleted from chromosome 10, PTEN)-ミオシンV相互作用 | 2009 | 2 | [114] | ||
| プロテアーゼ | カスパーゼ-3 | EGFP-DEVD-EBFP | 1998 | A | [115] |
| カスパーゼ-8 | CFP-c3-YFP-c6-mRFP | 2002 | A | [116] | |
| カスパーゼ-9 | SCAT9 | 2011 | A | [117] | |
| カスパーゼ-7 | VDEVDc | 2006 | A | [118] | |
| マトリックスメタロプロテアーゼ (MMP) | YFP-MSS-CFPdisplay, MTI-MMP-FRET biosensor | 2007, 2008 | A | [119][120] | |
| 第Xa因子 | 1996 | A | [121] | ||
| カルパイン活性 | pYSCS | 2000 | A | [122] | |
| プレセニリン | GFP-PSI-RFP | 2009 | 3-1 | [123] | |
| その他 | 細胞膜張力センサー | stFRET | 2008 | 3-1 | [124] |
| 膜電位 | VSFP, Mermaid, ArcLight, VSFP-Butterfly | 2001, 2008, 2012 | D | [125][126][127][128] | |
| ミオシンII | GSldCB | 1998, 2006 | 3-1 | [129][130] | |
| ヒト免疫不全ウイルスRevタンパク質 | YRGnC-11ad | 2005 | 3-1 | [131] | |
| 酸化還元 | Redoxfluor, Gaskins | 2010, 2011 | 3-1 | [132][133] |
プローブデザインA-Gは図4に対応している。京都大学医学部松田道行によるPhogemon Project などを参考に作成。
蛍光色素の選択
現在多くの場合GFPあるいはその関連タンパク質が用いられている。
蛍光強度比イメージングの場合は、GFPの色彩変異体であるシアン色蛍光タンパク質CFPと黄色蛍光タンパク質YFPのFRETペアがよく用いられている。CFPの中でも、Ceruleanが明るい蛍光を示すためこれを用いるべきである。YFPの変異体の中では、Venus、Ypetがよい。いずれも若干の凝集傾向が有り、これはタンパク質表面にある三つのアラニン残基のメチル基によるものとされており、それを変異させたmonomeric GFP (A206K変異体)で凝集を避ける事が出来る[134][135]。一方で、プロテアーゼプローブなどでは、FRETダイナミックレンジが改善するという報告もある[136] [137]。CFPとYFPはいずれもGFPの変異体で殆ど同一の配列である為か、トランスジェニック動物が作りにくい事が経験的に知られている{kamioka, 2012}。
近年、Clover(緑色域)とmRuby2(赤色域)がより良いFRETペアであると報告されている[138]。
一方、蛍光寿命イメージングとしてはドナーとしてmGFP、アクセプターとしてmRFPもしくはmCherryが用いられる。この場合、アクセプターの蛍光強度は問題ではないので蛍光を発しないREACh, darkVenus, superREAChなども用いられる[139][135][140]。
神経科学分野への応用例
1997年、宮脇らによって、CFPおよびYFPを利用した、細胞内カルシウム プローブ、カメレオンが開発され[141]、さらに、cAMP[142], cGMP[143]、 リン酸化[144]を初めとした主要な細胞内シグナル伝達分子のFRETプローブが次々と作製され、分子のリアルタイムな活性および局在の活性の解明に大きく貢献した。
2000年初期に記憶の形成に必須なシグナル分子、Ca2+/カルモデュリン依存性タンパク質キナーゼII (CaMKII)の活性化を評価するためのFRETプローブ、 Camuiが開発された[145]。CaMKIIはそれまでは、一旦活性化されたらその活性が自己リン酸化により持続する事で、長期に亘る記憶に必要なシナプス反応の増強を維持すると考えられてきたが、実際にはCaMKIIの活性化は一過性である事が示された[146]。
一方、樹状突起スパイン)の形態を制御するアクチンの重合を可視化するためのFRETプローブが開発され、アクチンの重合が長期増強現象に伴い引き起こされる事、またそれが長期間維持される事が示された[147]。その調節の上流にあるRho族低分子量Gタンパク質Cdc42、RhoAの活性も同様に維持される事が判った [148][149][150]。
個体においてもFRET測定法が導入されている。神経回路ネットワークにおけるシナプスの役割を解明する目的で、フェレットの大脳皮質視覚野にCaMKIIプローブを発現し、片眼剥奪によって、神経回路ネットワークに変化を起こした時のCaMKIIの活性化の変化を観測している[151]。また、神経活動をモニターする膜電位プローブを開発しマウスの洞毛刺激の投射先である体性感覚野バレル皮質での入力特異的な神経の活性化を観察している[152]。
病態との関係では、神経細胞内のカルシウム濃度を測定するために、オレゴングリーンBAPTAの蛍光寿命の変化から、カルシウム濃度を測定し、アストロサイトでのカルシウム濃度が、アルツハイマー病モデルマウスと正常マウスで異なることが報告されている[153]。
Homo-FRETも応用されている。CaMKIIは12量体を形成しているが、異方性の変化を基に、その構造中に二量体の単位が存在し、活性化に伴う二量体同士の位置関係が変化することが明らかにされている[154]。
将来展望
脳研究は、生動物の脳の神経細胞の活動を、広範囲で、より深部で観察したり、逆に神経細胞内の超微細構造を観察する方向に移るであろう。現在、FRETを基にしたin vivoイメージングは、応答の低さ、蛍光の弱さなどの難点はあるものの、蛍光タンパク質の蛍光強度や顕微鏡の性能の改良は日進月歩であり改善されていくであろう。また、神経活動に必要なシグナル伝達を同時に観察するために、マルチカラーイメージングの試みもなされるであろう。その際には、2つの波長を必要とする蛍光強度比変化を基にするFRET測定よりも、蛍光寿命イメージングが適している。
外部リンク
- Phogemon Project 京都大学医学部 松田道行よるFRETセンサー開発とイメージング方法の解説。文字化けする時にはブラウザーの設定をShift-JISにするよい。
- [1] 同英語版。
参考文献
- ↑
Förster, T. (2012).
Energy migration and fluorescence. 1946. Journal of biomedical optics, 17(1), 011002. [PubMed:22352636] [WorldCat] [DOI] - ↑ Förster, T.
Energiewanderung und Fluorescenz
Naturwissenscaft. 1946, 33:166–175 - ↑
Förster, T. (2012).
Energy migration and fluorescence. 1946. Journal of biomedical optics, 17(1), 011002. [PubMed:22352636] [WorldCat] [DOI] - ↑
Patterson, G.H., Piston, D.W., & Barisas, B.G. (2000).
Förster distances between green fluorescent protein pairs. Analytical biochemistry, 284(2), 438-40. [PubMed:10964438] [WorldCat] [DOI] 但しこの論文にはミスプリが有り、p. 439でκ2 とすべき所を、κとしている。 - ↑
Mitra, R.D., Silva, C.M., & Youvan, D.C. (1996).
Fluorescence resonance energy transfer between blue-emitting and red-shifted excitation derivatives of the green fluorescent protein. Gene, 173(1 Spec No), 13-7. [PubMed:8707050] [WorldCat] [DOI] - ↑
Xu, X., Gerard, A.L., Huang, B.C., Anderson, D.C., Payan, D.G., & Luo, Y. (1998).
Detection of programmed cell death using fluorescence energy transfer. Nucleic acids research, 26(8), 2034-5. [PubMed:9518501] [PMC] [WorldCat] [DOI] - ↑
Onuki, R., Nagasaki, A., Kawasaki, H., Baba, T., Uyeda, T.Q., & Taira, K. (2002).
Confirmation by FRET in individual living cells of the absence of significant amyloid beta -mediated caspase 8 activation. Proceedings of the National Academy of Sciences of the United States of America, 99(23), 14716-21. [PubMed:12409609] [PMC] [WorldCat] [DOI] - ↑
Joseph, J., Seervi, M., Sobhan, P.K., & Retnabai, S.T. (2011).
High throughput ratio imaging to profile caspase activity: potential application in multiparameter high content apoptosis analysis and drug screening. PloS one, 6(5), e20114. [PubMed:21637712] [PMC] [WorldCat] [DOI] - ↑
Li, I.T., Chiang, J.J., & Truong, K. (2006).
FRET evidence that an isoform of caspase-7 binds but does not cleave its substrate. Conference proceedings : ... Annual International Conference of the IEEE Engineering in Medicine and Biology Society. IEEE Engineering in Medicine and Biology Society. Annual Conference, 1, 531-4. [PubMed:17946841] [WorldCat] [DOI] - ↑
Okamoto, K., Nagai, T., Miyawaki, A., & Hayashi, Y. (2004).
Rapid and persistent modulation of actin dynamics regulates postsynaptic reorganization underlying bidirectional plasticity. Nature neuroscience, 7(10), 1104-12. [PubMed:15361876] [WorldCat] [DOI] - ↑
Takao, K., Okamoto, K., Nakagawa, T., Neve, R.L., Nagai, T., Miyawaki, A., ..., & Hayashi, Y. (2005).
Visualization of synaptic Ca2+ /calmodulin-dependent protein kinase II activity in living neurons. The Journal of neuroscience : the official journal of the Society for Neuroscience, 25(12), 3107-12. [PubMed:15788767] [PMC] [WorldCat] [DOI] - ↑
Newman, R.H., & Zhang, J. (2008).
Visualization of phosphatase activity in living cells with a FRET-based calcineurin activity sensor. Molecular bioSystems, 4(6), 496-501. [PubMed:18493642] [WorldCat] [DOI] - ↑
Terai, K., & Matsuda, M. (2005).
Ras binding opens c-Raf to expose the docking site for mitogen-activated protein kinase kinase. EMBO reports, 6(3), 251-5. [PubMed:15711535] [PMC] [WorldCat] [DOI] - ↑
Terai, K., & Matsuda, M. (2006).
The amino-terminal B-Raf-specific region mediates calcium-dependent homo- and hetero-dimerization of Raf. The EMBO journal, 25(15), 3556-64. [PubMed:16858395] [PMC] [WorldCat] [DOI] - ↑
Tsutsui, H., Karasawa, S., Okamura, Y., & Miyawaki, A. (2008).
Improving membrane voltage measurements using FRET with new fluorescent proteins. Nature methods, 5(8), 683-5. [PubMed:18622396] [WorldCat] [DOI] - ↑
Miyawaki, A., Llopis, J., Heim, R., McCaffery, J.M., Adams, J.A., Ikura, M., & Tsien, R.Y. (1997).
Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature, 388(6645), 882-7. [PubMed:9278050] [WorldCat] [DOI] - ↑
Yasuda, R., Harvey, C.D., Zhong, H., Sobczyk, A., van Aelst, L., & Svoboda, K. (2006).
Supersensitive Ras activation in dendrites and spines revealed by two-photon fluorescence lifetime imaging. Nature neuroscience, 9(2), 283-91. [PubMed:16429133] [WorldCat] [DOI] - ↑
Sato, M., Ozawa, T., Inukai, K., Asano, T., & Umezawa, Y. (2002).
Fluorescent indicators for imaging protein phosphorylation in single living cells. Nature biotechnology, 20(3), 287-94. [PubMed:11875431] [WorldCat] [DOI] - ↑
Ting, A.Y., Kain, K.H., Klemke, R.L., & Tsien, R.Y. (2001).
Genetically encoded fluorescent reporters of protein tyrosine kinase activities in living cells. Proceedings of the National Academy of Sciences of the United States of America, 98(26), 15003-8. [PubMed:11752449] [PMC] [WorldCat] [DOI] - ↑
Sato, M., Ueda, Y., & Umezawa, Y. (2006).
Imaging diacylglycerol dynamics at organelle membranes. Nature methods, 3(10), 797-9. [PubMed:16990811] [WorldCat] [DOI] - ↑
Sato, M., Ueda, Y., Takagi, T., & Umezawa, Y. (2003).
Production of PtdInsP3 at endomembranes is triggered by receptor endocytosis. Nature cell biology, 5(11), 1016-22. [PubMed:14528311] [WorldCat] [DOI] - ↑
Nishioka, T., Aoki, K., Hikake, K., Yoshizaki, H., Kiyokawa, E., & Matsuda, M. (2008).
Rapid turnover rate of phosphoinositides at the front of migrating MDCK cells. Molecular biology of the cell, 19(10), 4213-23. [PubMed:18685081] [PMC] [WorldCat] [DOI] - ↑
Nishioka, T., Aoki, K., Hikake, K., Yoshizaki, H., Kiyokawa, E., & Matsuda, M. (2008).
Rapid turnover rate of phosphoinositides at the front of migrating MDCK cells. Molecular biology of the cell, 19(10), 4213-23. [PubMed:18685081] [PMC] [WorldCat] [DOI] - ↑
Nishioka, T., Aoki, K., Hikake, K., Yoshizaki, H., Kiyokawa, E., & Matsuda, M. (2008).
Rapid turnover rate of phosphoinositides at the front of migrating MDCK cells. Molecular biology of the cell, 19(10), 4213-23. [PubMed:18685081] [PMC] [WorldCat] [DOI] - ↑
Miyawaki, A., Llopis, J., Heim, R., McCaffery, J.M., Adams, J.A., Ikura, M., & Tsien, R.Y. (1997).
Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature, 388(6645), 882-7. [PubMed:9278050] [WorldCat] [DOI] - ↑
Sato, M., Hida, N., Ozawa, T., & Umezawa, Y. (2000).
Fluorescent indicators for cyclic GMP based on cyclic GMP-dependent protein kinase Ialpha and green fluorescent proteins. Analytical chemistry, 72(24), 5918-24. [PubMed:11140757] [WorldCat] [DOI] - ↑
Honda, A., Adams, S.R., Sawyer, C.L., Lev-Ram, V., Tsien, R.Y., & Dostmann, W.R. (2001).
Spatiotemporal dynamics of guanosine 3',5'-cyclic monophosphate revealed by a genetically encoded, fluorescent indicator. Proceedings of the National Academy of Sciences of the United States of America, 98(5), 2437-42. [PubMed:11226257] [PMC] [WorldCat] [DOI] - ↑
Nikolaev, V.O., Gambaryan, S., & Lohse, M.J. (2006).
Fluorescent sensors for rapid monitoring of intracellular cGMP. Nature methods, 3(1), 23-5. [PubMed:16369548] [WorldCat] [DOI] - ↑
Thunemann, M., Wen, L., Hillenbrand, M., Vachaviolos, A., Feil, S., Ott, T., ..., & Feil, R. (2013).
Transgenic mice for cGMP imaging. Circulation research, 113(4), 365-71. [PubMed:23801067] [PMC] [WorldCat] [DOI] - ↑
Zaccolo, M., & Pozzan, T. (2002).
Discrete microdomains with high concentration of cAMP in stimulated rat neonatal cardiac myocytes. Science (New York, N.Y.), 295(5560), 1711-5. [PubMed:11872839] [WorldCat] [DOI] - ↑
Nikolaev, V.O., Bünemann, M., Hein, L., Hannawacker, A., & Lohse, M.J. (2004).
Novel single chain cAMP sensors for receptor-induced signal propagation. The Journal of biological chemistry, 279(36), 37215-8. [PubMed:15231839] [WorldCat] [DOI] - ↑
Tanimura, A., Nezu, A., Morita, T., Turner, R.J., & Tojyo, Y. (2004).
Fluorescent biosensor for quantitative real-time measurements of inositol 1,4,5-trisphosphate in single living cells. The Journal of biological chemistry, 279(37), 38095-8. [PubMed:15272011] [WorldCat] [DOI] - ↑
Sato, M., Ueda, Y., Shibuya, M., & Umezawa, Y. (2005).
Locating inositol 1,4,5-trisphosphate in the nucleus and neuronal dendrites with genetically encoded fluorescent indicators. Analytical chemistry, 77(15), 4751-8. [PubMed:16053285] [WorldCat] [DOI] - ↑
Matsu-ura, T., Michikawa, T., Inoue, T., Miyawaki, A., Yoshida, M., & Mikoshiba, K. (2006).
Cytosolic inositol 1,4,5-trisphosphate dynamics during intracellular calcium oscillations in living cells. The Journal of cell biology, 173(5), 755-65. [PubMed:16754959] [PMC] [WorldCat] [DOI] - ↑
Sato, M., Hida, N., & Umezawa, Y. (2005).
Imaging the nanomolar range of nitric oxide with an amplifier-coupled fluorescent indicator in living cells. Proceedings of the National Academy of Sciences of the United States of America, 102(41), 14515-20. [PubMed:16176986] [PMC] [WorldCat] [DOI] - ↑
Sato, M., Nakajima, T., Goto, M., & Umezawa, Y. (2006).
Cell-based indicator to visualize picomolar dynamics of nitric oxide release from living cells. Analytical chemistry, 78(24), 8175-82. [PubMed:17165805] [WorldCat] [DOI] - ↑
Imamura, H., Nhat, K.P., Togawa, H., Saito, K., Iino, R., Kato-Yamada, Y., ..., & Noji, H. (2009).
Visualization of ATP levels inside single living cells with fluorescence resonance energy transfer-based genetically encoded indicators. Proceedings of the National Academy of Sciences of the United States of America, 106(37), 15651-6. [PubMed:19720993] [PMC] [WorldCat] [DOI] - ↑
Awais, M., Sato, M., Sasaki, K., & Umezawa, Y. (2004).
A genetically encoded fluorescent indicator capable of discriminating estrogen agonists from antagonists in living cells. Analytical chemistry, 76(8), 2181-6. [PubMed:15080726] [WorldCat] [DOI] - ↑
Awais, M., Sato, M., Lee, X., & Umezawa, Y. (2006).
A fluorescent indicator to visualize activities of the androgen receptor ligands in single living cells. Angewandte Chemie (International ed. in English), 45(17), 2707-12. [PubMed:16555356] [WorldCat] [DOI] - ↑
Nishi, M., Tanaka, M., Matsuda, K., Sunaguchi, M., & Kawata, M. (2004).
Visualization of glucocorticoid receptor and mineralocorticoid receptor interactions in living cells with GFP-based fluorescence resonance energy transfer. The Journal of neuroscience : the official journal of the Society for Neuroscience, 24(21), 4918-27. [PubMed:15163683] [PMC] [WorldCat] [DOI] - ↑
Awais, M., Sato, M., & Umezawa, Y. (2007).
Optical probes to identify the glucocorticoid receptor ligands in living cells. Steroids, 72(14), 949-54. [PubMed:17897691] [WorldCat] [DOI] - ↑
Nakajima, T., Sato, M., Akaza, N., & Umezawa, Y. (2008).
Cell-based fluorescent indicator to visualize brain-derived neurotrophic factor secreted from living neurons. ACS chemical biology, 3(6), 352-8. [PubMed:18510313] [WorldCat] [DOI] - ↑
Awais, M., Sato, M., & Umezawa, Y. (2007).
Imaging of selective nuclear receptor modulator-induced conformational changes in the nuclear receptor to allow interaction with coactivator and corepressor proteins in living cells. Chembiochem : a European journal of chemical biology, 8(7), 737-43. [PubMed:17387660] [WorldCat] [DOI] - ↑
Carrillo, L.D., Krishnamoorthy, L., & Mahal, L.K. (2006).
A cellular FRET-based sensor for beta-O-GlcNAc, a dynamic carbohydrate modification involved in signaling. Journal of the American Chemical Society, 128(46), 14768-9. [PubMed:17105262] [WorldCat] [DOI] - ↑
Shimozono, S., Iimura, T., Kitaguchi, T., Higashijima, S., & Miyawaki, A. (2013).
Visualization of an endogenous retinoic acid gradient across embryonic development. Nature, 496(7445), 363-6. [PubMed:23563268] [WorldCat] [DOI] - ↑
Nakanishi, Y., Iida, S., Ueoka-Nakanishi, H., Niimi, T., Tomioka, R., & Maeshima, M. (2013).
Exploring dynamics of molybdate in living animal cells by a genetically encoded FRET nanosensor. PloS one, 8(3), e58175. [PubMed:23472155] [PMC] [WorldCat] [DOI] - ↑
Okumoto, S., Looger, L.L., Micheva, K.D., Reimer, R.J., Smith, S.J., & Frommer, W.B. (2005).
Detection of glutamate release from neurons by genetically encoded surface-displayed FRET nanosensors. Proceedings of the National Academy of Sciences of the United States of America, 102(24), 8740-5. [PubMed:15939876] [PMC] [WorldCat] [DOI] - ↑
Vinkenborg, J.L., Nicolson, T.J., Bellomo, E.A., Koay, M.S., Rutter, G.A., & Merkx, M. (2009).
Genetically encoded FRET sensors to monitor intracellular Zn2+ homeostasis. Nature methods, 6(10), 737-40. [PubMed:19718032] [PMC] [WorldCat] [DOI] - ↑
Kuner, T., & Augustine, G.J. (2000).
A genetically encoded ratiometric indicator for chloride: capturing chloride transients in cultured hippocampal neurons. Neuron, 27(3), 447-59. [PubMed:11055428] [WorldCat] [DOI] - ↑
Awaji, T., Hirasawa, A., Shirakawa, H., Tsujimoto, G., & Miyazaki, S. (2001).
Novel green fluorescent protein-based ratiometric indicators for monitoring pH in defined intracellular microdomains. Biochemical and biophysical research communications, 289(2), 457-62. [PubMed:11716495] [WorldCat] [DOI] - ↑
Fehr, M., Lalonde, S., Lager, I., Wolff, M.W., & Frommer, W.B. (2003).
In vivo imaging of the dynamics of glucose uptake in the cytosol of COS-7 cells by fluorescent nanosensors. The Journal of biological chemistry, 278(21), 19127-33. [PubMed:12649277] [WorldCat] [DOI] - ↑
Fehr, M., Frommer, W.B., & Lalonde, S. (2002).
Visualization of maltose uptake in living yeast cells by fluorescent nanosensors. Proceedings of the National Academy of Sciences of the United States of America, 99(15), 9846-51. [PubMed:12097642] [PMC] [WorldCat] [DOI] - ↑
Lager, I., Fehr, M., Frommer, W.B., & Lalonde, S. (2003).
Development of a fluorescent nanosensor for ribose. FEBS letters, 553(1-2), 85-9. [PubMed:14550551] [WorldCat] [DOI] - ↑
Takao, K., Okamoto, K., Nakagawa, T., Neve, R.L., Nagai, T., Miyawaki, A., ..., & Hayashi, Y. (2005).
Visualization of synaptic Ca2+ /calmodulin-dependent protein kinase II activity in living neurons. The Journal of neuroscience : the official journal of the Society for Neuroscience, 25(12), 3107-12. [PubMed:15788767] [PMC] [WorldCat] [DOI] - ↑
Lee, S.J., Escobedo-Lozoya, Y., Szatmari, E.M., & Yasuda, R. (2009).
Activation of CaMKII in single dendritic spines during long-term potentiation. Nature, 458(7236), 299-304. [PubMed:19295602] [PMC] [WorldCat] [DOI] - ↑ 56.0 56.1
Fujii, H., Inoue, M., Okuno, H., Sano, Y., Takemoto-Kimura, S., Kitamura, K., ..., & Bito, H. (2013).
Nonlinear decoding and asymmetric representation of neuronal input information by CaMKIIα and calcineurin. Cell reports, 3(4), 978-87. [PubMed:23602566] [WorldCat] [DOI] - ↑ 57.0 57.1
Piljić, A., de Diego, I., Wilmanns, M., & Schultz, C. (2011).
Rapid development of genetically encoded FRET reporters. ACS chemical biology, 6(7), 685-91. [PubMed:21506563] [WorldCat] [DOI] - ↑ 58.0 58.1 58.2
Ting, A.Y., Kain, K.H., Klemke, R.L., & Tsien, R.Y. (2001).
Genetically encoded fluorescent reporters of protein tyrosine kinase activities in living cells. Proceedings of the National Academy of Sciences of the United States of America, 98(26), 15003-8. [PubMed:11752449] [PMC] [WorldCat] [DOI] - ↑
Wang, Y., Botvinick, E.L., Zhao, Y., Berns, M.W., Usami, S., Tsien, R.Y., & Chien, S. (2005).
Visualizing the mechanical activation of Src. Nature, 434(7036), 1040-5. [PubMed:15846350] [WorldCat] [DOI] - ↑
Hitosugi, T., Sasaki, K., Sato, M., Suzuki, Y., & Umezawa, Y. (2007).
Epidermal growth factor directs sex-specific steroid signaling through Src activation. The Journal of biological chemistry, 282(14), 10697-706. [PubMed:17284441] [WorldCat] [DOI] - ↑
Violin, J.D., Zhang, J., Tsien, R.Y., & Newton, A.C. (2003).
A genetically encoded fluorescent reporter reveals oscillatory phosphorylation by protein kinase C. The Journal of cell biology, 161(5), 899-909. [PubMed:12782683] [PMC] [WorldCat] [DOI] - ↑
Braun, D.C., Garfield, S.H., & Blumberg, P.M. (2005).
Analysis by fluorescence resonance energy transfer of the interaction between ligands and protein kinase Cdelta in the intact cell. The Journal of biological chemistry, 280(9), 8164-71. [PubMed:15611119] [WorldCat] [DOI] - ↑
Kunkel, M.T., Toker, A., Tsien, R.Y., & Newton, A.C. (2007).
Calcium-dependent regulation of protein kinase D revealed by a genetically encoded kinase activity reporter. The Journal of biological chemistry, 282(9), 6733-42. [PubMed:17189263] [PMC] [WorldCat] [DOI] - ↑
Nagai, Y., Miyazaki, M., Aoki, R., Zama, T., Inouye, S., Hirose, K., ..., & Hagiwara, M. (2000).
A fluorescent indicator for visualizing cAMP-induced phosphorylation in vivo. Nature biotechnology, 18(3), 313-6. [PubMed:10700148] [WorldCat] [DOI] - ↑
Zhang, J., Ma, Y., Taylor, S.S., & Tsien, R.Y. (2001).
Genetically encoded reporters of protein kinase A activity reveal impact of substrate tethering. Proceedings of the National Academy of Sciences of the United States of America, 98(26), 14997-5002. [PubMed:11752448] [PMC] [WorldCat] [DOI] - ↑
Tunceroglu, A., Matsuda, M., & Birge, R.B. (2010).
Real-time fluorescent resonance energy transfer analysis to monitor drug resistance in chronic myelogenous leukemia. Molecular cancer therapeutics, 9(11), 3065-73. [PubMed:20817824] [PMC] [WorldCat] [DOI] - ↑
Terai, K., & Matsuda, M. (2005).
Ras binding opens c-Raf to expose the docking site for mitogen-activated protein kinase kinase. EMBO reports, 6(3), 251-5. [PubMed:15711535] [PMC] [WorldCat] [DOI] - ↑
Parrini, M.C., Camonis, J., Matsuda, M., & de Gunzburg, J. (2009).
Dissecting activation of the PAK1 kinase at protrusions in living cells. The Journal of biological chemistry, 284(36), 24133-43. [PubMed:19574218] [PMC] [WorldCat] [DOI] - ↑
Terai, K., & Matsuda, M. (2006).
The amino-terminal B-Raf-specific region mediates calcium-dependent homo- and hetero-dimerization of Raf. The EMBO journal, 25(15), 3556-64. [PubMed:16858395] [PMC] [WorldCat] [DOI] - ↑
Randriamampita, C., Mouchacca, P., Malissen, B., Marguet, D., Trautmann, A., & Lellouch, A.C. (2008).
A novel ZAP-70 dependent FRET based biosensor reveals kinase activity at both the immunological synapse and the antisynapse. PloS one, 3(1), e1521. [PubMed:18231606] [PMC] [WorldCat] [DOI] - ↑
Sasaki, K., Sato, M., & Umezawa, Y. (2003).
Fluorescent indicators for Akt/protein kinase B and dynamics of Akt activity visualized in living cells. The Journal of biological chemistry, 278(33), 30945-51. [PubMed:12773546] [WorldCat] [DOI] - ↑
Kunkel, M.T., Ni, Q., Tsien, R.Y., Zhang, J., & Newton, A.C. (2005).
Spatio-temporal dynamics of protein kinase B/Akt signaling revealed by a genetically encoded fluorescent reporter. The Journal of biological chemistry, 280(7), 5581-7. [PubMed:15583002] [PMC] [WorldCat] [DOI] - ↑ 73.0 73.1
Calleja, V., Alcor, D., Laguerre, M., Park, J., Vojnovic, B., Hemmings, B.A., ..., & Larijani, B. (2007).
Intramolecular and intermolecular interactions of protein kinase B define its activation in vivo. PLoS biology, 5(4), e95. [PubMed:17407381] [PMC] [WorldCat] [DOI] - ↑
Fujioka, A., Terai, K., Itoh, R.E., Aoki, K., Nakamura, T., Kuroda, S., ..., & Matsuda, M. (2006).
Dynamics of the Ras/ERK MAPK cascade as monitored by fluorescent probes. The Journal of biological chemistry, 281(13), 8917-26. [PubMed:16418172] [WorldCat] [DOI] - ↑
Sato, M., Kawai, Y., & Umezawa, Y. (2007).
Genetically encoded fluorescent indicators to visualize protein phosphorylation by extracellular signal-regulated kinase in single living cells. Analytical chemistry, 79(6), 2570-5. [PubMed:17261026] [WorldCat] [DOI] - ↑
Harvey, C.D., Ehrhardt, A.G., Cellurale, C., Zhong, H., Yasuda, R., Davis, R.J., & Svoboda, K. (2008).
A genetically encoded fluorescent sensor of ERK activity. Proceedings of the National Academy of Sciences of the United States of America, 105(49), 19264-9. [PubMed:19033456] [PMC] [WorldCat] [DOI] - ↑
Sato, M., Ozawa, T., Inukai, K., Asano, T., & Umezawa, Y. (2002).
Fluorescent indicators for imaging protein phosphorylation in single living cells. Nature biotechnology, 20(3), 287-94. [PubMed:11875431] [WorldCat] [DOI] - ↑
Johnson, S.A., You, Z., & Hunter, T. (2007).
Monitoring ATM kinase activity in living cells. DNA repair, 6(9), 1277-84. [PubMed:17428747] [WorldCat] [DOI] - ↑
Fuller, B.G., Lampson, M.A., Foley, E.A., Rosasco-Nitcher, S., Le, K.V., Tobelmann, P., ..., & Kapoor, T.M. (2008).
Midzone activation of aurora B in anaphase produces an intracellular phosphorylation gradient. Nature, 453(7198), 1132-6. [PubMed:18463638] [PMC] [WorldCat] [DOI] - ↑
Gavet, O., & Pines, J. (2010).
Progressive activation of CyclinB1-Cdk1 coordinates entry to mitosis. Developmental cell, 18(4), 533-43. [PubMed:20412769] [PMC] [WorldCat] [DOI] - ↑
Chew, T.L., Wolf, W.A., Gallagher, P.J., Matsumura, F., & Chisholm, R.L. (2002).
A fluorescent resonant energy transfer-based biosensor reveals transient and regional myosin light chain kinase activation in lamella and cleavage furrows. The Journal of cell biology, 156(3), 543-53. [PubMed:11815633] [PMC] [WorldCat] [DOI] - ↑
Fosbrink, M., Aye-Han, N.N., Cheong, R., Levchenko, A., & Zhang, J. (2010).
Visualization of JNK activity dynamics with a genetically encoded fluorescent biosensor. Proceedings of the National Academy of Sciences of the United States of America, 107(12), 5459-64. [PubMed:20212108] [PMC] [WorldCat] [DOI] - ↑ 83.0 83.1
Komatsu, N., Aoki, K., Yamada, M., Yukinaga, H., Fujita, Y., Kamioka, Y., & Matsuda, M. (2011).
Development of an optimized backbone of FRET biosensors for kinases and GTPases. Molecular biology of the cell, 22(23), 4647-56. [PubMed:21976697] [PMC] [WorldCat] [DOI] - ↑
Cai, X., Lietha, D., Ceccarelli, D.F., Karginov, A.V., Rajfur, Z., Jacobson, K., ..., & Schaller, M.D. (2008).
Spatial and temporal regulation of focal adhesion kinase activity in living cells. Molecular and cellular biology, 28(1), 201-14. [PubMed:17967873] [PMC] [WorldCat] [DOI] - ↑
Papusheva, E., Mello de Queiroz, F., Dalous, J., Han, Y., Esposito, A., Jares-Erijmanxa, E.A., ..., & Bunt, G. (2009).
Dynamic conformational changes in the FERM domain of FAK are involved in focal-adhesion behavior during cell spreading and motility. Journal of cell science, 122(Pt 5), 656-66. [PubMed:19208768] [WorldCat] [DOI] - ↑
Macůrek, L., Lindqvist, A., Lim, D., Lampson, M.A., Klompmaker, R., Freire, R., ..., & Medema, R.H. (2008).
Polo-like kinase-1 is activated by aurora A to promote checkpoint recovery. Nature, 455(7209), 119-23. [PubMed:18615013] [WorldCat] [DOI] - ↑
Tomida, T., Takekawa, M., O'Grady, P., & Saito, H. (2009).
Stimulus-specific distinctions in spatial and temporal dynamics of stress-activated protein kinase kinase kinases revealed by a fluorescence resonance energy transfer biosensor. Molecular and cellular biology, 29(22), 6117-27. [PubMed:19737916] [PMC] [WorldCat] [DOI] - ↑
Newman, R.H., & Zhang, J. (2008).
Visualization of phosphatase activity in living cells with a FRET-based calcineurin activity sensor. Molecular bioSystems, 4(6), 496-501. [PubMed:18493642] [WorldCat] [DOI] - ↑
Yasuda, R., Harvey, C.D., Zhong, H., Sobczyk, A., van Aelst, L., & Svoboda, K. (2006).
Supersensitive Ras activation in dendrites and spines revealed by two-photon fluorescence lifetime imaging. Nature neuroscience, 9(2), 283-91. [PubMed:16429133] [WorldCat] [DOI] - ↑ 90.0 90.1
Mochizuki, N., Yamashita, S., Kurokawa, K., Ohba, Y., Nagai, T., Miyawaki, A., & Matsuda, M. (2001).
Spatio-temporal images of growth-factor-induced activation of Ras and Rap1. Nature, 411(6841), 1065-8. [PubMed:11429608] [WorldCat] [DOI] - ↑ 91.0 91.1
Aoki, K., Nakamura, T., & Matsuda, M. (2004).
Spatio-temporal regulation of Rac1 and Cdc42 activity during nerve growth factor-induced neurite outgrowth in PC12 cells. The Journal of biological chemistry, 279(1), 713-9. [PubMed:14570905] [WorldCat] [DOI] - ↑
Kitano, M., Nakaya, M., Nakamura, T., Nagata, S., & Matsuda, M. (2008).
Imaging of Rab5 activity identifies essential regulators for phagosome maturation. Nature, 453(7192), 241-5. [PubMed:18385674] [WorldCat] [DOI] - ↑ 93.0 93.1
Murakoshi, H., Wang, H., & Yasuda, R. (2011).
Local, persistent activation of Rho GTPases during plasticity of single dendritic spines. Nature, 472(7341), 100-4. [PubMed:21423166] [PMC] [WorldCat] [DOI] - ↑
Yoshizaki, H., Ohba, Y., Kurokawa, K., Itoh, R.E., Nakamura, T., Mochizuki, N., ..., & Matsuda, M. (2003).
Activity of Rho-family GTPases during cell division as visualized with FRET-based probes. The Journal of cell biology, 162(2), 223-32. [PubMed:12860967] [PMC] [WorldCat] [DOI] - ↑
Takaya, A., Ohba, Y., Kurokawa, K., & Matsuda, M. (2004).
RalA activation at nascent lamellipodia of epidermal growth factor-stimulated Cos7 cells and migrating Madin-Darby canine kidney cells. Molecular biology of the cell, 15(6), 2549-57. [PubMed:15034142] [PMC] [WorldCat] [DOI] - ↑
Kawase, K., Nakamura, T., Takaya, A., Aoki, K., Namikawa, K., Kiyama, H., ..., & Matsuda, M. (2006).
GTP hydrolysis by the Rho family GTPase TC10 promotes exocytic vesicle fusion. Developmental cell, 11(3), 411-21. [PubMed:16950130] [WorldCat] [DOI] - ↑
Hao, Y., & Macara, I.G. (2008).
Regulation of chromatin binding by a conformational switch in the tail of the Ran exchange factor RCC1. The Journal of cell biology, 182(5), 827-36. [PubMed:18762580] [PMC] [WorldCat] [DOI] - ↑
Kurokawa, K., Mochizuki, N., Ohba, Y., Mizuno, H., Miyawaki, A., & Matsuda, M. (2001).
A pair of fluorescent resonance energy transfer-based probes for tyrosine phosphorylation of the CrkII adaptor protein in vivo. The Journal of biological chemistry, 276(33), 31305-10. [PubMed:11406630] [WorldCat] [DOI] - ↑
Lorenz, M., Yamaguchi, H., Wang, Y., Singer, R.H., & Condeelis, J. (2004).
Imaging sites of N-wasp activity in lamellipodia and invadopodia of carcinoma cells. Current biology : CB, 14(8), 697-703. [PubMed:15084285] [WorldCat] [DOI] - ↑
Ward, M.E., Wu, J.Y., & Rao, Y. (2004).
Visualization of spatially and temporally regulated N-WASP activity during cytoskeletal reorganization in living cells. Proceedings of the National Academy of Sciences of the United States of America, 101(4), 970-4. [PubMed:14732696] [PMC] [WorldCat] [DOI] - ↑ 101.0 101.1
Vilardaga, J.P., Bünemann, M., Krasel, C., Castro, M., & Lohse, M.J. (2003).
Measurement of the millisecond activation switch of G protein-coupled receptors in living cells. Nature biotechnology, 21(7), 807-12. [PubMed:12808462] [WorldCat] [DOI] - ↑
Corradi, G.R., & Adamo, H.P. (2007).
Intramolecular fluorescence resonance energy transfer between fused autofluorescent proteins reveals rearrangements of the N- and C-terminal segments of the plasma membrane Ca2+ pump involved in the activation. The Journal of biological chemistry, 282(49), 35440-8. [PubMed:17901055] [WorldCat] [DOI] - ↑
Lin, C.W., Jao, C.Y., & Ting, A.Y. (2004).
Genetically encoded fluorescent reporters of histone methylation in living cells. Journal of the American Chemical Society, 126(19), 5982-3. [PubMed:15137760] [WorldCat] [DOI] - ↑
Sasaki, K., Ito, T., Nishino, N., Khochbin, S., & Yoshida, M. (2009).
Real-time imaging of histone H4 hyperacetylation in living cells. Proceedings of the National Academy of Sciences of the United States of America, 106(38), 16257-62. [PubMed:19805290] [PMC] [WorldCat] [DOI] - ↑
Sato, M., Ueda, Y., Takagi, T., & Umezawa, Y. (2003).
Production of PtdInsP3 at endomembranes is triggered by receptor endocytosis. Nature cell biology, 5(11), 1016-22. [PubMed:14528311] [WorldCat] [DOI] - ↑ 106.0 106.1 106.2 106.3
Nishioka, T., Aoki, K., Hikake, K., Yoshizaki, H., Kiyokawa, E., & Matsuda, M. (2008).
Rapid turnover rate of phosphoinositides at the front of migrating MDCK cells. Molecular biology of the cell, 19(10), 4213-23. [PubMed:18685081] [PMC] [WorldCat] [DOI] - ↑
Nishioka, T., Frohman, M.A., Matsuda, M., & Kiyokawa, E. (2010).
Heterogeneity of phosphatidic acid levels and distribution at the plasma membrane in living cells as visualized by a Föster resonance energy transfer (FRET) biosensor. The Journal of biological chemistry, 285(46), 35979-87. [PubMed:20826779] [PMC] [WorldCat] [DOI] - ↑
Sato, M., Ueda, Y., & Umezawa, Y. (2006).
Imaging diacylglycerol dynamics at organelle membranes. Nature methods, 3(10), 797-9. [PubMed:16990811] [WorldCat] [DOI] - ↑
Okamoto, K., Nagai, T., Miyawaki, A., & Hayashi, Y. (2004).
Rapid and persistent modulation of actin dynamics regulates postsynaptic reorganization underlying bidirectional plasticity. Nature neuroscience, 7(10), 1104-12. [PubMed:15361876] [WorldCat] [DOI] - ↑
Murakoshi, H., Lee, S.J., & Yasuda, R. (2008).
Highly sensitive and quantitative FRET-FLIM imaging in single dendritic spines using improved non-radiative YFP. Brain cell biology, 36(1-4), 31-42. [PubMed:18512154] [PMC] [WorldCat] [DOI] - ↑
Haj, F.G., Verveer, P.J., Squire, A., Neel, B.G., & Bastiaens, P.I. (2002).
Imaging sites of receptor dephosphorylation by PTP1B on the surface of the endoplasmic reticulum. Science (New York, N.Y.), 295(5560), 1708-11. [PubMed:11872838] [WorldCat] [DOI] - ↑
Ni, Z., Mark, M.E., Cai, X., & Mao, Q. (2010).
Fluorescence resonance energy transfer (FRET) analysis demonstrates dimer/oligomer formation of the human breast cancer resistance protein (BCRP/ABCG2) in intact cells. International journal of biochemistry and molecular biology, 1(1), 1-11. [PubMed:20622991] [PMC] [WorldCat] - ↑
Homma, K., Niino, Y., Hotta, K., & Oka, K. (2008).
Ca(2+) influx through P2X receptors induces actin cytoskeleton reorganization by the formation of cofilin rods in neurites. Molecular and cellular neurosciences, 37(2), 261-70. [PubMed:17993279] [WorldCat] [DOI] - ↑
van Diepen, M.T., Parsons, M., Downes, C.P., Leslie, N.R., Hindges, R., & Eickholt, B.J. (2009).
MyosinV controls PTEN function and neuronal cell size. Nature cell biology, 11(10), 1191-6. [PubMed:19767745] [PMC] [WorldCat] [DOI] - ↑
Xu, X., Gerard, A.L., Huang, B.C., Anderson, D.C., Payan, D.G., & Luo, Y. (1998).
Detection of programmed cell death using fluorescence energy transfer. Nucleic acids research, 26(8), 2034-5. [PubMed:9518501] [PMC] [WorldCat] [DOI] - ↑
Onuki, R., Nagasaki, A., Kawasaki, H., Baba, T., Uyeda, T.Q., & Taira, K. (2002).
Confirmation by FRET in individual living cells of the absence of significant amyloid beta -mediated caspase 8 activation. Proceedings of the National Academy of Sciences of the United States of America, 99(23), 14716-21. [PubMed:12409609] [PMC] [WorldCat] [DOI] - ↑
Joseph, J., Seervi, M., Sobhan, P.K., & Retnabai, S.T. (2011).
High throughput ratio imaging to profile caspase activity: potential application in multiparameter high content apoptosis analysis and drug screening. PloS one, 6(5), e20114. [PubMed:21637712] [PMC] [WorldCat] [DOI] - ↑
Li, I.T., Chiang, J.J., & Truong, K. (2006).
FRET evidence that an isoform of caspase-7 binds but does not cleave its substrate. Conference proceedings : ... Annual International Conference of the IEEE Engineering in Medicine and Biology Society. IEEE Engineering in Medicine and Biology Society. Annual Conference, 1, 531-4. [PubMed:17946841] [WorldCat] [DOI] - ↑
Yang, J., Zhang, Z., Lin, J., Lu, J., Liu, B.F., Zeng, S., & Luo, Q. (2007).
Detection of MMP activity in living cells by a genetically encoded surface-displayed FRET sensor. Biochimica et biophysica acta, 1773(3), 400-7. [PubMed:17187878] [WorldCat] [DOI] - ↑
Ouyang, M., Lu, S., Li, X.Y., Xu, J., Seong, J., Giepmans, B.N., ..., & Wang, Y. (2008).
Visualization of polarized membrane type 1 matrix metalloproteinase activity in live cells by fluorescence resonance energy transfer imaging. The Journal of biological chemistry, 283(25), 17740-8. [PubMed:18441011] [PMC] [WorldCat] [DOI] - ↑
Mitra, R.D., Silva, C.M., & Youvan, D.C. (1996).
Fluorescence resonance energy transfer between blue-emitting and red-shifted excitation derivatives of the green fluorescent protein. Gene, 173(1 Spec No), 13-7. [PubMed:8707050] [WorldCat] [DOI] - ↑
Vanderklish, P.W., Krushel, L.A., Holst, B.H., Gally, J.A., Crossin, K.L., & Edelman, G.M. (2000).
Marking synaptic activity in dendritic spines with a calpain substrate exhibiting fluorescence resonance energy transfer. Proceedings of the National Academy of Sciences of the United States of America, 97(5), 2253-8. [PubMed:10688895] [PMC] [WorldCat] [DOI] - ↑
Uemura, K., Lill, C.M., Li, X., Peters, J.A., Ivanov, A., Fan, Z., ..., & Berezovska, O. (2009).
Allosteric modulation of PS1/gamma-secretase conformation correlates with amyloid beta(42/40) ratio. PloS one, 4(11), e7893. [PubMed:19924286] [PMC] [WorldCat] [DOI] - ↑
Meng, F., Suchyna, T.M., & Sachs, F. (2008).
A fluorescence energy transfer-based mechanical stress sensor for specific proteins in situ. The FEBS journal, 275(12), 3072-87. [PubMed:18479457] [PMC] [WorldCat] [DOI] - ↑
Sakai, R., Repunte-Canonigo, V., Raj, C.D., & Knöpfel, T. (2001).
Design and characterization of a DNA-encoded, voltage-sensitive fluorescent protein. The European journal of neuroscience, 13(12), 2314-8. [PubMed:11454036] [WorldCat] [DOI] - ↑
Tsutsui, H., Karasawa, S., Okamura, Y., & Miyawaki, A. (2008).
Improving membrane voltage measurements using FRET with new fluorescent proteins. Nature methods, 5(8), 683-5. [PubMed:18622396] [WorldCat] [DOI] - ↑
Jin, L., Han, Z., Platisa, J., Wooltorton, J.R., Cohen, L.B., & Pieribone, V.A. (2012).
Single action potentials and subthreshold electrical events imaged in neurons with a fluorescent protein voltage probe. Neuron, 75(5), 779-85. [PubMed:22958819] [PMC] [WorldCat] [DOI] - ↑
Akemann, W., Sasaki, M., Mutoh, H., Imamura, T., Honkura, N., & Knöpfel, T. (2013).
Two-photon voltage imaging using a genetically encoded voltage indicator. Scientific reports, 3, 2231. [PubMed:23868559] [PMC] [WorldCat] [DOI] - ↑
Suzuki, Y., Yasunaga, T., Ohkura, R., Wakabayashi, T., & Sutoh, K. (1998).
Swing of the lever arm of a myosin motor at the isomerization and phosphate-release steps. Nature, 396(6709), 380-3. [PubMed:9845076] [WorldCat] [DOI] - ↑
Zeng, W., Seward, H.E., Málnási-Csizmadia, A., Wakelin, S., Woolley, R.J., Cheema, G.S., ..., & Bagshaw, C.R. (2006).
Resonance energy transfer between green fluorescent protein variants: complexities revealed with myosin fusion proteins. Biochemistry, 45(35), 10482-91. [PubMed:16939200] [WorldCat] [DOI] - ↑
Endoh, T., Funabashi, H., Mie, M., & Kobatake, E. (2005).
Method for detection of specific nucleic acids by recombinant protein with fluorescent resonance energy transfer. Analytical chemistry, 77(14), 4308-14. [PubMed:16013840] [WorldCat] [DOI] - ↑
Yano, T., Oku, M., Akeyama, N., Itoyama, A., Yurimoto, H., Kuge, S., ..., & Sakai, Y. (2010).
A novel fluorescent sensor protein for visualization of redox states in the cytoplasm and in peroxisomes. Molecular and cellular biology, 30(15), 3758-66. [PubMed:20498274] [PMC] [WorldCat] [DOI] - ↑
Kolossov, V.L., Spring, B.Q., Clegg, R.M., Henry, J.J., Sokolowski, A., Kenis, P.J., & Gaskins, H.R. (2011).
Development of a high-dynamic range, GFP-based FRET probe sensitive to oxidative microenvironments. Experimental biology and medicine (Maywood, N.J.), 236(6), 681-91. [PubMed:21606117] [PMC] [WorldCat] [DOI] - ↑
Zacharias, D.A., Violin, J.D., Newton, A.C., & Tsien, R.Y. (2002).
Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells. Science (New York, N.Y.), 296(5569), 913-6. [PubMed:11988576] [WorldCat] [DOI] - ↑ 135.0 135.1
Murakoshi, H., Lee, S.J., & Yasuda, R. (2008).
Highly sensitive and quantitative FRET-FLIM imaging in single dendritic spines using improved non-radiative YFP. Brain cell biology, 36(1-4), 31-42. [PubMed:18512154] [PMC] [WorldCat] [DOI] - ↑
Nguyen, A.W., & Daugherty, P.S. (2005).
Evolutionary optimization of fluorescent proteins for intracellular FRET. Nature biotechnology, 23(3), 355-60. [PubMed:15696158] [WorldCat] [DOI] - ↑
Ohashi, T., Galiacy, S.D., Briscoe, G., & Erickson, H.P. (2007).
An experimental study of GFP-based FRET, with application to intrinsically unstructured proteins. Protein science : a publication of the Protein Society, 16(7), 1429-38. [PubMed:17586775] [PMC] [WorldCat] [DOI] - ↑
Lam, A.J., St-Pierre, F., Gong, Y., Marshall, J.D., Cranfill, P.J., Baird, M.A., ..., & Lin, M.Z. (2012).
Improving FRET dynamic range with bright green and red fluorescent proteins. Nature methods, 9(10), 1005-12. [PubMed:22961245] [PMC] [WorldCat] [DOI] - ↑
Ganesan, S., Ameer-Beg, S.M., Ng, T.T., Vojnovic, B., & Wouters, F.S. (2006).
A dark yellow fluorescent protein (YFP)-based Resonance Energy-Accepting Chromoprotein (REACh) for Förster resonance energy transfer with GFP. Proceedings of the National Academy of Sciences of the United States of America, 103(11), 4089-94. [PubMed:16537489] [PMC] [WorldCat] [DOI] - ↑
Kwok, S., Lee, C., Sánchez, S.A., Hazlett, T.L., Gratton, E., & Hayashi, Y. (2008).
Genetically encoded probe for fluorescence lifetime imaging of CaMKII activity. Biochemical and biophysical research communications, 369(2), 519-25. [PubMed:18302935] [PMC] [WorldCat] [DOI] - ↑
Miyawaki, A., Llopis, J., Heim, R., McCaffery, J.M., Adams, J.A., Ikura, M., & Tsien, R.Y. (1997).
Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature, 388(6645), 882-7. [PubMed:9278050] [WorldCat] [DOI] - ↑
Zaccolo, M., De Giorgi, F., Cho, C.Y., Feng, L., Knapp, T., Negulescu, P.A., ..., & Pozzan, T. (2000).
A genetically encoded, fluorescent indicator for cyclic AMP in living cells. Nature cell biology, 2(1), 25-9. [PubMed:10620803] [WorldCat] [DOI] - ↑
Sato, M., Hida, N., Ozawa, T., & Umezawa, Y. (2000).
Fluorescent indicators for cyclic GMP based on cyclic GMP-dependent protein kinase Ialpha and green fluorescent proteins. Analytical chemistry, 72(24), 5918-24. [PubMed:11140757] [WorldCat] [DOI] - ↑
Ting, A.Y., Kain, K.H., Klemke, R.L., & Tsien, R.Y. (2001).
Genetically encoded fluorescent reporters of protein tyrosine kinase activities in living cells. Proceedings of the National Academy of Sciences of the United States of America, 98(26), 15003-8. [PubMed:11752449] [PMC] [WorldCat] [DOI] - ↑
Takao, K., Okamoto, K., Nakagawa, T., Neve, R.L., Nagai, T., Miyawaki, A., ..., & Hayashi, Y. (2005).
Visualization of synaptic Ca2+ /calmodulin-dependent protein kinase II activity in living neurons. The Journal of neuroscience : the official journal of the Society for Neuroscience, 25(12), 3107-12. [PubMed:15788767] [PMC] [WorldCat] [DOI] - ↑
Lee, S.J., Escobedo-Lozoya, Y., Szatmari, E.M., & Yasuda, R. (2009).
Activation of CaMKII in single dendritic spines during long-term potentiation. Nature, 458(7236), 299-304. [PubMed:19295602] [PMC] [WorldCat] [DOI] - ↑
Okamoto, K., Nagai, T., Miyawaki, A., & Hayashi, Y. (2004).
Rapid and persistent modulation of actin dynamics regulates postsynaptic reorganization underlying bidirectional plasticity. Nature neuroscience, 7(10), 1104-12. [PubMed:15361876] [WorldCat] [DOI] - ↑
Lee, S.J., Escobedo-Lozoya, Y., Szatmari, E.M., & Yasuda, R. (2009).
Activation of CaMKII in single dendritic spines during long-term potentiation. Nature, 458(7236), 299-304. [PubMed:19295602] [PMC] [WorldCat] [DOI] - ↑
Harvey, C.D., Yasuda, R., Zhong, H., & Svoboda, K. (2008).
The spread of Ras activity triggered by activation of a single dendritic spine. Science (New York, N.Y.), 321(5885), 136-40. [PubMed:18556515] [PMC] [WorldCat] [DOI] - ↑
Murakoshi, H., Wang, H., & Yasuda, R. (2011).
Local, persistent activation of Rho GTPases during plasticity of single dendritic spines. Nature, 472(7341), 100-4. [PubMed:21423166] [PMC] [WorldCat] [DOI] - ↑
Mower, A.F., Kwok, S., Yu, H., Majewska, A.K., Okamoto, K., Hayashi, Y., & Sur, M. (2011).
Experience-dependent regulation of CaMKII activity within single visual cortex synapses in vivo. Proceedings of the National Academy of Sciences of the United States of America, 108(52), 21241-6. [PubMed:22160721] [PMC] [WorldCat] [DOI] - ↑
Akemann, W., Mutoh, H., Perron, A., Rossier, J., & Knöpfel, T. (2010).
Imaging brain electric signals with genetically targeted voltage-sensitive fluorescent proteins. Nature methods, 7(8), 643-9. [PubMed:20622860] [WorldCat] [DOI] - ↑
Kuchibhotla, K.V., Lattarulo, C.R., Hyman, B.T., & Bacskai, B.J. (2009).
Synchronous hyperactivity and intercellular calcium waves in astrocytes in Alzheimer mice. Science (New York, N.Y.), 323(5918), 1211-5. [PubMed:19251629] [PMC] [WorldCat] [DOI] - ↑
Thaler, C., Koushik, S.V., Puhl, H.L., Blank, P.S., & Vogel, S.S. (2009).
Structural rearrangement of CaMKIIalpha catalytic domains encodes activation. Proceedings of the National Academy of Sciences of the United States of America, 106(15), 6369-74. [PubMed:19339497] [PMC] [WorldCat] [DOI]





