カスパーゼ
三浦 正幸
東京大学大学院薬学系研究科 遺伝学教室
DOI:10.14931/bsd.5865 原稿受付日:2015年5月7日 原稿完成日:2015年6月2日
担当編集委員:村上 富士夫(大阪大学 大学院生命機能研究科)
英語名:caspase
カスパーゼは、インターロイキン1β変換酵素遺伝子と、線虫C. elegansの細胞死実行遺伝子ced-3と相同性を持つ、一群の細胞内システインプロテアーゼである。基質の切断部位P1にアスパラギン酸を要求する。基質を限定的に切断し、タンパク質の成熟、活性化、不活性化を引きおこす。活性化にはミトコンドリアから放出されたシトクロムcが関わる内因性経路と、FasやTNF受容体などの細胞死受容体が関わる外因性経路がある。1000以上の基質が知られており、アポトーシスに関わる基質の活性化の他、サイトカインの成熟・分泌、シグナル因子の分泌にも関わることで、細胞分化、移動、増殖、形態形成、シナプス機能調節といった様々な生命現象に関わっている。
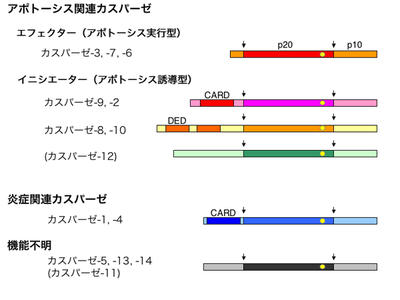
黒い矢印(↓)は活性化によって切断される部位。p20の右端近くの黄色い丸は活性中心のシステイン残基。括弧書きで示したカスパーゼ-11と12はヒトではまだ見つかっていない。図並び解説文はWikipediaより。CARD: caspase recruitment domain、DED: death effector domain。
カスパーゼとは
カスパーゼは、ヒト単球細胞からクローニングされたインターロイキン1 (IL-1)βを成熟・分泌させるインターロイキン1β変換酵素 (interleukin 1β-converting enzyme, ICE}遺伝子と、線虫C. elegansの全ての細胞死を実行する遺伝子ced-3と相同性を持つ一群の細胞内プロテアーゼである。基質の切断部位P1にアスパラギン酸を要求する特徴的なシステインプロテアーゼである(Cはシステインプロテアーゼ、aspaseはアスパラギン酸の後で切断するという特徴を示している)。
基質を限定的に切断し、タンパク質の成熟、活性化、不活性化を引きおこす[1]。これまでに1000以上の基質が知られている。カスパーゼが活性化されるとそれに引き続いてアポトーシスに特徴的な形態変化や分子の動きが見られるようになる。基質の中にはアポトーシスに特徴的な核凝縮・DNAの断片化促進に関わるICAD (inhibitor of caspase activated DNase)[2]、アポトーシス細胞膜上にフォスファチジルセリン(PS)が露出することに関わるスクランブラーゼXkr8[3]、PSの非対称性を制御するフリッパーゼATP11C[4]、アポトーシスに特徴的な細胞の形態変化に関わるキナーゼROCK-1[5]が含まれ、アポトーシス時の細胞変化を引き起こすプロテアーゼであることが判る[1]。
種類
哺乳類では18種が知られている[6]。ヒトからはカスパーゼ1〜10、12と14の12種類がクローニングされた。ヒトカスパーゼ12はエキソン4に終止コドンがあって分断されたタンパク質しかできないものに加え、全長型のタンパク質ができる遺伝子多型が報告されている。マウスカスパーゼ12は小胞体ストレスによって活性化される。マウスカスパーゼ11はヒトカスパーゼ4あるいは5と似ている。カスパーゼ1サブファミリー(カスパーゼ1、4、5、11、12)は遺伝子重複でできたと考えられ染色体上に連続して存在する[7]。線虫にはCED-3を含めて4種[8]、ショウジョウバエには7種がゲノム中に存在する[9]。
カスパーゼと類似の配列を持ったタンパク質が広く存在する[10]。メタカスパーゼは植物、菌、原生動物や細菌で、パラカスパーゼは動物、粘菌、細菌で見つかっている。植物、菌、原生動物のメタカスパーゼは細胞死に関わることが報告されているがパラカスパーゼの細胞死への関与は不明である。
構造
全てのカスパーゼは前駆体(プロカスパーゼ)として翻訳される。前駆体の酵素活性は非常に低い。N末端からプロドメイン、P20、P10をコードするサブユニットからなるが、プロドメインの構造と基質特異性からカスパーゼはサブタイプに分類することができる。短いプロドメインを持つ細胞死実行カスパーゼ3、6、7は、長いプロドメインを持つイニシエーターカスパーゼ(カスパーゼ2、8、9、10)によって切断されて活性化される[1]。細胞死実行カスパーゼは酵素活性が強く、活性化されると様々な基質を切断して細胞死を実行する。
活性型のカスパーゼはP20とP10がヘテロダイマーを作り、これが2つ合わさった4量をとる。イニシエーターカスパーゼの長いプロドメインはCARD (Caspase Recruitment Domain)あるいはDED (Death Effecter Domain)から構成され、タンパク質間相互作用に使われる。イニシエーターカスパーゼのDEDあるいはCARDにアダプタータンパク質が結合することによってイニシエーターカスパーゼが凝集し活性化する。これがカスパーゼによるタンパク質分解カスケードの開始シグナルとなる[1]。
活性化機構
ミトコンドリアが関わる内因性の仕組みと、細胞死受容体が関わる外因性の仕組みの2つに分類される。
ミトコンドリア経路
細胞死シグナルを受けたほ乳類細胞ではミトコンドリアからシトクロムcが細胞質に放出される。シトクロムcがアダプター分子Apaf-1に受容されると多量体化が引き起こされるが、その際にプロカスパーゼ9多量体に組み込まれ凝集することで活性化される[1]。このシトクロムc、Apaf-1、プロカスパーゼ9からなる複合体をアポプトソームと呼ぶ。これが引き金となり下流の細胞死実行カスパーゼが活性化される。線虫から細胞死遺伝子として同定されたCED-4はApaf-1とホモロジーを持つ分子であるが、シトクロムcを結合するC末端側のWD40リピートを欠いている。線虫の健常な細胞では、ミトコンドリアにあるCED-9 (Bcl-2ファミリー分子)にCED-4が結合している。ところが、死に行く細胞系譜においてBH3 onlyタンパク質であるEgl-1が発現するとCED-4と置き換わってCED-9と結合するるようになる。細胞質に放出されたCED-4はCED-3と結合し、CED-3の多量体化を促進することでCED-3を活性化する[11]。
細胞死受容体による外因性の経路
FasやTNF受容体に代表される細胞死誘導リガンドに対する受容体を介した細胞死では、リガンドの結合によって受容体の多量体化がおこり、受容体の細胞質領域にある細胞死ドメイン(death domain)にアダプタータンパク質とプロカスパーゼ8がリクルートされて活性化される。このリガンドの結合によって作られる受容体直下の複合体はDISC (death inducing signaling complex)と呼ばれる。FADDはC末端の細胞死ドメインで受容体と結合し、N末端のDED (death effector domain)でプロカスパーゼ8のDEDと結合する。凝集したプロカスパーゼ8は活性化型に変換し、プロカスパーゼ3を切断し活性化する。カスパーゼ8がプロカスパーゼ3を直接活性する細胞を1型細胞と呼ぶことがある。これに対し2型細胞と呼ばれる細胞では、活性化したカスパーゼ8がBH3-onlyファミリー Bidを切断し、切断された Bid (tBid)がミトコンドリア膜上にあるBax、Bakを凝集させてシトクロムcのミトコンドリアからの放出を促し、Apaf-1/カスパーゼ9/カスパーゼ3経路(上記ミトコンドリア経路)を活性化する[1]。
インフラマソームによる活性化
カスパーゼ1はインフラマソームと呼ばれるアポプトソームとは異なる構成因子によって形成される複合体で活性化される[12]。インフラマソームはカスパーゼ1、アダプタータンパク質ASC、Apaf-1に似たドメインを持つ分子NALP-1、-3といったNOD-like receptor (NLR)分子からなる複合体として形成される。インフラマソームは細菌感染やウイルス等の外来性の核酸による細胞内変化に応じて活性化されるほか、傷害細胞から放出されるATPなどの内在性危険信号物質によっても活性化される。さらに、様々なストレス応答としても活性化されることが明らかにされている。マクロファージで活性化されたカスパーゼ1はproIL-1βやproIL-18の切断による成熟と分泌を促す。生体イメージングによる解析から、マクロファージではカスパーゼ1の活性化とそれに引きいておこるIL-1βの分泌は全か無かの様式でおこることが観察されている[13]。
機能
カスパーゼはシステインプロテーアーゼであり、その認識配列で基質を限定分解することで機能を発揮する。基質は限定分解を受けることでその機能が失われたり、逆に亢進する(例えば、酵素の活性調節領域が切り離されて活性化する)。イニシエーターカスパーゼにはそのN末端側にタンパク質間相互作用に関わるCARDやDEDドメインがあり、このドメインを介したタンパク質相互作用によってカスパーゼ活性とは別に生理機能を果たすこともありうる。
細胞死誘導機能
カスパーゼは発生過程で過剰に作られた細胞や、ストレスを受けて傷害された細胞を生体から取り除くためのアポトーシスを誘導する。その一方で、死につつある細胞から周りの細胞に増殖シグナルを発する過程にも関わることが報告されている(代償性増殖)[14]。
カスパーゼ3、9、あるいはその活性化因子Apaf-1のノックアウトマウスは129系統において神経管閉鎖不全をともなう外脳症を発症する。神経管閉鎖時の細胞死動態を生体イメージングで解析した結果、アポトーシスが抑制された胚では神経管閉鎖速度が減少し、神経管閉鎖後に見られる脳室の拡大が、その時期になってもおきない。発生では一連の形態形成運動が適切なタイミングでおこることが必要であり、神経管閉鎖遅延が脳の形態異常の初期変化をもたらす原因と考えられる[15]。
ショウジョウバエにおける生体イメージングによる観察から、外感覚器前駆細胞(sensory organ precursor: SOP)が作られる時に分化に失敗した細胞がある頻度で出現するが、そのような細胞は選択的にアポトーシスによって除去されることが示されている[15]。
神経変性疾患の発症とカスパーゼとの関連も多く調べられている[16]。ハンチントン舞踏病患者およびそのマウスモデルにおいて、ポリグルタミン鎖の伸長したハンチンチンは カスパーゼによって切断を受ける。そしてハンチンチン切断断片が毒性を発揮することが示されている。アルツハイマー病の発症に関わるアミロイド前駆体タンパク質(APP)はカスパーゼ3で切断され、この切断されたAPPはアミロイド形成しやすいAβ42への切断が促進される。また、カスパーゼ3で切断されたC末端断片が細胞内で毒性をもつことも示されている。
非細胞死機能
カスパーゼ1はIL-1βやIL-18といった直接基質となるサイトカインの成熟・分泌に関わるだけではなく、bFGFのようなシグナル配列を持たないシグナル因子の分泌にも関わることが報告されている。カスパーゼは細胞内での局所的な活性化や、一過性の活性化によってアポトーシス実行から回避されることで、細胞分化、移動、増殖、細胞形態形成、シナプス機能調節といった様々な生命現象に関わっている[17]。
神経系でのカスパーゼによる非細胞死の働きを以下に示す。ショウジョウバエ幼虫期にSOPが未分化な神経前駆細胞集団から選ばれる数の調節に関しては、非細胞死機能としてのカスパーゼ活性が関わる[15]。
他にもショウジョウバエ変態期には、神経細胞の一部を除去することで神経系の再編成がおこる。樹状突起の刈り込みは樹状突起局所でのカスパーゼ活性化によって細胞死を伴わないで実行される[15]。樹状突起分岐形成に関しても、分岐点局所でのカスパーゼ活性が関与することがゼブラフィッシュで報告されている[15]。
哺乳類神経系において、網膜神経節細胞が外側膝状体に投射する際に軸索の刈り込みがおこるが、このプロセスにもカスパーゼが関わる可能性が示されている。
シナプス形成や機能とカスパーゼとの関連も報告されている。マウス嗅覚神経のシナプス成熟にカスパーゼ活性が関与することが示された[15]。キンカチョウの歌学習時には後シナプス終末でのカスパーゼ活性化がおこり、その阻害によって長期記憶形成が抑制される[18]。哺乳類では長期抑制(LTD)とAMPA型グルタミン酸受容体の内在化にカスパーゼ3の活性が使われている[19]。
関連項目
参考文献
- ↑ 1.0 1.1 1.2 1.3 1.4 1.5
Degterev, A., Boyce, M., & Yuan, J. (2003).
A decade of caspases. Oncogene, 22(53), 8543-67. [PubMed:14634618] [WorldCat] [DOI] - ↑
Sakahira, H., Enari, M., & Nagata, S. (1998).
Cleavage of CAD inhibitor in CAD activation and DNA degradation during apoptosis. Nature, 391(6662), 96-9. [PubMed:9422513] [WorldCat] [DOI] - ↑
Suzuki, J., Denning, D.P., Imanishi, E., Horvitz, H.R., & Nagata, S. (2013).
Xk-related protein 8 and CED-8 promote phosphatidylserine exposure in apoptotic cells. Science (New York, N.Y.), 341(6144), 403-6. [PubMed:23845944] [WorldCat] [DOI] - ↑
Segawa, K., Kurata, S., Yanagihashi, Y., Brummelkamp, T.R., Matsuda, F., & Nagata, S. (2014).
Caspase-mediated cleavage of phospholipid flippase for apoptotic phosphatidylserine exposure. Science (New York, N.Y.), 344(6188), 1164-8. [PubMed:24904167] [WorldCat] [DOI] - ↑
Coleman, M.L., Sahai, E.A., Yeo, M., Bosch, M., Dewar, A., & Olson, M.F. (2001).
Membrane blebbing during apoptosis results from caspase-mediated activation of ROCK I. Nature cell biology, 3(4), 339-45. [PubMed:11283606] [WorldCat] [DOI] - ↑
Eckhart, L., Ballaun, C., Hermann, M., VandeBerg, J.L., Sipos, W., Uthman, A., ..., & Tschachler, E. (2008).
Identification of novel mammalian caspases reveals an important role of gene loss in shaping the human caspase repertoire. Molecular biology and evolution, 25(5), 831-41. [PubMed:18281271] [WorldCat] [DOI] - ↑
Lamkanfi, M., Kalai, M., & Vandenabeele, P. (2004).
Caspase-12: an overview. Cell death and differentiation, 11(4), 365-8. [PubMed:14685161] [WorldCat] [DOI] - ↑
Shaham, S. (1998).
Identification of multiple Caenorhabditis elegans caspases and their potential roles in proteolytic cascades. The Journal of biological chemistry, 273(52), 35109-17. [PubMed:9857046] [WorldCat] [DOI] - ↑
Kumar, S., & Doumanis, J. (2000).
The fly caspases. Cell death and differentiation, 7(11), 1039-44. [PubMed:11139276] [WorldCat] [DOI] - ↑
Uren, A.G., O'Rourke, K., Aravind, L.A., Pisabarro, M.T., Seshagiri, S., Koonin, E.V., & Dixit, V.M. (2000).
Identification of paracaspases and metacaspases: two ancient families of caspase-like proteins, one of which plays a key role in MALT lymphoma. Molecular cell, 6(4), 961-7. [PubMed:11090634] [WorldCat] [DOI] - ↑
Yan, N., Chai, J., Lee, E.S., Gu, L., Liu, Q., He, J., ..., & Shi, Y. (2005).
Structure of the CED-4-CED-9 complex provides insights into programmed cell death in Caenorhabditis elegans. Nature, 437(7060), 831-7. [PubMed:16208361] [WorldCat] [DOI] - ↑
Lamkanfi, M., & Dixit, V.M. (2014).
Mechanisms and functions of inflammasomes. Cell, 157(5), 1013-22. [PubMed:24855941] [WorldCat] [DOI] - ↑
Liu, T., Yamaguchi, Y., Shirasaki, Y., Shikada, K., Yamagishi, M., Hoshino, K., ..., & Miura, M. (2014).
Single-cell imaging of caspase-1 dynamics reveals an all-or-none inflammasome signaling response. Cell reports, 8(4), 974-82. [PubMed:25127135] [WorldCat] [DOI] - ↑
Martín, F.A., Peréz-Garijo, A., & Morata, G. (2009).
Apoptosis in Drosophila: compensatory proliferation and undead cells. The International journal of developmental biology, 53(8-10), 1341-7. [PubMed:19247932] [WorldCat] [DOI] - ↑ 15.0 15.1 15.2 15.3 15.4 15.5
Yamaguchi, Y., & Miura, M. (2015).
Programmed cell death in neurodevelopment. Developmental cell, 32(4), 478-90. [PubMed:25710534] [WorldCat] [DOI] - ↑
D'Amelio, M., Sheng, M., & Cecconi, F. (2012).
Caspase-3 in the central nervous system: beyond apoptosis. Trends in neurosciences, 35(11), 700-9. [PubMed:22796265] [WorldCat] [DOI] - ↑
Shalini, S., Dorstyn, L., Dawar, S., & Kumar, S. (2015).
Old, new and emerging functions of caspases. Cell death and differentiation, 22(4), 526-39. [PubMed:25526085] [PMC] [WorldCat] [DOI] - ↑
Huesmann, G.R., & Clayton, D.F. (2006).
Dynamic role of postsynaptic caspase-3 and BIRC4 in zebra finch song-response habituation. Neuron, 52(6), 1061-72. [PubMed:17178408] [PMC] [WorldCat] [DOI] - ↑
Li, Z., Jo, J., Jia, J.M., Lo, S.C., Whitcomb, D.J., Jiao, S., ..., & Sheng, M. (2010).
Caspase-3 activation via mitochondria is required for long-term depression and AMPA receptor internalization. Cell, 141(5), 859-71. [PubMed:20510932] [PMC] [WorldCat] [DOI]