位置情報
笹井紀明
奈良先端科学技術大学院大学 バイオサイエンス研究科 発生医科学研究室
DOI:10.14931/bsd.7516 原稿受付日:2018年3月1日 原稿完成日:2018年3月27日
担当編集委員:大隅 典子(東北大学 大学院医学系研究科 附属創生応用医学研究センター 脳神経科学コアセンター 発生発達神経科学分野)
英語名:positional information
発生生物学における位置情報とは、胚発生期にある前駆細胞が組織内で自身の位置を認識するために細胞外部からもたらされる情報のことをいう。ここでいう「情報」は多くの場合、細胞外因子(分泌因子、細胞接着因子)の種類と濃度を意味する。細胞はこの情報によって、自らの器官内における相対的な位置を知り、どのような役割(機能)を担うのかを判断して分化する。ここでは、細胞外からもたらされるシグナル分子の種類や濃度勾配に対して細胞がどう反応するのかについて、発生期の脊髄神経における背腹軸パターン形成を中心に概説する。
位置情報とは
胚発生期の器官の形態形成(morphogenesis)においては、前駆細胞が器官内で様々な細胞へと分化し、それらが一定のルールに従って配置される。この細胞の配置はどの個体でも同じであり、このことを「パターン形成(pattern formation)」という[1][2]。器官内の種々の細胞が正確に配置されることは、器官が機能体として固有の機能を発揮するために重要である。器官が正確にパターン化されるためには、各前駆細胞が器官内における自らの相対的位置を認識する必要があるが、それを与えるのが位置情報である。
器官発生の初期には、組織は未分化細胞の塊だが、やがて細胞塊の一部にシグナルの源である細胞群(シグナリングセンター(signalling centre))が出現する[3]。この領域からは細胞外因子が分泌され、この分泌因子は多くの場合器官内で濃度勾配を形成する。器官内の各細胞(前駆細胞)はこの濃度によって自分が何の細胞に分化するのか(つまりどのような機能を分担するのか)の情報を獲得するのである。
モルフォゲンモデル
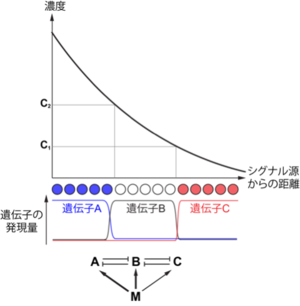
モルフォゲンの濃度によって多数の細胞が産生される様子を模式的に表した図(フランス国旗モデル[4][5]をもとに作成)
1969年、Lewis Wolpertは、シグナリングセンターから分泌された細胞外物質(モルフォゲン(morphogen))が濃度勾配を持って組織内に拡散しており、細胞の分化方向はその濃度に依存して決まるとする組織形成の原理を仮説的に提唱した[6][7][8][9]。モルフォゲンとはギリシャ語で「form giver(形を与えるもの)」という意味であり[10]、多くの研究から、組織形成に関わる液性因子(分泌タンパク質や化学低分子)のほとんど(線維芽細胞増殖因子(FGF)やWnt、トランスフォーミング増殖因子(TGFβ)、ソニック・ヘッジホッグ、レチノイン酸)がモルフォゲンとして働くことがこれまでに示されている[11]。濃度勾配が組織形成を制御するという概念は「モルフォゲンモデル(morphogen model)」と呼ばれ、現在に至るまで組織のパターン形成を分子的に説明する基本概念の1つである。
モルフォゲンにより器官内で多種類の細胞が生じる様子は、しばしば「フランス国旗」で表される[6][4]。これは、各細胞の分化方向の決定に濃度閾値(threshold)が存在することを前提にしており、濃度によって3つの異なる細胞が生じる様子をフランス国旗の青、白、赤の3色で表したものである(図1A)。このモデルは、濃度勾配を感知した各前駆細胞が、濃度に応じて3種類(実際にはそれ以上)の細胞に分化していく様子を表している。このことは、濃度依存的に発現する遺伝子が存在することを示している。さらに、隣接する領域に発現する遺伝子同士は互いにその発現を抑制する関係にあることも多い。
このようにして、細胞外からの情報(濃度勾配)が細胞内制御機構(遺伝子の転写制御ネットワーク)に変換され、異なる種類の細胞(つまり図1Aの赤と白、白と青)の境界が明確になり、結果的に正確なパターン形成が作り上げられる[8]。
しかし、組織内で形成される濃度勾配は静的ではなく(つまり発生過程のある時期に突然形成され、それが永久に保存されるものではなく)、器官発生の進行と連動して経時的に変化し、維持されるものであるから、各細胞が暴露される濃度も時々刻々と変化し、それにつれて細胞の分化状態も変化しながら徐々にパターン形成が確立されて行く(図1B)。
以下、濃度勾配の形成と細胞の分化状態の変化の関係を記述する。
神経管背腹軸における領域決定
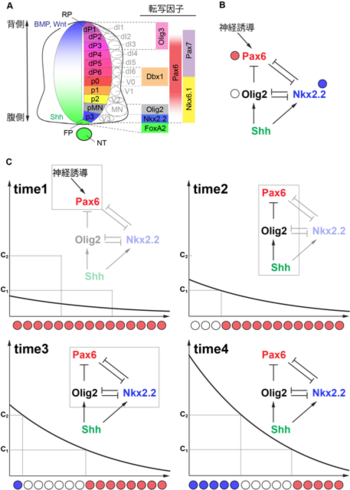
A. 脊髄レベルにおける神経管断面の模式図。神経管の中で、内側にはdP0-dP6, p0, p1, p2, pMN, p3の各領域が存在し、これらは神経前駆細胞である(つまり神経としての性質は持たず、増殖する)。領域名についているpはprogenitor(前駆細胞)の意味。一方外側の領域には神経(つまり機能性の神経細胞)領域dP0-dP6, V0, V1, V2, MN, V3が存在する。MNは運動神経(motor neuron)で、そのほかは介在神経の性質を持つ(各神経の性質については13を参照)。原則として、各領域は対応する前駆領域から分化する(例えば、dI1細胞はdp1細胞から、V1細胞はp1細胞から分化する)。この模式図はマウス10.5-11.5日胚、ニワトリの4-5日胚で見られるものであり、この後分化が進めば前駆領域は徐々に小さくなり、生まれる(孵化する)頃にはほとんどなくなってしまう。
FP: 底板(floor plate)、RP: 蓋板(roof plate)、NT: 脊索(notochord)。
B. 腹側神経前駆領域(Pax6, Olig2, Nkx2.2)の転写抑制の関係と、Shhによる転写誘導の関係。図中の青、白、赤のそれぞれの丸印は図2Cの細胞に対応する。
C. モルフォゲン(Shh)の濃度が徐々に上昇していきながらパターンが形成されていく様子。
(A)は[12][13]を、(B,C)は[14]を元に作成した。
モルフォゲンの動的な濃度勾配の変化がよく解析されているのは、脊髄神経管の断面(背腹軸)におけるシグナル分子と領域決定の関係についてである。胚発生期の神経管には背腹軸に沿って多数の神経前駆領域、神経領域が出現する[12][15] 。図2Aはその様子を模式的に表したもので、マウス10.5-11.5日胚、ニワトリ4-5日胚の脊髄レベルの神経管の断面を作成するとほぼ同様の様子が観察される。図中のdP1-dP6、p0-p3、pMNの各領域にはそれぞれ特有の性質を持つ神経前駆細胞が配置される。各前駆領域の細胞はさらに分化して、それぞれの前駆領域に対応する機能性の神経細胞(ニューロン)を産出する(dI1-dI6、V0-V3、MN:各神経細胞がもつ性質については[13]を参照)。
個々の領域を分子レベルで特徴付けることができるのは、領域特異的に発現するホメオボックス型またはbHLH型転写因子が同定されているためである(同定されている転写因子の一部を図2Aに掲載した:詳細については[13]を参照)。 これらの領域はどの個体でも配置が変わることがないため、「パターン」と呼ばれており、そのパターン決定は、蓋板(RP)や底板(FP)からそれぞれ分泌される骨形成因子(BMP)、Wnt、ソニック・ヘッジホッグ(Sonic Hedgehog; Shh)といったモルフォゲンの濃度勾配によっている。つまり、これら各領域の細胞の分化方向はモルフォゲンの種類と濃度という位置情報によって決定されるのであり、その意味で神経管の背腹軸は位置情報を解析する上で良いモデル系である。
腹側神経管とソニック・ヘッジホッグ
発生期におけるパターン形成は、発生過程のある時期に瞬間的に形成されるのではなく、モルフォゲンの時期的な濃度勾配の動的変化に従って徐々に形成されていくものである。この過程で、未分化な状態(または分化度が低い状態)で発現している遺伝子が徐々に他の遺伝子に置き換わっていく。
モルフォゲンのうち、神経管のパターン形成について解析が進んでいるのは、ソニック・ヘッジホッグ(Sonic Hedgehog; Shh)と腹側神経前駆領域のパターン形成である。Shhは、神経管の底板とその下部にある中胚葉性の組織、脊索(notochord; NT)に発現し、神経管の中で濃度勾配を形成して、主にp0領域から腹側の領域の決定に重要な役割を果たしている(図2A)[12]。
Shhのノックアウトマウスでは腹側神経領域のほとんどが消失し、胚性致死となる[12][16]。 しかし、Shhが神経管内で突然濃度勾配を形成するわけではない。神経管が形成された初期には、Shhは神経管の下部に位置する中胚葉由来の組織、脊策から分泌され、神経管の腹側のごく限られた領域に分布している。この領域には、まず低濃度領域に発現する遺伝子の発現(例えばNkx6.1やDbx1)が開始する[17]。その後、Shhの発現が持続するにつれて、発生源である底板領域の発現量は高くなって遠くまで濃度勾配が形成されるようになり、高濃度領域の遺伝子の発現が開始する。
この過程において、Nkx6.1、Nkx2.2、FoxA2の遺伝子の発現は、Shhシグナルを細胞内で仲介する転写因子Gliによって制御されているが、それぞれの遺伝子の発現制御領域に存在するGliの結合配列(DNA配列)が異なるために、アフィニティー(結合力)に違いがあることが示唆されている[18][19]。つまり、Shhシグナルに対する敏感さの違いが遺伝子発現とパターン形成を決定していると言える。
一方、転写因子のネットワークが重要だとするモデルも提唱されている(図2B)。神経管の発生初期には神経前駆細胞全体に転写因子の1つPax6が発現している。Pax6の発現自体は、神経誘導因子(細胞に「神経」という運命を与える因子)によって誘導される(図2C, time 1)。次に、Pax6はOlig2によって発現抑制される関係にあるため、Olig2がShhによって発現誘導されると、Pax6の発現が抑制される(図2C, time 2)。一方、Nkx2.2はOlig2と同じくShhのターゲット遺伝子であるが、初期にはPax6によってその発現が抑制されており、発現しない(図2C, time 2)。しかしOlig2がPax6の発現を抑制するとPax6がNkx2.2を抑制する作用が弱まり、結果的にNkx2.2の発現が開始する(図2C, time 3)。最後にNkx2.2とOlig2の相互抑制関係によってpMNとp3の領域が明確に分離し(図2C, time 4)、Pax6、Olig2(pMN領域)、Nkx2.2(p3領域)による神経管のパターン形成が完成するのである[14]。
この関係は常微分方程式によって数理モデル化されており、ShhによるOlig2とNkx2.2発現誘導効果が同等であってもパターン形成は成立する[14]。つまり、Gliのアフィニティー(結合力)の違いによる発現誘導とは異なるメカニズムで遺伝子発現が制御されていると言える。
上述のパターン形成で議論した細胞はすべて前駆細胞であり、機能的神経細胞に分化するにはNeurogeninなどの神経化転写因子が必要である。最近、Olig2がHes1,Hes5という転写因子の発現抑制を介してNeurogenin2の発現を誘導するという転写制御システムが提唱されるようになった[20]。
背側神経管とWnt、BMP
神経管の背側については、各領域に特異的に発現する遺伝子があまり同定されていないこともあり、蓋板から分泌される因子と個々の領域に発現する転写因子との関係は、腹側神経領域ほどは詳細に知られていない。しかし、主にWnt/BMPシグナルと背側神経管の領域決定の関係について、ノックアウトマウスやニワトリ胚を用いた研究が進んでいる。
まず、そもそも蓋板領域が背側神経管のパターン形成に必須であることが、蓋板領域を遺伝的に破壊したマウスで示されている[21]。次に、蓋板に発現する遺伝子として、Wnt1/3Aのほか、BMP4/5/6/7、さらにBMPと同じくTGFβファミリーに属するGDF7があり、これらが背側神経管のパターン形成に必須の役割を果たしている[21][22][23][24]。Wnt1/3Aのダブルノックアウトマウスでは、dI1、dI2領域が消失し、逆にdI3領域が拡大する[25]。このノックアウトマウスではBMPの濃度勾配の形成は影響を受けないことから、WntとBMPが独立に濃度勾配を形成することが示唆される。また、ノックアウトマウスの表現型からは、Wntは背腹軸全体のパターン形成に関与するのではなく、背側神経管内(dP1-dP3)のバランス維持に重要であることが示唆される。また、Wnt1/3aシグナルは、Shhシグナルの仲介因子であるGli3の発現に必須であるため[23]、WntはShhシグナルを、(たんぱく質同士の直接の相互作用ではなく)転写を介して制御していると言える。
一方、蓋板に発現するBMPシグナル分子に関しては、少なくともBMP4/5/6/7が発現しているほか、これらと同様にTGFβスーパーファミリーに属するGDF7が存在する[21][22][23]。BMPシグナルを全体をブロックするためにBMPレセプターのノックアウトが行われており、このノックアウトマウスはdI1/dI2領域が欠損することが明らかになっている[26]が、Wnt1/3aは(若干発現量が減少するものの)発現は維持されている。また、GDF7のノックアウトマウスでは背側の介在神経前駆細胞(dP1-dP6; 図2A)のほとんどの分化が抑制されるが、Wnt1/3Aの発現は影響を受けていない[27]。したがって、WntとBMPはいずれも背側神経管のパターン形成に重要だが、互いの発現には大きな影響を及ぼさず、独立に背側のパターン形成に役割を担っていると考えられる。
それでは濃度の勾配は重要であろうか。実際には、WntやBMPの濃度勾配が背側神経管のパターン形成に必須であるというデータがある[25][28] 。その一方最近、濃度勾配よりもシグナル因子の種類そのものの違いが細胞の分化方向を決定している(例えばBMP4 はdI1を強く誘導する一方で、BMP5はdI3を、BMP6とGDF7はdI1とdI3の分化を誘導する)という主張も存在する。したがって、蓋板から発現する様々なBMP/Wnt因子が独立に濃度勾配を形成し、その組み合わせが領域の多様性を生み出すと考えられる。
濃度勾配の形成と維持
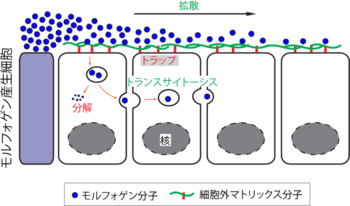
モルフォゲンの拡散と濃度勾配の形成・維持のメカニズム([29][30]をもとに作成)
一般に濃度勾配の形成は、分泌因子の拡散と、それを抑制(または制御)する効果のバランスによって成立している[29]。まず、シグナリングセンターで産生されたモルフォゲンは、そこから組織内へと放出され、細胞間隙(細胞外マトリックス)に浸透して組織内へと拡散する(図3A)。しかし、モルフォゲンが産出され、湧出するだけでは、器官のように閉鎖された空間では一定時間の後には濃度が一定になってしまう。したがって、その濃度勾配を維持するために、モルフォゲンの拡散(遠くに伝えようとする作用)と、拡散を制限する(遠くに行かせない)作用のバランスが保持されている。そのメカニズムには以下のようなものが知られている。
まずモルフォゲンを遠くに伝えるための方策としては、モルフォゲンが細胞間隙を浸潤・拡散していくほかに、モルフォゲンが細胞内を通過して拡散する「トランスサイトーシス(transcytosis)」の経路が知られている(図3)[31][32][30][33][34][35]。ショウジョウバエの羽の原基(wing disc)における分泌因子dppの拡散様式は、この原則に従うことが知られている。一方、拡散を抑制する作用の1つとしては、たんぱく質の分解やエンドサイトーシスによる細胞内への取り込みが挙げられる。タンパク質は一般に一定時間が過ぎると分解されるため、同時に産生されたたんぱく質の分子数(量)は時間とともに徐々に減少する。
濃度勾配を維持するための別の制御として、細胞外マトリックスたんぱく質の存在が知られている。これは、濃度勾配を時するために、拡散を補助することもあれば抑制することもある。
細胞外マトリックスたんぱく質のうち、主なものはヘパラン硫酸プロテオグリカン(heparin sulfate proteoglycan; HSPG)と総称される糖タンパク質で、このうちグリピカンやシンデカンがモルフォゲン産生細胞の周辺部に分布してモルフォゲンに結合し、モルフォゲンの拡散を助け、あるいは抑制して濃度勾配の維持に関与すると示唆されている[30]。例えばグリピカン3(GPC3)はShhやWntに結合することが示されているが、Wntシグナルを増強する一方で、Shhシグナルを弱化する[36][37]。特にShhはGPC3の糖鎖に結合してエンドサイトーシスによって細胞内に取り込まれ、リソソームによる分解系に送り込まれる[36]。逆に、GPC3のノックアウトマウスではShhシグナルの異常亢進を起こしている。
一方、他のグリピカンについては、ショウジョウバエを用いた実験から別のメカニズムが提唱されている。GPC4やGPC6のショウジョウバエホモログDally-like(Dlp)は、同じくHhやWg(Wntのショウジョウバエホモログ)に結合し、エンドサイトーシスによって細胞内に取り込む[38]が、Hhは分解されずトランスサイトーシスされ、Wgは分解系に送り込まれる[39]。このように、グリピカンなどの細胞外マトリックスタンパク質によるシグナル分子への結合、細胞内への取り込み、拡散・分解のシステムはシグナル分子特異的である。
エンドサイトーシスによるモルフォゲンの取り込みに関してはゼブラフィッシュにおけるリアルタイムイメージングによっても示されている。このように、細胞がモルフォゲン分子を取り込んで分解する様子は、分子を積極的に排除することを意味し、湧き出したモルフォゲンが流し台で排出される様子に例えることができるので、「source-sink model(湧き出しと排水モデル)」と呼ばれる[29][30][40]。以上のように、拡散と分解のバランスが組織内におけるモルフォゲンの濃度勾配を調節していると言える。
関連項目
参考文献
- ↑
Wolpert, L. (2011).
Positional information and patterning revisited. Journal of theoretical biology, 269(1), 359-65. [PubMed:21044633] [WorldCat] [DOI] - ↑
Wolpert, L. (2016).
Positional Information and Pattern Formation. Current topics in developmental biology, 117, 597-608. [PubMed:26970003] [WorldCat] [DOI] - ↑
Basson, M.A. (2012).
Signaling in cell differentiation and morphogenesis. Cold Spring Harbor perspectives in biology, 4(6). [PubMed:22570373] [PMC] [WorldCat] [DOI] - ↑ 4.0 4.1
Kerszberg, M., & Wolpert, L. (2007).
Specifying positional information in the embryo: looking beyond morphogens. Cell, 130(2), 205-9. [PubMed:17662932] [WorldCat] [DOI] - ↑
Fried, P., & Iber, D. (2014).
Dynamic scaling of morphogen gradients on growing domains. Nature communications, 5, 5077. [PubMed:25295831] [WorldCat] [DOI] - ↑ 6.0 6.1
Wolpert, L. (1969).
Positional information and the spatial pattern of cellular differentiation. Journal of theoretical biology, 25(1), 1-47. [PubMed:4390734] [WorldCat] [DOI] - ↑
Wolpert, L. (1994).
Positional information and pattern formation in development. Developmental genetics, 15(6), 485-90. [PubMed:7834908] [WorldCat] [DOI] - ↑ 8.0 8.1
Briscoe, J., & Small, S. (2015).
Morphogen rules: design principles of gradient-mediated embryo patterning. Development (Cambridge, England), 142(23), 3996-4009. [PubMed:26628090] [PMC] [WorldCat] [DOI] - ↑
Ashe, H.L., & Briscoe, J. (2006).
The interpretation of morphogen gradients. Development (Cambridge, England), 133(3), 385-94. [PubMed:16410409] [WorldCat] [DOI] - ↑
Tabata, T. (2001).
Genetics of morphogen gradients. Nature reviews. Genetics, 2(8), 620-30. [PubMed:11483986] [WorldCat] [DOI] - ↑
Bökel, C., & Brand, M. (2013).
Generation and interpretation of FGF morphogen gradients in vertebrates. Current opinion in genetics & development, 23(4), 415-22. [PubMed:23669552] [WorldCat] [DOI] - ↑ 12.0 12.1 12.2 12.3
Ribes, V., & Briscoe, J. (2009).
Establishing and interpreting graded Sonic Hedgehog signaling during vertebrate neural tube patterning: the role of negative feedback. Cold Spring Harbor perspectives in biology, 1(2), a002014. [PubMed:20066087] [PMC] [WorldCat] [DOI] - ↑ 13.0 13.1 13.2
Alaynick, W.A., Jessell, T.M., & Pfaff, S.L. (2011).
SnapShot: spinal cord development. Cell, 146(1), 178-178.e1. [PubMed:21729788] [PMC] [WorldCat] [DOI] - ↑ 14.0 14.1 14.2
Balaskas, N., Ribeiro, A., Panovska, J., Dessaud, E., Sasai, N., Page, K.M., ..., & Ribes, V. (2012).
Gene regulatory logic for reading the Sonic Hedgehog signaling gradient in the vertebrate neural tube. Cell, 148(1-2), 273-84. [PubMed:22265416] [PMC] [WorldCat] [DOI] - ↑
Le Dréau, G., & Martí, E. (2012).
Dorsal-ventral patterning of the neural tube: a tale of three signals. Developmental neurobiology, 72(12), 1471-81. [PubMed:22821665] [WorldCat] [DOI] - ↑
Chiang, C., Litingtung, Y., Lee, E., Young, K.E., Corden, J.L., Westphal, H., & Beachy, P.A. (1996).
Cyclopia and defective axial patterning in mice lacking Sonic hedgehog gene function. Nature, 383(6599), 407-13. [PubMed:8837770] [WorldCat] [DOI] - ↑
Dessaud, E., Ribes, V., Balaskas, N., Yang, L.L., Pierani, A., Kicheva, A., ..., & Sasai, N. (2010).
Dynamic assignment and maintenance of positional identity in the ventral neural tube by the morphogen sonic hedgehog. PLoS biology, 8(6), e1000382. [PubMed:20532235] [PMC] [WorldCat] [DOI] - ↑
Oosterveen, T., Kurdija, S., Alekseenko, Z., Uhde, C.W., Bergsland, M., Sandberg, M., ..., & Ericson, J. (2012).
Mechanistic differences in the transcriptional interpretation of local and long-range Shh morphogen signaling. Developmental cell, 23(5), 1006-19. [PubMed:23153497] [WorldCat] [DOI] - ↑
Oosterveen, T., Kurdija, S., Ensterö, M., Uhde, C.W., Bergsland, M., Sandberg, M., ..., & Ericson, J. (2013).
SoxB1-driven transcriptional network underlies neural-specific interpretation of morphogen signals. Proceedings of the National Academy of Sciences of the United States of America, 110(18), 7330-5. [PubMed:23589857] [PMC] [WorldCat] [DOI] - ↑
Sagner, A., Gaber, Z.B., Delile, J., Kong, J.H., Rousso, D.L., Pearson, C.A., ..., & Novitch, B.G. (2018).
Olig2 and Hes regulatory dynamics during motor neuron differentiation revealed by single cell transcriptomics. PLoS biology, 16(2), e2003127. [PubMed:29389974] [PMC] [WorldCat] [DOI] - ↑ 21.0 21.1 21.2
Lee, K.J., Dietrich, P., & Jessell, T.M. (2000).
Genetic ablation reveals that the roof plate is essential for dorsal interneuron specification. Nature, 403(6771), 734-40. [PubMed:10693795] [WorldCat] [DOI] - ↑ 22.0 22.1
Le Dréau, G., Garcia-Campmany, L., Rabadán, M.A., Ferronha, T., Tozer, S., Briscoe, J., & Martí, E. (2012).
Canonical BMP7 activity is required for the generation of discrete neuronal populations in the dorsal spinal cord. Development (Cambridge, England), 139(2), 259-68. [PubMed:22159578] [PMC] [WorldCat] [DOI] - ↑ 23.0 23.1 23.2
Alvarez-Medina, R., Cayuso, J., Okubo, T., Takada, S., & Martí, E. (2008).
Wnt canonical pathway restricts graded Shh/Gli patterning activity through the regulation of Gli3 expression. Development (Cambridge, England), 135(2), 237-47. [PubMed:18057099] [WorldCat] [DOI] - ↑
Andrews, M.G., Del Castillo, L.M., Ochoa-Bolton, E., Yamauchi, K., Smogorzewski, J., & Butler, S.J. (2017).
BMPs direct sensory interneuron identity in the developing spinal cord using signal-specific not morphogenic activities. eLife, 6. [PubMed:28925352] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1
Muroyama, Y., Fujihara, M., Ikeya, M., Kondoh, H., & Takada, S. (2002).
Wnt signaling plays an essential role in neuronal specification of the dorsal spinal cord. Genes & development, 16(5), 548-53. [PubMed:11877374] [PMC] [WorldCat] [DOI] - ↑
Wine-Lee, L., Ahn, K.J., Richardson, R.D., Mishina, Y., Lyons, K.M., & Crenshaw, E.B. (2004).
Signaling through BMP type 1 receptors is required for development of interneuron cell types in the dorsal spinal cord. Development (Cambridge, England), 131(21), 5393-403. [PubMed:15469980] [WorldCat] [DOI] - ↑
Lee, K.J., Mendelsohn, M., & Jessell, T.M. (1998).
Neuronal patterning by BMPs: a requirement for GDF7 in the generation of a discrete class of commissural interneurons in the mouse spinal cord. Genes & development, 12(21), 3394-407. [PubMed:9808626] [PMC] [WorldCat] [DOI] - ↑
Tozer, S., Le Dréau, G., Marti, E., & Briscoe, J. (2013).
Temporal control of BMP signalling determines neuronal subtype identity in the dorsal neural tube. Development (Cambridge, England), 140(7), 1467-74. [PubMed:23462473] [PMC] [WorldCat] [DOI] - ↑ 29.0 29.1 29.2
Schier, A.F., & Needleman, D. (2009).
Developmental biology: Rise of the source-sink model. Nature, 461(7263), 480-1. [PubMed:19779439] [WorldCat] [DOI] - ↑ 30.0 30.1 30.2 30.3
Yan, D., & Lin, X. (2009).
Shaping morphogen gradients by proteoglycans. Cold Spring Harbor perspectives in biology, 1(3), a002493. [PubMed:20066107] [PMC] [WorldCat] [DOI] - ↑
Ramel, M.C., & Hill, C.S. (2012).
Spatial regulation of BMP activity. FEBS letters, 586(14), 1929-41. [PubMed:22710177] [WorldCat] [DOI] - ↑
Müller, P., Rogers, K.W., Yu, S.R., Brand, M., & Schier, A.F. (2013).
Morphogen transport. Development (Cambridge, England), 140(8), 1621-38. [PubMed:23533171] [PMC] [WorldCat] [DOI] - ↑
Kicheva, A., Holtzer, L., Wartlick, O., Schmidt, T., & González-Gaitán, M. (2013).
Quantitative imaging of morphogen gradients in Drosophila imaginal discs. Cold Spring Harbor protocols, 2013(5), 387-403. [PubMed:23637364] [WorldCat] [DOI] - ↑
Kicheva, A., Pantazis, P., Bollenbach, T., Kalaidzidis, Y., Bittig, T., Jülicher, F., & González-Gaitán, M. (2007).
Kinetics of morphogen gradient formation. Science (New York, N.Y.), 315(5811), 521-5. [PubMed:17255514] [WorldCat] [DOI] - ↑
Bollenbach, T., Pantazis, P., Kicheva, A., Bökel, C., González-Gaitán, M., & Jülicher, F. (2008).
Precision of the Dpp gradient. Development (Cambridge, England), 135(6), 1137-46. [PubMed:18296653] [WorldCat] [DOI] - ↑ 36.0 36.1
Capurro, M.I., Xu, P., Shi, W., Li, F., Jia, A., & Filmus, J. (2008).
Glypican-3 inhibits Hedgehog signaling during development by competing with patched for Hedgehog binding. Developmental cell, 14(5), 700-11. [PubMed:18477453] [WorldCat] [DOI] - ↑
Capurro, M.I., Xiang, Y.Y., Lobe, C., & Filmus, J. (2005).
Glypican-3 promotes the growth of hepatocellular carcinoma by stimulating canonical Wnt signaling. Cancer research, 65(14), 6245-54. [PubMed:16024626] [WorldCat] [DOI] - ↑
Gallet, A., Staccini-Lavenant, L., & Thérond, P.P. (2008).
Cellular trafficking of the glypican Dally-like is required for full-strength Hedgehog signaling and wingless transcytosis. Developmental cell, 14(5), 712-25. [PubMed:18477454] [WorldCat] [DOI] - ↑
Yan, D., & Lin, X. (2008).
Opposing roles for glypicans in Hedgehog signalling. Nature cell biology, 10(7), 761-3. [PubMed:18591969] [WorldCat] [DOI] - ↑
Yu, S.R., Burkhardt, M., Nowak, M., Ries, J., Petrásek, Z., Scholpp, S., ..., & Brand, M. (2009).
Fgf8 morphogen gradient forms by a source-sink mechanism with freely diffusing molecules. Nature, 461(7263), 533-6. [PubMed:19741606] [WorldCat] [DOI]