「サイクリックAMP応答配列結合タンパク質」の版間の差分
Junko kurahashi (トーク | 投稿記録) 細 →標的遺伝子 |
|||
| (2人の利用者による、間の28版が非表示) | |||
| 1行目: | 1行目: | ||
<div align="right"> | <div align="right"> | ||
<font size="+1"> | <font size="+1">奥野浩行 </font><br> | ||
''鹿児島大学大学院 医歯学総合研究科''<br> | ''鹿児島大学大学院 医歯学総合研究科''<br> | ||
DOI:<selfdoi /> | DOI:<selfdoi /> 原稿受付日:年月日 原稿完成日:<br> | ||
担当編集委員: | 担当編集委員:<br> | ||
</div> | </div> | ||
英語名:cAMP responsive element binding protein | 英語名:cAMP responsive element binding protein | ||
略称(英語、日本語ともに): CREB | |||
{{box|text= CREBは転写制御因子のひとつであり、ゲノム上の遺伝子転写制御領域に存在するcAMP応答配列(CRE)を介した転写活性化に関わる中心的な核タンパク質である。CREBは生体のほとんどの細胞で恒常的に発現しており、細胞の増殖や分化、生存を含む多様な細胞応答に関与している。CREBの転写活性はさまざまなセカンドメッセンジャー経路を介して制御されており、神経細胞においては名前の由来であるサイクリックAMP依存的な活性調節に加えて、シナプス活性化にともなう細胞内カルシウム濃度上昇や神経栄養因子による受容体活性化などがCREB活性化の主要な因子として働いている。CREBの生理機能は神経系においてよく解析されており、アメフラシ、センチュウ、ショウジョウバエ、マウスなど広範な生物種において長期記憶の形成に必要であることが明らかになっている。また、記憶機能以外にも、アルコールや薬物などへの中毒、さらには鬱などの精神症状などの疾患プロセスにもCREBが関与していることが示唆されている。}} | |||
== CREBとは == | == CREBとは == | ||
脳において神経活動依存的な遺伝子発現は神経[[分化]]や長期[[シナプス可塑性]]、[[記憶]]の固定化・更新などさまざまな局面で重要な役割を果たしている<ref><pubmed>11691980</pubmed></ref>。神経細胞は[[神経伝達物質]]や[[細胞外シグナル分子]]を受け取ると、細胞内シグナル経路を介して[[mRNA]]転写を活性化させ、新たな遺伝子発現プログラムを開始する。このとき、核において細胞内シグナルを受け取る[[転写制御因子]]の一つが[[サイクリックAMP]]応答配列結合タンパク質、またはCREB(cAMP-responsive element binding protein)である<ref name=Altarejos2011><pubmed> 21346730 </pubmed></ref><ref name=Mayr2001><pubmed> 11483993 </pubmed></ref><ref name= Shaywitz1999><pubmed> 10872467 </pubmed></ref>。CREBは生体の様々な種類の細胞に広く発現しており、[[免疫]]細胞の分化・生存や肝臓細胞における[[糖新生]]などに関わることが知られるが<ref name=Altarejos2011/><ref name=Wen2010><pubmed>21084670</pubmed></ref>、神経系においては発生期の神経分化・生存および成熟神経細胞における多様な生理機能が明らかになっている<ref name=Barco2002><pubmed>11893339</pubmed></ref>。 | |||
CREBは核タンパク質であり、常に核内に存在していて一部は[[ゲノム]] | CREBは核タンパク質であり、常に核内に存在していて一部は[[ゲノム]]上に存在するcAMP応答配列CRE(cAMP-responsive element)に結合しているが、その転写促進活性はリン酸化によって厳密に制御されている<ref name= Shaywitz1999/>。CREBはもともと神経内分泌モデル細胞である[[PC12細胞]]において、細胞内cAMP濃度の上昇にともなう転写活性化に関わるDNA結合タンパク質として同定され、cAMP依存的タンパク質キナーゼ[[PKA]]によるリン酸化がその転写活性調節に重要であることが示されたが<ref><pubmed>2573431</pubmed></ref>、その後の研究により多様な細胞内シグナル伝達経路の複数のタンパク質リン酸化酵素によって調節されていることが明らかになった。<ref><pubmed>898022</pubmed></ref><ref><pubmed>1646483</pubmed></ref><ref><pubmed> 1512884 </pubmed></ref> | ||
== 構造 == | == 構造 == | ||
| 26行目: | 24行目: | ||
bZIP:塩基性アミノ酸+ロイシンジッパードメイン(2量体形成およびDNA結合ドメイン)]] | bZIP:塩基性アミノ酸+ロイシンジッパードメイン(2量体形成およびDNA結合ドメイン)]] | ||
CREBはcreb1遺伝子によってコードされている。[[哺乳類]](おそらく[[脊椎動物]])においては狭義のファミリー遺伝子(creb2遺伝子など)は存在しない。しかし、CREBと同じくCREに結合し転写調節を行う転写因子として[[CREM]](CRE modulator)や[[ATF1]](activating transcription factor1)などがあり、これらを含めて広義のCREBファミリーを形成している<ref name=Mayr2001/> | CREBはcreb1遺伝子によってコードされている。[[哺乳類]](おそらく[[脊椎動物]])においては狭義のファミリー遺伝子(creb2遺伝子など)は存在しない。しかし、CREBと同じくCREに結合し転写調節を行う転写因子として[[CREM]](CRE modulator)や[[ATF1]](activating transcription factor1)などがあり、これらを含めて広義のCREBファミリーを形成している<ref name=Mayr2001/>。また、creb1遺伝子からはオルタナティブスプライシングによって数種類のCREB[[アイソフォーム]]が生成される('''図1''')。そのうちのいくつかは転写抑制に関わるが、神経系における主要なアイソフォーム(α型と⊿型)は転写活性化に関わる。これらCREBアイソフォームの機能の差や使い分けについては不明な点が多い。 | ||
CREBおよびそのファミリータンパク質ではいくつかの機能モジュール構造を共通にもつ。中でもCREBファミリーを特徴づけるモジュールとしてキナーゼ誘導ドメイン(Kinase-inducible domain, KID)と呼ばれる60アミノ酸程度のドメイン構造が挙げられる('''図1''')。KID内のセリン残基がリン酸化修飾を受けることにより、CREB結合タンパク質(CBP)(後述)などの他のタンパク質との相互作用が促進され、その結果として転写活性が飛躍的に増加する。KID領域の両端にはグルタミン残基が豊富なQ1およびQ2ドメインがある。これらのドメインも転写活性化に関わる機能を持つと考えられ、例えばQ2ドメインには基本転写因子TFIIDの構成タンパク質の一つTAF4が結合する。CREBのC末側には塩基性アミノ酸・ロイシンジッパー(bZIP)ドメインがあり、このドメインを介してCREBは2量体を形成してCRE特異的にDNAと結合する。 | |||
== 発現 == | == 発現 == | ||
=== | === CREBの組織分布 === | ||
CREBファミリー遺伝子は進化的によく保存されており、単一細胞である酵母から多細胞生物である植物や動物にいたる生物まで広く存在する。哺乳類においてCREBはほとんどの臓器、そしてそれらを構成する多様な細胞で発現しており、広範な生体機能に関わっている。例えば、CREBは[[マクロファージ]]や[[T細胞]]および[[B細胞]]に発現し、分化、増殖、生存を調節することにより免疫系制御に重要な役割を果たしている<ref name=Wen2010/>。 | CREBファミリー遺伝子は進化的によく保存されており、単一細胞である酵母から多細胞生物である植物や動物にいたる生物まで広く存在する。哺乳類においてCREBはほとんどの臓器、そしてそれらを構成する多様な細胞で発現しており、広範な生体機能に関わっている。例えば、CREBは[[マクロファージ]]や[[T細胞]]および[[B細胞]]に発現し、分化、増殖、生存を調節することにより免疫系制御に重要な役割を果たしている<ref name=Wen2010/>。 | ||
神経系においては発生初期の[[神経板]]においてすでにCREBの発現が認められる。成熟した脳においては[[大脳]]、[[小脳]]、[[間脳]]、[[中脳]]などのほぼすべての脳領域でCREBの発現がみられる。また、これら脳領域において神経細胞および[[グリア細胞]]([[アストロサイト]]、[[オリゴデンドロサイト]])ともにCREBを恒常的に発現している。なお、CREBを活性化させる主要なシグナル経路は細胞種によって異なり、このため細胞種あるいは領域特異的なCREBの多様な機能が実現されていると考えられている<ref name=Lonze2002><pubmed>12194863</pubmed></ref>。 | 神経系においては発生初期の[[神経板]]においてすでにCREBの発現が認められる。成熟した脳においては[[大脳]]、[[小脳]]、[[間脳]]、[[中脳]]などのほぼすべての脳領域でCREBの発現がみられる。また、これら脳領域において神経細胞および[[グリア細胞]]([[アストロサイト]]、[[オリゴデンドロサイト]])ともにCREBを恒常的に発現している。なお、CREBを活性化させる主要なシグナル経路は細胞種によって異なり、このため細胞種あるいは領域特異的なCREBの多様な機能が実現されていると考えられている<ref name=Lonze2002><pubmed>12194863</pubmed></ref>。 | ||
=== | === CREBの細胞内分布 === | ||
恒常的に転写されているCREB mRNAは細胞体にて翻訳された後、合成されたCREBタンパク質は速やかに核に移行し、その後は核内に存在する。核内においてCREBは2量体を形成しDNAに結合した状態にあると考えられている。このため[[NF-κB]]や[[エストロゲン受容体]]など他の転写因子にみられるような細胞質-核移行による活性化様式ではなく、リン酸化などのタンパク質[[翻訳後修飾]]がCREBによる転写活性化を制御する主な機構となっている(次項参照)。 | 恒常的に転写されているCREB mRNAは細胞体にて翻訳された後、合成されたCREBタンパク質は速やかに核に移行し、その後は核内に存在する。核内においてCREBは2量体を形成しDNAに結合した状態にあると考えられている。このため[[NF-κB]]や[[エストロゲン受容体]]など他の転写因子にみられるような細胞質-核移行による活性化様式ではなく、リン酸化などのタンパク質[[翻訳後修飾]]がCREBによる転写活性化を制御する主な機構となっている(次項参照)。 | ||
== リン酸化による制御 == | == リン酸化による制御 == | ||
CREBは様々なタンパク質リン酸化酵素によって[[リン酸化修飾]]を受けることにより、転写活性が制御されている。特にKID内に存在する複数のセリン残基がリン酸化されることによりCREBと他のタンパク質との相互作用は正または負に制御される<ref name=Altarejos2011/>。中でも133番目のセリン残基(Ser133) | CREBは様々なタンパク質リン酸化酵素によって[[リン酸化修飾]]を受けることにより、転写活性が制御されている。特にKID内に存在する複数のセリン残基がリン酸化されることによりCREBと他のタンパク質との相互作用は正または負に制御される<ref name=Altarejos2011/>。中でも133番目のセリン残基(Ser133)は転写活性を促進するために最も重要な部位である。発見後初期の1990年代ではSer133はcAMP依存的タンパク質キナーゼ(PKA)によってのみリン酸化されると考えられていたが、その後の研究により神経細胞においては[[カルシウム]]・[[カルモジュリン]]タンパク質キナーゼIVや[[MAPキナーゼ]]ファミリーに属するMSK1/2, RSK1/2などによってもSer133はリン酸化されることが明らかになり、複数の細胞内シグナル経路によって制御されていることが明らかになった<ref name=Lonze2002/><ref><pubmed>12909086</pubmed></ref>。生体において、どのタンパク質リン酸化酵素がCREB Ser133の主要なリン酸化酵素であるかについては組織や細胞の種類、刺激の種類などの要因によって異なる。CREBのSer133はKID領域内に存在し、そのリン酸化は他のタンパク質との相互作用のスイッチとなる。KID領域にはSer133以外にも複数のセリン残基があり(Ser111, Ser121, Ser142/133など)、これらもリン酸化修飾を受けることからCREBに対する巧妙なリン酸化制御機構の存在が示唆されている<ref name=Altarejos2011/>。 | ||
一方、CREBの脱リン酸化による制御に関しては[[タンパク質フォスファターゼ]]1(PP1)やPP2Aの関与が示されているが不明な点が多い。 | |||
== CREBと結合する共活性化因子 == | |||
== | |||
CREBは転写因子であるが単独での転写活性化能力は低く、CREB転写活性の大部分はCREBのKID領域やbZIP領域と結合する共活性化因子([[コアクチベーター]])との相互作用によって担われている。CREBと共活性化因子の相互作用は刺激依存的に制御されている。 | CREBは転写因子であるが単独での転写活性化能力は低く、CREB転写活性の大部分はCREBのKID領域やbZIP領域と結合する共活性化因子([[コアクチベーター]])との相互作用によって担われている。CREBと共活性化因子の相互作用は刺激依存的に制御されている。 | ||
=== | === CREB結合タンパク質(CREB-binding protein, CBP)とp300 === | ||
細胞外からの刺激を受けない基底状態においてもCREBは標的遺伝子のプロモーター領域のCREに結合している<ref name=Impey2004><pubmed> 15620361 </pubmed></ref> <ref name=Zhang2005><pubmed>15753290</pubmed></ref>。刺激によってゲノムに結合しているCREBのKID領域のSer133がリン酸化修飾を受けると、CREB結合タンパク質CBPとの親和性が上昇し、CREBとCBPが複合体を形成する<ref name=Mayr2001/>。CBPは[[ヒストンアセチル化活性]]を持つため、ヒストンとDNAとの相互作用を弱めてCREB結合領域周辺の[[クロマチン]]構造を変化させて転写を促進する。また、CREB-CBP複合体は[[RNAポリメラーゼ]]II複合体との相互作用を介して転写を増強する。なお、脊椎動物ではCBP遺伝子(CREBBP)のパラローグとして[[p300]]遺伝子(EP300)が存在する。p300はCBPと構造的に類似しており、CREBに対してCBP同様に作用すると考えらえるが、生体におけるCBP/p300パラローグの機能差の有無や生理的意義については不明な点が多い。 | |||
=== CREBコアクチベーター(CREB-regulated transcription coactivators, CRTCs) === | |||
CREBのbZIP領域は2量体形成およびDNA結合に関わるドメインであるが、この領域に結合する共活性化因子としてCREBコアクチベーターCRTCsが知られる<ref name=Conkright2003><pubmed> 14536081 </pubmed></ref>。哺乳類においてCRTCsは3種(CRTC1、CRTC2およびCRTC3)からなる遺伝子ファミリーを形成しているが、神経系では主にCRTC1およびCRTC2が発現している。基底状態においてCRTCsはリン酸化修飾されており、14-3-3タンパク質と複合体を形成して細胞質に存在している。ここにcAMPやCa<sup>2+</sup>濃度上昇などの刺激が入ると、[[カルシニューリン]]によってCRTCsは脱リン酸化されて14-3-3から解離し、速やかに核内に移行してCREBと結合する。CRTCのbZIP領域への結合はCREBのCRE結合活性を増強し、また、CREB-CRTC複合体がTFIID複合体と効率的に相互作用することにより、CREBを介した転写活性を増強すると考えられるが<ref name=Conkright2003/>、詳細は不明である。なお、CREB-CRTC相互作用はKID領域のSer133リン酸化とは関係なく制御されており、CBP/p300とは独立した刺激依存的なCREB転写活性機構であると考えられている<ref><pubmed>17476304</pubmed></ref><ref><pubmed>15454081</pubmed></ref>。 | |||
=== CREBコアクチベーター | |||
== 標的遺伝子 == | == 標的遺伝子 == | ||
CREBは進化的に酵母、センチュウ、アメフラシ、ショウジョウバエから脊椎動物まで保存されている転写因子であり、神経細胞において多くの遺伝子の発現を調節している。CREBファミリータンパク質はCRE配列(-TGACGTCA-、またはその半分のTGACG or CGTCA)に結合し、CREをプロモーター領域にもつ遺伝子の転写を活性化させる<ref><pubmed>2885756</pubmed></ref>。特に、CRE配列がTATAボックス近傍にある遺伝子ではCREBを介した刺激依存的な転写誘導が効率的に引き起こされる。 | |||
CREBによって転写が制御される“CREB標的遺伝子”はCREBの発見以来精力的に探索されてきたが、近年の網羅的解析技術の発展によってゲノムワイドに包括的な解析が数多く行われている<ref name=Impey2004/><ref name=Zhang2005/><ref name=Kim2010><pubmed> 20393465 </pubmed></ref>。これらの研究によるとゲノム中には5000以上のCREB結合部位が存在し、多数の遺伝子の発現制御に関わっている可能性が示唆されている。例えば、細胞にcAMP刺激を加えると1000以上の遺伝子でmRNA発現が上昇するが、このうちの約半分の遺伝子においてCREB結合部位が存在する。しかしながら、CREB標的遺伝子は刺激や細胞の種類によって大きく異なる。神経活動依存的に発現量が変動する遺伝子群にはCREB標的遺伝子が特に多く含まれていることが報告されている('''表1''' | CREBによって転写が制御される“CREB標的遺伝子”はCREBの発見以来精力的に探索されてきたが、近年の網羅的解析技術の発展によってゲノムワイドに包括的な解析が数多く行われている<ref name=Impey2004/><ref name=Zhang2005/><ref name=Kim2010><pubmed> 20393465 </pubmed></ref>。これらの研究によるとゲノム中には5000以上のCREB結合部位が存在し、多数の遺伝子の発現制御に関わっている可能性が示唆されている。例えば、細胞にcAMP刺激を加えると1000以上の遺伝子でmRNA発現が上昇するが、このうちの約半分の遺伝子においてCREB結合部位が存在する。しかしながら、CREB標的遺伝子は刺激や細胞の種類によって大きく異なる。神経活動依存的に発現量が変動する遺伝子群にはCREB標的遺伝子が特に多く含まれていることが報告されている('''表1''')。これらのCREB標的遺伝子は古典的な前初期遺伝子を数多く含む。 | ||
興味深いことに、遺伝子の転写開始部位に近いプロモーター領域においては刺激の有無にかかわらずCREBはゲノムのCRE配列に結合しているが、遠位に存在するエンハンサー領域においてはCREB-CRE結合は刺激依存的に増強することが示されており<ref name=Kim2010/>、CREの存在する位置によって多様なCREBの転写制御機構が示唆されている。 | 興味深いことに、遺伝子の転写開始部位に近いプロモーター領域においては刺激の有無にかかわらずCREBはゲノムのCRE配列に結合しているが、遠位に存在するエンハンサー領域においてはCREB-CRE結合は刺激依存的に増強することが示されており<ref name=Kim2010/>、CREの存在する位置によって多様なCREBの転写制御機構が示唆されている。 | ||
{| class="wikitable" | |||
{| class="wikitable" | {| class="wikitable" | ||
|+ 表1 神経細胞におけるCREB標的遺伝子の一部 | |+ 表1 神経細胞におけるCREB標的遺伝子の一部 | ||
|- | |- | ||
| 転写制御因子 /核内受容体 || | | 転写制御因子 /核内受容体 || Btg2, Cebpb, Crem, Egr1, Fos, Fral, Fra2, Fosb, Hes1, Jun, Junb, Jund, Nr4a1, Nr4a2, Per1, Per2, Rb1 | ||
|- | |- | ||
| シナプス シナプス ・可塑性関連タンパク質 | | | シナプス シナプス ・可塑性関連タンパク質 || AchE, Arc, Bdnf, Inhba, Homer1, Ntrk2, Nptx2, Syn 1 | ||
|- | |- | ||
| シグナル伝達 || | | シグナル伝達 || Cdk5, Nf1, Nos1, Pgs2/Cox2, Sgk1 | ||
|- | |- | ||
| 神経ペブチド・神経伝達物質関連 || | | 神経ペブチド・神経伝達物質関連 || Adcyap1, Adrb1, Adrb2, Avp, Crh, Ddh, Gabral, Gal, Galr1, Pdyn, Penk, Pnoc, Sst, Sstr2, Tacr1, Vip | ||
|} | |||
プロモータ領域またはエンハサー領域にCRE配列が存在し、CREBによる転写調節を受けていることが報告されている、あるいは受ける可能性が高い遺伝子(マウスにおける遺伝子名)の一部を記した。 | |||
|} | |} | ||
== | == 長期シナプス可塑性および長期記憶形成におけるCREBの機能 == | ||
神経系におけるCREBの生理機能は、様々な生物種において様々なCREB機能阻害実験によって解析されている<ref><pubmed>9530494</pubmed></ref> | 神経系におけるCREBの生理機能は、様々な生物種において様々なCREB機能阻害実験によって解析されている<ref><pubmed>9530494</pubmed></ref>[20]。アメフラシにおいてエラ引き込み反射の長期増強には長期促通(long-term facilitation)と呼ばれる長期シナプス可塑性が関与しているが、この時、神経細胞に抗CREB抗体やデコイとなるCRE配列を注入すると長期促通が阻害される<ref><pubmed>2141668</pubmed></ref><ref><pubmed>9428516</pubmed></ref>[21, 22]。ショウジョウバエにおいては、ドミナントネガティブ型CREBを過剰発現させることによってCREBの機能阻害を引き起こすと、匂いと電気ショックの連合学習後に短期記憶は獲得できるものの、長期記憶は障害される<ref><pubmed>7923376</pubmed></ref>[23]。同様に、CREB欠損マウスにおける文脈依存的な恐怖条件付け課題では短期記憶は形成されるが長期記憶に障害があることが報告されている<ref><pubmed>7923378</pubmed></ref>[24]。ラットを用いた空間学習課題の実験では、CREBのアンチセンスオリゴを海馬に局所注入してCREBの発現を抑制すると、短期記憶は正常に形成されるが、長期記憶には障害が生じる<ref><pubmed>9122258</pubmed></ref>[25]。 | ||
マウスやラットにおいて海馬神経細胞におけるシナプスの長期増強(long-term potentiation, LTP)は海馬依存的な学習および記憶形成に重要な役割を果たしていると考えられている。特に、数時間以上持続する後期LTP(late-phase LTP)は長期記憶形成との関連が強く示唆されている。海馬神経細胞に活性化型CREBを発現させると、通常は後期LTPが起きない刺激条件でも後期LTPが成立することから、CREBあるいはCREBによって発現制御される遺伝子産物は海馬における後期LTPの成立に重要な役割を果たしていると考えられる<ref name=Barco2002/>[6]。 | |||
== 疾患との関わり == | == 疾患との関わり == | ||
CREBは長期記憶以外にも多様な神経機能において中核的役割を果しており、その機能破綻は様々な疾患の原因となりうる。CREBはCBPあるいはp300を介して転写調節を行うが、CBP/ | CREBは長期記憶以外にも多様な神経機能において中核的役割を果しており、その機能破綻は様々な疾患の原因となりうる。CREBはCBPあるいはp300を介して転写調節を行うが、CBP/p300の変異はヒトの精神運動発達障害を伴うRubinstein-Taybi症候群の原因となることが明らかになっている<ref><pubmed>7630403</pubmed></ref>[26]。また、ポリグルタミン病早期における病態の一つは、CREB依存的転写の阻害である可能性が指摘されている<ref><pubmed> 10958659 </pubmed></ref>[27]。また、アルコール中毒やコカインなどの薬物中毒となる過程においてもCREBによる転写調節が役割を果たしていることが動物実験において示されている<ref><pubmed> 15982754 </pubmed></ref>[28]。さらには、鬱病の治療薬である抗うつ剤の多くはCREB活性を上昇させることが示されており、鬱病におけるCREBの転写活性の低下の可能性が示唆されている<ref><pubmed> 12486182</pubmed></ref>[29]。しかし逆に、動物実験において側坐核などの特定の神経核においてCREB活性を強制的に上昇させた場合に鬱様の行動が惹起されることから<ref><pubmed>11549750</pubmed></ref>[30]、正常な脳機能を保つためには適切なレベルのCREB依存的な転写調節が必要であると考えられる。 | ||
== 参考文献 == | == 参考文献 == | ||
<references/> | <references/> | ||
2018年5月11日 (金) 14:51時点における版
奥野浩行
鹿児島大学大学院 医歯学総合研究科
DOI:10.14931/bsd.3025 原稿受付日:年月日 原稿完成日:
担当編集委員:
英語名:cAMP responsive element binding protein
略称(英語、日本語ともに): CREB
CREBは転写制御因子のひとつであり、ゲノム上の遺伝子転写制御領域に存在するcAMP応答配列(CRE)を介した転写活性化に関わる中心的な核タンパク質である。CREBは生体のほとんどの細胞で恒常的に発現しており、細胞の増殖や分化、生存を含む多様な細胞応答に関与している。CREBの転写活性はさまざまなセカンドメッセンジャー経路を介して制御されており、神経細胞においては名前の由来であるサイクリックAMP依存的な活性調節に加えて、シナプス活性化にともなう細胞内カルシウム濃度上昇や神経栄養因子による受容体活性化などがCREB活性化の主要な因子として働いている。CREBの生理機能は神経系においてよく解析されており、アメフラシ、センチュウ、ショウジョウバエ、マウスなど広範な生物種において長期記憶の形成に必要であることが明らかになっている。また、記憶機能以外にも、アルコールや薬物などへの中毒、さらには鬱などの精神症状などの疾患プロセスにもCREBが関与していることが示唆されている。
CREBとは
脳において神経活動依存的な遺伝子発現は神経分化や長期シナプス可塑性、記憶の固定化・更新などさまざまな局面で重要な役割を果たしている[1]。神経細胞は神経伝達物質や細胞外シグナル分子を受け取ると、細胞内シグナル経路を介してmRNA転写を活性化させ、新たな遺伝子発現プログラムを開始する。このとき、核において細胞内シグナルを受け取る転写制御因子の一つがサイクリックAMP応答配列結合タンパク質、またはCREB(cAMP-responsive element binding protein)である[2][3][4]。CREBは生体の様々な種類の細胞に広く発現しており、免疫細胞の分化・生存や肝臓細胞における糖新生などに関わることが知られるが[2][5]、神経系においては発生期の神経分化・生存および成熟神経細胞における多様な生理機能が明らかになっている[6]。
CREBは核タンパク質であり、常に核内に存在していて一部はゲノム上に存在するcAMP応答配列CRE(cAMP-responsive element)に結合しているが、その転写促進活性はリン酸化によって厳密に制御されている[4]。CREBはもともと神経内分泌モデル細胞であるPC12細胞において、細胞内cAMP濃度の上昇にともなう転写活性化に関わるDNA結合タンパク質として同定され、cAMP依存的タンパク質キナーゼPKAによるリン酸化がその転写活性調節に重要であることが示されたが[7]、その後の研究により多様な細胞内シグナル伝達経路の複数のタンパク質リン酸化酵素によって調節されていることが明らかになった。[8][9][10]
構造
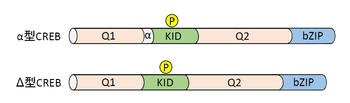
Q1, Q2:グルタミン酸残基が多い転写活性化ドメイン
α:オルタナティブスプライシングにより挿入される14アミノ酸残基からなるドメイン
KID:リン酸化制御をうけるドメイリン bZIP:塩基性アミノ酸+ロイシンジッパードメイン(2量体形成およびDNA結合ドメイン)
CREBはcreb1遺伝子によってコードされている。哺乳類(おそらく脊椎動物)においては狭義のファミリー遺伝子(creb2遺伝子など)は存在しない。しかし、CREBと同じくCREに結合し転写調節を行う転写因子としてCREM(CRE modulator)やATF1(activating transcription factor1)などがあり、これらを含めて広義のCREBファミリーを形成している[3]。また、creb1遺伝子からはオルタナティブスプライシングによって数種類のCREBアイソフォームが生成される(図1)。そのうちのいくつかは転写抑制に関わるが、神経系における主要なアイソフォーム(α型と⊿型)は転写活性化に関わる。これらCREBアイソフォームの機能の差や使い分けについては不明な点が多い。
CREBおよびそのファミリータンパク質ではいくつかの機能モジュール構造を共通にもつ。中でもCREBファミリーを特徴づけるモジュールとしてキナーゼ誘導ドメイン(Kinase-inducible domain, KID)と呼ばれる60アミノ酸程度のドメイン構造が挙げられる(図1)。KID内のセリン残基がリン酸化修飾を受けることにより、CREB結合タンパク質(CBP)(後述)などの他のタンパク質との相互作用が促進され、その結果として転写活性が飛躍的に増加する。KID領域の両端にはグルタミン残基が豊富なQ1およびQ2ドメインがある。これらのドメインも転写活性化に関わる機能を持つと考えられ、例えばQ2ドメインには基本転写因子TFIIDの構成タンパク質の一つTAF4が結合する。CREBのC末側には塩基性アミノ酸・ロイシンジッパー(bZIP)ドメインがあり、このドメインを介してCREBは2量体を形成してCRE特異的にDNAと結合する。
発現
CREBの組織分布
CREBファミリー遺伝子は進化的によく保存されており、単一細胞である酵母から多細胞生物である植物や動物にいたる生物まで広く存在する。哺乳類においてCREBはほとんどの臓器、そしてそれらを構成する多様な細胞で発現しており、広範な生体機能に関わっている。例えば、CREBはマクロファージやT細胞およびB細胞に発現し、分化、増殖、生存を調節することにより免疫系制御に重要な役割を果たしている[5]。
神経系においては発生初期の神経板においてすでにCREBの発現が認められる。成熟した脳においては大脳、小脳、間脳、中脳などのほぼすべての脳領域でCREBの発現がみられる。また、これら脳領域において神経細胞およびグリア細胞(アストロサイト、オリゴデンドロサイト)ともにCREBを恒常的に発現している。なお、CREBを活性化させる主要なシグナル経路は細胞種によって異なり、このため細胞種あるいは領域特異的なCREBの多様な機能が実現されていると考えられている[11]。
CREBの細胞内分布
恒常的に転写されているCREB mRNAは細胞体にて翻訳された後、合成されたCREBタンパク質は速やかに核に移行し、その後は核内に存在する。核内においてCREBは2量体を形成しDNAに結合した状態にあると考えられている。このためNF-κBやエストロゲン受容体など他の転写因子にみられるような細胞質-核移行による活性化様式ではなく、リン酸化などのタンパク質翻訳後修飾がCREBによる転写活性化を制御する主な機構となっている(次項参照)。
リン酸化による制御
CREBは様々なタンパク質リン酸化酵素によってリン酸化修飾を受けることにより、転写活性が制御されている。特にKID内に存在する複数のセリン残基がリン酸化されることによりCREBと他のタンパク質との相互作用は正または負に制御される[2]。中でも133番目のセリン残基(Ser133)は転写活性を促進するために最も重要な部位である。発見後初期の1990年代ではSer133はcAMP依存的タンパク質キナーゼ(PKA)によってのみリン酸化されると考えられていたが、その後の研究により神経細胞においてはカルシウム・カルモジュリンタンパク質キナーゼIVやMAPキナーゼファミリーに属するMSK1/2, RSK1/2などによってもSer133はリン酸化されることが明らかになり、複数の細胞内シグナル経路によって制御されていることが明らかになった[11][12]。生体において、どのタンパク質リン酸化酵素がCREB Ser133の主要なリン酸化酵素であるかについては組織や細胞の種類、刺激の種類などの要因によって異なる。CREBのSer133はKID領域内に存在し、そのリン酸化は他のタンパク質との相互作用のスイッチとなる。KID領域にはSer133以外にも複数のセリン残基があり(Ser111, Ser121, Ser142/133など)、これらもリン酸化修飾を受けることからCREBに対する巧妙なリン酸化制御機構の存在が示唆されている[2]。
一方、CREBの脱リン酸化による制御に関してはタンパク質フォスファターゼ1(PP1)やPP2Aの関与が示されているが不明な点が多い。
CREBと結合する共活性化因子
CREBは転写因子であるが単独での転写活性化能力は低く、CREB転写活性の大部分はCREBのKID領域やbZIP領域と結合する共活性化因子(コアクチベーター)との相互作用によって担われている。CREBと共活性化因子の相互作用は刺激依存的に制御されている。
CREB結合タンパク質(CREB-binding protein, CBP)とp300
細胞外からの刺激を受けない基底状態においてもCREBは標的遺伝子のプロモーター領域のCREに結合している[13] [14]。刺激によってゲノムに結合しているCREBのKID領域のSer133がリン酸化修飾を受けると、CREB結合タンパク質CBPとの親和性が上昇し、CREBとCBPが複合体を形成する[3]。CBPはヒストンアセチル化活性を持つため、ヒストンとDNAとの相互作用を弱めてCREB結合領域周辺のクロマチン構造を変化させて転写を促進する。また、CREB-CBP複合体はRNAポリメラーゼII複合体との相互作用を介して転写を増強する。なお、脊椎動物ではCBP遺伝子(CREBBP)のパラローグとしてp300遺伝子(EP300)が存在する。p300はCBPと構造的に類似しており、CREBに対してCBP同様に作用すると考えらえるが、生体におけるCBP/p300パラローグの機能差の有無や生理的意義については不明な点が多い。
CREBコアクチベーター(CREB-regulated transcription coactivators, CRTCs)
CREBのbZIP領域は2量体形成およびDNA結合に関わるドメインであるが、この領域に結合する共活性化因子としてCREBコアクチベーターCRTCsが知られる[15]。哺乳類においてCRTCsは3種(CRTC1、CRTC2およびCRTC3)からなる遺伝子ファミリーを形成しているが、神経系では主にCRTC1およびCRTC2が発現している。基底状態においてCRTCsはリン酸化修飾されており、14-3-3タンパク質と複合体を形成して細胞質に存在している。ここにcAMPやCa2+濃度上昇などの刺激が入ると、カルシニューリンによってCRTCsは脱リン酸化されて14-3-3から解離し、速やかに核内に移行してCREBと結合する。CRTCのbZIP領域への結合はCREBのCRE結合活性を増強し、また、CREB-CRTC複合体がTFIID複合体と効率的に相互作用することにより、CREBを介した転写活性を増強すると考えられるが[15]、詳細は不明である。なお、CREB-CRTC相互作用はKID領域のSer133リン酸化とは関係なく制御されており、CBP/p300とは独立した刺激依存的なCREB転写活性機構であると考えられている[16][17]。
標的遺伝子
CREBは進化的に酵母、センチュウ、アメフラシ、ショウジョウバエから脊椎動物まで保存されている転写因子であり、神経細胞において多くの遺伝子の発現を調節している。CREBファミリータンパク質はCRE配列(-TGACGTCA-、またはその半分のTGACG or CGTCA)に結合し、CREをプロモーター領域にもつ遺伝子の転写を活性化させる[18]。特に、CRE配列がTATAボックス近傍にある遺伝子ではCREBを介した刺激依存的な転写誘導が効率的に引き起こされる。
CREBによって転写が制御される“CREB標的遺伝子”はCREBの発見以来精力的に探索されてきたが、近年の網羅的解析技術の発展によってゲノムワイドに包括的な解析が数多く行われている[13][14][19]。これらの研究によるとゲノム中には5000以上のCREB結合部位が存在し、多数の遺伝子の発現制御に関わっている可能性が示唆されている。例えば、細胞にcAMP刺激を加えると1000以上の遺伝子でmRNA発現が上昇するが、このうちの約半分の遺伝子においてCREB結合部位が存在する。しかしながら、CREB標的遺伝子は刺激や細胞の種類によって大きく異なる。神経活動依存的に発現量が変動する遺伝子群にはCREB標的遺伝子が特に多く含まれていることが報告されている(表1)。これらのCREB標的遺伝子は古典的な前初期遺伝子を数多く含む。
興味深いことに、遺伝子の転写開始部位に近いプロモーター領域においては刺激の有無にかかわらずCREBはゲノムのCRE配列に結合しているが、遠位に存在するエンハンサー領域においてはCREB-CRE結合は刺激依存的に増強することが示されており[19]、CREの存在する位置によって多様なCREBの転写制御機構が示唆されている。
| 転写制御因子 /核内受容体 | Btg2, Cebpb, Crem, Egr1, Fos, Fral, Fra2, Fosb, Hes1, Jun, Junb, Jund, Nr4a1, Nr4a2, Per1, Per2, Rb1 |
| シナプス シナプス ・可塑性関連タンパク質 | AchE, Arc, Bdnf, Inhba, Homer1, Ntrk2, Nptx2, Syn 1 |
| シグナル伝達 | Cdk5, Nf1, Nos1, Pgs2/Cox2, Sgk1 |
| 神経ペブチド・神経伝達物質関連 | Adcyap1, Adrb1, Adrb2, Avp, Crh, Ddh, Gabral, Gal, Galr1, Pdyn, Penk, Pnoc, Sst, Sstr2, Tacr1, Vip |
プロモータ領域またはエンハサー領域にCRE配列が存在し、CREBによる転写調節を受けていることが報告されている、あるいは受ける可能性が高い遺伝子(マウスにおける遺伝子名)の一部を記した。
長期シナプス可塑性および長期記憶形成におけるCREBの機能
神経系におけるCREBの生理機能は、様々な生物種において様々なCREB機能阻害実験によって解析されている[20][20]。アメフラシにおいてエラ引き込み反射の長期増強には長期促通(long-term facilitation)と呼ばれる長期シナプス可塑性が関与しているが、この時、神経細胞に抗CREB抗体やデコイとなるCRE配列を注入すると長期促通が阻害される[21][22][21, 22]。ショウジョウバエにおいては、ドミナントネガティブ型CREBを過剰発現させることによってCREBの機能阻害を引き起こすと、匂いと電気ショックの連合学習後に短期記憶は獲得できるものの、長期記憶は障害される[23][23]。同様に、CREB欠損マウスにおける文脈依存的な恐怖条件付け課題では短期記憶は形成されるが長期記憶に障害があることが報告されている[24][24]。ラットを用いた空間学習課題の実験では、CREBのアンチセンスオリゴを海馬に局所注入してCREBの発現を抑制すると、短期記憶は正常に形成されるが、長期記憶には障害が生じる[25][25]。
マウスやラットにおいて海馬神経細胞におけるシナプスの長期増強(long-term potentiation, LTP)は海馬依存的な学習および記憶形成に重要な役割を果たしていると考えられている。特に、数時間以上持続する後期LTP(late-phase LTP)は長期記憶形成との関連が強く示唆されている。海馬神経細胞に活性化型CREBを発現させると、通常は後期LTPが起きない刺激条件でも後期LTPが成立することから、CREBあるいはCREBによって発現制御される遺伝子産物は海馬における後期LTPの成立に重要な役割を果たしていると考えられる[6][6]。
疾患との関わり
CREBは長期記憶以外にも多様な神経機能において中核的役割を果しており、その機能破綻は様々な疾患の原因となりうる。CREBはCBPあるいはp300を介して転写調節を行うが、CBP/p300の変異はヒトの精神運動発達障害を伴うRubinstein-Taybi症候群の原因となることが明らかになっている[26][26]。また、ポリグルタミン病早期における病態の一つは、CREB依存的転写の阻害である可能性が指摘されている[27][27]。また、アルコール中毒やコカインなどの薬物中毒となる過程においてもCREBによる転写調節が役割を果たしていることが動物実験において示されている[28][28]。さらには、鬱病の治療薬である抗うつ剤の多くはCREB活性を上昇させることが示されており、鬱病におけるCREBの転写活性の低下の可能性が示唆されている[29][29]。しかし逆に、動物実験において側坐核などの特定の神経核においてCREB活性を強制的に上昇させた場合に鬱様の行動が惹起されることから[30][30]、正常な脳機能を保つためには適切なレベルのCREB依存的な転写調節が必要であると考えられる。
参考文献
- ↑
Kandel, E.R. (2001).
The molecular biology of memory storage: a dialogue between genes and synapses. Science (New York, N.Y.), 294(5544), 1030-8. [PubMed:11691980] [WorldCat] [DOI] - ↑ 2.0 2.1 2.2 2.3
Altarejos, J.Y., & Montminy, M. (2011).
CREB and the CRTC co-activators: sensors for hormonal and metabolic signals. Nature reviews. Molecular cell biology, 12(3), 141-51. [PubMed:21346730] [PMC] [WorldCat] [DOI] - ↑ 3.0 3.1 3.2
Mayr, B., & Montminy, M. (2001).
Transcriptional regulation by the phosphorylation-dependent factor CREB. Nature reviews. Molecular cell biology, 2(8), 599-609. [PubMed:11483993] [WorldCat] [DOI] - ↑ 4.0 4.1
Shaywitz, A.J., & Greenberg, M.E. (1999).
CREB: a stimulus-induced transcription factor activated by a diverse array of extracellular signals. Annual review of biochemistry, 68, 821-61. [PubMed:10872467] [WorldCat] [DOI] - ↑ 5.0 5.1
Wen, A.Y., Sakamoto, K.M., & Miller, L.S. (2010).
The role of the transcription factor CREB in immune function. Journal of immunology (Baltimore, Md. : 1950), 185(11), 6413-9. [PubMed:21084670] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Barco, A., Alarcon, J.M., & Kandel, E.R. (2002).
Expression of constitutively active CREB protein facilitates the late phase of long-term potentiation by enhancing synaptic capture. Cell, 108(5), 689-703. [PubMed:11893339] [WorldCat] [DOI] - ↑
Gonzalez, G.A., & Montminy, M.R. (1989).
Cyclic AMP stimulates somatostatin gene transcription by phosphorylation of CREB at serine 133. Cell, 59(4), 675-80. [PubMed:2573431] [WorldCat] [DOI] - ↑
Simonian, S.J., Stuart, F.P., Hill, J.L., & Mahajan, S.K. (1977).
Conversion of a Scribner shunt to an arteriovenous fistula for chronic dialysis. Surgery, 82(4), 448-51. [PubMed:898022] [WorldCat] - ↑
Sheng, M., Thompson, M.A., & Greenberg, M.E. (1991).
CREB: a Ca(2+)-regulated transcription factor phosphorylated by calmodulin-dependent kinases. Science (New York, N.Y.), 252(5011), 1427-30. [PubMed:1646483] [WorldCat] [DOI] - ↑
Aohagi, Y., Shibahara, T., Machida, N., Yamaga, Y., Kagota, K., & Hayashi, T. (1992).
Natural infections of Clinostomum complanatum (Trematoda: Clinostomatidae) in wild herons and egrets, Tottori Prefecture, Japan. Journal of wildlife diseases, 28(3), 470-1. [PubMed:1512884] [WorldCat] [DOI] - ↑ 11.0 11.1
Lonze, B.E., & Ginty, D.D. (2002).
Function and regulation of CREB family transcription factors in the nervous system. Neuron, 35(4), 605-23. [PubMed:12194863] [WorldCat] [DOI] - ↑
Bito, H., & Takemoto-Kimura, S. (2003).
Ca(2+)/CREB/CBP-dependent gene regulation: a shared mechanism critical in long-term synaptic plasticity and neuronal survival. Cell calcium, 34(4-5), 425-30. [PubMed:12909086] [WorldCat] [DOI] - ↑ 13.0 13.1
Impey, S., McCorkle, S.R., Cha-Molstad, H., Dwyer, J.M., Yochum, G.S., Boss, J.M., ..., & Goodman, R.H. (2004).
Defining the CREB regulon: a genome-wide analysis of transcription factor regulatory regions. Cell, 119(7), 1041-54. [PubMed:15620361] [WorldCat] [DOI] - ↑ 14.0 14.1
Zhang, X., Odom, D.T., Koo, S.H., Conkright, M.D., Canettieri, G., Best, J., ..., & Montminy, M. (2005).
Genome-wide analysis of cAMP-response element binding protein occupancy, phosphorylation, and target gene activation in human tissues. Proceedings of the National Academy of Sciences of the United States of America, 102(12), 4459-64. [PubMed:15753290] [PMC] [WorldCat] [DOI] - ↑ 15.0 15.1
Conkright, M.D., Canettieri, G., Screaton, R., Guzman, E., Miraglia, L., Hogenesch, J.B., & Montminy, M. (2003).
TORCs: transducers of regulated CREB activity. Molecular cell, 12(2), 413-23. [PubMed:14536081] [WorldCat] [DOI] - ↑
Ravnskjaer, K., Kester, H., Liu, Y., Zhang, X., Lee, D., Yates, J.R., & Montminy, M. (2007).
Cooperative interactions between CBP and TORC2 confer selectivity to CREB target gene expression. The EMBO journal, 26(12), 2880-9. [PubMed:17476304] [PMC] [WorldCat] [DOI] - ↑
Screaton, R.A., Conkright, M.D., Katoh, Y., Best, J.L., Canettieri, G., Jeffries, S., ..., & Montminy, M. (2004).
The CREB coactivator TORC2 functions as a calcium- and cAMP-sensitive coincidence detector. Cell, 119(1), 61-74. [PubMed:15454081] [WorldCat] [DOI] - ↑
Montminy, M.R., & Bilezikjian, L.M. (1987).
Binding of a nuclear protein to the cyclic-AMP response element of the somatostatin gene. Nature, 328(6126), 175-8. [PubMed:2885756] [WorldCat] [DOI] - ↑ 19.0 19.1
Kim, T.K., Hemberg, M., Gray, J.M., Costa, A.M., Bear, D.M., Wu, J., ..., & Greenberg, M.E. (2010).
Widespread transcription at neuronal activity-regulated enhancers. Nature, 465(7295), 182-7. [PubMed:20393465] [PMC] [WorldCat] [DOI] - ↑
Silva, A.J., Kogan, J.H., Frankland, P.W., & Kida, S. (1998).
CREB and memory. Annual review of neuroscience, 21, 127-48. [PubMed:9530494] [WorldCat] [DOI] - ↑
Dash, P.K., Hochner, B., & Kandel, E.R. (1990).
Injection of the cAMP-responsive element into the nucleus of Aplysia sensory neurons blocks long-term facilitation. Nature, 345(6277), 718-21. [PubMed:2141668] [WorldCat] [DOI] - ↑
Martin, K.C., Casadio, A., Zhu, H., Yaping, E., Rose, J.C., Chen, M., ..., & Kandel, E.R. (1997).
Synapse-specific, long-term facilitation of aplysia sensory to motor synapses: a function for local protein synthesis in memory storage. Cell, 91(7), 927-38. [PubMed:9428516] [WorldCat] [DOI] - ↑
Yin, J.C., Wallach, J.S., Del Vecchio, M., Wilder, E.L., Zhou, H., Quinn, W.G., & Tully, T. (1994).
Induction of a dominant negative CREB transgene specifically blocks long-term memory in Drosophila. Cell, 79(1), 49-58. [PubMed:7923376] [WorldCat] [DOI] - ↑
Bourtchuladze, R., Frenguelli, B., Blendy, J., Cioffi, D., Schutz, G., & Silva, A.J. (1994).
Deficient long-term memory in mice with a targeted mutation of the cAMP-responsive element-binding protein. Cell, 79(1), 59-68. [PubMed:7923378] [WorldCat] [DOI] - ↑
Guzowski, J.F., & McGaugh, J.L. (1997).
Antisense oligodeoxynucleotide-mediated disruption of hippocampal cAMP response element binding protein levels impairs consolidation of memory for water maze training. Proceedings of the National Academy of Sciences of the United States of America, 94(6), 2693-8. [PubMed:9122258] [PMC] [WorldCat] [DOI] - ↑
Petrij, F., Giles, R.H., Dauwerse, H.G., Saris, J.J., Hennekam, R.C., Masuno, M., ..., & Peters, D.J. (1995).
Rubinstein-Taybi syndrome caused by mutations in the transcriptional co-activator CBP. Nature, 376(6538), 348-51. [PubMed:7630403] [WorldCat] [DOI] - ↑
McCampbell, A., Taylor, J.P., Taye, A.A., Robitschek, J., Li, M., Walcott, J., ..., & Fischbeck, K.H. (2000).
CREB-binding protein sequestration by expanded polyglutamine. Human molecular genetics, 9(14), 2197-202. [PubMed:10958659] [WorldCat] [DOI] - ↑
Carlezon, W.A., Duman, R.S., & Nestler, E.J. (2005).
The many faces of CREB. Trends in neurosciences, 28(8), 436-45. [PubMed:15982754] [WorldCat] [DOI] - ↑
Newton, S.S., Thome, J., Wallace, T.L., Shirayama, Y., Schlesinger, L., Sakai, N., ..., & Duman, R.S. (2002).
Inhibition of cAMP response element-binding protein or dynorphin in the nucleus accumbens produces an antidepressant-like effect. The Journal of neuroscience : the official journal of the Society for Neuroscience, 22(24), 10883-90. [PubMed:12486182] [PMC] [WorldCat] - ↑
Pliakas, A.M., Carlson, R.R., Neve, R.L., Konradi, C., Nestler, E.J., & Carlezon, W.A. (2001).
Altered responsiveness to cocaine and increased immobility in the forced swim test associated with elevated cAMP response element-binding protein expression in nucleus accumbens. The Journal of neuroscience : the official journal of the Society for Neuroscience, 21(18), 7397-403. [PubMed:11549750] [PMC] [WorldCat]