「Myocyte enhancer factor-2」の版間の差分
細編集の要約なし |
細編集の要約なし |
||
| 2行目: | 2行目: | ||
<font size="+1">[http://researchmap.jp/hiroyukiokuno 奥野 浩行]</font><br> | <font size="+1">[http://researchmap.jp/hiroyukiokuno 奥野 浩行]</font><br> | ||
''東京大学''<br> | ''東京大学''<br> | ||
DOI XXXX/XXXX 原稿受付日:2012年12月25日 原稿完成日:2013年4月19日<br> | |||
担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | 担当編集委員:[http://researchmap.jp/michisukeyuzaki 柚崎 通介](慶應義塾大学 医学部生理学)<br> | ||
</div> | </div> | ||
2013年6月21日 (金) 09:26時点における版
| myocyte enhancer factor-2 | |||||||||
|---|---|---|---|---|---|---|---|---|---|
 Structure of the MEF2A protein. Based on PyMOL rendering of PDB 1c7u. | |||||||||
| Identifiers | |||||||||
| Symbol | MEF2 | ||||||||
| Pfam | PF09047 | ||||||||
| InterPro | IPR015134 | ||||||||
| |||||||||
英語略:MEF2
MEF2は転写制御因子ファミリーの一つであり、筋細胞および神経細胞で特に高い発現を示す。近年、神経科学の分野ではMEF2は神経細胞の発達・分化や成熟神経細胞におけるシナプス機能の調節に関与していることが明らかになってきた。個体レベルにおいても記憶や学習への関与が示唆されている。
Myocyte enhancer factor-2とは
MEF2はもともと筋細胞分化に関わる因子として同定された転写因子であり、脊椎動物では異なる遺伝子によってコードされる4つのサブタイプ(MEF2A, MEF2B, MEF2C, MEF2D)が存在する。酵母、線虫、ショウジョウバエなどでは1種のMEF遺伝子が存在し、進化的に広く保存されている。発生期においてMEF2は、骨格筋や心筋の分化、神経堤形成、骨や血管形成など多様なイベントに重要な役割を果たしている。また、成体においては免疫系ではT細胞の分化・活性化や神経系ではシナプス機能の維持や調節に関与していることが明らかになってきた[1]。
MEF2はゲノム上の特定のDNA結合配列(MEF2 regulatory element、MRE)に結合して下流の遺伝子の転写活性化を促進する。また、MEF2は他の転写因子や補因子と多種多様な複合体を形成することにより、さらに巧妙な標的遺伝子の発現調節をおこなう。
構造
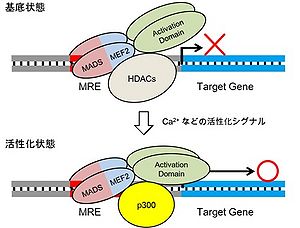
MEF2はMADS-MEF2ドメインと転写活性化ドメインの2つのドメインをもち、MADS-MEF2ドメインを介して2量体を形成しDNA認識配列(MRE)に結合する。定常状態ではMEF2はクラス2のヒストン脱アセチル酵素(HDACs)と結合しており、標的遺伝子の転写を抑制している。この抑制はカルシウムなどの活性化シグナルにより解除され、MEF2はヒストンアセチル化酵素(HAT)p300等と新たなタンパク複合体を形成することにより、標的遺伝子の転写を促進する。
MEF2は約500アミノ酸からなるタンパク質で、N末部の約90アミノ酸残基はファミリー内で相同性の高い領域であり、この部分はさらにMADS(MCM1-Agamous-Deficiens-Serum response factor)ボックスと呼ばれる他の転写因子と相同性のある領域とMEF2ファミリー間で保存されているMEF2相同領域に区分できる。一方、約400アミノ酸残基からなるC末側はMEFファミリー内および他の遺伝子とは相同性が低い領域であり、転写活性化ドメインとして機能している。核移行シグナルもC末部に存在する。
MEF2タンパク質はMADS-MEF2ドメインを介してホモ2量体およびサブタイプ間でのヘテロ2量体を形成し、DNA認識配列MRE(コンセンサス配列=[(C/T)TA(A/T)4TA(G/A)])に結合する。またMADS-MEF2ドメインはヒストン脱アセチル化酵素(HDACs)や転写因子等と相互作用することにより転写制御にも関与する(図)。
発現
MEF2の発現は発生から成体においてさまざまな器官・細胞に広く認められるが、その発現量は時期や細胞種によってサブタイプ毎に異なる。神経系においてはMEF2Cの発現がもっとも発生初期よりみられ、E11.5において終脳(telencephalon)の腹側部での発現がみとめられる。E13.5になると様々な脳領域において他のサブタイプも発現する[2]。また、成熟脳においてはMEF2A, MEF2CおよびMEF2Dが高い発現を示し、いずれも新皮質、海馬や小脳の神経細胞において発現しているが、各脳部位での発現量はサブタイプ毎に異なる[2]。しかしながら、現在のところサブタイプの違いによる標的遺伝子や転写活性化の調節機構(後述)など、サブタイプ間の機能差は不明である。
機能調節
MEF2による転写制御は神経機能に重要な役割を果たしていることが明らかになってきたが(次項参照)、神経細胞におけるMEF2の機能調節の詳細は不明である。現在のところ、MEF2の機能調節機構に関する多くの知見は筋細胞や免疫系細胞などの神経細胞以外を用いた研究から得られたものである。一般に、MEF2は活性化状態に関わらず核内でMREに結合しており、基底状態においてはHDACsと結合することによりヒストン修飾を介した積極的な転写抑性に関与していると考えられている[3]。ここにカルシウムシグナルやその他の活性化シグナルが核に伝わると、MEF2標的遺伝子の転写が活性化されるが、その活性化機構はリン酸化・脱リン酸化によって巧妙に制御されている。まず、リン酸化酵素カスケードによってHDACsがリン酸化されることによりMEF2から脱離し、替わりにヒストンアセチル化酵素(HAT)であるp300がMEF2と結合してクロマチンのリモデリングを引き起こし転写活性を増強させるというメカニズムが想定されている[3](図)。また、ERK5やJNK、p38キナーゼなどのMAPキナーゼ経路によりMEF2の転写活性化ドメインにある複数のアミノ酸残基(MEF2CにおいてはThr293, Thr300, Ser387等)がリン酸化されることにより、MEF2自身の転写活性化能が増強するという制御機構も存在する[4]。
さらに、カルシウム依存的な脱リン酸化酵素であるカルシニューリン(PP2B)による制御も報告されている。カルシニューリンは細胞体においてNFATを脱リン酸化することによりNFATの核移行を引き起こし、核内でのNFAT-MEF2複合体を形成促進することによりMREからの転写活性を上昇させる[3]。また、MEF2の転写活性化ドメインの特定の残基(例えばMEF2CのSer396やMEF2AのSer408)は基底状態においてリン酸化されており転写活性を抑性する方向に働いているが、カルシニューリン等によるこれら修飾残基の脱リン酸化は転写活性化を促進する[5] [6]。
神経細胞においてシナプス活動はカルシウムイオンの流入を引き起こし、カルシウム・カルモジュリン依存的キナーゼ(CaMK)やMAPキナーゼ(MAPK)、およびカルシニューリンの活性化が起こる。これらのリン酸化制御酵素によるMEF2およびMEF2相互作用タンパクのリン酸化状態の巧妙な制御により、MEF2による神経活動依存的な転写活性化が引き起こされていると考えられる[3]。さらに、MEF2はSUMO化などのリン酸化以外の翻訳後修飾も受けており、これらの修飾がMEF2の転写活性を調節している可能性が示唆されている[6]。
生物学的および生理的機能
MEF2は筋細胞においては、MyoDなどのbHLH型転写因子と協働して筋分化・維持に関わる多くの遺伝子を調節していることが知られていたが、神経細胞における標的遺伝子は不明であった。Flavellらは神経細胞を用いて活動依存的なMEF2の標的遺伝子をマイクロアレイ法やChIP-chip法などによりゲノムワイドで解析し、数百種類のMEF2標的遺伝子を同定した[7]。これらの中には神経活動によって発現が誘導される前初期遺伝子が多く含まれており、例えば転写因子をコードするc-fos, fosB, egr1等が含まれていた。また、シナプス機能を制御するArcやhomer1a, ynGAP、bndfなどもMEF2の標的遺伝子であった。
神経細胞においてMEF2AおよびMEF2Dを減少させると興奮性シナプスの数が増加し、逆に活性化型MEF2を発現させると興奮性シナプスの数および機能が低下する[8]。同様に、MEF2Cのノックアウトマウスではシナプス数およびシナプス伝達効率は上昇していた[9]。これらの結果より、成熟神経細胞においてMEF2は興奮性のシナプスの数および機能を負に制御する因子としてはたらいていると考えられる。
さらに、活性化型MEF2をマウス海馬や扁桃体に一過性に発現させた場合、記憶や学習に障害がみられることが報告されている[10]。また、MEF2の標的遺伝子の一部は脆弱性X症候群の原因遺伝子産物FMRPによって局所タンパク質翻訳制御を受けることによりシナプス機能調節に関与している可能性が示唆されている[11]。
関連項目
参考文献
- ↑
Potthoff, M.J., & Olson, E.N. (2007).
MEF2: a central regulator of diverse developmental programs. Development (Cambridge, England), 134(23), 4131-40. [PubMed:17959722] [WorldCat] [DOI] - ↑ 2.0 2.1
Lyons, G.E., Micales, B.K., Schwarz, J., Martin, J.F., & Olson, E.N. (1995).
Expression of mef2 genes in the mouse central nervous system suggests a role in neuronal maturation. The Journal of neuroscience : the official journal of the Society for Neuroscience, 15(8), 5727-38. [PubMed:7643214] [WorldCat] - ↑ 3.0 3.1 3.2 3.3
McKinsey, T.A., Zhang, C.L., & Olson, E.N. (2002).
MEF2: a calcium-dependent regulator of cell division, differentiation and death. Trends in biochemical sciences, 27(1), 40-7. [PubMed:11796223] [WorldCat] [DOI] - ↑
Han, J., & Molkentin, J.D. (2000).
Regulation of MEF2 by p38 MAPK and its implication in cardiomyocyte biology. Trends in cardiovascular medicine, 10(1), 19-22. [PubMed:11150724] [WorldCat] - ↑
Zhu, B., & Gulick, T. (2004).
Phosphorylation and alternative pre-mRNA splicing converge to regulate myocyte enhancer factor 2C activity. Molecular and cellular biology, 24(18), 8264-75. [PubMed:15340086] [PMC] [WorldCat] [DOI] - ↑ 6.0 6.1
Shalizi, A., Gaudillière, B., Yuan, Z., Stegmüller, J., Shirogane, T., Ge, Q., ..., & Bonni, A. (2006).
A calcium-regulated MEF2 sumoylation switch controls postsynaptic differentiation. Science (New York, N.Y.), 311(5763), 1012-7. [PubMed:16484498] [WorldCat] [DOI] - ↑
Flavell, S.W., Kim, T.K., Gray, J.M., Harmin, D.A., Hemberg, M., Hong, E.J., ..., & Greenberg, M.E. (2008).
Genome-wide analysis of MEF2 transcriptional program reveals synaptic target genes and neuronal activity-dependent polyadenylation site selection. Neuron, 60(6), 1022-38. [PubMed:19109909] [PMC] [WorldCat] [DOI] - ↑
Flavell, S.W., Cowan, C.W., Kim, T.K., Greer, P.L., Lin, Y., Paradis, S., ..., & Greenberg, M.E. (2006).
Activity-dependent regulation of MEF2 transcription factors suppresses excitatory synapse number. Science (New York, N.Y.), 311(5763), 1008-12. [PubMed:16484497] [WorldCat] [DOI] - ↑
Barbosa, A.C., Kim, M.S., Ertunc, M., Adachi, M., Nelson, E.D., McAnally, J., ..., & Olson, E.N. (2008).
MEF2C, a transcription factor that facilitates learning and memory by negative regulation of synapse numbers and function. Proceedings of the National Academy of Sciences of the United States of America, 105(27), 9391-6. [PubMed:18599438] [PMC] [WorldCat] [DOI] - ↑
Cole, C.J., Mercaldo, V., Restivo, L., Yiu, A.P., Sekeres, M.J., Han, J.H., ..., & Josselyn, S.A. (2012).
MEF2 negatively regulates learning-induced structural plasticity and memory formation. Nature neuroscience, 15(9), 1255-64. [PubMed:22885849] [WorldCat] [DOI] - ↑
Pfeiffer, B.E., Zang, T., Wilkerson, J.R., Taniguchi, M., Maksimova, M.A., Smith, L.N., ..., & Huber, K.M. (2010).
Fragile X mental retardation protein is required for synapse elimination by the activity-dependent transcription factor MEF2. Neuron, 66(2), 191-7. [PubMed:20434996] [PMC] [WorldCat] [DOI]