五嶋良郎
横浜市立大学大学院医学研究科分子薬理神経生物学
DOI:10.14931/bsd.4443 原稿受付日:2013年11月6日 原稿完成日:2013年12月26日
担当編集委員:林 康紀(独立行政法人理化学研究所 脳科学総合研究センター)
英語名:semaphorin
セマフォリンはセマドメインを持つリガンドの総称で、分泌型と膜結合型があり、現在20種類以上存在する。その主要な受容体であるプレキシンもセマドメインを細胞外に持ち、細胞内領域と相互作用する低分子量Gタンパク質やタンパク質リン酸化酵素等を介して情報伝達を行う。セマフォリンが生体内で担う役割は、神経軸索ガイダンス、血管新生、免疫、骨代謝制御と多岐にわたる。
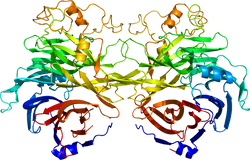
セマフォリン3A受容体結合領域の立体構造。1Q47による。 | |||||||||
| Identifiers | |||||||||
|---|---|---|---|---|---|---|---|---|---|
| Symbol | Sema | ||||||||
| Pfam | PF01403 | ||||||||
| InterPro | IPR001627 | ||||||||
| PROSITE | PDOC51004 | ||||||||
| SCOP | 1olz | ||||||||
| SUPERFAMILY | 1olz | ||||||||
| |||||||||
セマフォリンとは
セマフォリン分子群の先駆けは、特定の神経束を染め分けるモノクローナル抗体により同定されたファシクリンIVと、ニワトリ後根神経節細胞の成長円錐退縮応答を指標に同定されたコラプシン(後のセマフォリン3A, Sema3A)である。セマフォリン3Aは、当初、退縮活性を有することからコラプシンと命名されたが[1]、セマドメインを共有するセマフォリン3A以外のセマフォリンファミリー分子群が、必ずしも退縮活性を示さないことから、これらを総称して、セマフォリン(ギリシア語の手旗信号)ファミリー分子群と命名されるに至った[2]。
セマフォリンのプロトタイプであるセマフォリン3Aはその後、ニューロピリン1を受容体とすること、この受容体が、血管内皮細胞増殖因子 (vascular endothelial growth factor, VEGF)の受容体でもあることが発見されるに及び[3]、セマフォリンが血管系、免疫系等の神経系以外の様々な組織で重要な役割をすることが広く認識されるようになった。またアトピー性皮膚炎、がん、多発性硬化症、骨粗鬆症など[4]、セマフォリン分子が密接に関係する病態が発見されるようになった。最近では、COVID-19の病原ウイルスであるSARS-CoV2表面のスパイクタンパク質Sが、フリンプロテアーゼによって限定分解され、そのうちのS1と呼ばれる部位が、宿主細胞に発現するニューロピリン1に相互作用することが、SAR-CoV2の細胞内への取り込みを促進して感染効率を上昇させることを示す知見が報告された[5][6](Daly JL et al., 2020; Cantuti-Castelvetri L et al, 2020)。このように、現在ではセマフォリンおよびその受容体分子群は重要な創薬ターゲットとしても多くの研究者の注目を集めている[7] (Worzfeld and Offermanns, 2014)。
TIM-2:T cell immunoglobulin and mucin-domain-containing 2, CSPG: コンドロイチン硫酸プロテオグリカン, HSPG: ヘパラン硫酸プロテオグリカン
ファミリー分子
セマフォリンは構造の違いにより、8つのサブファミリー (セマフォリン1-7とV [ウイルス])に分類される(図1)[1]。全てのセマフォリンの N末端側はセマドメインとなっており、C末端側の構造によって、分泌型、膜貫通型、 GPIアンカー型等の性質が付与される。この内、セマフォリン1aは線虫やショウジョウバエ、セマフォリン2aはショウジョウバエ、ハチ等の昆虫に存在するセマフォリンである。
発現パターンと細胞内分布
脊椎動物に発現するセマフォリンセマフォリン3-7は各々固有の生体内分布を示す(表1)[8] [9]。セマフォリンは神経系に広く分布するのみならず、肺、消化器系、循環器系、免疫組織、骨組織を形成する細胞群にも発現し、その多彩な生理機能を有することを示唆している。セマフォリンの細胞内分布の詳細な解析は今後の課題である。
| 種類 | サブタイプ | 神経系 | 非神経系 |
| セマフォリン3 | A | 大脳皮質、梨状葉、海馬、尾状核、被核、扁桃体、小脳、分界条、 脊髄、後根神経節 | 肺、胎盤、胃、大腸、小腸、前立腺、骨芽細胞、腎臓 |
| B | 嗅球、小脳、中脳、橋、延髄 | ||
| C | 大脳皮質、梨状葉、内嗅領皮質、海馬、淡蒼球、扁桃体、小脳 | 脂肪細胞、肺、子宮、卵巣、膀胱、胃、大腸、骨芽細胞、骨格筋 | |
| D | 嗅球、脊髄、後根神経節 | 卵巣、膀胱、臍帯、表皮、骨芽細胞、脾臓、リンパ節、角膜、 レンズ | |
| E | 嗅球、網膜、大脳皮質、海馬、腹側線条体、基底前脳、 視床、中脳、橋、延髄 | 肺、胎盤、臍帯、上皮、胃、骨芽細胞 | |
| F | 大脳皮質、海馬、被殻、 腹側線条体、基底前脳、 扁桃体、視床、中脳、橋、延髄、小脳 | 肺、胎盤、子宮、下垂体 | |
| G | 後根神経節 | 肺、胎盤、卵巣、脂肪組織、腎臓 | |
| セマフォリン4 | A | 大脳皮質、梨状葉、内嗅領皮質、海馬、被殻、淡蒼球、黒質、扁桃体、小脳 | 脂肪組織、膀胱、胃、大腸、小腸、前立腺、T細胞、NK細胞、肥満細胞、樹状細胞、網膜色素上皮、角膜 |
| B | 後根神経節 | 胎盤、小腸、B細胞、形質細胞様樹状細胞 | |
| C | 後根神経節、小脳、嗅球、脊髄 | 胎盤、子宮、膀胱、肥満細胞、形質細胞様樹状細胞、ミクログリア、網膜、角膜、虹彩 | |
| D | B細胞、肥満細胞、マクロファージ、破骨細胞、ミクログリア、脾臓、リンパ節、骨髄、腎臓 | ||
| F | 大脳皮質、扁桃体、海馬、嗅球、脊髄、視床下部、小脳、側坐核、背側線条体、後根神経節 | 肺、胸腺細胞、T細胞、樹状細胞、顆粒球、網膜 | |
| G | 扁桃体、海馬、嗅球、視床下部、小脳 | 肝臓、腎臓、膀胱、胃、大腸、小腸 | |
| セマフォリン5 | A | 大脳皮質、扁桃体、海馬、嗅球、脊髄、視床下部、小脳、側坐核、線条体、後根神経節 | 子宮、卵巣、膀胱、骨芽細胞、水晶体 |
| B | 大脳皮質、扁桃体、海馬、嗅球、脊髄、側坐核、後根神経節 | 涙腺、膵臓、心臓、肝臓、ミクログリア | |
| セマフォリン6 | A | 大脳皮質、扁桃体、海馬、嗅球、脊髄、視床下部、小脳、側坐核、線条体、後根神経節 | 肺、胎盤、卵巣、小腸、膵臓、脂肪細胞、心臓、B細胞、肥満細胞、樹状細胞、脾臓、腎臓、網膜色素上皮、毛様体、水晶体 |
| B | 大脳皮質、扁桃体、海馬、嗅球、脊髄、視床下部、小脳、側坐核、線条体 | 膵臓、涙腺、唾液腺、マクロファージ | |
| C | 大脳皮質、扁桃体、海馬、嗅球、脊髄、視床下部、小脳、側坐核、線条体、後根神経節 | 肺、卵巣、心臓、樹状細胞、リンパ節、虹彩、眼杯 | |
| D | 脊髄、小脳 | 胃、小腸、膵臓、骨芽細胞、水晶体 | |
| セマフォリン7 | A | 脊髄、小脳 | 胃、小腸、膵臓、心臓、樹状細胞、骨芽細胞、破骨細胞、水晶体、角膜 |
サブタイプ名はAllen Brain Atlasへのリンク。
受容体と情報伝達
PI3K: ホスファチジルイノシトール3キナーゼ, Akt: v-akt murine thymoma viral oncogene homolog, GSK-3: glycogen synthase kinase 3, FARP2: FERM, RhoGEF and pleckstrin domain protein 2, Rac1: ras-related C3 botulinum toxin substrate 1, PAK: p21 protein (Cdc42/Rac)-activated kinase, Rnd1: Rho family GTPase 1, R-Ras: related RAS viral (r-ras) oncogene homolog, MICAL: microtubule associated monooxygenase, calponin and LIM domain containing 1, Fyn: FYN oncogene related to SRC, FGR, YES, Cdk5: cyclin-dependent kinase 5, CRMP: collapsin response mediator protein/dihydropyrimidinase-like
プレキシンは、細胞の上で二量体を作って不活性な状態になっている。プレキシンは、同じく二量体を形成するセマフォリンが結合すると分離して別々にセマフォリンに結合する。これにともなう位置関係・構造の変化がシグナルを発生する[11]。
ニューロピリン
藤澤らは、アフリカツメガエルの幼生視蓋組織から標品を調整し、これを抗原として、2つのモノクローナル抗体、MAb5A MAbB2を単離した。この二つの抗体は、それぞれ視蓋の特定の網状層を染め分けた。これがのちにニューロピリンと後述するプレキシンが同定される契機となった[12][13](Takagi et al, 1987; Fujisawa et al, 1997)。後に、これらがセマフォリンの受容体分子であることが明らかにされた[14][15] (Winberg et al, 1998; Takahashi et al, 1999)。
クラス3セマフォリンは分泌型であり、セマフォリン3A~3Gの7つが同定されている(表1)。このうちセマフォリン3EはプレキシンDを受容体とするが、それ以外はニューロピリン1、あるいはニューロピリン2を直接の受容体とする。ニューロピリン1、ニューロピリン2はプレキシンAと複合体を改正し、クラス3セマフォリンの機能的な受容体を形成する。またニューロピリン1およびニューロピリン2は血管内皮増殖因子 (vascular endothelial growth factor, VEGF)165の受容体としても作動する[16]。
プレキシン
セマフォリンの主要な受容体はプレキシンである。
プレキシンはA-Dの四つのサブファミリーに分類される。N末端はセマフォリンと同様にセマドメインとなっており、それ以外の細胞外ドメインも各サブタイプでほぼ共通である。立体構造の解析から、セマフォリンとプレキシンのセマドメインは七枚の羽のようなユニットからなるβプロペラ構造を持っていることが明らかとなった。セマフォリンとプレキシンの結合はお互いのセマドメイン同士を介して行われる。セマフォリン3サブファミリーは直接プレキシンと結合しないが、ニューロピリンとの結合を介してプレキシンAのセマドメインと相互作用する。プレキシンの中には、膜貫通型チロシンキナーゼや免疫グロブリンスーパーファミリー等と会合するものがある。これらの分子はプレキシンによる情報伝達を修飾する役割を担っている。
低分子量Gタンパク質
- プレキシンは、現在まで知られている膜貫通型受容体のうち、低分子量Gタンパク質と直接結合できる唯一の受容体ファミリーである(図2)[16] [17]。結合できる低分子量Gタンパク質は主にR-RasとRnd1である。プレキシンの細胞内領域はR-Rasを不活化するGTPase activating protein (GAP) ドメインがRnd1に対する結合領域であるRas-binding domain (RBD) で二つに分割されたような一次構造を持つ。しかし、立体構造の解析から、通常は細胞の上で二量体を作って不活性な状態になっているプレキシン(センサー)は、同じく二量体を形成するセマフォリン(信号)がやってくると分離して別々にセマフォリンに結合する。こうして、プレキシンからセマフォリンへとパートナーが替わることにより細胞内に信号が伝わると推定されている(図3)[18]。この際、タンパク質構造変化により、おそらくプレキシンの細胞内ドメイン同士の相互作用の変化をともなうと考えられるが、詳細はなお不明である。一方、細胞内領域の主要な機能は、GAPドメインとRBD領域がそれぞれひとかたまりとなった構造を持つため、リガンド依存的なRnd1の結合と、それに伴うGAP活性亢進によるR-Rasの不活化と考えられる(図2)。
- R-Rasの不活化により、ホスファチジルイノシトール3キナーゼ (PI3K) 活性が低下し、結果としてAkt-GSK3β系の活性化をもたらし、インテグリンを介した細胞接着が低下すると考えられている。
グアニンヌクレオチド交換因子
- プレキシンはグアニンヌクレオチド交換因子 (guanine nucleotide exchange factor: GEF) との結合を介して他の低分子量Gタンパク質の活性も制御できる。
- プレキシンAの場合は、膜直下にRacGEFの一種であるFARP2が結合する。受容体が刺激されると、FARP2がプレキシンAから解離し、結果として低分子量Gタンパク質Rac1及び下流のp21活性化キナーゼ (PAK) を活性化する[17]。
- 一方、プレキシンBの場合は、C末端にPDZドメイン結合配列があり、これを介してPDZ-RhoGEFやLARG等のRhoGEFが結合する。受容体が刺激されるとRhoGEFが活性化され、低分子量Gタンパク質Rhoと下流のRhoキナーゼ活性が亢進する。また、プレキシンBはRacとも相互作用することが知られている。
- これらをまとめると、プレキシンの基本的な機能は、Rnd1、R-Ras、Rac、Rhoの活性調節であり、これらの低分子量Gタンパク質を介して細胞骨格の再構成と細胞接着の制御を行っていると考えられる(図2)。
リン酸化酵素
- プレキシンAは低分子量Gタンパク質を介した情報伝達以外に、リン酸化酵素を介した情報伝達も行う。
- プレキシンAはSrcファミリーチロシンキナーゼの一種であるFynと相互作用する。セマフォリン3Aが結合するとFynによるチロシンリン酸化を介してCdk5が活性化する。Cdk5はコラプシン反応媒介タンパク質(collapsin response mediator protein, CRMP) をリン酸化する(図2)。一旦Cdk5によりリン酸化されると(プライミング)、GSK3βによりCRMPが認識されるようになり、追加的なリン酸化が行われる。これらのリン酸化を受けたCRMPと細胞内の他の様々なタンパク質との相互作用が変化し、微小管を含む細胞骨格の再構成に関与する[11]。また興味深いことに、アルツハイマー型認知症患者脳組織における神経原線維には、高度にリン酸化修飾を受けたCRMP2の集積が認められ、病態との関連が注目されている。
- GSK3βの基質には、CRMPのようなprimed substrateと 予めのリン酸化修飾を必要としないunprimed substrateが存在する。セマフォリン3Aシグナルの下流にはunprimed substrateであるAxin-1が関与し、GSK3β/Axin-1/β-カテニン複合体形成を経てエンドサイトーシスを誘起する[11]。また、プレキシンAは酸化還元酵素であるMolecules Interacting with CasL (MICAL) とも相互作用し、アクチンの重合を調節する。
- レーザー照射による局所分子不活化技術を用いて、成長円錐片側のCRMP1とCRMP2の活性を消失させると、照射部位とは逆側と同側の方向への、各々全く逆の軸索回旋が起こる。この事からCRMPサブファミリー分子の役割には相違があると考えられるが、詳細な機構は明らかではない[19]。
非プレキシン型受容体
いくつかのセマフォリンはプレキシン以外の受容体と結合することが知られている。CD72、Tim-2、インテグリン等が該当するが、それらはセマドメインを持たず、共通性が見られない。今後、これらの非プレキシン型受容体に関しても解明が進むものと思われる。
イオンチャネル
セマフォリンファミリー分子の細胞内情報伝達には、様々なカルシウム[20][21][22] (Sakai et al, 1999; Behar et al, 1999; Nishiyama et al, 2011)、カリウム、ナトリウムチャネル[23] (Yamashita et a, 2014) が関わる。神経系では、セマフォリン3Aがイオンチャネルの活性化を介してその受容体複合体とTrkAなどのニューロトロフィン受容体との相互作用や、順向性・逆行性の軸索内輸送を促す[24](Goshima et al, 2016)。このセマフォリン3Aによる細胞内応答は、樹状突起上のグルタミン酸受容体の輸送と樹状突起形成にもたらすことが示されている。しかしセマフォリンによって惹起される、様々な分子を含む細胞内膜の輸送と融合がどのように制御され、セマフォリン作用の発現に関わるかについては未解明である。
修飾因子
セマフォリンの作用は、上記の情報伝達系を基本とし、さらに細胞外のリガンド分子、細胞膜分子、そして細胞内の情報伝達分子などによって修飾される。
例えば、リガンド分子としてVEGFのスプライス変異体VEGF165はニューロピリン1に結合し、セマフォリン3Aに拮抗的に作用する。プレキシン-Aに直接結合するクラス6セマフォリンもセマフォリン3A作用を修飾する。
ニューロピリン1と相互作用する細胞膜タンパク質として、L1や類縁のCHL1が報告されている。ニューロピリン1と複合体を形成したL1は、接着班キナーゼとMAPキナーゼを活性化する。CHL1はセマフォリン3A/NRP1で制御される視床皮質路の軸索投射に関与する。
一方、プレキシン-Aはoff-Track、VEGFR2、TREM2、DAP12などの膜タンパク質と相互作用する[17]。これら相互作用分子の発現は組織や細胞により異なり、セマフォリンがそれぞれの細胞で引きおこす反応の違いに寄与する。細胞内分子cGMPはセマフォリン作用に影響する。セマフォリン3Aは末梢知覚神経軸索を反発するが、高濃度cGMPが存在する皮質錐体細胞の尖端樹状突起は、セマフォリン3A濃度勾配に沿って脳表方向へ誘引される。
生理機能
セマフォリンシグナリングの細胞生物学的な機能は細胞骨格の再構成と細胞接着の調節である。これらの結果、セマフォリンは細胞の形態や移動を制御すると考えられる。神経系においては、軸索伸長、軸索ガイダンス、樹状突起スパインの形態・機能の制御を介して神経回路網形成やシナプス可塑性に関与すると考えられる。セマフォリンはその発見の経緯から、当初は神経回路形成や軸索伸長制御因子としての役割が注目され、解明が進められたが、その後、セマフォリン3Aの遺伝子欠損マウスの表現型解析から、回路選択からシナプス形成に至る神経回路形成の各過程に広く関わることが示された[25]。神経回路選択やシナプス形成の過程が一つのガイダンス分子により制御されていることは興味深い。
この発見を経緯に、セマフォリンのシナプスや樹状突起パターン形成における作用が解析された。Kolodkinらのグループは同じクラス3セマフォリンに属するセマフォリン3Fの作用を追究する過程で、セマフォリン3Fが樹状突起スパインの数や興奮性シナプス電流の減少を引き起こすことを報告した。一方、同グループは、セマフォリン3Aとの結合を欠失したニューロピリン1をノックインしたマウスの解析から、セマフォリン3Aは樹状突起パターンには作用するが、スパインの数や形態には影響を与えないと報告しており、研究グループ間にセマフォリン3A作用の解釈をめぐって相違がある[25] [26]。
セマフォリンが学習・記憶等の生理学的過程にどのような役割を果たしているかは今後の重要課題の一つである。最近になり、海馬において神経活動依存性にセマフォリン3Aが分泌され、AMPA受容体のトラフィッキングを促進することが記憶学習の成立に必要であることが示された[27](Jitsuki-Takahashi et al, 2021)。
関連項目
参考文献
- ↑ 1.0 1.1
Raper, J.A. (2000).
Semaphorins and their receptors in vertebrates and invertebrates. Current opinion in neurobiology, 10(1), 88-94. [PubMed:10679438] [WorldCat] [DOI] - ↑
(1999).
Unified nomenclature for the semaphorins/collapsins. Semaphorin Nomenclature Committee. Cell, 97(5), 551-2. [PubMed:10367884] [WorldCat] [DOI] - ↑
Gagnon, M.L., Bielenberg, D.R., Gechtman, Z., Miao, H.Q., Takashima, S., Soker, S., & Klagsbrun, M. (2000).
Identification of a natural soluble neuropilin-1 that binds vascular endothelial growth factor: In vivo expression and antitumor activity. Proceedings of the National Academy of Sciences of the United States of America, 97(6), 2573-8. [PubMed:10688880] [PMC] [WorldCat] [DOI] - ↑
Negishi-Koga, T., & Takayanagi, H. (2012).
Bone cell communication factors and Semaphorins. BoneKEy reports, 1, 183. [PubMed:24171101] [PMC] [WorldCat] [DOI] - ↑
Daly, J.L., Simonetti, B., Klein, K., Chen, K.E., Williamson, M.K., Antón-Plágaro, C., ..., & Yamauchi, Y. (2020).
Neuropilin-1 is a host factor for SARS-CoV-2 infection. Science (New York, N.Y.), 370(6518), 861-865. [PubMed:33082294] [WorldCat] [DOI] - ↑
Cantuti-Castelvetri, L., Ojha, R., Pedro, L.D., Djannatian, M., Franz, J., Kuivanen, S., ..., & Simons, M. (2020).
Neuropilin-1 facilitates SARS-CoV-2 cell entry and infectivity. Science (New York, N.Y.), 370(6518), 856-860. [PubMed:33082293] [PMC] [WorldCat] [DOI] - ↑
Worzfeld, T., & Offermanns, S. (2014).
Semaphorins and plexins as therapeutic targets. Nature reviews. Drug discovery, 13(8), 603-21. [PubMed:25082288] [WorldCat] [DOI] - ↑ 8.0 8.1 http://biogps.org/#goto=welcome BioGPS
- ↑ 9.0 9.1 http://www.gensat.org/index.html The GENSAT Project at the Rockefeller University
- ↑
Skaliora, I., Singer, W., Betz, H., & Püschel, A.W. (1998).
Differential patterns of semaphorin expression in the developing rat brain. The European journal of neuroscience, 10(4), 1215-29. [PubMed:9749776] [WorldCat] [DOI] - ↑ 11.0 11.1 11.2
Yamashita, N., & Goshima, Y. (2012).
Collapsin response mediator proteins regulate neuronal development and plasticity by switching their phosphorylation status. Molecular neurobiology, 45(2), 234-46. [PubMed:22351471] [WorldCat] [DOI] - ↑
Takagi, S., Tsuji, T., Amagai, T., Takamatsu, T., & Fujisawa, H. (1987).
Specific cell surface labels in the visual centers of Xenopus laevis tadpole identified using monoclonal antibodies. Developmental biology, 122(1), 90-100. [PubMed:3297854] [WorldCat] [DOI] - ↑
Fujisawa, H., Ohta, K., Kameyama, T., & Murakami, Y. (1997).
Function of a cell adhesion molecule, plexin, in neuron network formation. Developmental neuroscience, 19(1), 101-5. [PubMed:9078440] [WorldCat] [DOI] - ↑
Winberg, M.L., Noordermeer, J.N., Tamagnone, L., Comoglio, P.M., Spriggs, M.K., Tessier-Lavigne, M., & Goodman, C.S. (1998).
Plexin A is a neuronal semaphorin receptor that controls axon guidance. Cell, 95(7), 903-16. [PubMed:9875845] [WorldCat] [DOI] - ↑
Takahashi, T., Fournier, A., Nakamura, F., Wang, L.H., Murakami, Y., Kalb, R.G., ..., & Strittmatter, S.M. (1999).
Plexin-neuropilin-1 complexes form functional semaphorin-3A receptors. Cell, 99(1), 59-69. [PubMed:10520994] [WorldCat] [DOI] - ↑ 16.0 16.1
Takamatsu, H., & Kumanogoh, A. (2012).
Diverse roles for semaphorin-plexin signaling in the immune system. Trends in immunology, 33(3), 127-35. [PubMed:22325954] [WorldCat] [DOI] - ↑ 17.0 17.1 17.2
Oinuma, I., Ishikawa, Y., Katoh, H., & Negishi, M. (2004).
The Semaphorin 4D receptor Plexin-B1 is a GTPase activating protein for R-Ras. Science (New York, N.Y.), 305(5685), 862-5. [PubMed:15297673] [WorldCat] [DOI] - ↑
Nogi, T., Yasui, N., Mihara, E., Matsunaga, Y., Noda, M., Yamashita, N., ..., & Takagi, J. (2010).
Structural basis for semaphorin signalling through the plexin receptor. Nature, 467(7319), 1123-7. [PubMed:20881961] [WorldCat] [DOI] - ↑
Higurashi, M., Iketani, M., Takei, K., Yamashita, N., Aoki, R., Kawahara, N., & Goshima, Y. (2012).
Localized role of CRMP1 and CRMP2 in neurite outgrowth and growth cone steering. Developmental neurobiology, 72(12), 1528-40. [PubMed:22378692] [WorldCat] [DOI] - ↑
Sakai, T., Furuyama, T., Ohoka, Y., Miyazaki, N., Fujioka, S., Sugimoto, H., ..., & Inagaki, S. (1999).
Mouse semaphorin H induces PC12 cell neurite outgrowth activating Ras-mitogen-activated protein kinase signaling pathway via Ca(2+) influx. The Journal of biological chemistry, 274(42), 29666-71. [PubMed:10514436] [WorldCat] [DOI] - ↑
Behar, O., Mizuno, K., Badminton, M., & Woolf, C.J. (1999).
Semaphorin 3A growth cone collapse requires a sequence homologous to tarantula hanatoxin. Proceedings of the National Academy of Sciences of the United States of America, 96(23), 13501-5. [PubMed:10557350] [PMC] [WorldCat] [DOI] - ↑
Nishiyama, M., Togashi, K., von Schimmelmann, M.J., Lim, C.S., Maeda, S., Yamashita, N., ..., & Hong, K. (2011).
Semaphorin 3A induces CaV2.3 channel-dependent conversion of axons to dendrites. Nature cell biology, 13(6), 676-85. [PubMed:21602796] [PMC] [WorldCat] [DOI] - ↑
Yamashita, N., Usui, H., Nakamura, F., Chen, S., Sasaki, Y., Hida, T., ..., & Goshima, Y. (2014).
Plexin-A4-dependent retrograde semaphorin 3A signalling regulates the dendritic localization of GluA2-containing AMPA receptors. Nature communications, 5, 3424. [PubMed:24599038] [WorldCat] [DOI] - ↑
Goshima, Y., Yamashita, N., Nakamura, F., & Sasaki, Y. (2016).
Regulation of dendritic development by semaphorin 3A through novel intracellular remote signaling. Cell adhesion & migration, 10(6), 627-640. [PubMed:27392015] [PMC] [WorldCat] [DOI] - ↑ 25.0 25.1
Morita, A., Yamashita, N., Sasaki, Y., Uchida, Y., Nakajima, O., Nakamura, F., ..., & Goshima, Y. (2006).
Regulation of dendritic branching and spine maturation by semaphorin3A-Fyn signaling. The Journal of neuroscience : the official journal of the Society for Neuroscience, 26(11), 2971-80. [PubMed:16540575] [PMC] [WorldCat] [DOI] - ↑
Tran, T.S., Rubio, M.E., Clem, R.L., Johnson, D., Case, L., Tessier-Lavigne, M., ..., & Kolodkin, A.L. (2009).
Secreted semaphorins control spine distribution and morphogenesis in the postnatal CNS. Nature, 462(7276), 1065-9. [PubMed:20010807] [PMC] [WorldCat] [DOI] - ↑
Jitsuki-Takahashi, A., Jitsuki, S., Yamashita, N., Kawamura, M., Abe, M., Sakimura, K., ..., & Takahashi, T. (2021).
Activity-induced secretion of semaphorin 3A mediates learning. The European journal of neuroscience, 53(10), 3279-3293. [PubMed:33772906] [WorldCat] [DOI]