「シングルセルRNAシーケンシング」の版間の差分
Masahitoyamagata (トーク | 投稿記録) 細 PMID:33393903をつかって、<ref name=Yamagata2021></ref>に変更 |
Masahitoyamagata (トーク | 投稿記録) 細 PMID:33393903をつかって、<ref name=Yamagata2021></ref>に変更 |
(相違点なし)
| |
2021年1月6日 (水) 10:18時点における最新版
山形方人
Harvard University
DOI:10.14931/bsd.8038 原稿受付日:2020年10月22日 原稿完成日:2020年12月23日
外部査読委員:京都大学メディカルイノベーションセンター 渡辺 亮
理化学研究所 生命機能科学研究センターバイオインフォマティクス研究開発チーム/東京医科歯科大学 難治疾患研究所 ゲノム応用医学部門 ゲノム機能情報分野 二階堂 愛
英:single-cell RNA sequencing, scRNA-seq
シングルセルRNAシーケンシング(single-cell RNA sequencing, 以下scRNA-seq)は、次世代シーケンサー(next generation sequencer、以下NGS)を用いることで、個々の細胞が保持しているmRNA全体を質的、量的に網羅的に調べる方法である。次元圧縮などの数理的な解析と組み合わせることで、遺伝子発現の状態に基づいた細胞の分類を行うことが可能であり、従来の組織学的、あるいは細胞生物学的手法では知られなかった新規の細胞種の同定や細胞状態の推定を行うことが可能になった。また、遺伝子発現プロファイルの変化に基づく擬時系列解析(pseudotime analysis)によって、刺激や発生に伴う細胞状態の遷移の描写ができる。神経系では、この方法により、神経細胞や非神経細胞の分類や状態についての知見が深まり、新しい神経細胞タイプ、細胞マーカー、病態の理解、更に機能的な遺伝子の同定などが系統的かつ網羅的に行われるようになった。scRNA-seqに、空間的情報、エピゲノム情報、タンパク質情報などの複数モダリティを取り入れた統合解析(multimodal single-cell omics)も行われている。
背景
トランスクリプトーム
トランスクリプトーム(transcriptome)は、細胞中に存在する全ての転写産物(タンパク質をコードするmRNA、タンパク質をコードしないノンコーディングRNA、マイクロRNAなど)の総体である[1][2]。トランスクリプトームは、ゲノムとは異なり、同一の個体でも、組織ごとに、更には発生段階や細胞外環境や刺激によって変化する。トランスクリプトームは、同質あるいは異質の多数の細胞集団(組織、培養細胞)からRNA抽出後、cDNAに変換し、それを1990年代に出現したDNAマイクロアレイのように数多くの既知mRNAを識別する技術によって解析されるようになった。その後、次世代シーケンサーの利用により、希少mRNAやノンコーディングRNAを含めた未知の転写産物の高感度検出が可能になるとともに、スプライシングで成熟していく過程のmRNAなど、転写産物の種類だけでなく、転写産物の構造的差異(スプライシングバリアント、SNPs、変異など)の解析もできるようになった。加えて、ヒトやモデル実験動物(マウス、ゼブラフィッシュ、ショウジョウバエ、線虫など)だけでなく、多種多様な生物のトランスクリプトームの把握も可能になった。従来から行われてきた組織全体などの多数の細胞を対象としたRNA-seq(バルクRNA-seq)では、複数の細胞における転写産物の平均を観察しているが、本項目では個々の細胞における転写産物を解析するscRNA-seqの原理とその応用について概説する。
開発史
1つの細胞の持つ生体物質を解明し、定量しようとする試みは古くからあった。1960年代になると、フローサイトメトリーを利用した蛍光活性化セルソーティング(Fluorescence-activated cell sorting, FACS)が発明され、標識抗体などのプローブと組み合わせることで、多数の細胞集団の中で1つの細胞が保持している生体分子の種類や量についての断片的な研究が可能になり、この方法は現在でも汎用されている[3]。その後、免疫組織化学やin situ hybridizationnなどにより、タンパク質やmRNAの種類や量が観察できるようになり、組織中に存在するそれぞれの細胞の同定などに活用されてきている。最近では、それぞれの細胞が持つ抗原分子を、異なった金属イオンで標識した抗体とフローサイトメトリーを組み合わせた方法で検出するマスサイトメトリー(CyTOFなど)も開発されてきている[4]。
細胞種にもよるが、1つの細胞内にある全RNA(ribosomal RNAを含む)は細胞種にもよるが1-50pgである。そのうち、mRNAの占める割合は1-5%程度である[5]。この微量のmRNAをcDNAに変換してから大幅に増幅できる方法が発明されたことで、1つの細胞が発現するmRNAを高感度で検出できるようになった[6][7] 。例えば、1991年、Linda BuckとRichard Axelは、嗅覚受容体がGタンパク質であると仮定し、個々の嗅覚細胞で特異的に観察されるGタンパク質mRNAを比較することで、嗅覚受容体の同定に成功した[8]。1995年になると、Catherine DulacとRichard Axelは、異なる鋤鼻神経細胞で特異的に発現する遺伝子を1つの細胞から作製したcDNAライブラリーを比較するディファレンシャル・スクリーニングを行うことで、フェロモン受容体を同定した[9]。同じ手法で異なる種類の神経細胞で発現している遺伝子も同定され[10][11]、1つの細胞の持つトランスクリプトームを比較するアプローチが神経細胞で特徴的に発現している遺伝子の同定に効果的なことが示された。
一方で多くの種類のmRNAを1細胞レベルで一挙に観察するための技術には感度やスループット、そしてコストの観点からブレークスルーが待たれた。1つの問題は多種類のcDNAを簡便に識別することを可能にする方法の開発であった。これを可能にしたのが、PCRなどのcDNA増幅法の改良とマイクロアレイの利用であった[12][13]。しかしながら、細胞ごとに高価なマイクロアレイを使用することは、多数の細胞のトランスクリプトームの観察には限界があった。2009年になると、これらの問題を解決できる可能性として、次世代シーケンサーを利用するscRNA-seqプロトコールがAzim Suraniのグループによって報告された[14]。しかしながら、多数のマイクロアレイでなく1回の次世代シーケンサー使用で済ませることができるものの、この報告でもわずか8個の細胞の解析に留まっており、1つの細胞ごとに処理を行うという操作が必要で、多数の細胞についてのトランスクリプームを一挙に理解することはできなかった。また、塩基配列の違うcDNAごとにPCR効率に差がある結果生じる増幅バイアス、また3’末端側が選択的に補足されることなどの課題があった。
現状
分子生物学的反応
その後、5’末端側の領域まで効率よく増幅するscRNA-seqのプロトコールが考案された[15]。特に、SMART-seq(Switching mechanism at the 5' End of RNA Templates)[16]およびその改良されたプロトコールであるSMART-seq2[17] [18]の使用例が多い(既に、SMART-seq3という改良プロトコールもある[19]が、以下SMART-seqと呼ぶ)。また、類似法としてSTRT(single-cell tagged reverse transcription)[20]などがある。
一方、CEL-seq(Cell Expression by Linear amplification and Sequencing)[21]、CEL-seq2[22] 、MARS-seq(Massively parallel single-cell RNA-seq)[23]では、T7 RNAポリメラーゼによるin vitro転写を用いることにより、PCRによる増幅で見られるバイアスを低減させようとしている。
また、Quartz-SeqやQuartz-Seq2ではPCR用のアダプターを付加する反応にポリAテーリングを利用することで、他の手法と比較して1.5-5倍程度の遺伝子を検出できる[15]。
バーコード技術
増幅バイアス除去のアプローチとして特に重要なのは、2011年に発表された核酸配列バーコードを利用した方法で、分子識別子(unique molecular identifiers: UMI)を持つcDNAを増幅させ、次世代シーケンサー後の情報処理を用いるものであると考えられる[24]。この方法では逆転写反応の際、ランダム塩基配列から構成されるUMIをcDNA末端に付加した後、増幅反応、次世代シーケンサーを行い、cDNA配列とUMI配列の両方を読む。cDNAにはRNA1分子に1つのUMIが付加されるので、同一のUMIを持っていれば、逆転写時に同一のcDNA由来とカウントする。UMIをカウントすることで、増幅前のmRNAのコピー数を知ることができる[20][25][26] [27][28][29]。
多様なプラットフォーム
細胞を分別するプラットフォームには、マイクロピペットによる捕獲、セルソーター、レーザー捕獲などを用いるマルチウェル法、あるいは半導体集積回路様の製作技術で作った流体回路を利用するFluidigm C1の装置(C1 Single- Cell Auto Prep)、更にドロップレット使用(下記)などがある[30][31]。これらは、SMART-seqと組み合わせて利用されることが多い。SMART-seqプロトコールの特徴は、全長mRNAのトランスクリプトーム情報を得ることができることであり、mRNAのスプライシングバリアントなどのアイソフォーム、SNPsの情報を利用したアリル特異的発現、変異の検出にも利用できる。また、それぞれ細胞ごとの反応を独立した場所で行うため、反応中に別の細胞の反応と混じる可能性が低い。小型のナノウェルを用いるSeq-Wellも同様に反応自体が混じる可能性が低い[26]。これらの点が、次に説明するドロップレットを使用して3’末端のみを標的にしたscRNA-seqと比べた場合の長所であるが、その高コスト(1細胞あたり数十ドル)と処理可能な細胞数の少なさが短所である。
これらとは別に、ハイスループットで安価な方法として、それぞれの細胞を独立に標識するのではなく、プールされた細胞を異なるウェルにランダムに振り分け、ウェル固有のバーコードで転写物を標識していく操作を複数回繰り返していくことで細胞を区別するSplit-seqやsci-RNA-seq3などの方法も用いられている[29][32]。
ドロップレット使用の3’エンドリード法
scRNA-seqのプラットフォームと方法について重要と考えられる進歩は、2015年、Harvard Medical Schoolの独立した2つのグループが、inDrops[33]そしてDrop-seq[34]という類似した2つのハイスループットな方法を開発したことであろう(inDropsはT7 RNAポリメラーゼ、Drop-seqはPCRで増幅)。これらの方法では、マイクロ流体力学 (microfluidics) 、 UMI(上述)と細胞ごとのバーコード(Cell Barcode)という2種類のDNAバーコーディング、そしてNGSと情報解析法を利用している。そして、多く細胞のサンプル調製の自動化と容易さから、1つの細胞あたりに要するコストを大幅に低下させることに成功した(Drop-seqは発表時で、1細胞あたり約5セント)。つまり、細胞1つずつをマイクロ流体力学によるエマルジョン作製技術を利用した装置に流入させ、その1細胞を1つのドロップレットに自動的に閉じ込める。そのドロップレット中には、ドロップレットごとにCell barcode/UMIとしてユニークなDNAバーコードを持つゲルビーズ(Gel Beads in Emulsion, GEMs)が入っており、それを足場に3’末端のみを標的にしたcDNA合成反応を実施することで、同じ細胞に含まれていた1分子のmRNAが同じCell barcodeを持つcDNAとして合成され、そのmRNA/cDNAが由来した細胞を識別できるということを利用している(図1)。
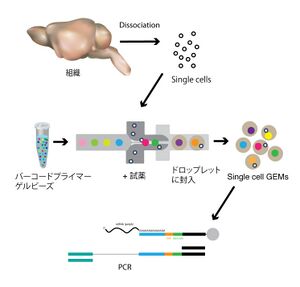
組織から解離させた細胞それぞれを、マイクロ流体力学を利用した装置で、バーコードプライマーが結合したゲルビーズとともにドロップレットに封じ込める。ドロップレット中には、ドロップレットごとにCell barcode/UMIとしてユニークなDNA配列を持つゲルビーズ(GEMs)が入っており、それを足場にcDNA合成反応を実施することで、同じ細胞に含まれていたmRNAが同じCell barcodeを持つDNAとして合成され、それを増幅する。
DropSeqはコストが低いが、細胞の取得率と検出感度が低い弱点がある。inDropsはDropSeqより細胞取得率が高く、パラメータを調整することにより、低レベルで発現される遺伝子の検出にも有利であるとされる[35]。DropSeqのセットアップはDolomite Bio、inDropは1 Cellbio社から販売されている。しかし、その後、10x Genomics社が同様の原理を用いたシングルセル遺伝子発現解析システムを市販することで、多くの研究者が利用できるようになっている[36]。Svenssonらによる最近のデータベース[37]では、scRNA-seqを用いた論文で用いられた方法について調査しており、この数年、10x Genomics社のプラットフォームを用いた論文が飛躍的に増加していることがわかる。10x Genomics社のプラットフォームは市販であるので導入が容易であり、DropSeqやinDropsに比べ多くの転写産物の検出が可能であるが、それらよりランニングコストは高価である[35]。
なお、3’エンドリード法だけでなく、抗体やT細胞レセプターのN末端側に位置する可変領域の配列決定が可能である5'末端のシーケンシングには5’エンドリード法が利用されることがある。
実際
ここでは主流になっている10x Genomics社のChromium controllerなどのドロップレットを用いた方法とSMART-seqなどを用いた他のプラットフォームに共通する方法の実際について概説する。scRNA-seqの利用には、4つのステップがある(図2)[38][39]。これらのうち、2.の段階については、上に記述したように市販の機器や試薬を利用する機会が多くなっているので、詳細は説明しない。
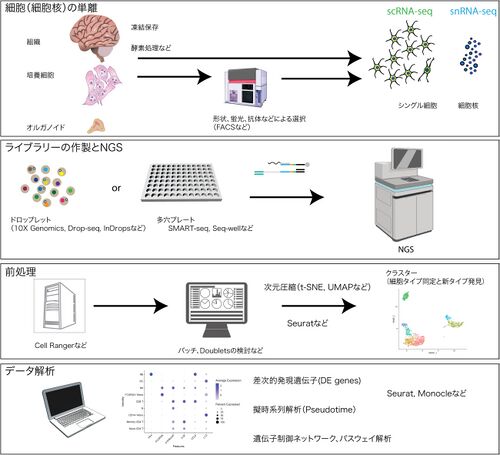
細胞の単離、ライブラリ作製とNGS、データの前処理から次元圧縮、データ解析。図の一部は2016 DBCLS TogoTV、あるいはSeuratを用いて10x Genomics社のPBMCデータから執筆者が作製。
- 個体や組織を採集し、そこから細胞あるいは細胞核を個別に解離された状態にすること。
- ドロップレット法やSMART-seq対応のプラットフォームなどによる個々の細胞からのライブラリーの作製とNGS。
- 得られた配列情報の前処理(preprocessing)。
- データ解析。
組織からの細胞、細胞核の分離
浮遊細胞(血液細胞など)ではない場合、物理的あるいは酵素処理などによって解離することで、生組織から状態の良い個々に分散した細胞を調製する必要がある。神経系組織の酵素処理には、パパインを用いる方法が広く用いられている[40]。ここで、しばしば問題となるのが、酵素処理による短時間加温や機械的刺激で、発現量が変化する遺伝子が存在することである[41]。特に、脳のミクログリアの解析には、低温下で組織をホモゲナイズするなどの工夫が必要であった[42]。また、このような現象を抑制するために、酵素処理時に転写阻害剤であるアクチノマイシンで処理したり[43]、ヒマラヤ氷河から得られた細菌Bacillus licheniformisから得られた低温プロテアーゼを用いる方法も報告されている[44][45]。また、細胞解離後に、メタノールで固定しscRNA-seqに使用したり[46]、クロスリンカーを用いる方法もある[47]。
単離した細胞は、そのまま10x Genomicsのシングルセル遺伝子発現解析のプラットフォームに導入することができるが、細胞表面分子マーカーに対する抗体や蛍光タンパク質レポーターなどを用いたFACS、パニング、MACS(磁気ビーズカラム)などによって、細胞の選択的濃縮や除去を行う場合もある。更に、抗体に抗体表示バーコードDNAをカップリングさせるCITE-seq(Cellular Indexing of Transcriptomes and Epitopes by Sequencing) については、下記の「統合解析」でも述べる。
なお、ヒト組織や希少生物などから生細胞を得ることは困難なことが多い。この場合、scRNA-seqの変法として、凍結した組織から、各細胞由来の核を調製し、核内のmRNAを分析するsnRNA-seq (single-nucleus RNA-seq)が利用されている。ただ、snRNA-seqでは、FACSなどによる特定細胞集団の分離が困難であることが多い。また、細胞質を持つ生細胞を利用したscRNA-seqとは違って、スプライシングの途上にある未成熟な核内転写産物を検出すること、更に検出できる遺伝子数も少なく、同等な結果が必ずしも得られない[48][49] [50][51][52][51][53][54][55][56] [15]。一方で、snRNA-seqでは、組織をそのまま凍結することから開始することが可能であるので、上述したscRNA-seqの問題である細胞解離酵素による処理などを避けることができる。更に、核を用いることで、大きな細胞体はマイクロ流体力学の流路で詰まりやすいなど、特に神経細胞で顕著である細胞の形状の多様性に伴うバイアスを減らすことができるといったメリットもある。こうしたプロトコールの一部は、protocols.ioのHuman Cell Atlasのグループで公開されている。
通常のscRNA-seqは、ポリアデニル化されたmRNAを検出しているが、MATQ-seq(multiple annealing and dC-tailing-based quantitative single-cell RNA-seq)、RamDA-seqなどを用いると、ポリアデニル化されていないRNAの検出も可能である[57] [58][1]。
更に、RNAを分析するscRNA-seqではないが、遺伝子発現状態との関係が想定されるオープンクロマチン領域をトランスポゾンを用いることで個々の細胞レベルで選択的に検出するsingle cell ATAC-seq (Assay for Transposase-Accessible Chromatin) [59][60][61], single cell THS-seq (transposome hypersensitive-site) [62]や DNAメチル化領域を観察するsnmC-seq、RRBSのような方法も利用されている[63][64][65]。
データ処理の流れ
総論
Illumina社に代表される次世代シーケンサーを用いて得られた結果は、ベースコールや細胞バーコードを用いたdemultiplexingなどの基礎解析を行うことで、各細胞における遺伝子の発現量のマトリックスを出力する。例えば、10XGenomics社のChromiumプラットフォームを用いた場合、10XGenomics社が提供するCell Rangerのmkrefコマンド(Linux上で作動)などにより、各生物種ごとのレファレンス配列リスト(マウスやヒトでは既製のものを利用できる)などを参考にしながら、細胞と転写産物量の対応マトリックスを作製する。その後のデータの処理についても、10x Genomics社がソフトウェアLoupeを提供している。しかしながら、その後のデータ解析を考慮して、R, Python, MATLABなどのデータ解析のための汎用プログラミング言語やコードで扱えるオブジェクトに変換するのが通常である。
scRNA-seq解析のためには、数多くのツールが公開されている。これらのツールは、バージョンが更新されたり、新しいものに置き換えられることがあるので、実際に利用する場合は最新の動向に注意を払う必要がある。scRNA-seqの解析に必要なツールは、scRNA-tools, Awesome single cell, Bioconductorなどで紹介されており、ほとんどがダウンロード可能である。また、bioRxivなどの査読前のプレプリントサーバで公開されて、随時試用、評価されていくものが多く、scRNA-seqのデータ(下記参考)とともに、オープンサイエンス実践の好例となっている。
Seurat
ここでは、scRNA-seqデータ解析のために最もよく利用されているRを用いたパッケージ「Seurat」[66] [67]を中心に紹介しておきたい。なお、一部の解析操作は、University of WashingtonのCole Trapnell研究室で開発されてきた軌道推定(下記参考)によく使用されるMonocle3でも可能である。Pythonを利用したものでは、ドイツ・ミュンヘンInstitute of Computational Biologyの Fabian Theisらが開発しているScanpyが有名である[68]。
New York UniversityのRahul Satija研究室が開発しているSeurat(画家スーラに由来)は、scRNA-seqデータ解析のために広く利用されているRパッケージであり、2020年秋現在、Seurat4のβバージョンが公開されている。論文の正式発表前から、サポート情報提供やコード修正なども頻繁に行っており、Satija研究室のウェッブサイト、Github、更にTwitterアカウントなどで最新情報を得ることできる。
品質の検討事項
最初に行うのは、scRNA-seqデータの品質管理である。ここでは、質の低い細胞のデータ(例えば、壊れた細胞では、転写産物の種類が少なくミトコンドリア由来の転写産物が多い)を取り除く。また、複数の試料を組み合わせる場合には、バッチごとの違いについて検討する[69][70][71] [72]。現実には、実験ごとのバッチの違いによる影響(Batch effect)がscRNA-seqの最大の問題であると示されてきており、試料の処理を同時に行うなど実験デザインを工夫する必要がある[73]。
また、ドロップレットを使用するscRNA-seqでしばしば問題になるのが、ドロップレットに2つ以上の細胞が封じ込められ、それらが同一のCell barcodeを持ってしまうアーティファクトである。通常「Doublet」「Multilet」と呼ばれるこの問題はデータ解析を混乱させるので、細胞単離の段階から注意する必要があるが、scRNA-seqデータ取得後にもデータ処理で解決できる可能性もある[74][75] [76][77] [78][79][80][81] [82][83][84][2]。なお、この手法を利用することで、バッチ効果を抑えるために、異なるバーコードを持つ複数の試料を混ぜて一つの試料として扱い、計算機的に再び分離、解析する手法が注目されている[85]。
scRNA-seqデータの次のノイズは、ある遺伝子の発現が低いために、本来同じタイプの細胞であっても、その遺伝子の発現が全く見られない「Dropout」と呼ばれる現象であり解析に影響を与えるので、これについても検討が必要である[86][87]。
次元圧縮
このような品質管理、ノーマライゼーションの過程を経て[88]、scRNA-seqのデータ解析において、最初に行うのが、次元圧縮 (dimensionality reduction)である[89][90][91][92]。主成分分析 (Principal component analysis, PCA)、更に発展させた均一マニフォールド近似と投影(Uniform Manifold Approximation and Projection, UMAP)、Diffusion maps[93], t分布型確率的近傍埋込み (t-distributed Stochastic Neighbor Embedding, tSNE)などの手法が用いられる。 特に、tSNEとUMAPは、高次元データを低次元の点の集合として可視化することで、それぞれの細胞の持つ遺伝子発現状態の類似度についての直観的な表示が可能でありしばしば用いられる(図3)。tSNEよりUMAPの方が迅速に類似集団間の関係が明確になるので、最近はUMAPを利用することが多くなってきている。次に、Louvainアルゴリズムなどでクラスタリング(コミュニティ分割)を行いグラフ上に表示できる(図3の色分け)。こうして、異なる転写状態を示す細胞の集合が別のクラスターとして表示され、同定可能になる[94]。

網膜(ニワトリ)の視細胞のデータを用いて執筆者が作製[95]。
データ解析
細胞クラスターの解釈とマーカー遺伝子候補の発見
scRNA-seqデータから得られる生物学的知見には、内在的に存在する細胞の種類、外部刺激や環境で変化した細胞の状態、そして種類や変化により特徴的に発現するマーカー遺伝子候補の発見がある[96][97]。クラスタリングにより、異なった細胞集団の存在が認識されると、それぞれのクラスター(群)に特徴的に発現している具体的な遺伝子を探索し、細胞集団の持つバイオマーカーによって、そのクラスター(群)の同定が可能になる。例えば、既に神経細胞とグリア細胞に特異的に発現する典型的マーカーはよく知られており、それぞれのクラスターの識別は容易である。更に、神経細胞のタイプ(下記参考)を区別できるマーカーや、外部刺激によって遺伝子発現状態が変化した神経細胞の状態は、In situ hybridizationや免疫組織化学などにより確認できる。このようなクラスターごとに発現が異なる遺伝子(差次的発現遺伝子)を見つけるためには(Differential expression analysis, DE analysis)、SeuratのFindMarkersコマンド中でも利用可能であるコード(MAST、DESeq2など[98][99][100])を用いることができる。細胞ごとの差次的発現遺伝子のVisualization(表示可視化)には、ドットプロット(dot plot)、ヴァイオリンプロット(violin plot)、リッジプロット(Ridge plot, joy plot)、UMAPなどの次元圧縮図上に転写物量をプロットするFeatureプロット(feature plot)などが、目的に応じて頻繁に用いられる(図4)。

A. ドットプロット。B.ヴァイオリンプロット。C. リッジプロット。D. UMAP(灰色)に転写物量(青)をプロットした Featureプロット。網膜の視細胞のデータを用いて執筆者が作製[95]。
擬時系列解析
実験的なノイズとは別に生物学的に意味のある遺伝子発現の変動には、位置情報、細胞周期、概日リズム、発現変動が大きい破裂型プロモーターの作動などの理由で 変動が見られるものもある[38][101]。特に、刺激・薬剤処理やさまざまな病態の進行や治療に伴う細胞の変化、発生途上にある細胞系譜や細胞分化といった細胞の遷移状態の解析(軌道推定(Trajectory inference);擬時系列解析(擬似時系列解析)、Pseudo-time analysis)には、scRNA-seqデータを用いることが効果的である[102][103][104]。しばしば用いられるMonocle3 [105][3]など、多くのコードを収集、比較しているサイトがある [4][5]。RNA velocityといったスプライシングされていく転写産物の量から細胞の分化状態を推定する方法もある[106][107]。しかし、これらの方法は、あくまで細胞系譜や細胞分化の推定に過ぎない。細胞系譜を更に確実に観察しつつ、scRNA-seqを行うことで、細胞タイプの系統関係を調べる方法として、CRISPR-Cas9を用いたゲノム編集による痕跡追跡記録法を導入したscGESTALT[108]、ScarTrace[109] 、LINNAEUS[110]、あるいはアデノシンデアミナーゼでRNA編集を行いタイムスタンプを入れる方法[111]がある。1塩基バリアント(Single-nucleotide variants: SNV)の系統的解析は、細胞の不均一性や系統的な関係を明らかにするための最も有望なアプローチの一つである[112]。
遺伝子制御ネットワーク、パスウェイ解析など
また細胞分化や刺激などによる変動に伴う特徴的な遺伝子発現状態をscRNA-seqで観察することは、遺伝子制御ネットワーク(例、SCENIC[113])、代謝経路やシグナル伝達系のためのパスウェイ解析(例、Metascape[114], [6]、Gene Ontolgoy[7])を理解するシステム生物学的な研究として有用である[115]。更に、scRNA-seqで得られた結果をもとに、細胞間相互作用の理解を深めるのを目的とするCellPhoneDB[116][8]、NicheNet[117]、SVCA[118]などがある。特に、Perturb-seq[119] やその変法[120]は、CRISPRライブラリーによるゲノム編集を施した細胞をscRNA-seqで解析することで、ゲノム編集で破壊された遺伝子の機能や遺伝子間の相互作用の理解を可能にしている後述する複数モダリティ情報を取り込んだscRNA-seqの1つであり、注目されている。
神経科学研究への適用
神経系細胞ビッグデータとしてのscRNA-seq
様々な神経・精神疾患について理解しその診断や治療に役立てるためには、神経細胞、グリア細胞を中心にした神経系にある細胞の種類や状態を識別し、それぞれの細胞における分子的な変化を観察することが重要である [121][122]。本項目で解説してきたscRNA-seq技術は、神経系に見られるそれぞれの細胞のトランスクリプトームについてビッグデータを提供することで、この細胞の種類や状態の識別に新たな判断材料を与えつつある。近年、中枢神経系のアストロサイト、オリゴデンドロサイト、ミクログリアといったグリア細胞も均一ではなく、内在的な多様性や外部因子による状態の変動が報告されてきている。神経細胞は、著しく多様であり、この多様性が神経系の多彩で複雑な機能発現の基盤となっている。従来の神経科学では、神経細胞の多様性は、それぞれの神経細胞の解剖学的な位置、発現している分子、電気生理学、結合性、形態、神経伝達物質、神経伝達物質受容体とシグナル伝達によって識別されてきている。こうした神経細胞の多様性を便宜的に記述するのに、タイプ(type)、クラス(class)、サブクラス(subclass)、サブタイプ(subtype) というような用語が用いられてきた。しかし、ここでは混乱を防ぐため、Masland(2004)[123]が提唱し、広く受けいれられている「クラス」と「タイプ」という単語を用いることとする[124]。タイプは、これ以上分類することができないとされる階層であり、共通性を持つタイプの集団がクラスである。例えば、大脳皮質の錐体細胞、網膜神経節細胞といった大雑把な区分はクラスである。大脳皮質の錐体細胞というクラスは、層や領野によって異なるタイプ、網膜神経節細胞には視覚情報に対して応答が異なるタイプが存在する。scRNA-seqは、「タイプ」の理解に新たな視点を提供している。
神経系へのscRNA-seqの適用
scRNA-seqの神経系での利用については、次々と新しい論文やプレプリントが発表されており、ここではscRNA-seqで得られてきた情報の典型例を紹介することにとどめる。
大脳皮質には、錐体細胞や非錐体細胞などの神経細胞や様々なグリア細胞などが見られ、古くから神経細胞タイプの識別が行われてきた。初期のFluidigm C1を用いたscRNA-seq技術でも、マウス皮質の小規模な細胞数を分類した研究で、これまで知られていた主要な神経細胞タイプとは違うタイプが見つかりscRNA-seqの有効性が示された[125]。その後のドロップレット使用の3’エンドリード法を利用した多数の細胞数の解析で、更に多数の神経細胞のタイプが見つかっている[124][51][126][127][128][129][130][9] [10]。特に、GABA作動性介在神経細胞タイプの多様性とその発生[131][132][133][134]についての、これまでの組織化学的な研究からは得られていなかった多くの情報は重要であろう。また、初期の発生過程[135][136][137][138][139]、老化[140]の理解が、scRNA-seq技術を利用することで進んでいる。更に、神経活動や臨界期に伴い変化するmRNAも細胞ごとに調査され興味深い[141] [142]。
ヒトを含めた霊長類の大脳についても発達段階を含めてscRNA-seqが適用されてきている[143][144][145][146][51][147][148][149][150][151] [152][11][12]。ヒトや霊長類に特徴的とされる島のvon Economo神経細胞(紡錘細胞)のような希少な神経細胞のscRNA-seqにも成功している[153]。
海馬[154][155][156][157]では、これまでの研究で記載されてきた神経細胞のタイプの存在が確認され、更に新規のタイプが見つかった。中枢神経系では、その他、外側膝状体[158]、大脳基底核[159]、視床下部[160][161] [162][163] [164][165]、線条体[166][167][168]、中脳[169][170][171]、手綱核[172]、発生中の間脳[173] 、さらに小脳[174][175][176]などの結果が報告されてきている。例えば、構成する細胞についての情報が詳細に研究されてきたと思われていたマウスの小脳においても、分子層にこれまでの星状細胞、バスケット細胞というカテゴリーとは違ったギャップジャンクションに特徴を持つ2種類の神経細胞があることが示唆されている[177]。
脳の外部では、運動神経[13]、感覚神経[178][179]、らせん神経節[180][181] 、嗅覚神経[182][183]、腸神経系 [184][185]、網膜[186][187][188][189][190][191][192][193][194][195][196][197][198][14][199][15][95][200]でのscRNA-seqデータがある。
またiPS細胞やES細胞由来の神経組織オルガノイドに含まれる神経細胞タイプを知る上でも利用されている[201][202][203][204][205][206]。このようなアプローチは、ネアンデルタール人型の遺伝子を持つ脳オルガノイドの解析[207]やSARS-CoV-2に感染する脳オルガノイド中の細胞の同定[208]など、新たな応用例が発表されてきており興味深い。
神経細胞以外の細胞
上衣細胞[209]は、神経幹細胞としての役割が示唆されてきたが、scRNA-seqによる解析ではその可能性が支持されなかった。グリア細胞では、ラジアルグリア[210][211][212][146][145]、アストロサイト[213][214][16]に多様性があることが示唆されてきている。また、オリゴデンドロサイト[215][216][217][218][219][17]については、これまで細胞生物学的に研究されてきた分化の過程がscRNA-seqにより詳細に解析されている。ミクログリアは、神経系の発達、老化、損傷などに伴う重要な遺伝子発現状態の変化がscRNA-seqにより詳細に明らかになった[220][221][222][42][223][224]。また、CNS境界関連マクロファージ(BAM) [225]や脳血管系[226]のscRNA-seqも実施されている。
疾患
scRNA-seqは、疾患の理解にも有用である。scRNA-seqでは、疾患に伴う遺伝子発現状態の変化を細胞タイプごとに観察することができるので、バルクRNA-seqでは埋もれていた遺伝子発現状態の変化や細胞ごとの変化を検出できるという長所がある。 例えば、筋萎縮性側索硬化症[227]、多発性硬化症[218][228][229]、アルツハイマー病やそのモデル動物[230][231][221] [18][232][233]、統合失調症[234][235]、てんかん[236]、自閉症やレット症候群[237][238]、シャルコー・マリー・トゥース病[239]、ダウン症[19]、パーキンソン病[240][241]、ハンチントン病[242]、がん[243][244]などに適用されている。最近、Perturb-seqにより、自閉症に関わる遺伝子の欠損に伴う細胞状態の変化などもscRNA-seqで報告されており[245]、疾患の理解のための新たな実験系の開発も始まりつつある。
scRNA-seqの展望
神経系の多様性と進化
NGSを用いることで、どんな生物種にも適用可能なscRNA-seqは、既に多様な生物の神経系の細胞の理解、更には種間の相同性や差異の研究に利用されており、神経系の進化を細胞レベルで考察するのに有用であろう(例、線虫[28]、ショウジョウバエ[246][247][248][187][249]、カタユウレイボヤCiona intestinalis[250][251]、ゼブラフィッシュ[252][253]、アカミミガメTrachemys scripta elegans、トカゲPogona vitticeps, PV[254]、ニワトリ[95]、霊長類[255][256][20])。ただ、遺伝子やトランスクリプトームの研究が進んでいる生物種では比較的容易であるが、遺伝子のアノテーションが十分でない生物種を用いる場合、scRNA-seqのデータ解析は困難を伴う。また種を超えた細胞タイプの相同性の理解には様々な工夫が必要である[257][191][21]。
データベースと統合
獲得されたscRNA-seqのデータは様々な目的で利用できるので、データベース化し利用できるようにする必要がある。神経系のトランスクリプトーム一般のデータベースが多数公開されており[258]、scRNA-seqのデータも基本的にNCBIのGene Expression Omnibusに登録されている。また、オープンサイエンス推進のためにcommon coordinate framework (CCF) やcentral annotation platform (CAP)という概念のもと、特にscRNA-seqを意識したものとして、米国のBRAIN Initiative Cell Census Consortium[259]、Human Cell Atlas ProjectのHuman Cell Atlas Data Portal、そのマウス版であるTabula Muris[260]やSten Linnarssonラボのマウス脳発生データベース、アレン脳研究所のAllen Brain Atlas、ブロード研究所のSingle Cell Portalなどのデータベースが稼働している。また、異なった方法や実験で得られたscRNA-seqのデータや後述の複数モダリティのシングルセルオミクスのデータを体系的に比較することも重要であり、CCA (Canonical correlation analysis)[66], Seurat 3.0以降に組み込まれたMMN (Mutual Nearest Neighbors)、LIGER[261] 、Harmony[262] 、MetaNeighber[263]、Conos[264][22]のようなアルゴリズムが開発され、後述の複数モダリティのシングルセルオミクスを組み込んだ統合サイトもでき始めている。またデータベースを利用して発現類似性検索も研究されている[265][266]。
空間トランスクリプトミクス
多数の細胞を扱うscRNA-seqの弱点は、組織から細胞や細胞核を解離する必要があるので、その細胞が存在していた解剖学的あるいは空間的な位置の情報を消去してしまうということである。組織切片におけるタンパク質などの分布は免疫組織化学、mRNAの分布はin situ hybridizationで検出することができるが、数多くのmRNAの分布を情報処理技術と組み合わせ一気に同定する方法がscRNA-seqと同様に開発されてきている(Slide-seq[267][268]、HDST[269]、Expansion sequencing[23]など[270], [271][227])、更に10x Genomics社が市販するVisium(現時点ではシングルセルレベルではない)などがある。現状では、大きな組織の空間トランスクリプトミクスは、空間解像度は限定されており、技術普及の観点からも課題が多い。しかし、そのデータを解析するためのアルゴリズム[272][273][274][24][25][26]、更にMerFish [275]、corrFISH[276]のように、subcellularレベルで多数のmRNAを検出する方法が多数開発されてきており([277] osmFISH[278]、STARmap (spatially-resolved transcript amplicon readout mapping) [279]、seqFISH+[280]、pciSeq(probabilistic cell typing by in situ sequencing)[27]、DSP(Digital Spatial Profiling) [281]、scRNA-seqと組み合わせることで、その弱点を補う空間トランスクリプトミクスにも利用され始め[163][28]、今後の発展が期待される分野である[282]。
統合解析
同一の細胞からscRNA-seqの情報だけでなく、ゲノム配列、ATAC-seqなどによるエピゲノム解析、少数のタンパク質、あるいはプロテオームなど複数のモダリティを同時に観察するオミクス(Single-cell multimodal omics)が注目されている[283][284]。2019年には、Nature Methodsの「Methods of the Year」に選ばれており、現状については、その特集号などを参考にされたい。例えば、細胞表面に提示されているマーカー分子に対する抗体にDNAを付加することで、そのマーカーを発現する細胞のトランスクリプトームを解析するCITE-seq[285]、 REAP-seq[286]も、既知の細胞表面マーカーの発現とscRNA-seqが同時に観察できる方法である。
複数モダリティのシングルセルオミクスとして、神経科学分野で注目されるのは、scRNA-seqをパッチクランプによる電気生理学的情報と組み合わせたPatch-seq[287] [288]である。また、ゲノムDNAとscRNA-seqを同時に観察することによって、近年、精神疾患の観点から注目されている発生途中で生じる遺伝子変異を研究するPRDD-seqは今後の展開が注目される[289]。最後に、BARseq (barcoded anatomy resolved by sequencing) [290]、CONNECTID[29]、Epi-Retro-seq[291]のような方法は、コネクトーム(神経細胞の結合性)と遺伝子発現状態を記録できるオミクスの新たな方向として興味深い。
関連項目
参考文献
- ↑
Wang, Z., Gerstein, M., & Snyder, M. (2009).
RNA-Seq: a revolutionary tool for transcriptomics. Nature reviews. Genetics, 10(1), 57-63. [PubMed:19015660] [PMC] [WorldCat] [DOI] - ↑
Stark, R., Grzelak, M., & Hadfield, J. (2019).
RNA sequencing: the teenage years. Nature reviews. Genetics, 20(11), 631-656. [PubMed:31341269] [WorldCat] [DOI] - ↑
Picot, J., Guerin, C.L., Le Van Kim, C., & Boulanger, C.M. (2012).
Flow cytometry: retrospective, fundamentals and recent instrumentation. Cytotechnology, 64(2), 109-30. [PubMed:22271369] [PMC] [WorldCat] [DOI] - ↑
Spitzer, M.H., & Nolan, G.P. (2016).
Mass Cytometry: Single Cells, Many Features. Cell, 165(4), 780-91. [PubMed:27153492] [PMC] [WorldCat] [DOI] - ↑
Livesey, F.J. (2003).
Strategies for microarray analysis of limiting amounts of RNA. Briefings in functional genomics & proteomics, 2(1), 31-6. [PubMed:15239941] [WorldCat] [DOI] - ↑
Eberwine, J., Yeh, H., Miyashiro, K., Cao, Y., Nair, S., Finnell, R., ..., & Coleman, P. (1992).
Analysis of gene expression in single live neurons. Proceedings of the National Academy of Sciences of the United States of America, 89(7), 3010-4. [PubMed:1557406] [PMC] [WorldCat] [DOI] - ↑
Sucher, N.J., & Deitcher, D.L. (1995).
PCR and patch-clamp analysis of single neurons. Neuron, 14(6), 1095-100. [PubMed:7541630] [WorldCat] [DOI] - ↑
Buck, L., & Axel, R. (1991).
A novel multigene family may encode odorant receptors: a molecular basis for odor recognition. Cell, 65(1), 175-87. [PubMed:1840504] [WorldCat] [DOI] - ↑
Dulac, C., & Axel, R. (1995).
A novel family of genes encoding putative pheromone receptors in mammals. Cell, 83(2), 195-206. [PubMed:7585937] [WorldCat] [DOI] - ↑
Tanabe, Y., William, C., & Jessell, T.M. (1998).
Specification of motor neuron identity by the MNR2 homeodomain protein. Cell, 95(1), 67-80. [PubMed:9778248] [WorldCat] [DOI] - ↑
Yamagata, M., Weiner, J.A., & Sanes, J.R. (2002).
Sidekicks: synaptic adhesion molecules that promote lamina-specific connectivity in the retina. Cell, 110(5), 649-60. [PubMed:12230981] [WorldCat] [DOI] - ↑
Kamme, F., Salunga, R., Yu, J., Tran, D.T., Zhu, J., Luo, L., ..., & Erlander, M. (2003).
Single-cell microarray analysis in hippocampus CA1: demonstration and validation of cellular heterogeneity. The Journal of neuroscience : the official journal of the Society for Neuroscience, 23(9), 3607-15. [PubMed:12736331] [PMC] [WorldCat] - ↑
Kurimoto, K., Yabuta, Y., Ohinata, Y., Ono, Y., Uno, K.D., Yamada, R.G., ..., & Saitou, M. (2006).
An improved single-cell cDNA amplification method for efficient high-density oligonucleotide microarray analysis. Nucleic acids research, 34(5), e42. [PubMed:16547197] [PMC] [WorldCat] [DOI] - ↑
Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., ..., & Surani, M.A. (2009).
mRNA-Seq whole-transcriptome analysis of a single cell. Nature methods, 6(5), 377-82. [PubMed:19349980] [WorldCat] [DOI] - ↑ 15.0 15.1 15.2
Mereu, E., Lafzi, A., Moutinho, C., Ziegenhain, C., McCarthy, D.J., Álvarez-Varela, A., ..., & Heyn, H. (2020).
Benchmarking single-cell RNA-sequencing protocols for cell atlas projects. Nature biotechnology, 38(6), 747-755. [PubMed:32518403] [WorldCat] [DOI] - ↑
Ramsköld, D., Luo, S., Wang, Y.C., Li, R., Deng, Q., Faridani, O.R., ..., & Sandberg, R. (2012).
Full-length mRNA-Seq from single-cell levels of RNA and individual circulating tumor cells. Nature biotechnology, 30(8), 777-82. [PubMed:22820318] [PMC] [WorldCat] [DOI] - ↑
Picelli, S., Björklund, Å.K., Faridani, O.R., Sagasser, S., Winberg, G., & Sandberg, R. (2013).
Smart-seq2 for sensitive full-length transcriptome profiling in single cells. Nature methods, 10(11), 1096-8. [PubMed:24056875] [WorldCat] [DOI] - ↑
Picelli, S., Faridani, O.R., Björklund, A.K., Winberg, G., Sagasser, S., & Sandberg, R. (2014).
Full-length RNA-seq from single cells using Smart-seq2. Nature protocols, 9(1), 171-81. [PubMed:24385147] [WorldCat] [DOI] - ↑
Hagemann-Jensen, M., Ziegenhain, C., Chen, P., Ramsköld, D., Hendriks, G.J., Larsson, A.J.M., ..., & Sandberg, R. (2020).
Single-cell RNA counting at allele and isoform resolution using Smart-seq3. Nature biotechnology, 38(6), 708-714. [PubMed:32518404] [WorldCat] [DOI] - ↑ 20.0 20.1
Islam, S., Kjällquist, U., Moliner, A., Zajac, P., Fan, J.B., Lönnerberg, P., & Linnarsson, S. (2011).
Characterization of the single-cell transcriptional landscape by highly multiplex RNA-seq. Genome research, 21(7), 1160-7. [PubMed:21543516] [PMC] [WorldCat] [DOI] - ↑
Hashimshony, T., Wagner, F., Sher, N., & Yanai, I. (2012).
CEL-Seq: single-cell RNA-Seq by multiplexed linear amplification. Cell reports, 2(3), 666-73. [PubMed:22939981] [WorldCat] [DOI] - ↑
Hashimshony, T., Senderovich, N., Avital, G., Klochendler, A., de Leeuw, Y., Anavy, L., ..., & Yanai, I. (2016).
CEL-Seq2: sensitive highly-multiplexed single-cell RNA-Seq. Genome biology, 17, 77. [PubMed:27121950] [PMC] [WorldCat] [DOI] - ↑
Jaitin, D.A., Kenigsberg, E., Keren-Shaul, H., Elefant, N., Paul, F., Zaretsky, I., ..., & Amit, I. (2014).
Massively parallel single-cell RNA-seq for marker-free decomposition of tissues into cell types. Science (New York, N.Y.), 343(6172), 776-9. [PubMed:24531970] [PMC] [WorldCat] [DOI] - ↑
Kivioja, T., Vähärautio, A., Karlsson, K., Bonke, M., Enge, M., Linnarsson, S., & Taipale, J. (2011).
Counting absolute numbers of molecules using unique molecular identifiers. Nature methods, 9(1), 72-4. [PubMed:22101854] [WorldCat] [DOI] - ↑
Islam, S., Zeisel, A., Joost, S., La Manno, G., Zajac, P., Kasper, M., ..., & Linnarsson, S. (2014).
Quantitative single-cell RNA-seq with unique molecular identifiers. Nature methods, 11(2), 163-6. [PubMed:24363023] [WorldCat] [DOI] - ↑ 26.0 26.1
Gierahn, T.M., Wadsworth, M.H., Hughes, T.K., Bryson, B.D., Butler, A., Satija, R., ..., & Shalek, A.K. (2017).
Seq-Well: portable, low-cost RNA sequencing of single cells at high throughput. Nature methods, 14(4), 395-398. [PubMed:28192419] [PMC] [WorldCat] [DOI] - ↑
Han, X., Wang, R., Zhou, Y., Fei, L., Sun, H., Lai, S., ..., & Guo, G. (2018).
Mapping the Mouse Cell Atlas by Microwell-Seq. Cell, 172(5), 1091-1107.e17. [PubMed:29474909] [WorldCat] [DOI] - ↑ 28.0 28.1
Cao, J., Packer, J.S., Ramani, V., Cusanovich, D.A., Huynh, C., Daza, R., ..., & Shendure, J. (2017).
Comprehensive single-cell transcriptional profiling of a multicellular organism. Science (New York, N.Y.), 357(6352), 661-667. [PubMed:28818938] [PMC] [WorldCat] [DOI] - ↑ 29.0 29.1
Rosenberg, A.B., Roco, C.M., Muscat, R.A., Kuchina, A., Sample, P., Yao, Z., ..., & Seelig, G. (2018).
Single-cell profiling of the developing mouse brain and spinal cord with split-pool barcoding. Science (New York, N.Y.), 360(6385), 176-182. [PubMed:29545511] [WorldCat] [DOI] - ↑
See, P., Lum, J., Chen, J., & Ginhoux, F. (2018).
A Single-Cell Sequencing Guide for Immunologists. Frontiers in immunology, 9, 2425. [PubMed:30405621] [PMC] [WorldCat] [DOI] - ↑
Svensson, V., da Veiga Beltrame, E., & Pachter, L. (2020).
A curated database reveals trends in single-cell transcriptomics. Database : the journal of biological databases and curation, 2020. [PubMed:33247933] [PMC] [WorldCat] [DOI] - ↑
Cao, J., Spielmann, M., Qiu, X., Huang, X., Ibrahim, D.M., Hill, A.J., ..., & Shendure, J. (2019).
The single-cell transcriptional landscape of mammalian organogenesis. Nature, 566(7745), 496-502. [PubMed:30787437] [PMC] [WorldCat] [DOI] - ↑
Klein, A.M., Mazutis, L., Akartuna, I., Tallapragada, N., Veres, A., Li, V., ..., & Kirschner, M.W. (2015).
Droplet barcoding for single-cell transcriptomics applied to embryonic stem cells. Cell, 161(5), 1187-1201. [PubMed:26000487] [PMC] [WorldCat] [DOI] - ↑
Macosko, E.Z., Basu, A., Satija, R., Nemesh, J., Shekhar, K., Goldman, M., ..., & McCarroll, S.A. (2015).
Highly Parallel Genome-wide Expression Profiling of Individual Cells Using Nanoliter Droplets. Cell, 161(5), 1202-1214. [PubMed:26000488] [PMC] [WorldCat] [DOI] - ↑ 35.0 35.1
Zhang, X., Li, T., Liu, F., Chen, Y., Yao, J., Li, Z., ..., & Wang, J. (2019).
Comparative Analysis of Droplet-Based Ultra-High-Throughput Single-Cell RNA-Seq Systems. Molecular cell, 73(1), 130-142.e5. [PubMed:30472192] [WorldCat] [DOI] - ↑
Zheng, G.X., Terry, J.M., Belgrader, P., Ryvkin, P., Bent, Z.W., Wilson, R., ..., & Bielas, J.H. (2017).
Massively parallel digital transcriptional profiling of single cells. Nature communications, 8, 14049. [PubMed:28091601] [PMC] [WorldCat] [DOI] - ↑
Svensson, V., da Veiga Beltrame, E., & Pachter, L. (2020).
A curated database reveals trends in single-cell transcriptomics. Database : the journal of biological databases and curation, 2020. [PubMed:33247933] [PMC] [WorldCat] [DOI] - ↑ 38.0 38.1
Luecken, M.D., & Theis, F.J. (2019).
Current best practices in single-cell RNA-seq analysis: a tutorial. Molecular systems biology, 15(6), e8746. [PubMed:31217225] [PMC] [WorldCat] [DOI] - ↑
Hwang, B., Lee, J.H., & Bang, D. (2018).
Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & molecular medicine, 50(8), 96. [PubMed:30089861] [PMC] [WorldCat] [DOI] - ↑
Ho, H., Both, M., Siniard, A., Sharma, S., Notwell, J.H., Wallace, M., ..., & Portmann, T. (2018).
A Guide to Single-Cell Transcriptomics in Adult Rodent Brain: The Medium Spiny Neuron Transcriptome Revisited. Frontiers in cellular neuroscience, 12, 159. [PubMed:29970990] [PMC] [WorldCat] [DOI] - ↑
Lacar, B., Linker, S.B., Jaeger, B.N., Krishnaswami, S.R., Barron, J.J., Kelder, M.J.E., ..., & Gage, F.H. (2016).
Nuclear RNA-seq of single neurons reveals molecular signatures of activation. Nature communications, 7, 11022. [PubMed:27090946] [PMC] [WorldCat] [DOI] - ↑ 42.0 42.1
Hammond, T.R., Dufort, C., Dissing-Olesen, L., Giera, S., Young, A., Wysoker, A., ..., & Stevens, B. (2019).
Single-Cell RNA Sequencing of Microglia throughout the Mouse Lifespan and in the Injured Brain Reveals Complex Cell-State Changes. Immunity, 50(1), 253-271.e6. [PubMed:30471926] [PMC] [WorldCat] [DOI] - ↑
Wu, Y.E., Pan, L., Zuo, Y., Li, X., & Hong, W. (2017).
Detecting Activated Cell Populations Using Single-Cell RNA-Seq. Neuron, 96(2), 313-329.e6. [PubMed:29024657] [WorldCat] [DOI] - ↑
Adam, M., Potter, A.S., & Potter, S.S. (2017).
Psychrophilic proteases dramatically reduce single-cell RNA-seq artifacts: a molecular atlas of kidney development. Development (Cambridge, England), 144(19), 3625-3632. [PubMed:28851704] [PMC] [WorldCat] [DOI] - ↑
O'Flanagan, C.H., Campbell, K.R., Zhang, A.W., Kabeer, F., Lim, J.L.P., Biele, J., ..., & Aparicio, S. (2019).
Dissociation of solid tumor tissues with cold active protease for single-cell RNA-seq minimizes conserved collagenase-associated stress responses. Genome biology, 20(1), 210. [PubMed:31623682] [PMC] [WorldCat] [DOI] - ↑
Alles, J., Karaiskos, N., Praktiknjo, S.D., Grosswendt, S., Wahle, P., Ruffault, P.L., ..., & Rajewsky, N. (2017).
Cell fixation and preservation for droplet-based single-cell transcriptomics. BMC biology, 15(1), 44. [PubMed:28526029] [PMC] [WorldCat] [DOI] - ↑
Attar, M., Sharma, E., Li, S., Bryer, C., Cubitt, L., Broxholme, J., ..., & Bowden, R. (2018).
A practical solution for preserving single cells for RNA sequencing. Scientific reports, 8(1), 2151. [PubMed:29391536] [PMC] [WorldCat] [DOI] - ↑
Grindberg, R.V., Yee-Greenbaum, J.L., McConnell, M.J., Novotny, M., O'Shaughnessy, A.L., Lambert, G.M., ..., & Lasken, R.S. (2013).
RNA-sequencing from single nuclei. Proceedings of the National Academy of Sciences of the United States of America, 110(49), 19802-7. [PubMed:24248345] [PMC] [WorldCat] [DOI] - ↑
Krishnaswami, S.R., Grindberg, R.V., Novotny, M., Venepally, P., Lacar, B., Bhutani, K., ..., & Lasken, R.S. (2016).
Using single nuclei for RNA-seq to capture the transcriptome of postmortem neurons. Nature protocols, 11(3), 499-524. [PubMed:26890679] [PMC] [WorldCat] [DOI] - ↑
Habib, N., Li, Y., Heidenreich, M., Swiech, L., Avraham-Davidi, I., Trombetta, J.J., ..., & Regev, A. (2016).
Div-Seq: Single-nucleus RNA-Seq reveals dynamics of rare adult newborn neurons. Science (New York, N.Y.), 353(6302), 925-8. [PubMed:27471252] [PMC] [WorldCat] [DOI] - ↑ 51.0 51.1 51.2 51.3
Habib, N., Avraham-Davidi, I., Basu, A., Burks, T., Shekhar, K., Hofree, M., ..., & Regev, A. (2017).
Massively parallel single-nucleus RNA-seq with DroNc-seq. Nature methods, 14(10), 955-958. [PubMed:28846088] [PMC] [WorldCat] [DOI] - ↑
Hu, P., Fabyanic, E., Kwon, D.Y., Tang, S., Zhou, Z., & Wu, H. (2017).
Dissecting Cell-Type Composition and Activity-Dependent Transcriptional State in Mammalian Brains by Massively Parallel Single-Nucleus RNA-Seq. Molecular cell, 68(5), 1006-1015.e7. [PubMed:29220646] [PMC] [WorldCat] [DOI] - ↑
Bakken, T.E., Hodge, R.D., Miller, J.A., Yao, Z., Nguyen, T.N., Aevermann, B., ..., & Tasic, B. (2018).
Single-nucleus and single-cell transcriptomes compared in matched cortical cell types. PloS one, 13(12), e0209648. [PubMed:30586455] [PMC] [WorldCat] [DOI] - ↑
Lake, B.B., Codeluppi, S., Yung, Y.C., Gao, D., Chun, J., Kharchenko, P.V., ..., & Zhang, K. (2017).
A comparative strategy for single-nucleus and single-cell transcriptomes confirms accuracy in predicted cell-type expression from nuclear RNA. Scientific reports, 7(1), 6031. [PubMed:28729663] [PMC] [WorldCat] [DOI] - ↑
Amamoto, R., Zuccaro, E., Curry, N.C., Khurana, S., Chen, H.H., Cepko, C.L., & Arlotta, P. (2020).
FIN-Seq: transcriptional profiling of specific cell types from frozen archived tissue of the human central nervous system. Nucleic acids research, 48(1), e4. [PubMed:31728515] [WorldCat] [DOI] - ↑
Ding, J., Adiconis, X., Simmons, S.K., Kowalczyk, M.S., Hession, C.C., Marjanovic, N.D., ..., & Levin, J.Z. (2020).
Systematic comparison of single-cell and single-nucleus RNA-sequencing methods. Nature biotechnology. [PubMed:32341560] [WorldCat] [DOI] - ↑
Sheng, K., Cao, W., Niu, Y., Deng, Q., & Zong, C. (2017).
Effective detection of variation in single-cell transcriptomes using MATQ-seq. Nature methods, 14(3), 267-270. [PubMed:28092691] [WorldCat] [DOI] - ↑
Hayashi, T., Ozaki, H., Sasagawa, Y., Umeda, M., Danno, H., & Nikaido, I. (2018).
Single-cell full-length total RNA sequencing uncovers dynamics of recursive splicing and enhancer RNAs. Nature communications, 9(1), 619. [PubMed:29434199] [PMC] [WorldCat] [DOI] - ↑
Buenrostro, J.D., Wu, B., Litzenburger, U.M., Ruff, D., Gonzales, M.L., Snyder, M.P., ..., & Greenleaf, W.J. (2015).
Single-cell chromatin accessibility reveals principles of regulatory variation. Nature, 523(7561), 486-90. [PubMed:26083756] [PMC] [WorldCat] [DOI] - ↑
Preissl, S., Fang, R., Huang, H., Zhao, Y., Raviram, R., Gorkin, D.U., ..., & Ren, B. (2018).
Single-nucleus analysis of accessible chromatin in developing mouse forebrain reveals cell-type-specific transcriptional regulation. Nature neuroscience, 21(3), 432-439. [PubMed:29434377] [PMC] [WorldCat] [DOI] - ↑
Cusanovich, D.A., Daza, R., Adey, A., Pliner, H.A., Christiansen, L., Gunderson, K.L., ..., & Shendure, J. (2015).
Multiplex single cell profiling of chromatin accessibility by combinatorial cellular indexing. Science (New York, N.Y.), 348(6237), 910-4. [PubMed:25953818] [PMC] [WorldCat] [DOI] - ↑
Lake, B.B., Chen, S., Sos, B.C., Fan, J., Kaeser, G.E., Yung, Y.C., ..., & Zhang, K. (2018).
Integrative single-cell analysis of transcriptional and epigenetic states in the human adult brain. Nature biotechnology, 36(1), 70-80. [PubMed:29227469] [WorldCat] [DOI] - ↑
Luo, C., Keown, C.L., Kurihara, L., Zhou, J., He, Y., Li, J., ..., & Ecker, J.R. (2017).
Single-cell methylomes identify neuronal subtypes and regulatory elements in mammalian cortex. Science (New York, N.Y.), 357(6351), 600-604. [PubMed:28798132] [PMC] [WorldCat] [DOI] - ↑
Luo, C., Rivkin, A., Zhou, J., Sandoval, J.P., Kurihara, L., Lucero, J., ..., & Ecker, J.R. (2018).
Robust single-cell DNA methylome profiling with snmC-seq2. Nature communications, 9(1), 3824. [PubMed:30237449] [PMC] [WorldCat] [DOI] - ↑
Harris, R.A., Wang, T., Coarfa, C., Nagarajan, R.P., Hong, C., Downey, S.L., ..., & Costello, J.F. (2010).
Comparison of sequencing-based methods to profile DNA methylation and identification of monoallelic epigenetic modifications. Nature biotechnology, 28(10), 1097-105. [PubMed:20852635] [PMC] [WorldCat] [DOI] - ↑ 66.0 66.1
Butler, A., Hoffman, P., Smibert, P., Papalexi, E., & Satija, R. (2018).
Integrating single-cell transcriptomic data across different conditions, technologies, and species. Nature biotechnology, 36(5), 411-420. [PubMed:29608179] [PMC] [WorldCat] [DOI] - ↑
Stuart, T., Butler, A., Hoffman, P., Hafemeister, C., Papalexi, E., Mauck, W.M., ..., & Satija, R. (2019).
Comprehensive Integration of Single-Cell Data. Cell, 177(7), 1888-1902.e21. [PubMed:31178118] [PMC] [WorldCat] [DOI] - ↑
Wolf, F.A., Angerer, P., & Theis, F.J. (2018).
SCANPY: large-scale single-cell gene expression data analysis. Genome biology, 19(1), 15. [PubMed:29409532] [PMC] [WorldCat] [DOI] - ↑
Haghverdi, L., Lun, A.T.L., Morgan, M.D., & Marioni, J.C. (2018).
Batch effects in single-cell RNA-sequencing data are corrected by matching mutual nearest neighbors. Nature biotechnology, 36(5), 421-427. [PubMed:29608177] [PMC] [WorldCat] [DOI] - ↑
Tung, P.Y., Blischak, J.D., Hsiao, C.J., Knowles, D.A., Burnett, J.E., Pritchard, J.K., & Gilad, Y. (2017).
Batch effects and the effective design of single-cell gene expression studies. Scientific reports, 7, 39921. [PubMed:28045081] [PMC] [WorldCat] [DOI] - ↑
Tran, H.T.N., Ang, K.S., Chevrier, M., Zhang, X., Lee, N.Y.S., Goh, M., & Chen, J. (2020).
A benchmark of batch-effect correction methods for single-cell RNA sequencing data. Genome biology, 21(1), 12. [PubMed:31948481] [PMC] [WorldCat] [DOI] - ↑
Hou, W., Ji, Z., Ji, H., & Hicks, S.C. (2020).
A systematic evaluation of single-cell RNA-sequencing imputation methods. Genome biology, 21(1), 218. [PubMed:32854757] [PMC] [WorldCat] [DOI] - ↑
Hicks, S.C., Townes, F.W., Teng, M., & Irizarry, R.A. (2018).
Missing data and technical variability in single-cell RNA-sequencing experiments. Biostatistics (Oxford, England), 19(4), 562-578. [PubMed:29121214] [PMC] [WorldCat] [DOI] - ↑
Wolock, S.L., Lopez, R., & Klein, A.M. (2019).
Scrublet: Computational Identification of Cell Doublets in Single-Cell Transcriptomic Data. Cell systems, 8(4), 281-291.e9. [PubMed:30954476] [PMC] [WorldCat] [DOI] - ↑
McGinnis, C.S., Murrow, L.M., & Gartner, Z.J. (2019).
DoubletFinder: Doublet Detection in Single-Cell RNA Sequencing Data Using Artificial Nearest Neighbors. Cell systems, 8(4), 329-337.e4. [PubMed:30954475] [PMC] [WorldCat] [DOI] - ↑
DePasquale, E.A.K., Schnell, D.J., Van Camp, P.J., Valiente-Alandí, Í., Blaxall, B.C., Grimes, H.L., ..., & Salomonis, N. (2019).
DoubletDecon: Deconvoluting Doublets from Single-Cell RNA-Sequencing Data. Cell reports, 29(6), 1718-1727.e8. [PubMed:31693907] [PMC] [WorldCat] [DOI] - ↑
Bernstein, N.J., Fong, N.L., Lam, I., Roy, M.A., Hendrickson, D.G., & Kelley, D.R. (2020).
Solo: Doublet Identification in Single-Cell RNA-Seq via Semi-Supervised Deep Learning. Cell systems, 11(1), 95-101.e5. [PubMed:32592658] [WorldCat] [DOI] - ↑
Kang, H.M., Subramaniam, M., Targ, S., Nguyen, M., Maliskova, L., McCarthy, E., ..., & Ye, C.J. (2018).
Multiplexed droplet single-cell RNA-sequencing using natural genetic variation. Nature biotechnology, 36(1), 89-94. [PubMed:29227470] [PMC] [WorldCat] [DOI] - ↑
Huang, Y., McCarthy, D.J., & Stegle, O. (2019).
Vireo: Bayesian demultiplexing of pooled single-cell RNA-seq data without genotype reference. Genome biology, 20(1), 273. [PubMed:31836005] [PMC] [WorldCat] [DOI] - ↑
Xu, J., Falconer, C., Nguyen, Q., Crawford, J., McKinnon, B.D., Mortlock, S., ..., & Coin, L.J.M. (2019).
Genotype-free demultiplexing of pooled single-cell RNA-seq. Genome biology, 20(1), 290. [PubMed:31856883] [PMC] [WorldCat] [DOI] - ↑
Stoeckius, M., Zheng, S., Houck-Loomis, B., Hao, S., Yeung, B.Z., Mauck, W.M., ..., & Satija, R. (2018).
Cell Hashing with barcoded antibodies enables multiplexing and doublet detection for single cell genomics. Genome biology, 19(1), 224. [PubMed:30567574] [PMC] [WorldCat] [DOI] - ↑
Gaublomme, J.T., Li, B., McCabe, C., Knecht, A., Yang, Y., Drokhlyansky, E., ..., & Regev, A. (2019).
Nuclei multiplexing with barcoded antibodies for single-nucleus genomics. Nature communications, 10(1), 2907. [PubMed:31266958] [PMC] [WorldCat] [DOI] - ↑
Heaton, H., Talman, A.M., Knights, A., Imaz, M., Gaffney, D.J., Durbin, R., ..., & Lawniczak, M.K.N. (2020).
Souporcell: robust clustering of single-cell RNA-seq data by genotype without reference genotypes. Nature methods, 17(6), 615-620. [PubMed:32366989] [WorldCat] [DOI] - ↑
Xi, N.M., & Li, J.J. (2020).
Benchmarking Computational Doublet-Detection Methods for Single-Cell RNA Sequencing Data. Cell systems. [PubMed:33338399] [WorldCat] [DOI] - ↑
Farouni, R., Djambazian, H., Ferri, L.E., Ragoussis, J., & Najafabadi, H.S. (2020).
Model-based analysis of sample index hopping reveals its widespread artifacts in multiplexed single-cell RNA-sequencing. Nature communications, 11(1), 2704. [PubMed:32483174] [PMC] [WorldCat] [DOI] - ↑
Brennecke, P., Anders, S., Kim, J.K., Kołodziejczyk, A.A., Zhang, X., Proserpio, V., ..., & Heisler, M.G. (2013).
Accounting for technical noise in single-cell RNA-seq experiments. Nature methods, 10(11), 1093-5. [PubMed:24056876] [WorldCat] [DOI] - ↑
Qiu, P. (2020).
Embracing the dropouts in single-cell RNA-seq analysis. Nature communications, 11(1), 1169. [PubMed:32127540] [PMC] [WorldCat] [DOI] - ↑
Vallejos, C.A., Risso, D., Scialdone, A., Dudoit, S., & Marioni, J.C. (2017).
Normalizing single-cell RNA sequencing data: challenges and opportunities. Nature methods, 14(6), 565-571. [PubMed:28504683] [PMC] [WorldCat] [DOI] - ↑
Kiselev, V.Y., Andrews, T.S., & Hemberg, M. (2019).
Challenges in unsupervised clustering of single-cell RNA-seq data. Nature reviews. Genetics, 20(5), 273-282. [PubMed:30617341] [WorldCat] [DOI] - ↑
Kobak, D., & Berens, P. (2019).
The art of using t-SNE for single-cell transcriptomics. Nature communications, 10(1), 5416. [PubMed:31780648] [PMC] [WorldCat] [DOI] - ↑
Tsuyuzaki, K., Sato, H., Sato, K., & Nikaido, I. (2020).
Benchmarking principal component analysis for large-scale single-cell RNA-sequencing. Genome biology, 21(1), 9. [PubMed:31955711] [PMC] [WorldCat] [DOI] - ↑
Sun, S., Zhu, J., Ma, Y., & Zhou, X. (2019).
Accuracy, robustness and scalability of dimensionality reduction methods for single-cell RNA-seq analysis. Genome biology, 20(1), 269. [PubMed:31823809] [PMC] [WorldCat] [DOI] - ↑
Haghverdi, L., Buettner, F., & Theis, F.J. (2015).
Diffusion maps for high-dimensional single-cell analysis of differentiation data. Bioinformatics (Oxford, England), 31(18), 2989-98. [PubMed:26002886] [WorldCat] [DOI] - ↑
Abdelaal, T., Michielsen, L., Cats, D., Hoogduin, D., Mei, H., Reinders, M.J.T., & Mahfouz, A. (2019).
A comparison of automatic cell identification methods for single-cell RNA sequencing data. Genome biology, 20(1), 194. [PubMed:31500660] [PMC] [WorldCat] [DOI] - ↑ 95.0 95.1 95.2 95.3
Yamagata, M., Yan, W., & Sanes, J.R. (2021).
A cell atlas of the chick retina based on single cell transcriptomics. eLife, 10. [PubMed:33393903] [WorldCat] [DOI] - ↑
Wagner, A., Regev, A., & Yosef, N. (2016).
Revealing the vectors of cellular identity with single-cell genomics. Nature biotechnology, 34(11), 1145-1160. [PubMed:27824854] [PMC] [WorldCat] [DOI] - ↑
Lähnemann, D., Köster, J., Szczurek, E., McCarthy, D.J., Hicks, S.C., Robinson, M.D., ..., & Schönhuth, A. (2020).
Eleven grand challenges in single-cell data science. Genome biology, 21(1), 31. [PubMed:32033589] [PMC] [WorldCat] [DOI] - ↑
Finak, G., McDavid, A., Yajima, M., Deng, J., Gersuk, V., Shalek, A.K., ..., & Gottardo, R. (2015).
MAST: a flexible statistical framework for assessing transcriptional changes and characterizing heterogeneity in single-cell RNA sequencing data. Genome biology, 16, 278. [PubMed:26653891] [PMC] [WorldCat] [DOI] - ↑
Love, M.I., Huber, W., & Anders, S. (2014).
Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome biology, 15(12), 550. [PubMed:25516281] [PMC] [WorldCat] [DOI] - ↑
Wang, T., Li, B., Nelson, C.E., & Nabavi, S. (2019).
Comparative analysis of differential gene expression analysis tools for single-cell RNA sequencing data. BMC bioinformatics, 20(1), 40. [PubMed:30658573] [PMC] [WorldCat] [DOI] - ↑
Kolodziejczyk, A.A., Kim, J.K., Svensson, V., Marioni, J.C., & Teichmann, S.A. (2015).
The technology and biology of single-cell RNA sequencing. Molecular cell, 58(4), 610-20. [PubMed:26000846] [WorldCat] [DOI] - ↑
Cembrowski, M.S., & Menon, V. (2018).
Continuous Variation within Cell Types of the Nervous System. Trends in neurosciences, 41(6), 337-348. [PubMed:29576429] [WorldCat] [DOI] - ↑
Marioni, J.C., & Arendt, D. (2017).
How Single-Cell Genomics Is Changing Evolutionary and Developmental Biology. Annual review of cell and developmental biology, 33, 537-553. [PubMed:28813177] [WorldCat] [DOI] - ↑
Telley, L., & Jabaudon, D. (2018).
A mixed model of neuronal diversity. Nature, 555(7697), 452-454. [PubMed:29565398] [WorldCat] [DOI] - ↑
Cao, J., Spielmann, M., Qiu, X., Huang, X., Ibrahim, D.M., Hill, A.J., ..., & Shendure, J. (2019).
The single-cell transcriptional landscape of mammalian organogenesis. Nature, 566(7745), 496-502. [PubMed:30787437] [PMC] [WorldCat] [DOI] - ↑
La Manno, G., Soldatov, R., Zeisel, A., Braun, E., Hochgerner, H., Petukhov, V., ..., & Kharchenko, P.V. (2018).
RNA velocity of single cells. Nature, 560(7719), 494-498. [PubMed:30089906] [PMC] [WorldCat] [DOI] - ↑
Bergen, V., Lange, M., Peidli, S., Wolf, F.A., & Theis, F.J. (2020).
Generalizing RNA velocity to transient cell states through dynamical modeling. Nature biotechnology, 38(12), 1408-1414. [PubMed:32747759] [WorldCat] [DOI] - ↑
Raj, B., Wagner, D.E., McKenna, A., Pandey, S., Klein, A.M., Shendure, J., ..., & Schier, A.F. (2018).
Simultaneous single-cell profiling of lineages and cell types in the vertebrate brain. Nature biotechnology, 36(5), 442-450. [PubMed:29608178] [PMC] [WorldCat] [DOI] - ↑
Alemany, A., Florescu, M., Baron, C.S., Peterson-Maduro, J., & van Oudenaarden, A. (2018).
Whole-organism clone tracing using single-cell sequencing. Nature, 556(7699), 108-112. [PubMed:29590089] [WorldCat] [DOI] - ↑
Spanjaard, B., Hu, B., Mitic, N., Olivares-Chauvet, P., Janjuha, S., Ninov, N., & Junker, J.P. (2018).
Simultaneous lineage tracing and cell-type identification using CRISPR-Cas9-induced genetic scars. Nature biotechnology, 36(5), 469-473. [PubMed:29644996] [PMC] [WorldCat] [DOI] - ↑
Rodriques, S.G., Chen, L.M., Liu, S., Zhong, E.D., Scherrer, J.R., Boyden, E.S., & Chen, F. (2020).
RNA timestamps identify the age of single molecules in RNA sequencing. Nature biotechnology. [PubMed:33077959] [WorldCat] [DOI] - ↑
Liu, F., Zhang, Y., Zhang, L., Li, Z., Fang, Q., Gao, R., & Zhang, Z. (2019).
Systematic comparative analysis of single-nucleotide variant detection methods from single-cell RNA sequencing data. Genome biology, 20(1), 242. [PubMed:31744515] [PMC] [WorldCat] [DOI] - ↑
Aibar, S., González-Blas, C.B., Moerman, T., Huynh-Thu, V.A., Imrichova, H., Hulselmans, G., ..., & Aerts, S. (2017).
SCENIC: single-cell regulatory network inference and clustering. Nature methods, 14(11), 1083-1086. [PubMed:28991892] [PMC] [WorldCat] [DOI] - ↑
Zhou, Y., Zhou, B., Pache, L., Chang, M., Khodabakhshi, A.H., Tanaseichuk, O., ..., & Chanda, S.K. (2019).
Metascape provides a biologist-oriented resource for the analysis of systems-level datasets. Nature communications, 10(1), 1523. [PubMed:30944313] [PMC] [WorldCat] [DOI] - ↑
Holland, C.H., Tanevski, J., Perales-Patón, J., Gleixner, J., Kumar, M.P., Mereu, E., ..., & Saez-Rodriguez, J. (2020).
Robustness and applicability of transcription factor and pathway analysis tools on single-cell RNA-seq data. Genome biology, 21(1), 36. [PubMed:32051003] [PMC] [WorldCat] [DOI] - ↑
Efremova, M., Vento-Tormo, M., Teichmann, S.A., & Vento-Tormo, R. (2020).
CellPhoneDB: inferring cell-cell communication from combined expression of multi-subunit ligand-receptor complexes. Nature protocols. [PubMed:32103204] [WorldCat] [DOI] - ↑
Jerome, F. (1988).
Off-the-wall principles of effective management. Hospitals, 62(22), 88. [PubMed:3181926] [WorldCat] - ↑
Arnol, D., Schapiro, D., Bodenmiller, B., Saez-Rodriguez, J., & Stegle, O. (2019).
Modeling Cell-Cell Interactions from Spatial Molecular Data with Spatial Variance Component Analysis. Cell reports, 29(1), 202-211.e6. [PubMed:31577949] [PMC] [WorldCat] [DOI] - ↑
Dixit, A., Parnas, O., Li, B., Chen, J., Fulco, C.P., Jerby-Arnon, L., ..., & Regev, A. (2016).
Perturb-Seq: Dissecting Molecular Circuits with Scalable Single-Cell RNA Profiling of Pooled Genetic Screens. Cell, 167(7), 1853-1866.e17. [PubMed:27984732] [PMC] [WorldCat] [DOI] - ↑
Replogle, J.M., Norman, T.M., Xu, A., Hussmann, J.A., Chen, J., Cogan, J.Z., ..., & Adamson, B. (2020).
Combinatorial single-cell CRISPR screens by direct guide RNA capture and targeted sequencing. Nature biotechnology. [PubMed:32231336] [WorldCat] [DOI] - ↑
Zeng, H., & Sanes, J.R. (2017).
Neuronal cell-type classification: challenges, opportunities and the path forward. Nature reviews. Neuroscience, 18(9), 530-546. [PubMed:28775344] [WorldCat] [DOI] - ↑
Tasic, B. (2018).
Single cell transcriptomics in neuroscience: cell classification and beyond. Current opinion in neurobiology, 50, 242-249. [PubMed:29738987] [WorldCat] [DOI] - ↑
Masland, R.H. (2004).
Neuronal cell types. Current biology : CB, 14(13), R497-500. [PubMed:15242626] [WorldCat] [DOI] - ↑ 124.0 124.1
Yuste, R., Hawrylycz, M., Aalling, N., Aguilar-Valles, A., Arendt, D., Arnedillo, R.A., ..., & Lein, E. (2020).
A community-based transcriptomics classification and nomenclature of neocortical cell types. Nature neuroscience. [PubMed:32839617] [WorldCat] [DOI] - ↑
Zeisel, A., Muñoz-Manchado, A.B., Codeluppi, S., Lönnerberg, P., La Manno, G., Juréus, A., ..., & Linnarsson, S. (2015).
Brain structure. Cell types in the mouse cortex and hippocampus revealed by single-cell RNA-seq. Science (New York, N.Y.), 347(6226), 1138-42. [PubMed:25700174] [WorldCat] [DOI] - ↑
Saunders, A., Macosko, E.Z., Wysoker, A., Goldman, M., Krienen, F.M., de Rivera, H., ..., & McCarroll, S.A. (2018).
Molecular Diversity and Specializations among the Cells of the Adult Mouse Brain. Cell, 174(4), 1015-1030.e16. [PubMed:30096299] [PMC] [WorldCat] [DOI] - ↑
Zeisel, A., Hochgerner, H., Lönnerberg, P., Johnsson, A., Memic, F., van der Zwan, J., ..., & Linnarsson, S. (2018).
Molecular Architecture of the Mouse Nervous System. Cell, 174(4), 999-1014.e22. [PubMed:30096314] [PMC] [WorldCat] [DOI] - ↑
Tasic, B., Yao, Z., Graybuck, L.T., Smith, K.A., Nguyen, T.N., Bertagnolli, D., ..., & Zeng, H. (2018).
Shared and distinct transcriptomic cell types across neocortical areas. Nature, 563(7729), 72-78. [PubMed:30382198] [PMC] [WorldCat] [DOI] - ↑
Chevée, M., Robertson, J.J., Cannon, G.H., Brown, S.P., & Goff, L.A. (2018).
Variation in Activity State, Axonal Projection, and Position Define the Transcriptional Identity of Individual Neocortical Projection Neurons. Cell reports, 22(2), 441-455. [PubMed:29320739] [PMC] [WorldCat] [DOI] - ↑
Lui, J.H., Nguyen, N.D., Grutzner, S.M., Darmanis, S., Peixoto, D., Wagner, M.J., ..., & Luo, L. (2020).
Differential encoding in prefrontal cortex projection neuron classes across cognitive tasks. Cell. [PubMed:33338423] [WorldCat] [DOI] - ↑
Paul, A., Crow, M., Raudales, R., He, M., Gillis, J., & Huang, Z.J. (2017).
Transcriptional Architecture of Synaptic Communication Delineates GABAergic Neuron Identity. Cell, 171(3), 522-539.e20. [PubMed:28942923] [PMC] [WorldCat] [DOI] - ↑
Frazer, S., Prados, J., Niquille, M., Cadilhac, C., Markopoulos, F., Gomez, L., ..., & Dayer, A. (2017).
Transcriptomic and anatomic parcellation of 5-HT3AR expressing cortical interneuron subtypes revealed by single-cell RNA sequencing. Nature communications, 8, 14219. [PubMed:28134272] [PMC] [WorldCat] [DOI] - ↑
Mi, D., Li, Z., Lim, L., Li, M., Moissidis, M., Yang, Y., ..., & Marín, O. (2018).
Early emergence of cortical interneuron diversity in the mouse embryo. Science (New York, N.Y.), 360(6384), 81-85. [PubMed:29472441] [PMC] [WorldCat] [DOI] - ↑
Mayer, C., Hafemeister, C., Bandler, R.C., Machold, R., Batista Brito, R., Jaglin, X., ..., & Satija, R. (2018).
Developmental diversification of cortical inhibitory interneurons. Nature, 555(7697), 457-462. [PubMed:29513653] [PMC] [WorldCat] [DOI] - ↑
Telley, L., Govindan, S., Prados, J., Stevant, I., Nef, S., Dermitzakis, E., ..., & Jabaudon, D. (2016).
Sequential transcriptional waves direct the differentiation of newborn neurons in the mouse neocortex. Science (New York, N.Y.), 351(6280), 1443-6. [PubMed:26940868] [WorldCat] [DOI] - ↑
Zywitza, V., Misios, A., Bunatyan, L., Willnow, T.E., & Rajewsky, N. (2018).
Single-Cell Transcriptomics Characterizes Cell Types in the Subventricular Zone and Uncovers Molecular Defects Impairing Adult Neurogenesis. Cell reports, 25(9), 2457-2469.e8. [PubMed:30485812] [WorldCat] [DOI] - ↑
Telley, L., Agirman, G., Prados, J., Amberg, N., Fièvre, S., Oberst, P., ..., & Jabaudon, D. (2019).
Temporal patterning of apical progenitors and their daughter neurons in the developing neocortex. Science (New York, N.Y.), 364(6440). [PubMed:31073041] [WorldCat] [DOI] - ↑
Loo, L., Simon, J.M., Xing, L., McCoy, E.S., Niehaus, J.K., Guo, J., ..., & Zylka, M.J. (2019).
Single-cell transcriptomic analysis of mouse neocortical development. Nature communications, 10(1), 134. [PubMed:30635555] [PMC] [WorldCat] [DOI] - ↑
Mizrak, D., Levitin, H.M., Delgado, A.C., Crotet, V., Yuan, J., Chaker, Z., ..., & Doetsch, F. (2019).
Single-Cell Analysis of Regional Differences in Adult V-SVZ Neural Stem Cell Lineages. Cell reports, 26(2), 394-406.e5. [PubMed:30625322] [PMC] [WorldCat] [DOI] - ↑
Ximerakis, M., Lipnick, S.L., Innes, B.T., Simmons, S.K., Adiconis, X., Dionne, D., ..., & Rubin, L.L. (2019).
Single-cell transcriptomic profiling of the aging mouse brain. Nature neuroscience, 22(10), 1696-1708. [PubMed:31551601] [WorldCat] [DOI] - ↑
Hrvatin, S., Hochbaum, D.R., Nagy, M.A., Cicconet, M., Robertson, K., Cheadle, L., ..., & Greenberg, M.E. (2018).
Single-cell analysis of experience-dependent transcriptomic states in the mouse visual cortex. Nature neuroscience, 21(1), 120-129. [PubMed:29230054] [PMC] [WorldCat] [DOI] - ↑
Kalish, B.T., Barkat, T.R., Diel, E.E., Zhang, E.J., Greenberg, M.E., & Hensch, T.K. (2020).
Single-nucleus RNA sequencing of mouse auditory cortex reveals critical period triggers and brakes. Proceedings of the National Academy of Sciences of the United States of America, 117(21), 11744-11752. [PubMed:32404418] [PMC] [WorldCat] [DOI] - ↑
Darmanis, S., Sloan, S.A., Zhang, Y., Enge, M., Caneda, C., Shuer, L.M., ..., & Quake, S.R. (2015).
A survey of human brain transcriptome diversity at the single cell level. Proceedings of the National Academy of Sciences of the United States of America, 112(23), 7285-90. [PubMed:26060301] [PMC] [WorldCat] [DOI] - ↑
Lake, B.B., Ai, R., Kaeser, G.E., Salathia, N.S., Yung, Y.C., Liu, R., ..., & Zhang, K. (2016).
Neuronal subtypes and diversity revealed by single-nucleus RNA sequencing of the human brain. Science (New York, N.Y.), 352(6293), 1586-90. [PubMed:27339989] [PMC] [WorldCat] [DOI] - ↑ 145.0 145.1
Zhong, S., Zhang, S., Fan, X., Wu, Q., Yan, L., Dong, J., ..., & Wang, X. (2018).
A single-cell RNA-seq survey of the developmental landscape of the human prefrontal cortex. Nature, 555(7697), 524-528. [PubMed:29539641] [WorldCat] [DOI] - ↑ 146.0 146.1
Nowakowski, T.J., Bhaduri, A., Pollen, A.A., Alvarado, B., Mostajo-Radji, M.A., Di Lullo, E., ..., & Kriegstein, A.R. (2017).
Spatiotemporal gene expression trajectories reveal developmental hierarchies of the human cortex. Science (New York, N.Y.), 358(6368), 1318-1323. [PubMed:29217575] [PMC] [WorldCat] [DOI] - ↑
Lake, B.B., Chen, S., Sos, B.C., Fan, J., Kaeser, G.E., Yung, Y.C., ..., & Zhang, K. (2018).
Integrative single-cell analysis of transcriptional and epigenetic states in the human adult brain. Nature biotechnology, 36(1), 70-80. [PubMed:29227469] [WorldCat] [DOI] - ↑
Polioudakis, D., de la Torre-Ubieta, L., Langerman, J., Elkins, A.G., Shi, X., Stein, J.L., ..., & Geschwind, D.H. (2019).
A Single-Cell Transcriptomic Atlas of Human Neocortical Development during Mid-gestation. Neuron, 103(5), 785-801.e8. [PubMed:31303374] [PMC] [WorldCat] [DOI] - ↑
Fan, X., Dong, J., Zhong, S., Wei, Y., Wu, Q., Yan, L., ..., & Tang, F. (2018).
Spatial transcriptomic survey of human embryonic cerebral cortex by single-cell RNA-seq analysis. Cell research, 28(7), 730-745. [PubMed:29867213] [PMC] [WorldCat] [DOI] - ↑
Hodge, R.D., Bakken, T.E., Miller, J.A., Smith, K.A., Barkan, E.R., Graybuck, L.T., ..., & Lein, E.S. (2019).
Conserved cell types with divergent features in human versus mouse cortex. Nature, 573(7772), 61-68. [PubMed:31435019] [PMC] [WorldCat] [DOI] - ↑
Khrameeva, E., Kurochkin, I., Han, D., Guijarro, P., Kanton, S., Santel, M., ..., & Khaitovich, P. (2020).
Single-cell-resolution transcriptome map of human, chimpanzee, bonobo, and macaque brains. Genome research. [PubMed:32424074] [WorldCat] [DOI] - ↑
Krienen, F.M., Goldman, M., Zhang, Q., C H Del Rosario, R., Florio, M., Machold, R., ..., & McCarroll, S.A. (2020).
Innovations present in the primate interneuron repertoire. Nature, 586(7828), 262-269. [PubMed:32999462] [WorldCat] [DOI] - ↑
Hodge, R.D., Miller, J.A., Novotny, M., Kalmbach, B.E., Ting, J.T., Bakken, T.E., ..., & Lein, E.S. (2020).
Transcriptomic evidence that von Economo neurons are regionally specialized extratelencephalic-projecting excitatory neurons. Nature communications, 11(1), 1172. [PubMed:32127543] [PMC] [WorldCat] [DOI] - ↑
Artegiani, B., Lyubimova, A., Muraro, M., van Es, J.H., van Oudenaarden, A., & Clevers, H. (2017).
A Single-Cell RNA Sequencing Study Reveals Cellular and Molecular Dynamics of the Hippocampal Neurogenic Niche. Cell reports, 21(11), 3271-3284. [PubMed:29241552] [WorldCat] [DOI] - ↑
Harris, K.D., Hochgerner, H., Skene, N.G., Magno, L., Katona, L., Bengtsson Gonzales, C., ..., & Hjerling-Leffler, J. (2018).
Classes and continua of hippocampal CA1 inhibitory neurons revealed by single-cell transcriptomics. PLoS biology, 16(6), e2006387. [PubMed:29912866] [PMC] [WorldCat] [DOI] - ↑
Hochgerner, H., Zeisel, A., Lönnerberg, P., & Linnarsson, S. (2018).
Conserved properties of dentate gyrus neurogenesis across postnatal development revealed by single-cell RNA sequencing. Nature neuroscience, 21(2), 290-299. [PubMed:29335606] [WorldCat] [DOI] - ↑
Zhong, S., Ding, W., Sun, L., Lu, Y., Dong, H., Fan, X., ..., & Wang, X. (2020).
Decoding the development of the human hippocampus. Nature, 577(7791), 531-536. [PubMed:31942070] [WorldCat] [DOI] - ↑
Kalish, B.T., Cheadle, L., Hrvatin, S., Nagy, M.A., Rivera, S., Crow, M., ..., & Greenberg, M.E. (2018).
Single-cell transcriptomics of the developing lateral geniculate nucleus reveals insights into circuit assembly and refinement. Proceedings of the National Academy of Sciences of the United States of America, 115(5), E1051-E1060. [PubMed:29343640] [PMC] [WorldCat] [DOI] - ↑
Wallace, M.L., Saunders, A., Huang, K.W., Philson, A.C., Goldman, M., Macosko, E.Z., ..., & Sabatini, B.L. (2017).
Genetically Distinct Parallel Pathways in the Entopeduncular Nucleus for Limbic and Sensorimotor Output of the Basal Ganglia. Neuron, 94(1), 138-152.e5. [PubMed:28384468] [PMC] [WorldCat] [DOI] - ↑
Campbell, J.N., Macosko, E.Z., Fenselau, H., Pers, T.H., Lyubetskaya, A., Tenen, D., ..., & Tsai, L.T. (2017).
A molecular census of arcuate hypothalamus and median eminence cell types. Nature neuroscience, 20(3), 484-496. [PubMed:28166221] [PMC] [WorldCat] [DOI] - ↑
Chen, R., Wu, X., Jiang, L., & Zhang, Y. (2017).
Single-Cell RNA-Seq Reveals Hypothalamic Cell Diversity. Cell reports, 18(13), 3227-3241. [PubMed:28355573] [PMC] [WorldCat] [DOI] - ↑
Romanov, R.A., Zeisel, A., Bakker, J., Girach, F., Hellysaz, A., Tomer, R., ..., & Harkany, T. (2017).
Molecular interrogation of hypothalamic organization reveals distinct dopamine neuronal subtypes. Nature neuroscience, 20(2), 176-188. [PubMed:27991900] [WorldCat] [DOI] - ↑ 163.0 163.1
Moffitt, J.R., Bambah-Mukku, D., Eichhorn, S.W., Vaughn, E., Shekhar, K., Perez, J.D., ..., & Zhuang, X. (2018).
Molecular, spatial, and functional single-cell profiling of the hypothalamic preoptic region. Science (New York, N.Y.), 362(6416). [PubMed:30385464] [PMC] [WorldCat] [DOI] - ↑
Rossi, M.A., Basiri, M.L., McHenry, J.A., Kosyk, O., Otis, J.M., van den Munkhof, H.E., ..., & Stuber, G.D. (2019).
Obesity remodels activity and transcriptional state of a lateral hypothalamic brake on feeding. Science (New York, N.Y.), 364(6447), 1271-1274. [PubMed:31249056] [WorldCat] [DOI] - ↑
Mickelsen, L.E., Bolisetty, M., Chimileski, B.R., Fujita, A., Beltrami, E.J., Costanzo, J.T., ..., & Jackson, A.C. (2019).
Single-cell transcriptomic analysis of the lateral hypothalamic area reveals molecularly distinct populations of inhibitory and excitatory neurons. Nature neuroscience, 22(4), 642-656. [PubMed:30858605] [PMC] [WorldCat] [DOI] - ↑
Gokce, O., Stanley, G.M., Treutlein, B., Neff, N.F., Camp, J.G., Malenka, R.C., ..., & Quake, S.R. (2016).
Cellular Taxonomy of the Mouse Striatum as Revealed by Single-Cell RNA-Seq. Cell reports, 16(4), 1126-1137. [PubMed:27425622] [PMC] [WorldCat] [DOI] - ↑
Muñoz-Manchado, A.B., Bengtsson Gonzales, C., Zeisel, A., Munguba, H., Bekkouche, B., Skene, N.G., ..., & Hjerling-Leffler, J. (2018).
Diversity of Interneurons in the Dorsal Striatum Revealed by Single-Cell RNA Sequencing and PatchSeq. Cell reports, 24(8), 2179-2190.e7. [PubMed:30134177] [PMC] [WorldCat] [DOI] - ↑
Märtin, A., Calvigioni, D., Tzortzi, O., Fuzik, J., Wärnberg, E., & Meletis, K. (2019).
A Spatiomolecular Map of the Striatum. Cell reports, 29(13), 4320-4333.e5. [PubMed:31875543] [WorldCat] [DOI] - ↑
La Manno, G., Gyllborg, D., Codeluppi, S., Nishimura, K., Salto, C., Zeisel, A., ..., & Linnarsson, S. (2016).
Molecular Diversity of Midbrain Development in Mouse, Human, and Stem Cells. Cell, 167(2), 566-580.e19. [PubMed:27716510] [PMC] [WorldCat] [DOI] - ↑
Hook, P.W., McClymont, S.A., Cannon, G.H., Law, W.D., Morton, A.J., Goff, L.A., & McCallion, A.S. (2018).
Single-Cell RNA-Seq of Mouse Dopaminergic Neurons Informs Candidate Gene Selection for Sporadic Parkinson Disease. American journal of human genetics, 102(3), 427-446. [PubMed:29499164] [PMC] [WorldCat] [DOI] - ↑
Tiklová, K., Björklund, Å.K., Lahti, L., Fiorenzano, A., Nolbrant, S., Gillberg, L., ..., & Perlmann, T. (2019).
Single-cell RNA sequencing reveals midbrain dopamine neuron diversity emerging during mouse brain development. Nature communications, 10(1), 581. [PubMed:30718509] [PMC] [WorldCat] [DOI] - ↑
Pandey, S., Shekhar, K., Regev, A., & Schier, A.F. (2018).
Comprehensive Identification and Spatial Mapping of Habenular Neuronal Types Using Single-Cell RNA-Seq. Current biology : CB, 28(7), 1052-1065.e7. [PubMed:29576475] [PMC] [WorldCat] [DOI] - ↑
Guo, Q., & Li, J.Y.H. (2019).
Defining developmental diversification of diencephalon neurons through single cell gene expression profiling. Development (Cambridge, England), 146(12). [PubMed:30872278] [PMC] [WorldCat] [DOI] - ↑
Carter, R.A., Bihannic, L., Rosencrance, C., Hadley, J.L., Tong, Y., Phoenix, T.N., ..., & Gawad, C. (2018).
A Single-Cell Transcriptional Atlas of the Developing Murine Cerebellum. Current biology : CB, 28(18), 2910-2920.e2. [PubMed:30220501] [WorldCat] [DOI] - ↑
Wizeman, J.W., Guo, Q., Wilion, E.M., & Li, J.Y. (2019).
Specification of diverse cell types during early neurogenesis of the mouse cerebellum. eLife, 8. [PubMed:30735127] [PMC] [WorldCat] [DOI] - ↑
Peng, J., Sheng, A.L., Xiao, Q., Shen, L., Ju, X.C., Zhang, M., ..., & Luo, Z.G. (2019).
Single-cell transcriptomes reveal molecular specializations of neuronal cell types in the developing cerebellum. Journal of molecular cell biology, 11(8), 636-648. [PubMed:30690467] [PMC] [WorldCat] [DOI] - ↑
Dagley, L.F., White, C.A., Liao, Y., Shi, W., Smyth, G.K., Orian, J.M., ..., & Purcell, A.W. (2014).
Quantitative proteomic profiling reveals novel region-specific markers in the adult mouse brain. Proteomics, 14(2-3), 241-61. [PubMed:24259518] [WorldCat] [DOI] - ↑
Usoskin, D., Furlan, A., Islam, S., Abdo, H., Lönnerberg, P., Lou, D., ..., & Ernfors, P. (2015).
Unbiased classification of sensory neuron types by large-scale single-cell RNA sequencing. Nature neuroscience, 18(1), 145-53. [PubMed:25420068] [WorldCat] [DOI] - ↑
Li, C.L., Li, K.C., Wu, D., Chen, Y., Luo, H., Zhao, J.R., ..., & Zhang, X. (2016).
Somatosensory neuron types identified by high-coverage single-cell RNA-sequencing and functional heterogeneity. Cell research, 26(1), 83-102. [PubMed:26691752] [PMC] [WorldCat] [DOI] - ↑
Shrestha, B.R., Chia, C., Wu, L., Kujawa, S.G., Liberman, M.C., & Goodrich, L.V. (2018).
Sensory Neuron Diversity in the Inner Ear Is Shaped by Activity. Cell, 174(5), 1229-1246.e17. [PubMed:30078709] [PMC] [WorldCat] [DOI] - ↑
Petitpré, C., Wu, H., Sharma, A., Tokarska, A., Fontanet, P., Wang, Y., ..., & Lallemend, F. (2018).
Neuronal heterogeneity and stereotyped connectivity in the auditory afferent system. Nature communications, 9(1), 3691. [PubMed:30209249] [PMC] [WorldCat] [DOI] - ↑
Hanchate, N.K., Kondoh, K., Lu, Z., Kuang, D., Ye, X., Qiu, X., ..., & Buck, L.B. (2015).
Single-cell transcriptomics reveals receptor transformations during olfactory neurogenesis. Science (New York, N.Y.), 350(6265), 1251-5. [PubMed:26541607] [PMC] [WorldCat] [DOI] - ↑
Li, H., Li, T., Horns, F., Li, J., Xie, Q., Xu, C., ..., & Luo, L. (2020).
Single-Cell Transcriptomes Reveal Diverse Regulatory Strategies for Olfactory Receptor Expression and Axon Targeting. Current biology : CB, 30(7), 1189-1198.e5. [PubMed:32059767] [WorldCat] [DOI] - ↑
Hockley, J.R.F., Taylor, T.S., Callejo, G., Wilbrey, A.L., Gutteridge, A., Bach, K., ..., & Smith, E.S.J. (2019).
Single-cell RNAseq reveals seven classes of colonic sensory neuron. Gut, 68(4), 633-644. [PubMed:29483303] [PMC] [WorldCat] [DOI] - ↑
Morarach, K., Mikhailova, A., Knoflach, V., Memic, F., Kumar, R., Li, W., ..., & Marklund, U. (2020).
Diversification of molecularly defined myenteric neuron classes revealed by single-cell RNA sequencing. Nature neuroscience. [PubMed:33288908] [WorldCat] [DOI] - ↑
Shekhar, K., Lapan, S.W., Whitney, I.E., Tran, N.M., Macosko, E.Z., Kowalczyk, M., ..., & Sanes, J.R. (2016).
Comprehensive Classification of Retinal Bipolar Neurons by Single-Cell Transcriptomics. Cell, 166(5), 1308-1323.e30. [PubMed:27565351] [PMC] [WorldCat] [DOI] - ↑ 187.0 187.1
Konstantinides, N., Kapuralin, K., Fadil, C., Barboza, L., Satija, R., & Desplan, C. (2018).
Phenotypic Convergence: Distinct Transcription Factors Regulate Common Terminal Features. Cell, 174(3), 622-635.e13. [PubMed:29909983] [PMC] [WorldCat] [DOI] - ↑
Rheaume, B.A., Jereen, A., Bolisetty, M., Sajid, M.S., Yang, Y., Renna, K., ..., & Trakhtenberg, E.F. (2018).
Single cell transcriptome profiling of retinal ganglion cells identifies cellular subtypes. Nature communications, 9(1), 2759. [PubMed:30018341] [PMC] [WorldCat] [DOI] - ↑
Buenaventura, D.F., Corseri, A., & Emerson, M.M. (2019).
Identification of Genes With Enriched Expression in Early Developing Mouse Cone Photoreceptors. Investigative ophthalmology & visual science, 60(8), 2787-2799. [PubMed:31260032] [PMC] [WorldCat] [DOI] - ↑
Clark, B.S., Stein-O'Brien, G.L., Shiau, F., Cannon, G.H., Davis-Marcisak, E., Sherman, T., ..., & Blackshaw, S. (2019).
Single-Cell RNA-Seq Analysis of Retinal Development Identifies NFI Factors as Regulating Mitotic Exit and Late-Born Cell Specification. Neuron, 102(6), 1111-1126.e5. [PubMed:31128945] [PMC] [WorldCat] [DOI] - ↑ 191.0 191.1
Peng, Y.R., Shekhar, K., Yan, W., Herrmann, D., Sappington, A., Bryman, G.S., ..., & Sanes, J.R. (2019).
Molecular Classification and Comparative Taxonomics of Foveal and Peripheral Cells in Primate Retina. Cell, 176(5), 1222-1237.e22. [PubMed:30712875] [PMC] [WorldCat] [DOI] - ↑
Collin, J., Queen, R., Zerti, D., Dorgau, B., Hussain, R., Coxhead, J., ..., & Lako, M. (2019).
Deconstructing Retinal Organoids: Single Cell RNA-Seq Reveals the Cellular Components of Human Pluripotent Stem Cell-Derived Retina. Stem cells (Dayton, Ohio), 37(5), 593-598. [PubMed:30548510] [PMC] [WorldCat] [DOI] - ↑
Voigt, A.P., Whitmore, S.S., Flamme-Wiese, M.J., Riker, M.J., Wiley, L.A., Tucker, B.A., ..., & Scheetz, T.E. (2019).
Molecular characterization of foveal versus peripheral human retina by single-cell RNA sequencing. Experimental eye research, 184, 234-242. [PubMed:31075224] [PMC] [WorldCat] [DOI] - ↑
Lo Giudice, Q., Leleu, M., La Manno, G., & Fabre, P.J. (2019).
Single-cell transcriptional logic of cell-fate specification and axon guidance in early-born retinal neurons. Development (Cambridge, England), 146(17). [PubMed:31399471] [WorldCat] [DOI] - ↑
Liang, Q., Dharmat, R., Owen, L., Shakoor, A., Li, Y., Kim, S., ..., & Chen, R. (2019).
Single-nuclei RNA-seq on human retinal tissue provides improved transcriptome profiling. Nature communications, 10(1), 5743. [PubMed:31848347] [PMC] [WorldCat] [DOI] - ↑
Laboissonniere, L.A., Goetz, J.J., Martin, G.M., Bi, R., Lund, T.J.S., Ellson, L., ..., & Trimarchi, J.M. (2019).
Molecular signatures of retinal ganglion cells revealed through single cell profiling. Scientific reports, 9(1), 15778. [PubMed:31673015] [PMC] [WorldCat] [DOI] - ↑
Menon, M., Mohammadi, S., Davila-Velderrain, J., Goods, B.A., Cadwell, T.D., Xing, Y., ..., & Hafler, B.P. (2019).
Single-cell transcriptomic atlas of the human retina identifies cell types associated with age-related macular degeneration. Nature communications, 10(1), 4902. [PubMed:31653841] [PMC] [WorldCat] [DOI] - ↑
Tran, N.M., Shekhar, K., Whitney, I.E., Jacobi, A., Benhar, I., Hong, G., ..., & Sanes, J.R. (2019).
Single-Cell Profiles of Retinal Ganglion Cells Differing in Resilience to Injury Reveal Neuroprotective Genes. Neuron, 104(6), 1039-1055.e12. [PubMed:31784286] [PMC] [WorldCat] [DOI] - ↑
Lu, Y., Shiau, F., Yi, W., Lu, S., Wu, Q., Pearson, J.D., ..., & Clark, B.S. (2020).
Single-Cell Analysis of Human Retina Identifies Evolutionarily Conserved and Species-Specific Mechanisms Controlling Development. Developmental cell, 53(4), 473-491.e9. [PubMed:32386599] [WorldCat] [DOI] - ↑ Shekhar K, Sanes JR (2021).
Generating and using transcriptomically based retinal cell atlases. Annu Rev Vis Sci 7: (in press) - ↑
Yao, Z., Mich, J.K., Ku, S., Menon, V., Krostag, A.R., Martinez, R.A., ..., & Ramanathan, S. (2017).
A Single-Cell Roadmap of Lineage Bifurcation in Human ESC Models of Embryonic Brain Development. Cell stem cell, 20(1), 120-134. [PubMed:28094016] [PMC] [WorldCat] [DOI] - ↑
Close, J.L., Yao, Z., Levi, B.P., Miller, J.A., Bakken, T.E., Menon, V., ..., & Lein, E. (2017).
Single-Cell Profiling of an In Vitro Model of Human Interneuron Development Reveals Temporal Dynamics of Cell Type Production and Maturation. Neuron, 93(5), 1035-1048.e5. [PubMed:28279351] [PMC] [WorldCat] [DOI] - ↑
Velasco, S., Kedaigle, A.J., Simmons, S.K., Nash, A., Rocha, M., Quadrato, G., ..., & Arlotta, P. (2019).
Individual brain organoids reproducibly form cell diversity of the human cerebral cortex. Nature, 570(7762), 523-527. [PubMed:31168097] [PMC] [WorldCat] [DOI] - ↑
Bhaduri, A., Andrews, M.G., Mancia Leon, W., Jung, D., Shin, D., Allen, D., ..., & Kriegstein, A.R. (2020).
Cell stress in cortical organoids impairs molecular subtype specification. Nature, 578(7793), 142-148. [PubMed:31996853] [WorldCat] [DOI] - ↑
Nickolls, A.R., Lee, M.M., Espinoza, D.F., Szczot, M., Lam, R.M., Wang, Q., ..., & Bönnemann, C.G. (2020).
Transcriptional Programming of Human Mechanosensory Neuron Subtypes from Pluripotent Stem Cells. Cell reports, 30(3), 932-946.e7. [PubMed:31968264] [PMC] [WorldCat] [DOI] - ↑
Xu, R., Li, X., Boreland, A.J., Posyton, A., Kwan, K., Hart, R.P., & Jiang, P. (2020).
Human iPSC-derived mature microglia retain their identity and functionally integrate in the chimeric mouse brain. Nature communications, 11(1), 1577. [PubMed:32221280] [PMC] [WorldCat] [DOI] - ↑
Dannemann, M., He, Z., Heide, C., Vernot, B., Sidow, L., Kanton, S., ..., & Camp, J.G. (2020).
Human Stem Cell Resources Are an Inroad to Neandertal DNA Functions. Stem cell reports, 15(1), 214-225. [PubMed:32559457] [PMC] [WorldCat] [DOI] - ↑
Pellegrini, L., Albecka, A., Mallery, D.L., Kellner, M.J., Paul, D., Carter, A.P., ..., & Lancaster, M.A. (2020).
SARS-CoV-2 Infects the Brain Choroid Plexus and Disrupts the Blood-CSF Barrier in Human Brain Organoids. Cell stem cell, 27(6), 951-961.e5. [PubMed:33113348] [PMC] [WorldCat] [DOI] - ↑
Shah, P.T., Stratton, J.A., Stykel, M.G., Abbasi, S., Sharma, S., Mayr, K.A., ..., & Biernaskie, J. (2018).
Single-Cell Transcriptomics and Fate Mapping of Ependymal Cells Reveals an Absence of Neural Stem Cell Function. Cell, 173(4), 1045-1057.e9. [PubMed:29727663] [WorldCat] [DOI] - ↑
Pollen, A.A., Nowakowski, T.J., Chen, J., Retallack, H., Sandoval-Espinosa, C., Nicholas, C.R., ..., & Kriegstein, A.R. (2015).
Molecular identity of human outer radial glia during cortical development. Cell, 163(1), 55-67. [PubMed:26406371] [PMC] [WorldCat] [DOI] - ↑
Johnson, M.B., Wang, P.P., Atabay, K.D., Murphy, E.A., Doan, R.N., Hecht, J.L., & Walsh, C.A. (2015).
Single-cell analysis reveals transcriptional heterogeneity of neural progenitors in human cortex. Nature neuroscience, 18(5), 637-46. [PubMed:25734491] [PMC] [WorldCat] [DOI] - ↑
Yuzwa, S.A., Borrett, M.J., Innes, B.T., Voronova, A., Ketela, T., Kaplan, D.R., ..., & Miller, F.D. (2017).
Developmental Emergence of Adult Neural Stem Cells as Revealed by Single-Cell Transcriptional Profiling. Cell reports, 21(13), 3970-3986. [PubMed:29281841] [WorldCat] [DOI] - ↑
Batiuk, M.Y., Martirosyan, A., Wahis, J., de Vin, F., Marneffe, C., Kusserow, C., ..., & Holt, M.G. (2020).
Identification of region-specific astrocyte subtypes at single cell resolution. Nature communications, 11(1), 1220. [PubMed:32139688] [PMC] [WorldCat] [DOI] - ↑
Bayraktar, O.A., Bartels, T., Holmqvist, S., Kleshchevnikov, V., Martirosyan, A., Polioudakis, D., ..., & Rowitch, D.H. (2020).
Astrocyte layers in the mammalian cerebral cortex revealed by a single-cell in situ transcriptomic map. Nature neuroscience. [PubMed:32203496] [WorldCat] [DOI] - ↑
Marques, S., Zeisel, A., Codeluppi, S., van Bruggen, D., Mendanha Falcão, A., Xiao, L., ..., & Castelo-Branco, G. (2016).
Oligodendrocyte heterogeneity in the mouse juvenile and adult central nervous system. Science (New York, N.Y.), 352(6291), 1326-1329. [PubMed:27284195] [PMC] [WorldCat] [DOI] - ↑
Marques, S., van Bruggen, D., Vanichkina, D.P., Floriddia, E.M., Munguba, H., Väremo, L., ..., & Castelo-Branco, G. (2018).
Transcriptional Convergence of Oligodendrocyte Lineage Progenitors during Development. Developmental cell, 46(4), 504-517.e7. [PubMed:30078729] [PMC] [WorldCat] [DOI] - ↑
Avey, D., Sankararaman, S., Yim, A.K.Y., Barve, R., Milbrandt, J., & Mitra, R.D. (2018).
Single-Cell RNA-Seq Uncovers a Robust Transcriptional Response to Morphine by Glia. Cell reports, 24(13), 3619-3629.e4. [PubMed:30257220] [PMC] [WorldCat] [DOI] - ↑ 218.0 218.1
Jäkel, S., Agirre, E., Mendanha Falcão, A., van Bruggen, D., Lee, K.W., Knuesel, I., ..., & Castelo-Branco, G. (2019).
Altered human oligodendrocyte heterogeneity in multiple sclerosis. Nature, 566(7745), 543-547. [PubMed:30747918] [PMC] [WorldCat] [DOI] - ↑
Perlman, K., Couturier, C.P., Yaqubi, M., Tanti, A., Cui, Q.L., Pernin, F., ..., & Antel, J.P. (2020).
Developmental trajectory of oligodendrocyte progenitor cells in the human brain revealed by single cell RNA sequencing. Glia. [PubMed:31958186] [WorldCat] [DOI] - ↑
Matcovitch-Natan, O., Winter, D.R., Giladi, A., Vargas Aguilar, S., Spinrad, A., Sarrazin, S., ..., & Amit, I. (2016).
Microglia development follows a stepwise program to regulate brain homeostasis. Science (New York, N.Y.), 353(6301), aad8670. [PubMed:27338705] [WorldCat] [DOI] - ↑ 221.0 221.1
Mathys, H., Adaikkan, C., Gao, F., Young, J.Z., Manet, E., Hemberg, M., ..., & Tsai, L.H. (2017).
Temporal Tracking of Microglia Activation in Neurodegeneration at Single-Cell Resolution. Cell reports, 21(2), 366-380. [PubMed:29020624] [PMC] [WorldCat] [DOI] - ↑
Sousa, C., Golebiewska, A., Poovathingal, S.K., Kaoma, T., Pires-Afonso, Y., Martina, S., ..., & Michelucci, A. (2018).
Single-cell transcriptomics reveals distinct inflammation-induced microglia signatures. EMBO reports, 19(11). [PubMed:30206190] [PMC] [WorldCat] [DOI] - ↑
Gunner, G., Cheadle, L., Johnson, K.M., Ayata, P., Badimon, A., Mondo, E., ..., & Schafer, D.P. (2019).
Sensory lesioning induces microglial synapse elimination via ADAM10 and fractalkine signaling. Nature neuroscience, 22(7), 1075-1088. [PubMed:31209379] [PMC] [WorldCat] [DOI] - ↑
Geirsdottir, L., David, E., Keren-Shaul, H., Weiner, A., Bohlen, S.C., Neuber, J., ..., & Prinz, M. (2019).
Cross-Species Single-Cell Analysis Reveals Divergence of the Primate Microglia Program. Cell, 179(7), 1609-1622.e16. [PubMed:31835035] [WorldCat] [DOI] - ↑
Van Hove, H., Martens, L., Scheyltjens, I., De Vlaminck, K., Pombo Antunes, A.R., De Prijck, S., ..., & Movahedi, K. (2019).
A single-cell atlas of mouse brain macrophages reveals unique transcriptional identities shaped by ontogeny and tissue environment. Nature neuroscience, 22(6), 1021-1035. [PubMed:31061494] [WorldCat] [DOI] - ↑
Vanlandewijck, M., He, L., Mäe, M.A., Andrae, J., Ando, K., Del Gaudio, F., ..., & Betsholtz, C. (2018).
A molecular atlas of cell types and zonation in the brain vasculature. Nature, 554(7693), 475-480. [PubMed:29443965] [WorldCat] [DOI] - ↑ 227.0 227.1
Maniatis, S., Äijö, T., Vickovic, S., Braine, C., Kang, K., Mollbrink, A., ..., & Phatnani, H. (2019).
Spatiotemporal dynamics of molecular pathology in amyotrophic lateral sclerosis. Science (New York, N.Y.), 364(6435), 89-93. [PubMed:30948552] [WorldCat] [DOI] - ↑
Falcão, A.M., van Bruggen, D., Marques, S., Meijer, M., Jäkel, S., Agirre, E., ..., & Castelo-Branco, G. (2018).
Disease-specific oligodendrocyte lineage cells arise in multiple sclerosis. Nature medicine, 24(12), 1837-1844. [PubMed:30420755] [PMC] [WorldCat] [DOI] - ↑
Giladi, A., Wagner, L.K., Li, H., Dörr, D., Medaglia, C., Paul, F., ..., & Mildner, A. (2020).
Cxcl10+ monocytes define a pathogenic subset in the central nervous system during autoimmune neuroinflammation. Nature immunology, 21(5), 525-534. [PubMed:32313246] [WorldCat] [DOI] - ↑
Mathys, H., Davila-Velderrain, J., Peng, Z., Gao, F., Mohammadi, S., Young, J.Z., ..., & Tsai, L.H. (2019).
Single-cell transcriptomic analysis of Alzheimer's disease. Nature, 570(7761), 332-337. [PubMed:31042697] [PMC] [WorldCat] [DOI] - ↑
Del-Aguila, J.L., Li, Z., Dube, U., Mihindukulasuriya, K.A., Budde, J.P., Fernandez, M.V., ..., & Harari, O. (2019).
A single-nuclei RNA sequencing study of Mendelian and sporadic AD in the human brain. Alzheimer's research & therapy, 11(1), 71. [PubMed:31399126] [PMC] [WorldCat] [DOI] - ↑
Keren-Shaul, H., Spinrad, A., Weiner, A., Matcovitch-Natan, O., Dvir-Szternfeld, R., Ulland, T.K., ..., & Amit, I. (2017).
A Unique Microglia Type Associated with Restricting Development of Alzheimer's Disease. Cell, 169(7), 1276-1290.e17. [PubMed:28602351] [WorldCat] [DOI] - ↑
Habib, N., McCabe, C., Medina, S., Varshavsky, M., Kitsberg, D., Dvir-Szternfeld, R., ..., & Schwartz, M. (2020).
Disease-associated astrocytes in Alzheimer's disease and aging. Nature neuroscience. [PubMed:32341542] [WorldCat] [DOI] - ↑
Skene, N.G., Bryois, J., Bakken, T.E., Breen, G., Crowley, J.J., Gaspar, H.A., ..., & Hjerling-Leffler, J. (2018).
Genetic identification of brain cell types underlying schizophrenia. Nature genetics, 50(6), 825-833. [PubMed:29785013] [PMC] [WorldCat] [DOI] - ↑
Jaffe, A.E., Hoeppner, D.J., Saito, T., Blanpain, L., Ukaigwe, J., Burke, E.E., ..., & Hyde, T.M. (2020).
Profiling gene expression in the human dentate gyrus granule cell layer reveals insights into schizophrenia and its genetic risk. Nature neuroscience, 23(4), 510-519. [PubMed:32203495] [WorldCat] [DOI] - ↑
Pfisterer, U., Petukhov, V., Demharter, S., Meichsner, J., Thompson, J.J., Batiuk, M.Y., ..., & Khodosevich, K. (2020).
Identification of epilepsy-associated neuronal subtypes and gene expression underlying epileptogenesis. Nature communications, 11(1), 5038. [PubMed:33028830] [PMC] [WorldCat] [DOI] - ↑
Velmeshev, D., Schirmer, L., Jung, D., Haeussler, M., Perez, Y., Mayer, S., ..., & Kriegstein, A.R. (2019).
Single-cell genomics identifies cell type-specific molecular changes in autism. Science (New York, N.Y.), 364(6441), 685-689. [PubMed:31097668] [WorldCat] [DOI] - ↑
Renthal, W., Boxer, L.D., Hrvatin, S., Li, E., Silberfeld, A., Nagy, M.A., ..., & Greenberg, M.E. (2018).
Characterization of human mosaic Rett syndrome brain tissue by single-nucleus RNA sequencing. Nature neuroscience, 21(12), 1670-1679. [PubMed:30455458] [PMC] [WorldCat] [DOI] - ↑
Sainio, M.T., Ylikallio, E., Mäenpää, L., Lahtela, J., Mattila, P., Auranen, M., ..., & Tyynismaa, H. (2018).
Absence of NEFL in patient-specific neurons in early-onset Charcot-Marie-Tooth neuropathy. Neurology. Genetics, 4(3), e244. [PubMed:29888333] [PMC] [WorldCat] [DOI] - ↑
Lang, C., Campbell, K.R., Ryan, B.J., Carling, P., Attar, M., Vowles, J., ..., & Wade-Martins, R. (2019).
Single-Cell Sequencing of iPSC-Dopamine Neurons Reconstructs Disease Progression and Identifies HDAC4 as a Regulator of Parkinson Cell Phenotypes. Cell stem cell, 24(1), 93-106.e6. [PubMed:30503143] [PMC] [WorldCat] [DOI] - ↑
Agarwal, D., Sandor, C., Volpato, V., Caffrey, T.M., Monzón-Sandoval, J., Bowden, R., ..., & Webber, C. (2020).
A single-cell atlas of the human substantia nigra reveals cell-specific pathways associated with neurological disorders. Nature communications, 11(1), 4183. [PubMed:32826893] [PMC] [WorldCat] [DOI] - ↑
Al-Dalahmah, O., Sosunov, A.A., Shaik, A., Ofori, K., Liu, Y., Vonsattel, J.P., ..., & Goldman, J.E. (2020).
Single-nucleus RNA-seq identifies Huntington disease astrocyte states. Acta neuropathologica communications, 8(1), 19. [PubMed:32070434] [PMC] [WorldCat] [DOI] - ↑
Neftel, C., Laffy, J., Filbin, M.G., Hara, T., Shore, M.E., Rahme, G.J., ..., & Suvà, M.L. (2019).
An Integrative Model of Cellular States, Plasticity, and Genetics for Glioblastoma. Cell, 178(4), 835-849.e21. [PubMed:31327527] [PMC] [WorldCat] [DOI] - ↑
Venteicher, A.S., Tirosh, I., Hebert, C., Yizhak, K., Neftel, C., Filbin, M.G., ..., & Suvà, M.L. (2017).
Decoupling genetics, lineages, and microenvironment in IDH-mutant gliomas by single-cell RNA-seq. Science (New York, N.Y.), 355(6332). [PubMed:28360267] [PMC] [WorldCat] [DOI] - ↑
Jin, X., Simmons, S.K., Guo, A., Shetty, A.S., Ko, M., Nguyen, L., ..., & Arlotta, P. (2020).
In vivo Perturb-Seq reveals neuronal and glial abnormalities associated with autism risk genes. Science (New York, N.Y.), 370(6520). [PubMed:33243861] [WorldCat] [DOI] - ↑
Davie, K., Janssens, J., Koldere, D., De Waegeneer, M., Pech, U., Kreft, Ł., ..., & Aerts, S. (2018).
A Single-Cell Transcriptome Atlas of the Aging Drosophila Brain. Cell, 174(4), 982-998.e20. [PubMed:29909982] [PMC] [WorldCat] [DOI] - ↑
Li, H., Horns, F., Wu, B., Xie, Q., Li, J., Li, T., ..., & Luo, L. (2017).
Classifying Drosophila Olfactory Projection Neuron Subtypes by Single-Cell RNA Sequencing. Cell, 171(5), 1206-1220.e22. [PubMed:29149607] [PMC] [WorldCat] [DOI] - ↑
Bates, A.S., Janssens, J., Jefferis, G.S., & Aerts, S. (2019).
Neuronal cell types in the fly: single-cell anatomy meets single-cell genomics. Current opinion in neurobiology, 56, 125-134. [PubMed:30703584] [WorldCat] [DOI] - ↑
Kurmangaliyev, Y.Z., Yoo, J., Valdes-Aleman, J., Sanfilippo, P., & Zipursky, S.L. (2020).
Transcriptional Programs of Circuit Assembly in the Drosophila Visual System. Neuron. [PubMed:33125872] [WorldCat] [DOI] - ↑
Horie, R., Hazbun, A., Chen, K., Cao, C., Levine, M., & Horie, T. (2018).
Shared evolutionary origin of vertebrate neural crest and cranial placodes. Nature, 560(7717), 228-232. [PubMed:30069052] [PMC] [WorldCat] [DOI] - ↑
Horie, T., Horie, R., Chen, K., Cao, C., Nakagawa, M., Kusakabe, T.G., ..., & Levine, M. (2018).
Regulatory cocktail for dopaminergic neurons in a protovertebrate identified by whole-embryo single-cell transcriptomics. Genes & development, 32(19-20), 1297-1302. [PubMed:30228204] [PMC] [WorldCat] [DOI] - ↑
Cosacak, M.I., Bhattarai, P., Reinhardt, S., Petzold, A., Dahl, A., Zhang, Y., & Kizil, C. (2019).
Single-Cell Transcriptomics Analyses of Neural Stem Cell Heterogeneity and Contextual Plasticity in a Zebrafish Brain Model of Amyloid Toxicity. Cell reports, 27(4), 1307-1318.e3. [PubMed:31018142] [WorldCat] [DOI] - ↑
Thyme, S.B., Pieper, L.M., Li, E.H., Pandey, S., Wang, Y., Morris, N.S., ..., & Schier, A.F. (2019).
Phenotypic Landscape of Schizophrenia-Associated Genes Defines Candidates and Their Shared Functions. Cell, 177(2), 478-491.e20. [PubMed:30929901] [PMC] [WorldCat] [DOI] - ↑
Tosches, M.A., Yamawaki, T.M., Naumann, R.K., Jacobi, A.A., Tushev, G., & Laurent, G. (2018).
Evolution of pallium, hippocampus, and cortical cell types revealed by single-cell transcriptomics in reptiles. Science (New York, N.Y.), 360(6391), 881-888. [PubMed:29724907] [WorldCat] [DOI] - ↑
Marchetto, M.C., Hrvoj-Mihic, B., Kerman, B.E., Yu, D.X., Vadodaria, K.C., Linker, S.B., ..., & Gage, F.H. (2019).
Species-specific maturation profiles of human, chimpanzee and bonobo neural cells. eLife, 8. [PubMed:30730291] [PMC] [WorldCat] [DOI] - ↑
Kanton, S., Boyle, M.J., He, Z., Santel, M., Weigert, A., Sanchís-Calleja, F., ..., & Camp, J.G. (2019).
Organoid single-cell genomic atlas uncovers human-specific features of brain development. Nature, 574(7778), 418-422. [PubMed:31619793] [WorldCat] [DOI] - ↑
Shafer, M.E.R. (2019).
Cross-Species Analysis of Single-Cell Transcriptomic Data. Frontiers in cell and developmental biology, 7, 175. [PubMed:31552245] [PMC] [WorldCat] [DOI] - ↑
Keil, J.M., Qalieh, A., & Kwan, K.Y. (2018).
Brain Transcriptome Databases: A User's Guide. The Journal of neuroscience : the official journal of the Society for Neuroscience, 38(10), 2399-2412. [PubMed:29437890] [PMC] [WorldCat] [DOI] - ↑
Ecker, J.R., Geschwind, D.H., Kriegstein, A.R., Ngai, J., Osten, P., Polioudakis, D., ..., & Zeng, H. (2017).
The BRAIN Initiative Cell Census Consortium: Lessons Learned toward Generating a Comprehensive Brain Cell Atlas. Neuron, 96(3), 542-557. [PubMed:29096072] [PMC] [WorldCat] [DOI] - ↑
Tabula Muris Consortium, Overall coordination, Logistical coordination, Organ collection and processing, Library preparation and sequencing, Computational data analysis, ..., & Principal investigators (2018).
Single-cell transcriptomics of 20 mouse organs creates a Tabula Muris. Nature, 562(7727), 367-372. [PubMed:30283141] [PMC] [WorldCat] [DOI] - ↑
Welch, J.D., Kozareva, V., Ferreira, A., Vanderburg, C., Martin, C., & Macosko, E.Z. (2019).
Single-Cell Multi-omic Integration Compares and Contrasts Features of Brain Cell Identity. Cell, 177(7), 1873-1887.e17. [PubMed:31178122] [PMC] [WorldCat] [DOI] - ↑
Korsunsky, I., Millard, N., Fan, J., Slowikowski, K., Zhang, F., Wei, K., ..., & Raychaudhuri, S. (2019).
Fast, sensitive and accurate integration of single-cell data with Harmony. Nature methods, 16(12), 1289-1296. [PubMed:31740819] [PMC] [WorldCat] [DOI] - ↑
Crow, M., Paul, A., Ballouz, S., Huang, Z.J., & Gillis, J. (2018).
Characterizing the replicability of cell types defined by single cell RNA-sequencing data using MetaNeighbor. Nature communications, 9(1), 884. [PubMed:29491377] [PMC] [WorldCat] [DOI] - ↑
Barkas, N., Petukhov, V., Nikolaeva, D., Lozinsky, Y., Demharter, S., Khodosevich, K., & Kharchenko, P.V. (2019).
Joint analysis of heterogeneous single-cell RNA-seq dataset collections. Nature methods, 16(8), 695-698. [PubMed:31308548] [PMC] [WorldCat] [DOI] - ↑
Kiselev, V.Y., Yiu, A., & Hemberg, M. (2018).
scmap: projection of single-cell RNA-seq data across data sets. Nature methods, 15(5), 359-362. [PubMed:29608555] [WorldCat] [DOI] - ↑
Sato, K., Tsuyuzaki, K., Shimizu, K., & Nikaido, I. (2019).
CellFishing.jl: an ultrafast and scalable cell search method for single-cell RNA sequencing. Genome biology, 20(1), 31. [PubMed:30744683] [PMC] [WorldCat] [DOI] - ↑
Rodriques, S.G., Stickels, R.R., Goeva, A., Martin, C.A., Murray, E., Vanderburg, C.R., ..., & Macosko, E.Z. (2019).
Slide-seq: A scalable technology for measuring genome-wide expression at high spatial resolution. Science (New York, N.Y.), 363(6434), 1463-1467. [PubMed:30923225] [PMC] [WorldCat] [DOI] - ↑
Stickels, R.R., Murray, E., Kumar, P., Li, J., Marshall, J.L., Di Bella, D.J., ..., & Chen, F. (2020).
Highly sensitive spatial transcriptomics at near-cellular resolution with Slide-seqV2. Nature biotechnology. [PubMed:33288904] [WorldCat] [DOI] - ↑
Vickovic, S., Eraslan, G., Salmén, F., Klughammer, J., Stenbeck, L., Schapiro, D., ..., & Ståhl, P.L. (2019).
High-definition spatial transcriptomics for in situ tissue profiling. Nature methods, 16(10), 987-990. [PubMed:31501547] [PMC] [WorldCat] [DOI] - ↑
Ståhl, P.L., Salmén, F., Vickovic, S., Lundmark, A., Navarro, J.F., Magnusson, J., ..., & Frisén, J. (2016).
Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science (New York, N.Y.), 353(6294), 78-82. [PubMed:27365449] [WorldCat] [DOI] - ↑
Moncada, R., Barkley, D., Wagner, F., Chiodin, M., Devlin, J.C., Baron, M., ..., & Yanai, I. (2020).
Integrating microarray-based spatial transcriptomics and single-cell RNA-seq reveals tissue architecture in pancreatic ductal adenocarcinomas. Nature biotechnology, 38(3), 333-342. [PubMed:31932730] [WorldCat] [DOI] - ↑
Edsgärd, D., Johnsson, P., & Sandberg, R. (2018).
Identification of spatial expression trends in single-cell gene expression data. Nature methods, 15(5), 339-342. [PubMed:29553578] [PMC] [WorldCat] [DOI] - ↑
Svensson, V., Teichmann, S.A., & Stegle, O. (2018).
SpatialDE: identification of spatially variable genes. Nature methods, 15(5), 343-346. [PubMed:29553579] [PMC] [WorldCat] [DOI] - ↑
Cang, Z., & Nie, Q. (2020).
Inferring spatial and signaling relationships between cells from single cell transcriptomic data. Nature communications, 11(1), 2084. [PubMed:32350282] [PMC] [WorldCat] [DOI] - ↑
Chen, K.H., Boettiger, A.N., Moffitt, J.R., Wang, S., & Zhuang, X. (2015).
RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science (New York, N.Y.), 348(6233), aaa6090. [PubMed:25858977] [PMC] [WorldCat] [DOI] - ↑
Coskun, A.F., & Cai, L. (2016).
Dense transcript profiling in single cells by image correlation decoding. Nature methods, 13(8), 657-60. [PubMed:27271198] [PMC] [WorldCat] [DOI] - ↑
Buxbaum, A.R., Haimovich, G., & Singer, R.H. (2015).
In the right place at the right time: visualizing and understanding mRNA localization. Nature reviews. Molecular cell biology, 16(2), 95-109. [PubMed:25549890] [PMC] [WorldCat] [DOI] - ↑
Codeluppi, S., Borm, L.E., Zeisel, A., La Manno, G., van Lunteren, J.A., Svensson, C.I., & Linnarsson, S. (2018).
Spatial organization of the somatosensory cortex revealed by osmFISH. Nature methods, 15(11), 932-935. [PubMed:30377364] [WorldCat] [DOI] - ↑
Wang, X., Allen, W.E., Wright, M.A., Sylwestrak, E.L., Samusik, N., Vesuna, S., ..., & Deisseroth, K. (2018).
Three-dimensional intact-tissue sequencing of single-cell transcriptional states. Science (New York, N.Y.), 361(6400). [PubMed:29930089] [PMC] [WorldCat] [DOI] - ↑
Shah, S., Lubeck, E., Zhou, W., & Cai, L. (2016).
In Situ Transcription Profiling of Single Cells Reveals Spatial Organization of Cells in the Mouse Hippocampus. Neuron, 92(2), 342-357. [PubMed:27764670] [PMC] [WorldCat] [DOI] - ↑
Merritt, C.R., Ong, G.T., Church, S.E., Barker, K., Danaher, P., Geiss, G., ..., & Beechem, J.M. (2020).
Multiplex digital spatial profiling of proteins and RNA in fixed tissue. Nature biotechnology, 38(5), 586-599. [PubMed:32393914] [WorldCat] [DOI] - ↑
Chen, W.T., Lu, A., Craessaerts, K., Pavie, B., Sala Frigerio, C., Corthout, N., ..., & De Strooper, B. (2020).
Spatial Transcriptomics and In Situ Sequencing to Study Alzheimer's Disease. Cell, 182(4), 976-991.e19. [PubMed:32702314] [WorldCat] [DOI] - ↑
Zhu, C., Preissl, S., & Ren, B. (2020).
Single-cell multimodal omics: the power of many. Nature methods, 17(1), 11-14. [PubMed:31907462] [WorldCat] [DOI] - ↑
Stuart, T., & Satija, R. (2019).
Integrative single-cell analysis. Nature reviews. Genetics, 20(5), 257-272. [PubMed:30696980] [WorldCat] [DOI] - ↑
Stoeckius, M., Hafemeister, C., Stephenson, W., Houck-Loomis, B., Chattopadhyay, P.K., Swerdlow, H., ..., & Smibert, P. (2017).
Simultaneous epitope and transcriptome measurement in single cells. Nature methods, 14(9), 865-868. [PubMed:28759029] [PMC] [WorldCat] [DOI] - ↑
Peterson, V.M., Zhang, K.X., Kumar, N., Wong, J., Li, L., Wilson, D.C., ..., & Klappenbach, J.A. (2017).
Multiplexed quantification of proteins and transcripts in single cells. Nature biotechnology, 35(10), 936-939. [PubMed:28854175] [WorldCat] [DOI] - ↑
Fuzik, J., Zeisel, A., Máté, Z., Calvigioni, D., Yanagawa, Y., Szabó, G., ..., & Harkany, T. (2016).
Integration of electrophysiological recordings with single-cell RNA-seq data identifies neuronal subtypes. Nature biotechnology, 34(2), 175-183. [PubMed:26689544] [PMC] [WorldCat] [DOI] - ↑
Cadwell, C.R., Palasantza, A., Jiang, X., Berens, P., Deng, Q., Yilmaz, M., ..., & Tolias, A.S. (2016).
Electrophysiological, transcriptomic and morphologic profiling of single neurons using Patch-seq. Nature biotechnology, 34(2), 199-203. [PubMed:26689543] [PMC] [WorldCat] [DOI] - ↑
Huang, A.Y., Li, P., Rodin, R.E., Kim, S.N., Dou, Y., Kenny, C.J., ..., & Walsh, C.A. (2020).
Parallel RNA and DNA analysis after deep sequencing (PRDD-seq) reveals cell type-specific lineage patterns in human brain. Proceedings of the National Academy of Sciences of the United States of America, 117(25), 13886-13895. [PubMed:32522880] [PMC] [WorldCat] [DOI] - ↑
Chen, X., Sun, Y.C., Zhan, H., Kebschull, J.M., Fischer, S., Matho, K., ..., & Zador, A.M. (2019).
High-Throughput Mapping of Long-Range Neuronal Projection Using In Situ Sequencing. Cell, 179(3), 772-786.e19. [PubMed:31626774] [WorldCat] [DOI] - ↑
Amberg, N., Laukoter, S., & Hippenmeyer, S. (2019).
Epigenetic cues modulating the generation of cell-type diversity in the cerebral cortex. Journal of neurochemistry, 149(1), 12-26. [PubMed:30276807] [PMC] [WorldCat] [DOI]